È necessario avere un abbonamento a JoVE per visualizzare questo. Accedi o inizia la tua prova gratuita.
Method Article
Preparazione di matrici di reclamo per Quantificare Contrazione Cellular
In questo articolo
Riepilogo
In questo video, dimostriamo le tecniche sperimentali utilizzate per fabbricare compatibile, della matrice extracellulare (ECM) substrati rivestiti adatto per colture cellulari e che sono suscettibili di microscopia a forza di trazione e di osservare gli effetti di rigidità ECM sul comportamento delle cellule.
Abstract
Il regolamento di adesione cellulare alla matrice extracellulare (ECM) è essenziale per la migrazione cellulare e rimodellamento ECM. Adesioni focali sono le assemblee macromolecolari che la coppia di F-contrattili actina del citoscheletro per l'ECM. Questo collegamento consente la trasmissione delle forze meccaniche intracellulare attraverso la membrana cellulare al supporto sottostante. Studi recenti hanno dimostrato le proprietà meccaniche della ECM regolare adesione focale e F-actina morfologia oltre a numerosi processi fisiologici, quali la differenziazione cellulare, la divisione, la proliferazione e la migrazione. Pertanto, l'uso di substrati di coltura cellulare è diventato un metodo sempre più diffuso per controllare con precisione e modulare le proprietà meccaniche ECM.
Per quantificare le forze di trazione in adesioni focali in una cella aderente, supporti compatibili sono utilizzati in combinazione con imaging ad alta risoluzione e tecniche computazionali in un microscopio chiamato metodo della forza di trazione (TFM). Questa tecnica si basa sulla misurazione della magnitudo locale e la direzione del substrato deformazioni indotte dalla contrazione cellulare. In combinazione con alta risoluzione microscopia a fluorescenza di proteine fluorescenti tag, è possibile correlare l'organizzazione del citoscheletro e rimodellamento con le forze di trazione.
Qui vi presentiamo un piano sperimentale dettagliato per la preparazione di due dimensioni, matrici compatibili al fine di creare un substrato di coltura cellulare con un ben caratterizzato, sintonizzabile rigidità meccanica, che è adatto per misurare la contrazione cellulare. Questi protocolli includono la realizzazione di idrogel di poliacrilamide, rivestimento di proteine ECM su gel di tali cellule placcatura in gel, e ad alta risoluzione, microscopia confocale utilizzando una camera di perfusione. Inoltre, mettiamo a disposizione un campione rappresentativo di dati che dimostrino posizione e la grandezza delle forze cellulare utilizzando protocolli citato TFM.
Protocollo
1. Attivazione della superficie coprioggetto
- Coprioggetti (# 1.5, 22x40 mm) vengono puliti con una serie di lavaggi sapone ed etanolo in un protocollo precedentemente descritto (Waterman-Storer, 1998) per pulire e rimuovere la polvere.
- Coprioggetto posto in un rack supporto in acciaio inox, in modo tale che coprioggetto sono distanziate tra loro e non toccare.
- In cappa (guanti di nitrile e occhiali consigliato), diluire piena forza 3-aminopropyltrimethoxysilane in isopropanolo per una concentrazione finale del 2% (2 ml silano / 100 ml di isopropanolo) per riempire un piatto quadrato di vetro (~ 350 ml di volume). A causa di reattività con la plastica, usare un bicchiere pipetta Pasteur di applicare 3-aminopropyltrimethoxysilane al isopropanolo.
- Coprioggetto immergere completamente da 1,2 in questa soluzione per 10 minuti agitando moderatamente su un piatto mescolare in cappa.
- Lavare coprioggetto immergendolo in DDH 2 O (4 scambi di acqua). Lasciare 10 min ammollo tempo per lo scambio finale, con agitazione. Soluzioni contenenti ammino-silano devono essere smaltiti come rifiuti pericolosi.
- Coprioggetto secco in stufa a temperatura calda (~ 37 ° C) per 10 minuti in un ambiente privo di polvere.
- Raffreddare a temperatura ambiente.
- Nella cappa, coprioggetto immergere in soluzione di glutaraldeide all'1% in DDH 2 O in un piatto di vetro quadrato sul piatto mescolare per 30 minuti.
- Lavare con 3 scambi di DDH 2 O per 10 minuti per scambio, mescolando. Smaltire glutaraldeide come rifiuti pericolosi.
- Asciutto a temperatura ambiente, coprendo con un foglio di alluminio per evitare la polvere di attaccarsi alla coprioggetto.
- Conservare in luogo asciutto, lontano dalla polvere, per un massimo di 2 mesi.
2. Preparazione di poliacrilamide (PAA) gel
- Preparare soluzioni di acrilamide / bis-acrilamide miscela dal 40% di acrilamide e il 2% bis-acrilamide, alla tabella 1. Manteniamo soluzioni madri diverse che sono ottimizzati per i gel di PAA rigidità diversa; esempi sono elencati nella tabella 1 e 2. Soluzioni madri possono essere conservati per diversi anni, finché il loro mantenimento in una bottiglia oscurata a 4 ° C.
- Soluzioni di lavoro che contiene le concentrazioni finali desiderato di acrilamide / bis-acrilamide sono ottenuti da soluzioni di riserva. Per esempio, preparare una soluzione di lavoro del 7,5% acrylamide/0.10% bis-acrilammide negli DDH 2 O per fare 2.8kPa gel PAA.
- Degas soluzione di acrilamide in una camera a vuoto per 20 minuti, per ridurre l'ossigeno all'interno della soluzione che impedisce la polimerizzazione PAA.
- Preparare il 10% di ammonio persolfato (APS) soluzione (0.5g/5mL). Usa fresca magazzino di lavoro entro 3 giorni. In alternativa, le scorte possono essere congelati per essere utilizzati in epoche successive.
- Mentre acrilamide è degasaggio, cancellare un 1x3 "vetrino da microscopio con Rain-X salviette vigorosamente per rendere scivolare vetro idrofobo superficie. Per rimuovere l'eccesso di pioggia-X, pulire vetrino con Kimwipe umido. Vetrini Mettere da parte, coperta.
- Rimuovere la soluzione di acrilammide da camera a vuoto e aggiungere perline fluorescenti (1% in volume, 5 microlitri per la soluzione di lavoro elencati nella Tabella 1). Aggiungi 0.75μl TEMED e 2,5 microlitri APS del 10%, che avvierà la polimerizzazione del gel. Mescolare bene pipettando per ~ 5 sec, per ridurre al minimo l'introduzione di bolle,
- Applicare 10-12 ml di soluzione di acrilamide per vetrino da microscopio idrofobiche (preparata al punto 2.4) e il luogo attivato coprioggetto 22x40mm sulla parte superiore della goccia. Soluzione gel dovrebbe coprioggetto cappotto intero. Appianare eventuali bolle che possono apparire all'interno della soluzione. Lasciare che la soluzione gel di polimerizzare a temperatura ambiente per circa 10 min.
- Il completamento della polimerizzazione può essere valutata invertendo soluzione rimanente lavora in provetta. Inoltre, il gel polimerizzato può tirare fuori dai bordi coprioggetto. Immediatamente dopo la polimerizzazione macroscopico si osserva, separare il coprioggetti dal vetrino. Utilizzando la punta fine di un paio di pinzette o una lama di rasoio, rimuovere con attenzione coprioggetto, con gel attaccato, dalla superficie del vetrino da microscopio e gel di immergersi in DDH 2 O, per mantenere l'idratazione.
3. Accoppiamento matrice extracellulare (ECM) proteine al gel PAA
Tre distinti metodi possono essere usati per collegare le proteine ECM sia per la superficie superiore del gel di PAA (3.1 e 3.2) o l'incorporazione di proteine ECM all'interno del volume di gel (3,3). Qui, si discute l'accoppiamento di fibronectina di gel PAA tradursi in una densità ligando superficie che è equivalente alla quantità adsorbita su vetro, dopo incubazione con 10 mcg / mL di soluzione di fibronectina per 1 ora. Considerazioni per la scelta di un metodo sono riportati nella discussione.
- Cross-linking proteina ECM di gel superficie PAA da Sulfo-SANPAH ammine reattiva sulle proteine covalentemente sono attaccati alla superficie del gel PAA dal heterobifunctional cross-linker Sulfo-SANPAH
- Preparare 40 aliquote microlitri di lavoro di Sulfo-SANPAH sciogliendo S ulfo-SANPAH polvere in anidro dimetilsolfossido (DMSO) (20 microlitri per mg di Sulfo-SANPAH). Scorte di congelare Flash in azoto liquido e conservare a -80 ° C per un uso successivo.
- Rimuovere DDH 2 O dalla superficie del gel mediante un filatore coprioggetto (<2 sec). Evitare l'essiccamento del gel.
- Diluire Sulfo-SANPAH-DMSO aliquote in DDH 2 O (2mg/ml, pH 7) immediatamente prima dell'uso e gel coat superficie (~ 200 l). Si noti che la reattività di dimezzamento di Sulfo-SANPAH è breve (~ 5 minuti) a temperatura ambiente in acqua, quindi, questa procedura dovrebbe essere fatto ad un ritmo rapido.
- Esporre superficie del gel ai raggi UV in un UV cross-linker forno (8W, 254 nm di lunghezza d'onda ad una distanza di 2-3 centimetri per 1,5 min). Solfo-SANPAH cambia di colore da arancione a marrone.
- Dip UV trattati con coprioggetto in un bicchiere con fresco DDH 2 O e rimuovere l'acqua in eccesso dalla superficie del gel mediante un filatore coprioggetto (<2 sec).
- Pipettare ~ 50 ml di freddo 1mg/ml fibronectina (FN) (in PBS, pH 7,4) su Parafilm in un contenitore piastra di Petri. Inverti coprioggetto sopra goccia FN, gel lato esposto a FN.
- Reagiscono a temperatura ambiente per 1-2 ore o a 4 ° C durante la notte.
- Coprioggetto posto in 6 piatti tessuto centimetri di coltura contenente PBS (pH 7,4), sufficiente a coprire coprioggetti, in condizioni di sterilità nella cappa del tessuto culturale.
- Lavare a lungo con numerosi lavaggi (3-5) di PBS (pH 7,4), in condizioni sterili.
- Sterilizzare i coprioggetti con l'uso di lampade germicide a cappuccio di coltura per 30 min.
- Incubare coprioggetto nei media delle cellule per 30-45 minuti prima di cellule placcatura.
- Cross-linking ECM alla superficie del gel PAA da gruppi idrato di idrazina carboidrati sulle proteine sono ossidate e abbinato al gel con idrato di idrazina.
- Preparare gel di poliacrilammide, come descritto nella sezione 2.
- Luogo PAA coprioggetto gel in plastica piastra di Petri in una cappa aspirante e guanti con pipetta circa 1 ml di idrato di idrazina non diluito sulla superficie di ogni gel PAA e incubare per almeno due ore, ma non più di 24 ore
- Aggiungi DDH 2 O alla piastra Petri; soluzione di idrato di idrazina rimuovere e smaltire come rifiuti pericolosi.
- Aggiungere 5% di acido acetico al Petri di immergersi coprioggetto. Coperchio e incubare per un'ora.
- Rimuovere l'acido acetico e lavare con DDH 2 O. Incubare in DDH 2 O per un'ora. I coprioggetti sono ora attivi e pronti a cross-link ossidato fibronectina (FN).
- Diluire 10 ml di 1 mg / ml soluzione FN in 940 microlitri di 50 mM tampone acetato di sodio (pH 4,5) in un tubo scuro centrifuga micro, facendo una concentrazione finale di 10 mg / ml.
- Fai la scorta di 20X sodio meta-periodato con l'aggiunta di 80 mg di sodio meta-periodato a 1 ml di 50 mM tampone acetato di sodio (pH 4,5).
- Aggiungere 50 ml di 20X sodio meta-periodato magazzino alla soluzione preparata in FN 3.2.6, in modo che la concentrazione di lavoro finale sono 10 mg / ml di FN e 4 mg / ml di sodio meta-periodato. Incubare nel tubo al buio a temperatura ambiente per 30 minuti.
- Rimuovere l'eccesso di DDH 2 O dalla superficie del gel attivato preparato in 3.2.5 utilizzando uno spinner coprioggetto (<2 sec). Evitare l'essiccamento del gel.
- Pipettare ~ 500 ml di soluzione di FN sulla superficie del gel attivato e incubare per 1 ora a temperatura ambiente.
- Coprioggetto posto in piatti contenenti PBS (pH 7,4), sufficiente a coprire coprioggetto.
- Lavare a lungo con numerosi lavaggi (3-5) di PBS (pH 7,4).
- Sterilizzare i coprioggetti con l'uso di lampade germicide a cappuccio di coltura per 30 min.
- Incubare coprioggetto nei media delle cellule per 30-45 minuti prima di cellule placcatura.
- Coniugazione maggior parte delle proteine ECM in PAA gel Acryloyl-X, estere succinimidile Questo protocollo è fatto prima a 2,5. Ammine reattiva sulle proteine sono accoppiate ad un monomero di acrilammide con NHS estere chimica e poi co-polimerizzato in la maggior parte dei gel di PAA.
- Coniugato proteine ECM di scelta per Acryloyl-X secondo le istruzioni del produttore. Soluzioni stock di proteina coniugata deve essere conservato a 4 ° C.
- Calcolare il volume di soluzione PAA di lavoro richiesto per gel di fabbricazione (ad esempio 10 uL per vetrino).
- Sottrarre 50 microlitri (ad esempio volume 10%) di acqua dalla quantità indicate nella ricetta soluzione di lavoro nella tabella 1 e avviare la polimerizzazione.
- Rimuovere volume calcolato al punto 3.3.2 e aggiungete ECM / Acryloyl-X soluzione al 10% del volume. (Es. 1 ml di ECM / Acryloyl-X a 9 ml di soluzione PAA). Questa operazione deve essere eseguita rapidamente, come gel è polimerizzazione.
- Completare i punti 2.6 e 2.7 come descritto in precedenza.
- Lavare a lungo con numerosi lavaggi (3-5) di PBS (pH 7,4), in condizioni sterili.
- Sterilizzare i coprioggetti con l'uso di lampade germicide a cappuccio di coltura per 30 min.
- Incubare coprioggetto nei media delle cellule per 30-45 minuti prima di cellule placcatura.
Questi passaggi sono condotti dopo le cellule sono stati autorizzati a diffondere su ECM rivestite substrato gel (~ ore 6-12). Per assemblare la camera di imaging confocale (RC-30WA), è utile consultare il sito Warner Strumenti per l'orientamento.
- Mezzi di comunicazione cellulare caldo e lo 0,5% o 0,25% tripsina e caricare in una siringa da 5 ml o 10 ml.
- Carica un coprioggetto 22x30mm sul supporto Coverslip dall'alto di un Instruments Warner confocale immagini da camera (RC-30WA) con grasso vuoto per mantenere il coprioggetto in posizione.
- Inserire una camera di formazione guarnizione in gomma sulla parte superiore del vetrino, consentendo l'accesso ad entrambi di ingresso e uscita tubazioni in polietilene. Questo permetterà una distanza di 150-1000 micron tra il vetrino superiore e il gel rivestita coprioggetti 22x40mm, a seconda delle dimensioni della guarnizione usata.
- Siringhe caricare sul tubo di ingresso, tramite kit di connettori e controllare che i media scorre attraverso i tubi e su Coverslip Top e bolle sono evidenti nelle linee di flusso.
- Ingrassare vuoto sulla base della camera e del carico gel rivestita coprioggetti 22x40mm, cellulare rivolta verso l'alto. Applicare i media caldi alle cellule.
- Luogo titolare Coverslip Top sulla base della camera, con la Camera guarnizione che separa dal gel rivestita coprioggetto. Assicurarsi che i perni di posizionamento all'interno della base della camera siedono all'interno del Fori Individuazione del Coverslip Top.
- Applicare la piastra di pressione alla base della camera e utilizzare la chiave piastra di pressione per avvitare e fissare la piastra di pressione.
- Controllare il flusso dei mezzi di comunicazione attraverso il tubo e la camera di monitorare eventuali perdite potenziali all'interno della camera e per eliminare qualsiasi supporto zone libere sulla superficie cellulare. Nota: utilizzando un ago di piccolo calibro per disegnare un leggero vuoto mentre infondendo con i media possono contribuire ad eliminare i media zone libere.
- Applicare la camera confocale di imaging per l'adattatore fase situata all'interno di un titolare microscopio per l'imaging.
- Immagine fluorescenza marcata con proteine e perline fluorescenti inserita all'interno del substrato di gel su un microscopio confocale a fluorescenza.
- Per ottenere un'immagine di posizioni senza forzature tallone all'interno del gel, profumato tripsina per staccare le aderenze cellulare dal gel, e scattare una foto delle perline fluorescenti nel campo dell'imaging stesso in cui la cellula aderito. Confronto di posizioni cordone teso e senza forzature permette per la quantificazione dello spostamento gel substrato in contrazione.
Rappresentante dei risultati:
Il protocollo sopra descrive la procedura sperimentale per la preparazione di gel compatibili PAA per studiare la contrattilità delle cellule ed è illustrato nella figura 1. La superficie del gel ottenuto con questo protocollo è relativamente piatta e liscia, con perline fluorescenti incorporate uniformemente (Figura 2A).
Se la misurazione contrazione gel nella posizione di adesioni focali, l'imaging della cella (Figura 3A) e superficie del gel (Figura 3B) deve essere fatto sul piano ottico confocale di adesioni focali. La contrazione di un gel possono essere visualizzati mediante lo spostamento di perline fluorescenti incorporati (Figura 3B) alla superficie del gel quando le cellule sono aderenti (tesi) rispetto distaccata (senza forzature). L'uso di algoritmi di calcolo può produrre trazione stress associato con lo spostamento di perline e corrispondente modulo elastico del gel (Figura 3C e 3D) (Sabass et al., 2008). Se l'imaging avviene più all'interno del gel, poi spostamenti tallone sarà più piccola e non rappresentativa delle forze di trazione esercitate adesioni focali.
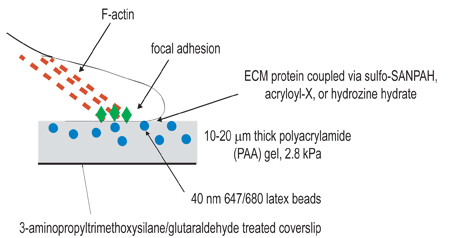
Figura 1. Illustrazione schematica del setup sperimentale. L'obiettivo generale di questa procedura è quello di creare matrici compatibili al fine di studiare la contrazione cellulare. Il primo passo della procedura sperimentale è quello di attivare vetrini dal trattamento amino-silane/glutaraldehyde allo scopo di ancoraggio gel polimerizzato. Il secondo passo è quello di polimerizzare un gel di poliacrilamide, contenente microsfere fluorescenti, sul coprioggetti attivato. La terza fase prevede la chimica cross-linking del ligando extracellulare sulla superficie del gel di poliacrilamide, utilizzando una delle tre tecniche di accoppiamento elencati al punto 3. Le cellule sono poi placcato sul gel e ha permesso di aderire e diffondere. Sotto attivo contrazione cellulare, perline incorporato nel gel spostano.

Figura 2. Ottica confocale fetta di superficie superiore del gel di PAA, come visualizzati con (A.) perline fluorescenti 40nm incorporati all'interno e gel (B.) immunofluorescenza fibronectina.

Figura 3. Risultato Rappresentante per un esperimento forza di trazione. (A.), aderenze focale in una cellula umana U2OS osteosarcoma sono marked da GFP-paxillina e (B) le posizioni di perline fluorescenti incorporati nel gel di PAA sottostante adesioni focali della tesi (verde) e senza forzature (rosso) stati. Le frecce indicano esempi di spostamento tallone. (C.) vettori di trazione e di stress (D.) corrispondente al calore scala della mappa delle tensioni di trazione derivato dalla contrazione del gel, utilizzando algoritmi di calcolo (Sabass et al., 2008). Barra di scala = 5 micron.
Tabella 1:
Archivi di esempio e di lavoro Solutions PAA (Dati in tabella 1 è stato ottenuto da Yeung et. Al. Ed indipendentemente confermato nel nostro laboratorio.)
| Magazzino PAA Soluzione | ||||
| Modulo di Taglio di PAA Gel (Pa) | 230 | 2833 | 8640 | 16344 |
| 40% di acrilammide (ml) | 1,25 | 3,12 | 2,34 | 2,50 |
| 2% Bis-acrilamide (ml) | 0,50 | 0,83 | 1,88 | 0,60 |
| Acqua (ml) | 3,25 | 1,04 | 0,78 | 1. 90 |
| Volume totale (ml): | 5 | 5 | 5 | 5 |
| Soluzione di lavoro PAA | ||||
| Soluzione madre Usato (Pa) | 230 | 2833 | 8640 | 16344 |
| Magazzino Volume Soluzione (mL) | 150 | 150 | 200 | 300 |
| Acqua (mL) | 341,75 | 341,75 | 291,75 | 191,75 |
| Perline (mL) | 5 | 5 | 5 | 5 |
| TEMED (mL) | 0,75 | 0,75 | 0,75 | 0,75 |
| 10% di APS (mL) | 2,5 | 2,5 | 2,5 | 2,5 |
| Volume totale (mL): | 500 | 500 | 500 | 500 |
| Acrilamide% finale | 3 | 7,5 | 7,5 | 12 |
| Finale Bis-acrilamide% | 0,06 | 0,1 | 0,3 | 0,15 |
Tabella 2:
Modulo di Taglio di PAA substrati di vari acrilamide finale e bis-acrilamide percentuali
| 12% di acrilamide | 7,5% di acrilammide | |||
| % Bis-acrilamide | Modulo di Taglio (Pa) | % Bis-acrilamide | Modulo di Taglio (Pa) | |
| 0,145 | 16344 | 0,01 | 689 | |
| 0,28 | 30067 | 0,03 | 1535 | |
| 0,45 | 34263 | 0,05 | 2286 | |
| 0,55 | 42375 | 0,075 | 2833 | |
| 0,575 | 50873 | 0,1 | 4069 | |
| 0,6 | 55293 | 0,2 | 5356 | |
| 0,3 | 8640 | |||
| 5% di acrilamide | 3% di acrilamide | |||
| % Bis-acrilamide | Modulo di Taglio (Pa) | % Bis-acrilamide | Modulo di Taglio (Pa) | |
| 0,05 | 430 | 0,02 | 1,3 | |
| 0,075 | 600 | 0,04 | 54 | |
| 0,1 | 1431 | |||
Discussione
La procedura descritta qui per la configurazione di un microscopio a forza di trazione (TFM) sperimentare, insieme con l'implementazione delle routine di tracciamento computazionale (Sabass et al., 2008), consente per la quantificazione delle forze cellulare con micron scala risoluzione spaziale. Per ottimizzare il protocollo sperimentale, è fondamentale per formare un substrato di gel puro ed uniforme con rivestimento uniforme di legante ECM. Discutiamo potenziali insidie di seguito:
Divulgazioni
Riconoscimenti
Ringraziamo il laboratorio di Ulrich Schwarz per il software di monitoraggio di calcolo utilizzati nella quantificazione delle forze di trazione cellulare (Sabass et al., 2008). Questo lavoro è stato sostenuto da un premio di carriera Burroughs Wellcome e Pioneer Award NIH direttore (DP10D00354) per ML Gardel e Medical Scientist Premio Nazionale delle Ricerche Servizio (5 T32 GM07281) a SP Winter.
Materiali
| Name | Company | Catalog Number | Comments |
| 3-aminopropyltrimethyoxysilane | Aldrich | 28, 177-8 | |
| 40% Acrylamide | Bio-Rad | 161-0140 | |
| 2% Bis-acrylamide | Fisher Scientific | BP1404 | |
| TEMED | Fisher Scientific | BP 150-20 | |
| Ammonium persulfate | Fisher Scientific | BP179 | |
| 40nm fluorescent micro-spheres | Invitrogen | F8789 | |
| Sulfo-SANPAH | Pierce, Thermo Scientific | 22589 | |
| Confocal imaging chamber (RC-30) | Warner Instruments | 64-0320 | |
| Coverslip spinner | Home made | NA | |
| Ultraviolet lamp CL1000 | UVP Inc. | 95-0228-01 | |
| Stainless steel rack | Electron Microscopy Sciences | 72239-04 | |
| acryloyl-X, SE (6-((acryloyl)amino)hexanoic acid) | Invitrogen | A-20770 | |
| Hydrazine hydrate | Sigma-Aldrich | 225819 | |
| Sodium meta-periodate | Thermo Fisher Scientific, Inc. | 20504 | |
| Isopropanol | Fisher Scientific | A416-4 | |
| Fibronectin | Sigma-Aldrich | F2006 | |
| Collagen | BD Biosciences | 354236 | |
| Coverslips (#1.5) | Corning | 2940‐224 | |
| Glutaraldehyde | Electron Microscopy Sciences | 16120 | |
| Rain-X | SOPUS Products | www.rainx.com | |
| Acetic Acid | Acros Organics | 64-19-7 |
Riferimenti
- Damljanovic, V., Lajerholm, B. C., Jacobson, K. Bulk and micropatterned conjugation of extracellular matrix proteins to characterized polyacrylamid substrates for cell mechanotransduction assays. Biotechniques. 39 (6), 847-851 (2005).
- Engler, A., Bacakova, L. N. e. w. m. a. n., Hategan, C., Griffin, A., M, D. D. i. s. c. h. e. r. Substrate compliance versus ligand in cell on gel responses. Biophys J. 86 ((1 Pt 1)), 617-628 (2004).
- Gardel, M. L., Sabass, B., Ji, L., Danuser, G., Schwarz, U. S., Waterman, C. M. Traction stress in focal adhesions correlates biphasically with actin retrograde flow speed. J Cell Biol. 183, 999-1005 (2008).
- Rajagopalan, P., Marganski, W. A., Brown, X. Q., Wong, J. Y. Direct comparison of the spread area, contractility, and migration of balb/c 3T3 fibroblasts adhered to fibronectin- and RGD-modified substrata. Biophys J. 87 (4), 2818-2827 (2004).
- Reinhart-King, C. A., Dembo, M., Hammer, D. A. The dynamics and mechanics of endothelial cell spreading. Biophys J. 89, 676-689 (2005).
- Stricker, J., Sabass, B., Schwarz, U. S., Gardel, M. L. Optimization of traction force microscopy for micron-sized focal adhesions. J. Phys: Condensed Matter. 22, 194104-194114 (2010).
- Sabass, B., Gardel, M. L., Waterman, C. M., Schwarz, U. S. High resolution traction force microscopy based on experimental and computational advances. Biophys J. 94, 207-220 (2008).
- Yeung, T. Effects of substrate stiffness on cell morphology, cytoskeletal structure, and adhesion. Cell Motil Cytoskeleton. 60 (1), 24-34 (2005).
- Waterman-Storer, C. M. Microtubule/organelle motility assays. Curr Protoc Cell Biol. , 13.1.1-13.1.21 (1998).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon