Method Article
Imaging verticale di Drosophila Embrioni
In questo articolo
Riepilogo
Qui vi presentiamo un protocollo di montaggio per macchiato Drosophila Embrioni in posizione verticale che permette l'imaging di sezioni trasversali usando la microscopia confocale.
Abstract
Alcuni noti gradienti morfogenetici e movimenti cellulari si verificano lungo l'asse dorsale / ventrale della
Protocollo
1. Metodologia di imaging
- Melt glicerina gelatina in un bagno d'acqua ° 55 ° C per 20-30 minuti fino a quando diventa un liquido omogeneo. Ricciolo, ma non agitare la bottiglia per evitare la formazione di bolle.
- Mentre la gelatina si scioglie, applicare un sottile strato di glicerolo con un Kimwipe su un vetrino pulito (opzionale).
- Pipettare 1 a 1,5 ml di glicerina gelatina sciolta con un P1000 punta tagliata della pipetta sulla superficie del vetrino per creare un sottile strato prima. Pipettare con attenzione per evitare bolle.
- Lasciate che la gelatina solidificare per 5 minuti su una superficie orizzontale, poi posto la diapositiva a -20 ° C per 5 minuti e procedere con l'allineamento degli embrioni. In alternativa, coprire il vetrino con pellicola trasparente e mettetelo a 4 ° C per un massimo di 5 giorni.
- Pipettare una piccola goccia di embrioni macchiato nel 70% glicerolo / PBS o altri mezzi di montaggio Antifade glicerolo-based (ad es Slowfade o Vectashield) sul lato del letto gelatina.
- Iniziare pre-allineamento degli embrioni da parte individualmente di trasferirli dal menù alla superficie gelatina. Trasferirli usando un ago ipodermico inclinato, usando il lato inclinato come un cucchiaio.
- Circa posto gli embrioni lungo 2-3 colonne. Se un embrione si blocca in dell'ago, questo aggiungete la goccia contenente gli embrioni altri per rilasciarlo. Non perdere tempo con cura allineando a questo punto. Lascia un pò di spazio tra le colonne.
- Allineare gli embrioni facendo scorrere orizzontalmente sul supporto gelatina con l'ausilio dell'ago ipodermico. Facendo scorrere, orientare le teste e le code nella stessa direzione. Rendili parallele tra loro lungo la colonna. Rimuovere l'eccesso di glicerolo PBS lasciato come una scia con un pezzo di Kimiwipe contorto.
Nota: glicerolo eccesso causerà embrioni di essere vagamente racchiusi all'interno di gel, con conseguente separazione dei due strati di gelatina, il che rende difficile da tagliare e montare. Al contrario, la rimozione di glicerolo troppo si secca e appiattire embrioni. - Utilizzare una punta di giallo tagliato a diffondere un sottile strato di gelatina negli embrioni (50-150 microlitri, a seconda di quanti erano allineati).
- Porre il vetrino in freezer -20 ° C per 10-20 minuti o conservarlo a 4 ° C per una notte. Assicurarsi che la gelatina è completamente rigido prima del taglio. In caso contrario, il blocco degli embrioni sarà molto difficile da accise. I migliori risultati per il taglio sono ottenuti il giorno successivo.
- In un ambito di dissezione, usare una lama di rasoio per tagliare completamente attraverso la gelatina su entrambi i lati degli embrioni allineate. Tenere la lama di rasoio in un angolo di 90 ° di effettuare tagli diritti. I tagli dovrebbero essere molto vicino ai poli anteriore e posteriore degli embrioni per evitare eccesso di gelatina che può ostacolare l'imaging.
- Aggiungere 100-200 ml di freddo PBT (4 ° C, PBS +0,1% Tween 20) accanto ai tagli e con attenzione raschiare la gelatina tra embrioni con una spatola.
Nota: Il detersivo in PBT aiuta la rimozione della gelatina dalla diapositiva senza danneggiare gli embrioni. - Utilizzando un ago, il trasferimento del blocco di gelatina con embrioni incorporato a un vetrino pulito per il taglio ulteriore. Tagliare i blocchi più piccoli con embrioni 5-7. Usa PBT di muoversi facilmente intorno al blocco di gelatina. In alternativa, posizionare il blocco su una superficie di vetro asciutta per farla attaccare al vetro, che crea una maggiore stabilità proprio per tagliare la gelatina, se necessario.
- Raddrizzarsi gli embrioni incorporato nel gelatina con l'aiuto di una lama di un rasoio e un ago. Per la visualizzazione al microscopio, procedere con uno dei due passaggi riportati di seguito, a seconda del tipo di microscopio stare in uso.
- Microscopio invertito: luogo finito blocchi embrione su un vetrino lungo, con gli embrioni in posizione verticale. Assicurarsi che il lato dell'embrione con la minor quantità di gelatina sarà a più stretto contatto con obiettivo.
- Microscopio verticale: fare due pile con quattro # 1 coprioggetto incollato con colla da utilizzare come "supporto" e posizionare il blocco embrione in glicerolo tra i "supporti". Colmare il pile con un coprioggetto lungo.
- Per l'analisi confocale, controllare la distanza di lavoro degli obiettivi da utilizzare per sapere se è possibile immagine tutto il percorso attraverso l'embrione o solo parzialmente. Per esempio, D. embrioni melanogaster hanno ~ 0,55 millimetri di lunghezza e possono essere interamente ripreso con un Zeiss 20x Plan-Apo o un 40x LD neo-Fluor obiettivo. Il sito contiene informazioni sui seguenti obiettivi Zeiss disponibili e vi dà la possibilità di ricercare gli obiettivi in base alle distanze di lavoro: https://www.micro-shop.zeiss.com/us/us_en/objektive.php
2. Rappresentante Risultati
Perché gli embrioni sono rimasti intatti, questo metodo può essere utilizzato per effettuare misurazioni precise delle dimensioni degli embrioni e distanze tra gli schemi di espressione genica. Nella Figura 1, ci mostrano una embrioni Blastoderm fase colorati con il nucleare macchia DAPI (blu), anti-dorsale delle proteine (rosso), mRNA SOG (giallo) e mRNA sna (verde). Proces morfologicases, come invaginazione del mesoderma (Figura 2A, B) e la migrazione di cellule mesodermiche (Figura 3 e 4) possono anche essere visualizzati utilizzando questo metodo. Z-posizioni diverse lungo l'asse DV può essere ottenuto cambiando il piano focale, come mostrato nelle Figure 2-4.
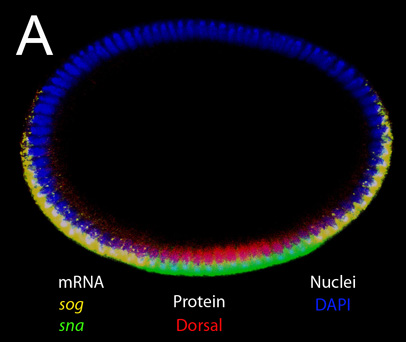
Figura 1. Verticale fetta ottica di un embrione intatto in fase Blastoderm. Colorazione doppia per mRNA e proteine. MRNA bersaglio per le sonde in situ (SOG e SNA) e di proteine colorate con l'anticorpo primario (anti-dorsali) sono indicati in figura. DAPI è stato usato per etichettare i nuclei. Si noti che l'espressione di SOG e SNA si trovano nel citoplasma apicale, mentre l'espressione della dorsale è nucleare. Segnali forti dalle trascrizioni sog nascente che si trovano nei nuclei può essere visto anche in questo ingrandimento.
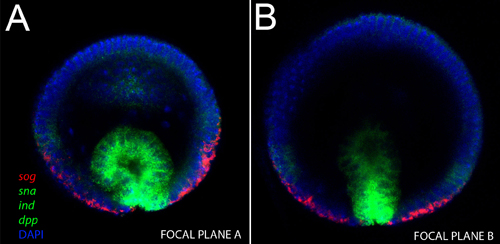
Figura 2. Immagine verticale di un embrione intatto gastrulating. Sonde mRNA usato e loro rispettivi colori sono indicate in figura. A) Si noti che in questa fase, tre geni colorati con lo stesso fluoro (sna, SOG e DPP, in verde), sono espressi in domini separati spazialmente. Il mesoderma invaginating esprime sna, lo strato di neuroblasti esprime ind e l'ectoderma esprime dpp, mentre l'espressione sog è ancora presente nelle regioni ventrale del sistema nervoso. B) In un piano più profondo confocale dell'embrione stesso, si nota la base del mesoderma invaginating esprime fortemente sna, ma la sua regione apicale esprime livelli più bassi.
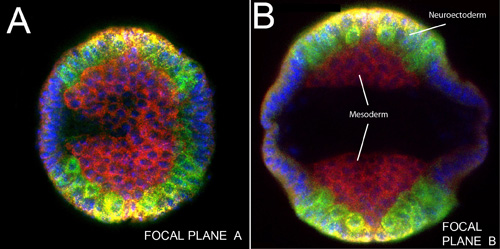
Figura 3. Immagine verticale di un embrione intatto con la band di germe esteso sog RNA (giallo);. Lumaca, ind e mRNA DPP (verde), proteine dorsali (rosso). I nuclei sono stati colorati con DAPI (blu). A) sul piano focale vicino alla testa dell'embrione. B) Un altro piano focale nella regione del tronco dell'embrione stesso. Massa nota di cellule mesodermiche prima della migrazione laterale (confronta con Figura 4) aderente alla linea mediana ventrale su entrambi i lati di un germe-band embrione esteso. Neuroblasti delaminazioni sono etichettati in verde (ind e SNA).
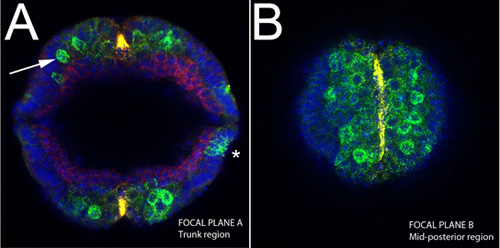
Figura 4. Immagine verticale di un embrione intatto durante l'estensione di germe di banda sog RNA (giallo);. Lumaca, ind e mRNA DPP (verde), proteine dorsali (rosso). A) Si noti la neuroblasti delaminazioni dal epitelio (freccia), macchiato sia per ind e SNA in questa fase (verde). Si noti anche l'espressione ristretto di SOG alla linea mediana ventrale (giallo). Espressione di DPP in questa fase può essere visto facce laterali dell'embrione (asterisco). B) sezione ottica nell'embrione stesso mostrato in A verso la fine della lunghezza dell'embrione, che corrisponde alla metà posteriore regione. Le cellule della linea mediana ventrale sono macchiati di sog.
Discussione
Prendendo immagini sezione di embrioni di Drosophila può essere impegnativo, perché richiede sia una dissezione accurata mano di fette di 2 o l'uso dei mezzi di comunicazione embedded per l'elaborazione microtomo, che di solito è dannoso per colorazioni fluorescenti. In alternativa, Z-stack fatto in un microscopio confocale può essere usato per fare la ricostruzione 3D della sezione immagini. Tuttavia, è tempo di fare varie fette sottili confocale per una accurata ricostruzione 3D e photobleaching diventa problematico. Un'altra preoccupazione quando gli embrioni di imaging che sono montati longitudinalmente è la dispersione della luce che si verifica quando si raggiunge più sezioni dell'embrione, che complica anche le misurazioni precise dei segnali fluorescenti a fini di quantificazione dei livelli di espressione della proteina o di mRNA 1. Anche con un microscopio a due fotoni confocale, dispersione della luce è ancora una preoccupazione a causa della opacità del materiale tuorlo al centro dell'embrione, che fa selezione di supporti di montaggio per la massima trasparenza del campione a essere limitata.
Per aggirare questi problemi, una nuova tecnica di imaging chiamata "End-on" degli embrioni posizionamento in verticale è stato recentemente sviluppato per l'immagine sia dal vivo che i campioni fisso 4. In questo lavoro, abbiamo sviluppato un nuovo protocollo per gli embrioni montate verticalmente che permette una semplice preparazione e la visualizzazione di embrioni fisso o tessuti di qualsiasi dimensione e forma che si sono macchiati con più sonde in situ 3. Il nostro metodo consiste nell'utilizzare un supporto di montaggio con consistenza gelatinosa che viene usato per racchiudere gli embrioni allineati al suo interno. Blocchi tagliati di questo mezzo di montaggio contenente gli embrioni incorporato può essere posizionato in posizione verticale prima immagine in qualsiasi tipo di microscopio confocale.
Integrando gli embrioni in gelatina e il posizionamento in verticale, siamo in grado di prendere orizzontale sezioni usando la microscopia confocale, in cui vengono presentati cellule lungo il dorso-ventrale asse dell'embrione contemporaneamente. Questo è un miglioramento significativo ai fini quantificazione perché i problemi di dispersione della luce e photobleaching di coloranti fluorescenti che si verifica in convenzionale Z-stack ricostruzione di embrioni montato longitudinalmente sono ora eliminati. Così, la quantificazione dei livelli di espressione del gene o della proteina lungo l'asse dorso-ventrale è preciso, dal momento che le cellule situato di fronte dorso-ventrale posizioni sono esposte nello stesso momento e nello stesso tempo confocale Z-posizione. Inoltre, il metodo descritto permette di ottenere una completa immagine 2-D in tutto il dorso-ventrale asse da un 3-D embrione senza l'utilizzo di complessi metodi computazionali svolgimento di visualizzare le cellule in posizioni diverse lungo l'asse DV 6.
Alcuni dei passaggi critici includere la rimozione glicerolo in eccesso dopo l'allineamento degli embrioni, per evitare di dividere tra i due strati di gelatina. Tuttavia, se il campione è troppo disidratato, l'immagine risultante sarà deformi e hanno una morfologia corretta. Inoltre, se il mezzo gelatina intorno l'embrione viene tagliato con un angolo piuttosto che direttamente, l'immagine risultante può apparire meno rotondo e più in forma ovulare, a causa di un non corretto posizionamento degli embrioni a un angolo di 90 gradi. Infine, se la gelatina non viene tagliato strettamente sufficiente per l'embrione sulle estremità anteriore / posteriore, non è possibile ottenere una immagine corretta distanza di lavoro, perché l'obiettivo sarebbe stato utilizzato principalmente per concentrarsi nella gelatina e non l'embrione.
Un altro passo importante che può essere necessaria quando l'imaging embrione gastrulating fine è quello di ordinare con attenzione le fasi di interesse nell'ambito di applicazione prima di allineare loro. Di solito, gli embrioni sono messo in scena in base alle caratteristiche morfologiche che possono essere più facilmente riconoscibile quando è in posizione longitudinale.
Tra le modifiche alternativi che possono essere fatte con questo metodo è quello di includere un anti-fade reagente (ad esempio p-fenilendiammina o PPD) nel secondo strato di gelatina per aiutare a proteggere contro photobleaching di coloranti fluorescenti.
Divulgazioni
Riconoscimenti
Gli autori sono particolarmente in debito con Deborah Harris e Danielle MacKay. Il supporto per questo lavoro è stato fornito dal Dipartimento di Biologia e il Collegio di Arti e Scienze di CWRU, e in base al numero 52005866 HHMI concedere per il supporto della formazione universitaria nelle scienze biologiche.
Materiali
| Name | Company | Catalog Number | Comments |
| Glycerin Jelly mounting media | Electron Microscopy Sciences | 17998-10 | Contains low concentration of phenol as preservative and should be used in hood or in well ventilated area. Recipe for making your own media is described in Zander, 1997. |
| Zeiss LSM 700 Confocal | Carl Zeiss, Inc. | The following objectives were used: Plan-Apo 20x 0.8 M27 (WD 0.55mm); EC Plan-Neofluar 40x 1.3 oil (WD 0.21mm); LD Plan-Neofluar 0.6 Korr 40X (WD 2.9mm). | |
| Phosphate buffered saline with 0.1% Tween 20 (PBT) | |||
| Glass microscope slides | Fisher Scientific | 12-544-7 | |
| Glass cover slips | Electron Microscopy Sciences | 72204-02 | Size 1 ½ (specific for the objectives used in this work). |
| Glass cover slips (for making supports) | Fisher Scientific | 12-544-10 | Use four coverslips glued with superglue. |
| Dissection microscope | M5 Wild Heerbrugg Wild Makroskop | M420 | |
| Razor blade | Steel back single edge industrial blades | ||
| Spatula | Fisher Scientific | 21-401-10 | |
| Hypodermic needles | Fisher Scientific | 1482610 | 26 Gauge |
| Pipettes | Labnet International |
Riferimenti
- Ay, A., Fakhouri, W. D., Chiu, C., Arnosti, D. N. Image processing and analysis for quantifying gene expression from early Drosophila embryos. Tissue Engineering. 14, 1517-1526 (2008).
- Grβhans, J., Wieschaus, E. A genetic link between morphogenesis and cell division during formation of the ventral furrow in Drosophila. Cell. 101, 523-531 (2000).
- Kosman, D., Mizutani, C. M., Lemons, D., Cox, W. G., McGinnis, W., Bier, E. Multiplex detection of RNA expression in Drosophila embryos. Science. 305, 846-846 (2004).
- Witzberger, M. M., Fitzpatrick, J. A. J., Crowley, J. C., Minden, J. S. End-on imaging: a new perspective on dorsoventral development in Drosophila embryos. Developmental Dynamics. 237, 3252-3259 (2008).
- Zander, R. H. On mounting delicate bryophytes in glycerol. The Bryologist. 100, 380-382 (1997).
- Liberman, L. M., Reeves, G. T., Stathopoulos, A. Quantitative imaging of the Dorsal nuclear gradient reveals limitations to threshold-dependent patterning in Drosophila. Proc Natl Acad Sci U S A. 106 (52), 22317-2222 (2009).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon