Method Article
Imagem vertical de Drosophila Os embriões
Neste Artigo
Resumo
Aqui apresentamos um protocolo de montagem para manchada Drosophila Embriões em posição vertical que permite que imagens de secções transversais através de microscopia confocal.
Resumo
Vários gradientes conhecidos morfogenéticos e movimentos celulares ocorrem ao longo do eixo dorsal / ventral da
Protocolo
1. Metodologia de imagens
- Derreter a glicerina em uma geléia de 55 ° C banho de água grau por 20-30 minutos até que se torne um líquido homogêneo. Redemoinho, mas não agitar o frasco para evitar a formação de bolhas.
- Enquanto a gelatina derrete, aplique uma camada fina de glicerol usando um Kimwipe em uma lâmina de vidro limpa (opcional).
- Pipeta mL 1-1,5 de glicerina derretida geléia usando um corte de ponta da pipeta P1000 na superfície da lâmina para criar uma camada fina em primeiro lugar. Pipeta com cuidado para evitar bolhas.
- Deixe a gelatina solidificar por 5 min em uma superfície horizontal, em seguida, coloque o slide a -20 ° C por 5 min e proceder ao alinhamento do embrião. Alternativamente, cobrir a lâmina com Saran Wrap e coloque-a 4 ° C por até 5 dias.
- Pipetar uma pequena gota de embriões manchada em glicerol 70% / PBS ou outros Antifade glicerol baseada meios de montagem (por exemplo, ou Slowfade Vectashield) no lado da cama de geléia.
- Começar a pré-alinhamento dos embriões por transferi-los individualmente a partir da queda para a superfície da geléia. Transferi-los usando uma agulha hipodérmica inclinada, usando o lado inclinado como uma colher.
- Cerca de colocar os embriões ao longo de 2-3 colunas. Se um embrião fica preso na agulha, mexa na gota contendo os embriões outros para liberá-lo. Não perca tempo alinhando-as com cuidado neste momento. Deixe algum espaço entre as colunas.
- Alinhar os embriões deslizando-los horizontalmente no meio de geléia com o auxílio da agulha hipodérmica. Enquanto desliza, orientar o cara e coroa na mesma direção. Torná-los paralelos um ao outro ao longo da coluna. Remover o excesso de glicerol PBS esquerda como uma trilha com um pedaço de Kimiwipe torcida.
Nota: glicerol em excesso causará embriões para ser vagamente envolto em gel, resultando na separação das duas camadas de geléia, tornando-o difícil de cortar e montar. Por outro lado, a remoção de glicerol demais vai secar e alisar os embriões. - Use uma ponta de corte amarelo para espalhar uma fina camada de geléia sobre os embriões (50-150 mL, dependendo de quantas foram alinhados).
- Slides lugar no freezer -20 ° C por 10-20 min ou guarde-a 4 ° C durante a noite. Certifique-se que a geléia é completamente rígida, antes de cortar. Caso contrário, o bloco de embriões será muito difícil especiais de consumo. Os melhores resultados são obtidos para o corte no dia seguinte.
- De acordo com um escopo de dissecação, use uma lâmina de barbear para cortar completamente através da geléia de ambos os lados dos embriões alinhados. Segure a lâmina de barbear em um ângulo de 90 ° para fazer cortes retos. Os cortes devem ser feitos muito perto dos pólos anterior e posterior dos embriões para evitar o excesso de geléia que pode obstruir imagem.
- Adicionar 100-200 mL de frio PBT (4 ° C, PBS 0,1% Tween 20) próximo aos cortes e com cuidado raspe a gelatina entre os embriões usando uma espátula.
Nota: O detergente em PBT ajuda a remoção da geléia a partir do slide sem danificar embriões. - Usando uma agulha, transferir o bloco de geléia com embriões incorporados a um slide limpa para cortar ainda mais. Cortar blocos menores com embriões 5-7. Use PBT facilmente mover o bloco de gelatina. Alternativamente, coloque o bloco sobre uma superfície de vidro seco para deixá-lo ligar para o vidro, o que gera mais estabilidade para cortar precisamente a geléia, se necessário.
- Virar na vertical os embriões embutido na geléia com o auxílio de uma lâmina de barbear e uma agulha. Para visualização no microscópio, prosseguir com qualquer uma das duas etapas abaixo, dependendo do tipo de microscópio estar sendo usado.
- Microscópio invertido: lugar terminou blocos de embrião para uma lamela de comprimento, com embriões em posição vertical. Verifique se o lado do embrião com a menor quantidade de geléia estará em contato mais próximo com o objectivo.
- Microscópio vertical: fazer duas pilhas com quatro # 1 lamínulas coladas com super cola para ser usado como "suporte" e colocar o bloco de embriões em glicerol entre as "suporta". Ponte das pilhas usando uma lamela de comprimento.
- Confocal para análise, verificar a distância de trabalho dos objectivos a ser usado para descobrir se você pode imaginar todo o caminho através do embrião ou apenas parcialmente. Por exemplo, D. embriões têm melanogaster ~ 0,55 milímetros de comprimento e pode ser inteiramente fotografada usando uma lente 20x Plano de Apo-ou 40x LD objetivo Neo-Fluor. O site a seguir tem informações sobre os objectivos Zeiss disponíveis e lhe dá a opção de pesquisar os objectivos de acordo com distâncias de trabalho: https://www.micro-shop.zeiss.com/us/us_en/objektive.php
2. Resultados representante
Porque os embriões são deixados intactos, este método pode ser usado para fazer medições precisas de tamanho do embrião e as distâncias entre os padrões de expressão gênica. Na Figura 1, mostramos uma embriões blastoderma estágio manchadas com o DAPI nuclear mancha (azul), anti-dorsais proteína (vermelho), mRNA sog (amarelo) e mRNA sna (verde). Proces morfológicasses, como a invaginação do mesoderma (Figura 2A, B) e migração das células mesodérmica (Figura 3 e 4) também pode ser visualizado utilizando este método. Diferentes posições-Z ao longo do eixo DV podem ser obtidas alterando o plano focal, como mostrado nas Figuras 2-4.
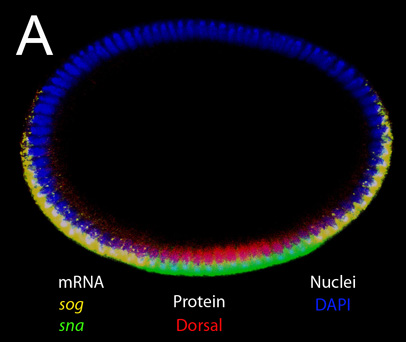
Figura 1. Vertical óptico fatia de um embrião intacto em estágio blastoderma. Coloração duplo mRNA e proteína. MRNAs alvo para sondas em situ (sog e sna) e proteína manchada pelo anticorpo primário (anti-Dorsal) são indicadas na figura. DAPI foi usado para rotular os núcleos. Note-se que a expressão de sog e sna estão localizados no citoplasma apical, enquanto a expressão de dorsais é nuclear. Sinais fortes de transcrições sog nascentes que estão localizadas em núcleos também podem ser vistos sob esta ampliação.
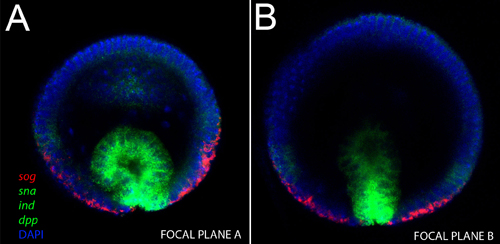
Figura 2. Testes de imagem na vertical de um embrião intacto gastrulating. MRNA utilizados e suas respectivas cores são indicadas na figura. A) Note-se que nesta fase, três genes corados com fluor mesmo (sna, sog e dpp, em verde), são expressos em domínios separados espacialmente. A mesoderme invaginante expressa sna, a camada de neuroblastos expressa ind eo ectoderma expressa dpp, enquanto expressão sog ainda está presente nas regiões ventral do sistema nervoso. B) Em um plano mais profundo confocal do embrião mesmo, notamos a base do mesoderma invaginante fortemente expressa sna, mas a sua região apical expressa níveis mais baixos.
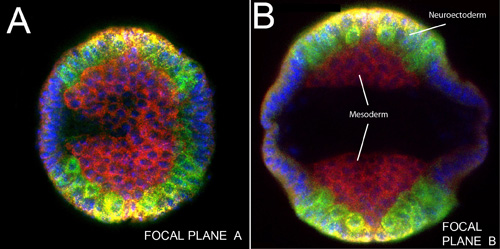
Figura 3. Imagem na vertical de um embrião intacto com a banda de germe estendida sog RNA (amarelo);. Caracol, ind e mRNAs dpp (verde), proteína dorsais (vermelho). Núcleos foram corados com DAPI (azul). A) plano focal perto da cabeça do embrião. B) Outro plano focal na região do tronco de embrião. Nota massa de células antes da migração mesodérmica lateral (compare a Figura 4) deitado próximo à linha média ventral em ambos os lados de um embrião de germe de banda estendida. Neuroblastos delaminação são rotulados em verde (ind e sna).
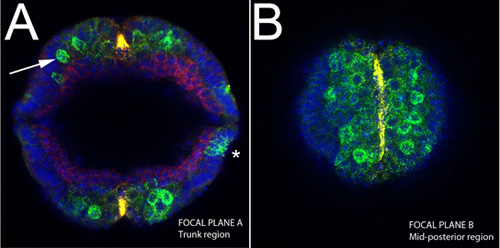
Figura 4. Imagem na vertical de um embrião intacto durante a extensão banda germe sog RNA (amarelo);. Caracol, ind e mRNAs dpp (verde), proteína dorsais (vermelho). A) Observe o neuroblastos delaminação do epitélio (seta), manchado tanto para ind e sna nesta fase (verde). Note-se também a expressão restrito de sog à linha média ventral (amarelo). Expressão de dpp nesta fase pode ser visto em laterais do embrião (asterisco). B) seção Optical no embrião mesmo mostrado na A perto do final do comprimento do embrião, o que corresponde a mid-posterior região. As células da linha média ventral estão manchadas de sog.
Discussão
Obtenção de imagens seção transversal de embriões Drosophila pode ser um desafio, pois exige tanto uma dissecção cuidadosa das mãos de fatias 2 ou o uso de meios embutido para micrótomo de processamento, que normalmente é prejudicial para colorações fluorescentes. Alternativamente, Z-stacks feita em um microscópio confocal pode ser usado para fazer a reconstrução 3D de seção transversal imagens. No entanto, é demorado para fazer várias fatias finas de Confocal para uma reconstrução 3D preciso e torna-se problemático fotobranqueamento. Outra preocupação quando embriões de imagem que são montados longitudinalmente é o espalhamento de luz que ocorre quando chegar seções mais profundas do embrião, o que também complica a tomar medidas precisas de sinais fluorescentes para fins de quantificação dos níveis de proteína ou mRNA expressão 1. Mesmo com um microscópio de dois fotões confocal, espalhamento de luz ainda é uma preocupação devido à opacidade do material gema no meio do embrião, o que faz seleção de meios de montagem de transparência ideal da amostra a ser limitado.
Para contornar esses problemas, uma nova técnica chamada "End-on" de imagens de embriões de posicionamento vertical foi recentemente desenvolvido para a imagem ao vivo e amostras fixas 4. Neste trabalho, desenvolvemos um novo protocolo para embriões montado verticalmente, que permite uma preparação simples e visualização de embriões fixo ou tecidos de qualquer tamanho e forma que são corados com múltiplas sondas situ 3. Nosso método consiste em utilizar uma mídia de montagem com consistência gelatinosa que é usado para envolver embriões alinhados dentro dele. Blocos cortados deste meios de montagem contendo embriões incorporado pode ser posicionada na vertical antes de imagens em qualquer tipo de microscópio confocal.
Ao incorporar os embriões para a geléia e posicioná-los na vertical, somos capazes de tomar horizontal cross-seções usando microscopia confocal, em que as células ao longo do eixo dorsal-ventral do embrião são apresentados simultaneamente. Esta é uma melhoria significativa para fins de quantificação desde problemas de espalhamento de luz e fotodegradação de corantes fluorescentes que ocorre na reconstrução Z-stack convencional de embriões montado longitudinalmente agora são eliminados. Assim, a quantificação dos níveis de expressão de gene ou proteína ao longo do eixo dorsal-ventral é precisa, uma vez que as células localizadas na frente dorsal-ventral posições são gravadas ao mesmo tempo e, ao mesmo confocal Z-position. Além disso, o método descrito nos permite obter uma imagem 2-D completo ao longo do eixo dorsal-ventral de um embrião de 3-D sem o uso de complexos métodos computacionais unrolling para visualizar as células em diferentes posições ao longo do eixo DV 6.
Alguns dos passos críticos incluem a remoção de glicerol em excesso após o alinhamento embriões, para evitar a divisão entre as duas camadas de geléia. No entanto, se a amostra é muito desidratado, a imagem resultante será disforme e têm morfologia incorreta. Além disso, se o meio de geléia de todo o embrião é cortado em um ângulo em vez de reto, a imagem resultante pode parecer menos redondo e mais ovular em forma, devido a um posicionamento incorreto dos embriões em um ângulo diferente de 90 graus. Finalmente, se a geléia não é cortado perto o suficiente para o embrião nas extremidades anterior / posterior, não é possível obter uma imagem apropriada porque a distância de trabalho o objetivo seria usado principalmente para se focalizar no jelly e não o embrião.
Outro passo importante que pode ser necessária quando embrião imagem final gastrulating é cuidadosamente classificar os estágios de interesse no âmbito antes de alinhar-los. Normalmente, os embriões são encenadas de acordo com características morfológicas que podem ser mais facilmente reconhecida quando em uma posição longitudinal.
Entre as modificações alternativa que pode ser feito com este método é incluir um reagente anti-fade (por exemplo, p-fenilenodiamina ou PPD) para a segunda camada de gelatina para ajudar a proteger contra a fotodegradação de corantes fluorescentes.
Divulgações
Agradecimentos
Os autores são especialmente gratos a Deborah Harris e MacKay Danielle. Suporte para este trabalho foi fornecida pelo Departamento de Biologia e da Faculdade de Artes e Ciências de CWRU, e por HHMI número de concessão 52005866 para apoio ao ensino de graduação em ciências biológicas.
Materiais
| Name | Company | Catalog Number | Comments |
| Glycerin Jelly mounting media | Electron Microscopy Sciences | 17998-10 | Contains low concentration of phenol as preservative and should be used in hood or in well ventilated area. Recipe for making your own media is described in Zander, 1997. |
| Zeiss LSM 700 Confocal | Carl Zeiss, Inc. | The following objectives were used: Plan-Apo 20x 0.8 M27 (WD 0.55mm); EC Plan-Neofluar 40x 1.3 oil (WD 0.21mm); LD Plan-Neofluar 0.6 Korr 40X (WD 2.9mm). | |
| Phosphate buffered saline with 0.1% Tween 20 (PBT) | |||
| Glass microscope slides | Fisher Scientific | 12-544-7 | |
| Glass cover slips | Electron Microscopy Sciences | 72204-02 | Size 1 ½ (specific for the objectives used in this work). |
| Glass cover slips (for making supports) | Fisher Scientific | 12-544-10 | Use four coverslips glued with superglue. |
| Dissection microscope | M5 Wild Heerbrugg Wild Makroskop | M420 | |
| Razor blade | Steel back single edge industrial blades | ||
| Spatula | Fisher Scientific | 21-401-10 | |
| Hypodermic needles | Fisher Scientific | 1482610 | 26 Gauge |
| Pipettes | Labnet International |
Referências
- Ay, A., Fakhouri, W. D., Chiu, C., Arnosti, D. N. Image processing and analysis for quantifying gene expression from early Drosophila embryos. Tissue Engineering. 14, 1517-1526 (2008).
- Grβhans, J., Wieschaus, E. A genetic link between morphogenesis and cell division during formation of the ventral furrow in Drosophila. Cell. 101, 523-531 (2000).
- Kosman, D., Mizutani, C. M., Lemons, D., Cox, W. G., McGinnis, W., Bier, E. Multiplex detection of RNA expression in Drosophila embryos. Science. 305, 846-846 (2004).
- Witzberger, M. M., Fitzpatrick, J. A. J., Crowley, J. C., Minden, J. S. End-on imaging: a new perspective on dorsoventral development in Drosophila embryos. Developmental Dynamics. 237, 3252-3259 (2008).
- Zander, R. H. On mounting delicate bryophytes in glycerol. The Bryologist. 100, 380-382 (1997).
- Liberman, L. M., Reeves, G. T., Stathopoulos, A. Quantitative imaging of the Dorsal nuclear gradient reveals limitations to threshold-dependent patterning in Drosophila. Proc Natl Acad Sci U S A. 106 (52), 22317-2222 (2009).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados