È necessario avere un abbonamento a JoVE per visualizzare questo. Accedi o inizia la tua prova gratuita.
Method Article
Saggi biofisici per sondare le proprietà meccaniche del nucleo della cellula Interphase: Strain supporto Applicazione e manipolazione microago
In questo articolo
Riepilogo
Vi presentiamo due indipendenti, microscopio a base di strumenti per misurare le deformazioni indotte nucleare e del citoscheletro in camere singole, cellule viventi aderenti in risposta alla applicazione dello sforzo globale o localizzato. Queste tecniche vengono utilizzate per determinare la rigidità nucleare (cioè, deformabilità) e di sondare la trasmissione intracellulare vigore tra il nucleo e il citoscheletro.
Abstract
Nella maggior parte delle cellule eucariotiche, il nucleo è l'organello più grande ed è tipicamente 2 a 10 volte più rigido del citoscheletro circostante, di conseguenza, le proprietà fisiche del nucleo contribuire in modo significativo il comportamento generale biomeccanico delle cellule in condizioni fisiologiche e patologiche. Per esempio, nella migrazione dei neutrofili ed invadere le cellule tumorali, rigidità nucleare può rappresentare un grosso ostacolo durante stravaso o il passaggio attraverso spazi ristretti all'interno dei tessuti 1. D'altra parte, il nucleo delle cellule nei tessuti meccanicamente attivi come muscolare richiede un adeguato sostegno strutturale resistere alle ripetute sollecitazioni meccaniche. È importante sottolineare che il nucleo è strettamente integrato nell'architettura di cellulari, ma è fisicamente collegato al citoscheletro circostante, che è un requisito fondamentale per il movimento intracellulare e il posizionamento del nucleo, per esempio, in cellule polarizzate, nuclei sinaptica a giunzioni neuromuscolari, o nella migrazione delle cellule. 2 Non sorprendentemente, mutazioni nelle proteine dell'involucro nucleare, come lamine e nesprins, che svolgono un ruolo critico nel determinare la rigidità nucleare e nucleo-citoscheletro accoppiamento, hanno dimostrato di recente per portare a una serie di malattie umane, tra cui Emery- Dreifuss distrofia muscolare, distrofia dei cingoli distrofia muscolare e cardiomiopatia dilatativa. 3 Per studiare la funzione di biofisica delle diverse proteine dell'involucro nucleare e l'effetto di mutazioni specifiche, abbiamo sviluppato metodi sperimentali per studiare le proprietà fisiche del nucleo in camere singole, le cellule viventi sottoposti a globale o localizzata perturbazione meccanica. Misura delle deformazioni indotte nucleari in risposta alla domanda di supporto proprio applicata ceppo rendimenti importanti informazioni sulla deformabilità del nucleo e permette di confronto quantitativo tra le diverse mutazioni o linee cellulari deficienti per specifiche proteine dell'involucro nucleare. Localizzata applicazione dello sforzo del citoscheletro con un microago viene utilizzato per integrare il dosaggio e può fornire ulteriori informazioni sulla trasmissione della forza intracellulare tra il nucleo e il citoscheletro. Studiare meccanica nucleare nelle cellule viventi conserva intatte l'architettura normale intracellulare ed evita artefatti potenziali che possono sorgere quando si lavora con nuclei isolati. Inoltre, lo sforzo di applicazione substrato presenta un buon modello per lo stress fisiologico sperimentato da cellule muscolari o di altri tessuti (per esempio, cellule muscolari lisce vascolari esposti al ceppo nave). Infine, mentre questi strumenti sono stati sviluppati principalmente per studiare la meccanica nucleare, possono anche essere applicato per studiare la funzione delle proteine del citoscheletro e di segnalazione meccanotrasduzione.
Protocollo
1. Substrato ceppo applicazione
La misurazione della pressione normalizzata nucleare comprende la preparazione di piatti tensioni con trasparente, membrane di silicone elastico come superficie di coltura cellulare di cellule placcatura sulle stoviglie, e l'acquisizione delle immagini delle cellule prima, durante e dopo (uniassiale o biassiale) applicazione dello sforzo.
Preparazione di piatti membrana di silicone e l'aderenza delle cellule
- Ogni piatto è costituito da un ceppo su misura piatto fondo di plastica con un diametro di 3 "e un O-ring in plastica per tenere una membrana di silicone, che funge da substrato di coltura cellulare. Per la preparazione dei piatti ceppo, pinza a 4" x 4 "pezzo di membrana di silicone tra l'O-ring e il piatto. accuratamente tagliato via la membrana in eccesso, sciacquare con acqua deionizzata, e autoclave i piatti tensioni.
- Segnare un punto di riferimento nel centro della parte inferiore della membrana (all'esterno) prima di rivestimento delle membrane di silicone con le molecole della matrice extracellulare (ad esempio, fibronectina). Questo punto di riferimento aiuterà a identificare le stesse cellule durante gli esperimenti di deformazione. (Opzionale: per applicazione dello sforzo uniassiale, due strisce parallele di scotch sono applicati in tutto il punto di riferimento per limitare le deformazioni della membrana in una dimensione.)
- Per fornire attaccamento ottimale delle cellule, il cappotto le membrane in silicone con 3 mg / ml fibronectina diluito in 10 ml di PBS o proteine della matrice extracellulare adatto. Coprire il piatto ceppo con un piatto capovolto polistirolo 10 cm, e incubare i piatti per tutta la notte a 4 ° C.
- Il giorno dopo, lavare le membrane una volta con tampone fosfato (PBS) per rimuovere le proteine in eccesso. Riempire piatto con 10 ml di mezzo di crescita (Dulbecco Modified Eagles Medium (glucosio) supplementato con 10% siero fetale bovino e 1% di penicillina / streptomicina) e mettere da parte.
- Topo fibroblasti embrionali sono trypsinized con il 0,05% tripsina e seminate nel terreno di crescita di circa il 30% confluenza sul piatto rivestito membrana di silicone e incubare per 24 - 48 ore in condizioni normali cultura.
Substrato ceppo esperimenti
- Set-up al microscopio per gli esperimenti. Gli esperimenti vengono eseguiti su un microscopio invertito con una fotocamera digitale adatto per la microscopia a fluorescenza, contrasto di fase o DIC, utilizzando un obiettivo 60x e adeguato software di acquisizione immagini (ad esempio, IPLab o Metamorph). Un microscopio in posizione verticale non è adatto per questa applicazione. Il dispositivo di tensione è costituito da una piastra di base che si adatta in scena microscopio e tiene un rullo cilindrico centrale che serve per applicare tensione alla sezione centrale della membrana di silicone, un piatto mobile che contiene il ceppo e piatto che può scorrere su e giù su quattro perni di guida, oltre a un 5-lb piastra del peso di applicare un carico.
- Al fine di visualizzare i nuclei, incubare le cellule del ceppo piatto con 1 mg / mL di Hoechst 33342 per 15 minuti a 37 ° C. Aspirare al largo della media e sostituire con 15 ml di fenolo rosso mezzo di crescita libera (fenolo-rosso senza Dulbecco Modified Eagles Media (glucosio) con 25 mM Hepes, supplementato con 10% di siero fetale bovino e 1% di penicillina / streptomicina). Avvitare il piatto versare nel portatarga piatto. Applicare attentamente grasso (Braycote 804 Grease vuoto) al perimetro della parte inferiore della membrana di silicone per assicurare scorrimento della membrana lungo la piastra centrale. Assicurarsi di mantenere la parte centrale della membrana trasparente.
- Posizionare la piastra di base sul palco microscopio. Montare portatarga piatto con attenzione sulla piastra di base. Assicurarsi che nella posizione iniziale di riposo, la membrana di silicone del piatto ceppo poggia liberamente sulla piastra centrale.
- In primo luogo, si concentrano sul fondo della membrana di silicone e trovare il punto centrale di riferimento nero. Il punto servirà come punto di partenza per tutte le acquisizioni delle immagini e aiuta a localizzare le cellule stesse durante e dopo l'allungamento. Usiamo un programma appositamente scritto automatizzato di imaging per memorizzare le posizioni di cellule e di trasferire queste cellule durante l'esperimento, ma questo può essere ottenuta anche manualmente.
- Partendo dal punto, regolare la messa per visualizzare le cellule e la parte superiore della membrana di silicone. Individuare ben diffusa cellule con nuclei in posizione centrale e di acquisire un contrasto di fase e una immagine di fluorescenza nucleare Hoechst macchia. L'immagine a contrasto di fase dovrebbe concentrarsi sul contorno delle cellule e la membrana di silicone, mentre le immagini a fluorescenza dovrebbe concentrarsi sul piano centrale del nucleo.
- Dopo l'acquisizione di immagini da 5 a 15 celle, tornare al punto centrale. Lentamente si applica il peso del piatto ceppo, con conseguente applicazione dello sforzo uniforme al centro del piatto. La deformazione massima applicata substrato è limitata da distanziali in nylon immessi sul perni di allineamento verticale (pin guida).
- Focus sul fondo della membrana di silicone e individuare il punto di riferimento nuovo. A partire dalpunto, spostare le stesse cellule e ancora acquisire un contrasto di fase ed una immagine fluorescenza delle cellule e nuclei sotto sforzo pieno, cercando di corrispondere al meglio alla piani focali delle immagini iniziali. Questo processo non deve superare i 10 minuti per evitare di rimodellamento attivo e di adattamento della cellula al substrato tesa.
- Dopo tutte le immagini corrispondenti sono stati acquisiti, spostare lo stadio microscopio torna al punto di partenza. Rimuovere con attenzione il peso dalla piastra di supporto piatto e lasciare la membrana di silicone per rilassarsi. Se necessario, spingere delicatamente il piatto ceppo fino a quando non si trova nella posizione iniziale. Quindi acquisire contrasto di fase e le immagini di fluorescenza del post-deformazione cellule come descritto sopra per le immagini ceppo.
Analisi
- Immagini di cellule e nuclei fluorescente prima, durante e dopo l'applicazione ceppo vengono analizzati per calcolare la tensione normalizzata nucleare. Nel nostro laboratorio, si usa una custom-scritto lo script MATLAB per l'analisi, ma diverse opzioni alternative sono disponibili. L'analisi viene eseguita in tre fasi.
- In primo luogo, per calcolare la tensione applicata substrato, le posizioni da 3 a 6 punti di controllo situati sulla membrana sono abbinati manualmente tra le corrispondenti pre-, full-, e post-deformazione delle immagini. Il programma MATLAB calcola quindi il ceppo membrana applicata mettendo a confronto le posizioni dei punti di controllo di corrispondenza tra le immagini pre-deformazione e full-tensione e anche la tensione residua tra il pre-deformazione e il post-deformazione delle immagini. Allo stesso tempo, i punti di controllo sono usati per registrare le coppie di immagini, che contribuirà a rilevare delle cellule danneggiate o distacco (vedi Figura 1).
- In una seconda fase, i nuclei vengono selezionati manualmente utilizzando un programma separato MATLAB che calcola ceppo nucleari per ogni nucleo individuale che corrisponde ad una dimensione nucleare o marcatori intranucleari tra corrispondenti pre-, full-, e post-deformazione delle immagini fluorescenti. Per tenere conto di piccole variazioni nella tensione della membrana applicata tra esperimenti diversi, esprimiamo risultati come Normalized Strain nucleare, definita come il rapporto di tensione indotto nucleare per la tensione della membrana applicata che viene calcolato per ogni nucleo. Gli script MATLAB sono disponibili presso il laboratorio Lammerding su richiesta.
- Infine, ogni nucleo è convalidato, escluse le misure a partire da cellule che si staccano o danneggiati durante l'applicazione ceppo (Figura 1).
2. Microago manipolazione test
Preparazione dei piatti, cellule aderenti, e microaghi
- Incubare 35 millimetri dal fondo di vetro piatti di coltura di cellule con una bassa concentrazione di fibronectina (0.5ug/ml) a Saline salina tampone di Hank (HBSS) o le proteine della matrice extracellulare adatto per 2 ore a 37 ° C. Lavare i piatti con HBSS due volte e aggiungere 2 ml di terreno di crescita per il piatto prima di procedere alla fase successiva.
- Topo fibroblasti embrionali sono trypsinized con il 0,05% tripsina e testa di serie in 2 ml di mezzo di crescita seminato a 7,5 x 10 4 cellule / ml sulle stoviglie in vetro fibronectina rivestite fondo. Cellule posto torna in incubatrice durante la notte. Cellule posto torna in incubatrice durante la notte. Si dovrebbe ottimizzare il numero di cellule per ottenere singolo aderente, non confluenti cellule per altri tipi di cellule.
- Tirare la microaghi, fatto di borosilicato capillari, alla punta diametro di circa 1-3 micron con una pipetta estrattore commerciale (ad esempio, Sutter Instrument Company).
Microago manipolazione esperimento
- Il giorno dopo, incubare le cellule con MitoTracker mitocondriale macchia (600 mM, Invitrogen) e Hoechst 33342 nucleare macchia (1 mg / mL) aggiunto al terreno di coltura per 30 minuti a 37 ° C incubatore.
- Lavare le cellule una volta in HBSS per 5 minuti a temperatura ambiente e poi aggiungere fenolo-rosso medio libera crescita alle cellule per l'imaging.
- Acquisire un'immagine della singola cellula, senza il microago inserito nel citoscheletro in contrasto di fase, una immagine fluorescente dell'immagine Hoechst 33342 e una macchia fluorescente dei mitocondriale macchia, con un obiettivo 60x (0,70 NA, Plan-Achromat) su una rovesciata con un microscopio digitale ad accoppiamento di carica fotocamera del dispositivo.
- Utilizzando un micromanipolatore (ad esempio, InjectMan NI 2, Eppendorf), inserire con cautela il microago nel citoplasma di una cellula una distanza fissa (di solito 5 micron) di distanza dalla periferia nucleare e prendere una immagine a contrasto di fase, una immagine fluorescente della Hoechst 33342 macchia e una immagine fluorescente della macchia mitocondriale. Per questa applicazione, aiuta a controllare il micromanipolatore attraverso un computer, ad esempio, Windows Hyperterminal, per raggiungere le procedure di micromanipolazione coerente.
- Spostare il microago, una distanza specifica (tipicamente 10 o 20 micron) verso la periferia cellulare a 1 micron / sec e contemporaneamente raccogliere immagini a contrasto di fase a fluorescenza e ogni 10secondi. Come il microago si sta muovendo per una distanza specifica verso la periferia cellulare. Con i parametri scelti, come il microago si sta muovendo per una distanza specifica verso la periferia cellulare, questo corrisponde a 2-3 frame durante il processo di manipolazione.
- Infine acquisire immagini supplementari dopo il microago viene rimosso dal citoscheletro.
Analisi
- Mappe di spostamento sono calcolati utilizzando una custom-script di MATLAB scritto sulla base di caratteristiche di tracciamento fluorescente del nucleo e nel citoplasma. (Lo script MATLAB è disponibile presso il laboratorio Lammerding su richiesta). Il programma utilizza un normalizzato algoritmo di cross-correlazione tra le regioni immagine di piccole dimensioni (circa 10 micron x 10 micron di dimensione e distanziati 5 micron a parte) in cornici immagini successive. Per ogni centro di regione, lo spostamento nelle direzioni x e y le direzioni vengono calcolate come il passaggio tra la posizione originale e la posizione recentemente identificati e visualizzati come un vettore di spostamento e memorizzati come valori numerici. Dalle mappe di spostamento, spostamenti di media nelle regioni predefinite possono essere calcolati. Da notare che gli spostamenti del citoscheletro si basano sul canale di fluorescenza per i marcatori del citoscheletro (ad esempio, MitoTracker mitocondriale macchia), mentre il nucleare spostamenti vengono calcolati dal canale fluorescenza corrispondente al segnale di Hoechst 33342. Per la nostra applicazione, abbiamo regolarmente esaminare le seguenti regioni: (i) ceppo del citoscheletro al sito di applicazione ceppo, cioè, il sito di inserzione microago, (ii) ceppo nucleare in una regione all'interno del nucleo verso il sito di applicazione ceppo, (iii) nucleare tensione in una regione nucleare di distanza dal sito di applicazione, e (iv) ceppo del citoscheletro in una regione citoplasmatica attraverso il nucleo. Inoltre, si può anche misurare direttamente allungamento nucleare da contrasto di fase o Hoechst 33342 sequenze di immagini a fluorescenza. In questo caso, applicata ceppo nucleare è calcolato dividendo l'allungamento nucleare (ΔL = L - L 0) per la lunghezza iniziale L 0, dove L è la lunghezza finale del nucleo, alla fine di applicazione dello sforzo e L 0 è il lunghezza iniziale del nucleo. Per le celle con un intatto nucleo-accoppiamento del citoscheletro, il nucleo si allungano verso il sito di applicazione dello sforzo. Al contrario, nelle cellule in cui si interrompe nucleo-citoscheletro accoppiamento, cioè, le forze sono trasmesse in modo meno efficiente tra il citoscheletro e il nucleo, il nucleo si prevede di allungare notevolmente inferiore in direzione del sito di applicazione dello sforzo. Così, diminuzione delle deformazioni nucleari in risposta alla applicazione dello sforzo del citoscheletro implica una (parziale) disaccoppiamento tra il nucleo e citoscheletro.
3. Rappresentante dei risultati:
Substrato ceppo applicazione
Abbiamo acquisito le immagini prima, durante e dopo l'applicazione dello sforzo di fibroblasti embrionali di topo da eterozigoti ed omozigoti lamina A / C-deficienti (LMNA + / - e LMNA - / -), e la wild-type (LMNA + / +) topo e successivamente calcolata la tensione normalizzata nucleare per ogni cella. Dopo l'analisi, i nuclei vengono convalidate e le cellule che diventano danneggiati o ritirare durante l'applicazione ceppo sono esclusi dall'analisi. La figura 1A mostra nuclei di tre cellule che sono validi, mentre Figura 1B mostra le celle che devono essere esclusi dall'analisi. Normalizzato i dati deformazione nucleare sono in pool di almeno tre esperimenti indipendenti (ciascuno contenente le misure da nuclei ~ 5-10) e confrontato con altre cellule o gruppi di trattamento con l'analisi statistica. Aumento della pressione normalizzata nucleare indica ridotta rigidità nucleare, come si è visto nelle cellule con ridotta espressione delle proteine dell'involucro nucleare lamina A / C (Figura 2).
Microago manipolazione test
Per l'analisi manipolazione microago, abbiamo ripreso spostamenti nucleare e del citoscheletro durante localizzata applicazione dello sforzo del citoscheletro. Le cellule che diventano danneggiati o staccati sono esclusi dall'analisi. Per l'analisi, si misura l'ampiezza dei movimenti nucleare e del citoscheletro verso il sito applicazione della forza in camere singole, cellule aderenti. Per esempio, in figura 3, si traccia mitocondriale (marker del citoscheletro) spostamenti prima e dopo la tensione del citoscheletro e poi tracciare gli spostamenti come vettori. Ogni vettore rappresenta lo spostamento calcolato come il passaggio tra la posizione originale e la posizione di nuova identificazione. Le regioni con un'immagine a bassa intensità o consistenza insufficiente (per esempio, le regioni al di fuori della cellula) sono esclusi dall'analisi. Gli spostamenti del citoscheletro e nucleare sono poi quantificati in aree selezionate a distanze crescenti dal sito di applicazione dello sforzo (Figura 4, le aree corrispondenti al colore boXES nel riquadro). In fibroblasti embrionali di topo con intatto nucleo-citoscheletro accoppiamento, le forze sono trasmesse attraverso le cellule intere, con conseguente indotto deformazioni nucleare e del citoscheletro che lentamente dissipare distanza dal sito di applicazione ceppo (Figura 4). Al contrario, i fibroblasti con disturbato nucleo-citoscheletro di accoppiamento (o alterati organizzazione del citoscheletro) display spostamenti localizzati vicino al sito di applicazione, come mostrato in figura 4 e solo deformazioni indotte poco più lontano. Comparabile applicazione dello sforzo del citoscheletro nel punto di inserimento microago (scatola arancione) si osserva per entrambi i fibroblasti di controllo (mCherry da sola) e fibroblasti con un nucleo perturbato-citoscheletro accoppiamento (DN KASH). Tuttavia, gli spostamenti indotti nucleare e del citoscheletro (blu, giallo, rosso e scatole) in altre regioni sono risultati significativamente più piccolo nel fibroblasti con interrotto nucleo-citoscheletro accoppiamento (DN KASH) rispetto alle cellule di controllo (solo mCherry) (Figura 4). Così, diminuzione spostamenti del citoscheletro e nucleare di distanza dal sito di applicazione dello sforzo, indica che la trasmissione della forza tra il citoscheletro e nucleo è stato disturbato.
È importante sottolineare che abbiamo anche convalidato che i mitocondri sono adatti marcatore del citoscheletro, effettuando la manipolazione microago sui fibroblasti embrionali di topo trasfettati con GFP-o mCherry actina e GFP-vimentina e fluorescente con Mitotracker verde o rosso. Mappe di spostamento del citoscheletro sono stati calcolati in maniera indipendente dal segnale fluorescente dei mitocondri e del citoscheletro di actina e vimentina. Lo spostamento medio assoluto è stata calcolata per quattro regioni distinte del citoscheletro a distanze crescenti di distanza dal sito di applicazione ceppo. I valori di pendenza e R-quadro sono stati calcolati dalla regressione lineare tra le misure ottenute dai mitocondri e da actina e vimentina, rispettivamente. Per actina, la pendenza è stato di 0,99 e il valore di R 2 è stato 0,986; per la vimentina, la pendenza è stato di 1,04 e il valore di R2 è stato 0,971, confermando che spostamenti mitocondriale servire come indicatori affidabili per le deformazioni del citoscheletro.
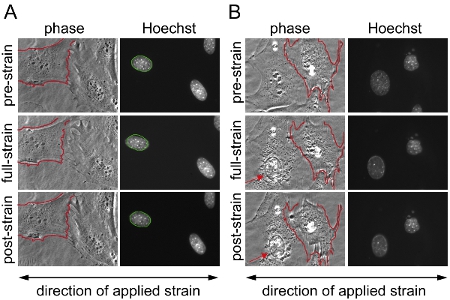
Figura 1. Ceppo applicazione substrato su fibroblasti embrionali di topo (MEF). Fibroblasti embrionali di topo si sviluppa su due aree distinte sulla membrana di silicio sono stati ripreso con contrasto di fase e microscopia a fluorescenza prima, durante e dopo l'applicazione del 20% di deformazione monoassiale. (A) Esempio di un esperimento di successo con nuclei valido dalle cellule che sopravvissero alla applicazione dello sforzo, senza alcun danno o distacco e (B) esempio di cellule che ritraggono / parzialmente staccata durante l'applicazione ceppo, i risultati dalle cellule rappresentato in (B) sono esclusi dall'analisi. In (B), la cella a sinistra mostra segni di danneggiamento e collasso del citoscheletro nucleare (freccia), mentre la cella a destra si stacca in parte e si ritrae durante l'applicazione dello sforzo. Questo può essere un'indicazione di applicazione dello sforzo eccessivo. Per una migliore confronto, in (A) e (B) il confine di una delle membrane cellulari non deformato è delineata in rosso e sovrapposta alla stessa cella durante e dopo l'applicazione dello sforzo. In (A) al confine del nucleo non deformato è delineata in verde e sovrapposta al nucleo stesso durante e dopo l'applicazione dello sforzo.
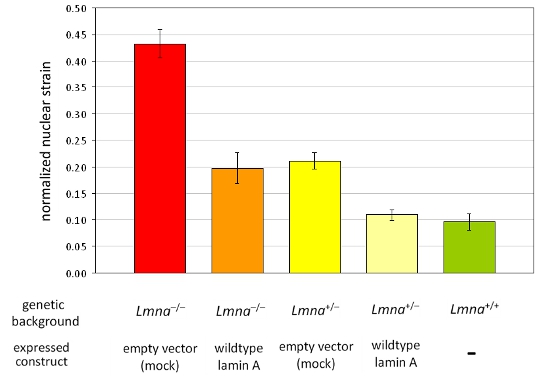
Figura 2. Analisi dei normalizzato ceppo nucleare in un pannello di diverse linee di cellule MEF MEF del LMNA -. / - E LMNA + / - background genetico ectopica che ha espresso un vettore vuoto o wild-type lamina A sono stati analizzati. In confronto al MEF da wild-type fratellini (LMNA + / +), perdita di lamina A / C espressione risultati in diminuzione rigidità nucleare che può essere completamente restaurato da reintroduzione di wild-type lamina A. In particolare, ha ridotto la rigidità nucleare si riflette valori aumentati di normalizzati ceppo nucleare. Le barre di errore rappresentano gli errori standard.

Figura 3. Test per misurare la manipolazione microago trasmissione intracellulare vigore. Contrasto di fase (A, B) e di fluorescenza (C, D) le immagini di un fibroblasto marcato con nucleare macchia (blu) e MitoTracker mitocondriale macchia (verde). Un microago è stato inserito nel citoscheletro ad una distanza definita dal nucleo (A e C) e successivamente spostato verso la periferia delle cellule (B, D). Spostamenti citoscheletro e nucleare sono stati quantificati tenendo traccia nucleo fluorescente e mitocondri utilizzando un custom-written cross-correlazione algoritmo. (E) Mappa di spostamento della finale del citoscheletro (verde) deformazioni calcolate da serie di immagini a fluorescenza; lunghezza della freccia è ingrandita di 2x per una migliore visilità. Barre di scala, a 10 micron.
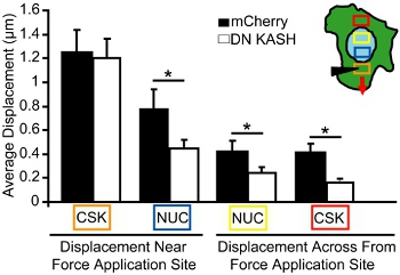
Figura 4. Analisi di trasmissione della forza durante la manipolazione intracellulare microago. Indotto spostamenti del citoscheletro e nucleare durante la manipolazione microago, misurata nelle aree corrispondenti alle caselle colorate (riquadro in A). L'Orange Box è il sito di applicazione dello sforzo. Nonostante applicazione dello sforzo simile nel citoscheletro (scatola arancione), ha indotto spostamenti nucleare e del citoscheletro (blu, giallo, rosso e scatole) erano significativamente minori nei fibroblasti embrionali di topo che, con un nucleo perturbato-citoscheletro accoppiamento (DN KASH) rispetto al controllo ( mCherry solo) delle cellule.
Access restricted. Please log in or start a trial to view this content.
Discussione
Substrato ceppo test
Applicazione ceppo è stato utilizzato con successo da noi e altri gruppi per lo studio delle deformazioni indotte nucleare in cellule sottoposte a stress meccanici e di indagare l'apporto di specifiche proteine dell'involucro nucleare alla rigidità nucleare. 4-8 Il vantaggio di questa tecnica è che le sonde proprietà meccaniche nuclei di vivere nel loro ambiente normale cellulare e del citoscheletro e che l'applicazione dello sforzo substrat...
Access restricted. Please log in or start a trial to view this content.
Divulgazioni
Nessun conflitto di interessi dichiarati.
Riconoscimenti
Questo lavoro è stato sostenuto dal National Institutes of Health (R01 e R01 HL082792 NS059348) e il Brigham and Women Hospital Cardiovascolare Award Leadership Group.
Access restricted. Please log in or start a trial to view this content.
Materiali
| Name | Company | Catalog Number | Comments |
| Nome del reagente | Azienda | Numero di catalogo | |
| Fibronectina | Millipore | FC010 | |
| MitoTracker FM Rosso e Verde FM | Invitrogen | M22425 e M-7514 | |
| Hoechst 33342 | Invitrogen | H3570 | |
| Sale di Hank Buffered Saline | Invitrogen | 14185 | |
| Fenolo libero, DMEM | Invitrogen | 21063 | |
| Siero fetale bovino | Aleken Biologicals | FBSS500 | |
| Penicillina / streptomicina | Sigma | P0781-100ML | |
| Vetro borosilicato con filamento | Sutter strumento | BF100-78-10 | |
| Lucido / lucido non armato fogli di silicone, 0.005 " | Specialità Manufacturing Inc. | ||
| Fosfato Dulbecco Buffered Saline | Invitrogen | 14200 | |
| 35 millimetri dal fondo di vetro piatti cultura (FluoroDish) | Mondo Strumenti di precisione, INC | FD35-100 | |
| Braycote 804 vuoto Grasso | Spi rifornimenti | 05133A-AB |
Riferimenti
- Friedl, P., Wolf, K., Lammerding, J. Nuclear mechanics during cell migration. Curr Opin Cell Biol. 23, 55-64 (2011).
- Mejat, A., Misteli, T. LINC complexes in health and disease. Nucleus. 1, 40-52 (2010).
- Worman, H. J., Fong, L. G., Muchir, A., Young, S. G. Laminopathies and the long strange trip from basic cell biology to therapy. J Clin Invest. 119, 1825-1836 (2009).
- Caille, N., Tardy, Y., Meister, J. J. Assessment of strain field in endothelial cells subjected to uniaxial deformation of their substrate. Ann Biomed Eng. 26, 409-416 (1998).
- Lammerding, J. Lamins A and C but not lamin B1 regulate nuclear mechanics. J Biol Chem. 281, 25768-25780 (2006).
- Lammerding, J. Abnormal nuclear shape and impaired mechanotransduction in emerin-deficient cells. J Cell Biol. 170, 781-791 (2005).
- Lammerding, J. Lamin A/C deficiency causes defective nuclear mechanics and mechanotransduction. J Clin Invest. 113, 370-378 (2004).
- Verstraeten, V. L., Ji, J. Y., Cummings, K. S., Lee, R. T., Lammerding, J. Increased mechanosensitivity and nuclear stiffness in Hutchinson-Gilford progeria cells: effects of farnesyltransferase inhibitors. Aging Cell. 7, 383-393 (2008).
- Brooks, S. V., Zerba, E., Faulkner, J. A. Injury to muscle fibres after single stretches of passive and maximally stimulated muscles in mice. J Physiol. 488, 459-469 (1995).
- Maniotis, A. J., Chen, C. S., Ingber, D. E. Demonstration of mechanical connections between integrins, cytoskeletal filaments, and nucleoplasm that stabilize nuclear structure. Proc Natl Acad Sci. 94, 849-854 (1997).
Access restricted. Please log in or start a trial to view this content.
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon