Method Article
Sesso Dimorfismo muscolo scheletrico da Proteomica
In questo articolo
Riepilogo
Un straight-forward insieme di metodi per isolare e determinare l'identità delle proteine più abbondanti espresse nel muscolo scheletrico. Circa 800 punti sono discernimento su un gel bidimensionale da 10 mg muscolare, che ci permette per la determinazione del sesso-specifico l'espressione della proteina. Questi metodi forniscono risultati equivalenti nella maggior parte dei tessuti.
Abstract
Lordo in contrazione del muscolo scheletrico è principalmente determinato da un numero relativamente piccolo di proteine contrattili, ma questo tessuto è anche straordinariamente adattabile a fattori ambientali 1 come l'ipertrofia da esercizi di resistenza e atrofia da disuso. Si presenta così il rimodellamento e adattamenti ai fattori di stress (calore, ischemia, metalli pesanti, ecc.) 2,3. Il danno può verificarsi al muscolo da una forza muscolare esercitando allo stesso tempo allunga, i cosiddetti 4 contrazione eccentrica. Le proteine contrattili può essere danneggiato in questi sforzi e hanno bisogno di essere riparato, degradati e / o sintetizzati, queste funzioni non fanno parte delle proteine contrattili, ma di altre proteine molto meno abbondante nella cellula. Per stabilire quali sottoinsieme di proteine è coinvolta nel miglioramento di questo tipo di danno, un proteoma globale deve essere stabilita prima dell'esercizio 5 e poi seguito successivamente l'esercizio per determinare il differenzialeespressione della proteina e, quindi, evidenziare proteine candidate negli adattamenti di danni e la sua riparazione. Inoltre, la maggior parte degli studi del muscolo scheletrico sono stati condotti sul maschio della specie e quindi non può essere rappresentativo del muscolo femminile.
In questo articolo vi presentiamo un metodo per l'estrazione di proteine riproducibile dai muscoli maschili e femminili, tra di loro di elettroforesi bidimensionale del gel seguito da digitale ad alta risoluzione di immagini 6. Questo fornisce un protocollo per i punti (e proteine successivamente identificati) che mostrano un aumento statisticamente significativo (p <0,05) due volte aumentare o diminuire, appaiono o scompaiono dallo stato di controllo. Questi vengono poi asportati, digerito con tripsina e separati da alta pressione cromatografia liquida accoppiata ad uno spettrometro di massa (LC / MS) per l'identificazione delle proteine (LC / MS / MS) 5. Questa metodologia (Figura 1) può essere utilizzato su molti tessuti con poca o nessuna modifica (fegato, cervello, sentiret, ecc.).
Protocollo
1. Preparazione del campione
- Usa fresca, tessuto flash RNAlater congelati o trattati dai topi come materiale di partenza. Blot via qualsiasi conservante in eccesso con un Kimwipe prima di procedere.
- Rimuovere tutti i tendini e la fascia intorno al muscolo e tritare il tessuto con singole lame di rasoio taglio per 2 minuti su lastre di vetro freddo in una stanza fredda (4 ° C) fino a quando il tessuto è ben tritato o in polvere. Raccogliere il campione in un pre-pesato provetta da microcentrifuga e determinare il peso del tessuto. Utilizzare non più di 10 mg di tessuto come materiale di partenza.
- Aggiungere 45 microlitri di buffer di estrazione (8 M urea, 50 mM DTT, 4% CHAPS, ampholytes vettore 0,2% 5 / 8, 0,0002% bromofenolo blu) per 1 mg di tessuto macinato, a temperatura ambiente. Se il volume è superiore a 350 microlitri, aggiungere 350 microlitri inizialmente e omogeneizzare (passo 1,4), quindi aggiungere il resto del buffer per ridurre schizzi.
- Omogeneizzare il tessuto sette volte con un pestello motorizzato (Kontes Corp) per 15 secondi a4 ° C, con due minuti di raffreddamento a ghiaccio tra ogni omogeneizzazione.
- Centrifugare i campioni a 15.600 xg per 30 minuti a 4 ° C. Salvare il surnatante e misurare il suo volume. La cella detriti pellet viene scartato.
- Precipitare le proteine del surnatante con gelida grado reagente acetone (3 volte v: rapporto v). Agitare per 15 secondi. Centrifugare per 15.600 xg per 10 secondi per formare pellet, e risospendere in tampone di estrazione con il motore Kontes e bacchette in polipropilene agitazione (Biospec Prod)
- Ripetere il passo 1,6.
- Continuare con stima concentrazione proteica dei campioni o conservarli a -20 ° C.
2. Concentrazione di proteine stima
Utilizzare il test Lowry 7 o il test BCA 8 (Pierce), lo scopo per circa 4,5 mg ml -1.
3. Isoelettrofocusing
- Rimuovere una striscia ReadyStrip IPG (11 cm, pH 5-8, Bio-Rad)da -20 ° C e lasciarlo equilibrare a temperatura ambiente per 10-15 min.
- Vortice del campione e pipetta 600-800 mcg (volume totale 200 mL) del campione di proteine in linea retta sul bordo posteriore di un canale nel cassetto reidratazione del campione, lasciando circa 1 cm ad ogni estremità.
- Con le pinzette e rimuovere la copertura in plastica della striscia di IPG - assicurarsi di gestire solo le estremità della striscia e di evitare qualsiasi contatto con il gel. Nota di base alla fine della striscia e posizionarla sul lato sinistro del vassoio. Posizionare la striscia IPG gel rivolto verso il basso sulla parte superiore del campione, evitare di intrappolare bolle sotto. Lasciare a reidratare per un'ora.
- Sovrapposizione con 2,5 ml di olio minerale (BioRad). Coprire il vassoio con il coperchio e lasciare a reidratarsi durante la notte a temperatura ambiente.
- Posto uno stoppino di carta su entrambe le estremità del canale vassoio messa a fuoco e bagnate ognuna con 10 microlitri di acqua Millipore.
- Estrarre IPG strip e tenere in verticale per circa 10 secondi lo scarico dell'olio. B asciugare la plasticaACKing con un Kimwipe.
- Posizionare il lato IPG gel striscia verso il basso e con l'estremità di base a sinistra nella barra delle applicazioni di messa a fuoco. Coprire la striscia con 2,5 ml di olio minerale.
- Collocare il vassoio nella cella PROTEAN IEF (Bio-Rad) e programmare un protocollo in 3 fasi (Tabella 1) alla temperatura della cella di default di 20 ° C, una corrente massima di 50 ìA / striscia, e nessun tempo reidratazione.
Tensione Tempo Volt-Ore Rampa Fase 1 250 20 min Lineare Fase 2 8000 2,5 ore Lineare Fase 3 8000 40.000 Rapido Totale 6,5 ore 40.000 Tabella 1. In tre fasi protocollo di cella PROTEAN IEF per 11 centimetri IPG strisce.
- Prendete le strisce IPG fuori dalla cella IEF con pinze. Asciugare il supporto di plastica con una Kimwipe e posizionare il lato gel IPG strip in un vassoio di reidratazione pulito. Si può coprire il vassoio reidratazione e avvolgerlo con pellicola trasparente e conservare a - 80 ° C o procedere.
4. Elettroforesi su gel poliacrilammide SDS
- Trasferire la striscia (verso l'alto gel) in un vassoio di 11 centimetri equilibrio.
- Aggiungere 4 ml di tampone riduzione (6 M urea, 2% SDS, 0,05 M Tris / HCl pH 8.8, 20% glicerolo, 2% DTT) sul canale ed equilibrare la striscia per 10 minuti con agitazione lieve.
- Rimuovere il tampone riduzione, aggiungere 4 ml di tampone alchilazione (6 M urea, 2% SDS, 0,05 M Tris / HCl pH 8.8, 20% glicerolo, 2.5% Iodoacetamide) ed equilibrare la striscia per 10 minuti con agitazione lieve.
- Sciacquare la striscia IPG immergendo il Stribrevemente p in 1 tampone X SDS in esecuzione (25 mM Tris, 192 mM glicina, 0,1% SDS, pH 8,2).
- Posare il lato gel striscia fino alla piastra posteriore del cm 11 Criterio prefabbricati 10,5% -14% Tris-HCl gel di poliacrilammide SDS (Bio-Rad) e spingere delicatamente in modo che rende pieno contatto con il gel SDS; accertarsi che non vi siano bolle intrappolate tra le due superfici del gel.
- Sovrapposizione la striscia IPG con 1 ml di soluzione di agarosio fuso (0,5% agarosio, 25 mM Tris, 192 mM glicina, 0,1% SDS, bromofenolo blu) e lasciare che l'agarosio solidificare per 5 minuti.
- Di carico 5 l di marker proteici 10-225 kDa (USB, P / N: 76740) nel singolo pozzo come standard di peso molecolare.
- Esegui il gel in SDS tampone (25 mM Tris, 192 mM glicina, 0,1% SDS, pH 8,2) a 200 V a temperatura ambiente o in una stanza fredda (4 ° C) utilizzando il bromofenolo migrazione fronte del colorante blu per monitorare la elettroforesi.
- Rimuovere il gel dal cast di plastica e macchie durante la notte con una leggera agitazione in Coomassie Brilliant BlueR-250 macchia (45,5% di metanolo, dH2O 45,5%, 9,0% di acido acetico, 0,25% Coomassie Brilliant Blue R-250).
- Agitare il gel in Decolorare soluzione 1 (45,5% metanolo, 45,5% dH 2 O, 9,0% di acido acetico) due volte per 3 ore ciascuna e in Decolorare 2 (5% di metanolo, 90% dH 2 O, 5% di acido acetico) durante la notte. Conservare il gel in 7% di acido acetico per l'analisi.
5. Gel di imaging e di analisi
- Catturare le immagini dei gel utilizzando il software Un programma Quantità VersaDoc che gestisce il sistema di imaging (BioRad). Ritaglia le aree dell'immagine in modo uniforme per tutti i gel nell'esperimento.
- Caricare le immagini ritagliate su PDQuest il programma 8,0 software per creare un set esperimento. Seguire la procedura guidata di configurazione delle prove per definire i parametri per la rilevazione di spot e punto di corrispondenza in tutto il gel. Ispezionare e perfezionare il punto di corrispondenza risultati manualmente, se necessario.
- Effettuare un'analisi quantitativa e statistica del gel per rilevare i punti di proteine abbinato con twdifferenze statisticamente significative o-fold o superiore di espressione tra i due gruppi gel. Crea un set di analisi di questi punti di interesse e tagliarli con l'ausilio di una taglierina posto EXQuest (BioRad) tripsina per la digestione e LC-MS-base l'identificazione delle proteine.
6. In-gel digestione trittico
Per questo passo un Pierce modificato In-Gel Tryptic Digest Kit (# 89871X, Pierce) viene utilizzato il protocollo.
- Aggiungere 200 ml di soluzione di decolorazione (25 bicarbonato di ammonio mM, 50% acetonitrile) per le spine gel. Vortex e incubare i campioni a 37 ° C per 30 minuti con agitazione. Rimuovere con cautela e scartare la soluzione.
- Ripetere il punto 6.1.
- Aggiungere 30 ml di preparati al momento tampone riduzione (50 mM Tris [2-carbossietil] fosfina, bicarbonato di ammonio 22,5 mm) per i tubi contenenti i tappi di gel e incubare a 60 ° C per 10 minuti.
- Lasciare raffreddare i campioni, poi rimuovere ed eliminare il buffer riducente.
- Aggiungere 30 ml di tampone alchilazione (100 Iodoacetamide mM, 20 mM di bicarbonato di ammonio, preparata immediatamente prima dell'uso, avvolto in un foglio di tubi. Incubare i campioni al buio a temperatura ambiente per 1 ora.
- Rimuovere e gettare il buffer alchilazione. Lavare le spine gel con l'aggiunta di 200 ml di soluzione di decolorazione ad ogni provetta. Incubare i campioni a 37 ° C per 15 minuti.
- Rimuovere e scartare la soluzione di decolorazione e ripetere il lavaggio.
- Aggiungere 50 ml di acetonitrile alle spine gel e incubare per 10 minuti a temperatura ambiente per permettere loro di ridursi a secco, poi rimuovere con attenzione il acetonitrile.
- Ripetere il passo 6,8 ancora una volta. Pezzi di gel dovrebbe apparire bianco e di piccole dimensioni.
- Lasciare pezzi gel ad asciugare in un concentratore Centrivap (Labconco, modello EX1245) per 10 minuti.
- Swell i pezzi di gel aggiungendo 10 ml di soluzione attivata tripsina (10 tripsina ng / mL, 25 mM di bicarbonato di ammonio). Incubare a temperatura ambiente per 15 minuti.
- Aggiungere 25 microlitri di buffer digestione (25 bicarbonato di ammonio mM) per i tubi. Incubare i campioni a temperatura ambiente per una notte con agitazione.
- Sonicare i campioni per 10 minuti (Branson, sonicatore modello da bagno 2510) e spin giù prima di rimuovere il surnatante in una nuova provetta. Salva il surnatante.
- Per ulteriori estrarre peptidi, aggiungere 10 ml di 0,1% di acido formico e incubare per 5 minuti a temperatura ambiente con agitazione. Sonicare i campioni per 10 minuti, raccogliere il surnatante e si combinano con surnatante salvato nel passaggio 6.13.
7. Dissalazione microscala di estratti peptide
- Rimuovere bicarbonato di ammonio e altri sali negli estratti peptide con l'aiuto di PepClean C-18 Colonne Spin (ThermoFisher) secondo le istruzioni del produttore.
- Peptidi eluire due volte con 20 microlitri acetonitrile 70%, evaporare a secco i campioni mediante centrifugazione del concentratore Centrivap per 1 ora e risospenderei peptidi in 10 microlitri di acido formico 0,1% per l'analisi MS.
8. L'identificazione delle proteine mediante HPLC-spettrometria di massa accoppiata
- Separato da 5 ml di miscela di peptidi su un cromatografo liquido ad alte prestazioni (HPLC) oltre 40 minuti, utilizzando un gradiente lineare di acetonitrile 2-50% con lo 0,2% di acido formico in una Pepswift monolitico PS-DVB colonna (Dionex) accoppiato ad un nanospray I origine su uno spettrometro di massa di ioni trappola (LCQ Deca XP Max, Thermo Fisher Scientific) ad un flusso di 400 nL / min alla punta.
- Rileva peptide MS1 segnali tra i 400 - 1400 m / z e consentono di 3 picchi più intensi di ioni ad essere isolati per MS2 sequenziamento da CID con esclusione dinamico.
- Per l'identificazione delle proteine, analizzare i risultati peptide attraverso una ricerca Sequest banca dati di riferimento del mouse (BioWorks software, Thermo-Fisher Scientific) con una modifica statica cisteina alchilati e modifiche dinamico per fosforilazione (serina, treonina, tirosina), metilazionezione (istidina), ossidazione (metionina), ADP-ribosilazione (arginina), e N-terminale acetilazione. Applicare criteri di corrispondenza conservativo (ad esempio, la tolleranza peptide impostato su 2 AMU e 1,00 tolleranza frammento, le proteine contengono almeno 2 peptidi superamento di un Xcorr vs carica dello Stato filtro [z = 1 / x = 1,5, z = 2 / x = 2.00, z = 3 / x = 2.50, z = 4 / x = 3.00] e una probabilità di p <0.05) 5 e ispezionare manualmente gli spettri MS per l'identificazione delle proteine fiducioso.
9. Rappresentante dei risultati:
Maschio e femmina non esercitati, murino muscolo scheletrico (bicipite brachiale) è stato estratto e separato in una mappa bidimensionale 5, il primo livello del muscolo proteoma comparativa (Figura 2). Una volta visualizzato usando ad alta risoluzione, immagini digitali, circa 800 spot proteici sono stati individuati in ciascuna. Utilizzando la mappa maschile proteoma come base, è chiaro che ci sono molti luoghi che cambiano in abbondanza nel proteoma femminile. Le intensità spot sono misure del cambiamento nella quantità di proteine (Figura 3). Le identità dei luoghi proteine che cambiano più di due volte (su o giù) e sono statisticamente significativi (p <0,05) con n = 5 topi, sono determinate con l'analisi della sequenza degli aminoacidi utilizzando cromatografia liquida-spettrometria di massa accoppiata. Questi risultati mostrano che le donne dimostrano una diminuzione dell'abbondanza degli enzimi del metabolismo degli zuccheri ed energia nelle enzima creatin chinasi (CK) isoforme, un diverso sistema di approvvigionamento energetico nei muscoli. Sia nell'uomo e negli animali superiori maschi visualizzare i livelli sierici di CK a riposo e dopo esercizio 9,10, ma i livelli sierici di CK non sono necessariamente correlati con la quantità di interruzione miofibrillare 11,12. Questo dimorfismo di genere nel abbondanza cellulare del CK in murino muscolo bicipite brachiale è un romanzo finding5 e può rispondere alla fisiologicamente diversi livelli sierici di CK.
Figure:
g1.jpg "/>
Figura 1. Una schematica generale del protocollo per la proteomica comparativa. Il protocollo è suddiviso in tre gruppi: preparazione del campione, l'analisi del campione e, l'identificazione delle proteine. Le estremità di ognuno di questi tre gruppi di protocolli sono ragionevoli luoghi pausa, anche se i risultati migliori sono guadagnato se il protocollo generale è svolta senza fermarsi.
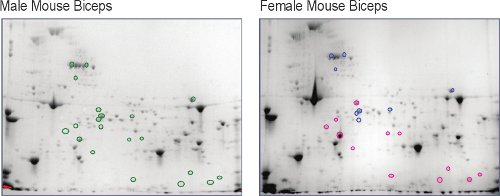
Figura 2. Confronto di femmina murini di proteine del bicipite brachiale punti rispetto ai luoghi identici nel maschio bicipite brachiale (verde circoli o). Punti che l'aumento maggiore o uguale a due volte nelle femmine sono cerchiati rosso (o) e quelli che la diminuzione minore o uguale a due volte sono cerchiati blu (o).

Figura 3 Analisi Spot Gel Intensity:. Asse X, femminapre-esercizio (controllo, n = 5); asse Y, singolo femminile attacco 0 gruppo di punti di tempo h (n = 5). Retta di regressione: coefficiente di correlazione = 0,919; pendenza = 0,976; intercettare = -0,0293. Punti al di sopra della linea rossa e sotto la linea blu cambiare più di + / - due volte.
Discussione
LC / MS metodo proteomica qui presentato è un protocollo più affidabile e riproducibile per una rapida analisi del primo livello del proteoma del muscolo scheletrico. Esso permette un confronto ragionevole espediente della specificità di genere. Proteine a bassa abbondanza richiederebbe un frazionamento del campione di muscolo di rimuovere il maggior numero di proteine contrattili possibile, aumentando così le proteine poco abbondanti. Sartoria del profilo proteomico può essere realizzato con strisce di gamma diversa di pH IPG e si alternano gel gradiente percentuale se lo si desidera. Macchie fluorescenti più sensibili esistono, ma si consiglia ad ogni laboratorio di determinare la linearità di queste macchie nel vostro sistema. Per un'analisi più rigorosa di espressione della proteina del sistema DIGE (bidimensionale In-gel elettroforesi sistema, GE Healthcare Life Sciences) può essere utilizzata, ma questo aggiunge un livello di complessità della metodologia. Inoltre, il protocollo qui descritto può essere utilizzato con la maggior parte aniMal tessuti per i quali un genoma è stato sequenziato, così come il sequenziamento de novo di una proteina da qualsiasi fonte, a condizione che esso possa essere risolto su un gel bidimensionale, in quanto si sovrappongono frammenti enzimatici può essere generato usando enzimi di elevata purezza, oltre di tripsina. Campioni provenienti da fonti di altri tessuti potrebbe richiedere alcune modifiche nella metodologia di estrazione, soprattutto se in cerca di proteine di membrana idrofobica e, comunque, abbiamo avuto buoni risultati con questo protocollo con cardiaco, fegato, reni e tessuti cerebrali. Altre modificazioni post-traduzionali possono essere rilevati modificando la massa spettro criteri di ricerca del database. In sintesi, abbiamo presentato un protocollo abbastanza dettagliata profilazione iniziale per l'analisi del proteoma.
Divulgazioni
Nessun conflitto di interessi dichiarati.
Riconoscimenti
Ringraziamo Yutian Gan per l'uso della figura 3. Questo lavoro è stato sostenuto dal National Science Foundation 0420971; la Smith College Blakeslee, Wilens e fondi Holmes, e l'Howard Hughes Medical Institute.
Materiali
| Name | Company | Catalog Number | Comments | |
| Nome del reagente | Tipo | Azienda | Numero di catalogo | Commenti |
|---|---|---|---|---|
| PepClean C-18 Spin Colonne | Di consumo | ThermoFisher Scientifico | PI-89870 | |
| Monolitico Pepswift colonna (100um x 5 cm) | Di consumo | Dionex | 162348 | |
| Criterio pre-cast 10,5% -14% Tris-HCl SDS poliacrilammide gel | Di consumo | BioRad | 345-0106 | |
| Readystrip strisce IPG | Di consumo | BioRad | Variare del pH gamme | |
| L'acido acetico | Reagente | Fisher Scientific | A465-1 | |
| Acetone | Reagente | Pharmco | 329000 | |
| Acetonitrile | Reagente | Fisher Scientific | A955 | |
| Agarose | Reagente | BioRad | 163-2111 | Copertura soluzione |
| Bicarbonato di ammonio | Reagente | Fluka | 40867 | |
| Bromofenolo blu | Reagente | Fisher Scientific | BP114 | |
| Bio-Lyte Ampholytes | Reagente | BioRad | Variare del pH gamme | |
| CHAPS | Reagente | USB | 13361 | |
| Commassie blu R-250 | Reagente | ThermoFisher Scientifico | 20278 | |
| Ditiotreitolo (DTT) | Reagente | USB | 15395 | |
| Acido formico | Reagente | ThermoFisher Scientifico | 28905 | |
| Glicerina | Reagente | Sigma-Aldrich | G6279 | |
| Glicina | Reagente | USB | 16407 | |
| Iodoacetamide | Reagente | Sigma-Aldrich | I6125 | |
| Metanolo | Reagente | Fisher Scientific | A452 | |
| Olio minerale | Reagente | BioRad | 163-2129 | |
| Sodio dodecil solfato | Reagente | Sigma-Aldrich | L6026 | |
| Tris di base | Reagente | Sigma-Aldrich | 93349 | |
| Tris [2-carboexyethyl] fosfina | Reagente | ThermoFisher Scientifico | 77720 | |
| Tripsina Endoproteinase, TPCK trattati, MS grado | Reagente | Perforare | 90055 | modificato |
| Urea | Reagente | USB | 75826 | |
| Acqua | Reagente | Burdick & Jackson | 365 | |
| Centrivap concentratore | TOOL | Labconco | ||
| Exquest posto taglierina | Strumento | BioRad | ||
| LCQ Deca XP Max ioni trappola spettrometro di massa | Strumento | ThermoFisher Scientifico | ||
| Motorizzato pestello | Strumento | Kontes Corp | ||
| Polipropilene mescolando | Strumento | Biospec Prod | ||
| aste | ||||
| PROTEAN IEF cellulare | Strumento | BioRad | ||
| Ultrasuoni | Strumento | Branson | ||
| Geometra Inoltre HPLC sistema | Strumento | ThermoFisher Scientifico | ||
| VersaDoc immagini sistema | Strumento | BioRad |
Riferimenti
- Fehrenbach, E., Mooren, F. C., Völker, K. . Molecular and Cellular Exercise Physiology. , 199-217 (2005).
- Mooren, F. C., Mooren, F. C., Völker, K. The Cell. Molecular and Cellular Exercise Physiology. , 3-18 (2005).
- Thompson, H. S., Clarkson, P. M., Scordilis, S. P. The repeated bout effect and heat shock proteins: intra-muscular HSP27 and HSP70 expression following two bouts of eccentric exercise in humans. Acta. Physiol. Scand. 174, 47-56 (2002).
- McHugh, M. P. Recent advances in the understanding of the repeated bout effect: the protective effect against muscle damage from a single bout of eccentric exercise. Scand. J. Med. Sci. Sports. 13, 88-97 (2003).
- Metskas, L. A., Kulp, M., Scordilis, S. P. Gender Dimorphism in the Exercise-Naïve Murine Skeletal Muscle Proteome. Cell Molec. Biol. Lett. 15, 507-516 (2010).
- Lopez, J. L. Two-dimensional electrophoresis in proteome expression analysis. J. Chromatogr. B. 849, 190-202 (2007).
- Lowry, O. H., Rosenburg, N. J., Farr, A. L., Randall, R. J. Protein measurement with the Folin phenol reagent. J. Biol. Chem. 193, 265-275 (1951).
- Smith, P. K., Krohn, R. I., Hermanson, G. T., Mallia, A. K., Gartner, F. H., Provenzano, M. D., Fujimoto, E. K., Goeke, N. M., Olson, B. J., Klenk, D. C. Measurement of Protein Using Bicinchoninic Acid. Anal. Biochem. 150, 76-85 (1985).
- Norton, J. P., Clarkson, P. M., Graves, J. E., Litchfield, P. L., Kirwan, J. Serum creatine kinase activity and body composition in males and females. Hum. Biol. 57, 591-598 (1985).
- Amelink, G. J., Kamp, H. H., Bär, P. R. Creatine kinase isoenzyme profiles after exercise in the rat: sex-linked differences in leakage of CK-MM. Pflügers. Arch. 412, 417-421 (1988).
- Kendall, B., Eston, R. Exercise-induced muscle damage and the potential protective role of estrogen. Sports Med. 32 (2), 103-123 (2002).
- Tiidus, P. M. Can oestrogen influence skeletal muscle damage, inflammation, and repair?. Br. J. Sports Med. 39, 251-253 (2005).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneEsplora altri articoli
This article has been published
Video Coming Soon