È necessario avere un abbonamento a JoVE per visualizzare questo. Accedi o inizia la tua prova gratuita.
Method Article
Anticorpo Microarray chimicamente bloccato per Multiplexed high-throughput Profiling di glicosilazione delle proteine specifico in campioni complessi
In questo articolo
Riepilogo
In questo studio, si descrive un protocollo migliorato per un multiplato ad alta resa microarray anticorpo con metodo di rilevazione lectina che può essere utilizzato in profili glicosilazione di proteine specifiche. Questo protocollo offre nuovi reagenti affidabili e riduce sensibilmente i tempi, i costi e alle attrezzature di laboratorio rispetto alla procedura precedente.
Abstract
In this study, we describe an effective protocol for use in a multiplexed high-throughput antibody microarray with glycan binding protein detection that allows for the glycosylation profiling of specific proteins. Glycosylation of proteins is the most prevalent post-translational modification found on proteins, and leads diversified modifications of the physical, chemical, and biological properties of proteins. Because the glycosylation machinery is particularly susceptible to disease progression and malignant transformation, aberrant glycosylation has been recognized as early detection biomarkers for cancer and other diseases. However, current methods to study protein glycosylation typically are too complicated or expensive for use in most normal laboratory or clinical settings and a more practical method to study protein glycosylation is needed. The new protocol described in this study makes use of a chemically blocked antibody microarray with glycan-binding protein (GBP) detection and significantly reduces the time, cost, and lab equipment requirements needed to study protein glycosylation. In this method, multiple immobilized glycoprotein-specific antibodies are printed directly onto the microarray slides and the N-glycans on the antibodies are blocked. The blocked, immobilized glycoprotein-specific antibodies are able to capture and isolate glycoproteins from a complex sample that is applied directly onto the microarray slides. Glycan detection then can be performed by the application of biotinylated lectins and other GBPs to the microarray slide, while binding levels can be determined using Dylight 549-Streptavidin. Through the use of an antibody panel and probing with multiple biotinylated lectins, this method allows for an effective glycosylation profile of the different proteins found in a given human or animal sample to be developed.
Introduction
Glycosylation of protein, which is the most ubiquitous post-translational modification on proteins, modifies the physical, chemical, and biological properties of a protein, and plays a fundamental role in various biological processes1-6. Because the glycosylation machinery is particularly susceptible to disease progression and malignant transformation, aberrant glycosylation has been recognized as early detection biomarkers for cancer and other diseases 7-12. In fact, most current cancer biomarkers, such as the L3 fraction of α-1 fetoprotein (AFP) for hepatocellular carcinoma 13-15, and CA199 for pancreatic cancer 16, 17 are all aberrant glycan moieties on glycoproteins. However, methods to study protein glycosylation have been complicated, and not suitable for routine laboratory and clinical settings. Chen et al. has recently invented a chemically blocked antibody microarray with a glycan-binding protein (GBP) detection method for high-throughput and multiplexed profile glycosylation of native glycoproteins in a complex sample 18. In this affinity based microarray method, multiple immobilized glycoprotein-specific antibodies capture and isolate glycoproteins from the complex mixture directly on the microarray slide, and the glycans on each individual captured protein are measured by GBPs. Because all normal antibodies contain N-glycans which could be recognized by most GBPs, the critical step of this method is to chemically block the glycans on the antibodies from binding to GBP. In the procedure, the cis-diol groups of the glycans on the antibodies were first oxidized to aldehyde groups by using NaIO4 in sodium acetate buffer avoiding light. The aldehyde groups were then conjugated to the hydrazide group of a cross-linker, 4-(4-N-MaleimidoPhenyl)butyric acid Hydrazide HCl (MPBH), followed by the conjugation of a dipeptide, Cys-Gly, to the maleimide group of the MPBH. Thus, the cis-diol groups on glycans of antibodies were converted into bulky none hydroxyl groups, which hindered the lectins and other GBPs bindings to the capture antibodies. This blocking procedure makes the GBPs and lectins bind only to the glycans of captured proteins. After this chemically blocking, serum samples were incubated with the antibody microarray, followed by the glycans detection by using different biotinylated lectins and GBPs, and visualized with Cy3-streptavidin. The parallel use of an antibody panel and multiple lectin probing provides discrete glycosylation profiles of multiple proteins in a given sample 18-20. This method has been used successfully in multiple different labs 1, 7, 13, 19-31. However, stability of MPBH and Cys-Gly, complicated and extended procedure in this method affect the reproducibility, effectiveness and efficiency of the method. In this new protocol, we replaced both MPBH and Cys-Gly with one much more stable reagent glutamic acid hydrazide (Glu-hydrazide), which significantly improved the reproducibility of the method, simplified and shorten the whole procedure so that the it can be completed within one working day. In this new protocol, we describe the detailed procedure of the protocol which can be readily adopted by normal labs for routine protein glycosylation study and techniques which are necessary to obtain reproducible and repeatable results.
Protocollo
1. Stampare un Microarray dell'anticorpo per il test
- Diluire tutti gli anticorpi a 0,5 mg / ml in soluzione salina tampone fosfato, pH 7,2 (PBS).
- Aliquotare 40 pl di ciascun anticorpo nella piastra a 384 pozzetti sorgente.
- Caricare la piastra a 384 pozzetti sorgente sul micro-distributore sciFLEXARRAYER Scienion.
- Caricare 20 vetrini microarray PATH sul micro-distributore come bersaglio.
- Impostare la micro-distributore per stampare 48 sottoarray identici, in cui sono macchiati 27 anticorpi e proteine di controllo in triplice copia in una configurazione 9x9 (Figura 1E, 1F).
- Avviare il micro-distributore per stampare le diapositive anticorpi microarray.
- Raccogliere i vetrini microarray anticorpi, e conservarli in cassette diapositive con essiccante. Aspirare sigillare la cassetta in un sacchetto di plastica sottovuoto utilizzando saldatrice (Foodsaver).
- Memorizzare i vetrini microarray sigillati a 4 ° C in frigorifero.
2. Chimicamente Bloccare il Microarray anticorpo per prevenire GBPIl legame con i anticorpi di cattura
Il dosaggio microarray inizia una volta che i vetrini microarray sono chimicamente bloccati e dura per circa 8 ore. Una volta avviato il saggio microarray deve essere completata (Procedura 2 a 8).
- Prendere il microarray scorre fuori dal frigorifero, ed equilibrare a temperatura ambiente per 30 minuti.
- Rimuovere il vetrino dal contenitore e brevemente sciacquare in soluzione salina tampone fosfato pH 7,2 con 0,1% Tween 20 (PBST0.1) una volta in una diapositiva lavabo, e poi in 15 mM tampone pH sodio acetato 5,0% di Tween 0,1 (CBT0 .1) in modo sequenziale. Incubare i vetrini in CBT0.1 per 10 minuti in vasca scorrevole lavaggio.
- Preparare fresca 150 mM Naio 4 in 15 mM tampone di acetato di sodio pH 5.0 (CB), e tenerlo in in una diapositiva lavandino in frigorifero, evitando la luce prima dell'uso.
- Rimuovere il vetrino dalla CB, e metterlo nella vasca contenente fresca Naio 4 con il lato anticorporivolto verso l'alto. Coprire il bacino con foglio di alluminio per evitare la luce, e incubare il bacino vetrino per 2 ore con delicata agitazione a 4 ° C in frigorifero.
- Preparare 300 ml di 10 mM di acido glutammico idrazide (il blocco) in CB.
- Rimuovere il vetrino dal bacino, e brevemente sciacquarlo in CB 3 volte per 5 minuti ogni volta in bacino diapositive lavaggio.
- Incubare le diapositive il blocco in una vasca di lavaggio per 2 ore a temperatura ambiente con delicata agitazione.
- Rimuovere i vetrini dal bacino, e lavarli con PBST0.1 per 3 minuti.
3. Blocco non specifici Associazioni per la Microarray con albumina sierica bovina (BSA)
- Preparare 300 ml di 1% BSA in tampone fosfato pH 7,2 con soluzione salina 0,5% Tween (PBST0.5) in un bacino diapositiva lavaggio, e incubare il vetrino microarray nel bacino per 1 ora a temperatura ambiente agitando delicatamente.
- Sciacquare i vetrini in PBST0.1 tre volte per 3 minuti ogni volta.
- Mettere il vetrinosu un rack scorrevole, e centrifugare a 1200 xg su una centrifugazione per 2 minuti per asciugare il vetrino microarray.
4. Imprint Griglia Cera sul vetrino Microarray per separare ogni sottomatrice
- Pre-riscaldamento del imprinter cera a 70 ° C per 5 minuti.
- Caricare il vetrino microarray bloccato nella imprinter cera con il lato di anticorpi rivolto verso la cera. Estrarre delicatamente la maniglia a cera imprimere sul vetrino in modo uniforme.
5. Applicare campioni di siero sul vetrino Microarray
- Durante la fase 2.4, preparare i campioni di siero per entrambi i test profiling glico in un campione (5.1.1), o singola glico misura epiptope tra campioni multipli (5.1.2).
- In un esperimento per le profilature glycan di più glicoproteine del siero in un unico campione di siero utilizzando più Sterline (vedi esperimento campione 1), uno i campioni di siero sarà applicato su tutti i sottoarray. In questo caso, 40 microlitri ampio siero viene diluito in 360 microlitri di PBS contenente 0,1%Tween-20, 0,1% Brij 35, 100 pg / ml di IgG di topo, 100 pg / ml di IgG di ratto, 100 pg / ml di IgG di coniglio, 100 pg / ml di IgG di capra e 100 pg / ml di asino IgG. Questo volume è sufficiente per l'applicazione di 6 microlitri di una soluzione diluita di siero su ogni sottomatrice.
- In un esperimento per la misurazione uno glicani su più proteine del siero tra più campioni di siero utilizzando uno rilevazioni GBP (vedere esperimento campione 2). In questo caso, 1 pl ampio siero viene diluito in 9 pl di PBS contenente 0,1% Tween-20, 0,1% Brij 35, 100 pg / ml di IgG di topo, 100 pg / ml di IgG di ratto, 100 pg / ml di IgG di coniglio , 100 pg / ml di IgG di capra e 100 pg / mL di asino IgG. Questo volume è sufficiente per l'applicazione di 6 microlitri di una soluzione diluita di siero su ogni sottomatrice.
- Dopo l'impronta di cera al punto 4, ad applicare attentamente 6 microlitri di campione diluito o di campioni di controllo (PBST0.1) ad ogni sottomatrice della diapositiva. Incubare il vetrino in una cassetta umidificato con tovaglioli di carta bagnati a temperatura ambienteper 1 ora.
- Sciacquare il vetrino con PBST0.1 tre volte per 3 minuti ogni volta.
- Asciugare il vetrino facendo girare è a 1200 xg per 2 minuti.
6. Applicare biotinilato GBP (Anticorpo o lectina Anti-glicano) nella diapositiva
- Durante la fase 2.4, preparare 10μg/ml delle lectine biotinilati / Gbps in PBST0.1.
- Nell'esperimento profiling glicano che sonda un campione con lectine multipli (campione esperimento 1), preparare 350 microlitri di lectina biotinilato che è sufficiente per tutti i sottoarray.
- In singolo glicano epitopo / biomarker di screening in campioni multipli utilizzando lectine più, preparare 10 pl di ogni lectina biotinilato che è sufficiente per un sottoarray.
- Applicare 6 microlitri della lectina diluito biotinilato (s) a ciascuna sottomatrice della slitta, e incubare nella scatola slitta umidificata con tovaglioli di carta umidi a temperatura ambiente per 1 ora.
- Sciacquare i vetrini con PBST0.1 tre volte per 3 minuti ciascuno Time.
- Asciugare il vetrino facendo girare è a 1200 xg in centrifugare per 2 minuti.
7. Applicare Dye neutravidina etichetta per il rilevamento della fluorescenza
- Preparare 350 microlitri di Dylight 549 neutravidina marcato che è sufficiente per tutti i sottoarray.
- Applicare 6 pl di Dylight 549 neutravidina etichettate su ciascun subarray, e incubare il vetrino nel cassetto scorrevole umidificato a temperatura ambiente per 1 ora.
- Sciacquare il vetrino con PBST0.1 tre volte per 3 minuti ogni volta.
- Asciugare il vetrino facendo girare l'ha a 1200 xg in centrifugare per 2 minuti.
8. Ottenere Immagine diapositiva Microarray a Scansione la diapositiva
- Eseguire la scansione della diapositiva utilizzando uno scanner a fluorescenza microarray a risoluzione di 10 micron. Le impostazioni di laser e PMT deve essere il più forte possibile, ma nessun punto di saturazione si osserva.
9. Estrazione dei dati e analisi
- Aprire l'immagine in ArrayPro 3.2.
- Impostare il modello di serie in base alla mappa matrice che mostra la posizione degli anticorpi macchie. Allineare con cura ogni cerchio modello sul punto corrispondente nell'immagine.
- Estrarre l'intensità di ogni spot in un file Excel per ulteriori analisi.
10. Risultati rappresentativi
Esempio di esperimento 1
Profiling glicosilazione delle glicoproteine sieriche più a campione del paziente carcinoma epatocellulare siero mediante microarray anticorpi chimicamente bloccato con più di rilevamento lectine.
L'obiettivo di questo esperimento è quello di esplorare il profilo individuale di 20 glicoproteine glicosilazione nel carcinoma epatocellulare (HCC) campione di siero del paziente mediante microarray anticorpi chimicamente bloccato con rilevamento lectina. Un microarray anticorpo, che contiene 48 sottoarray identici che comprendono 26 anticorpi e biotina-BSA, è stato progettato e realizzato come descritto nella Step 1. Questi anticorpi erano 26 contro 20 glicoproteine siero che identificati come promettenti valore diagnosi precoce per pazienti con carcinoma epatocellulare utilizzando Lectina immunoprecipitazione base combinato con l'identificazione di proteine spettrometria di massa 12, 32 come mostrato nella Tabella 1. Il modello e la disposizione dei punti anticorpo stampate in triplice copia in una sottomatrice rappresentante sono illustrati nella Figura 1E e 1F, rispettivamente. Due vetrini microarray identiche, non era chimicamente bloccato (Figura 1A), mentre l'altro è (figura 1B), sono stati utilizzati per effettuare lo stesso esperimento profilatura glicosilazione al fine di dimostrare l'importanza della procedura chimicamente blocco per l'analisi. Per la slitta chimicamente bloccata (figura 1B), l'esperimento avviato alla fase 2, per il none slitta chimicamente bloccato (Figura 1A), l'esperimento è iniziato dal punto 3. L'esperimento è stato portato da following tutti i passi descritti nel protocollo ad eccezione di Fase 5.1.2 e 6.1.2. Nella Fase 5,2, uno PBST0.1 campione di controllo è stato applicato su sottoarray in colonna 1 e 3, e un pool campione di HCC siero è stato applicato su sottoarray nella colonna 2 e 4, rispettivamente (Figura 1G). Questo confronto è per dimostrare l'efficacia, l'efficienza del procedimento, nonché l'affinità antigene legame degli anticorpi dopo chimicamente blocco. 22 lectine biotinilati (come mostrato in Tabella 1) che specifica per glicani differenti 18, 20 sono stati applicati su ciascuna sottomatrice come mostrato nella Figura 1G per profilatura glicosilazione. Le immagini dei chimicamente bloccati (Figura 1B) e non chimicamente bloccato (Figura 1A) microarray dopo il dosaggio glicosilazione profilatura seguendo il protocollo. Come mostrato nelle sottoarray nella colonna 1 e 3 in microarrays bloccati non chimicamente (Figura 1A e la Figura 1C), in cuisolo PBST0.1 è stata applicata, la maggior parte delle lectine destinata a catturare gli anticorpi, e ha mostrato di fondo molto elevato che paragonabili a quelle sottoarray nella colonna 2 e 4, in cui è stato applicato il campione di siero. È impossibile ottenere informazioni glicano profilo da questa diapositiva microarray. Al contrario, quando lo stesso esperimento è stato fatto su un vetrino anticorpo chimicamente bloccato microarray, i subarray nella colonna 1 e 3, su cui è stata applicata solo PBST0.1, la maggior parte delle lectine binding non mostrato o molto basso per catturare gli anticorpi, mentre antigene elevato associazioni sono mantenuti in subarray nella colonna 2 e 4, sul quale campione di siero è stato applicato (figura 1B e 1D). Questi risultati hanno mostrato la procedura chimicamente blocco è stato un passo fondamentale quindi la misura di glicani su anticorpi catturati glicoproteine. Seguendo il protocollo, profili di glicosilazione 22 glicoproteine HCC in siero può essere ottenuta.
Esperimento 2
Schermo per fucos alteratiylation su specifiche glicoproteine del siero come biomarker per cirrosi epatica discriminati e pazienti con carcinoma epatocellulare.
L'obiettivo di questo esperimento è quello di schermo per fucosylation alterato su specifiche glicoproteine del siero come biomarker che discriminano cirrosi epatica e carcinoma epatocellulare (HCC) pazienti. A differenza dell'esperimento 1, in cui è stato applicato un solo campione di siero su ogni sottoarray e sondato con lectine vari, in questo saggio, un totale di 40 campioni di siero diversi da HCC e cirrosi pazienti sono stati applicati su ciascun subarray, e sondati con una lectina (AAL ). L'analisi statistica, come il T test, ricevitore-operativa caratteristica (ROC) curve, è stato fatto per valutare le distribuzioni o la prestazione diagnostica del epiptope glicano / biomarker su ogni singola proteina in tutti i campioni di siero. Abbiamo utilizzato il microarray stesso anticorpo prodotto nell'Esperimento 1 eccetto per anti-CA19-9 e anticorpi anti-Lewis X in questo studio. Il compeiment è stata effettuata dal 2 settembre al punto 9 tranne il passaggio 5.1.1 e 6.1.1. Totale 40 campioni di siero da 20 cirrosi e 20 pazienti con carcinoma epatocellulare sono stati applicati al subarray casuale delle 48 sottoarray insieme ai campioni di controllo PBS come controllo negativo. Fucosylation di ciascuna delle proteine rilevati sono stati poi rilevato mediante biotinilato specifico lectina-fucosio. L'immagine microarray mostrato in Figura 1 ha dimostrato l'AAL lectina solo legati alle proteine plasmatiche catturati sul microarray (Figura 2D) invece di anticorpi catturati (figura 2E). Le intensità AAL vincolanti tutti gli spot sono stati poi estratti, e analizzati mediante test T e curve ROC per valutare le prestazioni del fucosylation (AAL intensità legame) di ciascuna proteina del siero del discriminazione tra HCC e gruppi cirrosi. I risultati hanno mostrato che la fucosylation di GP73 proteina ha la migliore discriminazione tra i due gruppi con p = 0,03 e area sottoCurva di curva ROC pari a 0,72. Questo esperimento dimostra questa procedura è un metodo rapido e efficiente per l'epitopo / biomarker glicano lo screening su campioni multipli all'interno proteine multiple.
| ID | Nome del reattivo | Abbreviazione | Azienda | Catalogo # |
| L1 | Biotinilato Concanavalina A | ConA | Vector Laboratories | BK-1000 |
| L2 | Biotinilato Sambucus Nigra lectina | SNA | Vector Laboratories | B-1305 |
| L3 | Biotinilato Lens Culinaris agglutinina | LCA | Vector Laboratories | BK-2000 |
| L4 | Biotinilato Ricinus Communis agglutinina I | RCA | Vector Laboratories | BK-1000 |
| L5 | Biotinilato Aleuria aurantia lectina | AAL | Vector Laboratories | B-1395 |
| L6 | Biotinilato Erythrina cristagalli lectina | ECL | Vector Laboratories | BK-3000 |
| L7 | Biotinilato Griffonia (Bandeiraea) Simplicifolia lectina II | GSL II | Vector Laboratories | BK-3000 |
| L8 | Biotinilato di Germe di Grano agglutinina | WGA | Vector Laboratories | BK-1000 |
| L9 | Biotinilato Erythroagglutinin Phaseolus vulgaris | PHA-E | Vector Laboratories | BK-2000 |
| L10 | Biotinilato Leucoagglutinin Phaseolus vulgaris | PHA-L | Vector Laboratories | BK-2000 |
| L11 | BioPeanut agglutinina tinylated | PNA | Vector Laboratories | BK-1000 |
| L12 | Biotinilato Pisum Sativum agglutinina | PSA | Vector Laboratories | BK-2000 |
| L13 | Biotinilato Dolichos biflorus agglutinina | DBA | Vector Laboratories | BK-1000 |
| L14 | Biotinilato lectina Datura Stramonium | DSL | Vector Laboratories | BK-3000 |
| L15 | Biotinilato agglutinina Sophora Japonica | SJA | Vector Laboratories | BK-2000 |
| L16 | Soia agglutinina biotinilato | SBA | Vector Laboratories | BK-1000 |
| L17 | Biotinilato Solanum tuberosum (patata) lectina | STL | Vector Laboratories | BK-3000 |
| L18 | Biotinilato Griffonia (Bandeiraea) Simplicifolia lectina I | GSL I | Vector Laboratories | BK-2000 |
| L19 | Biotinilato Vicia Villosa lectina | VVL | Vector Laboratories | BK-2000 |
| L20 | Biotinilato Lycopersicon esculentum (pomodoro) lectina | LEL | Vector Laboratories | BK-3000 |
| L21 | Biotinilato Ulex Europaeus agglutinina I | UEA I | Vector Laboratories | BK-1000 |
| L22 | Biotinilato Jacalin | JACALIN | Vector Laboratories | BK-3000 |
| A1 | Capra F (ab ') 2 Frammento anti-IgM umane, anticorpi Fc5μ | IgM | Jackson Immuno Research | 109-006-129 |
| A2 | Donkey F (ab ') 2Frag anti-IgG umane (H + L) anticorpo | AB1 | Jackson Immuno Research | 709-006-149 |
| A3 | Topo anti-IgG umane F (ab ') 2 anticorpo monoclonale | AB3 | Jackson Immuno Research | 209-005-097 |
| A4 | Capra anti-umano alfa 2 macroglobulina anticorpo policlonale | A2M | GeneTex | GTX62924 |
| A5 | Coniglio anti-umano alfa-1-antitripsina anticorpo policlonale | A1AT | Lee Biosiences | CA1T-80A |
| A6 | Topo anti-umano alfa-1-antitripsina anticorpo monoclonale | A1AT | Sigma Aldrich | SAB4200198 |
| A7 | Coniglio anti-umano alfa-1-antitripsina anticorpo policlonale | ACT | NeoMarkers | RB-367-A1 |
| A8 | Coniglio anti-umano alfa-1-antichymotrypsin anticorpo policlonale | ACT | Fisher Scientific | RB9213R7 |
| A9 | Topo anti-transferrina umana anticorpo monoclonale | Transferrina | GeneTex | GTX101035 |
| A10 | Coniglio anti-transferrina umana anticorpo policlonale | Transferrina | GeneTex | GTX77130 |
| A11 | Capra anti-umano apolipoproteina J anticorpo policlonale | ApoJ | Abcam | ab7610 |
| A12 | Topo anti-umano GP73 anticorpo monoclonale | GP73 | Abbott | 14H4-23 |
| A13 | Topo anti-umano GP73 anticorpo monoclonale | GP73 | Santa Cruz Biotechnology INC | sc-101275 |
| A14 | Coniglio anti-umano alfa-1 fetoproteina anticorpo policlonale | AFP | GenWay | GWB-41C966 |
| A15 | Murino anti-umano alfa-1 fetoproteina anticorpo monoclonale | AFP | Fitzgerald | 10-A05A |
| A16 | Topo anti-umano emopessina anticorpo monoclonale | Emopessina | Assaypro | 60.190-05.011 |
| A17 | Murino anti-umano glypican-3 (1g12) anticorpo monoclonale | GPL3 | Santa Cruz Bio | sc-65443 |
| A18 | Topo anti-umano chininogeno (LMW) anticorpo monoclonale | Chininogeno | Assaypro | 20.333-05.011 |
| A19 | Coniglio anti-umano MMP-21 anticorpo monoclonale | MMP21 | Epitomic | 1955-1 |
| A20 | Murino anti-umano CEACAM-1 anticorpo monoclonale | CEACAM | R & D Systems | MAB1180 |
| A21 | Ratto anti-umano DPPIV/CD26 anticorpo monoclonale | DPPIV | R & D Systems | MAB22441 |
| A22 | Topo anti-umano PIVKA II anticorpo monoclonale | PIVICA | Cristallo chem | 8040 |
| A23 | Topo anti-antigene carcinoembrionario | CEA | USA biologico | C1300 |
| A24 | Topo Antigen anti-cancro CA125 | CA125 | USA biologico | C0050-01D |
| A25 | Topo anti-CA19-9 Cancer antigene | CA19-9 | USA biologico | C0075-18 |
| A26 | Topo anti-Lewis x anticorpo monoclonale | Lewis x | Calbiochem | 434631 |
| bio | Biotinilato BSA (controllo positivo) | Bio | Fatto in casa | N / A |
Tabella 1. Elenco delle lectine e anticorpi utilizzati in questo protocollo.
| Nome del reattivo, s / attrezzature | Azienda | Numero di catalogo |
| Contatto micro-distributore non | BioDot Inc | sciFLEXARRAYER |
| 384 micropiastra | Pescatore | 14-230-243 |
| Foodsaver | Foodsaver | V3835 |
| Ultrasottile nitrocellulosa Coate microarray scivola | Gentel | PATH |
| Presentazione Imprinter (opzionale) | Il Gel Società | WSP60-1 |
| Shaker | Pescatore | 15-453-211 |
| Centrifuga | Eppendorf | 5804 000.013 |
| Far scorrere lavandino / Slide colorazione Dish with estraibile Rack | Pescatore | 08-812 |
| Diapositiva camera di incubazione / vetrino da microscopio box | Pescatore | 03-448-5 |
| Brij 35, 30 w / v% di soluzione in acqua | Acros Organics | AC32958-0025 |
| Tween-20 | Pescatore | P337-100 |
| Periodato di sodio (NAIO 4) | Sigma | 311448 |
| L-glutammico γ-idrazide | Sigma | G-7257 |
| Acetato di sodio anidro (CH 3 COONa) | Sigma | S2889 |
| Albumina sierica bovina (BSA) | Lampire Biological Labs | 7500804 |
| Tampone fosfato salino (PBS) (10X) | Denville scientifico | CP4390-48 |
| Dylight 549 neutravidina coniugato | Thermo | 22837 |
| Inibitori della proteasi Cocktail Tablets | Roche | 4693159001 |
| ChromPure IgG umane, frammento Fc | Jackson ImmunoResearch | 009-000-008 |
| ChromPure IgG umane, molecola intera | Jackson ImmunoResearch | 009-000-003 |
| ChromPure mouse IgG, molecola intera | Jackson ImmunoResearch | 015-000-003 |
| ChromPure mouse IgG, frammento Fc | Jackson ImmunoResearch | 015-000-008 |
| ChromPure IgG di coniglio, molecola intera | Jackson ImmunoResearch | 011-000-003 |
| ChromPure Donkey IgG, molecola intera | Jackson ImmunoResearch | 017-000-003 |
| Microarray Scanner | Tecan | LS Reloaded |
Tabella 2. Listdi attrezzature e reagenti utilizzati in questo protocollo.
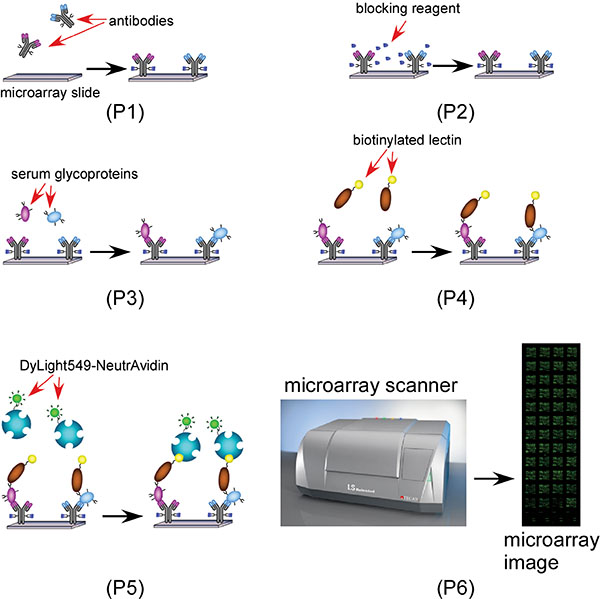
Schema 1 Uno schema che mostra la lectina anticorpo microarray basata processo di biomarker discovery glicano 1 (Step 2 a 4): Bloccare la microarray anticorpo con il blocco (Glu-idrazide) e BSA, 2 (punto 5):.. Applicare campioni di siero e la cattura glicoproteine specifiche con anticorpi specifici, 3 (Fase 6): si applicano lectina biotinilato (s), 4 (Fase 7): la sonda AAL biotinilato con Dylight 549 neutravidina etichettato per microarray imaging.
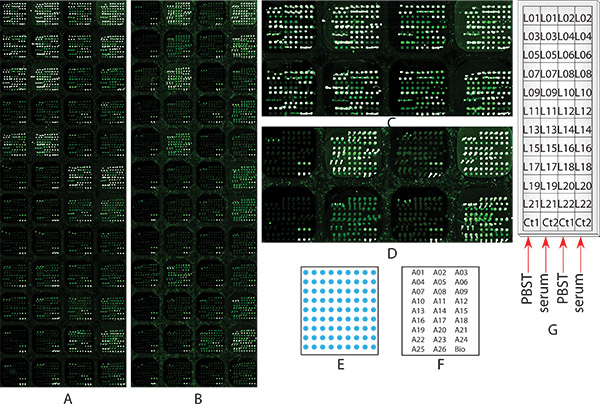
Figura 1. Immagini Microarray del profiling Sample glicosilazione Esperimento 1 di molteplici glicoproteine sieriche nel campione di siero del paziente HCC mediante Chemicmicroarray anticorpo alleato bloccato con più di rilevazione lectina. Due vetrini microarray identici, (A) none chimicamente bloccato, o (B) chimicamente bloccata come descritto nel passaggio 2, sia passato attraverso tutte le fasi da 2 a 9 per la profilatura glicosilazione, così come scopo di confronto. (A) e (B) sono le immagini microarray scansione durante la fase 8 in una risoluzione di 10 micron. (C) lo zoom in immagine delle prime due righe della none chimicamente bloccato slitta microarray (A), (D) lo zoom in immagine delle prime due righe della slitta non chimicamente bloccato microarray (B)), (E) lo schema della disposizione degli anticorpi all'interno di ogni sottomatrice; (F) mappe di matrice: la posizione di ciascun anticorpo all'interno del subarray, ogni nome anticorpo rappresenta 3 punti; (G), campione di siero e la posizione lectina: un diagramma che mostra sottomatrice ciascun campione di siero e è stata applicata su lectina.

Figura 2. Immagini Microarray dellacampione esperimento 2 schermo per fucosylation alterato su specifiche glicoproteine del siero come biomarker che discriminano cirrosi epatica e pazienti con carcinoma epatocellulare. Il saggio microarray è stata eseguita come descritto nella sezione Esempio Esperimento 2. (A) L'immagine intera diapositiva della diapositiva microarray dal punto 8; (B) lo schema della disposizione degli anticorpi all'interno di ogni sottomatrice; (C) mappe matrice: la posizione di ciascun anticorpo all'interno del subarray, ogni nome rappresenta l'anticorpo 3 punti; (D) uno zoom-in immagine di un subarray che sono state incubate con siero, (E) uno zoom-in immagine di un subarray che sono stati incubati con PBS controllo.
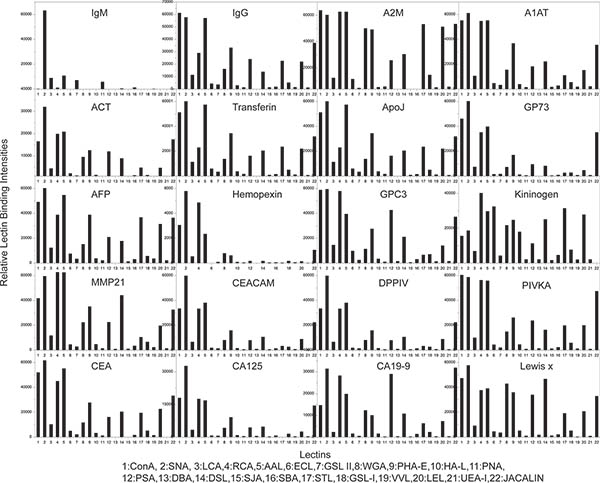
Figura 3. Risultati profiling glycan dell'esperimento campione 1. Ogni grafico a barre rappresentano lectina profilo legame (o profili glycan) di uno dei proteina testata 20. Trovati 22 lectine differenti sono stati utilizzati per analizzare i the glicano profilo di ciascuna proteina.
Discussione
1. Proteina bersaglio e selezione anticorpo di cattura
Prima del test microarray anticorpi, alcuni reagenti e materiali sono necessari per essere considerato e preparati. Per progettare un microarray per l'analisi degli anticorpi glicano o biomarker di screening glicano, un pannello di anticorpi specifici per i candidati della glicoproteina deve essere determinato secondo la letteratura o dai risultati precedenti. Questi anticorpi sono di solito acquistati da fornitori diversi, quali R &...
Divulgazioni
Gli autori non hanno nulla da rivelare.
Riconoscimenti
Questo lavoro è stato sostenuto dall'Istituto per l'epatite e la ricerca del virus.
Materiali
| Name | Company | Catalog Number | Comments | |
| ID | Nome del reattivo | Abbreviazione | Azienda | Catalogo # |
| L1 | Biotinilato Concanavalina A | ConA | Vector Laboratories | BK-1000 |
| L2 | Biotinilato Sambucus Nigra lectina | SNA | Vector Laboratories | B-1305 |
| L3 | Biotinilato Lens Culinaris agglutinina | LCA | Vector Laboratories | BK-2000 |
| L4 | Biotinilato Ricinus Communis agglutinina I | RCA | Vector Laboratories | BK-1000 |
| L5 | Biotinilato Aleuria aurantia lectina | AAL | Vector Laboratories | B-1395 |
| L6 | Biotinilato Erythrina cristagalli lectina | ECL | Vector Laboratories | BK-3000 |
| L7 | Biotinilato Griffonia (Bandeiraea) Simplicifolia lectina II | GSL II | Vector Laboratories | BK-3000 |
| L8 | Biotinilato di Germe di Grano agglutinina | WGA | Vector Laboratories | BK-1000 |
| L9 | Biotinilato Erythroagglutinin Phaseolus vulgaris | PHA-E | Vector Laboratories | BK-2000 |
| L10 | Biotinilato Leucoagglutinin Phaseolus vulgaris | PHA-L | Vector Laboratories | BK-2000 |
| L11 | Peanut agglutinina biotinilato | PNA | Vector Laboratories | BK-1000 |
| L12 | Biotinilato Pisum Sativum agglutinina | PSA | Vector Laboratories | BK-2000 |
| L13 | Biotinilato Dolichos biflorus agglutinina | DBA | Vector Laboratories | BK-1000 |
| L14 | Biotinilato lectina Datura Stramonium | DSL | Vector Laboratories | BK-3000 |
| L15 | Biotinilato agglutinina Sophora Japonica | SJA | Vector Laboratories | BK-2000 |
| L16 | Soia agglutinina biotinilato | SBA | Vector Laboratories | BK-1000 |
| L17 | Biotinilato Solanum tuberosum (patata) lectina | STL | Vector Laboratories | BK-3000 |
| L18 | Biotinilato Griffonia (Bandeiraea) Simplicifolia I lectina | GSL I | Vector Laboratories | BK-2000 |
| L19 | Biotinilato Vicia Villosa lectina | VVL | Vector Laboratories | BK-2000 |
| L20 | Biotinilato Lycopersicon esculentum (pomodoro) lectina | LEL | Vector Laboratories | BK-3000 |
| L21 | Biotinilato Ulex Europaeus agglutinina I | UEA I | Vector Laboratories | BK-1000 |
| L22 | Biotinilato Jacalin | JACALIN | Vector Laboratories | BK-3000 |
| A1 | Capra F (ab ') 2 Frammento anti-IgM umane, anticorpi Fc5μ | IgM | Jackson Immuno Research | 109-006-129 |
| A2 | Donkey F (ab ') 2 Frag anti-IgG umane (H + L) unotibody | AB1 | Jackson Immuno Research | 709-006-149 |
| A3 | Topo anti-IgG umane F (ab ') 2 anticorpo monoclonale | AB3 | Jackson Immuno Research | 209-005-097 |
| A4 | Capra anti-umano alfa 2 macroglobulina anticorpo policlonale | A2M | GeneTex | GTX62924 |
| A5 | Coniglio anti-umano alfa-1-antitripsina anticorpo policlonale | A1AT | Lee Biosiences | CA1T-80A |
| A6 | Topo anti-umano alfa-1-antitripsina anticorpo monoclonale | A1AT | Sigma Aldrich | SAB4200198 |
| A7 | Coniglio anti-umano alfa-1-antitripsina anticorpo policlonale | ACT | NeoMarkers | RB-367-A1 |
| A8 | Coniglio anti-umanoalfa-1-antichimotripsina anticorpo policlonale | ACT | Fisher Scientific | RB9213R7 |
| A9 | Topo anti-transferrina umana anticorpo monoclonale | Transferrina | GeneTex | GTX101035 |
| A10 | Coniglio anti-transferrina umana anticorpo policlonale | Transferrina | GeneTex | GTX77130 |
| A11 | Capra anti-umano apolipoproteina J anticorpo policlonale | ApoJ | Abcam | ab7610 |
| A12 | Topo anti-umano GP73 anticorpo monoclonale | GP73 | Abbott | 14H4-23 |
| A13 | Topo anti-umano GP73 anticorpo monoclonale | GP73 | Santa Cruz Biotechnology INC | sc-101275 |
| A14 | Coniglio anti-umano alfa-1 fetoprotein anticorpo policlonale | AFP | Genway | GWB-41C966 |
| A15 | Murino anti-umano alfa-1 fetoproteina anticorpo monoclonale | AFP | Fitzgerald | 10-A05A |
| A16 | Topo anti-umano emopessina anticorpo monoclonale | Emopessina | Assaypro | 60.190-05.011 |
| A17 | Murino anti-umano glypican-3 (1g12) anticorpo monoclonale | GPL3 | Santa Cruz Bio | sc-65443 |
| A18 | Topo anti-umano chininogeno (LMW) anticorpo monoclonale | Chininogeno | Assaypro | 20.333-05.011 |
| A19 | Coniglio anti-umano MMP-21 anticorpo monoclonale | MMP21 | Epitomic | 1955-1 |
| A20 | Topo anti-umano CEACAM-1 anticorpi monoclonaliy | CEACAM | R & D Systems | MAB1180 |
| A21 | Ratto anti-umano DPPIV/CD26 anticorpo monoclonale | DPPIV | R & D Systems | MAB22441 |
| A22 | Topo anti-umano PIVKA II anticorpo monoclonale | PIVICA | Cristallo chem | 8040 |
| A23 | Topo anti-antigene carcinoembrionario | CEA | USA biologico | C1300 |
| A24 | Topo Antigen anti-cancro CA125 | CA125 | USA biologico | C0050-01D |
| A25 | Topo anti-CA19-9 Cancer antigene | CA19-9 | USA biologico | C0075-18 |
| A26 | Topo anti-Lewis x anticorpo monoclonale | Lewis x | Calbiochem | 434631 |
| bio | Biotinilato BSA (controllo positivo) | Bio | Fatto in casa | N / A |
Tabella 1. Elenco delle lectine e anticorpi utilizzati in questo protocollo.
| Nome del reattivo, s / attrezzature | Azienda | Numero di catalogo |
| Contatto micro-distributore non | BioDot Inc | sciFLEXARRAYER |
| 384 micropiastra | Pescatore | 14-230-243 |
| Foodsaver | Foodsaver | V3835 |
| Ultrasottile nitrocellulosa Coate microarray scivola | Gentel | PATH |
| Presentazione Imprinter (opzionale) | Il Gel Società | WSP60-1 |
| Shaker | Pescatore | 15-453-211 |
| Centrifuga | Eppendorf | 5804 000.013 |
| Diapositiva lavandino / Slide Dish colorazione con Rack estraibile | Pescatore | 08-812 |
| Diapositiva camera di incubazione / vetrino da microscopio box | Pescatore | 03-448-5 |
| Brij 35, 30 w / v% di soluzione in acqua | Acros Organics | AC32958-0025 |
| Tween-20 | Pescatore | P337-100 |
| Periodato di sodio (NAIO 4) | Sigma | 311448 |
| L-glutammico γ-idrazide | Sigma | G-7257 |
| Acetato di sodio anidro (CH 3 COONa) | Sigma | S2889 |
| Albumina sierica bovina (BSA) | Lampire Biological Labs | 7500804 |
| Tampone fosfato salino (PBS) (10X) | Denville scientifico | CP4390-48 |
| Dylight 549 neutravidina coniugato | Thermo | 22837 |
| Inibitori della proteasi Cocktail Tablets | Roche | 4693159001 |
| ChromPure IgG umane, frammento Fc | Jackson ImmunoResearch | 009-000-008 |
| ChromPure IgG umane, molecola intera | Jackson ImmunoResearch | 009-000-003 |
| ChromPure mouse IgG, molecola intera | Jackson ImmunoResearch | 015-000-003 |
| ChromPure mouse IgG, frammento Fc | Jackson ImmunoResearch | 015-000-008 |
| ChromPure IgG di coniglio, molecola intera | Jackson ImmunoResearch | 011-000-003 |
| ChromPure Donkey IgG, molecola intera | Jackson ImmunoResearch | 017-000-003 |
| Microarray Scanner | Tecan | LS Reloaded |
Tabella 2. Elenco di attrezzature e reagenti utilizzati in questo protocollo.
Riferimenti
- Fang, M. The ER UDPase ENTPD5 promotes protein N-glycosylation, the Warburg effect, and proliferation in the PTEN pathway. Cell. 143, 711-724 (2010).
- Marino, K., Bones, J., Kattla, J. J., Rudd, P. M. A systematic approach to protein glycosylation analysis: a path through the maze. Nat. Chem. Biol. 6, 713-723 (2010).
- Shental-Bechor, D., Levy, Y. Effect of glycosylation on protein folding: a close look at thermodynamic stabilization. Proc. Natl. Acad. Sci. U. S. A. 105, 8256-8261 (2008).
- Hossler, P., Khattak, S. F., Li, Z. J. Optimal and consistent protein glycosylation in mammalian cell culture. Glycobiology. 19, 936-949 (2009).
- Nothaft, H., Szymanski, C. M. Protein glycosylation in bacteria: sweeter than ever. Nat. Rev. Microbiol. 8, 765-778 (2011).
- Sola, R. J., Griebenow, K. Effects of glycosylation on the stability of protein pharmaceuticals. J. Pharm. Sci. 98, 1223-1245 (2009).
- Li, C., Lubman, D. M. Analysis of serum protein glycosylation with antibody-lectin microarray for high-throughput biomarker screening. Methods Mol. Biol. 723, 15-28 (2011).
- Dwek, M. V., Jenks, A., Leathem, A. J. A sensitive assay to measure biomarker glycosylation demonstrates increased fucosylation of prostate specific antigen (PSA) in patients with prostate cancer compared with benign prostatic hyperplasia. Clin. Chim. Acta. 411, 1935-1939 (2010).
- Drake, P. M. Sweetening the pot: adding glycosylation to the biomarker discovery equation. Clin. Chem. 56, 223-236 (2010).
- Kim, Y. -. P., Park, S., Oh, E., Oh, Y. -. H., Kim, H. -. S. On-chip detection of protein glycosylation based on energy transfer between nanoparticles. Biosensors & Bioelectronics. 24, 1189-1194 (2009).
- Boland, M., Rudd, P. M. Disease related glycosylation changes and biomarker discovery: challenges and possibilities in an emerging field. Editorial. Dis. Markers. 25, 189-192 (2008).
- Norton, P. A. N-linked glycosylation of the liver cancer biomarker GP73. J. Cell Biochem. 104, 136-149 (2008).
- Nakagawa, T. Glycomic analysis of alpha-fetoprotein L3 in hepatoma cell lines and hepatocellular carcinoma patients. J. Proteome Res. 7, 2222-2233 (2008).
- Durazo, F. A. Des-gamma-carboxyprothrombin, alpha-fetoprotein and AFP-L3 in patients with chronic hepatitis, cirrhosis and hepatocellular carcinoma. J. Gastroenterol Hepatol. 23, 1541-1548 (2008).
- Kobayashi, M. Fucosylated fraction of alpha-fetoprotein, L3, as a useful prognostic factor in patients with hepatocellular carcinoma with special reference to low concentrations of serum alpha-fetoprotein. Hepatol. Res. 37, 914-922 (2007).
- Maisey, N. R. CA19-9 as a prognostic factor in inoperable pancreatic cancer: the implication for clinical trials. Br. J. Cancer. 93, 740-743 (2005).
- Talar-Wojnarowska, R. Clinical value of serum neopterin, tissue polypeptide-specific antigen and CA19-9 levels in differential diagnosis between pancreatic cancer and chronic pancreatitis. Pancreatology. 10, 689-694 (2010).
- Chen, S. Multiplexed analysis of glycan variation on native proteins captured by antibody microarrays. Nat. Methods. 4, 437-444 (2007).
- Shao, C. Antibody microarray analysis of serum glycans in esophageal spuamous cell carcinoma cases and controls. Proteomics Clinical Applications. 3, 923-931 (2009).
- Chen, S., Haab, B. B. Analysis of glycans on serum proteins using antibody microarrays. Methods Mol. Biol. 520, 39-58 (2009).
- Yue, T. The Prevalence and Nature of Glycan Alterations on Specific Proteins in Pancreatic Cancer Patients Revealed Using Antibody-Lectin Sandwich Arrays. Molecular & Cellular Proteomics. 8, 1697-1707 (2009).
- Wolf-Yadlin, A., Sevecka, M., MacBeath, G. Dissecting protein function and signaling using protein microarrays. Current Opinion in Chemical Biology. 13, 398-405 (2009).
- Richard, E. Proteomics as Applied to Inherited Metabolic Diseases. Current Proteomics. 6, 140-153 (2009).
- Nolen, B., Winans, M., Marrangoni, A., Lokshin, A. Aberrant tumor-associated antigen autoantibody profiles in healthy controls detected by multiplex bead-based immunoassay. Journal of Immunological Methods. 344, 116-120 (2009).
- Kuno, A. Focused Differential Glycan Analysis with the Platform Antibody-assisted Lectin Profiling for Glycan-related Biomarker Verification. Molecular & Cellular Proteomics. 8, 99-108 (2009).
- Hsu, K. -. L., Mahal, L. K. Sweet tasting chips: microarray-based analysis of glycans. Current Opinion in Chemical Biology. 13, 427-432 (2009).
- Borrebaeck, C. A. K., Wingren, C. High-throughput proteomics using antibody microarrays: an update. Expert Review of Molecular Diagnostics. 7, 673-686 (2007).
- Sanchez-Carbayo, M. Antibody array-based technologies for cancer protein profiling and functional proteomic analyses using serum and tissue specimens. Tumor Biology. 31, 103-112 (2010).
- Porter, A. A motif-based analysis of glycan array data to determine the specificities of glycan-binding proteins. Glycobiology. 20, 369-380 (2010).
- Maupin, K. A. Glycogene Expression Alterations Associated with Pancreatic Cancer Epithelial-Mesenchymal Transition in Complementary Model Systems. Plos One. 5, (2010).
- Sevecka, M., Wolf-Yadlin, A., MacBeath, G. Lysate Microarrays Enable High-throughput, Quantitative Investigations of Cellular Signaling. Molecular & Cellular Proteomics. 10, (2011).
- Wang, M. Novel fucosylated biomarkers for the early detection of hepatocellular carcinoma. Cancer Epidemiol. Biomarkers Prev. 18, 1914-1921 (2009).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon