Method Article
Senza contatto, Label-free monitoraggio delle cellule e matrice extracellulare mediante Spettroscopia Raman
In questo articolo
Riepilogo
Spettroscopia Raman è una tecnica adatta per il non-contatto, label-free analisi di cellule viventi, dell'ingegneria tissutale costrutti e tessuti nativi. Fonte specifici per le impronte digitali spettrali possono essere generati ed analizzati utilizzando l'analisi multivariata.
Abstract
Non distruttivo, non-contatto e label-free tecnologie per monitorare colture cellulari e tissutali sono necessari nel campo della ricerca biomedica. 1-5 metodi di routine, tuttavia, attualmente disponibili richiedono fasi di elaborazione e alterare l'integrità del campione. Spettroscopia Raman è un metodo rapido che permette la misurazione di campioni biologici senza necessità di ulteriori fasi di lavorazione. . Questo laser-tecnologia basata rileva la diffusione anelastica di luce monocromatica 6 In ogni vibrazione chimica è assegnato ad una specifica banda Raman (numero d'onda in cm -1), ciascun campione biologico presenta un tipico andamento spettrale a causa della loro intrinseca composizione biochimica 7. - 9 Entro spettri Raman, l'intensità dei picchi in correlazione con l'ammontare delle obbligazioni presenti molecolari. 1 Analogie e differenze dei set di dati spettrali possono essere rilevati utilizzando un'analisi multivariata (es. analisi delle componenti principali (PCA)) 10. Qui, noi dobbiamo fare spettroscopia Raman di cellule e tessuti viventi native. Le cellule sono seminate o sulle antenne fondo di vetro o mantenuto in sospensione in condizioni normali colture cellulari (37 ° C, 5% CO 2) prima della misurazione. Tessuti nativi sono sezionati e memorizzati in salina tamponata con fosfato (PBS) a 4 ° C misurazioni precedenti. A seconda del nostro set up sperimentale, abbiamo quindi sia focalizzata sul nucleo della cellula o della matrice extracellulare (ECM) proteine, quali il collagene e l'elastina. Per tutti gli studi, un minimo di 30 celle o 30 punti casuali di interesse all'interno del ECM vengono misurate. Fasi di elaborazione dei dati inclusi sottrazione del fondo e la normalizzazione.
Protocollo
1. Preparazione del campione biologico
- Preparazione delle cellule viventi
- Preparazione delle cellule aderenti
- Seme in cellule coltivate in vitro, o isolate di fresco su un piatto di vetro inferiore (Greiner BioOne / Germania) e incubare a 37 ° C e 5% CO 2 fino attaccamento cellulare è completata.
- Togliere terreno di coltura e lavare delicatamente tre volte con PBS misura preventiva. Conservare le cellule coperti sia con PBS o terreno di coltura cellulare durante tutta la procedura di misura.
- Preparazione delle cellule in sospensione
- Staccare in vitro-colture di cellule in base a protocolli comuni (ad esempio, tripsina-EDTA, scraping cella), centrifugare le cellule e risospendere il pellet cellulare ottenuta in PBS o terreno di coltura cellulare.
- Trasferire 100 microlitri della sospensione cellulare (concentrazione: massimo 100.000 cellule / ml) ad un piatto fondo di vetro.
- Preparazione delle cellule aderenti
- Preparazione di native tessuti
- Dopo la raccolta i tessuti, trasferirli sterile, ghiacciato PBS e tenerli non più di 12 ore a 4 ° C oppure attraverso una misurazione ghiaccio prima. Esperimenti precedenti hanno dimostrato che un tempo prolungato stoccaggio (tempo di ischemia fredda> 12 ore a 4 ° C) determinato cambiamenti spettrali dovute a processi di degradazione naturali.
- Le misurazioni devono essere effettuate con il tessuto rivestito in PBS o mezzo per evitare danneggiamenti delle proteine ECM e le cellule a causa di asciugatura del tessuto.
2. Spettrometro Raman
Il nostro spettrometro Raman personalizzata combina un microscopio a fluorescenza standard (Olympus IX 71), con uno spettrometro Raman che permette il confronto diretto delle immagini in campo chiaro e fluorescenza con gli spettri Raman. Il set di base è costituito da un diodo laser 784 nm (AG Toptica fotonica / Germania), un filtro notch per la separazione del Raman-luce diffusa dalla luce di eccitazione, un microscopio unond uno spettrografo (Kaiser Optical Systems Inc., Ann Arbor / USA) con un dispositivo ad accoppiamento di carica (CCD) ottimizzato per la rilevazione di informazioni spettrali (F-view from soft Imaging Systems / Germania).
3. Controllo della funzione Laser
- Avviare il software Andor Solis (Andor / Regno Unito) e impostare la temperatura della camera CCD a -60 ° C per ridurre al minimo il rumore provocato dalle correnti indotte termicamente nella fotocamera.
- Posizionare un wafer di silicio sul palco microscopio per la procedura di calibrazione.
- Accendere il laser e impostare la potenza a 85 mW.
- Utilizzare il software delle cellule B (Olympus / Germania) di focalizzare il laser sul wafer fino XY appare.
- Misurare il wafer di silicio con un unico tempo di integrazione di 1 sec usando un obiettivo 60x aria.
- Modificare l'unità della x-asse da alla variazione del numero di pixel Raman (cm -1) nel software Andor Solis.
- Variare il fuoco del laser al silicio del picco a 520 cm -1 inil raccolto spettro per trovare l'intensità massima possibile per questa banda Raman. La quantità minima di conteggi deve essere maggiore di 11.000 a una riuscita della calibrazione.
4. Raman misure spettroscopiche
Tutte le misure sono eseguite a temperatura ambiente.
- Impostazioni di base
- Utilizzare un obiettivo ad immersione in acqua 60x (Olympus / Germania) con un'apertura numerica di 1,2 per raccogliere lo spettro dei campioni.
- Modificare le impostazioni di acquisizione integrazioni a 10/10 secondi per un totale di 100 secondi per le misurazioni.
- Misurazione di cellule aderenti
- Prendete il piatto fondo di vetro con le celle e collocarlo sul palco microscopio.
- Per ottenere un segnale migliore e garantire la riproducibilità, focalizzare il laser sul nucleo della cellula, ruotare la luce microscopio e iniziare la raccolta dello spettro.
- Misurare uno spettro di riferimento del fondo emolto 10 spettri spostando il fuoco del laser accanto alla cella. E 'importante considerare che quando si cambia la messa a fuoco un nuovo sfondo devono essere raccolti per ogni profondità di messa a fuoco.
- Misurazione di cellule in sospensione
- Trasferire 100 microlitri della sospensione cellulare nel piatto inferiore vetro e posizionarlo sul microscopio scena.
- Fuoco del laser al centro della cella, ruotare la luce microscopio e iniziare la raccolta dello spettro.
- Misurare uno spettro di riferimento dello sfondo ogni 10 spettri spostando il fuoco del laser accanto alla cella. Quando si cambia la messa a fuoco, un nuovo sfondo devono essere rilevati per la profondità nuova attenzione.
- Misurazione di tessuti nativi
- Prendete il campione e trasferirli in un piatto fondo di vetro. La regione di interesse (ROI) dovrebbero essere orientati verso il fondo del recipiente.
- Riempire il piatto con PBS a sufficienza per coprire il campione.
- Posizionare un bicchiere di copertura sul campione per evitare qualsiasi movement del campione durante le misurazioni.
- Impostare il fuoco del laser nella struttura di interesse (risoluzione in profondità è laser e tessuto-dipendente) e iniziare a raccogliere gli spettri.
- Raccogliere uno spettro di riferimento dello sfondo ogni 10 spettri spostando il laser fuoco all'esterno della zona di tessuto intero. Quando si cambia la messa a fuoco, un nuovo sfondo devono essere rilevati per la profondità nuova attenzione.
- Misura di immunofluorescenza (IF)-etichettati criosezioni
- Sezione freschi, congelati snap-campioni di tessuto utilizzando un cryotom standard e li montano su silice rivestite con vetri di copertura.
- Colorare le criosezioni secondo un protocollo di routine per IF, impiegando soltanto un piccolo passo di fissaggio (max 10 minuti con paraformaldeide al 4%) e utilizzando anticorpi appropriati per la rilevazione della proteina di interesse.
- Eseguire misurazioni Raman concentrandosi nella zona in cui si verifica fluorescenza.
- Elastina degradazione esperimenti
- Place la ventricularis dei lembi della valvola aortica dissezionati suina (elastina ricco, sangue afflusso strato di lato del lembo della valvola cardiaca) di fronte al fondo del piatto inferiore vetro.
- Misurare il tessuto nativo come un 'non-incubate control' a 30 punti casuali in tutta la superficie del tessuto intera messa a fuoco nelle strutture fibrillari.
- Dividere in 3 sezioni e inserirli in separati 2.5 ml provette Eppendorf contenenti 2 ml di una soluzione elastasi (5 U / ml, Worthington / Germania).
- Incubare il tessuto per 15 o 30 minuti a 37 ° C.
- Dopo incubazione per 15 o 30 minuti, rimuovere i tessuti dalla provetta Eppendorf e lavare accuratamente con PBS al fine di interrompere completamente la reazione enzimatica.
- Misurare ogni campione a 30 punti casuali, concentrandosi nelle strutture fibrillari.
5. Elaborazione dati e analisi
- Spettri Raman di elaborazione
Il pre-trattamento deispettri generati è stata eseguita utilizzando il software OPUS (Bruker Optik GmbH / Germania).- Al fine di ridurre i segnali interferenti dal vetro e mezzo, nonché per evitare variazioni dovute variazioni del fuoco durante le misure, sottrarre lo spettro corrispondente sfondo dal spettri raccolti.
- Ridurre gli spettri nella regione tra il numero d'onda 400-1800 cm -1, che offre la più alta quantità di informazioni.
- Se necessario, normalizzare gli spettri al picco massimo. Fattori di normalizzazione indica le fluttuazioni di intensità e fallimenti sistematici, semplificando l'individuazione di cambiamenti strutturali nello spettro del campione.
- Eseguire una correzione al basale per aumentare la comparabilità tra i diversi esperimenti.
- Analisi di spettri Raman
Gli spettri Raman sono stati analizzati utilizzando PCA con la (CAMO / Norvegia) software Unscrambler. L'analisi multivariata rileva le differenze e le somiglianze all'interno dei dati spettraliset. Ogni spettro è tracciata come un unico punto in uno spazio multidimensionale in base ai conteggi raccolti per ogni turno Raman. Ciascun componente principale (PC) descrive una certa quantità di informazioni totale contenute nei dati originali. Il primo PC è quello che contiene la prima fonte di variabilità. Ogni PC che segue contiene, in ordine, meno informazioni rispetto al precedente. Ogni variabile ha un punteggio e un caricamento su ogni PC. Tracciando PC (= punteggi), correlazioni campione importanti possono essere esposti. I caricamenti descrivono il contributo di ciascuna variabile analizzata per la PCA.- Etichettare ogni gruppo di misure con la creazione di campi di riga per ogni gruppo di campioni.
- Utilizzare le seguenti impostazioni di base per la PCA: cross di convalida, l'algoritmo NIPALS, nessuna rotazione e avviare l'analisi. Queste impostazioni sono spettri dipendenti.
- Eseguire la PCA.
6. Risultati rappresentativi
Raman spECTRA generata da cellule aderenti spesso rivelano un basso rapporto segnale-rumore e una bassa intensità di segnale globale (Fig. 1). causa del fatto che il fuoco del laser deve essere impostata in prossimità del fondo di vetro, l'influenza del interferire 11 segnale bicchiere è piuttosto elevata, provocando mascheramento del segnale campione effettivo. Di conseguenza, il segnale campione potrebbe essere ridotta o addirittura eliminato durante una successiva sottrazione del fondo. Così, si preferisce utilizzare cellule in sospensione per la nostra analisi spettroscopica Raman, in quanto consentono il rilevamento di informazioni più dettagliate spettrale. Tuttavia, gli spettri di cellule aderenti e le sospensioni presentano le stesse vette principali che differiscono solo nella loro intensità.
Per la caratterizzazione di tipi cellulari diversi all'interno di una sospensione, senza pre-trattamento è richiesto. Gli spettri Raman media e la deviazione standard di fibroblasti umani, cellule staminali mesenchimali (MSC), condrociti e cheratinociti misurato in sospensione sonoillustrato nella figura 2. Tutti gli spettri Raman sono strutturati in modo simile, con picchi provenienti da biomolecole tipici quali proteine, acidi nucleici e lipidi (vedi Tabella 1). 12 Per questi tipi di cellule, la regione spettrale fra 600 e 1800 cm -1 contiene più pertinenti informazione spettrale, da chiare differenze che sono individuabili tra i differenti tipi di cellule (Fig. 2A). Esemplare, abbiamo evidenziato una regione spettrale (1280-1350 cm -1) presentano chiari differenze strutturali, che è assegnabile a vibrazioni molecolari di collagene e di lipidi. Al contrario, le analisi morfologiche non sono adatti per l'identificazione e la distinzione delle maggior parte delle cellule (Fig. 2B-I). Mentre la differenza tra condrociti e cellule della pelle è osservabile (Fig. 2D, H contro B, E e F, I), fibroblasti e MSC sono difficili da separare utilizzando esclusivamente in campo chiaro micrsocopia (Fig. 2B, F rispetto a C, G) 13.
Raman analisi spettroscopica di tessuto nativo, in particolare di proteine ECM, richiede che una ROI può essere visualizzato in campo chiaro immagini per essere in grado di concentrarsi sulla rispettiva struttura. Per l'assegnazione di una proteina di uno spettro impronta specifica, abbiamo generato spettri Raman di proteine pure disponibili in commercio e criosezioni immunoistologico macchiati. Qui, abbiamo individuato gli spettri delle impronte digitali di fibre elastiche all'interno dei tessuti nativi confronto elastina liofilizzato e immunofluorescenza macchiati criosezioni che impiegano un anticorpo contro l'elastina. Tuttavia, poiché elastina presenta un autofluorescenza alto, che si riflette nella spettri Raman, l'analisi dei dati è impegnativo (Fig. 3A). Per ridurre l'errore sistematico dovuto campione proprietà specifiche, come autofluorescenza, un trattamento adeguato dei set di dati è fondamentale. In uno i nostri datialyses, abbiamo utilizzato la normalizzazione per eliminare l'intensità del segnale significativamente maggiore della proteina pura elastina, e quindi, siamo stati in grado di generare comparabile spettri Raman (fig. 3B). L'elastina è una delle proteine ECM più stabili nel corpo ed è quindi molto difficile da degradare 14. Nel nostro set up sperimentale, abbiamo indotto la degradazione elastina in volantini sani suina della valvola aortica eseguendo una digestione enzimatica. Applicando la analisi multivariata PCA, abbiamo individuato differenze significative tra gli spettri Raman di enzimaticamente trattati con campioni ed i controlli nativi (Fig. 3C). Queste differenze sono state osservate spettrali nello spettro di carico a 861, 1003 e 1664 cm -1. Attesi cambiamenti strutturali nelle fibre a causa di tempi di esposizione prolungati a elastasi elastina contenenti sono stati mostrati mediante colorazione HART (Fig. 4), che sono riflesse anche in più gruppi distinti punteggio separabili (Fig. 3C).

Figura 1. Media spettri Raman dei fibroblasti staccati e aderente.
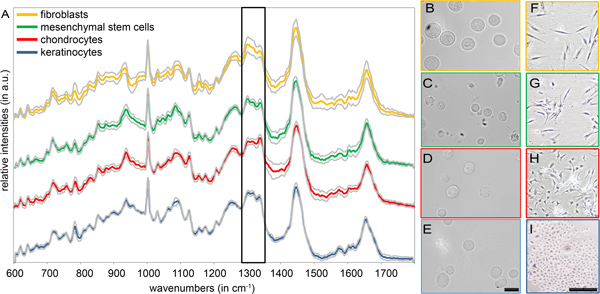
Figura 2. (A) media spettri Raman e deviazioni standard di quattro differenti tipi principali di cellule isolate (fibroblasti, CSM, condrociti e cheratinociti). Il telaio mette in evidenza la regione spettrale di 1280 - 1350 cm -1 con le differenze strutturali. (BE) in campo chiaro immagini di staccate (B), i fibroblasti (C) MSCS, (D) e condrociti (E) cheratinociti. Barra della scala è uguale a 20 micron. (FI) in campo chiaro immagini di aderenti (F), i fibroblasti (G) MSC, (H) e (i condrociti) cheratinociti. Barra della scala equivale a 200 micron.

Figura 3. (A) spettri Raman senza normalizzazione di lyophiliZed elastina (linea blu), immunofluorescenza (IF)-etichettati criosezioni (linea arancione) e le fibre elastiche (linea rossa) misurati all'interno lembi della valvola aortica nativa. L'intensità di segnale alto liofilizzata elastina è causato da autofluorescenza. (B) spettri Raman dopo la normalizzazione per eliminare guasti sistemici. (C) I punteggi ed i caricamenti del confronto tra i non-trattata di controllo (rosso) e-enzimaticamente degradata (blu e verde) fibre elastiche nel tessuto nativo.

Figura 4. HART's macchiati suina lembi valvolari aortici. Le fibre elastiche sono visualizzati in nero. (A) e (B) mostrano non trattati e di controllo (C) e (D) rappresentano campioni di tessuto che sono stati esposti alla elastina-enzima degradante elastasi per 30 minuti.
| Numero d'onda in cm -1 | Assegnazione 12 | |
| 717-719 | CN | Fosfolipidi |
| 785-788 | DNA / RNA basi, OPO backbone | DNA / RNA |
| 1003 - 1005 | Fenilalanina | Proteina |
| 1220-1280 | Ammide III | Proteina |
| 1445-1447 | CH 2 | Proteine / lipidi |
| 1655-1680 | Amide IC = C | Protein lipidi |
Tabella 1. Bande Raman che vengono rilevati in spettri di tutti i tipi cellulari (fibroblasti, CSM, condrociti e cheratinociti).
Discussione
Spettroscopia Raman è un attrezzo adatto per analizzare campioni biologici, come in vitro-colture di cellule e proteine ECM così come le cellule all'interno dei tessuti nativi. 11,15,16 Qui, abbiamo dimostrato che questo non-contatto, privo di etichetta tecnica permette discriminazione di differenti tipi di cellule e la rilevazione della degradazione proteica ECM esclusivamente in base alla composizione biomolecolare intrinseca di questi campioni biologici.
Il principale vantaggio della spettroscopia Raman è la capacità di non-invasivo quantificare l'impronta digitale biochimica di un campione dalla sua spettri Raman risultante. In contrasto con spettroscopia infrarossa, che produce informazioni simili, spettri Raman può essere raccolto da campioni acquosi, come la dispersione Raman dall'acqua è debole. Inoltre, spettroscopia Raman si basa unicamente sulla rilevazione di backscattering di luce monocromatica, pertanto, non è richiesta l'elaborazione del campione precedente misura. Questi attributi rendono Raspettroscopia uomo una promettente alternativa per il potenziale in applicazioni di imaging in vivo. A questo proposito, coerente anti-Stokes spettroscopia Raman (CARS) è una tecnica molto interessante in quanto permette l'acquisizione veloce e più sensibile dei dati in base ai segnali vibrazionali stessi utilizzati nei nostri esperimenti. 15,16 Altri metodi alternativi tra cui multiphoton indotta autofluorescenza e seconda immagini generazione armonica sono stati precedentemente dimostrato di essere adatto per il monitoraggio di campioni biologici non invasivo o minima. 17 Tuttavia, queste modalità di imaging sono associati con costi molto elevati e sono limitati a autofluorescenza generatrici molecole. Inoltre, spettrometro Raman è facile da abbinare con tradizionali microscopi ottici. Queste caratteristiche rendono la spettroscopia Raman uno strumento prezioso per studiare campioni biologici in ambienti fisiologici.
Uno dei limiti attuali della nostra spettroscopia Raman è istituitoil fuoco del laser relativamente piccola (250 nm intero massimo metà larghezza (FWHM) laterale e 700 nm FWHM assiale) che viene creato da un obiettivo ad alta apertura numerica (NA = 1,2). Sebbene un elevato apertura numerica permette di coprire una buona quantità di luce emessa Raman cedere in un alto rapporto segnale-a-rumore, l'elevata NA produce solo un piccolo fuoco raccolta all'interno del campione che è in genere molto più piccola di una cella. Per confrontare gli spettri Raman di cellule differenti, la raccolta di uno spettro rappresentativo è essenziale, che è difficile da ottenere con una piccola area di messa. Per risolvere questo problema, stiamo lavorando su un processo per automatizzare la raccolta del segnale in diversi punti all'interno della cellula (= mappatura spettroscopica Raman), con un conseguente spettrale media e cedere ad uno spettro rappresentativo. Inoltre, questa tecnica fornisce una panoramica della distribuzione delle specifiche bande Raman, ad esempio della distribuzione proteina all'interno di una cellula.
BiolI campioni ogical sono estremamente complessi e costituiti da una miscela eterogenea di biomolecole che contribuiscono alla raccolta spettri Raman. Pertanto, il modello spettrale è molto complessa e il controllo di un singolo tipo di una molecola all'interno di uno spettro Raman è difficile da realizzare con la sovrapposizione di segnali molecolari diversi. Inoltre, fluorescenza intrinseca del campione può mascherare preziose informazioni di deboli segnali Raman. È interessante notare che in alcuni dei nostri studi precedenti, abbiamo individuato in autofluorescenza gli spettri Raman come il principale fattore di differenziazione tra i tipi di cellule (fibroblasti e MSC) utilizzando uno strumento adeguato di analisi 13. Abbiamo anche individuato che i cambiamenti di intensità complessiva del segnale Raman può servire come un indicatore per lo stato di collagene e le fibre di collagene all'interno della ECM di lembi valvolari aortici. 9 Tuttavia, quando si analizza lo stato di elastina in questi tessuti, non siamo stati in grado di rilevare risultati simili. Come indicato nel risultatosezione s, si è invece in grado di rilevare alterazioni specifiche bande Raman elastasi nei campioni trattati rispetto ai controlli nativi. Non abbiamo visto una diminuzione complessiva del segnale Raman nei enzimaticamente trattati con campioni come previsto. Queste osservazioni portato in una trama punteggio che non ha rivelato una formazione di cluster chiaro come si vede nello studio precedente. 9 Al contrario, l'influenza del trattamento enzimatico è rilevabile nei risultati PCA. Partiamo dal presupposto che queste differenze tra i due proteine ECM, elastina e il collagene, si basano sulle differenze morfologiche e diversi processi di degradazione enzimatica: all'interno del lembo della valvola aortica, il collagene ricco di zone (fibrosa) è uno strato continuo che viene allentato a causa della trattamento enzimatico, per cui la zona contenente elastina (ventricularis) ha una configurazione di rete che appare frammentato dopo l'esposizione a elastasi (Fig. 4). Le misurazioni in loco singoli stati, pertanto, non approprimangiava per rilevare tali rotture piccoli all'interno della rete elastina. Qui, una mappatura Raman del tessuto aiuterebbe a identificare i guasti nelle reti.
Un'ulteriore sfida in spettroscopia Raman di campioni biologici è quello di ridurre i tempi di misura. Una soluzione è quella di aumentare la potenza del laser, che è adatto finché i campioni biologici non sono interessati da foto-danni. Tutti i nostri esperimenti in corso sono la prova di principio studi incentrati sulla ricerca di base, tuttavia, il nostro obiettivo generale è quello di implementare la spettroscopia Raman per applicazioni cliniche compresa la medicina rigenerativa (ad esempio: controllo della qualità dei prodotti dell'ingegneria tissutale), pre-trapianto e trapianto di monitoraggio la diagnostica del cancro.
Divulgazioni
Non ci sono conflitti di interesse dichiarati.
Riconoscimenti
Ringraziamo Steffen Koch per il suo supporto tecnico e Shannon Lee Layland (sia IGB Fraunhofer di Stoccarda) per i suoi utili suggerimenti sul manoscritto. Questo lavoro è stato sostenuto finanziariamente dal Attrarre programma del Fraunhofer-Gesellschaft e la BMBF (sia per KS-L.).
Materiali
| Name | Company | Catalog Number | Comments |
| Nome del reattivo | Azienda | Numero di catalogo | Comments |
| elastasi | Worthington | LS006363 | |
| anticorpi anti-elastina | Sigma | HPA018111 | 1:75; tampone citrato |
| PBS | Lonza | 17-512F | |
| fondo di vetro piatti | Greiner BioOne | 627860 | |
| Il Unscrambler | CAMO | ||
| Opus | Bruker |
Riferimenti
- Chan, J. W., Lieu, D. K. Label-free biochemical characterization of stem cells using vibrational spectroscopy. J. Biophotonics. 2, 656-668 (2009).
- Downes, A., Mouras, R., Elfick, A. Optical spectroscopy for noninvasive monitoring of stem cell differentiation. J. Biomed. Biotechnol. 2010, 101864-101864 (2010).
- Gentleman, E. Comparative materials differences revealed in engineered bone as a function of cell-specific differentiation. Nat. Mater. 8, 763-770 (2009).
- Notingher, I., Hench, L. L. Raman microspectroscopy: a noninvasive tool for studies of individual living cells in vitro. Expert. Rev. Med. Devices. 3, 215-234 (2006).
- Schenke-Layland, K. Optimized preservation of extracellular matrix in cardiac tissues: implications for long-term graft durability. Ann. Thorac. Surg. 83, 1641-1650 (2007).
- Raman, C. V., Krishnan, K. S. A new type of secondary radiation. Nature. 122, 12(1928).
- Frushour, B. G., Koenig, J. L. Raman scattering of collagen, gelatin, and elastin. Biopolymers. 14, 379-391 (1975).
- Pudlas, M., Koch, S., Bolwien, C., Walles, H. Raman spectroscopy as a tool for quality and sterility analysis for tissue engineering applications like cartilage transplants. Int. J. Artif. Organs. 33, 228-237 (2010).
- Votteler, M. Raman spectroscopy for the non-contact and non-destructive monitoring of collagen damage within tissues. J. Biophotonics. , (2011).
- Wold, S., Esbensen, K., Geladi, P. Principal component analysis. Chemometrics and Intelligent Laboratory Systems. 2, 37-52 (1987).
- Pudlas, M. Raman Spectroscopy: A Noninvasive Analysis Tool For The Discrimination of Human Skin Cells. Tissue Eng. Part C Methods. 10, (2011).
- Movasaghi, Z., Rehman, S., Rehman, I. U. Raman Spectroscopy of Biological Tissues. Applied Spectroscopy Reviews. 42, 493-541 (1080).
- Pudlas, M. Non-contact discrimination of human bone marrow-derived mesenchymal stem cells and fibroblasts using Raman spectroscopy. Medical Laser Application. 26, 119-125 (2011).
- Mecham, R. P. Methods in elastic tissue biology: elastin isolation and purification. Methods. 45, 32-41 (2008).
- Downes, A., Mouras, R., Bagnaninchi, P., Elfick, A. Raman spectroscopy and CARS microscopy of stem cells and their derivatives. Journal of Raman Spectroscopy. 42, 1864-1870 (2011).
- Krafft, C., Dietzek, B., Popp, J. Raman and CARS microspectroscopy of cells and tissues. Analyst. 134, 1046-1057 (2009).
- Schenke-Layland, K. Non-invasive multiphoton imaging of extracellular matrix structures. J. Biophotonics. 1, 451-462 (2008).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon