Method Article
Un protocollo per l'identificazione di interazioni proteina-proteina Sulla base di
In questo articolo
Riepilogo
Vi presentiamo una variante del RAPIDO (immunoprecipitazione quantitativi combinazione con smontabile), approccio che è stato introdotto in precedenza per distinguere tra vero e falso interazioni proteina-proteina. Il nostro approccio è basato sulla 15N Marcatura metabolica, la modulazione di affinità di interazioni proteina-proteina dalla presenza / assenza di ATP, immunoprecipitazione e spettrometria di massa quantitativa.
Abstract
Interazioni proteina-proteina sono fondamentali per molti processi biologici nella cellula. Pertanto, la loro caratterizzazione svolge un ruolo importante nella ricerca attuale e una pletora di metodi per la loro indagine è disponibile 1. Interazioni proteina-proteina sono spesso altamente dinamico e potrebbe dipendere localizzazione subcellulare, modificazioni post-traduzionali di proteine e dell'ambiente locale 2. Pertanto, essi devono essere esaminati nel loro ambiente naturale, per cui co-immunoprecipitazione approcci sono il metodo di scelta 3. Co-precipitate partner di interazione sono identificati mediante immunoblotting in un approccio mirato, o spettrometria di massa (LC-MS/MS) in modo non mirati. Quest'ultima strategia spesso è influenzata negativamente da un gran numero di falsi positivi scoperte, derivano principalmente dalla sensibilità di moderni spettrometri di massa che con sicurezza rilevare tracce di aspecifico proteine precipitanti. A Recent approccio per superare questo problema si basa sull'idea che ridotte quantità di partner di interazione specifici sarà co-precipitato con una proteina bersaglio in cui la concentrazione cellulare è diminuito di RNAi, mentre le quantità di proteine aspecifico precipitanti dovrebbe essere influenzata. Questo approccio, chiamato VELOCE per immunoprecipitazione quantitativi combinazione con atterramento 4, impiega Etichettatura degli isotopi stabili di aminoacidi nelle cellule in coltura (SILAC) 5 e MS per quantificare la quantità di proteine immunoprecipitate da ceppi wild-type e knock-down. Proteine che si trovano in un rapporto di 1:1 può essere considerata come contaminanti, tali arricchito in precipitati dal tipo selvaggio come partner di interazione specifici della proteina bersaglio. Anche se innovativo, QUICK porta alcune limitazioni: in primo luogo, SILAC è costosa e limitata per gli organismi che idealmente sono auxotrophic per arginina e / o lisina. Risultati di interconversione Inoltre, quando viene alimentato arginina pesante, arginina-to-prolina in additmassa ional sposta per ciascun prolina in un peptide e diluisce leggermente pesante con arginina luce, che rende la quantificazione più noioso e meno accurato 5,6. Secondo, VELOCE richiede che gli anticorpi vengono titolati in modo tale che essi non si satura con proteina bersaglio in estratti di knock-down mutanti.
Qui vi presentiamo un protocollo modificato VELOCE che supera i limiti di cui sopra, con Quick sostituendo SILAC per l'etichettatura 15 N metabolica e sostituendo RNAi mediato knock-down per la modulazione affinità di interazioni proteina-proteina. Dimostriamo l'applicabilità del protocollo in base alle unicellulari Chlamydomonas verde alga reinhardtii come organismo modello e il tuttofare HSP70B cloroplasto come proteina bersaglio 7 (Figura 1). HSP70s sono noti per interagire con specifiche co-accompagnatori e substrati solo nello stato ADP 8. Sfruttiamo questa proprietà come mezzo per verificare la specificainterazione di HSP70B con il suo fattore di scambio nucleotidico CGE1 9.
Protocollo
1. Anticorpo adsorbimento
- Pesare 120 mg di proteina A Sepharose in 15 ml tubo conico (Falcon). A 15 mg di proteina A Sepharose è necessario per ogni immunoprecipitazione (IP) tale importo è sufficiente per 8 IP. Aggiungere 5 ml di tampone fosfato 0,1 M (pH 7,4) e lasciare la Proteina A Sepharose swell per 30 min a 4 ° C.
(Si noti che tutti i passi da questo momento devono essere effettuate con i guanti per evitare la contaminazione con cheratina e sul ghiaccio per evitare la degradazione delle proteine / dissociazione complesso.)
- Centrifugare per 60 sec a 1000 xga 4 ° C a pellet della Proteina A Sepharose gonfio. Rimuovere con cura il supernatante e risospendere le sfere in 5 ml di tampone fosfato 0,1 M (pH 7,4). Ripetere questa operazione per tre volte di lavare accuratamente le perline.
- Dopo l'ultima fase di centrifugazione, rimuovere supernatante e lasciare circa 0,5 ml di tampone fosfato. Aggiungere 0,9 ml di tampone fosfato 0,5 M (pH 7,4), 400affinità microlitri purificati anticorpi primari (50 microlitri per IP) contro la proteina bersaglio (qui HSP70B), e 16 pl di anticorpi contro una proteina di controllo (qui CF1β). Riempire con DDH 2 O ad un volume totale di 5 ml.
(Si noti che gli anticorpi purificati per affinità dovrebbero essere utilizzati per ridurre la contaminazione da IgG non specifiche, che interferiscono con nano-LC-MS analisi - per un protocollo di vedere Willmund et al (2005) 10 CF1β viene precipitato come un controllo di carico ed è stato scelto.. perché è abbondante e, dopo lisi cellulare, presenti nelle frazioni solubili e di membrana. In alternativa, i livelli di proteine contaminanti possono essere utilizzati per normalizzare per carico disuguale.)
- Consentire Proteina A Sepharose perline per IgG adsorbono durante una incubazione di 1 ora a 25 ° C su un rullo tubo (CAT RM5W, 36 rpm).
- Centrifugare per 60 sec a 1000 xga 4 ° C a pellet della Proteina A Sepharose perline. Rimuovere con attenzione il surnatante e resuspend le perle in 5 ml 0,1 M tampone borato di sodio (pH 9,0). Ripetere questa operazione tre volte per rimuovere a fondo le ammine che appagare la reticolante.
- Pesare 25,9 mg di fresco, solido dimethylpimelimidate e risospendere in 5 ml di tampone borato di sodio 0,1 M (pH 9,0) per ottenere una concentrazione finale di 20 mM. Aggiungere questa soluzione alla Proteina A Sepharose perline.
- Permettono IgG di cross-link a proteina A per 30 min a 25 ° C su un rullo tubo.
- Centrifugare per 60 sec a 1000 xga 4 ° C per agglomerare le perline. Rimuovere con attenzione il surnatante e risospendere le sfere in 5 ml 1 M Tris-HCl (pH 7.5) per placare reticolante libero. Ripetere questa operazione una volta ed incubare per 2 ore a 25 ° C o 12-24 ore a 4 ° C su un rullo tubo.
- Opzionale: se Proteina A Sepharose perline accoppiate a IgG non sono direttamente utilizzati per IP, per memorizzare fino a una settimana è possibile. Per questo, centrifugare per 60 sec a 1000 xga 4 ° C per agglomerare le perline, rimuovere attentamente il supernperline atant e risospendere in 5 ml di tampone fosfato 0,1 M (pH 7,5) contenente 0,02% di sodio azide e conservare a 4 ° C fino ad ulteriore utilizzo.
2. Lisi cellulare, reticolazione e preparazione del campione
- Crescere due culture Chlamydomonas in terreno contenente 7,5 mM NH 4 Cl 14 o 15 NH 4 Cl come fonte di azoto ad una densità di ~ 5 x 10 6 cellule / ml. Le cellule hanno bisogno di passare attraverso almeno dieci generazioni per l'etichettatura completa. Qui, le cellule sono state coltivate in terreno photomixotrophically TAP 11 su un agitatore rotatorio a 25 ° C sotto irradiazione continua con luce bianca (30 μE m -2 s -1).
- Trasferire due aliquote di 14-N e 15 N-cellule marcate a quattro tubi GSA e cellule raccolto da un 4 min centrifugazione a 4000 xg e 4 ° C (il numero di cellule raccolte per ciascuna aliquota dipende dalla concentrazione cellulare del bersaglio proteine e ha bisogno di essere determinati empirically in anticipo per garantire sufficiente proteina bersaglio viene precipitato. Un buon punto di partenza è di 10 9 cellule per aliquota, cioè 200 ml di coltura con 5 x 10 6 cellule / ml.)
Per reticolazione solo: nel caso di complessi proteici saranno reticolato prima IP, le cellule devono essere lavati per rimuovere ammine presenti nel terreno. Per questo, risospendere le cellule in 40 ml pre-raffreddato KH tampone (20 mM HEPES-KOH (pH 7,2), 80 mM KCl) e trasferirli a 50 ml provette Falcon. Centrifugare per 60 sec a 1.000 xg e 4 ° C. Ripetere questa operazione una sola volta. - Risospendere le cellule in 2 ml di tampone di lisi (20 mM HEPES-KOH (pH 7,2), 1 mM MgCl 2, 10 mM KCl, 154 mM NaCl) pre-raffreddata a 4 ° C e trasferirli 15-ml provette Falcon. Raccogliere le cellule rimanenti in tubi di GSA con un ulteriore 1 tampone di lisi ml ciascuno. Aggiungere 50 microlitri 25 x inibitore della proteasi e 12,5 microlitri 1 M MgCl 2 (ad una concentrazione finale di 3,5 mM) per ciascuna aliquota.
- Aggiungere 150Lysis microlitri di buffer, 12,5 microlitri 1 M ATP, 833 microlitri creatina fosfato 270 mM e 7 microlitri 5 pg / pl fosfochinasi (concentrazione finale 2,5 mM ATP creatina, creatina fosfato 45 mM, e 7 pg / ml fosfochinasi) ad uno dei le aliquote contenenti N-14 e N-15 cellule marcate (questi sono i + ATP aliquote).
- Aggiungere 930 microlitri di buffer di lisi e 70 microlitri 1 U / pl apirasi alle altre aliquote contenenti 14-N e 15 cellule N-etichettati (queste sono le aliquote-ATP).
- Incubare per 2 min a 25 ° C su un rullo avvolgitore per stabilire ATP-impoveriscono e ATP-piene stati. Se la fase di reticolazione viene omesso, aggiungere un altro 1 ml di tampone di lisi.
Per reticolazione solo: nel caso in cui studiate interazioni proteina-proteina sono transitori è consigliabile catturarli da una fase di reticolazione. Per questo, aggiungere 500 microlitri 20 mM ditio-bis (succinimidil propionato) (DSP) disciolto in DMSO (concentrazione finale di 2 mM) ad etubo ach direttamente prima sonicazione. - Sonicare quattro volte 20 secondi sul ghiaccio per rompere le cellule con 20 sec pause in mezzo per il raffreddamento. (Usiamo il Bandelin Sonoplus HD2070 con una punta KE76 al controllo di uscita del 75% e duty cycle del 60%. Le impostazioni necessarie per le altre macchine / dispositivi / suggerimenti devono essere determinato in anticipo per garantire la completa lisi cellulare e per evitare fuoriuscite. )
Solo per la reticolazione: consentono complessi proteici di reticolazione mediante incubazione per 1 ora a 4 ° C su un rullo tubo. Dopo la reticolazione, integrare ogni provetta con 500 microlitri 1 M glicina e incubare in un rullo tubo per altri 15 min a 4 ° C per saziare reticolante libera. - Preparare quattro a 6 ml cuscini saccarosio (20 mM HEPES-KOH (pH 7,2), 0,6 M saccarosio) in SW41 Ti tubi a parete sottile (Beckman Coulter Articolo No: 344059), adagiare l'intero ~ 5,5 ml di lisati cellulari sul saccarosio cuscini (equilibrio con tampone di lisi) e centrifugare per 30 min a 200.000 xg e 4 ° C in un SW41 Ti rotor.
- Trasferire la cima del gradiente contenente complessi proteici solubili in quattro provette da 15 ml (Falcon evitare di trasferire parti del cuscino di saccarosio), aggiungere 350 microlitri 10% Triton X-100 ad una concentrazione finale di 0,5% per ognuno di essi, miscelare accuratamente e aggiungere tampone di lisi per un volume totale di 7 ml ciascuna.
(Trasferimento 70 pl di ciascun estratto cellulare solubile freschi 1,5 ml provette coniche (provette Eppendorf) e aggiungere 70 microlitri 2 x tampone campione SDS-(4% SDS, glicerolo 125 mM Tris-HCl (pH 6,8), 20%, 10 % 2-mercaptoetanolo) a ciascuno per SDS-PAGE e analisi immunoblot.) - Eliminare i cuscini saccarosio e risospendere il pellet di membrana in 3 ml di tampone di lisi ciascuno. Aggiungere a ciascun ml 1 10% Triton X-100 ad una concentrazione finale del 2%, sonicazione in ghiaccio per sciogliere pellets, e aggiungere tampone di lisi per un volume totale di 5 ml ciascuna.
- Preparare altre quattro a 6 ml cuscini saccarosio in SW41 Ti tubi a parete sottile, porre le 5 ml ~ delle membrane solubilizzati dal passaggio 2,10 attentamente sul saccarosiocuscini, e centrifugare per 30 min a 200.000 xg e 4 ° C in un rotore SW41 Ti.
- Trasferire la cima del gradiente contenente complessi di proteine di membrana in quattro provette da 15 ml e aggiungere Falcon tampone di lisi (contenente 2% Triton X-100) ad un volume finale di 7 ml ciascuna. (Trasferimento 70 pl di ciascun estratto cellulare solubile a fresco provette Eppendorf e aggiungere 70 microlitri 2 x SDS-campione ciascuno per SDS-PAGE e immunoblot analisi.)
3. Immunoprecipitazione
- Pellet la Proteina A Sepharose perline contenenti anticorpi accoppiati (da 1,8 o 1,9 gradini) da un 60-sec centrifugazione a 1000 xg e 4 ° C, con attenzione rimuovere le perline surnatante e risospendere in 4 ml di tampone di lisi. Ripetere questa operazione due volte per equilibrare perline in tampone di lisi.
- Riempire fino a 8 ml di tampone di lisi e trasferire 1 ml della sospensione di ciascuna delle otto 15-ml provette Falcon contenenti complessi proteici solubili o di membrana da ATP-ricolma e riducono ATP-14 N-15 e N-marcato cellule (dal punto 2.9 e 2.12).
- Incubare per 2 ore a 4 ° C su un rullo tubo di complessi proteici precipitare.
- Pellet le perle di un 60-sec centrifugazione a 1000 xga 4 ° C e scartare il surnatante. Lasciare un piccolo volume di liquido sopra le perline per facilitare il trasferimento.
- Trasferire le perle da ciascuna provetta Falcon a 1,5 ml conica provette (provette Eppendorf). Raccogliere tutti perline rimanenti nelle provette Falcon, aggiungere un altro 0,8 ml di tampone di lisi contenente 0,1% Triton a ciascuno, vortex delicatamente, centrifugare per 60 sec a 1000 xga 4 ° C e trasferire il buffer con perline residui verso i tubi Eppendorf. Scambio tubo è necessario per evitare contaminazioni da proteine aderenti alle pareti di plastica.
- Pellet le perle di un 15-sec centrifugazione a 16.100 xg e 4 ° C, rimuovere accuratamente i supernatanti e Risospendere le sfere in 1,3 ml di tampone di lisi contenente 0,1% Triton. Ripetere questa operazione due volte con buf lisifer contenente Triton e due volte con tampone di lisi Triton manca di lavare accuratamente le perline. Lasciare un piccolo volume di liquido sopra le perline per facilitare il trasferimento.
- Trasferire nuovamente le microsfere freschi 1,5 ml provette Eppendorf per rimuovere le proteine aderenti alle pareti di plastica. Lavare le provette vecchi con 1 ml di tampone di lisi Triton mancano e trasferire tutti i cordoni residui ai tubi freschi.
- Centrifugare per 15 sec a 16.100 xg e 4 ° C, rimuovere i supernatanti prima con una pipetta normale, quindi rimuovere tutto il supernatante residuo completamente con un 50-microlitri siringa Hamilton.
4. Preparazione del campione per nano-LC-MS/MS
- Aggiungere 100 microlitri di buffer di eluizione appena preparata (8 M urea, 25 mM NH 4 HCO 3) a ciascun tubo, utilizzare il 100 microlitri di buffer di eluizione da lavare perline attaccati alla siringa Hamilton e incubare per 10 min in una Thermomixer a 800 rpm e 65 ° C, e per altri 20 min a 30 ° C. (A molto più completoeluizione di proteine legate eluizione si ottiene con il 2% SDS precipitazioni e successiva con 80% acetone.)
- Centrifugare per 15 secondi a 16.100 xg e 25 ° C. Trasferire supernatanti di tubi freschi con 50 microlitri siringa Hamilton.
- Aggiungere 50 microlitri di buffer di eluizione ai talloni, utilizzare il 50 microlitri di buffer di eluizione per lavare via perline attaccati alla siringa Hamilton e ripetere le fasi di incubazione e centrifugazione 4.1 e 4.2, rispettivamente. Piscina gli eluati rispettivi.
(Trasferimento 30 pl di eluati per provette Eppendorf fresco, aggiungere 30 microlitri 2 x SDS-campione tampone di SDS-PAGE per e analisi immunoblot.) - Combinare l'eluito precipitati da + /-ATP-trattati, 14 e 15 N-N-marcato proteine solubili e di membrana come segue:
120 microlitri 15 N / + ATP e 120 microlitri 14 N /-ATP
120 microlitri 14 N / + ATP e 120 microlitri 15 N /-ATP
120 microlitri 15 N / + ATP e 120 microlitri 14 N /-ATP
120 microlitri 14 N / + ATP e 120 microlitri 15 N /-ATP - Aggiungere 1,5 microlitri preparata DTT 1 M ad una concentrazione finale di 6,5 mM per ciascuno dei quattro combinazioni di ridurre i legami disolfuro (compresi quelli in reticolante) e incubare per 30 min a 25 ° C.
- Aggiungere 10,5 microlitri preparata iodoacetamide 0,6 M ad una concentrazione finale di 25 mM di carboxymethylate i tioli ridotte e incubare per 20 min a 25 ° C al buio.
- Aggiungere 256 microlitri 40 mM NH 4 HCO 3 e 4 microlitri Lys-C (0,1 mcg / pl), tubi di tenuta con parafilm e incubare per almeno 16 ore durante la notte su una ruota di rotazione a 37 ° C.
- Aggiungere 470 microlitri 20 mM NH 4 HCO 3, 10 microlitri acetonitrile 100% (ad una concentrazione finale di 1%) e 8 pl perline tripsina, e incubare in rotazione una ruota per almeno 16 ore a 37 ° C.
- Centrifugare per 5 minuti a 16.100 xg e 4 ° C, e supernatanti trasferimento di fresco 2-vasca ml Eppendorfes. Lavare le provette vecchi con 50 microlitri 20 mM NH 4 HCO 3, 0,5% di acido acetico, e piscina con supernatanti prima.
- Per la dissalazione, preparare in casa C 18-StageTips tagliando fuori due dischi da Empore C 18 del materiale con un ago da siringa e metterli in un 200-pl punta della pipetta. In questo modo preparare quattro 200-pl punte. Punch buchi nei coperchi di quattro 2 ml provette Eppendorf e suggerimenti inserto.
- Presupposto il C 18-StageTips con 50 microlitri soluzione B (80% di acetonitrile, 0,5% di acido acetico). Centrifugare per 3 minuti a 800 g e 25 ° C.
- Equilibrare il C-18 StageTips con 100 ul di soluzione A (acido acetico 0,5%, 2% acetonitrile). Centrifugare per 3 minuti a 800 g e 25 ° C. Ripetere questa operazione una sola volta.
- 100 pl di caricare i supernatanti dalle digestioni triptici (4.9) sul C-18 StageTips e centrifugare per 3 minuti a 800 g e 25 ° C. Ripetere questa operazione fino a quando il surnatante completi sono stati applicati ale colonne.
- Lavare il C 18-StageTips con 100 microlitri Centrifuga A. Soluzione per 3 min a 800 g e 25 ° C. Ripetere questa operazione due volte.
- Eluire peptidi triptici in un nuovo 1,5 ml tubo Eppendorf con 50 microlitri Centrifuga B. Soluzione per 3 min a 800 g e 25 ° C. Ripetere questa operazione una sola volta. Peptidi secco al completamento di una velocità di vac.
- Optional: Guarnizione provette Eppendorf con parafilm e conservare a -80 ° C fino all'uso successivo.
- Risospendere i peptidi essiccate con 20 ul di soluzione A e incubare per almeno 1 ora in ghiaccio, interrotto da due a 15 min incubazioni in un bagno sonicatore. Centrifugare per 20 min a 16.100 xga 4 ° C, e applicare surnatante nano-LC-MS/MS.
5. Risultati rappresentativi
Come mostrato esemplarmente per i 14 estratti cellulari N-marcati in figura 2A, HSP70B e CGE1 sono quasi esclusivamente localizzata nella frazione solubile, indipendente dallo stato ATP. Incontrasto, CF1β è localizzato alle frazioni solubili e di membrana arricchito, come sonicazione cesoie parte dalla membrana-CF si trova o, e serve quindi come carico di controllo per entrambe le frazioni. Come mostrato nella figura 2B, quantità simili di HSP70B stati precipitati con gli anticorpi anti-HSP70B anticorpi da 14-N e 15 N-marcato estratti solubili, indipendenti dallo stato ATP. Al contrario, solo HSP70B poco è stato precipitato da frazioni di membrana con importi leggermente più grandi proveniente da ATP-depleti frazioni di membrana rispetto a frazioni ATP piene, quindi corroborando risultati precedenti 9. No CGE1 è stato co-precipitato con HSP70B in frazioni solubili o di membrana ATP-piene, mentre le grandi quantità di CGE1 sono stati co-precipitati con HSP70B da ATP-impoverito frazioni solubili, e poco da ATP-impoverito frazioni di membrana.
L'interazione di CGE1 con HSP70B solo nello stato ADP si osserva anche nelAnalisi MS: in figura 3, rappresentante MS1 spettri di HSP70B e CGE1 peptidi generati da precipitati con l'antisiero HSP70B da estratti cellulari solubili sono mostrati. Nell'esperimento mostrato in Figura 3A, sono precipitati da miscugli di 14 N-etichettati estratti privi di ATP e 15 N-marcato estratti contenenti ATP. Mentre la forma pesante e leggera etichettato del peptide HSP70B stata rilevata a intensità uguali, solo la forma della luce etichettato peptide CGE1 (da ATP-estratti) è stata trovata. In figura 3B stessi peptidi del anti-HSP70B precipitato derivato da miscele di mutuamente etichettati estratti cellulari solubili sono mostrati. Di conseguenza, questa volta solo la forma pesante etichetta del peptide CGE1 (da-ATP estratti) è stato rilevato, mentre questo era ancora il caso per entrambi, leggeri e pesanti HSP70B peptidi marcati.
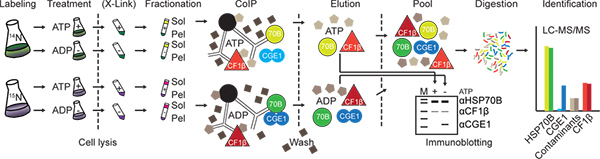
Figura 1. Sperimentali workflow. Cellule vengono marcate metabolicamente con 14 N e 15 N per almeno 10 generazioni, raccolto e fornito con o impoverito da ATP. Dopo lisi delle cellule complessi proteici può essere opzionalmente reticolato (X-link) con DSP. Cellule lisate vengono poi separati in solubile (Sol) e membrana arricchiti (Pel) frazioni. Proteine bersaglio (in questo caso HSP70B) e una proteina di controllo (qui CF1β) sono immunoprecipitati con anticorpi specifici accoppiati alla proteina A sefarosio perle (nero). Dopo il lavaggio, proteine precipitate sono eluiti e direttamente analizzati mediante immunoblotting, o le rispettive 14 e 15 N-N-etichettato frazioni in + e ATP-ATP sono stati raggruppati, digeriti e analizzati da nano-LC-MS/MS. Nel caso Nell'esempio fatto qui, la 15 frazione N-marcato è stato impoverito da ATP. Peptidi conseguenza, il rapporto di intensità di pesanti etichettati (colori scuri) alla luce etichettati (colori chiari) dalla proteina di controllo(CF1β), la proteina bersaglio (HSP70B) e non specificamente legati contaminanti dovrebbe essere intorno a uno, mentre questo rapporto dovrebbe essere molto alto per le proteine che interagiscono specificamente con la proteina target (CGE1). Clicca qui per ingrandire la figura .

Figura 2. Una analisi di input per immunoprecipitazione HSP70B. Proteina totale è stato estratto da solubile (Sol) e la membrana arricchito (Pel) frazioni sia impoverito da ATP (-ATP) o integrato con ATP e ATP un sistema di rigenerazione (+ ATP). 0,01% degli estratti proteici sono stati separati su un 10% SDS-poliacrilammide, e livelli di HSP70B e CGE1 relativa proteina di controllo CF1β carico sono stati analizzati mediante immunoblotting. Analisi B di immunoprecipitati. HSP70B stato immunoprecipitato da 14 N-e15 N-marcato solubile e di membrana arricchita di estratti cellulari contenenti o prive ATP. Le proteine corrispondenti al 3,3% del immunoprecipitati sono stati separati su un 10% di SDS-poliacrilammide gel e livelli di HSP70B e CGE1 rispetto al carico CF1β controllo sono stati analizzati mediante immunoblotting. Clicca qui per ingrandire la figura .
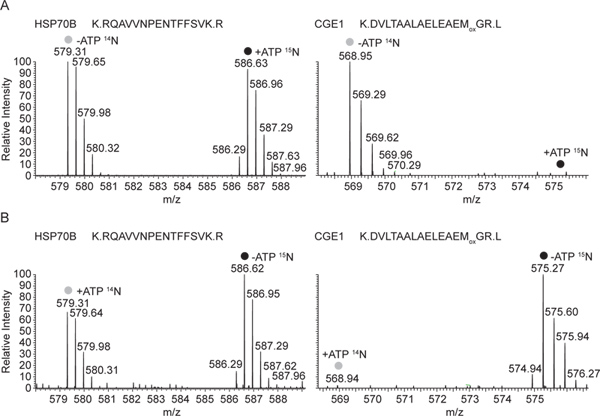
Figura 3. Un spettri di massa e rappresentativa di HSP70B CGE1 peptidi anti-HSP70B immunoprecipitati eseguita misti frazioni solubili (14 N-ATP / 15 N + ATP). Integrale spettri MS di 14 N e 15 N peptidi marcati, corrispondenti al-ATP e + ATP stati, rispettivamente, da HSP70B e co-immunoprecipitate CGE1 sono mostrati. Entrambi i peptidi sono tre volte pagano, il peptide HSP70B contiene 22 atomi di azoto, il PEPT CGE1ide 19, corrispondente ad uno spostamento di massa di 7,33 e 6,33 m / z, rispettivamente. spettri di massa rappresentativa B dall'esperimento reciproca (14 N + ATP / 15 N-ATP). Spettri MS degli stessi 14 N e 15 N peptidi marcati qui, corrispondenti alla ATP + e ATP-stati, rispettivamente, da HSP70B e co-immunoprecipitate CGE1 sono mostrati. Clicca qui per ingrandire figura .
Discussione
Abbiamo recentemente introdotto due miglioramenti del metodo VELOCE: una fase di reticolazione per catturare transitori interazioni proteina-proteina (QUICK-X), e la precipitazione di controllo di normalizzare l'efficienza precipitazioni disuguali 6. Qui vi presentiamo un protocollo contenente due miglioramenti in più di RAPIDO: in primo luogo, si sostituisce SILAC 5 per 15 N marcatura metabolica. I vantaggi sono che 15 N marcatura metabolica è molto più economico rispetto SILAC, se 15 N viene fornito come semplice sale inorganico. Inoltre, con 15 N metabolica etichettatura RAPIDO può essere applicato a prototrofici organismi per tutti gli aminoacidi, come la maggior parte delle piante, funghi e batteri. E infine, arginina-to-prolina interconversione inerente SILAC 5,6 non rappresenta un problema per la quantificazione di 15 N peptidi marcati. Esempi di strumenti idonei per la valutazione quantitativa di 15 proteomica N dati sono 12 MSQUANT o IOMIQS 13.
In secondo luogo, si introduce affinità modulazione come mezzo per specificatamente ridurre la quantità di proteine che interagiscono con una proteina bersaglio in un dato campione rispetto all'altro. I vantaggi di questo approccio sono che aggira la costruzione di mutanti knockdown, che per alcuni sistemi modello sono difficili da generare o non può essere generato affatto in caso di proteine bersaglio essenziali. Inoltre, evita fraintendimenti dovuti espressione differenziale proteine potenzialmente presenti come una risposta della cellula a bussare-giù una proteina bersaglio: se altre proteine sono down-regolati come bene e cross-reagiscono con l'antisiero utilizzato per immunoprecipitazione, verrebbero interpretati come partner vera interazione della proteina bersaglio. Finalmente, affinità modulazione sopprime la necessità di trovare un buon anticorpo antigene-to-rapporto.
Anche se applichiamo il nostro protocollo di Chlamydomonas reinhardtii come organismo modello, Può essere facilmente adattato a qualsiasi altro organismo che può essere coltivata in coltura cellulare ed è in grado di utilizzare o nitrato di ammonio come fonte di azoto. Modulazione affinità di complessi proteici da ATP / ADP può essere direttamente applicato ad altri accompagnatori cui interazione con i substrati e proteine coorte dipende dallo stato ATP, come i sistemi GroEL/HSP60/Cpn60 o HSP90 chaperone 14,15, o qualsiasi altro sistema in cui affinità di legame sono modulate da ATP. Affinità modulazione dovrebbe funzionare anche per i casi in cui sono alterati affinità tra le interazioni proteina da farmaci specifici, come radicicol geldanamicina o nel caso di sistemi Hsp90 15.
Un limite evidente di questo protocollo è che richiede purificati per affinità di anticorpi contro una proteina bersaglio noto per essere sensibili ad un trattamento specifico / farmaco che modula la sua affinità per le proteine partner. Pertanto, non è elevato throughput metodo.
Divulgazioni
Nessun conflitto di interessi dichiarati.
Riconoscimenti
Ringraziamo Olivier Vallon per l'antisiero contro CF1β. Questo lavoro è stato sostenuto dalla Max Planck Society e sovvenzioni della Deutsche Forschungsgemeinschaft (Schr 617/5-1) e il Bundesministerium für Bildung und Forschung (la biologia dei sistemi FORSYS iniziativa, GoFORSYS progetto).
Materiali
| Name | Company | Catalog Number | Comments |
| Nome del reagente | Azienda | Numero di catalogo | Commenti (opzionale) |
| Proteina Sepharose | Sigma-Aldrich | P3391 | |
| DMP (Dimetil pimelimidate) | Sigma-Aldrich | D8388 | Conservare essiccato a -20 ° C, sciogliere direttamente prima dell'uso |
| Inibitore della proteasi (completo, EDTA-free) | Roche Applied Science | 11873580001 | |
| ATP (adenosina-5'-triphosphat) | Carl Roth | K054 | |
| Creatina fosfato | Sigma-Aldrich | 27920 | |
| Fosfochinasi creatina | Sigma-Aldrich | C7886 | ; |
| DSP ditiobis [succinimidil propionato] | Sientific Thermo | 22585 | Conservare essiccato a 4 ° C |
| 15 NH 4 Cl | Cambridge isotopo laboratori, Andover, MA | NLM-467 | |
| Lys-C (endoproteinasi Lys-C) | Roche Applied Science | 11047825001 | |
| Tripsina perline (Poroszyme Immobilized tripsina) | Applied Biosystems | 2-3127-00 | Mescolare energicamente direttamente prima dell'uso |
| Empore C 18 47 millimetri disco | Varian | 12145004 |
Riferimenti
- Perrakis, A., Musacchio, A., Cusack, S., Petosa, C. Investigating a macromolecular complex: the toolkit of methods. J. Struct. Biol. 175, 106-112 (2011).
- Gavin, A. C., Maeda, K., Kuhner, S. Recent advances in charting protein-protein interaction: mass spectrometry-based approaches. Curr. Opin. Biotechnol. 22, 42-49 (2011).
- Markham, K., Bai, Y., Schmitt-Ulms, G. Co-immunoprecipitations revisited: an update on experimental concepts and their implementation for sensitive interactome investigations of endogenous proteins. Anal. Bioanal. Chem. 389, 461-473 (2007).
- Selbach, M., Mann, M. Protein interaction screening by quantitative immunoprecipitation combined with knockdown (QUICK. Nat Methods. 3, 981-983 (2006).
- Ong, S. E., Mann, M. A practical recipe for stable isotope labeling by amino acids in cell culture (SILAC). Nat Protoc. 1, 2650-2660 (2006).
- Heide, H. Application of quantitative immunoprecipitation combined with knockdown and cross-linking to Chlamydomonas reveals the presence of vesicle-inducing protein in plastids 1 in a common complex with chloroplast HSP90C. Proteomics. 9, 3079-3089 (2009).
- Nordhues, A., Miller, S. M., Muhlhaus, T., Schroda, M. New insights into the roles of molecular chaperones in Chlamydomonas and Volvox. International review of cell and molecular biology. 285, 75-113 (2010).
- Mayer, M. P., Bukau, B. Hsp70 chaperones: cellular functions and molecular mechanism. Cell Mol. Life Sci. 62, 670-684 (2005).
- Schroda, M., Vallon, O., Whitelegge, J. P., Beck, C. F., Wollman, F. A. The chloroplastic GrpE homolog of Chlamydomonas: two isoforms generated by differential splicing. The Plant Cell. 13, 2823-2839 (2001).
- Willmund, F., Schroda, M. HEAT SHOCK PROTEIN 90C is a bona fide Hsp90 that interacts with plastidic HSP70B in Chlamydomonas reinhardtii. Plant Physiol. 138, 2310-2322 (2005).
- Harris, E. H. The Chlamydomonas Sourcebook: Introduction to Chlamydomonas and Its Laboratory Use. , Elsevier, Academic Press. San Diego, CA. (2008).
- Mortensen, P. MSQuant, an open source platform for mass spectrometry-based quantitative proteomics. J. Proteome Res. 9, 393-403 (2010).
- M?hlhaus, T., Weiss, J., Hemme, D., Sommer, F., Schroda, M. Quantitative Shotgun Proteomics Using a Uniform 15N-Labeled Standard to Monitor Proteome Dynamics in Time Course Experiments Reveals New Insights into the Heat Stress Response of Chlamydomonas reinhardtii. Mol. Cell Proteomics. 10, (2011).
- Bukau, B., Horwich, A. L. The Hsp70 and Hsp60 chaperone machines. Cell. 92, 351-366 (1998).
- Wandinger, S. K., Richter, K., Buchner, J. The Hsp90 chaperone machinery. J. Biol. Chem. 283, 18473-18477 (2008).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon