Method Article
Generazione di monomero Exon1 huntingtina nativo, senza tag e fibrille usando una strategia di fusione di SUMO
In questo articolo
Riepilogo
Qui, presentiamo un protocollo robusto e ottimizzato per la produzione di quantità di milligrammo di monomeri nativi, privo di etichetta e fibrille di exon1, della proteina huntingtina (Httex1), basata sulla fusione transitoria del piccolo ubiquitina relative al modificatore (SUMO).
Abstract
Malattia di Huntington (HD) è una malattia neurodegenerative ereditato fatale causata da un'espansione CAG (≥36) nel primo esone del gene MH, conseguente l'espressione della proteina huntingtina (Htt) o N-terminale frammenti della stessa con un espanso di polyglutamine ( tratto poliQ).
La exon1 della proteina huntingtina (Httex1) è il più piccolo frammento di Htt che riassume molte delle caratteristiche di HD in cellulare e animale modelli ed è uno dei frammenti più ampiamente studiati di Htt. Le piccole dimensioni del Httex1 rendono sperimentalmente più favorevoli alla caratterizzazione biofisica utilizzando tecniche standard e ad alta risoluzione rispetto ai frammenti più lunghi o Htt full-length. Tuttavia, la propensione ad alta aggregazione del mutante Httex1 (mHttex1) con aumentata polyQ contenuto (≥42) ha reso difficile sviluppare espressione efficienti e sistemi di purificazione a produrre queste proteine in quantità sufficienti e renderli accessibili al scienziati di diverse discipline senza l'uso di proteine di fusione o altre strategie che alterano la sequenza nativa della proteina. Qui presentiamo un metodo robusto e ottimizzato per la produzione di quantità di milligrammo di nativo, tag-gratuita Httex1 basata sulla fusione transitoria del piccolo ubiquitina relative al modificatore (SUMO). La semplicità e l'efficienza della strategia eliminerà la necessità di utilizzare sequenze di non-nativi di Httex1, così rendendo questa proteina più accessibile ai ricercatori e migliorare la riproducibilità degli esperimenti in laboratori diversi. Noi crediamo che questi progressi faciliterà anche futuri studi volti a chiarire la relazione struttura-funzione di Htt, nonché sviluppare nuovi strumenti diagnostici e terapie per curare o rallentare la progressione della HD.
Introduzione
Htt è una proteina di 348 kDa ed è stato implicato in varie funzioni fisiologiche1. Quando Htt contiene una regione di polyQ espanso di più di 36 residui nella sua estremità N-terminale, che provoca HD2,3. Patologia di HD è caratterizzata da inclusioni cellulari nel corpo striato e corteccia, che conduce alla morte neuronale e l'atrofia dei tessuti colpiti4,5. Parecchi frammenti N-terminale Htt che contengono il tratto ripetere polyQ sono stati rilevati in cervelli post mortem da pazienti con MH e sono pensati per essere generati dal processamento proteolitico della proteina huntingtina6. Gli studi recenti suggeriscono che Httex1 potrebbe anche essere formato dovuto l'impionbatura del mRNA aberrante. Httex1 contiene la mutazione patologica poliQ e sua sovraespressione in animali possa ricapitolare molte delle caratteristiche chiave di HD7, evidenziando così un possibile ruolo centrale di questo frammento in HD patologia e malattia progressione6, 8,9.
A causa l'aggregazione alta propensione del mutante Httex1 (mHttex1) con tratto poliQ espanso, la maggior parte degli esistenti sistemi di espressione si basata sulla fusione transitoria di Httex1 alle proteine (come la glutatione-S-transferasi (GST), thioredoxin (TRX) o maltosio--proteina (MBP) e/o peptidi (poli-istidina) che differenzialmente migliorano l'espressione, stabilità, purificazione e/o solubilità10,11,12,13,14 ,15,16,17,18,19,20,21,22,23 ,24,25,26,27,28. Il partner di fusione è legato al Httex1 con una breve sequenza contenente un sito di clivaggio per proteasi come la tripsina, tabacco etch proteasi virus (TEV) o PreScission per consentire la scissione e il rilascio di Httex1 prima dell'inizio dell'aggregazione o purificazione. Carenze di questi metodi includono la possibilità di lasciare ulteriori residui a causa di non-traceless scissione e la creazione di frammenti troncate a causa di miscleavage all'interno della sequenza di Httex1, oltre alla eterogeneità dovuto fenditura incompleta ( Vedi Vieweg et per più approfondita discussione su vantaggi e limiti di questo approccio)10. Per risolvere queste limitazioni, abbiamo recentemente sviluppato una strategia di espressione che consente la generazione di Httex1 privo di tag nativo per la prima volta utilizzando una transitoria fusione del N-terminale di Synechocystis SP. (Ssp) DnaB intein a Httex110. Mentre il clivaggio di intein è traceless e specifici e produce quantità di mg di proteine, soffre ancora due inconvenienti che potrebbero ridurre il rendimento: vale a dire, prematuro fenditura di intein che può verificarsi durante l'espressione e il fatto che la scissione si verifica sopra parecchie ore, che potrebbero portare alla perdita di proteine a causa di aggregazione, soprattutto per Httex1 con ripetizioni di polyQ espanso.
Per affrontare queste limitazioni e di affinare la nostra strategia per la produzione di nativi, privo di tag Httex1, abbiamo sviluppato un nuovo sistema di espressione basato sulla fusione transitoria di SUMO, più esattamente il lievito dell'omologo Smt3 a Httex1. L'applicazione del sistema SUMO per la produzione di proteine ricombinanti in primo luogo è stato pubblicato nel 200429, dove è stati dimostrati un tasso aumentato di espressione e la solubilità della proteina di fusione di SUMO. Il tag SUMO può essere clivato da ubiquitina come proteine specifiche proteasi 1 (ULP1), che non richiedono un sito di riconoscimento, ma riconosce la struttura terziaria di SUMO e praticamente eliminano la possibilità di miscleavage30. Inoltre, la fenditura di ULP1-mediata è veloce e traceless e non lascia residui aggiuntivi. La scissione precoce del tag fusion, come osservato con l'autocatalitica intein10, è completamente evitata mediante la richiesta di una proteasi esterna. Mentre la strategia SUMO è oggi ampiamente usata per la produzione di proteine ricombinanti31,32,33, dimostriamo in questa carta che è particolarmente utile per la generazione di un intrinsecamente disordinati, aggregazione-inclini, proteine amiloidogeniche come Httex1. Noi crediamo che la semplicità, l'efficienza e la solidità del nostro metodo basati su SUMO-fusione renderà più accessibili ai ricercatori di diverse discipline Httex1 nativo, privo di etichetta ed eliminano la necessità di utilizzare sequenze di non-nativi del Httex1 in vitro . Si tratta di un importante passo avanti che faciliterà gli studi futuri per delucidare il rapporto struttura-funzione di Httex1.
Il protocollo descrive la purificazione di Httex1 da 12 L di coltura batterica, ma il protocollo potrebbe essere facilmente adattato per produzioni su scala più piccola o più grande. Il protocollo descrive la produzione di tipo selvaggio Httex1 (wtHttex1) con una lunghezza di ripetizione di poliQ sotto (23Q) e mutante Httex1 (mHttex1) con un polyQ ripetere lunghezza sopra (43Q) la soglia di patogena (36Q).
Protocollo
1. espressione di Httex1 ricombinante 23Q e 43Q
- Preparare i buffer richiesto e soluzioni. Preparare 1000 x soluzione stock di ampicillina (AMP, 100 mg/mL), filtro (0,2 µm), aliquota e conservare a-20 ° C. Preparare supporto di brodo (LB) di Lisogenesi (25 g LB Miller per 1 L H2O), autoclave. Preparare la soluzione di riserva di 1m isopropilico ß-D-1thiogalactopyranoside (IPTG), filtro (0,2 µm), aliquotare e negozio a-20 ° C.
- Trasformare chimica competente Escherichia coli B ER 2566 con un vettore di pTWIN1, contenente Httex1 umana fusa ad un tag N-terminale His6-SUMO con il metodo scossa di calore34.
Nota: Il ceppo di e. coli BL21 DE3 inoltre è stato utilizzato. Tuttavia, in questo caso è stata osservata una maggiore quantità di troncamenti. - Inoculare 200 mL di LB-medio con 1 x AMP in una beuta da 1 L con l'aggiunta di una singola Colonia dalla piastra di agar con una pipetta sterile. Incubare la cultura a 30 ° C e 180 rpm per 20 h (una notte) in un incubatore batterico.
- Prelevare un campione di 1 mL della coltura con una pipetta sterile. Misurare la densità ottica a 600 nm (OD600) del campione con una cuvetta di plastica usa e getta e un fotometro (rispetto la misura compresa tra 0,1 e 1, diluire con LB-medio se necessario). Calcolare la quantità di coltura che si tradurrà in una partenza OD600 di 0.05 in una cultura di 3L (con una coltura di OD600 = 3 che significherebbe 50 mL).
- Inoculare quattro culture (ogni 3 L di LB-medio con 1 x AMP in un pallone da 5 L), aggiungendo la quantità calcolata di coltura con una pipetta sterile. Incubare le colture a 37 ° C e 180 giri/min in un incubatore batterico.
- Ogni 30 min, prelevare un campione di 1 mL della coltura con una pipetta sterile. Misurare il OD600 del campione con una cuvetta di plastica usa e getta e un fotometro. Quando ha raggiunto OD600 0,1 (in genere dopo 1-2 h), impostare la temperatura dell'incubatore batterica a 14 ° C e continuare l'incubazione durante il raffreddamento. Ogni 30 min, prelevare un campione di 1 mL della coltura con una pipetta sterile. Misurare il OD600 del campione con una cuvetta di plastica usa e getta e un fotometro.
Nota: Il tempo per raffreddare le culture può variare con l'incubatore utilizzato, quindi il momento di iniziare il raffreddamento potrebbe essere necessario essere adattato a seconda del tipo di incubatore utilizzato. Tuttavia, cambiando il gradiente di temperatura dovrebbe avere solo un piccolo impatto sulla resa come la proteina di fusione di SUMO sembra essere abbastanza stabile. - Quando OD600 ha raggiunto 0.3-0.4 (in genere dopo 1-2 h), prelevare un campione di pre-induzione della cultura per analisi SDS-PAGE della sovraespressione. Calcolare la dimensione del campione che dà una quantità paragonabile di celle e un buon segnale il Coomassie macchiati SDS-PAGE: per un 10 ben gel: volume = 0,2 mL/OD600; prendere la metà di un gel di ben 15.
- Per una coltura batterica con un OD600= 0,4, prendere 500 µ l. Prendere il volume calcolato di coltura batterica con una pipetta sterile. Rotazione verso il basso del campione (18000 x g, 4 ° C, 2 min) e scartare il surnatante. Tenere il pellet a-20 ° C fino all'uso per l'analisi (punto 1.11).
- Indurre l'espressione della proteina di pipettaggio 1,2 mL di una soluzione di riserva di IPTG 1 M per ogni soluzione di cultura 3L (concentrazione finale di 0,4 mM). Continuare incubando la coltura a 14 ° C per 16 h (una notte).
Nota: La temperatura avrà in genere raggiunto ~ 20 ° C con il tempo che viene aggiunto IPTG, a seconda delle prestazioni dell'incubatore. - Prelevare un campione di post-induzione della cultura per l'analisi di SDS-PAGE di sovraespressione, seguendo la procedura descritta al punto 1.7.
- Raccogliere le cellule mediante centrifugazione in tubi di 1 L (3993 x g, 4 ° C, 10 min). Eliminare il supernatante, tenere il pellet cellulare sul ghiaccio e procedere direttamente alla purificazione.
- Analizzare la sovraespressione di SDS-PAGE35,36. Risospendere i campioni pre- e post-induzione in 20 µ l di esecuzione buffer e 20 µ l di 2 x della tintura di caricamento. Riscaldare i campioni per 5 min a 95 ° C in un blocco di calore e caricare 20 µ l su un gel di 15%, mentre è ancora caldo. Esegua il gel per 90 min a 180 V. macchia il gel con Coomassie tintura secondo le istruzioni del produttore. Confrontare i risultati con i risultati rappresentativi Figura 1C.
Nota: Il protocollo può essere fermato qui, il pellet cellulare possa essere congelato e conservato a-80 ° C per diverse settimane. Per risultati ottimali, è consigliabile utilizzare il pellet batterico fresco ed evitare il congelamento. Gelo-disgelo può portare a lisi delle cellule e la degradazione di Httex1. Questo potrebbe ridurre la resa e la qualità della proteina.
2. cellula Lisi e purificazione del suo6-proteina di fusione di SUMO Httex1 immobilizzati di cromatografia di affinità del metallo (IMAC)
- Preparare 2 L di tampone A in una bottiglia di vetro (50mm Tris (idrossimetil)-aminomethan (Tris), 500 mM NaCl, imidazolo 15 mM). Preparare 1 L di tampone B (50 mM Tris, 500 mM NaCl, pH di 500 mM imidazolo 7.4) in una bottiglia di vetro. Dopo aver sciolto i sali, regolare il pH con 10 N HCl e filtrare le soluzioni in bottiglie fresca con un filtro superiore bottiglia (0,65 µm). Preparare un fluoruro di x phenylmethylsulfonyl 1000 (PMSF, 0.3 M) soluzione, aliquota di 100 µ l e conservare a-20 ° C.
Nota: Il protocollo è progettato per consentire completato tutti i passaggi dalla lisi delle palline batteriche a fase inversa ad alte prestazioni cromatografia liquida (RP-HPLC) purificazione e liofilizzazione entro 8-9 h. Per limitare l'aggregazione e la proteolisi, è consigliabile per lavorare rapidamente senza mettere in pausa ed eseguire tutti i passaggi a 4 ° C o su ghiaccio. - Aggiungere 100 µ l di soluzione madre PMSF e cinque compresse (1 / 30 mL di volume finale) dell'inibitore delle proteasi per 100 mL di tampone pre-refrigerati r. Add il pellet batterico nel buffer e omogeneizzare la sospensione mediante agitazione con un ancoretta magnetica e pipettando su e giù per wi TH una pipetta sterile da 10 mL (~ 30 min).
- Dividere la sospensione di batteri in aliquote di 40 mL in provette di plastica usa e getta da 50 mL. Sonicare ogni aliquota in un batch di acqua/ghiaccio per lisi cellulare (70% ampiezza, tempo di sonicazione totale 5 min, intervalli di sonicazione di 30 s, 30 s pausa).
Nota: È importante che il campione non si riscalda durante il passaggio di sonicazione. Si consiglia di aggiungere un po' acqua per il bagno di ghiaccio per migliorare la dissipazione del calore durante la sonicazione. La procedura di sonicazione potrebbe dover essere adattato se viene utilizzato uno strumento diverso. Altri metodi di lisi come una pressa francese o un microfluidizer dovrebbero funzionare bene e potrebbero essere utile per evitare il riscaldamento dell'aggregazione della proteina e del campione. Questi dispositivi non erano disponibili nel nostro laboratorio e abbiamo ottenuto buoni risultati con il nostro protocollo di sonicazione. - Prelevare un campione di 50 µ l del lisato per analisi di sodio dodecil solfato del gel di poliacrilammide elettroforesi (SDS-PAGE). Centrifugare il campione (18000 x g, 4 ° C, 2 min) e dispensare la frazione solubile in una nuova provetta. Risospendere la frazione insolubile in 50 µ l di tampone A con una pipetta. Mantenere i campioni su ghiaccio fino all'analisi SDS-PAGE (punto 2.6).
- Chiarire il lisato mediante centrifugazione (39191 x g, 4 ° C, 60 min).
- Durante la fase di centrifugazione, analizzare il passo di lisi delle cellule da SDS-PAGE. Aggiungere 50 µ l di 2 x tintura alla frazione solubile e insolubile del lisato di carico rispettivamente. Calore per 5 min a 95 ° C e carico 2 µ l su un gel di 15%, mentre è ancora caldo. Esegua il gel per 90 min a 180 V. macchia il gel con Coomassie tintura secondo le istruzioni del produttore. Confrontare i risultati con i risultati rappresentativi Figura 1C.
- Filtrare il surnatante (0,45 µm, filtri per siringa). Prelevare un campione di 20 µ l del surnatante filtrato per analisi di SDS-PAGE (punto 2.11).
Nota: In genere, si ottiene un volume di 90 a 100 mL di surnatante chiarificato e filtrato. In genere, 3 filtri per siringa sono sufficienti. Se la filtrazione è ingombrante, provare ad aumentare la velocità di centrifugazione e/o tempo. - Isolare la proteina di fusione His6-SUMO Httex1 da Lisato batterico chiarificato mediante cromatografia di affinità del metallo immobilizzato (IMAC) su un sistema di cromatografia in fase liquida (FPLC) prestazioni veloci a 4 ° C37.
- Riempire il chiarificato lisato in un superloop e il carico sulla colonna Ni-NTA (spogliato, puliti e ricaricato secondo il manuale del produttore, un "bianco" precedente è consigliato) a 2 mL/min passano 10 volumi di colonna (CV, 200 mL) di tampone A 10 mL/min per lavare fuori le proteine non associate.
- Eluire la proteina di fusione con 2.5 CV (50 mL) del 100% di tampone B a 2 mL/min utilizzano una dimensione di frazione di 50 mL per il carico e lavaggio e 5 mL per l'eluizione. Confrontare i risultati con i risultati rappresentativi in Figura 1,D.
- Prelevare un campione di ciascuna frazione per analisi di SDS-PAGE (20 µ l) e la piscina le frazioni contenenti la proteina di fusione secondo il picco del cromatogramma IMAC. Aggiungere (2S, 3S) - 1,4 - Bis (sulfanyl) butano-2,3-diolo (DTT) e L-cisteina (concentrazione finale di 100 mM ciascuno) come polvere e sciogliere delicatamente capovolgendo la provetta.
Nota: Nella nostra esperienza la purezza della proteina di fusione nelle diverse frazioni è paragonabile. Come misura precauzionale, le frazioni della proteina di fusione purificata dovrebbero essere riunite rapidamente dopo IMAC per impedire l'aggregazione delle frazioni altamente concentrate. Inoltre, è consigliabile procedere direttamente per la fenditura del tag di SUMO e la purificazione di HPLC. Se necessario, il protocollo può essere fermato qui. La soluzione diluita della proteina di fusione è stata congelata in azoto liquido, conservata a-80 ° C e purificata dopo lo scongelamento senza una riduzione significativa del rendimento. Conservazione della soluzione diluita della proteina di fusione a 4 ° C per 24 h anche dà risultati simili. - Analizzare l'IMAC da SDS-PAGE. Aggiungere 20 µ l di colorante per ciascun campione di carico. Caricare 2 µ l di materiale grezzo (2,7), la frazione non legata, la frazione di lavare e ciascuna frazione del picco di eluizione su un gel di 15%. Esegua il gel per 90 min a 180 V. macchia il gel con Coomassie tintura secondo le istruzioni del produttore. Confrontare i risultati con i risultati rappresentativi in Figura 1,D.
3. la scissione della sua6-SUMO-tag e purificazione HPLC
Attenzione: L'acido trifluoroacetico (TFA) è un liquido volatile e causa gravi ustioni possono così gestire con cura. Effettuare tutte le manipolazioni in una cappa aspirante e indossare adeguati dispositivi di protezione individuale (cioè, guanti monouso in nitrile, occhiali di sicurezza e un camice da laboratorio).
- In una bottiglia da 5 L, aggiungere 5 mL di TFA con una siringa di plastica da 5 L di acqua (solvente a: H2O, 0.1% (TFA). Aggiungere 2,5 mL di TFA con una siringa di plastica in una bottiglia da 2,5 L di acetonitrile (solvente b: acetonitrile, 0,1% TFA).
- Preparare il sistema HPLC come suggerito dal produttore. Eseguire un'esecuzione in bianco per garantire una colonna pulita.
- Prelevare un campione di 100 µ l della proteina di fusione prima dell'aggiunta di ULP1 per monitorare la reazione di scissione da UPLC (punto 3.5).
- Trasferire 20 mL della proteina di fusione in un nuovo tubo da 50 mL e aggiungere 0,4 mL di soluzione di riserva di ULP1, Incubare in ghiaccio. Mantenere la proteina di fusione rimanenti sul ghiaccio.
Nota: Il His-Tag catalitico frammento 403-621 della proteasi Ubiquitin-like-specifico 1 (qui denominato "ULP1") venne utilizzato per fendere il tag SUMO. La proteina di fusione è più stabile rispetto al Httex1 spaccati. Si raccomanda di non fendere il tag di SUMO dell'intero batch. Al contrario, continuare con aliquote di dimensioni che possono essere applicate direttamente e completamente alla colonna HPLC. - Ogni 10 minuti, prelevare un campione di 100 µ l del clivaggio per monitorare l'avanzamento di ultra-prestazioni cromatografia liquida (UPLC). Centrifugare i campioni (18000 giri/min, 4 ° C, 2 min) e analizzare 2 µ l del surnatante da UPLC (pendenza dal 10% al 90% solvente B nella A 0.25 a 3 min, 10% B per 1 min, consultare le istruzioni del produttore per l'utilizzo dello strumento). Paragonare i cromatogrammi ottenuti per il campione prima dell'aggiunta di ULP1 e i campioni prelevati. Confrontare i risultati con i risultati rappresentativi Figura 2B.
- Una volta completata la scissione di SUMO (il picco della proteina di fusione è scomparso nel cromatogramma UPLC e viene completamente convertito al picco di SUMO e Httex1 appena apparsa), filtrare il campione con un filtro per siringa (0,22 µm).
Nota: Il clivaggio SUMO è in genere molto veloce (10-20 min a 4 ° C) quindi analisi UPLC con un tempo di funzionamento di 4 min è uno strumento prezioso per monitorare la reazione. Filtrare il campione prima di HPLC purificazione è principalmente una misura preventiva per aumentare la durata della colonna. Il campione non dovrebbe girare torbido. - Purificare il campione filtrato da RP-HPLC (gradiente di 25-35% solvente B in solvente A, oltre 40 min a 15 mL/min (0-10 min: 5%; 10-12.5 min: dal 5 al 25%; 12,5-52,5 min: 25 al 35%; 52,5-57,5 min 35 e 95%; consultare le istruzioni del produttore per l'utilizzo dello strumento). Confrontare i risultati con i risultati rappresentativi Figura 2C.
Nota: Httex1 e sua6-SUMO separato anche da RP-HPLC. Tuttavia, ci possono essere piccole quantità di Httex1 troncato all'inizio e la fine del picco. Raccogliere piccole frazioni per ottenere la massima quantità di materiale puro.
Attenzione: Utilizzare le attrezzature di sicurezza adeguati (cioè, camice da laboratorio, guanti isolanti e uno schermo facciale) quando movimentazione di fluidi criogenici. - Analizzare l'HPLC-frazioni di spettrometria totale di ionizzazione di electro-spray (ESI-MS, autocampionatore, iniettare 10 µ l, portata 0,6 mL/min, solvente: 20% B in A, nessuna colonna, consultare le istruzioni del produttore per l'utilizzo dello strumento) e UPLC (pendenza dal 10% al 90% solvente B nella A per 0.25 a 3 min, 10% B per 1 min, consultare le istruzioni del produttore per l'utilizzo dello strumento). Piscina le frazioni di purezza in provette in plastica da 50 mL, congelato in azoto liquido e lyophilize. Pesare e trasferire la proteina liofilizzata in provette in plastica da 2 mL e conservare a-20 ° C.
- Caratterizzare il materiale purificato da UPLC, ESI-MS e SDS-PAGE. Sciogliere 100 µ g di liofilizzato Httex1 in 8 µ l di TFA ordinata in una provetta da 1,5 mL e incubare per 20 minuti a temperatura ambiente nella provetta chiusa. Attentamente evaporare il TFA sotto cappa aspirante con un flusso di azoto o argon. Utilizzare bassa pressione di azoto/argon per evitare la perdita del campione.
- Sciogliere la proteina in 100 µ l di H2O. analizzare 2 µ l di UPLC e 5 µ l di ESI-MS come descritto al punto 3.8. Mix 20 µ l di soluzione proteica con 20 µ l di 2 x della tintura di caricamento.
- Analizzare la quantità di 1 µ g a 10 µ g di SDS-PAGE. Esegua il gel per 90 min a 180 V. macchia il gel con Coomassie tintura secondo le istruzioni del produttore. Confrontare i risultati con i risultati rappresentativi Figura 2D.
4. disaggregazione e Resolubilization delle proteine Httex1
Attenzione: TFA è un liquido volatile e causa gravi ustioni possono così gestire con cura. Effettuare tutte le manipolazioni in una cappa aspirante e indossare adeguati dispositivi di protezione individuale (cioè guanti monouso in nitrile, occhiali di sicurezza e un camice da laboratorio).
- Preparare 10 mL di soluzione salina tamponato fosfato di Dulbecco (DPBS) (137 mM NaCl, 2.7 mM KCl, 10mm Na2HPO4, 2mm KH2PO4, pH 7.4) dalla polvere premiscelata in un tubo da 50 mL. Filtrare la soluzione DPBS attraverso un filtro da 0,2 µm prima di ogni utilizzo.
- Sciogliere 150 µ g di liofilizzato Httex1 in 12 µ l di TFA ordinata in una provetta da 1,5 mL e incubare per 20 minuti a temperatura ambiente nella provetta chiusa. Attentamente evaporare il TFA sotto cappa aspirante con un flusso di azoto o argon. Utilizzare bassa pressione di azoto/argon per evitare la perdita del campione38.
Nota: In generale, utilizzare 4 µ l TFA per sciogliere e disaggregare 50 µ g di proteina. Questa procedura consente di creare un film della proteina all'interno pareti della provetta. Per impedire l'aggregazione immediata di Httex1 nei passaggi seguenti, lavorare con buffer pre-raffreddata, mantenere la proteina sempre su ghiaccio ed evitare alte concentrazioni. - Sciogliere la proteina disaggregata in 1 mL di DPBS pre-raffreddato e regolare il pH a 7,2-7,4 con 1 M NaOH. Filtrare la soluzione della proteina attraverso un filtro centrifugo 100 kDa in tubi di plastica da 1,5 mL (20000 x g, 4 ° C, 20 minuti).
Nota: La concentrazione teorica calcolata di Httex1 è superiore alla concentrazione finale desiderata per tenere conto di possibili perdite. Il passo di filtrazione è necessario rimuovere qualsiasi aggregazione che potrebbero essersi formata durante la dissoluzione della proteina. - Determinare la concentrazione di Httex1 mediante una curva di calibrazione UPLC basata su analisi dell'amminoacido (rilevazione a λ214) e invia 2 µ g di proteina per l'analisi dell'amminoacido per convalidare la concentrazione10. Calcolare la quantità di DPBS che deve essere aggiunto per ottenere una concentrazione di 3 µM Httex1.
- Diluire la proteina a 3 µM aggiungendo la quantità calcolata di DPBS alla provetta. Tenere il tubo sul ghiaccio fino all'inizio del protocollo di aggregazione.
Nota: Httex1 43Q non devono essere conservati in soluzione. Sempre preparare una soluzione di proteine fresco basata sul protocollo di cui sopra. Httex1 proteine sono meglio conservati come una polvere liofilizzata a-20 ° C.
5. monitoraggio della cinetica di aggregazione di Httex1 43Q UPLC e dicroismo circolare (spettroscopia) e caratterizzazione degli aggregati di microscopia elettronica in trasmissione (TEM)
- Preparare soluzione di formiato di uranile per TEM come precedentemente segnalati39.
- Avviare l'aggregazione di Httex1 43Q incubando una soluzione di 3 µM in DPBS a 37 ° C (uso 1 mL di soluzione preparata come descritto nel protocollo di disaggregazione).
Nota: Può essere eseguita l'aggregazione di Httex1 alle più alte concentrazioni a seconda delle esigenze e finalità dell'esperimento. - Quantificare la quantità di proteina solubile utilizzando UPLC intervalli di tempo indicati (a 0, 1, 2, 4, 6, 8, 12, 24, 48 e 120 h). Per fare questo, prendete un'aliquota del 35 µ l e rimuovere gli aggregati insolubili mediante centrifugazione (20000 x g, 4 ° C, 20 min). Iniettare 4 µ l del surnatante il UPLC. Calcolare la percentuale di monomero solubile basato sul cambiamento della zona di picco utilizzando lo strumento software 40. Confrontare i risultati con i risultati rappresentativi in Figura 3A.
- Caratterizzano i cambiamenti nella struttura secondaria usando la spettroscopia CD a 0 e 48 h. Prendere un'aliquota di 100 µ l e misurare l'ellitticità (cuvette in quarzo di 1 mm, 195 nm a 250 nm, 20 ° C, dati punti ogni 0,2 nm, velocità 10 nm/min, integrazione digitale tempo 2 s, larghezza di banda pari a 1,0 nm). Acquisire 6 spettri del campione e media e liscia con un filtro binomio con larghezza di convoluzione di 99. Tracciare gli spettri come il residuo medio ellitticità molare (θMRE)41. Confrontare i risultati con i risultati rappresentativi Figura 3B.
- Caratterizzare le proprietà morfologiche e strutturali degli aggregati di TEM. Mettere 3 µ l della soluzione della proteina su una Formvar/carbonio-rivestito 200-mesh, Scarica a bagliore rame griglia per 1 min. lavare la griglia due volte con acqua μL 15, una volta con 15 μL di formiato di uranile 0,7% (p/v) e la macchia per 30 s con 15 μL di formiato di uranile 0,7% w/v. Eseguire un'analisi TEM delle griglie. Confrontare i risultati con i risultati rappresentativi nella Figura 3C.
Risultati
Httex1 è espressa in e. coli con un N-terminale sua6-tag SUMO. I risultati rappresentativi della espressione e purificazione della proteina di fusione sono riassunti nella Figura 1. La sequenza di Httex1 è costituito da residui di 2-90 di Htt e inizia con Ala2, perché Met1 è completamente spaccati in vivo42. La numerazione degli aminoacidi si riferisce alla variante 23Q, la sequenza completa della proteina di fusione espressa è mostrata in Figura 1A. I plasmidi saranno depositati presso Addgene nel prossimo futuro da condividere con la Comunità. Un disegno schematico del plasmide e la proteina di fusione espressa è illustrato nella Figura 1B. His6-SUMO Httex1 esprime a un livello medio (Figura 1,C) e la maggior parte della proteina di fusione è presente nella frazione solubile dopo lisi, sia per il 23Q e la variante 43Q. La proteina di fusione migra più in alto del previsto, sulla base del peso molecolare. Questo è parzialmente dovuto la piega forte di SUMO, ma principalmente a causa della composizione insolita sequenza di Httex1, contenente principalmente residui di glutamina e prolina. Il mutante (43Q) e il wildtype (23Q) proteina di fusione può essere arricchita di ~ 80% purezza da IMAC (Figura 1D) la presenza di co-purificazione proteina ospite può essere spiegata dal livello comparativamente bassa espressione di Httex1 e il grande campione volume applicato alla colonna.
La scissione del suo6-tag di SUMO e la purificazione di Httex1 è illustrato nella Figura 2A. UPLC è uno strumento efficace per monitorare la scissione del suo6-tag SUMO (Figura 2B). Il picco originale della proteina di fusione è consumato e due picchi di nuovi e ben separati, corrispondente al suo6-SUMO tag e Httex1 vengono visualizzati. La reazione di scissione è rifinita in 10-20 min. Il Western Blot (WB) è troppo lento per monitorare in modo efficiente la reazione di scissione, ma è stato incluso nella figura per riferimento e per dimostrare la completezza del clivaggio SUMO. Entrambi Httex1 23Q e 43Q possono essere separati anche dal suo6-SUMO tag da RP-HPLC (Figura 2C) e sono stati ottenuti a elevata purezza, come indicato dall'analisi UPLC, MS e SDS-PAGE (Figura 2D).
Per illustrare che le proteine Httex1 preparate da questo metodo mantengono le proprietà di aggregazione previsto di Httex1, abbiamo valutato la cinetica di fibrillazione del mutante Httex1 a 37 ° C da un'analisi di sedimentazione, monitorare i cambiamenti nella struttura secondaria di CD spettroscopia e caratterizza la morfologia degli aggregati di TEM. Un set di dati rappresentativo delle cinetiche di aggregazione di formazione di fibrille di mHttex1 come determinato mediante un test di sedimentazione è mostrato inFigura 3A. La perdita di sostanza solubile Httex1 43Q nel tempo, a causa di formazione della fibrilla è stata quantificata tramite UPLC. Osserviamo un completo depauperamento delle proteine solubili dopo 48 ore di incubazione. Inoltre, abbiamo determinato la struttura secondaria della proteina di spettroscopia CD (Figura 3B). Httex1 43Q turni da non strutturati (λmin 205 nm) alla conformazione ricca soprattutto β-foglio (λmin 215 nm) dopo 48 ore di incubazione. Questo cambiamento strutturale è accompagnato dalla formazione di aggregati fibrillari lungo come osservabile da TEM a 48 ore (Figura 3C).
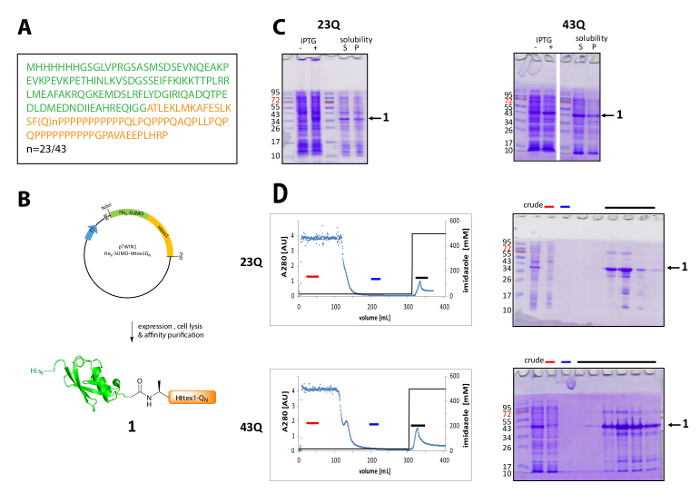
Figura 1 . Espressione e purificazione del suo6-proteina di fusione di SUMO Httex1.
(A), la sequenza aminoacidica del suo6-SUMO-Httex1-QN fusione costrutti (sua6-SUMO in verde e Httex1-QN in arancione); (B) descrizione schematica dell'espressione e purificazione della proteina di fusione; (C) analisi SDS-PAGE dell'espressione e la solubilità della proteina di fusione dopo la lisi; (D) cromatogramma della IMAC purificazione della proteina di fusione e analisi delle frazioni da SDS-PAGE (barra rossa: barra di frazione non legata, blu: frazione di lavaggio, barra nera: le frazioni contenenti il picco di eluizione); Clicca qui per visualizzare una versione più grande di questa figura.

Figura 2 . Fenditura del suo6 SUMO tag e purificazione delle proteine di Httex1-QN privo di etichetta.
Descrizione schematica (A); (B) analisi di fenditura del tag SUMO con ULP1 di UPLC (blu: prima dell'aggiunta della ULP1; nero: 20 min (23Q), rispettivamente 10 min (43Q) dopo l'aggiunta di ULP1) e WB (MAB5492 1: 2000, secondario goat anti-mouse anticorpo 1: 5000); (C) cromatogramma della purificazione RP-HPLC preparativa di Httex1; D: analisi della Httex1 purificata da UPLC, SDS-PAGE ed ESI-MS; il peso molecolare previsto è Da 9943 (23Q) e Da 12506 (43Q) rispettivamente. Clicca qui per visualizzare una versione più grande di questa figura.
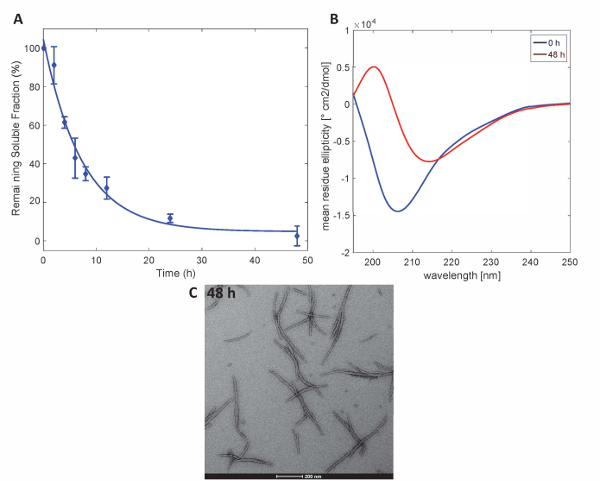
Figura 3 . Aggregazione di Httex1-43Q: saggio di sedimentazione (A) basato su UPLC. (B) spettri CD della struttura secondaria a 0 h e micrografie TEM 48 h. (C) degli aggregati a 48 h (barre della scala sono 200 nm). Clicca qui per visualizzare una versione più grande di questa figura.
Discussione
In questo protocollo, abbiamo delineato una procedura efficace per ottenere quantità di milligrammo di nativo, senza tag Httex1 contenenti residui di glutamina 23 o 43. Questo è stato ottenuto esprimendo Httex1 come una fusione di C-terminale a un suo6-tag SUMO, che viene utilizzato per isolare la proteina di fusione dalla cella lysata di IMAC e viene scisso prima purificazione HPLC di Httex1. Mentre la strategia SUMO è stata utilizzata nella produzione di parecchie altre proteine, il nostro metodo indica che le proprietà uniche di che Sumo potrebbe essere utilizzato anche per generare intrinsecamente disordinata, aggregazione-inclini, proteine amiloidogeniche che precedentemente hanno dimostrato di essere estremamente difficile da gestire e produrre43,44. Vi presentiamo un protocollo che è semplice, facile da usare e paragonabile a un protocollo per la generazione di una proteina "ben educata". La fusione di SUMO solubilizza e stabilizza Httex1 durante l'espressione e la fase di purificazione di IMAC. Scissione precoce del tag, come osservato con il intein strategia10 e l'aggregazione non erano più un problema.
Proteine intrinsecamente disordinati sono particolarmente vulnerabili alla degradazione. Mentre la degradazione del N-terminale della regione N17 non è un problema usando questo protocollo, possono verificarsi troncamenti in PRD di Httex1. Come le proteine tronche sono molto simile a Httex1 di idrofobicità, carica e dimensione, rimuoverli con mezzi cromatografici è impegnativo, quindi è meglio prevenire la loro formazione in primo luogo. Attenersi strettamente al protocollo, sempre lavorando sul ghiaccio e utilizzando una quantità sufficiente di inibitore della proteasi dovrebbe aiutare a mantenere molto basso il livello di troncamento osservato. L'applicazione di un tag di fusione al C-terminale di Httex1 potrebbe rimuovere troncamenti in PRD facilmente come la proteina tronca perderebbe anche il tag di affinità. Tuttavia, se la sequenza nativa deve essere mantenuto che questa opzione non può essere applicata come Httex1 termina con prolina e al meglio della nostra conoscenza non ci sono nessun tag di fusione di C-terminale che è noti per indurre il clivaggio traceless ed efficiente dopo prolina.
La parte più critica del protocollo è la gestione della Httex1 liberata dopo la scissione del tag SUMO da ULP1. La proteina deve essere purificata immediatamente da RP-HPLC. Fortunatamente, questa è una reazione efficiente e veloce che di solito è completata in 10-20 min a 4 ° C. Al contrario, la strategia di intein richiesto diverse ore per la scissione completa di intein, richiedendo così un compromesso tra la fenditura incompleta e inizio aggregazione al fine di massimizzare il rendimento. Un workup veloce è necessario per mutante Httex1, come si inizierà ad aggregare alla concentrazione relativamente alta presente nella reazione di scissione, mentre la variante 23Q è stabile per un tempo più lungo. Durante la purificazione di RP-HPLC, un altro vantaggio di SUMO diventa evidente: mentre il Ssp DnaB intein è idrofobo e bastoni fortemente alla colonna, SUMO è più idrofilo ed eluisce completamente dalla colonna a fase inversa C4. Anche se ULP1 commerciale è piuttosto costoso, la proteina può essere prodotto facilmente nell'alto rendimento seguendo protocolli precedentemente pubblicati29.
L'importanza critica di applicare un protocollo di disaggregazione prima utilizzando Httex1 non può essere sottolineato abbastanza. Liofilizzato polyQ proteine quali Httex1 sono stabili e possono essere memorizzati lunghi periodi, ma non sono completamente solubili in acqua e buffer. La presenza di oligomeri preformati o fibrille potrebbe avere un impatto significativo sulla cinetica di aggregazione e proprietà biofisiche del proteina45. Il protocollo di disaggregazione descritto qui permette la disgregazione della proteina, rimozione degli aggregati preformati e generazione di una soluzione di Httex1 monomerico da un campione di liofilizzato. Abbiamo osservato la simile morfologia di cinetica e fibrilla di aggregazione per Httex1 ottenuti con il SUMO e la strategia di intein.
Rispetto ai precedenti metodi per la produzione di Httex1, la strategia SUMO descritta qui offre diversi vantaggi e si espande la gamma di possibili studi per indagare la struttura e le proprietà funzionali di questa proteina nella salute e nella malattia. La proteina di fusione di SUMO-Httex1 è facile da gestire, può essere congelato e memorizzato o mantenuto in soluzione per 24 h a temperatura ambiente, mentre il mHttex1 gratuito sarebbe aggregare rapidamente. La stabilità e l'alta solubilità delle proteine di fusione di SUMO-Httex1 offrono una maggiore flessibilità per modificare la proteina e/o introdurre modifiche enzimatiche e chimiche in mHttex1 che altrimenti non sarebbe possibile dopo fenditura. Ciò comprende l'introduzione di modificazioni post-traduzionali, fluorofori, spin etichette, tag di biotina, ecc i progressi presentati qui dovrebbero 1) facilitare futuri studi per delucidare la relazione struttura-funzione di Httex1; 2) generare nuovi strumenti per studiare l'aggregazione Htt e patologia diffusione; 3) consentire lo sviluppo di nuovi test per identificare molecole che stabilizzano mutante Httex1 e prevenire la sua aggregazione; e 4) incoraggiare gli scienziati di altri campi per portare a lavorare su questa proteina e unire la nostra ricerca per trovare cure per la malattia di Huntington.
Divulgazioni
Gli autori dichiarano di non avere conflitti di interesse con il contenuto di questo articolo.
Riconoscimenti
Questo lavoro è stato finanziato principalmente da sovvenzioni dalla fondazione CHDI e Swiss National Science Foundation. Ringraziamo Dr. Sophie Vieweg per utili discussioni durante lo sviluppo di questo nuovo sistema di espressione e altri membri del gruppo Lashuel per condividere la loro esperienza con questo sistema di espressione e per il loro input e prezioso feedback. Ringraziamo anche la Prof. ssa Oliver Hantschel per fornire il plasmide ULP1. Gli autori ringraziano il Dr. John B. Warner e Dr. Senthil K. Thangaraj per revisione critica del manoscritto
Materiali
| Name | Company | Catalog Number | Comments |
| Uranyl formate (UO2(CHO2)2) | EMS | 22450 | |
| Formvar/carbon 200 mesh, Cu 50 grids | EMS | FCF200-Cu-50 | |
| High Precision Cell made of Quartz SUPRASIL 1 mm light path from Hellma Analytics | HellmaAnalytics | 110-1-40 | |
| Buffer Substance Dulbecco's (PBS w/o Ca and Mg) ancinne ref 47302 (RT) SERVA | Witech | SVA4730203 | |
| Ampicillin | AxonLab | A0839.0100 | |
| Luria Broth (Miller's LB Broth) | Chemie Brunschwig | 1551.05 | |
| Isopropyl β-D-1-thiogalactopyranoside (IPTG) | AxonLab | A1008.0025 | |
| E. coli B ER2566 | NEB | NEB# E4130 | |
| Imidazole | Sigma | 56750-500G | |
| cOmplete Protease Inhibitor Cocktail | Roche | 4693116001 | |
| Anti-Huntingtin Antibody, a.a. 1-82 | Merck Millipore Corporation | MAB5492 | |
| IRDye 680RD Goat anti-Mouse IgG (H + L) | Licor | 925-68070 | |
| PMSF | AxonLab | A0999.0005 | |
| HisPrep 16/10 column | GE Healthcare | 28936551 | |
| C4 HPLC column | Phenomenex | 00G-4168.P0 | 10 µm C4 300 Å, LC Column 250 x 21.2 mm, Phenomenex, 19x10 mm guard column, not temperature jacketed |
| Acetonitrile HPLC | MachereyNagel | C2502 | |
| Filtre seringue Filtropur S 0,45 ul sans prefiltre sterile | Sarstedt AG | 83.1826 | |
| Spectrophotometer semi-micro cuvette | Reactolab S.A. | 2534 | |
| Superloop, 1/16" fittings (ÄKTAdesign), 50 ml | GE Healthcare | 18111382 | |
| Trifluoroacetic acid | Sigma | 302031 | |
| GREINER Tubes fo FPLC 16 x 100 mm, cap. 12.0 ml | Greiner Bio-One | 7.160 102 | |
| 100 kD Microcon fast flow filters | Merck Millipore Corporation | MRCF0R100 | |
| Vibra-cell VCX130 ultrasonic liquid processor | Sonics | ||
| Äkta 900 equipped with a fraction collector | GE Healthcare | ||
| Jasco J-815 Circular Dichroism | Jasco | ||
| Waters UPLC system | Waters | C8 BEH acquity 2.1x100 mm 1.7 micron column , preheated column (40 °C), flow rate of 0.6 mL/min, injection volume of 4 μL | |
| waters HPLC system | Waters | 2489 UV detector and 2535 quaternary gradient module, 20 mL loop in a FlexInject housing | |
| ESI-MS: Finnigan LTQ | Thermo Fisher Scientific | ||
| lyophylizer instrument | FreeZone 2.5 Plus | ||
| Tecnai Spirit BioTWIN | FEI | electron microscope equipped with a LaB6 gun and a 4K x 4K FEI Eagle CCD camera (FEI) and operated at 80 kV | |
| 37 °C shaking incubator | Infors HT multitron Standard | ||
| Biophotometer plus | Eppendorf |
Riferimenti
- Saudou, F., Humbert, S. The Biology of Huntingtin. Neuron. 89 (5), 910-926 (2016).
- MacDonald, M. E., Gines, S., Gusella, J. F., Wheeler, V. C. Huntington's disease. Neuromolecular Medicine. 4 (1-2), 7-20 (2003).
- Li, S., Li, X. J. Multiple pathways contribute to the pathogenesis of Huntington disease. Molecular Neurodegeneration. 1, 19 (2006).
- DiFiglia, M. Aggregation of Huntingtin in Neuronal Intranuclear Inclusions and Dystrophic Neurites in Brain. Science. 277 (5334), 1990-1993 (1997).
- Atwal, R. S., et al. Huntingtin has a membrane association signal that can modulate huntingtin aggregation, nuclear entry and toxicity. Human Molecular Genetics. 16 (21), 2600-2615 (2007).
- Sathasivam, K., et al. Aberrant splicing of HTT generates the pathogenic exon 1 protein in Huntington disease. Proceedings of the National Academy of Sciences U S A. 110 (6), 2366-2370 (2013).
- Mangiarini, L., et al. Exon 1 of the HD gene with an expanded CAG repeat is sufficient to cause a progressive neurological phenotype in transgenic mice. Cell. 87 (3), 493-506 (1996).
- El-Daher, M. T., et al. Huntingtin proteolysis releases non-polyQ fragments that cause toxicity through dynamin 1 dysregulation. EMBO Journal. 34 (17), 2255-2271 (2015).
- Lunkes, A., et al. Proteases acting on mutant Huntingtin generate cleaved products that differentially build up cytoplasmic and nuclear inclusions. Molecular Cell. 10 (2), 259-269 (2002).
- Vieweg, S., Ansaloni, A., Wang, Z. M., Warner, J. B., Lashuel, H. A. An Intein-based Strategy for the Production of Tag-free Huntingtin Exon 1 Proteins Enables New Insights into the Polyglutamine Dependence of Httex1 Aggregation and Fibril Formation. Journal of Biological Chemistry. 291 (23), 12074-12086 (2016).
- Georgalis, Y., et al. Huntingtin aggregation monitored by dynamic light scattering. Proceedings of the National Academy of Sciences U S A. 95 (11), 6118-6121 (1998).
- Scherzinger, E., et al. Huntingtin-encoded polyglutamine expansions form amyloid-like protein aggregates in vitro and in vivo. Cell. 90 (3), 549-558 (1997).
- Scherzinger, E., et al. Self-assembly of polyglutamine-containing huntingtin fragments into amyloid-like fibrils: implications for Huntington's disease pathology. Proceedings of the National Academy of Sciences U S A. 96 (8), 4604-4609 (1999).
- Muchowski, P. J., et al. Hsp70 and hsp40 chaperones can inhibit self-assembly of polyglutamine proteins into amyloid-like fibrils. Proceedings of the National Academy of Sciences U S A. 97 (14), 7841-7846 (2000).
- Heiser, V., et al. Inhibition of huntingtin fibrillogenesis by specific antibodies and small molecules: implications for Huntington's disease therapy. Proceedings of the National Academy of Sciences U S A. 97 (12), 6739-6744 (2000).
- Bennett, E. J., Bence, N. F., Jayakumar, R., Kopito, R. R. Global impairment of the ubiquitin-proteasome system by nuclear or cytoplasmic protein aggregates precedes inclusion body formation. Molecular Cell. 17 (3), 351-365 (2005).
- Tam, S., et al. The chaperonin TRiC blocks a huntingtin sequence element that promotes the conformational switch to aggregation. Nature Structural & Molecular Biology. 16 (12), 1279-1285 (2009).
- Nekooki-Machida, Y., et al. Distinct conformations of in vitro and in vivo amyloids of huntingtin-exon1 show different cytotoxicity. Proceedings of the National Academy of Sciences U S A. 106 (24), 9679-9684 (2009).
- Wacker, J. L., Zareie, M. H., Fong, H., Sarikaya, M., Muchowski, P. J. Hsp70 and Hsp40 attenuate formation of spherical and annular polyglutamine oligomers by partitioning monomer. Nature Structural & Molecular Biology. 11 (12), 1215-1222 (2004).
- Legleiter, J., et al. Monoclonal antibodies recognize distinct conformational epitopes formed by polyglutamine in a mutant huntingtin fragment. Journal of Biological Chemistry. 284 (32), 21647-21658 (2009).
- Legleiter, J., et al. Mutant huntingtin fragments form oligomers in a polyglutamine length-dependent manner in vitro and in vivo. Journal of Biological Chemistry. 285 (19), 14777-14790 (2010).
- Nucifora, L. G., et al. Identification of novel potentially toxic oligomers formed in vitro. from mammalian-derived expanded huntingtin exon-1 protein. Journal of Biological Chemistry. 287 (19), 16017-16028 (2012).
- Dahlgren, P. R., et al. Atomic force microscopy analysis of the Huntington protein nanofibril formation. Nanomedicine. 1 (1), 52-57 (2005).
- Poirier, M. A., et al. Huntingtin spheroids and protofibrils as precursors in polyglutamine fibrilization. Journal of Biological Chemistry. 277 (43), 41032-41037 (2002).
- Duim, W. C., Chen, B., Frydman, J., Moerner, W. E. Sub-diffraction imaging of huntingtin protein aggregates by fluorescence blink-microscopy and atomic force microscopy. Chemphyschem. 12 (13), 2387-2390 (2011).
- Pieri, L., Madiona, K., Bousset, L., Melki, R. Fibrillar alpha-synuclein and huntingtin exon 1 assemblies are toxic to the cells. Biophysical Journal. 102 (12), 2894-2905 (2012).
- Monsellier, E., Redeker, V., Ruiz-Arlandis, G., Bousset, L., Melki, R. Molecular interaction between the chaperone Hsc70 and the N-terminal flank of huntingtin exon 1 modulates aggregation. Journal of Biological Chemistry. 290 (5), 2560-2576 (2015).
- Isas, J. M., Langen, R., Siemer, A. B. Solid-State Nuclear Magnetic Resonance on the Static and Dynamic Domains of Huntingtin Exon-1 Fibrils. Biochemistry. 54 (25), 3942-3949 (2015).
- Malakhov, M. P., et al. SUMO fusions and SUMO-specific protease for efficient expression and purification of proteins. Journal of Structural Function Genomics. 5 (1-2), 75-86 (2004).
- Mossessova, E., Lima, C. D. Ulp1-SUMO crystal structure and genetic analysis reveal conserved interactions and a regulatory element essential for cell growth in yeast. Molecular Cell. 5 (5), 865-876 (2000).
- Kumari, S., Pal, R. K., Gupta, R., Goel, M. High Resolution X-ray Diffraction Dataset for Bacillus licheniformis Gamma Glutamyl Transpeptidase-acivicin complex: SUMO-Tag Renders High Expression and Solubility. Protein Journakl. 36 (1), 7-16 (2017).
- Zhang, J., Sun, A., Dong, Y., Wei, D. Recombinant Production and Characterization of SAC, the Core Domain of Par-4, by SUMO Fusion System. Applied Biochemistry and Biotechnology. , (2017).
- Reif, A., et al. Semisynthesis of biologically active glycoforms of the human cytokine interleukin 6. Angewandte Chemie International Edition English. 53 (45), 12125-12131 (2014).
- Froger, A., Hall, J. E. Transformation of plasmid DNA into E. coli using the heat shock method. Journal of Visualized Experiments. (6), 253 (2007).
- Smith, B. J. SDS Polyacrylamide Gel Electrophoresis of Proteins. Methods in Molecular Biology. 1, 41-55 (1984).
- Lawrence, A. M., Besir, H. U. Staining of proteins in gels with Coomassie G-250 without organic solvent and acetic acid. Journal of Visualized Experiments. (30), (2009).
- Block, H., et al. Immobilized-metal affinity chromatography (IMAC): a review. Methods in Enzymology. 463, 439-473 (2009).
- Chen, S. M., Wetzel, R. Solubilization and disaggregation of polyglutamine peptides. Protein Science. 10 (4), 887-891 (2001).
- Booth, D. S., Avila-Sakar, A., Cheng, Y. Visualizing proteins and macromolecular complexes by negative stain EM: from grid preparation to image acquisition. Journal of Visualized Experiments. (58), (2011).
- O'Nuallain, B., et al. Kinetics and thermodynamics of amyloid assembly using a high-performance liquid chromatography-based sedimentation assay. Amyloid, Prions, and Other Protein Aggregates, Pt C. 413, 34-74 (2006).
- Greenfield, N. J. Analysis of circular dichroism data. Methods in Enzymology. 383, 282-317 (2004).
- Aiken, C. T., et al. Phosphorylation of threonine 3: implications for Huntingtin aggregation and neurotoxicity. Journal of Biological Chemistry. 284 (43), 29427-29436 (2009).
- Satakarni, M., Curtis, R. Production of recombinant peptides as fusions with SUMO. Protein Expression and Purification. 78 (2), 113-119 (2011).
- Davies, H. A., Wilkinson, M. C., Gibson, R. P., Middleton, D. A. Expression and purification of the aortic amyloid polypeptide medin. Protein Expression and Purification. 98, 32-37 (2014).
- Chen, S., Wetzel, R. Solubilization and disaggregation of polyglutamine peptides. Protein Science. 10 (4), 887-891 (2001).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon