Method Article
Geração de nativos, sem marcas de formatação da huntingtina Exon1 monômero e fibrilas usando uma estratégia de fusão de sumô
Neste Artigo
Resumo
Aqui, apresentamos um protocolo robusto e otimizado para a produção de quantidades de miligrama de monômeros nativos, livre de marca e fibrilas do exon1 da proteína huntingtina (Httex1) com base na fusão transitória do ubiquitin pequeno modificador relacionados (SUMO).
Resumo
A doença de Huntington (HD) é uma doença hereditária neurodegenerativa fatal causada por uma expansão de CAG (≥36) no primeiro exon do gene HD, resultando na expressão da proteína do Huntingtin (Htt) ou N-terminal respectivos fragmentos com um (polyglutamine expandido trecho do polyQ).
O exon1 da proteína huntingtina (Httex1) é o menor fragmento de Htt que recapitula a muitos dos recursos de HD em celular e animal modelos e é um dos mais amplamente estudados fragmentos de Htt. O pequeno tamanho do Httex1 torna experimentalmente mais receptivos a caracterização biofísica, usando técnicas padrão e de alta resolução, em comparação com mais fragmentos ou Htt completo. No entanto, a agregação alta propensão de mutantes Httex1 (mHttex1) com conteúdo polyQ aumentada (≥42) tem dificultado a desenvolver expressão eficiente e sistemas de purificação para produzir essas proteínas em quantidades suficientes e torná-los acessíveis para cientistas de diferentes disciplinas, sem o uso de proteínas da fusão ou outras estratégias que alteram a sequência nativa da proteína. Apresentamos aqui um método robusto e otimizado para a produção de quantidades de miligrama de nativo, marca isento de Httex1 com base na fusão transitória do ubiquitin pequeno modificador relacionados (SUMO). A simplicidade e a eficiência da estratégia vão eliminar a necessidade de usar sequências não-nativas de Httex1, assim, tornando esta proteína mais acessível aos investigadores e melhorar a reprodutibilidade de experimentos em laboratórios diferentes. Acreditamos que estes avanços também facilitará estudos futuros vistos elucidar a relação estrutura-função de Htt, bem como desenvolver novas ferramentas de diagnósticos e terapias para tratar ou retardar a progressão da DH.
Introdução
HTT é uma proteína de 348 kDa e tem sido implicado em várias funções fisiológicas1. Quando Htt contém uma região expandida polyQ de mais de 36 resíduos em seu N-terminal, que provoca HD2,3. Patologia de HD é caracterizada por inclusões celulares no corpo estriado e córtex, que leva à morte neuronal e atrofia dos tecidos afetados4,5. Vários fragmentos de Htt N-terminal que contém o intervalo de repetição polyQ foram detectados no post-mortem de cérebros de doentes de Huntington e são pensados para ser gerado pelo processamento proteolítica da proteína huntingtina6. Estudos recentes sugerem que Httex1 também pode ser formada devido a emenda de mRNA aberrante. Httex1 contém a mutação polyQ patológica e sua superexpressão em animais pode recapitular muitas das principais características do HD7, destacando, assim, um possível papel central deste fragmento em HD patologia e doença progressão6, 8,9.
Devido a agregação alta propensão de mutantes Httex1 (mHttex1) com polyQ expandida do trato, a maioria dos sistemas de expressão existentes baseiam-se a fusão transiente de Httex1 às proteínas (como a glutationa S-transferase (GST), tioredoxina (TRX) ou maltose-proteína (MBP) e/ou peptídeos (poli-histidina) que diferencialmente melhorar sua expressão, estabilidade, purificação e/ou solubilidade10,11,12,13,14 ,15,16,17,18,19,20,21,22,23 ,24,25,26,,27,28. O parceiro de fusão está ligado à Httex1 com uma sequência curta, contendo um local de clivagem por proteases como a tripsina, tabacco etch protease do vírus (TEV) ou PreScission para permitir a clivagem e a libertação de Httex1 antes do início da agregação ou purificação. Deficiências desses métodos incluem a possibilidade de deixar resíduos adicionais devido à não-traceless clivagem e a criação de fragmentos truncados devido miscleavage dentro da sequência de Httex1, além de heterogeneidade devido à clivagem incompleta ( Ver Vieweg et al . para uma discussão mais aprofundada, sobre as vantagens e limitações desta abordagem)10. Para lidar com essas limitações, recentemente desenvolvemos uma estratégia de expressão, possibilitando a geração de Httex1 livre de marca nativo pela primeira vez, utilizando uma fusão de N-terminal transiente do Synechocystis SP. (Ssp) DnaB intein a Httex110. Enquanto a clivagem intein é traceless e específico e produz quantidade mg de proteínas, que ainda sofre de dois inconvenientes que poderiam reduzir o rendimento: ou seja, prematura clivagem do intein que pode ocorrer durante a expressão e o facto de clivagem ocorre sobre várias horas, que podem levar à perda de proteína devido à agregação, especialmente para Httex1 com repetições polyQ expandido.
Para resolver essas limitações e refinar a nossa estratégia para a produção de nativo, livre de marca Httex1, desenvolvemos um novo sistema de expressão baseado em fusão transiente de sumô, mais exatamente o fermento do homólogo Smt3 para Httex1. A aplicação do sistema para a produção de proteínas recombinantes de sumô foi publicado pela primeira vez em 200429, onde demonstrou-se um aumento da taxa de expressão e solubilidade da proteína de fusão de sumô. A tag de sumô pode ser clivada pela ubiquitina como específicos da proteína protease 1 (ULP1), que não necessita de um site de reconhecimento, mas reconhece a estrutura terciária de sumô e praticamente elimina a possibilidade de miscleavage30. Além disso, a clivagem mediada por ULP1 é rápido e traceless e não deixa resíduos adicionais para trás. A clivagem prematura da marca de fusão, como observado com a autocatalytic intein10, completamente é evitada pela exigência de uma protease externo. Enquanto a estratégia de sumô é hoje amplamente utilizada para a produção de proteínas recombinantes a31,32,33, demonstramos neste artigo o que é especialmente útil para a geração de uma intrinsecamente desordenado, sujeitos a agregação amiloidogénicos proteína tais como Httex1. Acreditamos que a simplicidade, eficiência e robustez de nosso método baseada em SUMO-fusão vão fazer Httex1 nativo, livre de marca mais acessível aos pesquisadores de diferentes disciplinas e eliminar a necessidade de usar sequências não-nativas de Httex1 em vitro . Este é um avanço importante que facilitará futuros estudos para elucidar a relação estrutura-função de Httex1.
O protocolo descreve a purificação do Httex1 de 12 L de cultura bacteriana, mas o protocolo pode ser facilmente adaptado para as produções de menor ou maior escala. O protocolo descreve a produção de tipo selvagem Httex1 (wtHttex1) com um comprimento de repetição polyQ abaixo (23Q) e mutante Httex1 (mHttex1) com um polyQ repetir comprimento acima (43Q) o limiar patogénico (36Q).
Protocolo
1. expressão de recombinação Httex1 23Q e 43Q
- Prepare os buffers exigidos e soluções. Preparar 1000 x solução estoque de ampicilina (AMP, 100 mg/mL), filtro (0,2 µm), alíquota e loja a-20 ° C. Preparar o meio de caldo de carne (LB) lisogenia (25g LB Miller por 1 L H2O), autoclave. Preparar a solução-mãe 1 M isopropílico ß-D-1thiogalactopyranoside (IPTG), filtro (0,2 µm), alíquota e loja a-20 ° C.
- Transformação química competentes Escherichia coli B ER 2566 com um vetor de pTWIN1, contendo Httex1 humana fundida a uma marca de N-terminal His6-sumô com o de método de choque do calor34.
Nota: A e. coli BL21 DE3 estirpe também tem sido usada. No entanto, neste caso uma maior quantidade de truncamentos foi observada. - Inocular 200 mL de LB-médio com 1 x AMP em um Erlenmeyer de 1L, adicionando uma única colônia da placa de ágar com uma ponta de pipeta estéril. Incube a cultura a 30 ° C e 180 rpm para 20 h (overnight) numa incubadora bacteriana.
- Tome uma amostra de 1 mL da cultura com uma pipeta estéril. Medir a densidade óptica em 600 nm (OD600) de amostra uma cubeta de plástico descartável e um fotômetro (respeito a medição variam entre 0,1 e 1, diluído com LB-médio se necessário). Calcular a quantidade de preculture que resultará em uma partida de OD600 de 0,05 em uma cultura de 3L (com um preculture de OD600 = 3 isso significaria 50 mL).
- Inocular quatro culturas (cada 3 L de meio LB com 1 x AMP em um balão de 5 L), adicionando a quantidade calculada de preculture com uma pipeta estéril. Incube as culturas a 37 ° C e 180 rpm numa incubadora bacteriana.
- Cada 30 min, tire uma amostra de 1 mL da cultura com uma pipeta estéril. Medir o OD600 de amostra uma cubeta de plástico descartável e um fotômetro. Quando OD600 atingiu 0,1 (normalmente após 1-2 h), definir a temperatura da incubadora bacteriana a 14 ° C e continuar a incubação arrefecendo. Cada 30 min, tire uma amostra de 1 mL da cultura com uma pipeta estéril. Medir o OD600 de amostra uma cubeta de plástico descartável e um fotômetro.
Nota: O tempo para esfriar as culturas pode variar com a incubadora usada, então a hora de começar a refrigeração pode ter que ser adaptados dependendo do tipo de incubadora usada. No entanto, alterar o gradiente de temperatura deve ter apenas um pequeno impacto sobre o rendimento como a proteína de fusão de sumô parece ser bastante estável. - Quando OD600 atingiu 0.3-0.4 (normalmente após 1-2 h), tire uma amostra de pre-indução da cultura para análise de SDS-PAGE da superexpressão. Calcular o tamanho da amostra que dá uma quantidade comparável de células e um bom sinal no Coomassie manchado de SDS-PAGE: gel de bem para um 10: volume = 0,2 mL/OD600; Tome metade de um gel bem 15.
- Para uma cultura bacteriana com uma OD600= 0,4, tomar 500 µ l. Tome o volume calculado de cultura bacteriana com uma pipeta estéril. Girar a amostra (18000 x g, 4 ° C, 2 min) e descartar o sobrenadante. Manter a pelota a-20 ° C até que esteja pronto a ser usado para análise (passo 1.11).
- Induzir a expressão da proteína por pipetagem 1,2 mL de uma solução de reserva de IPTG 1m para cada solução de cultura 3L (concentração final 0,4 mM). Continue a incubacao a cultura a 14 ° C, durante 16 h (durante a noite).
Nota: A temperatura normalmente atingirá ~ 20 ° C pelo tempo que IPTG é adicionado, dependendo do desempenho da incubadora. - Tome uma amostra de pós-indução da cultura para análise de SDS-PAGE de superexpressão, seguindo o procedimento descrito na etapa 1.7.
- Recolher as células por centrifugação em tubos de 1 L (3993 habitantes x g, 4 ° C, 10 min). Desprezar o sobrenadante, manter o centrifugado no gelo e ir diretamente para a purificação.
- Analise a superexpressão por SDS-PAGE35,36. Resuspenda as amostras pré e pós-indução em 20 µ l de tampão e 20 µ l de 2 x tintura de carregamento. Aqueça as amostras por 5 min a 95 ° C, em um bloco de calor e carregar 20 µ l em um gel de 15%, enquanto ainda está quente. Funcione o gel para 90 min em 180 V. mancha o gel com Coomassie tingir-se de acordo com as instruções do fabricante. Compare os resultados com resultados representativos na Figura 1C.
Nota: O protocolo pode ser parado aqui, o centrifugado pode ser congelado e armazenado a-80 ° C durante várias semanas. Para obter melhores resultados, é recomendável usar o pellet bacteriano fresco e evitar o congelamento. Congelamento-descongelamento pode levar a Lise das células e a degradação do Httex1. Isso pode reduzir o rendimento e a qualidade da proteína.
2. pilha Lysis e purificação de seus6-proteína da fusão do sumô Httex1 imobilizados por cromatografia de afinidade de Metal (IMAC)
- Prepare-se 2 L de tampão A num frasco de vidro (50 mM Tris (hidroximetil)-aminomethan (Tris), 500 mM de NaCl, imidazol 15 mM). Prepare 1 L de tampão B (50 mM Tris, 500 mM de NaCl, pH 7.4 do imidazole de 500 milímetros) em um frasco de vidro. Depois de dissolver os sais, ajustar o pH com 10 N HCl e filtrar as soluções para garrafas frescas com um frasco de filtro superior (0,65 µm). Preparar um fluoreto de x phenylmethylsulfonyl 1000 (PMSF, 0.3 M) solução, alíquota de 100 µ l e loja a-20 ° C.
Nota: O protocolo é projetado para permitir concluir todas as etapas da lise das pelotas bacterianas para purificação de cromatografia líquida (RP-HPLC) de fase reversa de alta performance e liofilização dentro de 8-9 h. Para limitar a agregação e proteólise, recomenda-se trabalhar rapidamente sem parar e executar todas as etapas, a 4 ° C ou em gelo. - Adicionar 100 µ l de solução-mãe de PMSF e cinco comprimidos (1 por 30 mL de volume final) de inibidor de protease para 100 mL de tampão pre-refrigerado r. Add a pelota bacteriana para o buffer e homogeneizar a suspensão por agitação com uma barra de agitação magnética e pipetando subindo e descendo wi th uma pipeta estéril 10 mL (~ 30 min).
- Divida a suspensão de bactérias em alíquotas de 40 mL em tubos de plástico descartável de 50 mL. Proceda à sonicação cada alíquota em um lote de água e gelo para lise celular (amplitude de 70%, tempo total sonication 5min, intervalos de 30 sonication s, 30 pausa de s).
Nota: É importante que a amostra não aquece durante a etapa de sonication. Recomenda-se adicionar um pouco de água para o banho de gelo para melhorar a dissipação de calor durante sonication. O procedimento sonication talvez precise ser adaptado se for utilizado um instrumento diferente. Outros métodos de Lise como uma imprensa francesa ou um microfluidizer devem funcionar tão bem e podem ser benéficos para evitar o aquecimento da amostra e proteína agregação. Estes dispositivos não estavam disponíveis em nosso laboratório e obtivemos bons resultados com nosso protocolo sonication. - Tome uma amostra de 50 µ l do lisado para análise de eletroforese (SDS-PAGE) de sódio Dodecil sulfato gel de poliacrilamida. Centrifugar a amostra (18000 x g, 4 ° C, 2 min) e pipetar a fração solúvel em um novo tubo de ensaio. Resuspenda a fração insolúvel em 50 µ l de tampão A com uma pipeta. Mantenha as amostras no gelo até análise de SDS-PAGE (passo 2.6).
- Clarificar o lisado por centrifugação (39191 x g, 4 ° C, 60 min).
- Durante a etapa de centrifugação, analise o passo de lise celular por SDS-PAGE. Adicione 50 µ l de 2 x tintura para a fração solúvel e insolúvel do lisado de carregamento, respectivamente. Aqueça por 5 min a 95 ° C e carregar 2 µ l em um gel de 15%, enquanto ainda está quente. Funcione o gel para 90 min em 180 V. mancha o gel com Coomassie tingir-se de acordo com as instruções do fabricante. Compare os resultados com resultados representativos na Figura 1C.
- Filtre o sobrenadante (0,45 µm, filtros seringa). Tome uma amostra de 20 µ l do sobrenadante filtrado para análise de SDS-PAGE (etapa 2.11).
Nota: Normalmente, um volume de 90 a 100 mL de sobrenadante clarificado e filtrado é obtido. Normalmente, 3 filtros seringa são suficientes. Se a filtragem é complicada, tente aumentar a velocidade de centrifugação e/ou tempo. - Isole a proteína de fusão de Httex1 de His6-SUMO da bacteriano esclareceu lisado por cromatografia de afinidade de metal imobilizado (IMAC) em um sistema de cromatografia líquida (FPLC) desempenho rápido em 4 ° C37.
- Preencher o esclareceu lisado em um superloop e a carga sobre a coluna do Ni-NTA (despojado, limpos e recarregados de acordo com o manual do fabricante, um espaço em branco anterior executar é recomendado) para volumes de coluna de 2 mL/min. passar 10 (CV, 200 mL) de tampão A 10 mL/min para lavar fora as proteínas unbound.
- Eluir a proteína de fusão com 2.5 CV (50 mL) de 100% do buffer B em 2 mL/min. Use um tamanho de fração de 50 mL para carregar e de lavagem e 5 mL para a eluição. Compare os resultados com resultados representativos em Figura 1D.
- Tirar uma amostra de cada fração para análise de SDS-PAGE (20 µ l) e as frações que contêm a proteína de fusão, de acordo com o pico do cromatograma IMAC da piscina. Adicionar (2S, 3S) - 1,4 - Bis (Sulfanilo) butano-2,3-diol (TDT) e L-cisteína (concentração final de 100 mM cada) como um pó e dissolver por inversão suavemente o tubo.
Nota: Na nossa experiência é comparável a pureza da proteína de fusão em diferentes frações. Como medida de precaução, as frações da proteína purificada fusão devem ser agrupadas rapidamente após IMAC para evitar a agregação das frações altamente concentradas. Além disso, é recomendável proceder diretamente para a clivagem da marca de sumô e purificação de HPLC. Se necessário, o protocolo pode ser parado aqui. Solução diluída da proteína de fusão foi congelada em nitrogênio líquido, armazenada a-80 ° C e purificada após o descongelamento, sem uma redução significativa no rendimento. Armazenamento da solução diluída da proteína de fusão a 4 ° C por 24 h também dá resultados semelhantes. - Analise o IMAC por SDS-PAGE. Adicione 20 μL de tintura para cada amostra de carregamento. Carrega 2 µ l do material bruto (2.7), a fração não-acoplada, a fração de lavagem e cada fração do pico da eluição em um gel de 15%. Funcione o gel para 90 min em 180 V. mancha o gel com Coomassie tingir-se de acordo com as instruções do fabricante. Compare os resultados com resultados representativos em Figura 1D.
3. segmentação de seus6-sumô-tag e purificação de HPLC
Cuidado: O ácido trifluoroacético (TFA) é um líquido volátil e podem causa queimaduras severas para segurar com cuidado. Realizar qualquer manuseio em uma coifa e usar equipamento de proteção pessoal adequado (i.e., luvas de nitrilo descartáveis, óculos de segurança e um jaleco).
- Em uma garrafa de 5 L, adicionar 5 mL de TFA com uma seringa de plástico de 5 L de água (solvente r: H2O, 0,1% (TFA). Adicionar 2,5 mL de TFA com uma seringa plástica de uma garrafa de 2,5 L de acetonitrilo (solvente b: acetonitrilo, 0,1% TFA).
- Prepare o sistema HPLC como sugerido pelo fabricante. Realize uma corrida em branco para garantir uma coluna limpa.
- Tome uma amostra de 100 µ l da proteína de fusão antes da adição de ULP1 para monitorar a reação de clivagem por UPLC (passo 3.5).
- 20 mL da proteína de fusão de transferência para um novo tubo de 50 mL e adicionar 0,4 mL da solução ULP1, incubar no gelo. Manter a proteína de fusão restantes no gelo.
Nota: Com sua tag catalítico fragmento 403-621 da protease do Ubiquitin-tipo-específico 1 (aqui referido como "ULP1") foi usado para cravar a marca SUMO. A proteína de fusão é mais estável que o Httex1 entalhadas. Não é aconselhável decompor a tag SUMO de todo o lote. Em vez disso, continue com alíquotas de um tamanho que pode ser diretamente e totalmente aplicado à coluna HPLC. - A cada 10 minutos, tire uma amostra de 100 µ l da reação de clivagem para monitorar o progresso por cromatografia líquida de ultra (UPLC). Centrifugar as amostras (18000 rpm, 4 ° C, 2 min) e analisar 2 µ l do sobrenadante por UPLC (gradiente de 10% a 90% do solvente B na de 0,25 a 3 min, 10% B por 1 min, consulte as instruções do fabricante para o uso do instrumento). Compare os cromatogramas obtidos para a amostra antes da adição de ULP1 e das amostras colhidas. Compare os resultados com resultados representativos na Figura 2B.
- Uma vez que o sumô-clivagem é completa (o pico da proteína de fusão desapareceu no cromatograma UPLC e é totalmente convertido para o pico de sumô e Httex1 aparecendo recentemente), a amostra de filtragem com um filtro de seringa (0,22 µm).
Nota: A clivagem de sumô é tipicamente muito rápida (10-20 min a 4 ° C), portanto, a análise UPLC com um tempo de execução de 4 min é uma ferramenta valiosa para monitorar a reação. A amostra antes de HPLC de filtragem purificação é principalmente uma medida preventiva para aumentar a vida útil da coluna. A amostra não deve tornar turva. - Purificar a amostra filtrada por RP-HPLC (gradiente de 25-35% solvente B em A solvente, mais de 40 min a 15 mL/min. (0-10 min: 5%; 10-12,5 min: 5 a 25%; 12.5-52,5 min: 25 a 35%; 52,5 57,5 min 35 a 95%; consulte as instruções do fabricante para o uso do instrumento). Compare os resultados com resultados representativos na Figura 2C.
Nota: Httex1 e seu6-sumô separado bem por RP-HPLC. No entanto, pode haver pequenas quantidades de Httex1 truncado no início e no final do pico. Colete pequenas frações para obter a máxima quantidade de material puro.
Atenção: Utilize o equipamento de segurança adequado (ou seja, jaleco, luvas isoladas e um protetor de cara) quando manipular fluidos criogênicos. - Analisar os HPLC-frações por espectrometria de massa de ionização electro-pulverizador (ESI-MS, mostruário, injectar 10 µ l, vazão de 0,6 mL/min, solvente: 20% B em A, nenhuma coluna, consulte as instruções do fabricante para o uso do instrumento) e UPLC (gradiente de 10% a 90% solvente B na de 0,25 a 3 min, 10% B por 1 min, consulte as instruções do fabricante para o uso do instrumento). Piscina de fracções de pureza similar em tubos plásticos de 50 mL, congelar em nitrogênio líquido e lyophilize. Pesar e transferir a proteína liofilizada para tubos de plástico de 2 mL e armazenar a-20 ° C.
- Caracteriza o material purificado por UPLC, ESI-MS e SDS-PAGE. Dissolver 100 µ g de Httex1 liofilizado em 8 µ l de TFA puro em um tubo de 1,5 mL e incubar durante 20 min à temperatura no tubo de ensaio fechado. Evapora-se cuidadosamente o TFA sob uma coifa com um fluxo de nitrogênio ou argônio. Use baixa pressão de argônio/nitrogênio para evitar a perda da amostra.
- Dissolva a proteína em 100 µ l de H2O. analisar 2 µ l por UPLC e 5 µ l por ESI-MS etapa 3.8. Mix 20 µ l de solução de proteína com 20 µ l de 2 x tintura de carregamento.
- Analise quantidades de 1 µ g para 10 µ g por SDS-PAGE. Funcione o gel para 90 min em 180 V. mancha o gel com Coomassie tingir-se de acordo com as instruções do fabricante. Compare os resultados com resultados representativos na Figura 2,D.
4. desagregação e Resolubilization de Httex1 de proteínas
Atenção: O TFA é um líquido volátil e podem causa queimaduras severas para segurar com cuidado. Realizar qualquer manuseio em uma coifa e usar equipamento de proteção pessoal adequado (ou seja, luvas de nitrilo descartáveis, óculos e um jaleco).
- Prepare-se 10 mL de solução salina de tampão fosfato de Dulbecco (DPBS) (137 mM NaCl, 2,7 mM KCl, 10 mM Na2HPO4, 2mm KH2PO4, pH 7,4) do pó pré-misturado em um tubo de 50 mL. Filtre a solução DPBS através de um filtro de 0,2 µm antes de cada utilização.
- Dissolver 150 µ g de Httex1 liofilizado em 12 µ l de TFA puro em um tubo de 1,5 mL e incubar durante 20 min à temperatura no tubo de ensaio fechado. Evapora-se cuidadosamente o TFA sob uma coifa com um fluxo de nitrogênio ou argônio. Use baixa pressão de argônio/nitrogênio para evitar a perda da amostra38.
Nota: em geral, use 4 µ l TFA para dissolver e desagregar a 50 µ g de proteína. Este procedimento irá criar uma película de proteína no interior as paredes do tubo de ensaio. Para evitar agregação imediata de Httex1 nas etapas a seguir, trabalhar com buffers pre-cooled, mantenha a proteína sempre no gelo e evitar altas concentrações. - Dissolver a proteína desagregada em 1 mL de DPBS pre-cooled e ajustar o pH para 7,2-7,4 com 1 M de NaOH. Filtre a solução da proteína através de um filtro centrífugo de 100 kDa para tubos plásticos de 1,5 mL (20000 x g, 4 ° C, 20 minutos).
Nota: A concentração teórica calculada de Httex1 é maior do que a concentração final desejada para compensar eventuais perdas. A etapa de filtração é necessária remover quaisquer agregados que podem ter se formado durante a dissolução da proteína. - Determinar a concentração de Httex1 usando uma curva de calibração de UPLC com base na análise de aminoácidos (deteção λ214) e enviar de 2 µ g da proteína para análise de aminoácidos para validar a concentração10. Calcular a quantidade de DPBS que precisa ser adicionado para obter uma concentração de 3 µM Httex1.
- Dilua a proteína a 3 µM, adicionando a quantidade calculada de DPBS para o tubo de ensaio. Mantenha o tubo no gelo até o início do protocolo de agregação.
Nota: Httex1 43Q não devem ser armazenadas em solução. Sempre prepare uma solução de proteína fresca baseada no protocolo acima. Httex1 proteínas são armazenadas melhor como um pó liofilizado a-20 ° C.
5. monitoramento de agregação cinética de Httex1 43Q usando UPLC e circular Dicroísmo (CD) espectroscopia e caracterização dos agregados por microscopia eletrônica de transmissão (TEM)
- Prepare a solução de formiato de uranilo para TEM como relatado anteriormente39.
- Iniciar a agregação de Httex1 43Q incubando uma solução de 3 µM em DPBS a 37 ° C (uso 1 mL da solução preparada conforme descrito acima no protocolo de desagregação).
Nota: A agregação de Httex1 pode ser executada em altas concentrações, dependendo das necessidades e objetivos do experimento. - Quantificar a quantidade de proteína solúvel usando UPLC na hora indicada pontos (0, 1, 2, 4, 6, 8, 12, 24, 48 e 120 h). Para fazer isso, tomar uma alíquota de 35 µ l e remover os agregados insolúveis por centrifugação (20000 x g, 4 ° C, 20 min). Injete o UPLC 4 µ l do sobrenadante. Calcule a proporção de monômero solúvel com base na mudança da área de pico, usando o instrumento software 40. Compare os resultados com resultados representativos na Figura 3.
- Caracteriza as mudanças na estrutura secundária usando espectroscopia de CD em 0 e 48 h. Tomar uma alíquota de 100 µ l e medir a elipticidade (cubeta de quartzo de 1 mm, 195 nm a 250 nm, 20 ° C, dados aponta cada 0,2 nm, velocidade de 10 nm/min, integração digital tempo de 2 s, largura de banda de 1.0 nm). Adquirir 6 espectros da amostra e média e alise usando um filtro binomial com largura de convolução de 99. Traça os espectros como o resíduo significa elipticidade molar (θMRE)41. Compare os resultados com resultados representativos em Figura 3B.
- Caracteriza as propriedades estruturais e morfológicas dos agregados pela TEM. Coloque 3 µ l da solução da proteína para uma Formvar/carbono-revestido 200-malha, brilho-descarregada cobre a grelha para 1 min. Lave a grade duas vezes com água de 15 µ l, uma vez com 15 μL de formiato de uranilo 0,7% (p/v) e mancha por 30 s, com 15 μL de formiato de uranilo 0,7% w/v. Execute uma análise TEM das grades. Compare os resultados com resultados representativos na Figura 3C.
Resultados
Httex1 é expresso em e. coli com um N-terminal dele6-marca SUMO. Os resultados representativos da expressão e purificação da proteína de fusão são resumidos na Figura 1. A sequência de Httex1 consiste dos resíduos 2-90 de Htt e começa com Ala2, porque Met1 é totalmente clivada em vivo42. A numeração dos aminoácidos refere-se a variante 23Q, a sequência completa da proteína expressa fusão é mostrada na Figura 1A. Os plasmideos serão depositados no Addgene em um futuro próximo para ser compartilhado com a Comunidade. Um esquema do plasmídeo e a proteína de fusão expressa é mostrado na Figura 1B. Httex1 His6-SUMO expressa a um nível médio (Figura 1C) e maior parte da proteína de fusão está presente na fração solúvel após Lise, tanto para o 23Q e a variante de 43Q. A proteína de fusão migra mais alto do que o esperado, com base no peso molecular. Isto é parcialmente devido a forte dobra de sumô, mas principalmente devido à composição de sequência incomum de Httex1, contendo principalmente resíduos de glutamina e prolina. Tanto a sua (23Q) e o mutante (43Q) proteína de fusão pode ser enriquecida a ~ 80% de pureza por IMAC (Figura 1D), a presença de co, purificação da proteína do hospedeiro pode ser explicada pelo nível comparativamente baixa expressão de Httex1 e o grande exemplo volume aplicado à coluna.
O decote de seus6-marca de sumô e a purificação de Httex1 é mostrado na Figura 2. UPLC é uma ferramenta eficiente para monitorar o decote de seus6-marca de sumô (Figura 2B). O pico original da proteína de fusão é consumido e dois picos de novos e bem separados do seu correspondente6-marca de sumô e Httex1 aparecem. A reação de clivagem é terminada em 10-20 min. O Western Blot (WB) é muito lento para acompanhar a reação de clivagem com eficiência, mas foi incluída na figura para referência e para demonstrar a completude da clivagem do sumô. Ambos Httex1 23Q e 43Q podem ser separados bem desde os seus6-sumô tag por RP-HPLC (Figura 2C) e foram obtidos em elevado grau de pureza, como demonstrado pela análise UPLC, MS e SDS-PAGE (Figura 2D).
Para ilustrar que as proteínas Httex1 preparadas por este método mantêm as propriedades de agregação esperado do Httex1, avaliamos a cinética de fibrillization da mutante Httex1 a 37 ° C por um ensaio de sedimentação, monitorado as mudanças na estrutura secundária por CD espectroscopia e caracteriza-se a morfologia dos agregados pela TEM. Um conjunto de dados representativo da cinética de agregação de mHttex1 formação de fibrila conforme determinado por um ensaio de sedimentação é mostrado naFigura 3. A perda de Httex1 solúvel 43Q ao longo do tempo, devido à formação de fibrila foi quantificada por UPLC. Observamos uma completa depleção de proteína solúvel após 48 horas de incubação. Além disso, nós determinamos a estrutura secundária da proteína por espectroscopia de CD (Figura 3B). Httex1 43Q turnos de não-estruturados (λmin 205 nm) para conformação rica principalmente β-folha (λmin 215 nm) após 48 horas de incubação. Esta mudança estrutural é acompanhada pela formação de agregados tempo fibrillar como observável pela TEM 48 horas (Figura 3C).
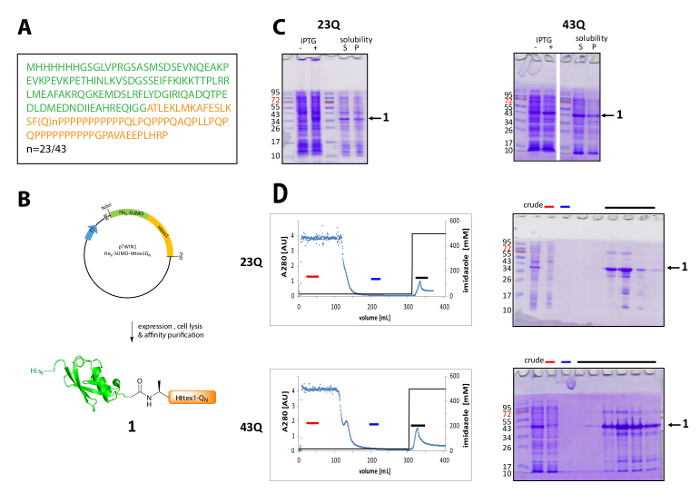
Figura 1 . Expressão e purificação de seus6-proteína de fusão de sumô Httex1.
(A), a sequência de aminoácidos de seus6-sumô-Httex1-QN fusão construções (seu6-sumô em verde e Httex1-QN em laranja); (B) visão geral esquemática da expressão e purificação da proteína de fusão; (C) análise de SDS-PAGE de expressão e a solubilidade da proteína de fusão após lise; (D) cromatograma da purificação da proteína de fusão do IMAC e análise das frações por SDS-PAGE (barra vermelha: barra de fração desacoplado, azul: fração de lavagem, preto bar: frações contendo o pico de eluição); Clique aqui para ver uma versão maior desta figura.

Figura 2 . Clivagem de seus6 sumô tag e purificação de proteínas de Httex1-QN marca-livre.
Visão esquemática do (A); (B) análise da clivagem da marca de sumô com ULP1 por UPLC (azul: antes da adição de ULP1; preto: 20 min (23Q), respectivamente de 10 min (43Q) após a adição de ULP1) e BM (MAB5492 1: 2000, secundário de cabra anti rato anticorpo 1:5000); (C) cromatograma da purificação RP-HPLC preparativa de Httex1; D: análise do Httex1 purificado por UPLC, SDS-PAGE e ESI-MS; o peso molecular esperado é Da 9943 (23Q) e Da 12506 (43Q) respectivamente. Clique aqui para ver uma versão maior desta figura.
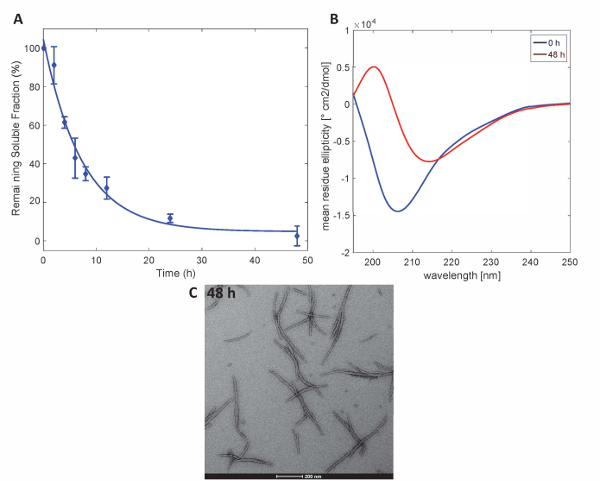
Figura 3 . Agregação de Httex1-43Q: ensaio de sedimentação (A) com base em UPLC. (B) CD espectros da estrutura secundária às 0 h e micrografias de 48 h. (C) temperatura dos agregados a 48 h (barras de escala são 200 nm). Clique aqui para ver uma versão maior desta figura.
Discussão
Neste protocolo, descrevemos um procedimento eficiente para obter quantidades de miligrama de nativo, untagged Httex1 que contenham resíduos de glutamina 23 ou 43. Isto foi conseguido por expressar Httex1 como uma fusão do C-terminal para seus6-marca de sumô, que é usado para isolar a proteína de fusão da célula lisada por IMAC e é clivado antes da purificação de HPLC de Httex1. Enquanto a estratégia de sumô tem sido usada na produção de várias outras proteínas, nosso método mostra que as propriedades únicas de que sumô também poderia ser usado para gerar intrinsecamente desordenado, sujeitos a agregação da proteína amiloidogénicos que anteriormente demonstraram ser extremamente difícil de manipular e produzir43,44. Apresentamos um protocolo que é simples, fácil de usar e comparável a um protocolo para a geração de uma proteína "bem comportada". A fusão de sumô solubiliza e estabiliza o Httex1 durante a expressão e a etapa de purificação do IMAC. Clivagem prematura da tag, como observado com a estratégia de intein10 e agregação já não eram um problema.
Proteínas intrinsecamente desordenadas são especialmente vulneráveis à degradação. Enquanto a degradação do N-terminal da região N17 não é um problema usando este protocolo, truncamentos no PRD de Httex1 podem ocorrer. Como as proteínas truncadas são muito semelhantes aos Httex1 em tamanho, carga e hidrofobicidade, removendo-os por meio de cromatografia em fase gasosa é um desafio, assim é melhor evitar a sua formação em primeiro lugar. Aderindo intimamente ao protocolo, sempre trabalhando no gelo e usando uma quantidade suficiente de inibidor de protease devem ajudar a manter o nível de truncamento observado muito baixa. Aplicar uma tag de fusão no C-terminal de Httex1 poderia remover truncamentos no PRD facilmente como a proteína truncada perderia a tag afinidade também. No entanto, não se a sequência nativa precisa ser mantida que esta opção não pode ser aplicada como Httex1 termina com prolina e o melhor de nosso conhecimento há nenhum C-terminal de fusao que é conhecidos por induzir a clivagem traceless e eficiente depois de prolina.
A parte mais crítica do protocolo é a manipulação do Httex1 libertado após a clivagem da marca de sumô por ULP1. A proteína deve ser purificada imediatamente por RP-HPLC. Felizmente, esta é uma reação eficiente e rápida que normalmente é concluída em 10-20 min a 4 ° C. Em contraste, a estratégia de intein necessárias várias horas para clivagem completa de intein, exigindo um trade-off entre a clivagem incompleta e agregação de início a fim de maximizar o rendimento. Um rápido exame é necessário para o mutante Httex1, como ele vai começar a agregar na comparativamente alta concentração presente na reação de clivagem, Considerando que a variante 23Q é estável por mais tempo. Durante a purificação RP-HPLC, outra vantagem do sumô torna-se evidente: enquanto o Ssp DnaB intein é hidrofóbico e adere fortemente à coluna, sumô é mais hidrofílico e elutes completamente da coluna de fase reversa de C4. Embora ULP1 comercial é muito caro, a proteína pode ser facilmente produzida em alto rendimento seguindo protocolos publicados anteriormente29.
A importância crítica da aplicação de um protocolo de desagregação antes usando o Httex1 não pode ser suficiente sublinhado. Liofilizado polyQ proteínas tais como Httex1 são estáveis e podem ser armazenados longos períodos, mas não são completamente solúveis em água e buffers. A presença de oligômeros pré-formadas ou fibrilas poderia ter um impacto significativo na cinética de agregação e propriedades biofísicas do proteína45. O protocolo de desagregação descrito aqui permite a desagregação da proteína, remoção dos agregados pré-formadas e geração de uma solução de Httex1 monomérica de uma amostra liofilizada. Observamos morfologia de cinética e fibrila agregação similar obtidos com o SUMO e a estratégia de intein de Httex1.
Em comparação com os métodos anteriores para a produção de Httex1, a estratégia de sumô descrita aqui oferece diversas vantagens e expande a gama de possíveis estudos para investigar a estrutura e propriedades funcionais desta proteína na saúde e na doença. A proteína de fusão de sumô-Httex1 é fácil de manusear, pode ser congelado e armazenado ou mantido em solução por 24 horas à temperatura ambiente, enquanto o mHttex1 livre iria agregar rapidamente. A estabilidade e alta solubilidade das proteínas de fusão de sumô-Httex1 fornecem maior flexibilidade para manipular a proteína e/ou introduzir modificações enzimáticas e químicas em mHttex1 que não seriam possíveis após a clivagem. Isso inclui a introdução de modificações borne-translational fluorophores, rotação de rótulos, etiquetas de biotina, etc os avanços apresentados aqui devem 1) facilitar futuros estudos para elucidar as relações estrutura-função de Httex1; 2) gerar novas ferramentas para investigar Htt agregação e patologia espalhando; 3) permitem o desenvolvimento de novos ensaios para identificar moléculas que estabilizar mutante Httex1 e impedem sua agregação; e 4) incentivar os cientistas de outras áreas para trazer para o trabalho sobre esta proteína e se juntar a nossa busca para encontrar a cura para a doença de Huntington.
Divulgações
Os autores declaram que têm não há conflitos de interesse com o conteúdo deste artigo.
Agradecimentos
Este trabalho foi financiado principalmente pelos subsídios da Fundação CHDI e o Swiss National Science Foundation. Agradecemos a Dr. Sophie Vieweg para discussões útil durante o desenvolvimento deste novo sistema de expressão e outros membros do grupo Lashuel por compartilhar sua experiência com este sistema de expressão e para os seus comentários valiosos e entrado. Agradecemos também o Prof Oliver Hantschel para fornecer o plasmídeo ULP1. Os autores agradecer Dr. John B. Warner e Dr. Senthil K. Thangaraj revisão crítica do manuscrito
Materiais
| Name | Company | Catalog Number | Comments |
| Uranyl formate (UO2(CHO2)2) | EMS | 22450 | |
| Formvar/carbon 200 mesh, Cu 50 grids | EMS | FCF200-Cu-50 | |
| High Precision Cell made of Quartz SUPRASIL 1 mm light path from Hellma Analytics | HellmaAnalytics | 110-1-40 | |
| Buffer Substance Dulbecco's (PBS w/o Ca and Mg) ancinne ref 47302 (RT) SERVA | Witech | SVA4730203 | |
| Ampicillin | AxonLab | A0839.0100 | |
| Luria Broth (Miller's LB Broth) | Chemie Brunschwig | 1551.05 | |
| Isopropyl β-D-1-thiogalactopyranoside (IPTG) | AxonLab | A1008.0025 | |
| E. coli B ER2566 | NEB | NEB# E4130 | |
| Imidazole | Sigma | 56750-500G | |
| cOmplete Protease Inhibitor Cocktail | Roche | 4693116001 | |
| Anti-Huntingtin Antibody, a.a. 1-82 | Merck Millipore Corporation | MAB5492 | |
| IRDye 680RD Goat anti-Mouse IgG (H + L) | Licor | 925-68070 | |
| PMSF | AxonLab | A0999.0005 | |
| HisPrep 16/10 column | GE Healthcare | 28936551 | |
| C4 HPLC column | Phenomenex | 00G-4168.P0 | 10 µm C4 300 Å, LC Column 250 x 21.2 mm, Phenomenex, 19x10 mm guard column, not temperature jacketed |
| Acetonitrile HPLC | MachereyNagel | C2502 | |
| Filtre seringue Filtropur S 0,45 ul sans prefiltre sterile | Sarstedt AG | 83.1826 | |
| Spectrophotometer semi-micro cuvette | Reactolab S.A. | 2534 | |
| Superloop, 1/16" fittings (ÄKTAdesign), 50 ml | GE Healthcare | 18111382 | |
| Trifluoroacetic acid | Sigma | 302031 | |
| GREINER Tubes fo FPLC 16 x 100 mm, cap. 12.0 ml | Greiner Bio-One | 7.160 102 | |
| 100 kD Microcon fast flow filters | Merck Millipore Corporation | MRCF0R100 | |
| Vibra-cell VCX130 ultrasonic liquid processor | Sonics | ||
| Äkta 900 equipped with a fraction collector | GE Healthcare | ||
| Jasco J-815 Circular Dichroism | Jasco | ||
| Waters UPLC system | Waters | C8 BEH acquity 2.1x100 mm 1.7 micron column , preheated column (40 °C), flow rate of 0.6 mL/min, injection volume of 4 μL | |
| waters HPLC system | Waters | 2489 UV detector and 2535 quaternary gradient module, 20 mL loop in a FlexInject housing | |
| ESI-MS: Finnigan LTQ | Thermo Fisher Scientific | ||
| lyophylizer instrument | FreeZone 2.5 Plus | ||
| Tecnai Spirit BioTWIN | FEI | electron microscope equipped with a LaB6 gun and a 4K x 4K FEI Eagle CCD camera (FEI) and operated at 80 kV | |
| 37 °C shaking incubator | Infors HT multitron Standard | ||
| Biophotometer plus | Eppendorf |
Referências
- Saudou, F., Humbert, S. The Biology of Huntingtin. Neuron. 89 (5), 910-926 (2016).
- MacDonald, M. E., Gines, S., Gusella, J. F., Wheeler, V. C. Huntington's disease. Neuromolecular Medicine. 4 (1-2), 7-20 (2003).
- Li, S., Li, X. J. Multiple pathways contribute to the pathogenesis of Huntington disease. Molecular Neurodegeneration. 1, 19 (2006).
- DiFiglia, M. Aggregation of Huntingtin in Neuronal Intranuclear Inclusions and Dystrophic Neurites in Brain. Science. 277 (5334), 1990-1993 (1997).
- Atwal, R. S., et al. Huntingtin has a membrane association signal that can modulate huntingtin aggregation, nuclear entry and toxicity. Human Molecular Genetics. 16 (21), 2600-2615 (2007).
- Sathasivam, K., et al. Aberrant splicing of HTT generates the pathogenic exon 1 protein in Huntington disease. Proceedings of the National Academy of Sciences U S A. 110 (6), 2366-2370 (2013).
- Mangiarini, L., et al. Exon 1 of the HD gene with an expanded CAG repeat is sufficient to cause a progressive neurological phenotype in transgenic mice. Cell. 87 (3), 493-506 (1996).
- El-Daher, M. T., et al. Huntingtin proteolysis releases non-polyQ fragments that cause toxicity through dynamin 1 dysregulation. EMBO Journal. 34 (17), 2255-2271 (2015).
- Lunkes, A., et al. Proteases acting on mutant Huntingtin generate cleaved products that differentially build up cytoplasmic and nuclear inclusions. Molecular Cell. 10 (2), 259-269 (2002).
- Vieweg, S., Ansaloni, A., Wang, Z. M., Warner, J. B., Lashuel, H. A. An Intein-based Strategy for the Production of Tag-free Huntingtin Exon 1 Proteins Enables New Insights into the Polyglutamine Dependence of Httex1 Aggregation and Fibril Formation. Journal of Biological Chemistry. 291 (23), 12074-12086 (2016).
- Georgalis, Y., et al. Huntingtin aggregation monitored by dynamic light scattering. Proceedings of the National Academy of Sciences U S A. 95 (11), 6118-6121 (1998).
- Scherzinger, E., et al. Huntingtin-encoded polyglutamine expansions form amyloid-like protein aggregates in vitro and in vivo. Cell. 90 (3), 549-558 (1997).
- Scherzinger, E., et al. Self-assembly of polyglutamine-containing huntingtin fragments into amyloid-like fibrils: implications for Huntington's disease pathology. Proceedings of the National Academy of Sciences U S A. 96 (8), 4604-4609 (1999).
- Muchowski, P. J., et al. Hsp70 and hsp40 chaperones can inhibit self-assembly of polyglutamine proteins into amyloid-like fibrils. Proceedings of the National Academy of Sciences U S A. 97 (14), 7841-7846 (2000).
- Heiser, V., et al. Inhibition of huntingtin fibrillogenesis by specific antibodies and small molecules: implications for Huntington's disease therapy. Proceedings of the National Academy of Sciences U S A. 97 (12), 6739-6744 (2000).
- Bennett, E. J., Bence, N. F., Jayakumar, R., Kopito, R. R. Global impairment of the ubiquitin-proteasome system by nuclear or cytoplasmic protein aggregates precedes inclusion body formation. Molecular Cell. 17 (3), 351-365 (2005).
- Tam, S., et al. The chaperonin TRiC blocks a huntingtin sequence element that promotes the conformational switch to aggregation. Nature Structural & Molecular Biology. 16 (12), 1279-1285 (2009).
- Nekooki-Machida, Y., et al. Distinct conformations of in vitro and in vivo amyloids of huntingtin-exon1 show different cytotoxicity. Proceedings of the National Academy of Sciences U S A. 106 (24), 9679-9684 (2009).
- Wacker, J. L., Zareie, M. H., Fong, H., Sarikaya, M., Muchowski, P. J. Hsp70 and Hsp40 attenuate formation of spherical and annular polyglutamine oligomers by partitioning monomer. Nature Structural & Molecular Biology. 11 (12), 1215-1222 (2004).
- Legleiter, J., et al. Monoclonal antibodies recognize distinct conformational epitopes formed by polyglutamine in a mutant huntingtin fragment. Journal of Biological Chemistry. 284 (32), 21647-21658 (2009).
- Legleiter, J., et al. Mutant huntingtin fragments form oligomers in a polyglutamine length-dependent manner in vitro and in vivo. Journal of Biological Chemistry. 285 (19), 14777-14790 (2010).
- Nucifora, L. G., et al. Identification of novel potentially toxic oligomers formed in vitro. from mammalian-derived expanded huntingtin exon-1 protein. Journal of Biological Chemistry. 287 (19), 16017-16028 (2012).
- Dahlgren, P. R., et al. Atomic force microscopy analysis of the Huntington protein nanofibril formation. Nanomedicine. 1 (1), 52-57 (2005).
- Poirier, M. A., et al. Huntingtin spheroids and protofibrils as precursors in polyglutamine fibrilization. Journal of Biological Chemistry. 277 (43), 41032-41037 (2002).
- Duim, W. C., Chen, B., Frydman, J., Moerner, W. E. Sub-diffraction imaging of huntingtin protein aggregates by fluorescence blink-microscopy and atomic force microscopy. Chemphyschem. 12 (13), 2387-2390 (2011).
- Pieri, L., Madiona, K., Bousset, L., Melki, R. Fibrillar alpha-synuclein and huntingtin exon 1 assemblies are toxic to the cells. Biophysical Journal. 102 (12), 2894-2905 (2012).
- Monsellier, E., Redeker, V., Ruiz-Arlandis, G., Bousset, L., Melki, R. Molecular interaction between the chaperone Hsc70 and the N-terminal flank of huntingtin exon 1 modulates aggregation. Journal of Biological Chemistry. 290 (5), 2560-2576 (2015).
- Isas, J. M., Langen, R., Siemer, A. B. Solid-State Nuclear Magnetic Resonance on the Static and Dynamic Domains of Huntingtin Exon-1 Fibrils. Biochemistry. 54 (25), 3942-3949 (2015).
- Malakhov, M. P., et al. SUMO fusions and SUMO-specific protease for efficient expression and purification of proteins. Journal of Structural Function Genomics. 5 (1-2), 75-86 (2004).
- Mossessova, E., Lima, C. D. Ulp1-SUMO crystal structure and genetic analysis reveal conserved interactions and a regulatory element essential for cell growth in yeast. Molecular Cell. 5 (5), 865-876 (2000).
- Kumari, S., Pal, R. K., Gupta, R., Goel, M. High Resolution X-ray Diffraction Dataset for Bacillus licheniformis Gamma Glutamyl Transpeptidase-acivicin complex: SUMO-Tag Renders High Expression and Solubility. Protein Journakl. 36 (1), 7-16 (2017).
- Zhang, J., Sun, A., Dong, Y., Wei, D. Recombinant Production and Characterization of SAC, the Core Domain of Par-4, by SUMO Fusion System. Applied Biochemistry and Biotechnology. , (2017).
- Reif, A., et al. Semisynthesis of biologically active glycoforms of the human cytokine interleukin 6. Angewandte Chemie International Edition English. 53 (45), 12125-12131 (2014).
- Froger, A., Hall, J. E. Transformation of plasmid DNA into E. coli using the heat shock method. Journal of Visualized Experiments. (6), 253 (2007).
- Smith, B. J. SDS Polyacrylamide Gel Electrophoresis of Proteins. Methods in Molecular Biology. 1, 41-55 (1984).
- Lawrence, A. M., Besir, H. U. Staining of proteins in gels with Coomassie G-250 without organic solvent and acetic acid. Journal of Visualized Experiments. (30), (2009).
- Block, H., et al. Immobilized-metal affinity chromatography (IMAC): a review. Methods in Enzymology. 463, 439-473 (2009).
- Chen, S. M., Wetzel, R. Solubilization and disaggregation of polyglutamine peptides. Protein Science. 10 (4), 887-891 (2001).
- Booth, D. S., Avila-Sakar, A., Cheng, Y. Visualizing proteins and macromolecular complexes by negative stain EM: from grid preparation to image acquisition. Journal of Visualized Experiments. (58), (2011).
- O'Nuallain, B., et al. Kinetics and thermodynamics of amyloid assembly using a high-performance liquid chromatography-based sedimentation assay. Amyloid, Prions, and Other Protein Aggregates, Pt C. 413, 34-74 (2006).
- Greenfield, N. J. Analysis of circular dichroism data. Methods in Enzymology. 383, 282-317 (2004).
- Aiken, C. T., et al. Phosphorylation of threonine 3: implications for Huntingtin aggregation and neurotoxicity. Journal of Biological Chemistry. 284 (43), 29427-29436 (2009).
- Satakarni, M., Curtis, R. Production of recombinant peptides as fusions with SUMO. Protein Expression and Purification. 78 (2), 113-119 (2011).
- Davies, H. A., Wilkinson, M. C., Gibson, R. P., Middleton, D. A. Expression and purification of the aortic amyloid polypeptide medin. Protein Expression and Purification. 98, 32-37 (2014).
- Chen, S., Wetzel, R. Solubilization and disaggregation of polyglutamine peptides. Protein Science. 10 (4), 887-891 (2001).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados