È necessario avere un abbonamento a JoVE per visualizzare questo. Accedi o inizia la tua prova gratuita.
Method Article
Misura di rigidità dei substrati in morbido Silicone per gli studi di Mechanobiology usando un microscopio a fluorescenza Widefield
In questo articolo
Riepilogo
Substrati con rigidità nella gamma kilopascal sono utili per studiare la risposta delle cellule a rigidità di micro-ambiente fisiologicamente rilevanti. Utilizzando solo un microscopio a fluorescenza widefield, il modulo di Young di gel di silicone morbido può essere determinato utilizzando un rientro con una sfera adatta.
Abstract
Tessuti molli del corpo umano in genere hanno rigidità nell'intervallo kilopascal (kPa). Di conseguenza, substrati flessibili in silicone e idrogel hanno dimostrati di essere utile substrati per la coltura delle cellule in un microambiente fisico che imita parzialmente condizioni in vivo . Qui, presentiamo un semplice protocollo per la caratterizzazione di moduli di Young dei substrati elastici lineare isotropici in genere utilizzati per gli studi mechanobiology. Il protocollo è costituito da un substrato morbido silicone su una piastra di Petri o in silicone rigido la preparazione, ricoprire la superficie superiore del substrato in silicone con perline fluorescenti, usando una sfera di scala millimetrata per far rientrare la superficie superiore (per gravità), il fluorescente di imaging perline sulla superficie frastagliata in silicone utilizzando un microscopio a fluorescenza e analizzando le immagini risultanti per calcolare il modulo di Young del substrato del silicone. Superficie superiore del substrato del giunto con una proteina della matrice extracellulare di moduli (oltre le perline fluorescenti) consente il substrato del silicone per poter essere facilmente utilizzate per placcatura delle cellule e gli studi successivi utilizzando gli esperimenti di microscopia di forza di trazione. L'uso di silicone rigido, invece di una capsula di Petri, come base di morbido silicone, consente di utilizzare mechanobiology studi che coinvolgono il tratto esterno. Un vantaggio specifico di questo protocollo è che un microscopio a fluorescenza widefield, che è comunemente disponibile in molti laboratori, sia le principali attrezzature necessarie per questa procedura. Dimostriamo questo protocollo misurando il modulo di Young di substrati di morbido silicone di diversi moduli elastici.
Introduzione
Cellule in tessuti molli risiedono in un micro-ambiente in cui la rigidità è in kilopascal gamma1, contrariamente ai piatti di coltura del tessuto cui rigidità è diversi ordini di grandezza superiore. I primi esperimenti con cellule su substrati molli rivestite con proteine di matrice extracellulare ha mostrato che la rigidità del substrato influenza come cellule spostare e come aderiscano alla matrice extracellulare sotto2,3. Infatti, la rigidità del substrato influenza fondamentalmente il cellulare funzione4 in un modo simile a segnali biochimici pervasivi. Gel di poliacrilammide (rivestito con proteine della matrice extracellulare) sono (acqua-permeando) idrogel che sono stati ampiamente utilizzati come substrati di coltura di cella per mechanobiology studi5. Polidimetilsilossano (PDMS), il silicone più comune (polisilossano), è stato ampiamente usato come un silicone rigido con rigidità megapascal-gamma per micron-scala fabbricazione6. Più recentemente, in morbido silicone substrati con rigidità nella gamma kilopascal più fisiologicamente rilevanti sono state impiegate come substrati di coltura cellulare per mechanobiology studi7,8.
Diversi metodi sono stati utilizzati per misurare la rigidità di substrati flessibili, tra cui la microscopia a forza atomica, deformazione macroscopica di interi campioni su stretching, reologia e i rientri utilizzando sfere e sfericamente con punta microindentors9 . Mentre ogni tecnica ha i suoi vantaggi e svantaggi, rientro con una sfera è un metodo particolarmente semplice ma abbastanza preciso che richiede solo l'accesso a un microscopio a fluorescenza widefield. Rientro con una sfera metallica è stato utilizzato per misurare la rigidità degli idrogeli lavoro anteriore3,9,10. Primi lavori che hanno dimostrato l'importanza della rigidità del substrato al movimento cellulare utilizzarono questo metodo per determinare idrogel substrato rigidità3. Più recentemente, la microscopia confocale è stata utilizzata anche per un elegante caratterizzazione10.
Qui, presentiamo un protocollo dettagliato per la preparazione di un substrato morbido silicone, perline fluorescenti di accoppiamento (e una proteina della matrice extracellulare quali il collagene ho) solo per la superficie superiore, una sfera di rientro e il top con superficie di imaging di fase e fluorescenza di imaging, rispettivamente e infine analizzando le immagini per calcolare il modulo di Young del substrato del silicone. Il substrato di silicone morbido preparato in questo modo può essere prontamente utilizzato per gli esperimenti di microscopia di forza di trazione. L'uso di silicone rigido (anziché una capsula di Petri) come base per il silicone morbido consente inoltre studi mechanobiology utilizzando un tratto esterno. Nei casi giustificati, sono indicati anche considerazioni pratiche necessarie per evitare possibili complicazioni.
Protocollo
1. fabbricazione di substrato morbido Silicone
- Pesare 1,75 g di un componente e 1,75 g di componente B (A:B = 1:1) dal kit di elastomero di silicone morbido utilizzando (polistirolo) pesatura vassoi.
- Aggiungere un componente a componente B nel vassoio di pesatura e mescolarli insieme per 5 min usando un bastoncino di appropriato.
- Aggiungere la miscela di cui sopra una piastra di Petri da 35 mm. Lasciare il composto diffondere in modo uniforme attraverso la piastra di Petri per un paio di minuti.
Nota: La scelta del diametro di Petri e la quantità di silicone morbido determinerà lo spessore di silicone morbido. Qui, lo spessore sarà circa 3,5 mm; Ulteriori informazioni sulla scelta dello spessore di elastomero nella sezione discussione . - Posto di Petri con la miscela di silicone, con il coperchio fuori, in una camera a vuoto per 15 min rimuovere eventuali bolle d'aria. Durante questo tempo, pre-riscaldare un piatto caldo a 70 ° C.
- Una volta che la piastra riscaldante raggiunge i 70 ° C, inserire una lastra di vetro su di esso e quindi inserire la capsula di Petri con la miscela di silicone sul vetrino. Lasciate che il silicone della cura a 70 ° C per 30 min. Non posizionare il piatto di polistirolo direttamente sulla piastra calda, come la capsula di Petri potrebbe sciogliersi.
2. accoppiamento di microsfere fluorescenti al Silicone morbido
- Posizionare il curato silicone morbido (in di Petri scoperti) in una camera di UV profonda (un recinto con una lampada UV profonda di lunghezze d'onda luminose di 185 e 254 nm). Esporre il campione di morbido silicone (~ 5-10 cm dalla lampada UV) a profonda luce UV per 5 min.
- Mentre il silicone è essendo esposto a luce UV profonda, procedere con passaggi 2.2-2.6 sotto. Dopo l'esposizione ai raggi UV profonda, degassare il profondo camera UV per almeno 5 min prima di recuperare il campione.
- Nel frattempo, pesare 19 mg di 1-ethyl-3-(3-dimethylaminopropyl) carbodiimide (EDC) in una provetta da microcentrifuga da 1,5 mL e aggiungere 500 μL di acqua deionizzata (DI) ad esso. Sciogliere l'EDC agitando delicatamente il tubo.
- In un tubo del microcentrifuge separata 1,5 mL, pesare 11 mg di N- Idrossisulfosuccinimide (sulfo-NHS), aggiungere 500 μL di acqua deionizzata ad esso e sciogliere il sulfo-NHS agitando delicatamente il tubo. Quindi, combinare le soluzioni EDC e sulfo-NHS in un tubo del microcentrifuge singolo.
- Per questa soluzione EDC/sulfo-NHS, aggiungere 30 μL di 0,44 μm diametro rosso (o qualsiasi altro colore fluorescenza basato sui cubi di filtro disponibili nel microscopio di fluorescenza) carbossilato per volta microsfere fluorescenti (con una concentrazione stock 1% p/v).
- Aggiungere la miscela EDC/sulfo-NHS/tallone, 0,02 mg di collagene I (da una coda di topo, stock concentrazione di 4 mg/mL in 0.02 M di acido acetico) per ottenere una concentrazione di circa 0,02 mg/mL.
- Vortice il EDC/NHS/tallone/collagene miscela brevemente per garantire che le perle sono uniformemente dispersi, prima dell'accoppiamento.
- Pipettare 1 mL di EDC/NHS/tallone/collagene ho miscela su un pezzo di parafilm posizionato sopra un altro coperchio poco profondo, piatto (di diametro inferiore). Capovolgere la capsula di Petri con silicone morbido su questa miscela in modo che la superficie in morbido silicone Contatta la miscela ma non tocca direttamente la superficie del coperchio più piccolo di Petri qui sotto. Per alzare il piatto di Petri invertito, utilizzare uno o due vetrini sotto entrambi i lati dei distanziali invertito di Petri.
Nota: Fare riferimento alla Figura 1 per vedere come viene eseguita passo 2.7. - Coprire il campione con carta stagnola e incubare a temperatura ambiente per 30 min.
- Rimuovere la capsula di Petri con silicone morbido e metterlo in posizione verticale (in silicone-lato alto).
- Lavare la superficie in morbido silicone con tampone fosfato salino (PBS) aggiungere 2 mL di PBS (pH 7.4) al piatto. Lasciate riposare per un paio di minuti. Aspirare la PBS e lavare il silicone nuovo con 2 mL di PBS. Lasciate che la cura di silicone ulteriormente per circa un giorno. Per questo motivo, posizionare il campione di morbido silicone in PBS a 37 ° C durante la notte.
3. misurazione della rigidità del Silicone con sfera rientro utilizzando un microscopio a fluorescenza Widefield
- Recuperare la capsula di Petri con silicone morbido e assicurarsi che contenga almeno 1 mL di PBS per avere la superficie in silicone diversi mm sotto la superficie del liquido.
- Usando la pinzetta appuntita, cadere cinque penetratori di sfera zirconio 1 mm sopra il silicone morbido. Immergere le sfere nel liquido di coltura e rilasciarli, lontano dai bordi di almeno 5 indentor diametri distanza dalla posizione di altri i penetratori e lo strato di silicone.
Nota: Quando è sceso di sopra della superficie del liquido, le sfere riesca a inserire il mezzo liquido (galleggiante) a causa della tensione superficiale del liquido di coltura. - Posizionare la capsula di Petri con silicone morbido sul tavolino del microscopio in modo che è possibile immagine attraverso la base di Petri.
- Usando la formazione immagine di fase con un obiettivo da 10x (ad esempio un secco 10 X oggettiva di NA 0,30), individuare e mettere a fuoco un penetratore a sfera.
- Prendere un'immagine di fase di una parte o la totalità del penetratore e salvare quest'immagine. Se disponibile, utilizzare una scansione delle mattonelle. Se il penetratore ha eventuali difetti visibili, scartare e sostituirlo con un altro penetratore.
- Sotto imaging fase dal vivo, pan a sinistra del bordo del penetratore modo che il bordo sinistro della cornice è almeno una distanza di ~1.5 R dal centro del penetratore. Assicurarsi che il centro del penetratore rimane visibile sul lato destro, vicino al bordo destro del fotogramma dell'immagine. Prendere un'immagine di fase e salvarlo.
- Passare alla sorgente di luce microscopio l'illuminazione per il canale rosso fluorescente. Con x- e y-Coordinate invariate ( x-y posizione del centro indentor all'interno, ma vicino al bordo destro della cornice), messa a fuoco verso il basso (diminuzione Z) fino a quando le microsfere fluorescenti rosse sotto centro di penetratore della sfera Basta andare fuori fuoco.
- Prendere un z-stack con un'immagine per ogni z-incremento di 0,5 µm fino alle microsfere nello strato superiore del silicone lontano il penetratore (vicino al bordo sinistro della cornice imaging) andare fuori fuoco.
- Ripetere i passaggi da 3.4-3.8 con le altre penetratori sul campione.
4. calcolo rigidezza del Silicone (modulo di Young)
- Aprire l'immagine di fase del penetratore usando ImageJ, fare clic sullo strumento linea e misurare il diametro del penetratore in pixel. Cliccare e tenere premuto su un punto sul bordo penetratore, spostare il cursore in un punto diametralmente opposto sul bordo e nota la lunghezza in pixel visualizzati sulla barra di stato della finestra principale di ImageJ prima di rilasciare il cursore.
- Verificare che l'unità di lunghezza è impostata su pixel facendo clic Analyze | Impostare la scala e controllando l' unità di lunghezza.
- Raggio di penetratore in pixel per convertire in μm tenendo conto l'ingrandimento dell'obiettivo e la dimensione in pixel fotocamera CCD (R in μm = R in pixel x la dimensione in pixel fotocamera CCD in μm / l'ingrandimento dell'obiettivo).
- Aprire il canale rosso z-stack di immagini della microperla (se le microsfere sono rosso fluorescente) in ImageJ cliccando sul File | Importazione | Sequenza di immagini e selezionare qualsiasi immagine nello stack e fare clic su OK per aprire lo stack.
Nota: F1 è il numero del fotogramma in cui le microsfere sotto il centro del penetratore sono nella migliore messa a fuoco possibile e F2 è il numero del fotogramma in cui le microsfere (in una regione di ~1.5 R dal centro della perlina) vicino al bordo sinistro della cornice sono nella migliore messa a fuoco possibile. La z-differenza tra i due telai è il δ di profondità di rientro.- Utilizzando lo strumento di riga di ImageJ, tracciare una linea attraverso una ben definito della microperla nell'immagine. Fare clic su analizzare | Tracciare il profilo e fare clic sul pulsante Live per ottenere l'intensità di scansione linea aggiornata attraverso il tallone mentre si selezionano diversi fotogrammi. Il telaio che dà il valore massimo dell'intensità massima può essere scelto come il telaio a fuoco.
- Poiché l'incremento di z tra i frame nello stack di z è 0,5 μm, calcolare la profondità di rientro in μm come δ = (F2-F1) x 0,5.
- Calcolare la forza esercitata sul gel dal penetratore grazie al suo peso (meno la forza avversaria capace di galleggiare), vale a dire, il rientro della forza F, come il volume del penetratore x (la densità del penetratore - la densità del mezzo liquido) x accelerazione di gravità. Utilizzare l'equazione F = (4/3) x 3.142 x (R3) x (ρpenetratore - ρmedio) x g dove R è il raggio del penetratore,penetratore ρ è la densità del penetratore, ρmedio è la densità del mezzo liquido e g è l'accelerazione di gravità (9,81 m/s2). Esprimere tutte le quantità sul lato destro in unità di SI di ottenere F (in N).
- Calcolare il modulo di Young (E) del silicone utilizzando un modificato11 equazione12 di Hertz modello:
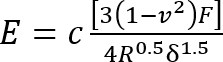
Dove:
c = fattore di correzione che consente di modificare l'espressione del modello di Hertz che lo segue;
v = rapporto di Poisson del gel del silicone (preso come 0,5 per quanto riguarda materiali incomprimibile7);
F = la forza di rientro;
R = raggio del penetratore; e
Δ = la profondità di rientro.
Esprimere tutte le quantità sul lato destro in unità di SI di ottenere E in PA.- Calcolare il fattore di correzione c come segue3:
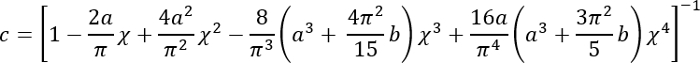
Dove: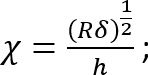
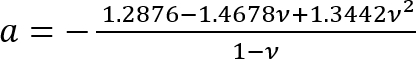 ; e
; e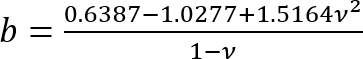 .
.
Dovrebbe essere notato che questo fattore di correzione è specificamente per essere utilizzato solo quando il silicone morbido aderisce bene ai Petri (o in silicone rigido) di sotto di esso (che è il caso qui). - Calcolare l'altezza h dello strato di morbido silicone basato sulla quantità di silicone aggiunto e il diametro della piastra di Petri. In alternativa, ottenere h direttamente determinando la coordinata z delle superfici superiore e inferiore dello strato in silicone da formazione immagine di fase (minore impurità vengono messi a fuoco a entrambe le superfici). Si noti che per un grande h (h2 > Rδ), il fattore di correzione c è vicino a 1.
- Calcolare il fattore di correzione c come segue3:
- Ripetere i passaggi 4.1-4.4 per ogni penetratore. Media di elasticità ottenuti da ogni penetratore per ottenere il media modulo di Young per il campione del silicone.
Risultati
Utilizzando il protocollo di cui sopraelencato, abbiamo preparato in silicone morbido in una capsula di Petri da 35 mm, guarito a 70 ° C per 30 min e accoppiato microsfere fluorescenti (collagene e io) sulla superficie superiore, come schematicamente rappresentato in Figura 1. UV profondo è stato utilizzato in precedenza per la proteina eventuale accoppiamento a substrati13. Nota che (I) le condizioni di reticolazione utilizzate...
Discussione
Mentre il metodo di rientro di sfera è facile da implementare, attenzione attenta deve essere pagata per la scelta del penetratore e lo spessore del campione in morbido silicone. L'equazione utilizzata per calcolare il modulo di Young è valida sotto una serie di condizioni11e questi sono in genere soddisfatti quando lo spessore del campione del silicone è > 10% del raggio penetratore e < ~ 13 x il raggio del penetratore. Abbiamo trovato che uno spessore del silicone di 5-10 volte il raggio del ...
Divulgazioni
Gli autori non hanno nulla a rivelare.
Riconoscimenti
Vi ringraziamo di generosamente permettendo l'uso di reometro Margaret Gardel. Riconosciamo il supporto dal NIH (1R15GM116082) che ha attivato questo lavoro.
Materiali
| Name | Company | Catalog Number | Comments |
| CY 52-276 A/B silicone elastomer kit | Dow Corning | CY 52-276 | Store at room temperature |
| Thermo Scientific Pierce EDC | Fisher Scientific | PI22980 | Store at -20°C |
| Thermo Scientific Pierce Sulfo-NHS crosslinker | Fisher Scientific | PI-24510 | Store at 4°C |
| Carboxyl fluorescent pink particles, 0.4-0.6 µm, 2 mL | Spherotech, Inc. | CFP-0558-2 | Store at 4°C, do not freeze |
| 1.0 mm Acid washed Zirconium beads | OPS Diagnostics LLC | BAWZ 1000-250-33 | |
| Deep UV chamber with ozone evacuator | Novascan Technologies, Inc. | PSD-UV4, OES-1000D | |
| Wide field fluorescence microscope | Leica Microsystems | DMi8 | |
| Collagen I, from rat tail | Corning | 354236 | Stock concentration = 4 mg/ml; store at 4°C |
| ImageJ-NIH | N/A | N/A | public-domain software |
Riferimenti
- Handorf, A. M., Zhou, Y., Halanski, M. A., Li, W. J. Tissue stiffness dictates development, homeostasis, and disease progression. Organogenesis. 11 (1), 1-15 (2015).
- Pelham, R. J., Wang, Y. -. L. Cell locomotion and focal adhesions are regulated by substrate flexibility. Proceedings of the National Academy of Sciences. 94 (25), 13661-13665 (1997).
- Lo, C. M., Wang, H. B., Dembo, M., Wang, Y. L. Cell movement is guided by the rigidity of the substrate. Biophysical Journal. 79, 144-152 (2000).
- Discher, D. E., Janmey, P., Wang, Y. -. L. Tissue cells feel and respond to the stiffness of their Substrate. Science. 310, 1139-1143 (2005).
- Kandow, C. E., Georges, P. C., Janmey, P. A., Beningo, K. A. Polyacrylamide hydrogels for cell mechanics: steps toward optimization and alternative uses. Methods in Cell Biology. 83, 29-46 (2007).
- Johnston, I. D., McCluskey, D. K., Tan, C. K. L., Tracey, M. C. Mechanical characterization of bulk Sylgard 184 for microfluidics and microengineering. Journal of Micromechanics and Microengineering. 24 (3), 035017 (2014).
- Style, R. W., et al. Traction force microscopy in physics and biology. Soft Matter. 10 (23), 4047-4055 (2014).
- Lee, E., et al. Deletion of the cytoplasmic domain of N-cadherin reduces, but does not eliminate, traction force-transmission. Biochemical and Biophysical Research Communications. 478 (4), 1640-1646 (2016).
- Frey, M. T., Engler, A., Discher, D. E., Lee, J., Wang, Y. L. Microscopic methods for measuring the elasticity of gel substrates for cell culture: microspheres, microindenters, and atomic force microscopy. Methods Cell Biol. 83, 47-65 (2007).
- Lee, D., Rahman, M. M., Zhou, Y., Ryu, S. Three-dimensional confocal microscopy indentation method for hydrogel elasticity measurement. Langmuir. 31 (35), 9684-9693 (2015).
- Dimitriadis, E. K., Horkay, F., Maresca, J., Kachar, B., Chadwick, R. S. Determination of elastic moduli of thin layers of soft material using the atomic force microscope. Biophysical Journal. 82 (5), 2798-2810 (2002).
- Hertz, H. Über die Berührung fester elastischer Körper. Journal für die reine und angewandte Mathematik. 92, 156-171 (1882).
- Azioune, A., Carpi, N., Tseng, Q., Théry, M., Piel, M., Cassimeris, L., Tran, P. Protein micropatterns: a direct printing protocol using deep UVs. Microtubules: In Vivo. , 133-146 (2010).
- Bashirzadeh, Y., Qian, S., Maruthamuthu, V. Non-intrusive measurement of wall shear stress in flow channels. Sensors and Actuators A: Physical. 271, 118-123 (2018).
- Muhamed, I., Chowdhury, F., Maruthamuthu, V. Biophysical tools to study cellular mechanotransduction. Bioengineering (Basel). 4 (1), 12 (2017).
- Dumbali, S. P., Mei, L., Qian, S., Maruthamuthu, V. Endogenous sheet-averaged tension within a large epithelial cell colony. Journal of Biomechanical Engineering. 139 (10), 101008 (2017).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon