Method Article
Dissezione del locale Ca2 + segnali in cellule coltivate di targeting per membrana Ca2 + indicatori
In questo articolo
Riepilogo
Qui presentiamo un protocollo per Ca2 + imaging in neuroni e cellule gliali, che consente la dissezione di Ca2 + segnali a risoluzione subcellulare. Questo processo è applicabile a tutti i tipi di cellulari che permettono l'espressione di codificato geneticamente indicatori di Ca2 + .
Abstract
Ione calcio (Ca2 +) è una molecola universale messaggero intracellulare che spinge le vie multiple di segnalazione, che conduce alle uscite biologici diversi. Il coordinamento delle sorgenti di segnale due Ca2 + — "Ca2 + afflusso" all'esterno della cellula e memorizzare "Rilascio2 + Ca" da Ca2 + intracellulare del reticolo endoplasmatico (ER) — è considerato per essere alla base i diversi modelli spazio-temporali di Ca 2 + segnali che causano funzioni biologiche nelle cellule. Lo scopo del presente protocollo è di descrivere un nuovo Ca2 + metodo di imaging che consente il monitoraggio del momento di "Afflusso di Ca2 +" e "Ca2 + release". OER-GCaMP6f è un codificato geneticamente Ca2 + indicatore (GECI) composto da GCaMP6f, che si rivolge alla membrana esterna ER. OER-GCaMP6f può monitorare il rilascio di Ca2 + a una risoluzione temporale superiore convenzionale GCaMP6f. Combinato con membrana plasmatica targeting GECIs, lo spazio-temporali modello Ca2 + segnale può essere descritto in una risoluzione subcellulare. I targeting subcellulare Ca2 + indicatori descritti qui sono, in linea di principio, disponibile per tutti i tipi di cellulari, anche per in vivo imaging dei neuroni di Caenorhabditis elegans . In questo protocollo, introduciamo il Ca2 + imaging in cellule da linee cellulari, neuroni e cellule gliali in colture primarie dissociate e descrivere la preparazione di azione congelate dei neuroni corticali del ratto.
Introduzione
CA2 + segnali rappresentano l'elevazione della concentrazione intracellulare di Ca2 + . CA2 + è il secondo messaggero universale per le cellule eucariotiche. Utilizzo di Ca2 +, cellule funzionano tramite diverse vie di segnalazione intracellulare e inducono varie uscite biologici. Ad esempio, in neuroni, rilascio delle vescicole sinaptiche al terminale presinaptico, espressione genica nel nucleo e induzione di plasticità sinaptica presso la postsynapse sono regolati da distinti segnali di Ca2 + che precisamente attivano l'appropriato enzimi a valle in siti adatti e con una tempistica precisa1.
Modelli specifici spatiotemporal di Ca2 + segnali attivano gli enzimi specifici a valle. CA2 + segnali vengono generati dal coordinamento tra le due diverse Ca2 + fonti: afflusso di Ca2 + dallo spazio extracellulare e il rilascio di Ca2 + dal reticolo endoplasmico (ER), che serve come una Ca intracellulare2 + negozio. Il spatiotemporal Ca2 + segnalazione modello significativo per indurre una funzione specifica delle cellule è supportato anche da nanodomini di 10 – 100 µM Ca2 + generato in prossimità di canali del Ca2 + sulla membrana plasmatica o ER membrana2. D'importanza, la fonte di Ca2 + segnali è uno dei fattori più critici che determinano l'uscita biologico a valle. Nei neuroni, afflusso di Ca2 + e il rilascio di Ca2 + hanno effetti opposti sul clustering di acido gamma - aminobutirrico (GABA) recettoriA (GABAAR) alle sinapsi GABAergica, che è responsabile dell'inibizione della eccitabilità di un neurone3. Afflusso di CA2 + accompagnato da massiccia eccitazione neuronale induce la dispersione di sinaptica GABAAR cluster, mentre persistente Ca2 + rilasciare dall'ER promuove il clustering di sinaptica GABAARs. Altri gruppi hanno anche riferito che la direzione ottimizzazione dei coni di crescita è dipenda criticamente sulla fonte del Ca2 + segnale: afflusso di Ca2 + induce repulsione, mentre il rilascio di Ca2 + guide l'attrazione della crescita neuronale cono4. Di conseguenza, per comprendere appieno il Ca2 + sottostante uscite cellulari specifiche vie di segnalazione, è importante identificare la fonte di Ca2 + segnali descrivendo il Ca2 + segnali alla risoluzione subcellulare.
In questo protocollo, descriviamo un Ca2 + metodo di imaging per segnalare Ca2 + segnali alla risoluzione subcellulare, che consente la stima del Ca2 + di sorgenti di segnale (Figura 1). CA2 + microdomini appena sotto la membrana plasmatica con successo sono monitorati da codificato geneticamente Ca2 + indicatori (GECIs) mirati alla membrana del plasma tramite il collegamento del segnale della membrana plasmatica-localizzazione Lck all'interno di Src chinasi a N-termini di GECIs5. Per rilevare il modello Ca2 + segnale nelle vicinanze di ER con una migliore risoluzione spaziale e temporale, abbiamo recentemente sviluppato OER-GCaMP6f, in cui GCaMP6f6 obiettivi ER membrana esterna, utilizzando la proteina transmembrana ER. OER-GCaMP6f sensibile può segnalare il rilascio di Ca2 + dall'ER una migliore risoluzione spatiotemporal rispetto convenzionale GCaMP6f immaturi di cellule COS-77 e di cellule HEK2938, evitando la diffusione di Ca2 + e GECIs . Inoltre abbiamo confermato che la spontanea Ca2 + elevazione in astrociti ippocampali coltivati segnalato da OER-GCaMP6f ha mostrato un modello spatiotemporal diverso rispetto a quella monitorata da GCaMP6f membrana plasmatica targeting (Lck-GCaMP6f)7 ,9, che indicano che il Ca2 + imaging con OER-GCaMP6f in combinazione con Lck-GCaMP6f contribuisce alla dissezione di Ca2 + segnali alla risoluzione subcellulare per identificare le loro fonti.
Attualmente, abbiamo dettaglio il protocollo per la dissezione Ca2 + segnale in cellule HeLa e neuroni-astrociti placcati su vetrini coprioggetti colture miscelati. Il Ca2 + tecnica di imaging con GECIs indicato qui, Lck-GCaMP6f, plasma-membrana-mirati RCaMP210 (Lck-RCaMP2), e OER-GCaMP6f (Figura 1) sono applicabili a tutte le celle in cui questi GECIs può essere espresso.
Protocollo
Tutti gli esperimenti descritti qui sono stati approvati dal comitato di sicurezza RIKEN e Comitato di esperimento sugli animali, secondo le linee guida emessa dal Ministero giapponese dell'educazione, cultura, sport, scienza e tecnologia.
1. preparazione delle celle
- Preparazione di poli (ethyleneimine)-rivestito vetrini coprioggetti
Nota: Rivestimento di Poly(Ethyleneimine) (PEI) è raccomandato per l'apparecchiatura di vetro, in quanto permette di neuroni e astrociti per collegare strettamente a lamelle senza impedire il loro sviluppo. Tuttavia, altri metodi di rivestimento (ad es., poli-ornitina, poli L-lisina, laminina rivestimento) sono anche disponibili, se necessario, per piatti con fondo di vetro.- Posizionare un vetrino coprioggetti di 18 mm di diametro in ciascun pozzetto di una piastra 12-pozzetti. Preparare la soluzione di PEI 0,04% (12,5 mL/12-pozzetti) utilizzando acqua sterilizzata.
- Aggiungere 1 mL di soluzione di PEI 0,04% ad ogni pozzetto. Assicurarsi che non ci sono bolle sotto i coprioggetti.
- Incubare le piastre in incubatore a CO2 durante la notte a 37 ° C.
- Il giorno dopo, lavare i vetrini coprioggetti rivestiti 3 x con 1 mL di acqua sterilizzata. Rimuovere la soluzione di PEI con un aspiratore, aggiungere 1 mL di acqua sterilizzata in ciascun pozzetto e agitare la piastra 12-pozzetti, in modo che la soluzione di PEI tra il vetrino coprioggetto e la piastra può essere lavata accuratamente. Come il PEI rimanenti è tossico per le cellule, assicurarsi che l'acqua dopo il lavaggio finale viene aspirato completamente.
- Asciugare e sterilizzare i coprioggetti all'interno della cappa con raggi ultravioletti (UV) luce per almeno 15 min. Il piatto di PEI-rivestiti sono memorizzabili a 4 ° C fino a 2 mesi. Illuminare i piatti con luce UV per 15 min prima dell'uso.
- Aggiungere 5 mL di acqua distillata sterile nello spazio tra i pozzetti per evitare l'evaporazione del terreno di coltura.
- Linee cellulari di placcatura
Nota: Questo protocollo fornisce solo un esempio per la transfezione in cellule da linee cellulari di mammifero, come le cellule HeLa e cellule COS-7. Gli utenti possono applicare altri protocolli di transfezione che sono ottimizzati per i loro esperimenti. In questa sezione, descriveremo il protocollo di coltura delle cellule HeLa, che è applicabile anche alle cellule COS-7.- Il giorno prima la transfezione, coltura le cellule in una piastra di coltura 10 cm fino a quando non raggiungono la confluenza di 70 – 90%.
- Preriscaldare il terreno di coltura (Vedi Tabella materiali) a 37 ° C.
- Lavare le cellule 2 x con tampone fosfato salino senza Ca2 + e Mg2 + (PBS [-]).
- Aspirare il PBS(-), aggiungere 1 mL di soluzione 0,5% tripsina-etilendiamminotetraacetico (EDTA) l'acido e incubare le cellule a 37 ° C per 90 s fino a raggiungere una forma rotonda.
- Aggiungere 9 mL di preriscaldati terreno di coltura per fermare il trypsinization. Diluire le cellule con terreno di coltura ad un rapporto di 1:6.
- Semi di 1 mL delle cellule diluite sulle lamelle rivestite su PEI nelle piastre 12-pozzetti.
- Preparazione della cultura hippocampal del neurone-Astrocita miscelato da ratti o topi
Nota: Sezioni 1.3 e 1.4 in primo luogo deve essere rivisto e approvato da un comitato di utilizzare e istituzionali Animal Care e deve seguire le procedure approvate ufficialmente per la cura e l'uso degli animali da laboratorio. Il diagramma di flusso del protocollo coltura neuronale è illustrato nella Figura 2.- Preparare tutti i reagenti sotto la cappa a flusso laminare. Medio dell'Aquila di luogo Dulbecco modificato (DMEM) in due 100 mm cultura piatti (circa 20 mL/piatto) che sono in una ghiacciaia.
- Preparare 50 mL di terreno dissezione composto di DMEM e 20 mM 4-(2-hydroxyethyl)-1-piperazineethanesulfonic acido (HEPES) (Vedi Tabella materiali) e dispensare il mezzo in tre piatti di cultura di 60 mm (circa 7 mL/piatto) e otto 35 mm piastre di coltura (circa 2 mL/piatto). Posizionare i piatti in un altro frigo.
- Preparare 50 mL di soluzione fisiologica l'incubazione, composta da soluzione salina bilanciata di Hanks' completati con 20 mM HEPES (Vedi Tabella materiali) e posto 8 mL di salino in una provetta conica da 15 mL sul ghiaccio.
- Preparare il supporto di placcatura con mezzo minimo essenziale (MEM) completato con B-27, glutamina e penicillina-streptomicina (Vedi Tabella materiali). Mantenere questo mezzo a temperatura ambiente (20 – 28 ° C).
- Sterilizzare gli strumenti chirurgici con etanolo al 70%.
- Posizionare un asciugamano di carta in un vaso di vetro con un coperchio e 1ml di isoflurane. Lasciate l'isoflurano evaporare per 1 min.
- Mettere un ratto incinto o un mouse nel recipiente di preparati secondo punto 1.3.6 e tenere l'animale nel vaso fino a quando non si è anestetizzato (circa 30 s a 1 min).
- Prendere l'animale anestetizzato dal vaso e disinfettare l'animale e l'apparecchiatura di dissezione di spruzzare con etanolo al 70%. Tagliare la linea ventrale mediana con pinzette e forbici per dissezione standard ed estrarre l'utero dal ratto incinto o del mouse.
- E18 – 19 embrioni dall'utero di un ratto femmina anestetizzato o il mouse, usando le forbici per dissezione delicate, estratti e gli embrioni estratti con una pinza anello in DMEM ghiacciata in un piatto di 10 cm per l'anestesia freddo.
- Decapitare l'embrione con le forbici dissezione bene e posizionare la testa in DMEM ghiacciata in un piatto di 10 cm.
- Estrarre il cervello da ogni embrione con un 13 cm curvata Semken forcipe ed il forcipe con estremità sottili. Mantenere il cervello in mezzo dissezione ghiacciata in un piatto di 60 mm.
- Rimuovere ippocampi, utilizzando due pinze con punte fini, nel mezzo di dissezione ghiacciata in piastre da 35 mm e mantenere il tessuto isolato in incubazione saline collocato in una provetta conica da 15 mL sul ghiaccio.
- Lavare ippocampi con soluzione fisiologica incubazione e incubare con tripsina (1,25 mg/mL) e dnasi I (0,25 mg/mL) in una soluzione salina l'incubazione per 5 min a 37 ° C. Il volume di incubazione raccomandata è 2,7 mL di salino incubazione, 150 µ l di 20x stock tripsina e 150 µ l di brodo di 20 x dnasi (Vedi Tabella materiali).
- Lavare ippocampi 3 x con soluzione salina ghiacciata incubazione.
- Aspirare la soluzione salina di incubazione e aggiungere 1 mL di terreno placcatura contenente dnasi I (10 µ l di soluzione madre; Vedi Tabella materiali). Sospendere il tessuto di pipettaggio non più di 20 x e misurare la densità di cellule vitali, utilizzando un contatore di cellule e il dosaggio del blu di Trypan.
- Diluire le cellule ippocampali ad una densità di 1.4 x 105 vitali cellule/mL per i ratti e 2,5 x 105 vitali cellule/mL per i topi. Semi di 1 mL delle sospensioni delle cellule diluito su lamelle rivestite su PEI in piastre di coltura 12-pozzetti.
- Mantenere le cellule a 37 ° C in un incubatore a CO2 per 2 – 3 giorni.
- Togliere la soluzione di placcatura. Non lasciare che le celle a secco. Delicatamente e rapidamente aggiungere il supporto di manutenzione preriscaldata (Vedi Tabella materiali).
- Preparazione del ratto corticali del neurone-Astrocita mescolato cultura e cellule congelate e la rinascita delle colture congelate
Nota: Un metodo di crioconservazione per le cellule corticali era descritto previously11. Qui, viene fornito un protocollo modificato in cui cellule corticali possono essere conservate a-80 ° C per almeno 3 mesi. Il diagramma di flusso per questo protocollo è illustrato nella Figura 2.- Preparare DMEM, dissezione media, saline di incubazione e placcatura medio come indicato nella procedura 1.3.1–1.3.4 e la Tabella materiali.
- Preparare il supporto di lavaggio costituito da DMEM, siero bovino fetale inattivati e penicillina-streptomicina (Vedi Tabella materiali), se necessario.
- Estrarre gli embrioni E18 – 19 dall'utero di un ratto femmina anestetizzato o il mouse, utilizzando le forbici di dissezione delicata e utilizzare pinze anello per inserire ogni embrione estratti in DMEM ghiacciata per anestesia freddo.
- Rimuovere i cervelli da embrioni e tenerli nel mezzo di dissezione ghiacciata. Rimuovere il cortexes e mantenerli in incubazione saline collocata in una provetta conica da 15 mL sul ghiaccio.
- Lavare il cortexes con soluzione fisiologica incubazione e incubare la cortexes con tripsina (1,25 mg/mL) e dnasi I (0,25 mg/mL) in soluzione salina di incubazione per 5 min a 37 ° C. Il volume di incubazione consigliati per 12 cortexes è 5,4 mL di soluzione fisiologica di incubazione, 300 µ l di 20x stock tripsina e 300 µ l di brodo di 20 x dnasi I.
- Lavare il cortexes 3 x con soluzione salina ghiacciata incubazione.
- Rimuovere il supernatante e aggiungere 2 mL di terreno di placcatura completati con 150 µ l di dnasi I stock. Dissociare le cellule pipettando meno di 20 colpi e filtrare le celle utilizzando un colino di cella con un formato del poro di 70 µm.
- Lavare il filtro cella con 20 mL di terreno placcatura placcatura. Per la preparazione di cellula congelati stock, lavare le cellule con 20 mL di terreno di lavaggio (Vedi Tabella materiali)
- Misurare la densità delle cellule vitali, utilizzando un contatore di cellule e il metodo del blu di Trypan.
- Diluire le cellule corticali ad una densità di 1.4 x 105 vitali cellule/mL con il mezzo di placcatura e aggiungere 1 mL di sospensione cellulare diluito alle lamelle rivestite su PEI nelle piastre di coltura 12-pozzetti.
- Mantenere le cellule a 37 ° C in un incubatore a CO2 per 2 – 3 giorni e modificare il terreno di coltura per il mezzo di manutenzione.
- Dopo passo 1.4.9, preparare il brodo di cellule corticali congelato mediante centrifugazione le cellule a 187 x g per 3 minuti, utilizzando un rotore di oscillazione.
- Aspirare il supernatante e aggiungere il mezzo di crioconservazione (Vedi Tabella materiali) conservati a 4 ° C, per ottenere una densità cellulare di 1 x 107 cellule/mL. Aliquotare 1 mL di sospensione cellulare in provette criogeniche.
- Posizionare i tubi in una cella di congelamento contenitore con un tasso di congelamento di-1 ° C/min, fino a quando non viene raggiunta una temperatura di-80 ° C e trasferire il contenitore di congelamento per un congelatore-80 ° C. Le cellule possono essere conservate per almeno 3 mesi a-80 ° C.
- Per rivivere le cellule congelate, preriscaldare il lavaggio medio (circa 13 mL per ogni tubo criogenico) e mezzo di mantenimento per cellule corticali congelate (Vedi Tabella materiali).
- Scongelare le cellule congelate rapidamente a 37 ° C in un bagno d'acqua.
- Diluire le cellule scongelate delicatamente con il mezzo di lavaggio preriscaldati. Centrifugare le cellule a 187 x g per 3 minuti, utilizzando un rotore di oscillazione.
- Sospendere il pellet in 1 mL di lavaggio medio e misurare la densità di cellule vitali.
- Diluire le cellule con il mezzo di mantenimento per cellule corticali congelate per produrre una densità cellulare di 3.0 x 105 vitali cellule/mL ed il seme di 1 mL di sospensione cellulare nelle piastre rivestite con PEI 12-pozzetti.
2. espressione di membrana-mirati GECIs
-
Transfezione delle celle
- Aggiungere 250 ng del plasmide GECI (cioè, Lck-GCaMP6f, Lck-RCaMP2 o OER-GCaMP6f con promotore CMV)7, 8,9 a 100 µ l del mezzo riduttore del siero (Vedi Tabella materiali) per pozzetto. Per il cotransfection di Lck-RCaMP2 e OER-GCaMP6f, uso 250 ng di ogni plasmide in 100 µ l di terreno di riduttore del siero in ciascun pozzetto.
- Aggiungere 0,5 µ l di reagente di transfezione (Vedi Tabella materiali) per pozzetto nella miscela medio del siero plasmide-ridotto. Per il cotransfection di Lck-RCaMP2 e OER-GCaMP6f, aggiungere 0,5 µ l di reagente di transfezione per pozzetto.
- Incubare la miscela per 30 min a temperatura ambiente (20 – 28 ° C).
- Aggiungere 100 µ l della miscela per ogni vetrino coprioggetti in modo sospensione.
- Incubare le cellule per 48 – 72 ore in incubatore a CO2 a 37 ° C per consentire l'espressione della GECIs.
-
Transfezione e infezione da virus adeno-associato di neuroni ippocampali o corticali
Nota: Transfezione per 3 – 5 giorni in vitro (DIV) si traduce in un più alto tasso di transfezione per i neuroni. La transfezione a 6 – 8 DIV è comodo per l'espressione ottima di GECIs negli astrociti. Per l'espressione di GECIs in neuroni dissociati cultura dopo 9 DIV, l'infezione dei vettori di virus adeno-associato (AAV) fornisce una migliore efficacia di espressione. Vettori AAV per l'espressione di Lck-GCaMP6f, Lck-RCaMP2 e OER-GCaMP6f sotto il EF1a promotori sono stati preparati come descritto in precedenza, utilizzando HEK293 celle12 (Vedi Tabella materiali).- Per la transfezione 3 – 8 giorni dopo il placcaggio, etichetta due tubi, uno per il DNA del plasmide e l'altra per reagente di transfezione.
- Aggiungere 50 µ l di siero riduttore terreno (Vedi Tabella materiali) per pozzetto in ogni provetta.
- Aggiungere 0,5 µ g di plasmide DNA per vetrino coprioggetto e 1 µ l di integratore accompagnato da reagente di transfezione per i neuroni (Vedi Tabella materiali) per pozzetto per il tubo di DNA del plasmide. Per il cotransfection di Lck-RCaMP2 e OER-GCaMP6f, 0,5 µ g di ogni plasmide e 1 µ l di supplemento sono mescolati in 50 µ l di siero riduttore terreno per pozzetto.
- Aggiungere 1 µ l di reagente di transfezione (Vedi Tabella materiali) per pozzetto nella provetta del reagente di transfezione. La stessa quantità di reagente di transfezione è utilizzata per cotransfection.
- Vortice entrambe le provette per 1 – 2 s.
- Aggiungere la miscela di reagente di transfezione (dal punto 2.2.4) alla miscela di DNA (dal punto 2.2.3). Mescolare pipettando delicatamente e incubare la miscela (100 µ l per vetrino coprioggetti) per 5 min a temperatura ambiente.
- Questa miscela sulle celle di carico in modo sospensione.
- Incubare le cellule in un incubatore a CO2 per 2 – 3 giorni fino a quando sono espresse le proteine marker.
- Per infezione AAV, aggiungere 3 µ l di AAV alla cultura mista neuroni-astrociti. Mescolare delicatamente a dondolo il piatto. Per la doppia infezione di Lck-RCaMP2 e OER-GCaMP6f, 3 µ l di ogni AAV è stato introdotto per pozzetto. Nel caso in cui i numeri delle cellule che esprimono GECIs sono insufficienti, la quantità ottimale di AAV per l'infezione dovrebbe essere determinata.
- Mantenere la cultura per 1 – 2 settimane fino a quando il GECIs sono espressi.
3. Ca2 + Imaging
-
La formazione immagine simultanea delle cellule che esprimono Lck-RCaMP2 e OER-GCaMP6f
Nota: Per registrare simultaneamente i segnali Lck-RCaMP2 e OER-GCaMP6f, suddivisione dell'immagine ottica sono necessari. Le ottiche permettono la separazione tra RCaMP2 e GCaMP6f e la loro proiezione sulla stessa cornice fotografica della macchina fotografica (Figura 3A). Formazione immagine simultanea inoltre richiede (1) i sorgenti che contemporaneamente possono emettere luce nel blu (450-490 nm) di eccitazione e spettri di verde (500 – 560 nm), filtro (2) doppia banda e specchio dicroico imposta nel microscopio (3) emissione filtri per RCaMP2 e GCaMP6f. Per informazioni dettagliate, fare riferimento alla Tabella materiali.- Accendere i dispositivi di imaging e computer almeno 30 min prima della registrazione. Preriscaldare il microscopio riscaldanti a 37 ° C. Impostare il dispositivo di immagine-splitting, filtri e fonte di luce. Allineare l'ottica di suddivisione dell'immagine in modo che lo stesso campo di vista appare sulla fotocamera. Scegliere l'obiettivo appropriato (Vedi Tabella materiali).
- Montare il vetrino coprioggetto contenente le cellule transfected con Lck-RCaMP2 e OER-GCaMP6f nella camera di registrazione, aggiungere il supporto imaging appropriato o il buffer (400 µ l per vetrini coprioggetto 18 mm) in aula e posizionarlo sulla fase di microscopio. Mettere un coperchio sulla camera di registrazione per evitare l'evaporazione del fluido.
- Individuare le cellule che esprimono sia Lck-RCaMP2 e OER-GCaMP6f da formazione immagine di fluorescenza. Ridurre al minimo l'intensità della luce di eccitazione per evitare photobleaching e fototossicità.
- Togliere il coperchio e iniziare una registrazione time-lapse a 10 Hz. Durante questa registrazione, è necessario aggiungere l'agonista alla camera per evocare Ca2 + risposte (ad es., istamina per cellule HeLa, ATP per cellule COS-7).
- Salvare i dati di time-lapse in unità disco rigido (HDD).
- Analizzare i dati utilizzando software di analisi di immagine.
-
Registrazione attività spontanea dei astrocytes esprimendo Lck-RCaMP2 e OER-GCaMP6f
Nota: Senza suddivisione dell'immagine ottica, i Ca2 + segnali di membrana del plasma e quelli intorno al pronto soccorso possono essere monitorati nella stessa cella. Qui, la registrazione sequenziale di Lck-RCaMP2 e OER-GCaMP6f in astrociti stessi è descritto. Un obiettivo a immersione in olio con un'apertura numerica più grande 1.3 è altamente raccomandato per attività spontanea di Ca2 + .- Accendere il microscopio, fotocamera, fonte di luce e il riscaldamento del microscopio dell'alloggiamento almeno 30 min prima della registrazione.
- Montare il vetrino coprioggetto contenente le cellule transfettate con Lck-RCaMP2 e OER-GCaMP6f nella camera di registrazione e aggiungere 400 µ l del mezzo imaging. Mettere un coperchio sulla parte superiore della camera.
- Scegliere il filtro impostato per GCaMP6f e la sorgente di luce (luce di eccitazione, ad es. 470– 490 blu nm; si veda Tabella materiali). Individuare gli astrociti esprimendo OER-GCaMP6f.
- Scegliere un set di filtri e la sorgente luminosa per RCaMP2 (luce di eccitazione, per esempio, 510 – 560 nm verde; Vedi Tabella materiali) e confermare se Lck-RCaMP2 è espresso in astrociti stessi. Evitare la lunga esposizione alla sorgente luminosa per prevenire photobleaching.
- Registrare immagini time-lapse di Lck-RCaMP2 a 2 Hz per 2 min. salvare i dati di imaging su disco fisso.
- Cambiare il filtro impostato che per GCaMP6f. Registrare immagini time-lapse di OER-GCaMP6f nello stesso campo di vista, a 2 Hz per 2 min. salvare i dati sul disco rigido.
- Analizzare i dati utilizzando il software di analisi di immagine.
-
Registrazione attività neuronale spontanea ed indotta Ca2 + elevazione nei neuroni
Nota: Il programma di installazione di microscopio per l'imaging di un neurone è lo stesso come descritto nella sezione 3.2. Qui, l'imaging del Ca2 + elevazione spontanea a causa di Lck-GCaMP6f e Ca2 + elevazione indotta dall'attivazione di mGluRs causa OER-GCaMP6f è descritto.- Per registrare l'attività neuronale spontanea, montare il vetrino coprioggetto che contiene le cellule che esprimono Lck-GCaMP6f nella camera di registrazione e aggiungere 400 µ l di medie di imaging. Mettere un coperchio sulla parte superiore della camera.
- Impostare il filtro e la sorgente di luce (blu eccitazione, ad es. 470-790 nm; si veda Tabella materiali) a quelli per GCaMP6f. Trovare i neuroni che esprimono Lck-GCaMP6f e mostrando attività spontanea.
- Acquisire immagini a 2 Hz o più veloce. Salvare i dati sul disco rigido.
- Analizzare i dati utilizzando il software di analisi di immagine.
- Per registrare il Ca2 + elevazione indotta, montare i vetrini coprioggetti contenente le cellule che esprimono OER-GCaMP6f con 400 µ l mezzo di imaging. Mettere un coperchio sulla parte superiore della camera.
- Utilizzando il filtro impostato per GCaMP6f, trovare i neuroni che esprimono OER-GCaMP6f.
- Rimuovere il coperchio. Avviare la registrazione time-lapse a 2 Hz o più veloce. Durante la registrazione, aggiungere gli agonisti per un recettore accoppiato a proteine Gq (ad es., mGluR5 agonista [RS] - 3,5 - dihydroxyphenylglycine [DHPG]) per evocare un rilascio di Ca2 + dall'ER.
- Salvare le immagini time-lapse sul disco rigido e analizzare i dati.
Risultati
LCK-RCaMP2 e OER-GCaMP6f sono stati espressi in cellule HeLa, ed entrambi i segnali sono stati registrati simultaneamente mediante suddivisione dell'immagine ottica, 24 ore dopo la trasfezione (Figura 3A e Video 1). Le immagini sono state acquisite a 10 Hz. istamina (sua, 1 µM), che induce il rilascio di Ca2 + dall'ER, è stato aggiunto durante la registrazione. Al momento dell'applicazione del suo, è aumentato l'intensità del segnale di Lck-RCaMP2 e OER-GCaMP6f, come mostrato dal display pseudocolor ΔF/F0, che rappresenta il cambiamento dall'intensità di fluorescenza iniziale (Figura 3B). I corsi di tempo di Ca2 + elevazione (ΔF/F0) segnalati da Lck-RCaMP2 e OER-GCaMP6f sono stati confrontati nella stessa regione di interesse (ROI) (Figura 3). I valori ΔF/F0 sono stati normalizzati i valori di picco per consentire il confronto di corso di tempo tra le due GECIs diversi, che hanno livelli di espressione diversa e distribuzioni. Entrambi i sensori segnalato un'oscillazione-come Ca2 + elevazione. LCK-RCaMP2 e OER-GCaMP6f ha mostrato lo stesso corso di tempo per Ca2 + quota altimetrica in due cellule tra i tipi di cinque cella esaminato (Figura 3, ROI 1 e 3). Tuttavia, il Ca2 + elevazioni mostrate da Lck-RCaMP2 è rimasto a un livello superiore rispetto a quella mostrata da OER-GCaMP6f (Figura 3 C, ROI 2, 4 e 5). I risultati indicano che il Ca2 + quota altimetrica è prolungata in prossimità della membrana plasmatica, mentre viene terminato prima nei dintorni di ER, che è la fonte di questo Ca2 + segnale indotto dalla sua stimolazione.
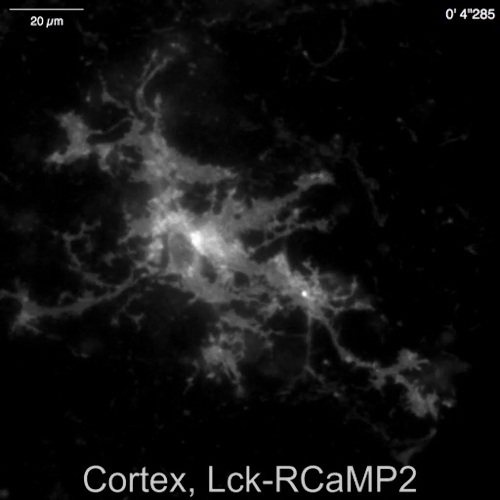
Spontaneo Ca2 + segnali dagli astrociti in neuroni-astrociti mescolato cultura da ippocampi di ratto (Figura 4A) e cortexes (Figura 4B) sono stati indicati da Lck-RCaMP2 e OER-GCaMP6f (Figura 4, Video 2, Video 3 , Video 4e 5). Colture corticali sono stati fatti rivivere da stock congelati che è stato preparato come descritto in questo protocollo. LCK-RCaMP2 e OER-GCaMP6f segnali sono stati registrati in sequenza a 2 Hz dalle cellule stesse. Tre ROIs sono stati selezionati nell'area che ha mostrato Ca2 + altezza da ogni GECI, e il corso di tempo del ΔF/Fbase (cioè, l'intensità di fluorescenza) cambiato dall'intensità di fluorescenza media durante il periodo di registrazione intera (Fbase ). Quando la fluorescenza di base è stabile e Ca2 + elevazioni sono meno frequenti, Fbase diventa una linea di base utile per rilevare gli eventi di Ca2 + elevazione. Ca2 + elevazioni spontanee erano visibili solo al processo astrocytic, non al corpo cellulare. Questo risultato è coerente con le precedenti relazioni su astrocytic spontanea Ca2 + segnali da altri GECIs visualizzati in vitro13 e vivo in14. Negli astrociti ippocampali sia corticali, Ca2 + elevazioni mostrate da Lck-RCaMP2 (in alto) erano più frequenti rispetto a quelli indicati da OER-GCaMP6f. Questo risultato è coerenza con la nostra precedente dimostrazione che Ca2 + le elevazioni in astrociti a causa di Lck-GCaMP6f sono state rilevate più frequentemente rispetto a quelli a causa di OER-GCaMP6f7 e suggerisce che questa nozione è anche applicabile presso il livello di singola cellula.
A 2 Hz (Figura 5A e Video 6) sono stati osservati spontanea Ca2 + aumenti di Lck-GCaMP6f in neuroni ippocampali di ratto acerbo (10 DIV). I corsi di tempo di ΔF/F0 in cinque differenti ROIs suggeriscono che queste elevazioni di Ca2 + localmente sono confinate ai domini subcellulari. Figura 5B (Video 7) Mostra il Ca2 + risposte in neuroni ippocampali matura del topo (30 DIV) infettate con vettori AAV OER-GCaMP6f-espressione. I neuroni sono stati stimolati con 100 µM DHPG, che è l'agonista per i recettori metabotropici del glutammato, inducendo il rilascio di Ca2 + . DHPG Ca2 + rilascio indotto dovuto OER-GCaMP6f è stato rilevato.
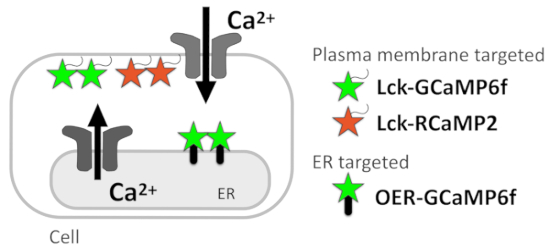
Figura 1: diagramma mostra targeting membrana GECIs. Diagramma schematico di GECIs targeting della membrana plasmatica (Lck-GCaMP6f e Lck-RCaMP2) ed esterno ER targeting membrana GCaMP6f (OER-GCaMP6f).
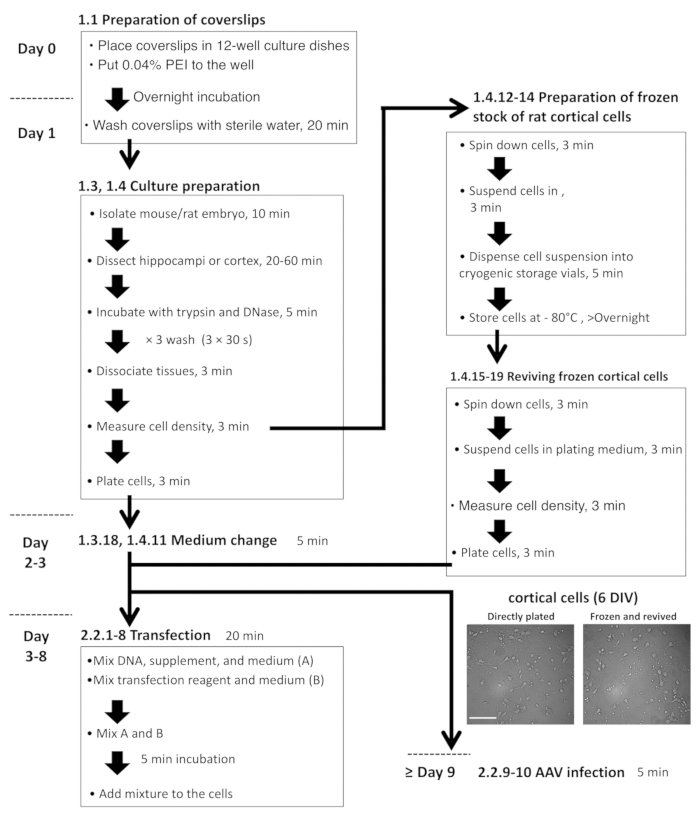
Figura 2: diagramma di flusso per la preparazione delle cellule ippocampali e corticali, trasfezione di plasmide e infezione AAV. Le immagini al microscopio sono rappresentativi, appena placcato DIV-6 cellule corticali (a sinistra) e rivivere le cellule dallo stock congelati (a destra). Barra della scala = 100 µm. Clicca qui per visualizzare una versione più grande di questa figura.
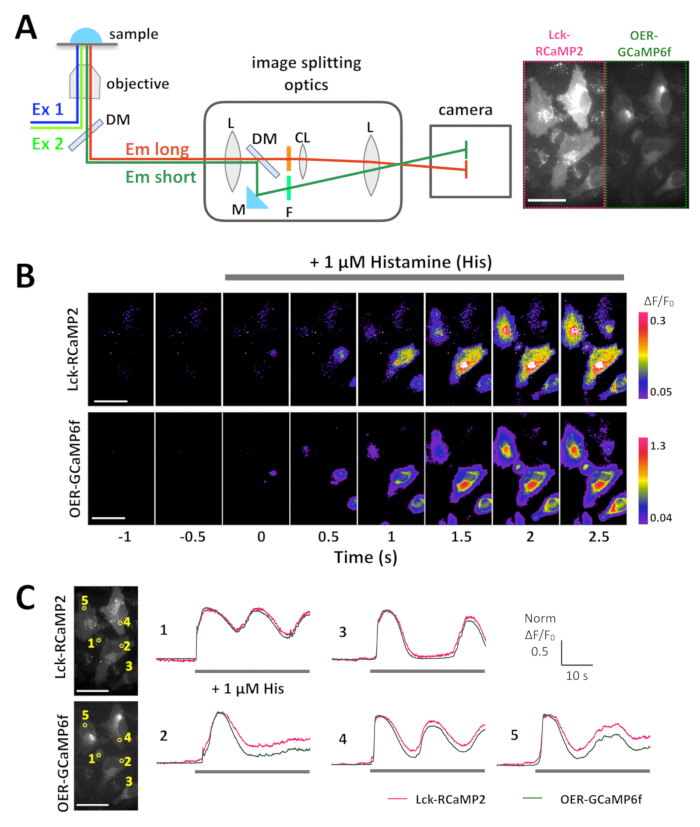
Figura 3: esempio della formazione immagine simultanea di Lck-RCaMP2 e OER-GCaMP6f in cellule HeLa. (A) rappresentazione schematica della separazione del segnale con suddivisione dell'immagine ottica. Lo stesso campo di vista per Lck-RCaMP2 e OER-GCaMP6f contemporaneamente è proiettata sulla fotocamera. Una registrazione rappresentanza acquistata a 10 Hz da una telecamera CMOS è fornita nel Video 1e un frame di questa registrazione viene visualizzato nel pannello A (a destra). (B) immagini di pseudo-colore ΔF/F0 per Lck-GCaMP2 (in alto) e OER-GCaMP6f (in basso). Istamina (sua, 1 µM) è stato aggiunto a 0 s. (C) rappresentante tempo normalizzato ΔF/F0 corso di Lck-GCaMP2 (magenta) e OER-GCaMP6f (verde). I dati sono stati normalizzati al valore massimo di ΔF/F0 per ciascuna parcella. Le barre grigie indicano i tempi della sua applicazione. I dati sono stati analizzati con un software su misura TI Workbench15. Barra della scala = 50 µm (immagine microscopica). Clicca qui per visualizzare una versione più grande di questa figura.
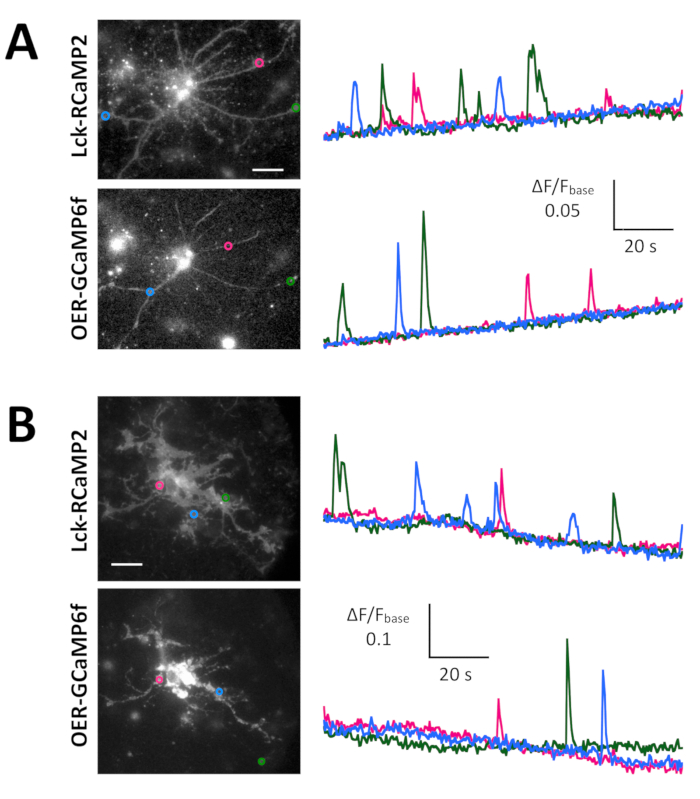
Figura 4: Spontanea Ca2 + elevazione negli astrociti monitorati per espressione Lck-RCaMP2 e OER-GCaMP6f. (A) rappresentante hippocampal e astrociti corticali (B) transfettati con Lck-RCaMP2 (in alto) e OER-GCaMP6f (in basso). Cellule corticali sono stati fatti rivivere da colture stock congelate. LCK-RCaMP2 e OER-GCaMP6f immagini in sequenza sono stati acquisiti nella stessa cella, a 2 Hz, con una telecamera EM-CCD. Le trame sulla sinistra mostrano i corsi di tempo di ΔF/Fbase misurati nella ROIs indicato nell'immagine microscopica. I dati sono stati analizzati con TI Workbench. Film reali sono forniti in Video 2e Video 3, Video 4 Video 5. La barra della scala = 20 µm (immagine microscopica). La deriva della linea di base suggerisce i cambiamenti in Ca2 + livello globale nella cella. Clicca qui per visualizzare una versione più grande di questa figura.
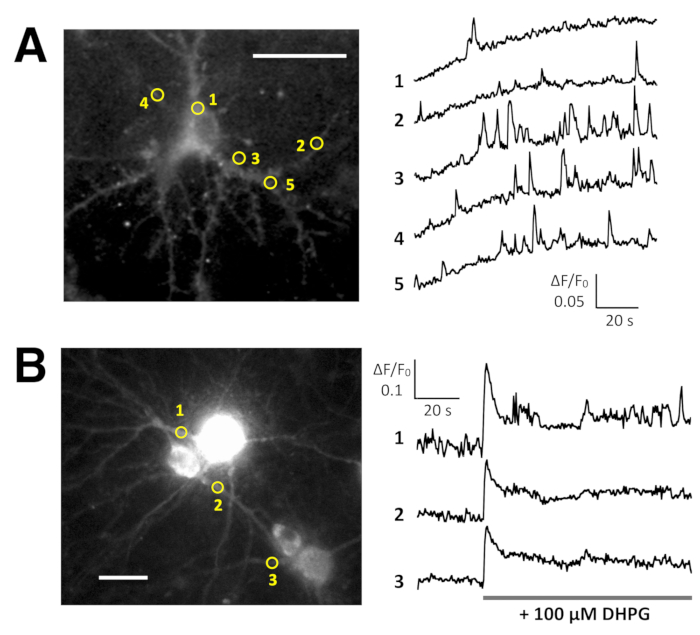
Figura 5: Esempi di Ca2 + imaging in neuroni con Lck-GCaMP6f e OER-GCaMP6f. Neuroni ippocampali di ratto rappresentativo di (A) che esprime Lck-GCaMP6f a 10 DIV (a sinistra) e trame mostrando il tempo corsi di ΔF/F0 misurata in ROIs (cerchi gialli) sono state indicate nell'immagine (a destra). I numeri nel corso tempo corrispondono ai numeri ROI nell'immagine. Si noti che lo schema temporale di Ca2 + quota altimetrica è diverso tra le varie regioni di interesse. La deriva della linea di base suggerisce l'aumento del Ca2 + livello globale in questo neurone. (B) un esempio di neuroni ippocampali di matura del topo (DIV 30) infettati con vettori di AAV espressione OER-GCaMP6f (a sinistra). Il tempo terreno di corso di ΔF/F0 misurato Mostra il Ca2 + risposta a 100 µM (RS) - 3,5 - dihydroxyphenylglycine (DHPG) applicato la tempistica indicata dalla barra grigia. Cerchi gialli indicano la posizione di ROIs dove il corso di tempo è stato ottenuto. Le immagini sono state acquisite a 2 Hz con una telecamera CCD raffreddata (pannello A) o una telecamera di EM-CCD (pannello B) e analizzate con Workbench TI. Barra della scala = 20 µm (immagine microscopica). Clicca qui per visualizzare una versione più grande di questa figura.
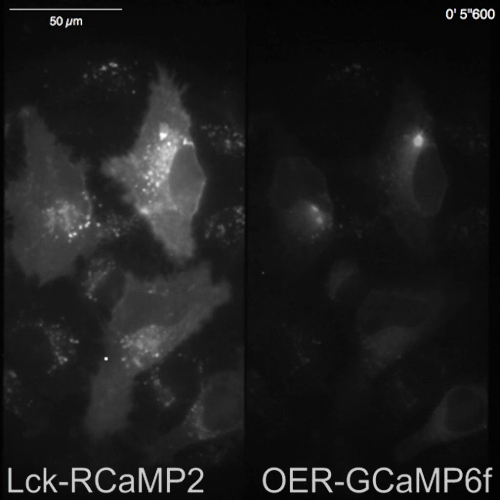
Video 1: esempio di formazione immagine simultanea di Lck-RCaMP2 e OER-GCaMP6f in cellule HeLa. Rappresentante registrazione acquisita a 10 Hz e presentato in Figura 3. Barra della scala = 50 µm. Per favore clicca qui per scaricare questo video.

Video 2: Transitorio2 + Ca spontanea osservata nel Lck-RCaMP2 in astrociti ippocampali. Rappresentante registrazione acquisita a 2 Hz (Figura 4A), registrato lo stesso campo di vista come Video 3. Barra della scala = 20 µm. per favore clicca qui per scaricare questo video.
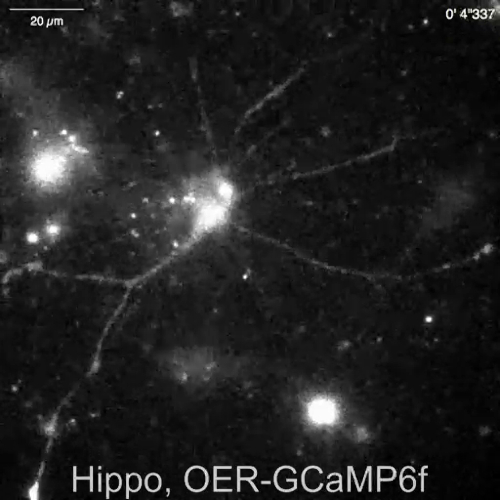
Video 3: Transitorio2 + Ca spontanea osservata in OER-GCaMP6f in un Astrocita hippocampal. Rappresentanza registrazione acquisita a 2 Hz (Figura 4A), nello stesso campo di vista come Video 2. Barra della scala = 20 µm. per favore clicca qui per scaricare questo video.
Video 4: Transitorio2 + Ca spontanea osservata nel Lck-RCaMP2 in un Astrocita corticale rappresentante registrazione acquisita a 2 Hz (Figura 4B), registrata nello stesso campo di vista come Video 5. Barra della scala = 20 µm. per favore clicca qui per scaricare questo video.
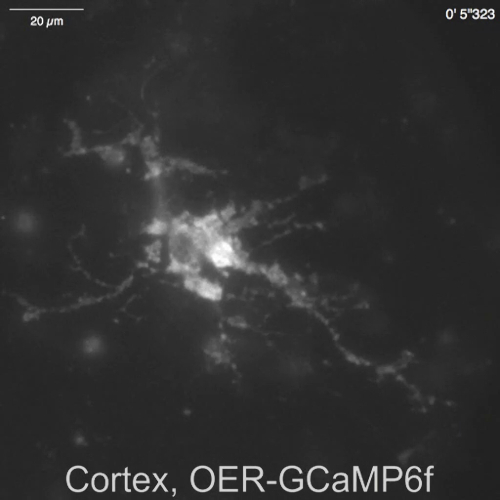
Video 5: Transitorio2 + Ca spontanea osservata in OER-GCaMP6f in un Astrocita corticale. Rappresentanza registrazione acquisita a 2 Hz (Figura 4B), nello stesso campo di vista come Video 4. La barra della scala = 20 µm. per favore clicca qui per scaricare questo video.
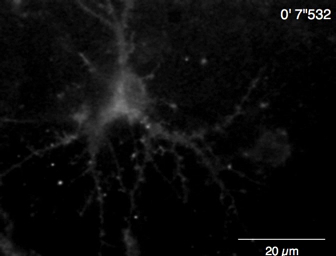
Video 6: Esempio di Ca2 + imaging in un neurone hippocampal del ratto (DIV 10) da Lck-GCaMP6f. Esempio di un neurone Ca2 + segnali registrati a 2 Hz (Figura 5A). Barra della scala = 20 µm. per favore clicca qui per scaricare questo video.
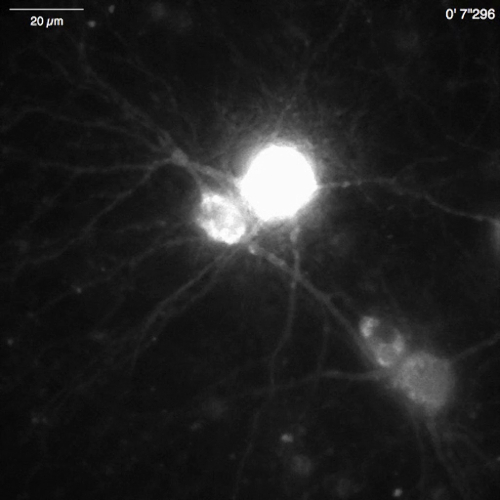
Video 7: Ca2 + rilascio in un neurone ippocampale con mouse (DIV 30) espressione OER-GCaMP6f. Esempio di Ca2 + segnali neuronali registrato in un neurone hippocampal del mouse infettato con vettori di AAV espressione OER-GCaMP6f (figura 5B). Il neurone è stato stimolato con 100 µM dihydroxyphenylglycine (DHPG) applicato a 30 s per evocare il rilascio di Ca2 + dall'ER. Barra della scala = 20 µm. per favore clicca qui per scaricare questo video.
Discussione
Diverse uscite biologici vengono avviate da Ca2 + segnali. CA2 + è un versatile Messaggero di segnalazione intracellulare. Decodifica di Ca2 + segnali per evocare uscite specifiche è stata una questione biologica fondamentale, e Ca2 + tecniche di imaging per descrivere la diversità di Ca2 + segnali sono necessari. Il protocollo attualmente dettagliato consente la rilevazione di Ca2 + segnali distinti alla membrana plasmatica ed ER (Figura 3 e Figura 4) e Ca2 + microdomini locali all'interno di una cella (Figura 4 e Figura 5). Ciò contribuisce a descrivere la diversità di Ca2 + segnali intracellulari. La risoluzione temporale di Ca2 + segnali inoltre è stata migliorata di mira GECIs nella membrana plasmatica ed ER perché può evitare l'effetto di una diffusione tridimensionale di Ca2 + e GECIs se stessi, e ha il potenziale per rilevare la momento di afflusso di Ca2 + o Ca2 + release, che si verifica sulla membrana.
Il protocollo presenta alcune limitazioni. Gli utenti dovrebbero tenere a mente che i segnali rilevati sono la sommatoria del "momento di afflusso di Ca2 + o rilascio" e "Ca2 + diffusa fuori dall'origine Ca2 +" , soprattutto per grandi Ca2 + segnali. Ad esempio, anche se la sua stimolazione in cellule HeLa evoca Ca2 + rilascio dall'ER, sua risultante Ca2 + segnali vengono rilevati non solo di OER-GCaMP6f ER-mirati, ma anche di plasma-membrana-mirati Lck-RCaMP2 (Figura 3). Un'altra limitazione è che il modello spatiotemporal di Ca2 + segnali potrebbe non essere l'unico fattore dell'uscita di Ca2 + segnali. La distribuzione delle proteine effettrici a valle (ad esempio Ca2 +-dipendente chinasi e fosfatasi) può anche essere una determinazione fattore2. Per decodificare completamente i Ca2 + segnali intracellulari, analisi del comportamento degli enzimi a valle, che non rientra in questo protocollo, sono assolutamente necessario.
Uno degli aspetti più critici per il successo Ca2 + formazione immagine è l'imaging acquisizione immagine e installazione condizioni, anche per quanto riguarda altri studi di formazione immagine live. Precedentemente abbiamo indicato che Ca2 + risposte nella cella sono altamente dipendenti sulla durata e l'intensità dell'eccitazione e su condizioni di acquisizione immagine, tra cui l'esposizione tempo e acquisizione frequenza16. Potenza di illuminazione di eccitazione è il fattore più critico, in quanto può causare tossicità luce e photobleaching di GECIs. Le condizioni di registrazione del tempo di esposizione, frequenza, intensità della luce di eccitazione e la durata di registrazione di registrazione dovrebbero essere ottimizzate in base allo scopo dell'esperimento. Si consiglia di ridurre il tempo di esposizione e l'intensità della luce di eccitazione per quanto possibile di evitare photobleaching e fototossicità alla cella. La frequenza di registrazione e la durata di registrazione dovrebbe essere sufficiente a coprire i Ca2 + elevazione eventi di interesse ma dovrebbe essere tenuti più bassi possibile evitare anche photobleaching e fototossicità. Si consiglia di determinare la frequenza di registrazione e la durata prima e ottimizzando l'intensità della luce e il tempo di esposizione, in modo che il photobleaching del GECIs è ridotto al minimo. Un altro fattore importante è il livello di espressione della GECIs. GECIs hanno un Ca2 +-buffering effetto come sono Ca2 +-proteine leganti. Di conseguenza, la sovraespressione di GECIs provoca il buffering di Ca2 +, che è fisiologicamente necessari per le cellule. La quantità di espressione GECI dovrebbe essere minimizzata per evitare imaging cellule che esprimono quantità elevate di GECIs.
In conclusione, la dissezione di Ca2 + segnali ad una risoluzione subcellulare è uno dei passi più importanti per la decodifica Ca2 + segnali intracellulari che determinano il fenomeno biologico di uscita. Questo protocollo fornisce un nuovo metodo per la dissezione di Ca2 + segnali per descrivere la diversità tra questi segnali. Attualmente, questa tecnica è limitata per gli esperimenti in vitro. Tuttavia, Lck-GCaMP6f è già utilizzato per in vivo Ca2 + imaging in topi17e OER-GCaMP6f è stata confermata per monitorare Ca2 + segnali in vivo nei neuroni di motore VD in c. elegans7. Di conseguenza, GECIs nel compartimento subcellulare di targeting ha il potenziale per essere ampliato in vivo imaging in futuro, quindi permettente Ca2 + dissezione in vivo.
Divulgazioni
Gli autori non hanno nulla a rivelare.
Riconoscimenti
Questo lavoro è supportato dalle seguenti sovvenzioni: il Giappone Science and Technology Agency (JST) / Precursory ricerca per embrionale di scienza e tecnologia (PRESTO) (JPMJPR15F8, Giappone); la società giapponese per la promozione della scienza (JSPS) / sovvenzioni in aiuti per la ricerca scientifica (KAKENHI) (JP18H05414, JP17H05710, JP16K07316), Takeda Foundation. Gli autori ringraziano Haruhiko Bito (Università di Tokyo) per la fornitura di RCaMP2 e Arthur J.Y. Huang e Thomas McHugh (RIKEN CBS) per la fornitura di vettori AAV e per le istruzioni per quanto riguarda la preparazione di AAV. Gli autori vorrei anche ringraziare gli editor presso la Gazzetta di esperimenti visualizzati per il loro aiuto con riprese video e montaggio.
Materiali
| Name | Company | Catalog Number | Comments |
| (RS)-3,5-Dihydroxyphenylglycine (DHPG) | Tocris | #0342 | |
| 0.5% DNase I stock solution | Sigma-Aldrich | #11284932001 | Prepare 0.5% DNase I (w/v) in Hanks' Balanced Salt Solution supplemented with 120 mM MgSO4. Prepare 160 µL aliquots and store at -30 °C. |
| 0.5% Trypsin-EDTA solution | Thermo Fisher Scientific | #25300054 | |
| 100 mM L-glutamine (×100 stock) | Thermo Fisher Scientific | #25030081 | Preparing small aliquots of 250-750 µL and store at -30 °C. |
| 100 mM Sodium pyruvate (×100 stock) | Thermo Fisher Scientific | #11360070 | Aliquots (10 mL) can be stored at -20 °C. After thawing, the solution can be maintained at 4 °C for 2 months. |
| 12-Well multiwell culture plates with low-evaporation lid | Falcon | #353043 | Low-evaporation lid is critical for culturing neuron-glia mixed culture. For cell line cells, alternative culture dishes can be used. |
| 18 mm diameter circular coverslips | Karl Hecht "Assistent" | #41001118 | Thickness 1, 18 mm diameter circular coverslips; alternative coverslips can be used. |
| 1 M HEPES | Thermo Fisher Scientific | #15630080 | pH 7.2-7.6 |
| 2.5% Trypsin stock solution (×20 stock) | Sigma-Aldrich | #T4674 | Prepare 150 µL aliquot and store at -30 °C. |
| 50% Poly(ethyleneimine) (PEI) solution | Sigma-Aldrich | #P3143 | Prepare 2% (V/V) PEI stock solution (×50) with distilled water sterilized by filtration. Store stock solution at -30 °C after preparing small aliquots of 250-750 µL. Prepare 0.04% PEI solution with distilled water on the day of coverslip coating. |
| 70% Ethanol | Kept in a spray bottle to be used for surface disinfection. | ||
| Adeno-associated virus (AAV) for Lck-GCaMP6f, Lck-RCaMP2, and OER-RCaMP2 expression under the direction of the EF1a promoter | AAV can be prepared using AAV Helper Free System (Agilent Technologies) and HEK293 cells, or alternative methods. pAAV.EF1a.Lck-GCaMP6f, pAAV.EF1a.Lck-RCaMP2, and pAAV.EF1a.OER-GCaMP6f are available upon request. | ||
| B-27 supplement (×50 stock) | Thermo Fisher Scientific | #17504044 | This can be replaced by B-27 plus supplement (Thermo Fisher Scientific; #A3582801) or MACS NeuroBrew-21 (Miltenyi Biotec, Bergisch Gladbach, Germany; #130-093-566). |
| B57BL/6 | Japan SLC, Inc. | ||
| Camera for microscopic image recording | The following cameras were available for use: cooled-CCD camera (e.g., Hamamatsu Photonics, OECA-ER), EM-CCD camera (e.g., Hamamatsu Photonics, ImagEM; Andor, iXon) or CMOS camera (e.g., Hamamatsu Photonics ORCA-Flash4.0) | ||
| Cell freezing container | Sarstedt K.K. | #95.64.253 | Alternative cell freezing container can be used. |
| Cell strainer | Falcon | #352350 | |
| CO2 incubator | Maintain at 37 °C, 5% CO2. | ||
| Cryogenic tube | Corning | #430661 | Alternative cryogenic tubes can be used. |
| Cryopreservation medium | Zenoaq | "CELLBANKER1" | |
| Culture medium (for HeLa cells) | Dulbecco's modified Eagle's medium (DMEM) supplemented with 10% heat-inactivated fetal bovine serum, and penicillin-streptomycin solution (final concentration: Penicillin 100 U/mL and Streptomycin 100 µg/mL) | ||
| Dissection medium | One milliliter of 1 M HEPES (final concentration 20 mM) to 49 mL DMEM | ||
| DMEM | Nacalai | #08456-65 | Alternative DMEM can be used. |
| DMEM | Nacalai | #08456-65 | Low glucose |
| DNA transfection reagent | Sigma-Aldrich | #6366244001 | "X-tremegene HP DNA transfection reagent" Alternative transfection reagents can be used. |
| Glass jar with a lid | 500 mL jar (for mouse) or 1,500 mL jar (for rat) to anesthetize the animal | ||
| HBSS | Thermo Fisher Scientific | #14170161 | HBSS free of calcium and magnesium |
| Heat inactivated bovine serum | Thermo Fisher Scientific | #10100147 | |
| HeLa cells | RIKEN BioResource Center | #RCB0007 | |
| Histamine | Sigma-Aldrich | #H7125 | |
| Image analysis software | Such as Metamorph (Molecular Devices), ImageJ (NIH), and TI Workbench14 (custom made) | ||
| Image splitting optics | Hamamatsu Photonics | #A12801-01 | W-view GEMINI |
| Image splitting optics dichroic mirror | Semrock | #FF560-FDi01-25×36 | For separation of green fluorescent protein/red fluorescent protein (GFP/RFP) signal |
| Image splitting optics emission filters | Semrock | #FF01-512/25-25, #FF01-630/92-25 | For emission of GFP/RFP signal, respectively |
| Imaging medium and buffer | Use optimal medium or buffer for the experiment. When medium is used, medium without phenol red is desirable to reduce background fluorescence. Add 20 mM HEPES to maintain pH outside of CO2 incubator. | ||
| Incubation saline | Add 1 mL of 1 M HEPES (20 mM) to 49 mL HBSS | ||
| Inverted fluorescence microscope | Such as IX73 (Olympus) or Eclipse TI (Nikon Instech) | ||
| Isoflurane | Pfizer | Used for anesthesia | |
| Maintenance medium (for 4 × 12 well dishes) | 48.5 mL Neurobasal-A medium supplemented with 1 mL B-27, 500 µL of L-glutamine stock and 25 µL Penicillin-Streptomycin solution. | ||
| Maintenance medium for frozen cortical cells (for 1 × 12 well dishes) | 12.2 mL Neurobasal plus medium supplemented with 250 µL B-27, 125 µL of L-glutamine stock and 6.2 µL Penicillin-Streptomycin solution. | ||
| MEM (Minimum Essential Medium) | Thermo Fisher Scientific | #11090-081 | |
| Microscope filter set for GCaMP6f imaging | Appropriate filter for GFP (excitation, 480 ± 10 nm; emission, 530 ± 20 nm) | ||
| Microscope filter set for RCaMP2 imaging | Appropriate filter for RFP (excitation, 535 ± 50 nm; emission, 590 nm long pass) | ||
| Microscope filter sets for double imaging of RCaMP2 and GCaMP6f | Semrock | #FF01-468/553-25, #FF493/574-FDi01-25×36, #FF01-512/630-25 | Dual excitation filter, Dual dichroic mirror, and emission filter for GFP/RFP imaging. |
| Microscope heating system | A heating system to maintain cells at 37 °C during the imaging. To avoid drift caused by thermal expansion, heating systems covering the entire microscope itself (e.g., Tokai Hit, Thermobox) are recommended. | ||
| Microscope light source for excitation | Mercury lamp (100 W), xenon lamp (75 W), Light-emitting diode (LED) illumination system (e.g., CoolLED Ltd., precisExcite; Thorlabs Inc., 4-Wavelength LED Source; Lumencor, SPECTRA X light engine). In case of mercury lump and xenon lamp, use ND filter to reduce the excitation intensity. | ||
| Microscope objective lens | Plan-Apochromat oil immersion objective with numerical aperture higher than 1.3 is highly recommended for the recording of spontaneous Ca2+ activity in neurons and astrocytes. | ||
| Neurobasal plus medium | Thermo Fisher Scientific | #A3582901 | |
| Neurobasal-A Medium | Thermo Fisher Scientific | #10888022 | Neurobasal plus medium (Thermo Fisher, A3582901) can be used instead of Neurobasal-A medium. |
| PBS(-): Phosphate-buffered saline free of Ca2+ and Mg2+ | Fujifilm Wako Pure Chemical Cooperation | #164-23551 | The absence of Ca2+ and Mg2+ is critical not to inhibit the trypsin activity. An alternative to PBS(-) can be used. |
| PC and image acquisition software | Such as Metamorph (Molecular Devices); Micromanager; TI Workbench14. | ||
| Penicillin-Streptomycin solution | Thermo Fisher Scientific | #15140122 | Penicillin 10,000 U/mL and Streptomycin 10,000 µg/mL |
| Plasmid for Lck-GCaMP6f, Lck-RCaMP2, and OER-RCaMP2 expression under cytomegalovirus promoter 7-9 | Available upon request | ||
| Plating medium (for 4 × 12 well dishes) | 48 mL MEM supplemented with 1 mL B-27 supplement, 500 µL L-glutamine stock (final concentration: 2 mM), 500 µL of sodium pyruvate stock (1 mM) and 25 µL Penicillin-Streptomycin solution (penicillin 5 U/mL, streptomycin 5 µg/mL). This concentration of Penicillin-Streptomycin, which is 1/20 of the concentration recommended by the manufacturer, is critical for neuronal survival. | ||
| Recording chamber | Elveflow | Ludin Chamber | This recording chamber is for 18 mm diameter round coverslips. |
| Reduced serum media | Thermo Fisher | #11058021 | Opti-MEM |
| Stereomicroscope | Used to dissect hippocampi. Olympus SZ60 or equivalent stereomicroscopes are available. | ||
| Surgical instruments | Standard dissecting scissors to cut the abdomen of a mouse or a rat, tweezers to pinch the uterus, delicate dissecting scissors to cut the uterus and the head of embryo, ring forceps to pinch the embryos, 13 cm curved Semken forceps (Fine Science Tools #11009-13) to extract brains, 3 forceps with fine tips (Dumont Inox #5) | ||
| Transfection reagent for neuron | Thermo Fisher Scientific | #L3000008 | "Lipofectamine 3000" reagent. It is composed of the the "supplement (P3000)" that should be mixed with plasmid DNA in the step 2.2.3, and the "transfection reagent (lipofectamine 3000)" used in the step 2.2.4. |
| Trypan blue (0.4%) | Thermo Fisher Scientific | #15250061 | |
| Wash medium for frozen cortical cells | 25 mL DMEM, supplemented with 250 µL heat-inactivated fetal bovine serum + 12.5 µL Penicillin Streptomycin. | ||
| Wistar rats | Japan SLC, Inc | Pregnant rats (E18) |
Riferimenti
- Clapham, D. E. Calcium signaling. Cell. 131 (6), 1047-1058 (2007).
- Bagur, R., Hajnoczky, G. Intracellular Ca(2+) Sensing: Its Role in Calcium Homeostasis and Signaling. Molecular Cell. 66 (6), 780-788 (2017).
- Bannai, H., et al. Bidirectional Control of Synaptic GABAAR Clustering by Glutamate and Calcium. Cell Reports. 13 (12), 2768-2780 (2015).
- Tojima, T., Hines, J. H., Henley, J. R., Kamiguchi, H. Second messengers and membrane trafficking direct and organize growth cone steering. Nature Reviews Neuroscience. 12 (4), 191-203 (2011).
- Shigetomi, E., Kracun, S., Sofroniew, M. V., Khakh, B. S. A genetically targeted optical sensor to monitor calcium signals in astrocyte processes. Nature Neuroscience. 13 (6), 759-766 (2010).
- Chen, T. W., et al. Ultrasensitive fluorescent proteins for imaging neuronal activity. Nature. 499 (7458), 295-300 (2013).
- Niwa, F., et al. Dissection of local Ca(2+) signals inside cytosol by ER-targeted Ca(2+) indicator. Biochemical and Biophysical Research Communications. 479 (1), 67-73 (2016).
- Vervliet, T., et al. Basal ryanodine receptor activity suppresses autophagic flux. Biochemical Pharmacology. 132, 133-142 (2017).
- Sakuragi, S., Niwa, F., Oda, Y., Mikoshiba, K., Bannai, H. Astroglial Ca2+ signaling is generated by the coordination of IP3R and store-operated Ca2+ channels. Biochemical and Biophysical Research Communications. 486 (4), 879-885 (2017).
- Inoue, M., et al. Rational design of a high-affinity, fast, red calcium indicator R-CaMP2. Nature Methods. 12 (1), 64-70 (2015).
- Quasthoff, K., et al. Freshly frozen E18 rat cortical cells can generate functional neural networks after standard cryopreservation and thawing procedures. Cytotechnology. 67 (3), 419-426 (2015).
- Boehringer, R., et al. Chronic Loss of CA2 Transmission Leads to Hippocampal Hyperexcitability. Neuron. 94 (3), 642-655 (2017).
- Arizono, M., et al. Receptor-selective diffusion barrier enhances sensitivity of astrocytic processes to metabotropic glutamate receptor stimulation. Science Signaling. 5 (218), ra27 (2012).
- Kanemaru, K., et al. In vivo visualization of subtle, transient, and local activity of astrocytes using an ultrasensitive Ca(2+) indicator. Cell Reports. 8 (1), 311-318 (2014).
- Inoue, T. TI Workbench, an integrated software package for electrophysiology and imaging. Microscopy (Oxford, UK). 67 (3), 129-143 (2018).
- Miyamoto, A., Bannai, H., Michikawa, T., Mikoshiba, K. Optimal microscopic systems for long-term imaging of intracellular calcium using a ratiometric genetically-encoded calcium indicator. Biochemical and Biophysical Research Communications. 434 (2), 252-257 (2013).
- Srinivasan, R., et al. New Transgenic Mouse Lines for Selectively Targeting Astrocytes and Studying Calcium Signals in Astrocyte Processes In Situ and In Vivo. Neuron. 92 (6), 1181-1195 (2016).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneEsplora altri articoli
This article has been published
Video Coming Soon