Method Article
Il nanopatterndifid antistrato DNA Mediato delle strutture inorganiche per applicazioni di rilevamento
In questo articolo
Riepilogo
Qui, descriviamo un protocollo per creare nanostrutture inorganiche discrete e accurate sui substrati utilizzando forme di origami di DNA come modelli guida. Il metodo è dimostrato dalla creazione di antenne plasmoniche a forma di papillon in oro su un substrato trasparente (sapphire).
Abstract
La nanotecnologia del DNA strutturale fornisce un percorso percorribile per la costruzione dal basso verso l'alto utilizzando il DNA come materiale da costruzione. La tecnica di nanofabbricazione del DNA più comune è chiamata origami del DNA e consente la sintesi ad alta velocità di strutture accurate e altamente versatili con precisione a livello di nanometri. Qui, è mostrato come le informazioni spaziali degli origami di DNA possano essere trasferite alle nanostrutture metalliche combinando gli origami di DNA bottom-up con gli approcci litografici top-down utilizzati convenzionalmente. Ciò consente la fabbricazione di miliardi di piccole nanostrutture in un solo passaggio su substrati selezionati. Il metodo è dimostrato utilizzando origami di DNA bowtie per creare strutture di antenne metalliche a forma di bowtie su nitride di silicio o substrati zaffiro. Il metodo si basa sulla crescita selettiva di uno strato di ossido di silicio sopra il substrato di deposizione origami, ottenendo così una maschera di patterning per seguire i passaggi litografici. Queste superfici dotate di nanostruttura possono essere ulteriormente utilizzate come sensori molecolari (ad esempio, spettroscopia Raman (SERS)) potenziata dalla superficie e in varie altre applicazioni ottiche a causa delle piccole dimensioni delle caratteristiche (sotto-10 nm). La tecnica può essere estesa ad altri materiali attraverso modifiche metodologiche; pertanto, le superfici otticamente attive risultanti possono trovare un uso nello sviluppo di metamateriali e metasuperfici.
Introduzione
La nanotecnologia del DNA strutturale si è rapidamente evoluta nel corso del recente decennio1,2, e lo sviluppo più influente nel campo è stato probabilmente l'invenzione di DNA origami3,4. La tecnica degli origami del DNA permette la fabbricazione di praticamente qualsiasi nanoforma con accurate caratteristiche strutturali3,4. Questo potente metodo può essere utilizzato nella disposizione spaziale (sub)nanometro-precisa e l'ancoraggio di altri nano-oggetti, come nanotubi di carbonio5, nanoparticelle metalliche6,7,8, 9, enzimi/proteine10,11,12,13 e materiali terapeutici14,15,16,17 . È importante sottolineare che queste strutture non sono solo statiche, ma possono anche essere programmate per agire in modo dinamico18,19. Le innumerevoli applicazioni degli origami di DNA vanno dalla somministrazione di farmaci20,21,22 all'elettronica molecolare/plasmonica5,23,24, 25 e dalla scienza dei materiali26,27 alle nuove tecniche di imaging e calibrazione28.
Oltre alle applicazioni di cui sopra, l'estrema risoluzione spaziale delle forme di origami del DNA potrebbe essere sfruttata in nanopatterning e litografia su nanoscala29,30. Questo protocollo descrive un metodo di litografia per la creazione di nanostrutture inorganiche discrete e accurate su substrati utilizzando modelli di origami di DNA. Questi modelli possono essere prodotti in modo efficiente in varie forme e in grandi quantità31, e depositati senza sforzo su substrati scelti su grandi scale32. Queste proprietà consentono una fabbricazione altamente parallela di miliardi di nanostrutture in un unico passaggio rispetto alla litografia a fascio di elettroni comunemente usata, ma piuttosto lenta, o ad altre tecniche di nanofabbricazione basate sulla scansione.
Qui, il processo di fabbricazione è dimostrato creando strutture a forma di bowtie in oro su nitrati di silicio e substrati di zaffiro; in altre parole, le informazioni spaziali degli origami del DNA vengono trasferite in nanostrutture interamente metalliche. Come discusso qui, la tecnica non è limitata alla struttura di origami del DNA bowtie selezionata poiché il metodo consente l'uso di praticamente qualsiasi forma di origami di DNA. Inoltre, con modifiche metodiche, la tecnica può essere estesa a diversi metalli e substrati spianando la strada verso la fabbricazione di metasuperfici33.
Le superfici modellate con la fabbricazione mediata dall'origami del DNA possono fungere da sensori versatili; ad esempio, possono essere utilizzati in spettroscopia Raman migliorata dalla superficie (SERS). Come risultato delle piccole dimensioni delle singole nanoforme, le superfici create possono trovare usi in applicazioni ottiche e plasmoniche alla gamma di lunghezza d'onda visibile.
Protocollo
1. Progettazione di origami di DNA
NOTA: In questo protocollo, un processo di nanopatterning viene descritto utilizzando una struttura di origami del DNA bowtie bidimensionale(Figura 1)34. Per progettare una nuova forma di origami di DNA, seguire le linee guida riportate di seguito:
- Progettare la forma desiderata e le sequenze di filamento di pinzatura necessarie dell'origami del DNA utilizzando caDNAno35. Per produrre un origami piatto a strato singolo, utilizzare l'opzione del reticolo quadrato di caDNAno e regolare manualmente la spaziatura crossover saltando alcune basi nel progetto (vedere Figura 1 e il file caDNAno supplementare) per rimuovere la torsione strutturale risultante dall'imballaggio a reticolo quadrato36,37.
- Estendere le estremità di ogni elica del DNA con filamenti contenenti sporgenza poli-T (8 nt); ciò impedirà la multimerizzazione degli oggetti tramite interazioni di stack di base smussato(Figura 1 e file caDNAno supplementare).
- Eseguire un'analisi computazionale del progetto. CanDo38,39 può essere utilizzato per prevedere la forma tridimensionale (3D) e la rigidità strutturale dell'origami del DNA. CanDo è anche uno strumento utile per scorrere il numero di salti di base necessari per la correzione della torsione e per regolare il disegno di conseguenza.
- In caDNAno, scegliere la lunghezza dell'impalcatura preferita e generare i fili di pinzatura necessari per piegare la struttura. Per la struttura a papillon, vengono utilizzati lo scaffold M13mp18 lungo 7249 nt e 205 fili di pinzatura unici (vedere il file caDNAno supplementare).
NOTA: Ci sono anche altri strumenti computazionali disponibili per la progettazione di strutture di origami di DNA40,41,42,43. A seconda dello strumento/software scelto, possono essere utilizzati anche altri strumenti di simulazione43,44.
2. Assemblaggio di origami di DNA
- Fare il brodo di fili di pinzatura mescolando uguali quantità di tutte le oligonucleotidi necessarie per la struttura bowtie (in totale 205 graffette)34. Gli oligonucleotidi dovrebbero avere tutti la stessa concentrazione iniziale (ad es., 100 M in acqua libera da RNase).
- Preparare la miscela di reazione di piegatura dell'origami del DNA in quantità di 100 ,l in un tubo PCR da 0,2 mL, mescolando 20 -L di filo di scaffold M13mp18 (tipo p7249, a 100 nM), 40 l of staple stock solution, dove ogni filamento è a 500 nM (che produce all'impalcatura) e 40 luna di 2,5 volte buffer di piegatura (FOB). FOB contiene Tris - acido acetico - etilenediaminetraicaicaa acida (EDTA) buffer (TAE) integrato con MgCl2. Vedere la tabella 1 per le concentrazioni dei componenti FOB.
- Anneal la miscela di reazione in un termociclista da 90 a 27 gradi centigradi. Utilizzare la rampa di piegatura termica presentata nella Tabella 2.
3. Purificazione degli origami di DNA
NOTA: la quantità in eccesso di filamenti di pinzatura può essere rimossa dalla soluzione di origami del DNA utilizzando un metodo di purificazione del polilulo di etilene (PEG) non distruttivo. Il protocollo è adattato da Stahl et al.45.
- Diluire 200 - L di strutture di origami di DNA assemblato con 600 L di 1x FOB (vedi Tabella 1) per ottenere un volume iniziale di 800 l.
- Mescolare la soluzione diluita di origami di DNA 1:1 con 800 - L di buffer di precipitazione PEG (15% PEG 8000 (w/v), 1x TAE, 505 mM NaCl) e mescolare accuratamente pipetting avanti e indietro.
- Centrifugare la miscela per 30 min a 14.000 x g e temperatura ambiente.
- Rimuovere con cautela il supernatante utilizzando una pipetta.
- Aggiungere 200 - L of 1x FOB e mescolare delicatamente pipettando. Una diversa quantità di 1x FOB può anche essere aggiunto per ottenere la concentrazione desiderata di origami di DNA.
- Per riscioglierare le strutture degli origami di DNA (piccolo pellet trasparente nella parte inferiore del tubo), incubare le strutture di origami di DNA purificato PEG durante la notte a temperatura ambiente.
- Stimare la concentrazione di origami di DNA dopo la purificazione del PEG misurando l'assorbimento ad una lunghezza d'onda di 260 nm utilizzando uno spettrofotometro UV/Vis. Utilizzare la legge Di birra-Lambert e un coefficiente di estinzione di 1,1 x 108 M-1 cm-1 per il calcolo6. La tipica concentrazione di origami di DNA dopo la purificazione del PEG è di 15-20 nM.
- Conservare le strutture di origami di DNA purificato del PEG a 4 gradi centigradi. Le strutture di origami del DNA sono di solito stabili per mesi in modo da grandi quantità di magazzino possono essere preparati per un uso successivo.
NOTA: La quantità in eccesso di filamenti di pinzatura può anche essere rimossa utilizzando altre tecniche di purificazione46, come la filtrazione di spin47, la centrifugazione zonale del tasso48 e l'estrazione gel agarose49. Le strutture di origami di DNA sono stabili in una varietà di soluzioni tampone50, e se necessario, il mezzo di stoccaggio può essere cambiato dopo la purificazione PEG attraverso la filtrazione spin51.
4. Elettroforesi gel di agarose
NOTA: La qualità del piegamento e la rimozione dei fili di pinzatura in eccesso possono essere verificati utilizzando l'elettroforesi gel di agarose.
- Preparare un gel di agarose (2% (w/v) aggiungendo 1 g di agarose e 45 mL di 1x TAE a una fiaschetta erinemeyera. Riscaldare la miscela in un forno a microonde fino a quando l'agarose è completamente dissolta, e viene prodotta una soluzione chiara.
- Raffreddare la soluzione sotto l'acqua corrente fino a quando il pallone è comodo da toccare (50-60 gradi centigradi).
- Aggiungere alla soluzione 5 mL di 110 mM MgCl2 e 40 -L di bromuro di etidio (0,58 mg mL-1) e agitare delicatamente la miscela.
AVVISO: Il bromuro di etidio è un potenziale cancerogeno e deve essere maneggiato con cura. - Impostare il vassoio di colata gel e versare l'agarose liquido nel vassoio di colata. Lasciare che il gel si solidifici a temperatura ambiente per almeno 30 min.
- Rimuovere il gel dal vassoio di colata e metterlo in una camera di elettroforesi gel. Riempire la camera con buffer in esecuzione (1x TAE con 11 mM MgCl2).
- Aggiungere 1 x luna di 6x gel di carico per 5 - L di soluzione campione e mescolare accuratamente. Caricare i campioni con la cura pipettando la quantità desiderata delle soluzioni campione in tasche di gel separate.
- Eseguire il gel di agarose ad una tensione costante di 95 V per 45 min.
- Visualizzare il gel sotto la luce ultravioletta utilizzando un sistema di imaging gel (Figura 2A).
5. Preparazione del substrato (Figura 3A)
NOTA: i seguenti passaggi vengono eseguiti tutti all'interno di una camera pulita, ad eccezione della crescita di SiO2 (passaggio 9). Le fasi di pulizia possono anche essere sostituite con una pulizia standard basata su piranha-soluzione se questo processo non è sufficiente per rimuovere tutti i residui dal substrato.
- Tagliare chip da 7 mm x 7 mm da un wafer da utilizzare come substrato. Per SiN, utilizzare una sega in silicio, una penna a diamante o un attrezzo simile. Lo zaffiro a dicing (Al2O3) richiederà uno strumento specializzato o una lama di sega. La dimensione del chip non deve essere esatta.
- Pulire le patatine.
- Immergere i trucioli tagliati a dadini in un bicchiere con acetone caldo (acetone riscaldato a 52 gradi centigradi) e tenerli riscaldati per almeno 15 min. A seconda della pulizia iniziale del substrato, potrebbe essere necessario un tempo più lungo.
- Mentre sono ancora nel bagno di acetone caldo, strofinare delicatamente i trucioli con un tampone di cotone per rimuovere meccanicamente eventuali pellicole residui.
- Utilizzando una pinzetta, sollevare i trucioli dall'acetone caldo e utilizzare una bottiglia di lavaggio per sciacquarli con acetone a temperatura ambiente.
- Immergere le patatine in un bicchiere con isopropanol e ultrasonica per 2 min.
- Sollevare i trucioli fuori dall'isopropanolo con una pinzetta e asciugarli immediatamente e accuratamente utilizzando un flusso di azoto. Toccare e tenere premuti solo i lati e i bordi dei trucioli, poiché le aree coperte dalle pinzette non si asciugano correttamente, lasciando potenzialmente residui e altre contaminazioni sulle aree di contatto. Per ottenere risultati ottimali, utilizzare il flusso più elevato possibile e tenere le superfici del chip parallele alla direzione del flusso.
- Conservare i trucioli in un contenitore coperto all'interno della camera di pulizia per un uso successivo.
6. Deposizione di vapore chimico potenziato al plasma (PECVD) dello strato di silicio amorfo (a-Si) (Figura 3B)
- Posizionare i trucioli nell'apparecchiatura PECVD.
- Impostare i parametri di deposizione in modo che crescano circa 50 nm di silicio amorfo (a-Si). Le impostazioni esatte variano in base al modello e alla calibrazione dell'apparecchiatura. Vedere la tabella 3 per i parametri utilizzati qui. Eseguire il programma di deposizione a-si per far crescere il livello.
- Dopo la lavorazione, conservare i chip in un contenitore coperto in condizioni standard di camera pulita.
7. Trattamento al plasma di ossigeno dello strato a-Si (Figura 3B)
NOTA: Questo passaggio renderà la superficie del substrato leggermente carica negativamente e idrofila, in modo che le strutture di origami del DNA possano essere successivamente efficacemente assorbite in superficie con l'aiuto di ioni di magnesio aggiuntivi.
- Collocare i trucioli nell'apparecchiatura RIE (Ion Etching) reattiva.
- Impostare i parametri di incisione per generare plasma di ossigeno. Anche in questo caso, le impostazioni esatte variano in base al modello e alla calibrazione dell'apparecchiatura. Vedere la tabella 3 per i parametri utilizzati qui. Eseguire il programma di trattamento del plasma di ossigeno.
- Continuare immediatamente con il passo successivo, poiché gli effetti del trattamento si deteriorano rapidamente. Tipicamente, i substrati devono essere utilizzati entro i prossimi 30 min dopo il trattamento al plasma.
8. Deposizione di origami di DNA (Figura 3C)
- Preparare una miscela di origami di DNA per la deposizione mescolando 5 origami di origami di DNA piegato/purificato (20 nM) con 4 lof1 FOB e 1 l di 1 M MgCl2. La soluzione risultante contiene 10 nM di origami di DNA e circa 100 mM di Mg2.
- Depositare 10 l della miscela di origami di DNA su un chip con trattamento al plasma di ossigeno e incubare coperto per 5 min a temperatura ambiente. La copertura previene l'essiccazione involontaria e aiuta a rimuovere in seguito le strutture di sale e origami estranei.
- Dopo l'incubazione, lavare la superficie mediante il primo tubo di acqua distillata (ad esempio MilliQ) sul chip. Sciacquare l'acqua avanti e indietro un paio di volte con la pipetta, evitando di toccare il centro del chip. Rimuovere la maggior parte dell'acqua dalla superficie con la pipetta. Questo fa sì che solo l'origami correttamente adsorbito di rimanere sulla superficie.
- Ripetere questo ciclo di lavaggio (passaggi 8.3) da 3 a 4 volte.
- Dopo il lavaggio, asciugare immediatamente il campione con un flusso di azoto. Eseguire questa operazione allo stesso modo dell'essiccazione nella preparazione del substrato (passaggio 5). È importante asciugare il campione il più accuratamente possibile.
NOTA: La densità delle strutture depositate e quindi la densità delle nanostrutture metalliche possono essere modificate regolando la concentrazione di origami di DNA e Mg2 nella soluzione di deposizione. Una maggiore concentrazione di Mg2 migliora l'adesione all'origami del DNA e quindi aumenta la densità, ma alla fine causerà anche l'agglomerazione delle strutture di origami del DNA. Pertanto, la concentrazione di origami di DNA dovrebbe essere regolata prima.
9. Crescita della maschera SiO2 (Figura 3D)
NOTA: questo passaggio può essere eseguito al di fuori della camera di pulizia. La seguente versione produrrà un modello di tono negativo, ma è possibile modificare il processo per produrre un modello di tono positivo invece. Il processo di crescita di SiO2 è adattato da Surwade et al.52, sviluppato ulteriormente dagli autori53, e infine ottimizzato per questo protocollo.
- Prendere un desiccatore sigillabile (1,5 L), un piatto Petri che si inserisce all'interno del desiccatore (opzionale) e una piastra perforata che può funzionare come una piattaforma all'interno del desiccatore.
- Prendere 100 g di gel di silice e mescolare con 30 g di acqua distillata nel piatto Petri o direttamente nel desiccatore. Fare questo passo preferibilmente almeno 24 h in anticipo per consentire al gel di silice di stabilizzarsi.
NOTA: Questo viene utilizzato per controllare l'umidità all'interno del desiccatore e quindi anche il tasso di crescita e la morfologia del film SiO2. Una maggiore umidità si traduce in un tasso più elevato e in una struttura grossolana. In alternativa, il gel di silice può essere curato in una camera di prova climatica. - Mettere il gel di silice nel desiccatore e separarlo con la piastra perforata.
- Posizionare i chip con origami di DNA adsorbito e una fiala aperta di (fresco) 10 mL di ortosilicate tetraetile (TEOS) e un'altra fiala di 10 mL del 25% di idrossido di ammonio (NH4OH) nel desiccatore, sulla piattaforma perforata. Impostare le fiale vicino e su lati opposti dei campioni. Preferibilmente utilizzare un tappo di fiaschetta o un piedistallo piatto simile per sollevare leggermente i trucioli dalla piattaforma.
AVVISO: Sia NH4OH che TEOS sono dannosi in caso di contatto con la pelle e i loro vapori possono causare irritazione sia agli occhi che agli organi respiratori. Utilizzare in una zona ben ventilata e indossare guanti protettivi, protezione degli occhi e indumenti protettivi. - Sigillare la camera e incubare per 20 ore a temperatura ambiente. Questo farà crescere una pellicola SiO2 sulle aree in cui non si trovano le strutture degli origami del DNA, creando una maschera modellata da 10-20 nm con fori a forma di origami di DNA (Figura 4).
- Rimuovere i campioni dalla camera dopo l'incubazione. Conservare in un contenitore coperto. L'elaborazione può essere sospesa qui. Smaltire il TEOS usato e NH4OH. Il lotto di gel di silice può essere utilizzato 2-3 volte se viene tenuto sigillato all'interno del desiccatore tra gli usi e utilizzato entro 2-3 settimane.
10. Incisione reattiva (RIE) di SiO2 e a-Si(Figura 3E)
- Collocare i trucioli nell'apparecchiatura RIE (Ion Etching) reattiva.
- Impostare i parametri di incisione solo per etch 2-5 nm di SiO2 al fine di rivelare il livello a-Si sotto i fori nella maschera SiO2. Le impostazioni esatte devono essere determinate sperimentalmente per le singole apparecchiature. I parametri qui utilizzati sono presentati nella tabella 3. Eseguire il programma di incisione al plasma anisotropico SiO2.
- Impostare i parametri di incisione per eseguire il foro attraverso il livello a-Si di 50 nm. I parametri qui utilizzati sono nuovamente presentati nella tabella 3. Eseguire il programma di incisione al plasma isotropico a-Si.
- Rimuovere i campioni dalle apparecchiature RIE e conservarli. L'elaborazione può essere sospesa di nuovo qui.
11. Deposizione di vapore fisico (PVD) di metalli (Figura 3F)
- Caricare i trucioli nella camera di evaporazione dello strumento PVD.
- Scegliere un metallo di destinazione. In primo luogo, scegliere un metallo adesivo. In questo caso, vengono utilizzati 2 nm di cromo (Cr).
- Impostare il programma di controllo dello spessore per il materiale e lo spessore di destinazione. Il metodo di controllo dipende dallo strumento. In questo caso, viene utilizzata una microbilancia di cristallo di quarzo (QCM). Lo spessore misurato viene regolato in base alla densità del materiale di destinazione e al fattore z e deve essere corretto da un fattore di utensile determinato sperimentalmente specifico per il dispositivo e per ogni materiale di destinazione.
- Avviare il fascio di elettroni, allineare il fascio al bersaglio e aumentare la corrente del fascio fino a raggiungere una velocità di deposizione di 0,05 nm/s. Evaporare fino a raggiungere uno spessore finale di 2 nm.
- Scegliere un secondo metallo bersaglio (ad es. oro) senza sfiatare la camera o interrompere il processo. Interruzioni o sfiato permetteranno al metallo adesivo di iniziare a ossidare e diminuire la sua usabilità come adesivo.
- Ripetere i passaggi da 11.3 a 11.4. Evaporare fino a raggiungere 20 nm. Questo creerà una struttura metallica a forma di origami di DNA attraverso i fori della maschera SiO2 con un'altezza totale di 22 nm.
- Sfogare la camera e rimuovere i campioni.
- L'elaborazione può essere sospesa qui se i campioni sono immagazzinati coperti.
12. Sollevamento con acido idrofluorico (HF) (Figura 3G)
- Versare il 50% di soluzione etchant basata su HF in un contenitore di plastica adatto. Nessun HCl deve essere utilizzato per la miscela, dal momento che HCl inciderebbe il Cr nel campione.
AVVISO: L'HF è estremamente corrosivo, causa gravi irritazioni e ustioni e può essere fatale a contatto con la pelle o se inalato. Utilizzare HF solo in un cofano di fumi dedicato o panca bagnata ventilata con un grembiule protettivo, guanti resistenti alle sostanze chimiche e visiera per il viso o una protezione chimica comunque completa. - Immergere i campioni nell'etchant basato su HF e mescolare delicatamente con una pinzetta di plastica.
- Attendere che lo strato SiO2 si incida completamente e che lo strato metallico si stacchi. Il tempo varia notevolmente a seconda della densità dei fori della maschera. Un numero maggiore di fori si tradurrà in un'incisione più veloce. Se lo strato metallico è difficile da staccare, è possibile utilizzare una breve ultrasonica per 5 a 10 s.
- Una volta staccata la pellicola metallica, sciacquare i campioni con acqua a doppia distillazione e isopropanolo.
- Dopo il risciacquo, asciugare i campioni con un flusso di azoto allo stesso modo come indicato per la preparazione del substrato (passaggio 5). Evitare il contatto delle pinzette con il centro chip, in quanto ciò potrebbe distruggere le nanostrutture formate.
NOTA: i campioni possono essere memorizzati ed elaborazione sospesa qui.
13. RIE di restante a-Si(Figura 3H)
- Collocare i trucioli nell'apparecchiatura RIE (Ion Etching) reattiva.
- Impostare i parametri di incisione per la rimozione completa di tutti i 50 nm di a-Si. I parametri possono essere gli stessi del passaggio 10, ma è possibile utilizzare un tempo di incisione leggermente più lungo (40 s) per garantire la rimozione di tutti gli a-Si. Vedere la tabella 3 per i parametri utilizzati qui. Eseguire il programma di incisione al plasma isotropico a-Si per rimuovere gli a-Si rimanenti.
- Rimuovere i campioni dalle apparecchiature RIE e conservarli. Questo concluderà l'elaborazione del campione.
14. Microscopia a forza atomica (AFM)
NOTA: la microscopia a forza atomica e la microscopia elettronica a scansione possono essere utilizzate per monitorare il successo della crescita e del patterning della pellicola, nonché per immagini di strutture di origami di DNA piegate (Figura 2B,C). Il passaggio di preparazione di esempio seguente può essere ignorato se vengono imageti i campioni elaborati dai passaggi 5-13.The following sample preparation step can be skipped if processed samples from Steps 5-13 are imaged.
- Preparazione del campione per AFM
- Per immaginare l'origami del DNA piegato, prendi un chip di substrato di mica.
- Fissare il chip mica a uno scivolo del microscopio di vetro utilizzando un adesivo.
- Preparare 10 l di origami di DNA diluindo lo stock di origami di DNA da 20 nM 50 volte in 1 foB ad una concentrazione di circa 0,4 nM. La diluizione viene effettuata al fine di evitare il sovraffollamento del substrato.
- Sbucciare lo strato superiore del foglio di mica con nastro adesivo debole per ottenere una superficie appena screpolata e carica.
- Depositare la soluzione diluita per origami di DNA sulla mica appena scissa e incubare il campione coperto per 1 min a temperatura ambiente.
- Dopo l'incubazione, lavare la superficie 3-4 volte con 100 l di acqua distillata utilizzando una pipetta. Questo fa sì che solo l'origami correttamente adsorbito di rimanere sulla superficie.
- Depositare 100 l di acqua distillata sulla superficie della mica.
- Inclinare e toccare bruscamente il vetrino del microscopio sul tavolo per staccare la maggior parte dell'acqua.
- Ripetere questo ciclo di lavaggio 3-4 volte.
- Asciugare accuratamente il campione con un flusso di azoto subito dopo il lavaggio. Il campione è quindi pronto per l'imaging AFM.
- Inserire i campioni di origami di DNA o i trucioli trasformati in un AFM ed eseguire scansioni. Una dimensione di scansione di 1-10 m è adatta per risolvere correttamente le strutture.
15. Microscopia elettronica a scansione (SEM)
- Collocare i campioni in un SEM. I chip elaborati possono essere utilizzati così come sono (non è necessaria un'ulteriore preparazione del campione).
- Scegliere la tensione di accelerazione. Utilizzare basse tensioni (5-10 kV) per ridurre gli effetti di ricarica poiché il substrato campione (Al2O3 o SiN) è un isolante.
- Eseguire la scansione di tutte le aree di interesse. Ridurre al minimo i tempi di scansione per ridurre la ricarica ed evitare la deposizione di contaminazione.
Risultati
Una figura schematica della progettazione di origami di DNA bowtie e dei suoi dettagli strutturali sono mostrati nella Figura 1. L'elettroforesi gel di Agarose e l'AFM sono utilizzati per analizzare il ripiegamento degli origami del DNA e la qualità della purificazione del PEG (Figura 2). Il flusso di processo dei passaggi di nanolitografia viene visualizzato nella Figura 3. Le immagini AFM rappresentative dopo la crescita della maschera SiO2 sono mostrate nella Figura 4 (questo passaggio è illustrato nella Figura 3D),mentre le immagini SEM delle nanostrutture metalliche finali sono visibili nella Figura 5 (questo passaggio è illustrato in Figura 3H). La Figura 6 illustra la funzionalità ottica delle nanostrutture metalliche basate sull'origami del DNA del bowtie.
| Concentrazioni dei componenti del buffer di piegatura (FOB) [mM] | |||||
| Tris | Acido acetico | Edta | Cloruro di magnesio | Ph | |
| 2,5 x FOB | 100 | 47.5 | 2.5 | 31.25 | 8,3 USD |
| 1x FOB | 40 | 19 | 1 | 12.5 | 8,3 USD |
Tabella 1: Composizione del buffer di piegatura (FOB).
| Intervallo di temperatura [oC] | Velocità di raffreddamento |
| 90-70 | -0,2 gradi centigradi / 8 s |
| 70-60 | -0,1 Gradi centigradi / 8 s |
| 60-27 | -0,1 S / 2 s |
| 12 | Tenere premuto fino a quando non si ferma |
Tabella 2: rampa termica per il ripiegamento degli origami bowtie. Dopo l'annealing, l'origami verrà conservato a 12 oC fino a quando il programma non viene arrestato manualmente.
| Parametri PECVD e RIE | ||||||
| gas | Flusso di gas [sccm] | Pressione della camera [mTorr] | Potenza RF [W] | Temperatura [oC] | Durata [s] | |
| PECVD di a-Si | 5% SiH4 in N2 | 500 | 1000 | 15 | 250 | 90 |
| O2 trattamento al plasma | O2 | 50 | 40 | 200 | 30 | 1200 |
| RIE di SiO2 | CHF3 | 25 | ||||
| Ar | 25 | 30 | 100 | 25 | 10-22 | |
| RIE di a-Si | O2 | 8 | ||||
| SF6 | 100 | 90 | 50 | 30 | 35 | |
| RIE di rimanere a-Si | O2 | 8 | ||||
| SF6 | 100 | 90 | 50 | 30 | 35-40 |
Tabella 3: Parametri di processo per la deposizione di vapore chimico potenziato al plasma (PECVD) e l'incisione reattiva (RIE). I parametri di processo per questi dispositivi sono specifici per i singoli strumenti e potrebbe essere necessario adattarli quando utilizzati.
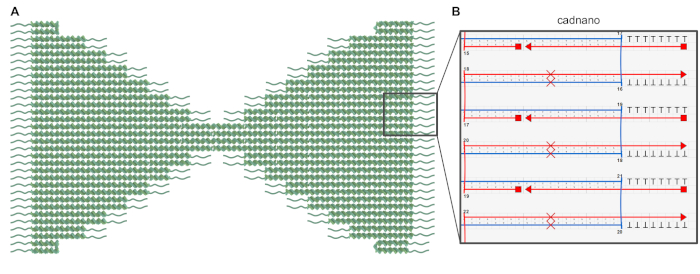
Figura 1: Progettazione dell'origami del DNA bowtie. (A) Rappresentazione schematica del disegno degli origami bowtie in cui la struttura del nucleo è mostrata come doppie eliche e le sporgenpite polit sono rappresentate come linee ondulate. (B) Screenshot di una parte del design degli origami di papillon nel software caDNAno. Le croci rosse indicano il salto della coppia di basi per la correzione della torsione e vengono aggiunti i montanti T8per evitare il smussato di base. Fare clic qui per visualizzare una versione più grande di questa figura.

Figura 2: Caratterizzazione della struttura degli origami del DNA delbowtie. (A) Agarose gel elettroforesi della struttura bowtie prima e dopo la purificazione di poli(etilene glicole) (PEG). Lo scaffold lungo 7249 nucleotidi viene utilizzato come riferimento. (B) Immagine di microscopia a forza atomica (AFM) delle strutture del papillon prima della purificazione. (C) Immagine AFM delle strutture del papillon dopo la purificazione PEG. Fare clic qui per visualizzare una versione più grande di questa figura.
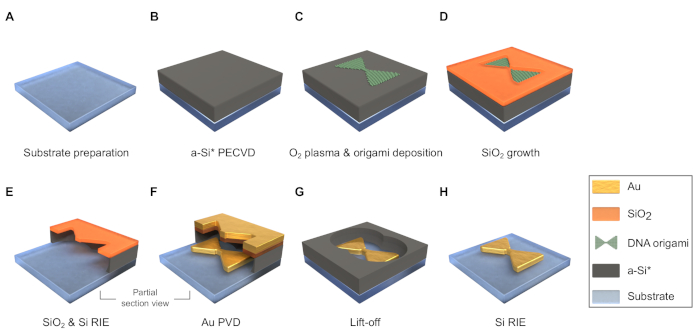
Figura 3: Schema del flusso del processo di fabbricazione (le dimensioni non sono in scala). (A) Dadi e pulire il substrato. (B) Depositare uno strato a-Si mediante deposizione di vapore chimico potenziata al plasma (PECVD). È possibile impiegare uno strato sacrificale aggiuntivo sotto l'a-Si per consentire il decollo con etchant diverso da HF. (C) Trattare la superficie del campione con O2 plasma e depositare origami di DNA su di esso. (D) Far crescere la maschera SiO2 in desiccatore. (E) Incidere uno strato sottile di SiO2 e attraverso l'A-Si sotto di esso da incisione iosgione reattiva (RIE). (F) Depositare il metallo attraverso la maschera per deposizione fisica del vapore (PVD). (G) Il sollevamento con HF. (H) rimuove l'a-Si rimanente da RIE. Fare clic qui per visualizzare una versione più grande di questa figura.

Figura 4: Immagini AFM rappresentative della pellicola SiO2 con il modello a forma di origami di DNA. (A) l'area di scansione di 10 m x 10 m dimostra l'elevato rendimento della formazione del modello. (B) Una scansione più vicina di 3 m x 3 m mostra i singoli modelli precisi nella pellicola SiO2. Fare clic qui per visualizzare una versione più grande di questa figura.

Figura 5: Immagini rappresentative dimicroscopia elettronica a scansione(SEM) di nanostrutture metalliche basate su origami di DNA strutturalmente diversi. (A) Origami di DNA a forma di croce, cioè la cosiddetta piastrella Seeman origami54. (B) Antenne Bowtie. (C) Strutture chiral doppia L (CDL). I rientri mostrano singole strutture con dimensioni di scatola di 150 nm x 150 nm. La resa di fabbricazione di strutture esatte è fino al 76% per l'origami bowtie e al 50% per le altre strutture qui esposte34. Questa cifra è stata adattata e modificata da Shen et al.34. La figura è riprodotta con il permesso degli autori e pubblicata da The American Association for the Advancement of Science, 2018. Fare clic qui per visualizzare una versione più grande di questa figura.
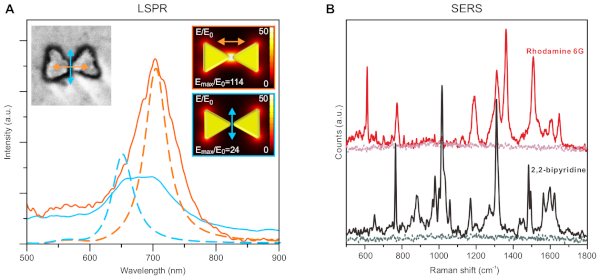
Figura 6: Proprietà ottiche/funzionali rappresentative delle nanostrutture risultanti. (A) Misurazioni localizzate della risonanza dei plasmoni di superficie (LSPR) di una singola struttura a pezze d'oro con polarizzazione diversa (colore codificato come arancione e blu). Le linee solide vengono misurate spettrale e le linee tratteggiate sono risultati di simulazione. Gli insiemi mostrano l'immagine SEM della particella misurata (a sinistra) e il modello utilizzato per la simulazione (a destra). (B) Superficie migliorata spettroscopia Raman (SERS) di rhodamine 6G e 2,2-bipyridine misurata su una superficie coperta con nanostrutture bowtie. La linea di base di ogni campione mostra il livello del segnale quando le nanostrutture erano assenti. Questa cifra è stata adattata e modificata da Shen et al.34. La figura è riprodotta con il permesso degli autori e pubblicata da The American Association for the Advancement of Science, 2018. Fare clic qui per visualizzare una versione più grande di questa figura.

Supplemental File 1: File CaDNAno Fare clic qui per scaricare questo file.

File supplementare 2: sequenza m13mp18 Fare clic qui per scaricare questo file.
File supplementare 3: Sequenza di filamento di pinzatura di pinzatura di pinzatura Fare clic qui per scaricare questo file.
Discussione
Il protocollo offre grande libertà e precisione sotto forma di nanostrutture prodotte. Modificando il design degli origami del DNA, la forma delle nanostrutture metalliche può essere controllata. La forma finale ed esatta delle strutture metalliche è inoltre determinata dalla fase di crescita della maschera (Passaggio 9) e in misura minore dall'incisione della maschera (Passaggio 10) se non dovesse essere anisotropica. Se il tempo di crescita della maschera è sufficientemente prolungato, i fori nella maschera inizieranno a chiudersi. Questo può essere utilizzato per omettere le caratteristiche più sottili di alcune strutture e controllare le dimensioni dello spazio, come dimostrato in Shen et al.34 con triangoli separati dell'origami bowtie (Figure 5B). Al contrario, le forme più sottili possono essere meglio conservate accorciando il tempo di crescita dell'ossido. Ciò significa che è possibile ottimizzare le proprietà ottiche visualizzate in Figura 6, non solo cambiando il design dell'origami utilizzato, ma anche ottimizzando la crescita del film SiO2.
Se lo spessore della maschera viene modificato in modo significativo, tale modifica deve essere riflessa anche nel passo SiO2 RIE. Solo uno strato molto sottile di SiO2 deve essere inciso (2-5 nm) per penetrare a malapena attraverso i fori della maschera. Questa è la parte più sensibile e cruciale dell'intero processo. Poiché il tempo di incisione è estremamente breve, solo 10-20 s, le impostazioni esatte devono essere determinate sperimentalmente quando si tenta per la prima volta con nuove attrezzature. Questo vale anche per il passo 10.4 come alcuni SiO2 è anche inciso durante l'incisione a-Si. L'estensione del SiO2 inciso è determinato dalla selettività dei parametri di incisione a-Si utilizzati, delle attrezzature e persino delle singole calibrazioni delle apparecchiature. Bisogna fare attenzione a non incidere via l'intero strato SiO2 durante questi due processi.
Un altro passo sensibile è la crescita di SiO2. Il processo di crescita dipende sia dall'umidità della camera che dall'attività attuale del TEOS usato. TEOS si degrada come adsorbe l'acqua dall'aria, facendolo diventare meno efficace con l'età. Questo può manifestarsi come un tasso di crescita significativamente più lento, meno controllabile in pochi mesi, anche con un adeguato stoccaggio della sostanza chimica. 34 Se lo strato SiO2 risultante è più sottile del previsto, questo può indicare un problema con TEOS piuttosto che con l'umidità della camera. Mentre una minore umidità può anche provocare un tasso di crescita più basso e pellicola più sottile, la pellicola risultante dovrebbe anche essere più liscia del normale. Nel frattempo uno strato grossolano a grana e ruvido indicherebbe al contrario un problema con un'elevata umidità.
È anche possibile eseguire questo protocollo su qualsiasi altro substrato liberamente scelto con due requisiti: deve tollerare sia l'incisione HF (passo 12) che le temperature di 200-300 gradi centigradi del PECVD (fase 6). La temperatura può essere abbassata in modo sicuro a 100 gradi centigradi per il PECVD di a-Si se viene utilizzato un substrato più sensibile, ma HF non può essere evitato se il protocollo viene seguito esattamente come descritto. Per eludere HF, sarebbe necessaria l'applicazione di un ulteriore strato sacrificale. Se il requisito dell'incisione HF venisse rimosso, questo protocollo diverrebbe compatibile con una più ampia selezione di materiali e metalli del substrato.
Poiché questo protocollo è costituito da processi di micro e nanofabbricazione comunemente usati e robusti, potrebbe essere combinato con un numero qualsiasi di altri protocolli di microfabbricazione in cui si desiderano piccole dimensioni di feature e forme metalliche complesse. Nel prossimo futuro, soprattutto con l'arrivo della produzione di massa di origami di DNA a basso costo31, c'è il potenziale per questo metodo per facilitare sia l'uso generale che la nanopatterning ad alta velocità per nanofotonica basata su interfaccia e plasmonica55 .
Divulgazioni
Gli autori non hanno nulla da rivelare.
Riconoscimenti
Questo lavoro è stato sostenuto dall'Accademia di Finlandia (progetti 286845, 308578, 303804, 267497), Jane e Aatos Erkko Foundation e La Sigrid Jusélius Foundation. Questo lavoro è stato svolto nell'ambito del programma Academy of Finland Centers of Excellence (2014-2019). Riconosciamo la fornitura di strutture e supporto tecnico da parte di Aalto University Bioeconomy Facilities e OtaNano – Nanomicroscopy Center (Aalto-NMC) e Micronova Nanofabrication Center.
Materiali
| Name | Company | Catalog Number | Comments |
| Acetone | Honeywell | 40289H | Semiconductor grade ULSI, ≥ 99.5 % |
| Agarose | Fisher Bioreagents | 1036603 | Low-EEO, multi-purpose and molecular biology grade |
| Ammonium hydroxide | Fisher Chemical | 10652251 | 25 % ammonia solution, Certified AR for Analysis, d = 0.91 |
| BRANSON 5510 | Branson | Ultrasonic bath | |
| Dimension Icon | Bruker | Atomic force microscope | |
| Electron-beam evaporator IM-9912 | Instrumentti Mattila | Evaporator for PVD | |
| Ethidium bromide | Sigma Aldrich | E8751 | Fluorescent dye for DNA staining |
| Eon Microplate spectrophotometer | BioTek | UV/Vis spectrophotometer used for DNA origami concentration measurements | |
| Gel Doc XR+ Documentation System | BioRad | Gel imaging system | |
| Gel Loading Dye, Blue (6×) | New England Biolabs | B7021S | Bromophenol blue-based loading dye for agarose gel electrophoresis |
| G-storm GS1 Thermal cycler | Gene Technologies | ||
| HBR 4 | IKA | Heating bath | |
| Hydrofluoric acid | Honeywell | 40213H | Semiconductor grade, 49.5-50.5 % |
| Isopropanol | Honeywell | 40301H | Semiconductor grade VLSI, ≥ 99.8 % |
| Magnesium chloride | Sigma Aldrich | M8266 | Anhydrous, ≥ 98 % |
| Mini-Sub Cell GT Horizontal Electrophoresis System | BioRad | ||
| Plasmalab 80+ PECVD | Oxford Instruments | PECVD system | |
| Plasmalab 80+ RIE | Oxford Instruments | RIE system | |
| Poly(ethylene glycol) | Sigma Aldrich | 89510 | BioUltra, 8,000 |
| PowerPac HC High-Current Power Supply | BioRad | ||
| Sapphire substrate (Al2O3) | University Wafer | Thickness: 430 μm, Polish: DSP, Size: 50.8 mm | |
| Sigma VP | Zeiss | Scanning electron microscope | |
| Silica gel | Merck | 1019691000 | With indicator (orange gel), granulate ~1-3 mm |
| Single-stranded Scaffold DNA, type p7249 | Tilibit Nanosystems | At 100 nM concentration | |
| Sodium chloride | Sigma Aldrich | S9888 | ACS reagent, ≥ 99.0 % |
| Staple strands (oligonucleotides) | Integrated DNA Technologies | Sequences can be ordered e.g. at 100 micromolar in Rnase-free water | |
| TAE buffer (50×) pH 8.0 | VWR Chemicals | 444125D | Electran Electrophoresis grade |
| Take3 micro-volume plate | BioTek | Used for DNA origami concentration measurements | |
| Tetraethyl orthosilicate | Sigma Aldrich | 86578 | ≥ 99.0 % (GC) |
Riferimenti
- Seeman, N. C., Sleiman, H. F. DNA nanotechnology. Nature Reviews Materials. 3 (1), 17068 (2017).
- Linko, V., Dietz, H. The enabled state of DNA nanotechnology. Current Opinion in Biotechnology. 24 (4), 555-561 (2013).
- Rothemund, P. W. K. Folding DNA to create nanoscale shapes and patterns. Nature. 440 (7082), 297-302 (2006).
- Hong, F., Zhang, F., Liu, Y., Yan, H. DNA Origami: Scaffolds for Creating Higher Order Structures. Chemical Reviews. 117 (20), 12584-12640 (2017).
- Maune, H. T., et al. Self-assembly of carbon nanotubes into two-dimensional geometries using DNA origami templates. Nature Nanotechnology. 5 (1), 61-66 (2010).
- Hung, A. M., et al. Large-area spatially ordered arrays of gold nanoparticles directed by lithographically confined DNA origami. Nature Nanotechnology. 5 (2), 121-126 (2010).
- Kuzyk, A., et al. DNA-based self-assembly of chiral plasmonic nanostructures with tailored optical response. Nature. 483 (7389), 311-314 (2012).
- Zhang, T., et al. 3D DNA Origami Crystals. Advanced Materials. 30 (28), 1800273 (2018).
- Julin, S., et al. DNA origami directed 3D nanoparticle superlattice via electrostatic assembly. Nanoscale. 11 (10), 4546-4551 (2019).
- Fu, J., Liu, M., Liu, Y., Yan, H. Spatially-Interactive Biomolecular Networks Organized by Nucleic Acid Nanostructures. Accounts of Chemical Research. 45 (8), 1215-1226 (2012).
- Linko, V., et al. DNA-based enzyme reactors and systems. Nanomaterials. 6 (8), 139 (2015).
- Ramakrishnan, S., Subramaniam, S., Stewart, A. F., Grundmeier, G., Keller, A. Regular Nanoscale Protein Patterns via Directed Adsorption through Self-Assembled DNA Origami Masks. ACS Applied Materials & Interfaces. 8 (45), 31239-31247 (2016).
- Grossi, G., Jaekel, A., Andersen, E. S., Saccà, B. Enzyme-functionalized DNA nanostructures as tools for organizing and controlling enzymatic reactions. MRS Bulletin. 42 (12), 920-924 (2017).
- Douglas, S. M., Bachelet, I., Church, G. M. A logic-gated nanorobot for targeted transport of molecular payloads. Science. 335 (6070), 831-834 (2012).
- Li, S., et al. A DNA nanorobot functions as a cancer therapeutic in response to a molecular trigger in vivo. Nature Biotechnology. 36 (3), 258-264 (2018).
- Zhao, Y. -. X., et al. DNA origami delivery system for cancer therapy with tunable release properties. ACS Nano. 6 (10), 8684-8691 (2014).
- Kollmann, F., et al. Superstructure-Dependent Loading of DNA Origami Nanostructures with a Groove-Binding Drug. ACS Omega. 3 (8), 9441-9448 (2018).
- Zhang, D. Y., Seelig, G. Dynamic DNA nanotechnology using strand-displacement reactions. Nature Chemistry. 3 (2), 103-113 (2011).
- Ijäs, H., Nummelin, S., Shen, B., Kostiainen, M. A., Linko, V. Dynamic DNA Origami Devices: from Strand-Displacement Reactions to External-Stimuli Responsive Systems. International Journal of Molecular Sciences. 19 (7), 2114 (2018).
- Li, J., Fan, C., Pei, H., Shi, J., Huang, Q. Smart Drug Delivery Nanocarriers with Self-Assembled DNA Nanostructures. Advanced Materials. 25 (32), 4386-4396 (2013).
- Linko, V., Ora, A., Kostiainen, M. A. DNA Nanostructures as Smart Drug-Delivery Vehicles and Molecular Devices. Trends in Biotechnology. 33 (10), 586-594 (2015).
- Jiang, Q., Liu, S., Liu, J., Wang, Z. G., Ding, B. Rationally Designed DNA-Origami Nanomaterials for Drug Delivery In Vivo. Advanced Materials. , (2018).
- Shen, B., Linko, V., Dietz, H., Toppari, J. J. Dielectrophoretic trapping of multilayer DNA origami nanostructures and DNA origami-induced local destruction of silicon dioxide. Electrophoresis. 36 (2), 255-262 (2015).
- Kuzyk, A., Jungmann, R., Acuna, G. P., Liu, N. DNA Origami Route for Nanophotonics. ACS Photonics. 5 (4), 1151-1163 (2018).
- Liu, N., Liedl, T. DNA-Assembled Advanced Plasmonic Architectures. Chemical Reviews. 118 (6), 3032-3053 (2018).
- Bathe, M., Rothemund, M. DNA Nanotechnology: A Foundation for Programmable Nanoscale Materials. MRS Bulletin. 42 (12), 882-888 (2017).
- Pilo-Pais, M., Acuna, G. P., Tinnefeld, P., Liedl, T. Sculpting light by arranging optical components with DNA nanostructures. MRS Bulletin. 42 (12), 936-942 (2017).
- Graugnard, E., Hughes, W. L., Jungmann, R., Kostiainen, M. A., Linko, V. Nanometrology and super-resolution imaging with DNA. MRS Bulletin. 42 (12), 951-959 (2017).
- Zhong, J., et al. Metallized DNA nanolithography for encoding and transferring spatial information for graphene patterning. Nature Communications. 4, 1663 (2013).
- Zhang, G., Surwade, S. P., Zhou, F., Liu, H. DNA nanostructure meets nanofabrication. Chemical Society Reviews. 42 (7), 2488-2496 (2013).
- Praetorius, F., Kick, B., Behler, K. L., Honemann, M. N., Weuster-Botz, D., Dietz, H. Biotechnological mass production of DNA origami. Nature. 552 (7683), 84-87 (2017).
- Linko, V., et al. One-step large-scale deposition of salt-free DNA origami nanostructures. Scientific Reports. 5, 15634 (2015).
- Arbabi, A., Horie, Y., Bagheri, M., Faraon, A. Dielectric metasurfaces for complete control of phase and polarization with subwavelength spatial resolution and high transmission. Nature Nanotechnology. 10 (11), 937-943 (2015).
- Shen, B., et al. Plasmonic nanostructures through DNA-assisted lithography. Science Advances. 4 (2), (2018).
- Douglas, S. M., et al. Rapid prototyping of 3D DNA-origami shapes with caDNAno. Nucleic Acids Research. 37 (26), 5001-5006 (2009).
- Ke, Y., et al. Multilayer DNA Origami Packed on a Square Lattice. Journal of the American Chemical Society. 131 (43), 15903-15908 (2009).
- Dietz, H., Douglas, S. M., Shih, W. M. Folding DNA into twisted and curved nanoscale shapes. Science. 325 (5941), 725-730 (2009).
- Castro, C. E., et al. A primer to scaffolded DNA origami. Nature Methods. 8 (3), 221-229 (2011).
- Kim, D. -. N., Kilchherr, F., Dietz, H., Bathe, M. Quantitative prediction of 3D solution shape and flexibility of nucleic acid nanostructures. Nucleic Acids Research. 40 (7), 2862-2868 (2011).
- Benson, E., et al. DNA rendering of polyhedral meshes at the nanoscale. Nature. 523 (7561), 441-444 (2015).
- Veneziano, R., et al. Designer nanoscale DNA assemblies programmed from the top down. Science. 352 (6923), 1534 (2016).
- Linko, V., Kostiainen, M. A. Automated design of DNA origami. Nature Biotechnology. 34 (8), 826-827 (2016).
- Nummelin, S., Kommeri, J., Kostiainen, M. A., Linko, V. Evolution of Structural DNA Nanotechnology. Advanced Materials. 30 (24), 1703721 (2018).
- Maffeo, C., Yoo, J., Aksimentiev, A. De novo reconstruction of DNA origami structures through atomistic molecular dynamics simulation. Nucleic Acids Research. 44 (7), 3013-3019 (2016).
- Stahl, E., Martin, T. G., Praetorius, F., Dietz, H. Facile and Scalable Preparation of Pure and Dense DNA Origami Solutions. Angewandte Chemie International Edition. 53 (47), 12735-12740 (2014).
- Shaw, A., Benson, E., Högberg, B. Purification of Functionalized DNA Origami Nanostructures. ACS Nano. 9 (5), 4968-4975 (2015).
- Kuzyk, A., Yurke, B., Toppari, J. J., Linko, V., Törmä, P. Dielectrophoretic Trapping of DNA Origami. Small. 4 (4), 447-450 (2008).
- Lin, C., Perrault, S. D., Kwak, M., Graf, F., Shih, W. M. Purification of DNA-origami nanostructures by rate-zonal centrifugation. Nucleic Acids Research. 41 (2), 40 (2013).
- Douglas, S. M., et al. Self-assembly of DNA into nanoscale three-dimensional shapes. Nature. 459 (7245), 414-418 (2009).
- Ramakrishnan, S., Ijäs, H., Linko, V., Keller, A. Structural stability of DNA origami nanostructures under application-specific conditions. Computational and Structural Biotechnology Journal. 16, 342-349 (2018).
- Kielar, C., et al. On the Stability of DNA Origami Nanostructures in Low-Magnesium Buffers. Angewandte Chemie International Edition. 57 (30), 9470-9474 (2018).
- Surwade, S. P., et al. Nanoscale growth and patterning of inorganic oxides using DNA nanostructure templates. Journal of the American Chemical Society. 135 (18), 6778-6781 (2013).
- Shen, B., Linko, V., Tapio, K., Kostiainen, M. A., Toppari, J. J. Custom-shaped metal nanostructures based on DNA origami silhouettes. Nanoscale. 7 (26), 11267-11272 (2015).
- Liu, W., Zhong, H., Wang, R., Seeman, N. C. Crystalline Two-Dimensional DNA-Origami Arrays. Angewandte Chemie International Edition. 50 (1), 264-267 (2011).
- Shen, B., Kostiainen, M. A., Linko, V. DNA Origami Nanophotonics and Plasmonics at Interfaces. Langmuir. 34 (49), 14911-14920 (2018).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon