È necessario avere un abbonamento a JoVE per visualizzare questo. Accedi o inizia la tua prova gratuita.
Method Article
Quantificazione dei livelli di etanolo negli embrioni di pesce zebra utilizzando la cromatografia dello spazio della testa
In questo articolo
Riepilogo
Questo lavoro descrive un protocollo per quantificare i livelli di etanolo in un embrione di pesce zebra utilizzando la cromatografia del gas dello spazio della testa da metodi di esposizione adeguati alla lavorazione degli embrioni e all'analisi dell'etanolo.
Abstract
I disturbi dello spettro dell'alcolfe fetale (FASD) descrivono un continuum altamente variabile di difetti dello sviluppo indotti dall'etanolo, tra cui dismofomorfologie facciali e disturbi neurologici. Con una patologia complessa, FASD colpisce circa 1 bambino su 100 nati negli Stati Uniti ogni anno. A causa della natura altamente variabile della FASD, i modelli animali si sono dimostrati critici nella nostra attuale comprensione meccanicistica dei difetti di sviluppo indotti dall'etanolo. Un numero crescente di laboratori si è concentrato sull'utilizzo del pesce zebra per esaminare i difetti dello sviluppo indotti dall'etanolo. I pesci zebra producono un gran numero di embrioni emblari traslucidi, geneticamente trattivi, che sono esportati esternamente. Ciò consente ai ricercatori di controllare con precisione la tempistica e il dosaggio dell'esposizione all'etanolo in più contesti genetici e di quantificare l'impatto dell'esposizione all'etanolo embrionale attraverso tecniche di imaging dal vivo. Questo, combinato con l'elevato grado di conservazione della genetica e dello sviluppo con gli esseri umani, ha dimostrato che il pesce zebra è un modello potente in cui studiare la base meccanicistica della teratogenicità dell'etanolo. Tuttavia, i regimi di esposizione all'etanolo hanno variato tra diversi studi sui pesci zebra, il che ha confuso l'interpretazione dei dati dei pesci zebra in questi studi. Ecco un protocollo per quantificare le concentrazioni di etanolo negli embrioni di pesce zebra utilizzando la cromatografia del gas dello spazio della testa.
Introduzione
I disturbi dello spettro dell'alcolfe fetale (FASD) descrivono una vasta gamma di menomazioni neurologiche e dismorfologie craniofacciali associate all'esposizione all'etanolo embrionale1. Molteplici fattori, tra cui la tempistica e il dosaggio dell'esposizione all'etanolo e il background genetico, contribuiscono alla variazione del FASD2,3. Nell'uomo, la complessa relazione di queste variabili rende difficile lo studio e la comprensione dell'eziologia della FASD. I modelli animali si sono dimostrati cruciali nello sviluppo della nostra comprensione della base meccanicistica della teratogenicità dell'etanolo. Un'ampia varietà di sistemi di modelli animali è stata utilizzata per studiare molteplici aspetti della FASD e i risultati sono stati notevolmente coerenti con quanto si trova nell'esposizionenell'uomo 4. I sistemi modello Roditori sono utilizzati per esaminare molti aspetti del FASD, con i topi che sono i più comuni5,6,7. La maggior parte di questo lavoro si è concentrata sui difetti dello sviluppo per l'esposizione precoce all'etanolo8, anche se in seguito l'esposizione all'etanolo ha dimostrato di causare anomalie dello sviluppo pure9. Inoltre, le capacità genetiche dei topi hanno notevolmente aiutato nella nostra capacità di sondare le basi genetiche di FASD10,11. Questi studi sui topi suggeriscono fortemente che ci sono interazioni gene-etanolo con il percorso riccio sonoro, segnalazione dell'acido retinoico, dismutasi Superossido, sintesi di ossido nitrico I, Aldh2 e Fancd28,10,11, 12,13,14,15,16,17,18 19,20,21. Questi studi dimostrano che i modelli animali sono fondamentali per far progredire la nostra comprensione della FASD e dei suoi meccanismi sottostanti.
Il pesce zebra è emerso come un potente sistema modello per esaminare molti aspetti della teratogenesi dell'etanolo22,23. A causa della loro fecondazione esterna, dell'elevata fecondità, della trattatibilità genetica e delle capacità di imaging dal vivo, i pesci zebra sono ideali per studiare fattori come tempismo, dosaggio e genetica della teratogenesi dell'etanolo. L'etanolo può essere somministrato a embrioni messi in scena con precisione e gli embrioni possono quindi essere immagine per esaminare l'impatto diretto dell'etanolo durante i processi di sviluppo. Questo lavoro può essere correlato direttamente agli esseri umani, perché i programmi genetici di sviluppo sono altamente conservati tra il pesce zebra e gli esseri umani e può quindi aiutare a guidare gli studi umani FASD24. Mentre il pesce zebra è stato utilizzato per esaminare la teratogenesi dell'etanolo, la mancanza di consenso nel segnalare concentrazioni di etanolo embrionale rende difficile il confronto con gli esseri umani25. Nei sistemi dei mammiferi, i livelli di alcol nel sangue sono correlati direttamente ai livelli di etanolo tissutale26. Molti degli studi sui pesci zebra trattano gli embrioni prima della formazione completa del loro sistema circolatorio. Senza un campione materno da esaminare, è necessario un processo per valutare le concentrazioni di etanolo per quantificare i livelli di etanolo all'interno dell'embrione. Qui descriviamo un processo per quantificare le concentrazioni di etanolo in un embrione di pesce zebra in via di sviluppo utilizzando la cromatografia del gas dello spazio della testa.
Access restricted. Please log in or start a trial to view this content.
Protocollo
Tutti gli embrioni di pesce zebra utilizzati in questa procedura sono stati allevati e allevati seguendo i protocolli IACUC stabiliti27. Questi protocolli sono stati approvati dall'Università del Texas ad Austin e dall'Università di Louisville.
NOTA: La linea di pesce zebra Tg(fli1:EGFP)y1 è stata utilizzata in questo studio28. Tutta l'acqua utilizzata in questa procedura è acqua sterile di osmosi inversa. Tutte le analisi statistiche sono state eseguite utilizzando Graphpad Prism v8.2.1.
1. Fare i media embrionali
- Per fare uno stock 20x di supporti embrionali, sciogliere 17,5 g di NaCl, 0,75 g di KCl, 2,9 g di CaCl2, 0,41 g di K2HPO4, 0,142 g di NA2HPO4e 4,9 g di MgSO4-7H2O in 1 L di acqua. Ignorare il precipitato bianco che si forma; ciò non avrà alcun impatto sui media. Filtrare sterilizzare la soluzione di magazzino e conservare a 4 gradi centigradi.
- Per creare la soluzione di supporto embrionale funzionante, sciogliere 1,2 g di NaHCO3 in 1 L di 20x di brodo di media embrionali e aggiungere 19 L di acqua. Mantenere la soluzione di supporto embrionale funzionante a 28 gradi centigradi.
2. Misurazione del volume embrionale mediante lo spostamento dell'acqua
NOTA: In questo protocollo vengono utilizzati 24 embrioni successivi alla fecondazione (hpf)(Figura 1). Gli embrioni utilizzati nelle misurazioni del volume non vengono utilizzati nell'analisi dell'etanolo.
- Mettere 10 embrioni e fluidi extraembryonici in un tubo di microcentrifuga da 1,5 mL contrassegnato con un volume di 250 l (Figura 2A). Aggiungere acqua alla linea di riempimento di 250 L (vedere esempio con acqua tinta nella figura 2B).
- Ripetere il passaggio 2.1 per impostare i recipienti per gli embrioni a cui sono stati rimossi i courioni.
- Per rimuovere il chorion, mettere gli embrioni nei loro chorions in un piatto Petri da 100 mm con 2 mg/mL di cocktail proteasi in embriomi a temperatura ambiente (RT) per 10 min. Ogni pochi minuti, ruotare delicatamente gli embrioni per rompere il chorion.
- Una volta che tutti gli embrioni sono liberi dal loro coro, rimuovere gli embrioni dechorionati dal cocktail proteasi/ mezzi di embrione e mettere in un nuovo piatto Petri 100 mm con mezzi embrionali freschi per lavare gli embrioni. Ripetere questo lavaggio passo 1 più tempo (per un totale di 2 lavaggi). Trasferire gli embrioni in una parabola Petri fresca di 100 mm.
- Utilizzando la punta più piccola possibile e senza danneggiare gli embrioni, rimuovere con attenzione tutto il liquido da circa gli embrioni utilizzando un micropipettore p200 (Figura 2C) e pesare l'acqua con una scala con precisione <0,1 mg. Per determinare il volume del campione di 10 embrioni, sottrarre il peso/volume dell'acqua (1 mL di acqua e 1 g di acqua.) rimossa da 250 l. Per determinare il volume di un singolo embrione, dividere la differenza tra 250 l e il peso dell'acqua rimossa da 10.
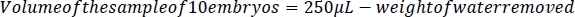
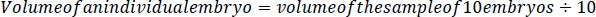
3. Trattamento di embrioni con etanolo
- Raccogliere gli embrioni dai serbatoi di accoppiamento e metterli in una parabola Petri standard da 100 mm. Contare non più di 100 embrioni per singola parabola Petri e incubare a 28,5 gradi centigradi.
NOTA: Se il chorion deve essere rimosso (passaggio 2.2), deve essere rimosso prima di aggiungere l'etanolo. - A 6 hpf, aggiungere fino a 100 embrioni a un nuovo piatto Petri standard da 100 mm con mezzi embrionali o multimediali embrionali , ovvero l'1% di etanolo (v/v). Coprire, ma NON sigillare il piatto Petri. Collocare gli embrioni in un'incubatrice a bassa temperatura impostata a 28,5 gradi centigradi per 18 ore, o fino a quando gli embrioni raggiungono il punto di tempo di sviluppo di 24 hpf.
4. Preparazione del flusso di lavoro prima dell'elaborazione degli embrioni per la cromatografia del gas spaziale della testa
- Fare una soluzione di 5 M NaCl in acqua (450 s sarà necessario per ogni tubo campione) per denaturare tutte le proteine e prevenire il metabolismo dell'etanolo. Preparare il cocktail proteasi (Tabella dei Materiali) ad una concentrazione di 2 mg/mL nei mezzi embrionali.
- Etichettare un tubo di microcentrifuga da 1,5 ml e una fiala cromatografica a gas da 2 mL per ogni campione. Etichettare ulteriori fiale di cromatografo a gas da 2 mL per gli standard di etanolo descritti di seguito, così come aria, acqua e gli spazi vuoti da cocktail 5 M NaCl/proteasi.
- Impostare tre micropipettori p200 due impostati su 50 l, e il terzo impostato su 200 L; un micropipettore p1000 impostato su 450 l e un micropipettore p2 impostato su 2 L. Per le pipette di vetro utilizzate per trasferire gli embrioni dai piatti Petri ai tubi di microcentrifuga da 1,5 mL, passare rapidamente la punta della pipetta attraverso una fiamma per lisciare i bordi per non danneggiare gli embrioni quando li si disegna nella pipetta.
5. Lavorazione degli embrioni per la cromatografia del gas spaziale capo
NOTA: Entrambi gli embrioni nei loro chorion e quelli precedentemente rimossi dai loro chorions sono trattati allo stesso modo per coerenza nel calcolo dei fattori di diluizione.
- Utilizzando i due micropipettori p200 impostati su 50 l, si innunci 50 - l della soluzione cocktail proteasi e disegna 50 l di acqua nel secondo.
- Utilizzando la pipetta di vetro e il micropipettore, posizionare rapidamente 10 embrioni (dal punto 3.2) in un tubo di microcentrifuga da 1,5 mL e chiudere il tappo (come descritto nella Figura 2A). Ripetere l'operazione per tutti i campioni da testare, i controlli e gli embrioni trattati con etanolo.
- Utilizzando il micropipettore p200 impostato su 200 , aprire rapidamente il tappo e rimuovere tutti i supporti embrionali residui (come descritto nella Figura 2C). Posizionare rapidamente il pipetta contenente 50 l di acqua nel tubo e aggiungere rapidamente (per non danneggiare gli embrioni), quindi rimuovere l'acqua. Aggiungere rapidamente 50 l della soluzione di cocktail protease dal pipettor in attesa e chiudere il tappo del tubo (Figura 2D).
NOTA: eseguire questo processo un campione alla volta. - Lasciare che il campione si sieda a RT per 10 min per consentire al cocktail di proteasi di degradare il chorion. Quindi, aggiungere rapidamente 450 luna di 5 M NaCl e chiudere il tappo del tubo (Figura 2E). Vortice i campioni per 10 min. Per accelerare il processo, impostare il mixer per vortice più campioni contemporaneamente.
NOTA: Aggiungere una piccola quantità di una miscela di perline di silice (2 dimensioni, 0,5 mm e 1 mm di perline) a qualsiasi tubo con embrioni più vecchi di 24 cvf. Negli embrioni più vecchi, il notocordo rimarrà intatto indipendentemente da quanto tempo sono omogeneizzati. - Dopo aver omogeneizzato per 10 min, rimuovere rapidamente 2 -L di supernatante embrionale omogeneizzato e aggiungere a una fiala cromatografo a gas. Sigillare rapidamente la fiala con il cappuccio in politetrafluoroetilene.
6. Preparazione delle norme sui supporti e sugli etanoli
- Per preparare gli standard dei media, diluire i supporti con un fattore 10 con una soluzione cocktail NaCl/protease da 5 M. Aggiungere 2 l di ciascun campione a una fiala cromatografica a gas e sigillare con un tappo in politetrafluoroetilene.
- Per preparare gli standard di etanolo, creare una diluizione seriale di 100% etanolo nella soluzione cocktail NaCl/protease da 5 M alle seguenti concentrazioni: 0.3125, 0.625, 1.25, 2.5, 5, 10, 20 e 40 mM. Aggiungete 2 l di ogni standard in una fiala cromatografica a gas e si guarniscono con un cappuccio in politetrafluoroetilene.
7. Preparazione del cromatografo del gas dello spazio della testa
NOTA: questa configurazione e protocollo potrebbe essere necessario modificare a seconda del cromatografo a gas utilizzato. La cromatografia del gas spaziale della testa viene utilizzata per quantificare i livelli di etanolo, non per la separazione.
- Impostare il riscaldatore per l'autocampionatore a 58 gradi centigradi e accenderlo. Lasciare che il riscaldatore raggiunga i 58 gradi centigradi e accendere le linee di gas ad aria e idrogeno che alimentano il cromatografo a gas (per la ionizzazione della fiamma utilizzata per quantificare l'etanolo).
NOTA: il psi deve essere impostato per azionare correttamente il cromatografo secondo le specifiche del produttore. Assicurarsi che la linea dell'elio sia accesa e impostata sul psi corretto. - Per il setto, assicurarsi che il numero di campioni iniettati non sia >100. Per la fibra di iniezione, assicurarsi che il numero di campioni iniettati non è >500.
NOTA: Se uno dei due è superiore alla quantità di iniezioni, dovranno essere modificate prima di eseguire i campioni di prova. - Accendere il software di analisi e assicurarsi che la workstation sia configurata correttamente. Memorizzare tutti i dati in base agli standard di laboratorio/reparto. Creare un nuovo elenco di esempio per gli esempi da eseguire.
- Avviare il metodo di avvio e attendere che il rilevatore di ionizzazione della fiamma si stabilizzi prima di eseguire i campioni. Una volta stabile, eseguire il metodo di avvio del software per pulire la fibra.
8. Misurazioni campione con cromatografia a gas dello spazio della testa
- Una volta completato il metodo di avvio, creare 1 nuova riga per ogni campione nell'elenco di esempio. Dal menu dei metodi nel software di analisi, caricare 436 corrente spme etanolo 2013 3min assorbire 2_5min corsa rg. METH per ogni campione nell'elenco di esempio.
- Compilare l'elenco dei campioni, a partire dall'aria, dall'acqua, dagli spazi vuoti dei cocktail 5 M NaCl/protease. Quindi immettere gli standard in ordine, da 0,3125 a 40 mM. Seguire gli standard con un secondo giro di aria, acqua, e 5 M NaCl / protease cocktail spazi vuoti.
- Inserire tutti i campioni di supernata dell'embrione omogeneizzato da testare, dalla concentrazione di etanolo più bassa a quella più alta. Terminare inserendo un terzo e ultimo giro dell'aria, acqua, e 5 M NaCl / protease cocktail vuoti.
- Aggiungere le fiale del cromatografo a gas all'autocampionatore nell'ordine in cui sono stati immessi i campioni. Consentire il riscaldamento dei campioni per 10-15 min. Avviare l'esecuzione dell'esempio nel software.
- Dopo aver eseguito tutti i campioni e gli spazi vuoti finali, attivare il metodo di arresto nel software aggiungendo un campione finale nell'elenco dei campioni ed eseguendo standby. METH. Eseguire il backup di tutti i dati acquisiti durante le esecuzioni dell'esempio. Spegnere l'apparecchiatura nel seguente ordine: riscaldatore autocampionatore, serbatoio di idrogeno e, solo dopo che la temperatura del cromatografo raggiunge i 30 gradi centigradi, il serbatoio dell'aria. Lasciare il serbatoio di elio per preservare la colonna di cera.
9. Esempio di integrazione dei picchi di etanolo e analisi della concentrazione dei campioni
NOTA: tutti i valori a partire da 9.3 sono stati calcolati in un file Excel precompilato.
- Una volta completato il metodo di spegnimento, fare clic su Apri cromatogramma. Aprire la cartella contenente i risultati. I campioni vengono integrati automaticamente in questo programma.
- Nei risultati, assicurarsi che siano stati integrati i picchi corretti (picchi di etanolo tra 2 e 2,5). Una volta che tutti i campioni sono stati confermati, stampare o esportare i risultati.
- Tracciare l'altezza massima degli standard di etanolo su un grafico. Calcolare i valori di pendenza, intercetta Y e R2 per gli standard etanolo (R2 deve essere >0.99).
NOTA: questi valori verranno utilizzati per determinare la concentrazione di etanolo dall'altezza massima del campione. - Per ogni campione, sottrarre l'altezza massima del campione dall'intercetta Y degli standard di etanolo. Dividere questo valore per la pendenza degli standard di etanolo per ottenere il valore di etanolo per ogni campione nelle fiale GC.
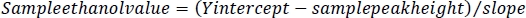
- Per calcolare la concentrazione di etanolo negli embrioni, calcolare innanzitutto il fattore di diluizione per ogni campione. Prendere la misura del volume calcolata al punto 2.3 di questo protocollo e dividerla per la misura del volume più 500 L. Questo rappresenta la soluzione cocktail NaCl/protease da 5 M aggiunta ad ogni campione durante l'elaborazione dell'embrione (passaggi 5.3 e 5.4).
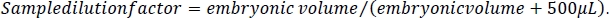
- Usando questo fattore di diluizione del campione, moltiplicare per il valore di etanolo del campione per ogni campione. I risultati saranno in concentrazione mM. Calcolare i campioni di riferimento dei supporti moltiplicando il valore dell'etanolo del supporto per il fattore di diluizione pari a 10.
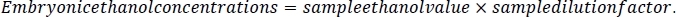
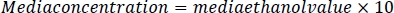
Access restricted. Please log in or start a trial to view this content.
Risultati
I livelli di etanolo sanguigno non possono essere determinati nei primi pesci zebra embrionali, perché non hanno un sistema circolatorio completamente formato. Per determinare il livello di concentrazione di etanolo negli embrioni di pesce zebra, i livelli di etanolo sono misurati direttamente dal tessuto embrionale omogeneizzato. Per misurare correttamente le concentrazioni di etanolo embrionale, è necessario tener conto del volume embrionale. L'embrione (tuorlo attaccato) si trova all'interno del chorion (guscio d'uo...
Access restricted. Please log in or start a trial to view this content.
Discussione
Come sistema di modelli di sviluppo, il pesce zebra è ideale per studiare l'impatto dei fattori ambientali sullo sviluppo. Producono un gran numero di embrioni fecondati esternamente, il che consente di tempistica precisa e paradigmi di dosaggio negli studi sull'etanolo. Questo, combinato con le capacità di imaging dal vivo e la conservazione genetica e dello sviluppo con gli esseri umani, rende il pesce zebra un potente sistema modello per gli studi di teratologia. Descritto è un protocollo per misurare le concentraz...
Access restricted. Please log in or start a trial to view this content.
Divulgazioni
Gli autori non hanno nulla da rivelare.
Riconoscimenti
La ricerca presentata in questo articolo è stata supportata da precedenti sovvenzioni da National Institutes of Health/National Institute of Dental and Craniofacial Research (NIH/NIDCR) R01DE020884 a J.K.E. e National Institutes of Health/National Institute on Alcohol Abuse and Alcoholism (NIH/NIAAA) F32AA021320 a C.B.L. e con l'attuale sovvenzione da parte dei National Institutes of Health/National Institute on Alcohol Abuse (NIH/NIAAA) R00AA023560 a C.B.L. Ringraziamo Rueben Gonzales per aver fornito e assistito l'analisi del cromatografo a gas. Ringraziamo Tiahna Ontiveros e la dott.ssa Gina Nobles a scrivere assistenza.
Access restricted. Please log in or start a trial to view this content.
Materiali
| Name | Company | Catalog Number | Comments |
| Air | Provided by contract to the university | ||
| Analytical Balance | VWR | 10204-962 | |
| AutoSampler, CP-8400 | Varian | Gas Chromatograph Autosampler | |
| Calcium Chloride | VWR | 97062-590 | |
| Ethanol | Decon Labs | 2701 | |
| Gas chromatograph vial with polytetrafluoroethylene/silicone septum and plastic cap 2 mL | Agilent | 8010-0198 | Can reuse the vials after cleaning, but not the caps/septa |
| Gas Chromatograph, CP-3800 | Varian | ||
| Helium | Provided by contract to the university | ||
| HP Innowax capillary column | Agilent | 19095N-123I | 30 m x 0.53 mm x 1.0 μm film thick |
| Hyrdogen | Provided by contract to the university | ||
| Magnesium Sulfate (Heptahydrate) | Fisher Scientific | M63-500 | |
| Microcentrifuge tube 1.5 mL | Fisher Scientific | 2682002 | |
| Micropipette tips 10 μL | Fisher Scientific | 13611106 | |
| Micropipette tips 1000 μL | Fisher Scientific | 13611127 | |
| Micropipette tips 200 μL | Fisher Scientific | 13611112 | |
| Petri dishes 100 mm | Fisher Scientific | FB012924 | |
| Pipetman L p1000L Micropipette | Gilson | FA10006M | |
| Pipetman L p200L Micropipette | Gilson | FA10005M | |
| Pipetman L p2L Micropipette | Gilson | FA10001M | |
| Polytetrafluoroethylene/silicone septum and plastic cap | Agilent | 5190-7021 | Replacement caps/septa for gas chromatograph vials |
| Potassium Chloride | Fisher Scientific | P217-500 | |
| Potassium Phosphate (Dibasic) | VWR | BDH9266-500G | |
| Pronase | VWR | 97062-916 | |
| Silica Beads .5 mm | Biospec Products | 11079105z | |
| Silica Beads 1.0 mm | Biospec Products | 11079110z | |
| Sodium Bicarbonate | VWR | BDH9280-500G | |
| Sodium Chloride | Fisher Scientific | S271-500 | |
| Sodium Phosphate (Dibasic) | Fisher Scientific | S374-500 | |
| Solid-phase microextraction fiber assembly Carboxen/Polydimethylsiloxane | Millipore Sigma | 57343-U | Replacement fibers |
| Star Chromatography Workstation | Varian | Chromatography software | |
| Thermogreen Low Bleed (LB-2) Septa | Millipore Sigma | 23154 | Replacement inlet septa |
Riferimenti
- Elliott, E. J., Payne, J., Morris, A., Haan, E., Bower, C. Fetal alcohol syndrome: a prospective national surveillance study. Archive of Diseases in Childhood. 93 (9), 732-737 (2008).
- Cudd, T. A. Animal model systems for the study of alcohol teratology. Experimental Biology and Medicine. 230 (6), 389-393 (2005).
- Williams, J. F., Smith, V. C. Committee on Substance Abuse. Fetal Alcohol Spectrum Disorders. Pediatrics. 136 (5), 1395-1406 (2015).
- Patten, A. R., Fontaine, C. J., Christie, B. R. A comparison of the different animal models of fetal alcohol spectrum disorders and their use in studying complex behaviors. Frontiers in Pediatrics. 2, 93(2014).
- Petrelli, B., Weinberg, J., Hicks, G. G. Effects of prenatal alcohol exposure (PAE): insights into FASD using mouse models of PAE. Biochemistry and Cell Biology. 96 (2), 131-147 (2018).
- Mayfield, J., Arends, M. A., Harris, R. A., Blednov, Y. A. Genes and Alcohol Consumption: Studies with Mutant Mice. International Review Neurobiology. 126, 293-355 (2016).
- Marquardt, K., Brigman, J. L. The impact of prenatal alcohol exposure on social, cognitive and affective behavioral domains: Insights from rodent models. Alcohol. 51, 1-15 (2016).
- Sulik, K. K. Genesis of alcohol-induced craniofacial dysmorphism. Experimental Biology and Medicine. 230 (6), 366-375 (2005).
- Lipinski, R. J., et al. Ethanol-induced face-brain dysmorphology patterns are correlative and exposure-stage dependent. PLoS One. 7 (8), 43067(2012).
- Eberhart, J. K., Parnell, S. The genetics of fetal alcohol spectrum disorders. Alcoholism: Clinical and Experimental Research. 40 (6), 1154-1165 (2016).
- Becker, H. C., Diaz-Granados, J. L., Randall, C. L. Teratogenic actions of ethanol in the mouse: a minireview. Pharmacology, Biochemistry and Behavior. 55 (4), 501-513 (1996).
- Ahlgren, S. C., Thakur, V., Bronner-Fraser, M. Sonic hedgehog rescues cranial neural crest from cell death induced by ethanol exposure. Proceedings of the National Academy of Sciences of the United States of America. 99 (16), 10476-10481 (2002).
- Loucks, E. J., Ahlgren, S. C. Deciphering the role of Shh signaling in axial defects produced by ethanol exposure. Birth Defects Research Part A: Clinical and Molecular Teratology. 85 (6), 556-567 (2009).
- Hong, M., Krauss, R. S. Cdon mutation and fetal ethanol exposure synergize to produce midline signaling defects and holoprosencephaly spectrum disorders in mice. PLoSGenetics. 8 (10), 1002999(2012).
- Aoto, K., Shikata, Y., Higashiyama, D., Shiota, K., Motoyama, J. Fetal ethanol exposure activates protein kinase A and impairs Shh expression in prechordal mesendoderm cells in the pathogenesis of holoprosencephaly. Birth Defects Research Part A: Clinical and Molecular Teratology. 82 (4), 224-231 (2008).
- Deltour, L., Ang, H. L., Duester, G. Ethanol inhibition of retinoic acid synthesis as a potential mechanism for fetal alcohol syndrome. The FASEB Journal. 10 (9), 1050-1057 (1996).
- Wentzel, P., Eriksson, U. J. Ethanol-induced fetal dysmorphogenesis in the mouse is diminished by high antioxidative capacity of the mother. Toxicological Sciences. 92 (2), 416-422 (2006).
- Karacay, B., Mahoney, J., Plume, J., Bonthius, D. J. Genetic absence of nNOS worsens fetal alcohol effects in mice. II: microencephaly and neuronal losses. Alcoholism: Clinical and Experimental Research. 39 (2), 221-231 (2015).
- Bonthius, D. J. Jr, Winters, Z., Karacay, B., Bousquet, S. L., Bonthius, D. J. Importance of genetics in fetal alcohol effects: null mutation of the nNOS gene worsens alcohol-induced cerebellar neuronal losses and behavioral deficits. Neurotoxicology. 46, 60-72 (2015).
- Bonthius, D. J., et al. Deficiency of neuronal nitric oxide synthase (nNOS) worsens alcohol-induced microencephaly and neuronal loss in developing mice. Brain Research. Developmental Brain Research. 138 (1), 45-59 (2002).
- Langevin, F., Crossan, G. P., Rosado, I. V., Arends, M. J., Patel, K. J. Fancd2 counteracts the toxic effects of naturally produced aldehydes in mice. Nature. 475 (7354), 53-58 (2011).
- Lovely, C. B., Fernandes, Y., Eberhart, J. K. Fishing for Fetal Alcohol Spectrum Disorders: Zebrafish as a Model for Ethanol Teratogenesis. Zebrafish. 13 (5), 391-398 (2016).
- Fernandes, Y., Buckley, D. M., Eberhart, J. K. Diving into the world of alcohol teratogenesis: a review of zebrafish models of fetal alcohol spectrum disorder. Biochemistry and Cell Biology. 96 (2), 88-97 (2018).
- McCarthy, N., et al. Pdgfra protects against ethanol-induced craniofacial defects in a zebrafish model of FASD. Development. 140 (15), 3254-3265 (2013).
- Lovely, C. B., Nobles, R. D., Eberhart, J. K. Developmental age strengthens barriers to ethanol accumulation in zebrafish. Alcohol. 48 (6), 595-602 (2014).
- Harris, R. A., Trudell, J. R., Mihic, S. J. Ethanol's molecular targets. Science Signaling. 1 (28), (2008).
- Westerfield, M. The Zebrafish Book: A guide for the laboratory use of zebrafish Danio (Brachydanio) rerio. , University of Oregon Press. Eugene, OR. (1993).
- Lawson, N. D., Weinstein, B. M. In vivo imaging of embryonic vascular development using transgenic zebrafish. Developmental Biology. 248 (2), 307-318 (2002).
- Hagedorn, M., Kleinhans, F. W., Artemov, D., Pilatus, U. Water Distribution and permeability of zebrafish embryos, Brachydanio rerio. Journal of Experimental Zoology. 278 (6), 356-371 (1997).
- Lippi, G., et al. The alcohol used for cleansing the venipuncture site does not jeopardize blood and plasma alcohol measurement with head-space gas chromatography and an enzymatic assay. Biochemia Medica. 27 (2), 398-403 (2017).
- Poklis, J. L., Wolf, C. E., Peace, M. R. Ethanol concentration in 56 refillable electronic cigarettes liquid formulations determined by headspace gas chromatography with flame ionization detector (HS-GC-FID). Drug Testing and Analysis. 9 (10), 1637-1640 (2017).
- Heit, C., et al. Quantification of Neural Ethanol and Acetaldehyde Using Headspace GC-MS. Alcoholism: Clinical and Experimental Research. 40 (9), 1825-1831 (2016).
- Chun, H. J., Poklis, J. L., Poklis, A., Wolf, C. E. Development and Validation of a Method for Alcohol Analysis in Brain Tissue by Headspace Gas Chromatography with Flame Ionization Detector. Journal of Analytical Toxicology. 40 (8), 653-658 (2016).
- Schlatter, J., Chiadmi, F., Gandon, V., Chariot, P. Simultaneous determination of methanol, acetaldehyde, acetone, and ethanol in human blood by gas chromatography with flame ionization detection. Human and Experimental Toxicology. 33 (1), 74-80 (2013).
- Schier, C. J., Mangieri, R. A., Dilly, G. A., Gonzales, R. A. Microdialysis of ethanol during operant ethanol self-administration and ethanol determination by gas chromatography. Journal of Visualized Experiments. (67), e4142(2012).
- Adalsteinsson, E., Sullivan, E. V., Mayer, D., Pfefferbaum, A. In vivo quantification of ethanol kinetics in rat brain. Neuropsychopharmacology. 31 (12), 2683-2691 (2006).
- Quertemont, E., Green, H. L., Grant, K. A. Brain ethanol concentrations and ethanol discrimination in rats: effects of dose and time. Psychopharmacology. 168 (3), 262-270 (2003).
- Flentke, G. R., Klinger, R. H., Tanguay, R. L., Carvan, M. J., Smith, S. M. An evolutionarily-conserved mechanism of calcium-dependent neurotoxicity. Alcoholism: Clinical and Experimental Research. 38 (5), 1255-1265 (2014).
- Reimers, M. J., Flockton, A. R., Tanguay, R. L. Ethanol- and acetaldehyde-mediated developmental toxicity in zebrafish. Neurotoxicology and Teratology. 26 (6), 769-781 (2004).
- Zhang, C., Ojiaku, P., Cole, G. J. Forebrain and hindbrain development in zebrafish is sensitive to ethanol exposure involving agrin, Fgf, and sonic hedgehog function. Birth Defects Research Part A: Clinical and Molecular Teratology. 97 (1), 8-27 (2013).
Access restricted. Please log in or start a trial to view this content.
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon