Method Article
Visualizzazione di organelli in situ mediante tomografia crio-STEM
In questo articolo
Riepilogo
La tomografia crio-STEM fornisce un mezzo per visualizzare organelli di cellule intatte senza incorporamento, sezionamento o altre preparazioni invasive. La risoluzione 3D ottenuta è attualmente nell'intervallo di pochi nanometri, con un campo visivo di diversi micrometri e uno spessore accessibile nell'ordine di 1 μm.
Abstract
La microscopia elettronica criogenica (cryo-EM) si basa sull'imaging di campioni biologici o organici incorporati nel loro mezzo acquoso nativo; L'acqua viene solidificata in un vetro (cioè vetrificato) senza cristallizzazione. Il metodo crio-EM è ampiamente utilizzato per determinare la struttura di macromolecole biologiche recentemente ad una risoluzione quasi atomica. L'approccio è stato esteso allo studio di organelli e cellule utilizzando la tomografia, ma la modalità convenzionale di imaging EM a trasmissione ad ampio campo subisce una grave limitazione nello spessore del campione. Ciò ha portato a una pratica di fresatura di lamelle sottili utilizzando un fascio ionico focalizzato; L'alta risoluzione è ottenuta mediante la media dei sottotomogrammi dalle ricostruzioni, ma le relazioni tridimensionali al di fuori dello strato rimanente vengono perse. La limitazione dello spessore può essere aggirata mediante imaging a scansione di sonda, simile al EM a scansione o al microscopio a scansione laser confocale. Mentre la microscopia elettronica a trasmissione a scansione (STEM) nella scienza dei materiali fornisce la risoluzione atomica in singole immagini, la sensibilità dei campioni biologici criogenici all'irradiazione elettronica richiede considerazioni speciali. Questo protocollo presenta una configurazione per la criotomografia utilizzando STEM. La configurazione topica di base del microscopio è descritta sia per i sistemi a due che a tre condensatori, mentre l'automazione è fornita dal software SerialEM non commerciale. Vengono inoltre descritti i miglioramenti per l'acquisizione dei lotti e l'allineamento correlativo alle mappe di fluorescenza acquisite in precedenza. Ad esempio, mostriamo la ricostruzione di un mitocondrio, evidenziando la membrana interna ed esterna e i granuli di fosfato di calcio, nonché i microtubuli circostanti, i filamenti di actina e i ribosomi. La tomografia crio-STEM eccelle nel rivelare il teatro degli organelli nel citoplasma e, in alcuni casi, anche la periferia nucleare delle cellule aderenti in coltura.
Introduzione
La visualizzazione tridimensionale (3D) degli organelli è un compito fondamentale nella moderna biologia cellulare. Date le scale coinvolte, che vanno da decine di nanometri per le vescicole secretorie a molti micron per il nucleo cellulare, è difficile trovare una singola tecnica di microscopia adatta a tutte le applicazioni. Mentre la moderna microscopia a fluorescenza può coprire gran parte della gamma in termini di risoluzione, appaiono solo le molecole etichettate. Il teatro cellulare rimane il regno della microscopia elettronica. I metodi convenzionali di fissazione chimica, incorporazione della plastica e colorazione con metalli pesanti sono fortemente invasivi, quindi i risultati possono dipendere dai dettagli della preparazione del campione. Cryo-EM, d'altra parte, è vincolato dalla necessità di vetrificare il mezzo acquoso; cristalli di ghiaccio che formano diffratto l'illuminazione elettronica, causando artefatti di contrasto di contrasto superiore rispetto al materiale organico di interesse.
L'ultimo decennio ha visto una proliferazione di tecniche di imaging EM sviluppate o adattate per studi cellulari1. Il congelamento ad alta pressione combinato con la fresatura iterativa a fascio ionico focalizzato (FIB) e l'imaging seriale della superficie utilizzando il microscopio elettronico a scansione (cioè FIB-SEM) è attualmente il metodo di scelta per campioni di grandidimensioni 2. La tomografia criogenica a raggi X molli (cryo-SXT) è adatta per campioni di diverse dimensioni micron, limitata dalla caratteristica lunghezza di assorbimento dei raggi X molli in acqua 3,4,5. Questa scala include molti tipi di cellule intatte e la natura quantitativa del contrasto di assorbimento dei raggi X aggiunge un aspetto della misurazione della concentrazione6 o della spettroscopia7. Se combinata con la media dei sottotomogrammi, la tomografia elettronica a criotrasmissione (cryo-ET), basata sulla microscopia elettronica a trasmissione di contrasto di fase (TEM), offre la massima risoluzione per macromolecole o complessi 8,9,10. Tuttavia, è raro che gli organelli intatti siano così regolari da poter essere mediati interi. Inoltre, il modo convenzionale di TEM a campo largo è limitato per lo spessore del campione a poche centinaia di nanometri dalla combinazione di scattering anelastico (che comporta perdita di energia) nel campione e aberrazione cromatica nella lente magnetica dell'obiettivo11,12. La grande diffusione di energia impone l'uso di un filtro di energia per rimuovere la foschia fuori fuoco risultante, ma il campione sensibile subisce ancora danni da radiazioni mentre il segnale dell'immagine diventa esponenzialmente più debole con l'aumentare dello spessore.
La modalità di imaging alternativa, la trasmissione a scansione EM (STEM), aggira la necessità di filtraggio dell'energia e mantiene gli elettroni sparsi anelasticamente per la formazione dell'immagine, anche se attualmente a una risoluzione inferiore rispetto alla tomografia TEM (Figura 1). In realtà, non si forma alcuna immagine reale. Invece, come in un EM di scansione, le misurazioni vengono effettuate punto per punto e l'immagine viene assemblata dal computer. L'ingrandimento è determinato solo dalla dimensione dei passaggi di scansione senza modificare le correnti dell'obiettivo. Se configurato correttamente, l'intervallo utile di spessore del campione per la tomografia crio-STEM (CSTET) può raggiungere 1,5 o anche 2 μm, sebbene la zona di comfort, dove l'intensità del segnale utile rimane una frazione significativa dell'illuminazione, sia di circa 600-900 nm11,13. Questo è sufficiente per vedere una grande frazione del citoplasma e occasionalmente un bordo del nucleo cellulare. In pratica, troviamo che la vetrificazione con il metodo standard di immersione nel fluido criogenico impone un vincolo più severo sullo spessore rispetto all'imaging STEM. L'obiettivo di questo articolo video è facilitare l'incorporazione di CSTET nella cassetta degli strumenti per l'imaging di cellule e organelli nei laboratori di ricerca e nelle strutture di microscopia.
La prima sfida è che le operazioni al microscopio in CSTET non sono ancora standardizzate per le applicazioni delle scienze della vita come lo sono state per la tomografia crio-TEM. L'hardware STEM è stato raramente (se non mai) destinato al mercato crio-EM. Tuttavia, questo sta cambiando con la nuova generazione di microscopi e molti strumenti esistenti possono essere adattati. STEM come tecnica è decollata e in gran parte presa in consegna nelle scienze dei materiali, dove c'è anche un interesse nascente per i metodi criogenici e a basso dosaggio14,15. La letteratura scientifica dei materiali abbonda di acronimi BF-STEM, ADF-STEM, HAADF-STEM, 4D-STEM, DPC-STEM, ecc., Che aggiungono confusione. Offriamo qui un punto di partenza consigliato che, nella nostra esperienza collettiva presso il Weizmann Institute of Science, fornisce il protocollo più generale per risultati utili basati sull'imaging STEM a campo luminoso (BF)16. In nessun modo esaurisce o addirittura esplora la gamma di possibilità, ma servirà come base per ulteriori miglioramenti. Mentre enfatizziamo il crio-STEM, la maggior parte del protocollo è ugualmente rilevante per la tomografia STEM a temperatura ambiente di sezioni incorporate in plastica.
L'essenza di STEM è scansionare il campione con una sonda elettronica focalizzata (Figura 1), il cono di illuminazione, e registrare i segnali dal piano di diffrazione (scattering) in trasmissione, pixel per pixel, per produrre immagini 2D17,18. I campioni amorfi, compresa la maggior parte dei materiali cellulari, produrranno un modello di diffusione diffuso nella trasmissione. La configurazione STEM pratica più semplice è quella di posizionare un rilevatore circolare per registrare il disco centrale (cioè l'illuminazione della sonda che verrebbe trasmessa senza un campione). Il campione disperde gli elettroni lontano da questo cono di illuminazione nella misura in cui il segnale diminuisce. Questo produce un'immagine BF: il campione appare scuro su uno sfondo luminoso. Un rivelatore anulare può anche (o invece) essere utilizzato per rilevare la dispersione dal campione al di fuori del cono di illuminazione. Con il campione rimosso, non c'è segnale. Quando un campione è sul posto, gli oggetti appaiono luminosi su uno sfondo scuro nell'immagine in campo scuro (DF). La nomenclatura per STEM (BF, campo scuro anulare [ADF], campo scuro anulare ad alto angolo [HAADF], ecc.) si riferisce principalmente agli intervalli di angoli di raccolta per i rivelatori.
L'angolo di convergenza dell'illuminazione rappresenta un adattamento essenziale dello STEM alla tomografia cellulare. Quando la priorità assoluta è l'alta risoluzione, l'angolo di convergenza dovrebbe essere il più ampio possibile. (Questo è simile alla microscopia a scansione laser confocale; la risoluzione è determinata dal diametro della sonda, che scala come la lunghezza d'onda divisa per l'apertura numerica. Si noti che ci riferiamo all'angolo di semiangolo o semiconvergenza per EM.) Quando la priorità è la profondità di campo, invece, un compromesso nella risoluzione offre un grande vantaggio, in quanto il fascio focalizzato rimane approssimativamente parallelo per una distanza pari al doppio della lunghezza d'onda divisa per semi-angolo al quadrato. Idealmente, l'intero volume della cella rimane a fuoco19. Ad esempio, a 300 keV, la lunghezza d'onda dell'elettrone di deBroglie è 0,002 nm, quindi una convergenza di 1 mrad produce una risoluzione di 2 nm e una profondità di campo di 4 micron. In queste condizioni, la tomografia può essere eseguita anche senza messa a fuoco durante il processo di raccolta dei dati, ma solo una volta all'inizio dell'acquisizione. Una STEM convenzionale con capacità di tomografia può raggiungere un angolo di semiconvergenza di 7 o 8 mrad; Pertanto, in linea di principio, potremmo raggiungere una risoluzione dell'ordine di 0,25 nm, ma poi con una profondità focale di soli 62 nm. Questo è chiaramente troppo sottile per l'imaging cellulare. I microscopi più avanzati con tre lenti a condensatore offrono una regolazione continua dell'angolo di semiconvergenza su un intervallo considerevole. Con la più tradizionale configurazione a due condensatori, la convergenza è fissata discretamente dall'apertura del condensatore (C2).
Per campioni robusti e incorporati in plastica, è possibile registrare una serie focale ad ogni inclinazione e combinarle per un'alta risoluzione20, ma per i campioni criogenici, il bilancio della radiazione è troppo severamente limitato. Infine, nel valutare i vantaggi dell'imaging BF o DF, per campioni spessi, si dovrebbero considerare gli effetti della diffusione elastica multipla nel campione. Il segnale BF è meno corrotto dallo scattering multiplo e mostra una risoluzione più elevata per campioni spessi 16,21.
Un'utile regola empirica è stata quella di impostare angoli di raccolta molte volte più grandi della convergenza. Più spesso è il campione, più grande dovrebbe essere il disco di raccolta. Un disco troppo piccolo fornirà una bassa intensità del segnale; Un disco troppo grande comporterà uno scarso contrasto dell'immagine, poiché solo la dispersione dell'angolo più alto contribuirà. Gli angoli di raccolta devono essere ottimizzati per un determinato campione. Gli angoli del rilevatore in funzione della lunghezza (diffrazione) della telecamera devono essere calibrati in modo indipendente. Possono essere visualizzati comodamente dal software del microscopio. In pratica, un fattore da due a cinque nel rapporto tra semiangoli di raccolta e illuminazione, rispettivamente da θ a α (Figura 1), è un punto di partenza raccomandato per CSTET di campioni cellulari.
Il seguente protocollo descrive l'operazione di tomografia STEM utilizzando il popolare software SerialEM per il controllo del microscopio22,23. SerialEM non è legato a un produttore specifico ed è ampiamente utilizzato nella tomografia TEM. La maggior parte delle operazioni di allestimento per la tomografia possono essere eseguite direttamente dal TEM. La strategia di SerialEM consiste nel modellare il sistema di scansione come una telecamera. Ciò consente il semplice crossover da TEM a STEM. Si dovrebbe tenere presente, tuttavia, che parametri come l'ingrandimento e il binning sono interamente artificiali. I parametri importanti sono il campo visivo in micron, il numero di pixel nel campo visivo e il tempo di esposizione. La spaziatura dei pixel, o campionamento, è il campo lineare diviso per il numero di pixel, mentre il tempo di permanenza è il numero di pixel diviso per il tempo di esposizione.
La configurazione minima per STEM e CSTET comporta tre caratteristiche sul microscopio: un generatore di scansione, un rilevatore STEM e un software di controllo della tomografia. Il protocollo fa riferimento alla nomenclatura di FEI/Thermo Fisher Scientific (TFS), ma i concetti sono generici. Il software proprietario di TFS è stato descritto in JoVE per TEM24 e il funzionamento STEM è molto simile.
Partiamo dal presupposto che il microscopio sia stato allineato in anticipo dal team di assistenza o da personale esperto e che un allineamento di colonne possa essere richiamato caricando un file. Le regolazioni minori sono chiamate allineamenti diretti e possono essere memorizzate nei cosiddetti registri FEG (microscopi TFS). Gli allineamenti diretti includono il centro di rotazione, i punti di rotazione, l'allineamento di diffrazione e la compensazione per l'astigmatismo del condensatore. Le regolazioni devono essere eseguite in modo iterativo. Si noti che i microscopi TFS implementano modalità distinte nanosonda (nP) e microsonda (μP); per una data apertura del condensatore, questi forniscono un campo visivo relativamente stretto o ampio con illuminazione parallela in TEM e un fascio di elettroni più o meno convergente (strettamente focalizzato) in STEM, rispettivamente. Altri produttori utilizzano schemi diversi per coprire la gamma di angoli di convergenza.
Prima di iniziare, è necessario scegliere il campo visivo, L, e il campionamento (larghezza dei pixel), l, a seconda del campione in studio. Ad esempio, per l = 1 nm/pixel, è necessario scegliere un'immagine di 4.000 x 4.000 pixel che coprirà un campo visivo di 4 μm2 . La risoluzione sarà, nella migliore delle ipotesi, il doppio del campionamento spaziale, quindi 2 nm, e il diametro della sonda, d, dovrebbe corrispondere a quello. La calibrazione dell'angolo della sonda va oltre lo scopo di questo protocollo, quindi supponiamo che sia disponibile una tabella o una lettura dello schermo. Il diametro della sonda è approssimativamente la lunghezza d'onda dell'elettrone divisa per l'angolo di semiconvergenza (in radianti): d = λ/θ. La lunghezza d'onda, λ, è 0,002 nm per 300 keV e 0,0025 nm per elettroni da 200 keV, quindi θ di 1 mrad fornirà un diametro spot di 2 o 2,5 nm, rispettivamente.
Il protocollo è presentato in una progressione di complessità crescente. Il primo compito è quello di produrre un'immagine STEM, che dipende dal software del produttore del microscopio, e poi una serie di inclinazione, per la quale utilizziamo SerialEM. Molti lettori avranno senza dubbio familiarità con SerialEM, quindi i compiti più complicati verranno naturalmente. Non è necessario seguire rigorosamente le procedure. Gli sviluppi relativi all'automazione possono essere implementati direttamente per STEM e per TEM. Gli utenti esperti probabilmente invertiranno il protocollo, iniziando con la registrazione correlativa delle mappe di fluorescenza e continuando a impostare la tomografia batch. Ulteriori dettagli possono essere trovati nella vasta documentazione e nelle librerie di tutorial per SerialEM stesso, incluso un recente articolo di JoVE sugli ultimi sviluppi nell'automazione25.
Protocollo
1. Configurazione STEM
- Allineamenti preliminari: caricare il file di allineamento delle colonne, quindi aprire Valvole a colonna. Se si utilizza un crio-supporto ad ingresso laterale, aprire Cryo-shield. Avviare in modalità TEM. Il raggio dovrebbe apparire sullo schermo. In caso contrario, ridurre l'ingrandimento.
- Portare il microscopio alla messa a fuoco eucentrica premendo il pulsante sul pannello di controllo.
- Impostare la dimensione dello spot su un valore conveniente (ad esempio, 6) per visualizzare lo schermo fluorescente, direttamente o con la fotocamera integrata (a seconda del modello di microscopio).
- Impostare il microscopio in modalità STEM e verificare che la messa a fuoco utilizzi le lenti del condensatore (intensità) anziché l'obiettivo. Imposta l'attenzione eucentrica. Quindi, uscire dalla modalità di diffrazione per le regolazioni iniziali.
- Assicurarsi che il raggio non sia oscurato. Ridurre l'ingrandimento fino a quando il raggio non appare sullo schermo. Regolare lo spostamento del fascio verso il centro e aumentare l'ingrandimento in passi fino a circa 70kx mantenendo il raggio al centro.
- Inserire l'apertura del condensatore desiderata, tipicamente 50 μm. Controllare la centratura dell'apertura. Quando si ruota leggermente la manopola di messa a fuoco avanti e indietro, il punto dovrebbe espandersi e contrarsi ma rimanere in posizione, come se un piano tagliasse una clessidra verticale immaginaria. Se l'apertura non è centrata, l'illuminazione si sposterà lateralmente, come se la clessidra fosse inclinata.
- Porta il raggio a fuoco e premi Intensity List Focus (se disponibile) nella scheda Allineamenti o torna alla messa a fuoco eucentrica come in 1.2. Regolate nuovamente la posizione del fascio al centro.
- Regolare il centro di rotazione. Ruotare la rotellina di messa a fuoco al minimo o un gradino sopra, in modo che il raggio pulsi delicatamente e assicurarsi che rimanga fermo mentre la messa a fuoco si sposta su e giù.
- Selezionare i punti pivot e riunire i due punti con le regolazioni X e Y.
NOTA: se una piastra di fase è installata su uno strumento TFS, è necessario utilizzare solo la regolazione X. - Defocalizzare leggermente il fascio e regolare gli stigma del condensatore per rendere il disco rotondo. Vai su e giù attraverso la messa a fuoco per ottimizzare; Non ci dovrebbe essere alcuna tendenza ad allungarsi in una direzione o nell'altra quando si passa la messa a fuoco.
- Normalizzare le lenti. Quindi, aumentare progressivamente l'ingrandimento a circa 240 kx utilizzando lo spostamento del fascio per mantenere il punto centrato e ripetere le regolazioni del centro di rotazione e del punto di rotazione (passaggi 1,6-1,8).
- Tornare alla modalità di diffrazione. Il fascio dovrebbe apparire come un disco uniforme sullo schermo fluorescente. La lunghezza della telecamera (CL) ora controlla efficacemente la distanza ottica dal rivelatore, come nella cristallografia a raggi X. Cambialo e osserva come il disco si contrae e si ingrandisce, come se la posizione dello schermo si spostasse verso o lontano dal campione. Questo cono rappresenta l'illuminazione BF.
NOTA: Per ogni CL, c'è un allineamento di diffrazione da regolare per portare il fascio proiettato sull'asse centrale. Normalmente non è necessario perfezionarli tutti, solo uno (o pochi) da utilizzare per il rilevamento. - Attiva la modalità STEM ad alto ingrandimento.
- Se è disponibile un rilevatore BF STEM, iniziare con quello. Portate lo stage in un'area con un foro vuoto e regolate l'allineamento della diffrazione per centrare il fascio utilizzando il CL desiderato.
- Molti microscopi sono dotati solo di un rivelatore HAADF; c'è un trucco per usarlo come rilevatore BF. Per prima cosa, ritrarre il rilevatore, centrare il fascio come sopra negli allineamenti diretti, inserire un'apertura dell'obiettivo, tipicamente piccola come 20 μm, e regolare la sua posizione per circondare il fascio in modo uniforme. (Il modo più semplice per farlo è inserire un campione o una griglia di prova per vedere un alone intorno all'illuminazione). Quindi, utilizzare l'allineamento di diffrazione per spostare il fascio fuori asse e sull'area del rilevatore. Inserire e ritrarre il rilevatore mentre si guarda il raggio sullo schermo per confermare che il raggio è bloccato dal rilevatore.
- Avviare una scansione nel software del microscopio e regolare le impostazioni di luminosità e contrasto (B/C) in modo che il segnale sia vicino a 0 quando il raggio è oscurato e vicino alla saturazione quando l'illuminazione completa cade sul rilevatore. Utilizzare la visualizzazione dell'oscilloscopio per assistenza. Le impostazioni possono essere accoppiate in modi imprevisti, quindi la regolazione deve essere iterata più volte.
- Riportare il microscopio a un ingrandimento relativamente basso nel registro ad alto ingrandimento (modalità SA), senza passare alla modalità low mag (LM).
- Prendere nota della corrente dello schermo per riferimento in seguito (vedere il passaggio 3.1.1). La corrente può essere modificata con l'obiettivo della pistola e le impostazioni delle dimensioni dello spot, come in TEM, con numeri crescenti corrispondenti a una corrente ridotta.
- A questo punto, salvare un registro FEG per facilitare il ritorno ai valori standard.
NOTA: la struttura può preparare un set predefinito di registri FEG in modo che gli utenti possano iniziare da una configurazione funzionante.
2. Posizionamento del campione
- In modalità LM TEM, assicurarsi che la griglia non sia opaca. Trova un quadrato della griglia per effettuare le regolazioni iniziali. È conveniente trovare un'area vuota, un buco o un quadrato della griglia strappato. Registra le posizioni del palcoscenico per tornare a loro comodamente.
- Portare il campione ad altezza eucentrica. Esistono diversi metodi per farlo. Ad esempio, usate lo stage wobbler per inclinare la griglia mentre spostate l'altezza del campione lungo l'asse Z fino a quando l'immagine non smette di spostarsi lateralmente. In alternativa, contrassegnare alcune caratteristiche sullo schermo di visualizzazione e inclinare il palco di 30°. La feature si sposterà lateralmente. Regolate l'altezza del campione per riportarlo nella posizione originale. Aumentare l'ingrandimento o l'inclinazione dello stage per perfezionarlo. Tornare all'inclinazione di 0°.
- Tornare alla modalità STEM e inserire il rilevatore STEM. Passare alla modalità di ingrandimento (SA) più basso. Assicurarsi che un'immagine venga osservata sullo schermo del computer. Scansione rapida per evitare esposizioni non necessarie; 1 μs/pixel o meno sarebbe tipico. Si noti che l'immagine può apparire distorta sul bordo sinistro quando la scansione è eccessivamente veloce (vedere la Figura 2).
- Premere Eucentric Focus, aumentare l'ingrandimento e perfezionare la messa a fuoco durante la scansione. È conveniente utilizzare la lente di messa a fuoco fornita dal software del microscopio.
- Sintonizzare l'astigmatismo del condensatore. Questo può essere fatto più precisamente con il metodo Ronchigram. Con il fascio su un'area di campionamento sottile, mettere a fuoco il punto in cui il raggio trasmesso esplode, tra le immagini in ombra del campione su entrambi i lati. Quindi, regolare la sintonizzazione del condensatore per rendere il disco centrale rotondo. Ciò richiede una certa pratica, specialmente per i campioni criogenici.
NOTA: Un'alternativa è trovare alcune particelle d'oro (spesso usate come marcatori fiduciali per l'allineamento in tomografia). Aumentare l'ingrandimento e mettere a fuoco su e giù, regolando l'astigmatismo in modo che le particelle rimangano rotonde senza allungamento in entrambe le direzioni. - Vai alla modalità LM STEM e continua la scansione per trovare un'area interessante. Se necessario, regolare nuovamente le impostazioni B/C del rilevatore (punto 1.13.3), approssimativamente a questo punto.
3. Registrazione di un'immagine
- Stimare la dose per la registrazione. Come regola generale, mirare a 100-150 e-/Å2 per l'intero tomogramma. Controllare la tolleranza di dose per campione. La corrente in Ampere (A) divisa per la carica per elettrone rappresenta un conteggio di e-/s, che viene moltiplicato per il tempo di esposizione, T, in secondi e diviso per il campo visivo in Å2. Per comodità, esprimere la corrente in nanoampere (come letto dallo schermo), l'ampiezza del campo visivo in micrometri e il tempo di esposizione del fotogramma in secondi, con fattori adeguati:
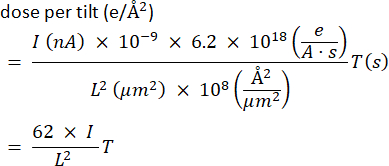
Questo numero deve essere moltiplicato per il numero di inclinazioni per ottenere l'esposizione totale nella serie. Ad esempio, con una corrente I = 0,04 nA, un campo L = 4 μm e un tempo di esposizione di 12 s, otteniamo 1,9 e-/Å 2 per inclinazione, o circa 110 e-/Å2 per una serie di 61 proiezioni. - Riportare lo stage in un foro (o ritrarre la griglia) e regolare la corrente del fascio utilizzando le dimensioni dello spot e/o le impostazioni dell'obiettivo della pistola per raggiungere la corrente dello schermo desiderata. Se la misurazione è 0, inserire un'apertura C2 maggiore per aumentare la corrente. Quindi, correggere dividendo la misura per il quadrato del rapporto dei diametri e infine tornare all'apertura più piccola.
- Perfezionare le impostazioni B/C in base al passaggio 1.13.3, quindi riportare lo stadio in un'area di interesse e ricontrollare gli allineamenti diretti e l'astigmatismo nelle condizioni di imaging.
- Acquisire un'immagine di prova.
4. Tomografia con SerialEM
- Avviare il server SerialEM sul computer microscopio (SerialEM è normalmente installato su uno diverso). Quindi, aprire SerialEM. STEM appare in SerialEM come una telecamera, con la stessa interfaccia. Assicurarsi che le comunicazioni siano eseguite in modo appropriato per l'installazione. Ad esempio, la scheda del microscopio dovrebbe visualizzare l'ingrandimento corretto. Se questo non funziona, fermati qui e risolvi i problemi.
- Trova la scheda Fotocamera e script e apri Configurazione. Assicurati che la telecamera STEM sia selezionata e per ogni modalità scegli i tempi di binning e di permanenza adatti. Assicurati che il rilevatore (o i rilevatori) appropriati appaiano nella casella "canali da acquisire" in basso a sinistra. Premi Acquisisci in basso per testare. Ancora una volta, non continuare se non succede nulla o se il raggio non si spegne. Fermati e controlla le comunicazioni.
NOTA: Le modalità sono utilizzate in modo simile alla tomografia TEM. Il binning è un artificio per la compatibilità con TEM. Il parametro importante è il numero di pixel; Diversi sistemi di microscopi utilizzano binning diversi per raggiungere lo stesso conteggio.- Visualizza e cerca dovrebbero essere scansioni veloci con relativamente pochi pixel (ad esempio, 512 x 512 pixel a 1 s).
- La messa a fuoco è generalmente una piccola area con registrazione veloce. Pertanto, in modalità a basso dosaggio, scegli Messa a fuoco per avere un ingrandimento diverso dalla modalità di registrazione, che può aiutare in termini di precisione. Lasciare invariata la dimensione dello spot.
- Per la modalità Registrazione, utilizzare i parametri scelti sopra nella stima dell'esposizione. Se disponibile, selezionate anche Messa a fuoco dinamica, che corregge la messa a fuoco riga per riga in un'immagine inclinata.
- Per comodità in seguito, scegliete il binning per l'anteprima in modo che sia identico a quello per View (vedere Miglioramento #1, passaggio 5.5). La dimensione dell'immagine (numero di pixel) non deve necessariamente essere la stessa.
- Utilizzare la modalità Prova per mantenere l'area Record centrata durante l'acquisizione. Può essere simile a Vista, ma fate attenzione che non vi siano distorsioni di scansione evidenti sul lato sinistro del fotogramma (vedere la Figura 2 per un esempio). Anche in questo caso, in modalità Low Dose, la versione di prova può avere un ingrandimento diverso rispetto alla modalità Record.
- Utilizzare immediatamente la modalità Montage per la scansione della griglia. Dovrebbe essere sufficientemente denso di pixel per trovare aree di interesse; Raccomandazione: 512 x 512 o 1024 x 1024. Assicurati che l'immagine sia buona, con una gamma dinamica decente (vista nei controlli di visualizzazione delle immagini), poiché questa modalità viene utilizzata per trovare le aree del campione.
- Salvare il file delle impostazioni in modo che queste scelte vengano mantenute come predefinite per la sessione successiva (che potrebbe verificarsi presto nel caso in cui il programma si arresti in modo anomalo).
- Testare le calibrazioni dello spostamento dell'immagine e dello spostamento dello stage. Nella modalità microsonda (o nanosonda), fare clic su Visualizza immagine, passare il mouse su di essa, tenere premuto il pulsante e trascinare diagonalmente di circa un quarto del campo visivo. Quindi, prendi un'altra vista. Le immagini dovrebbero sovrapporsi perfettamente. Ripeti trascinando di nuovo tenendo premuto il tasto Maiusc (per imporre un movimento di scena). Le immagini dovrebbero sovrapporsi bene, anche se forse in modo meno preciso.
- Vai alla modalità LM e prova lo spostamento di scena. Se questi test falliscono, arrestare e risolvere i problemi. Il resto non funzionerà.
- Crea una mappa LM di montaggio dell'intera griglia. Questo può essere fatto anche in TEM.
- Vai alla modalità STEM con ingrandimento più basso in cui l'immagine non è bloccata da aperture, in genere intorno a 185x.
- Fare clic sul menu Navigatore > Apri. Quindi, fare clic sul menu Navigatore > Opzioni di navigazione. > deselezionare Converti mappe in byte.
- Seleziona il menu Navigatore > Montaggio e griglie > Imposta montaggio completo. Si aprirà un menu. Le opzioni da controllare sono: Sposta stage invece di spostare l'immagine, Crea mappa da ogni montaggio se Navigatore è aperto e Usa mappatura montaggio, non Registra parametri. La griglia dell'immagine dovrebbe essere approssimativamente 6 x 6 o 7 x 7. Cerca l'area totale da registrare. Se sono necessarie troppe immagini, l'ingrandimento potrebbe non essere letto correttamente dal microscopio (Figura 3). Fermati e controlla.
- Per avviare l'acquisizione del montaggio, premere Start su Controlli di montaggio o Montaggio in Fotocamera e script. Si aprirà un altro menu per scegliere i parametri del file: mrc, store as integers e, in un'altra finestra di dialogo, scegliere il nome del file per l'archiviazione. Assicurati di memorizzare i dati nell'area dati adatta e non nelle impostazioni utente di SerialEM. Si consiglia di non memorizzare dati di grandi dimensioni sul disco C in cui risiede il sistema.
- Al termine dell'acquisizione, il montaggio apparirà nella finestra principale (Figura 4). Ora è possibile navigare intorno alla griglia usando l'indicatore e i punti sul Navigatore, proprio come in TEM.
- Ricontrollare la correzione dell'astigmatismo. Di solito, è sufficiente scansionare rapidamente una piccola area e regolare insieme alla messa a fuoco in modo che le caratteristiche puntiformi come le nanoparticelle d'oro rimangano completamente rotonde mentre passano dentro e fuori fuoco (come su un SEM) ad alto ingrandimento. Più conservativamente, si possono ripetere gli allineamenti diretti dei passaggi 1,5-1,8 prima di fissare l'astigmatismo.
- Allineare l'imaging ad alto ingrandimento al montaggio LM.
- Vai a una posizione sul montaggio LM con alcune caratteristiche facilmente riconoscibili. Aggiungete un punto, fate clic sulla finestra Navigatore > Aggiungi punti e fate clic sulla feature. Spingere di nuovo (smettere di aggiungere) per disattivare.
- Scatta un'immagine Vista o Prova con l'ingrandimento HM (High Mag) più basso e trova l'elemento indicato. Posizionate l'indicatore (croce verde) e, con il punto corrispondente evidenziato nella finestra Navigatore, nel menu Navigatore > Maiusc su marcatore..., nella finestra di dialogo visualizzata, scegliete Tutte le mappe e Salva spostamento, come illustrato nella Figura 5.
- Prova spostando lo stage in altre posizioni e verificando che l'immagine HM sia centrata nelle stesse posizioni selezionate nel montaggio LM. Se la feature non è visibile nell'immagine HM, lo spostamento tra le modalità LM e HM potrebbe essere troppo ampio. In questo caso, esegui un montaggio poligonale su un'area più ampia. Fare clic sulla finestra Navigatore > Aggiungi poligono, scegliere alcuni punti, quindi fare nuovamente clic su Aggiungi poligono per chiudere. Fate clic sul menu Navigatore > Montaggio e griglie > Imposta montaggio poligono e Controlli montaggio > Avvia. Quindi, trova i punti corrispondenti come sopra e fai clic su Maiusc su marcatore ....
- Impostare la modalità a basso dosaggio.
- Aprire la scheda Controllo dose bassa e selezionare la casella Modalità dose bassa .
- Seleziona Aggiornamento continuo e vai su Visualizza. Scegli l'ingrandimento del microscopio per questa modalità, in genere il più basso o vicino al più basso. Scegli ciascuna delle modalità successive e imposta l'ingrandimento appropriato. La registrazione deve essere impostata per ottenere la dimensione dei pixel desiderata e l'ingrandimento della messa a fuoco deve essere abbastanza alto da risolvere chiaramente i marcatori fiduciali o altre funzioni ad alto contrasto per la messa a fuoco. Quindi, deseleziona immediatamente Aggiornamento continuo.
NOTA: la modalità di prova può essere impostata in modo simile a Record, nel qual caso l'area di tracciamento dovrà essere spostata dall'area di registrazione per ridurre al minimo l'esposizione, o a basso ingrandimento, che comprende gran parte dell'ambiente circostante. - Scatta un'immagine Visualizza e imposta la posizione di prova utilizzando Definisci posizione dell'area: Prova.
NOTA: Raccomandazione #1: Dato il cambiamento nell'area e nel tempo campionati, l'esposizione aggiuntiva per uno studio a basso mago potrebbe essere minima. Finché la barra della griglia non entra nel campo visivo, questo metodo di tracciamento è in genere più affidabile. Raccomandazione #2: È anche possibile aggiornare la dimensione dello spot per regolare la dose per diverse modalità, come Focus. Per la stabilità nelle impostazioni dell'ottica del microscopio, si consiglia di non farlo, ma piuttosto di lasciare la dimensione dello spot come appropriato per Record e quindi di regolare il tempo di esposizione, se necessario, per le modalità di scansione più veloci. - Fare clic sulla scheda Controllo a bassa dose > Vai a: Rec., quindi fare clic sul menu Navigatore > Apri stati di imaging > Aggiungi stato corrente. Dagli un nome in modo che possa essere richiamato facilmente (ad esempio, μP STEM per microsonda STEM). È possibile definire diversi stati per compiti diversi.
- Sposta il palco in una posizione di interesse per la tomografia (maggiori informazioni sull'uso del Navigatore di seguito).
- Aggiungere un punto nel Navigatore facendo clic sulla finestra Navigatore > Aggiungi punti.
- Perfezionate l'altezza eucentrica selezionando Attività > Eucentricità > Approssimativa. Finestra Navigatore > Aggiornamento Z.
- Impostare le aree per Focus e Trial. Innanzitutto, scatta un'immagine View. Quindi, nella scheda Controllo a basso dosaggio , in Definisci posizione dell'area, selezionare Focus o Trial. Regolate le posizioni per le due aree lungo l'asse di inclinazione. Lo stato attivo non deve sovrapporsi all'area Record.
NOTA: tenere presente che c'è un certo over-shoot, in particolare sul bordo sinistro, quindi nessuno dei due deve essere impostato immediatamente adiacente all'area di registrazione. L'entità dell'over-shoot può essere valutata su un'area di test eseguendo ripetutamente la scansione all'ingrandimento del record e quindi riducendo l'ingrandimento per ottenere una visione più ampia.
- Per una buona misura (opzionale con esperienza), inclinare lo stage a +60° e poi a -60°, ogni volta che si scatta un'immagine Vista o Anteprima per assicurarsi che la barra della griglia non entri nel campo di visualizzazione Area record mostrato in verde.
- Impostare la serie di inclinazione.
- Aprire un nuovo file per salvare i dati facendo clic su File > Apri nuovo e scegliere un nome file. Selezionare il menu Serie inclinazione > Impostazione serie inclinazione (Tilt Series) (Figura 6). Risoluzione dei problemi: se il pulsante Apri nuovo è grigio, verificare che la serie di inclinazione precedente sia stata terminata. In caso contrario, terminare facendo clic su Inclina serie > Termina.
- Impostate gli angoli di inclinazione estremi nelle caselle Inclina fino a e Fine a , in genere rispettivamente -60° e +60°, nonché l'incremento, in genere 2°.
- Per la modalità simmetrica della dose (consigliata - assicurarsi che la modalità Low Dose sia ancora attiva), selezionare Run series in two directions from ___. Il grezzo è normalmente 0, ma può essere utilizzato per compensare lamelle FIB pre-inclinate (ad esempio, a 20°).
- Per semplicità, selezionare Mantieni costante il tempo di esposizione. La modifica richiede alcune rielaborazioni del calcolo dell'esposizione e può anche interferire con le ricostruzioni algebriche (ad esempio, ART/SART/SIRT), che confrontano le intensità proiettate con i dati originali (vedi passo 7.1).
- Seleziona Salta messa a fuoco automatica. Un piccolo angolo di semiconvergenza, come 1 mrad, implica una profondità di campo così lunga che potrebbe non essere necessario mettere a fuoco. Come test, mettere a fuoco a 0°, inclinare a 60° o -60° e premere Record. Mantenere la concentrazione è principalmente una questione di precisa altezza eucentrica. Per una maggiore convergenza, potrebbe essere necessario concentrarsi durante la serie. In questo caso, prendere una vista e nella scheda Controllo a basso dosaggio , definire la posizione dell'area e dello stato attivo e regolare l'area di messa a fuoco.
- Azioni iniziali e finali: se la regolazione eucentrica dell'altezza è stata eseguita accuratamente, non è necessario ripeterla. Deselezionare Chiudi valvole a colonna alla fine della serie, a meno che non vi siano buone ragioni per non farlo.
- Per tenere traccia dei parametri di controllo, ci sono molte opzioni. Per la raccolta automatica dei dati, la priorità assoluta è evitare che il programma si arresti per l'input dell'utente. Utilizzare le impostazioni consigliate: ripetere Record se la percentuale di campo perso è superiore a 5. Quindi, traccia dopo e seleziona Allinea con anteprima prima di ottenere un nuovo riferimento traccia e Ottieni nuovo riferimento traccia se l'allineamento record differisce del 2%. Per la raccolta dati assistita, applicare parametri più rigorosi per evitare il rischio di perdere ore di tempo dello strumento.
- Quando sei pronto per iniziare, premi GO nella parte inferiore della finestra. Alla fine, selezionare Inclina serie > Termina.
5. Acquisizione batch in più punti utilizzando il Navigatore (Miglioramento #1)
NOTA: Il protocollo è rudimentale perché a) è identico a TEM e b) stanno diventando disponibili nuovi strumenti che probabilmente renderanno obsoleta una descrizione completa.
- Caricare il montaggio completo della griglia nella finestra principale.
- Scegli una serie di aree che sembrano promettenti. Fare clic su Aggiungi punti e aggiungere ai centri dei quadrati della griglia selezionati. Fare nuovamente clic su Aggiungi punti per disattivare la modalità.
- Con lo stato di imaging a basso dosaggio attivato, selezionare la finestra Navigatore > selezionare Acquisisci e Nuovo file all'elemento per ciascuno dei quadrati selezionati. Per il primo, si aprirà una finestra di dialogo per le proprietà del file (Figura 7) e quindi il nome del file. Scegliete le immagini montate, quindi nella finestra di dialogo Imposta montaggio (Figura 8), selezionate Usa parametri vista in modalità Dose bassa e create una griglia sufficientemente grande da coprire il quadrato (circa 100 x 100 μm).
- Fare clic sul menu Navigatore > Acquisisci agli elementi e selezionare i parametri per la mappatura. Ancora più importante, in Azioni primarie, selezionare Crea mappa navigatore, quindi Eucentricità approssimativa ed Eucentricità fine e premere Vai (Figura 9). Per una griglia a 200 mesh, si otterrà una mappa di circa l'intero quadrato (Figura 10)
- Preparati per le selezioni della posizione del tomogramma. Per aprire un nuovo file, fare clic su File > Apri nuovo. Assegnagli un nome file (ad esempio, AnchorMaps.mrc).
- Con la modalità Low Dose attiva, accedere a Camera & Script > Setup e assicurarsi che View e Preview si trovino nello stesso binning (altrimenti non possono essere salvati nello stesso file).
- Visita i punti nelle mappe quadrate facendo clic sulla finestra Navigatore > Vai a XYZ.
- Scegli una posizione per il primo tomogramma con il pennarello. Scatta un'immagine di anteprima e perfeziona la posizione trascinandola con il tasto destro del mouse. Quindi, selezionare la finestra Navigatore > Nuova mappa e fare clic su Sì alla finestra di dialogo da salvare. Quindi, scatta un'immagine Visualizza della stessa area e salva nuovamente come nuova mappa. Assicurati che Visualizza mappa sia evidenziata e seleziona Per lo stato di ancoraggio nella finestra Navigatore in alto a destra.
- Scegli la seconda posizione, prendi di nuovo un'anteprima e perfeziona la posizione come sopra, e questa volta premi Mappa di ancoraggio nella finestra Navigatore. In questo modo sia l'anteprima che la vista verranno salvate come nuove mappe nel navigatore.
- Ripetere il passaggio 5.5.4 per tutte le posizioni desiderate. Spostarsi da un quadrato della griglia a un altro selezionando il punto e premendo Vai a XYZ nella finestra Navigatore.
- Per la messa a fuoco di riferimento, scegliete un'area libera della griglia e mettete a fuoco con attenzione, manualmente o con la messa a fuoco automatica. Premere Reset defocus sul pannello del microscopio. Creare uno script in SerialEM facendo clic su Script > Modifica > Modifica 15 (o un altro numero libero) e immettere due righe: NomeScript SetDefocus e SetDefocus 0.
- Nella finestra Navigatore, evidenziare la prima posizione (Anteprima o Visualizza). Premere Maiusc-T, quindi evidenziare l'ultima posizione e premere nuovamente Maiusc-T. Si aprirà la finestra di dialogo Proprietà file. Scegli immagini e parametri a fotogramma singolo da salvare, incluso il nome del file. Modifica il nome del file ma lascia l'etichetta dell'elemento alla fine.
NOTA: i nomi dei file che terminano con numeri verranno aggiornati automaticamente per le serie di inclinazione successive. È conveniente utilizzare l'opzione Menu Navigatore > Navigazione. Opzioni > Usa etichette di elementi nei nomi dei file. - Successivamente, il menu Configurazione serie Tilt si aprirà automaticamente. Riempite come prima (Figura 6), ma non scegliete Perfeziona eucentricità. I punti di anteprima nel Navigatore saranno ora contrassegnati come TS. Si può tornare a questo menu utilizzando il pulsante nella finestra Navigatore sopra l'elenco degli elementi.
- Scegliere il menu Navigatore > Acquisisci agli elementi. Questa volta, selezionare i parametri per Dati finali. Selezionare Azione primaria: acquisire serie inclinazione e scegliere Gestisci dewar/vuoto, Riallinea a elemento, Eucentricità fine ed Esegui script prima dell'azione, con Script da eseguire: SetDefocus. Selezionare le opzioni generali: nessuna finestra di messaggio quando si verifica un errore e chiudere le valvole della colonna alla fine, quindi premere GO (Figura 11). SerialEM visiterà ora tutte le mappe di ancoraggio e registrerà una serie di inclinazioni in ogni posizione (Figura 12).
6. Registrazione della mappa CLEM (Miglioramento #2)
- Se non è già stato fatto, caricare il montaggio completo della griglia nella finestra principale, quindi creare una nuova finestra facendo clic su Finestra > Nuova finestra e assegnarle un nome. Si noti che il montaggio completo della griglia viene visualizzato in Registrazione 1.
- Importare l'immagine della mappa CLEM facendo clic sul menu Navigatore > Importa mappa. Dovrebbe andare in Registrazione 2. Può anche essere assegnata una nuova finestra come sopra.
- Esaminate la mappa CLEM per determinare la rotazione relativa rispetto al montaggio completo della griglia. È più facile farlo su una mappa in cui appaiono le barre della griglia. Si noti che la mappa può anche essere capovolta. Allinealo approssimativamente usando il menu Navigatore > Ruota mappa, con Capovolgi, se necessario.
NOTA: il passaggio seguente può anche ospitare un capovolgimento, ma farlo in anticipo rende molto più semplice quanto segue. Questo passaggio può essere ripetuto fino a quando l'allineamento sembra soddisfacente, ma non è necessario essere precisi. - Introdurre punti di registrazione per un allineamento preciso. Posizionare un numero di punti corrispondenti su una e poi sull'altra finestra. Per ciascuno di questi punti, selezionare Punto di registrazione nella parte superiore della finestra del navigatore. I punti corrispondenti saranno etichettati con un indice crescente, seguito da R1 o R2 rispettivamente per il montaggio della griglia o la mappa importata.
NOTA: l'uso delle griglie del Finder rende questo passaggio molto semplice. In caso contrario, scegliete feature solide come l'indicatore centrale o gli angoli dei quadrati della griglia. In linea di principio, tre punti sono sufficienti per un allineamento grezzo, ma più punti aiutano a compensare piccole discrepanze nelle costruzioni di montaggio. - Se si desiderano diversi canali di una mappa CLEM, ad esempio diversi colori fluorescenti o fluorescenza senza uno sfondo a campo chiaro, importarli come nel passaggio 6.2 precedente e modificare le registrazioni in 2. In questo modo, erediteranno i punti di registrazione dalla prima mappa.
- Imporre la registrazione tramite il menu Navigatore > le voci Trasforma. I punti di registrazione corrispondenti appariranno ora su tutte le mappe, che saranno elencate nella registrazione 1. Per allineare visivamente, fare clic sul menu Navigatore > Ruota mappa. Si noti che quando una mappa viene caricata dalla finestra Navigatore, non viene ruotata automaticamente a meno che non sia selezionata la casella Ruota quando carico è selezionata. Può sempre essere ruotato dalla finestra Navigatore.
- Ora dovrebbe essere possibile posizionare il marcatore o un punto sulla mappa di fluorescenza e spostare lo stadio nella posizione corrispondente sulla griglia (Figura 13).
7.3D ricostruzione
- Ciò comporta gli stessi passaggi di base della tomografia TEM: allineamento della serie di inclinazione seguito tipicamente da retroproiezione. Sono disponibili diverse piattaforme software e i dati possono essere trattati in modo simile ai dati TEM, con l'eccezione che la correzione CTF dovrebbe essere saltata.
- Applicare normalizzazioni dell'intensità per migliorare il contributo delle proiezioni ad alta inclinazione o per bilanciare un cambiamento di illuminazione durante la serie, con l'avvertenza che le informazioni quantitative sono influenzate. IMOD26 funziona bene, ad esempio, mentre AreTomo27 è molto utile per l'allineamento senza fiducia; dove sono presenti particelle fiduciali, ClusterAlign28 può seguirle come cluster piuttosto che singoli punti; ciò è particolarmente utile per i dati STEM in cui le macchie possono essere perse o nascoste da funzionalità ad alto contrasto.
- Segui la ricostruzione con la deconvoluzione 3D29 per migliorare il contrasto e il denoising. Una volta ricostruiti, rappresentano i dati del volume STEM con uno qualsiasi dei metodi standard, come le ortosezioni o la segmentazione isosuperficiale. In particolare, la distribuzione dell'intensità è unipolare, senza inversioni di contrasto o frange di Fourier che appaiono nel TEM convenzionale a contrasto di fase. Un mezzo interessante di rappresentazione dei tomogrammi STEM, in generale, è invertire il campo luminoso (Figura 14). L'effetto è quello di "occhi a raggi X", vedendo gli oggetti densi di interesse attraverso la cella chiarificata30.
Risultati
Un montaggio completo della griglia preparato in STEM mostra le aree con celle di interesse (Figura 4). Si noti che l'immagine è in campo scuro, quindi i fori vuoti appaiono scuri. Le celle appaiono parzialmente luminose, dove gli elettroni sono dispersi verso il rivelatore HAADF. Nelle parti più spesse, tipicamente al centro o vicino alle barre della griglia, il contrasto diventa di nuovo scuro. Ciò è dovuto allo scattering multiplo, per cui gli elettroni raggiungono angoli non catturati dal rivelatore. In pratica, queste aree saranno troppo spesse per la tomografia.
La fase successiva comprende mappe di ingrandimento intermedio (Figura 10). Queste sono spesso chiamate mappe di montaggio medio o mappe quadrate, riferendosi ai quadrati della griglia piuttosto che alla forma. Anche con la modalità STEM microprobe più bassa, saranno acquisiti anche come immagini di montaggio. Facendo clic sull'elemento nella finestra Navigatore l'immagine della mappa viene caricata nella finestra principale. All'interno di quest'area vengono scelti punti specifici per l'acquisizione del tomogramma. Utilizzare la modalità Anteprima per individuare l'area in corrispondenza dell'ingrandimento della registrazione. Tieni presente che potrebbe esserci uno spostamento laterale tra Anteprima e Registra, a seconda delle velocità di scansione. Qui può essere registrata una singola serie di inclinazione. I mitocondri, ad esempio, possono essere spesso identificati nell'immagine Anteprima e registrazione (Figura 12).
Per la tomografia batch, lo stadio viaggerà intorno alla griglia e potrebbe non tornare esattamente nella stessa posizione. Le mappe di ancoraggio vengono utilizzate per garantire che le aree di registrazione desiderate vengano identificate in modo affidabile abbinando l'immagine di anteprima a una vista di ingrandimento inferiore, anche se il movimento dello stage si è spostato. PyEM23,24 offre un'alternativa più veloce che è certamente consigliata, ma non fa ancora parte dell'installazione standard.
Nell'approccio CLEM, una mappa di fluorescenza può essere utilizzata per identificare le aree di interesse per la tomografia. La fluorescenza viene acquisita esternamente e deve essere registrata sul montaggio completo della griglia. È utile avere le barre della griglia e i fori visibili, in modo che un composito di fluorescenza + campo chiaro unito possa essere preparato prima dell'importazione, ad esempio nel software GIMP (https://www.gimp.org) o ImageJ31 (fare attenzione che ImageJ inverta l'asse verticale). Quando la gamma dinamica della fluorescenza è ampia, può essere difficile creare una tale fusione senza saturazione. In questo caso, le due mappe possono essere importate separatamente e quindi registrate insieme come descritto (Figura 13). In questo modo, un punto sulla mappa di fluorescenza nella finestra del navigatore, seguito da "Vai a XY", porta il palco nella posizione corrispondente sulla griglia montata nel TEM. Fate attenzione che piccole incongruenze nella creazione del montaggio (o della mappa di fluorescenza, se anch'essa è un montaggio) porteranno a piccoli spostamenti; Pertanto, per una precisione ottimale, la fluorescenza deve essere nuovamente registrata specificamente nella mappa quadrata. Spesso è sufficiente fare questa registrazione ad occhio, sulla base di fori nella pellicola di supporto o caratteristiche condivise con l'immagine luminosa a campo luminoso.
La ricostruzione della serie di inclinazione produce un volume 3D (Figura 14). Questo esempio ha una dimensione in pixel di 2,042 nm, che si traduce in un campo visivo di ~4 μm. I depositi di fosfato di calcio (punte di freccia arancione) si distinguono, a causa del numero atomico più elevato rispetto all'ambiente circostante. I microtubuli (punte di freccia rossa) possono essere tracciati in tutto il campo visivo. Inoltre, le membrane mitocondriali interne ed esterne (punte di freccia verdi) possono essere viste chiaramente. L'actina può essere osservata come fasci o come singoli filamenti. Per ottenere una risoluzione più isotropa, la ricostruzione può essere elaborata mediante deconvoluzione.
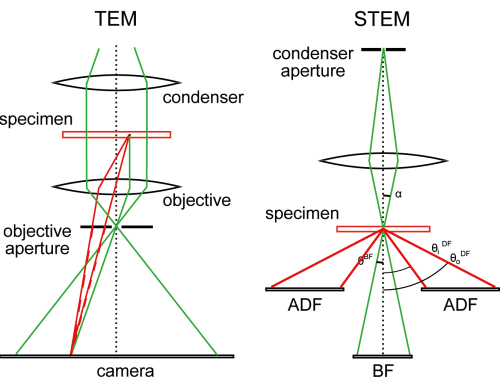
Figura 1: Confronto tra TEM e STEM. In TEM, il campo visivo è illuminato da un fascio quasi parallelo e l'obiettivo forma un'immagine ingrandita sulla telecamera. Per il crio-TEM, l'apertura dell'obiettivo viene aperta per far passare le onde elettroniche diffuse (linea rossa tratteggiata), che, con la sfocatura, producono contrasto di fase per interferenza con le onde non diffuse (verdi). STEM, d'altra parte, raster un fascio focalizzato attraverso il campione e raccoglie gli elettroni diffusi in modo pixel per pixel. Più rivelatori possono raccogliere elettroni sparsi a diverse angolazioni. Questa cifra è stata modificata da30. Fare clic qui per visualizzare una versione ingrandita di questa figura.

Figura 2: Esempio di distorsione dell'immagine sul lato sinistro a causa dell'elevata velocità di scansione. Si noti la distorsione contrassegnata da punte di freccia gialle, così come i fori di forma ovale distorti nel Quantifoil. Fare clic qui per visualizzare una versione ingrandita di questa figura.
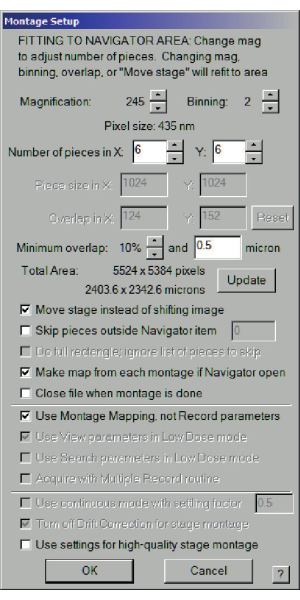
Figura 3: Finestra di dialogo di impostazione del montaggio per l'intero montaggio della griglia. Scegli l'ingrandimento e il numero di pezzi in X e Y in modo che l'area totale raggiunga circa 2.000 μm per ottenere una mappa per la maggior parte della griglia. Scegliete anche Sposta stage invece di spostare l'immagine e Usa mappatura montaggio, non Registra parametri. Fare clic qui per visualizzare una versione ingrandita di questa figura.

Figura 4: GridMap a basso ingrandimento registrato in modalità STEM. Le aree spezzate appaiono completamente nere nell'immagine in campo scuro. Le aree grigio scuro sono probabilmente troppo spesse e scarsamente vetrificate. Le buone aree candidate per la tomografia sono bianche o grigio chiaro in questa scansione. Barra di scala = 100 μm. Fare clic qui per visualizzare una versione ingrandita di questa figura.
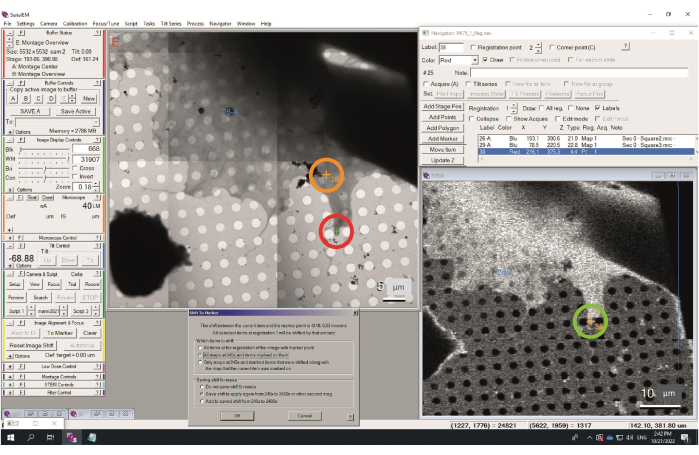
Figura 5: Passaggio al processo del marcatore. Un oggetto riconoscibile è contrassegnato nella mappa a bassa risoluzione da Aggiungi punti (38 in questa immagine). Si noti lo spostamento tra la mappa a bassa risoluzione (cerchio verde) e la mappa a media risoluzione (cerchio arancione). Quindi, l'oggetto viene contrassegnato dal marcatore (croce verde, cerchio rosso) e viene avviata la finestra di dialogo Sposta su marcatore. L'opzione scelta sposta tutte le mappe a 245x della stessa quantità. Fare clic qui per visualizzare una versione ingrandita di questa figura.
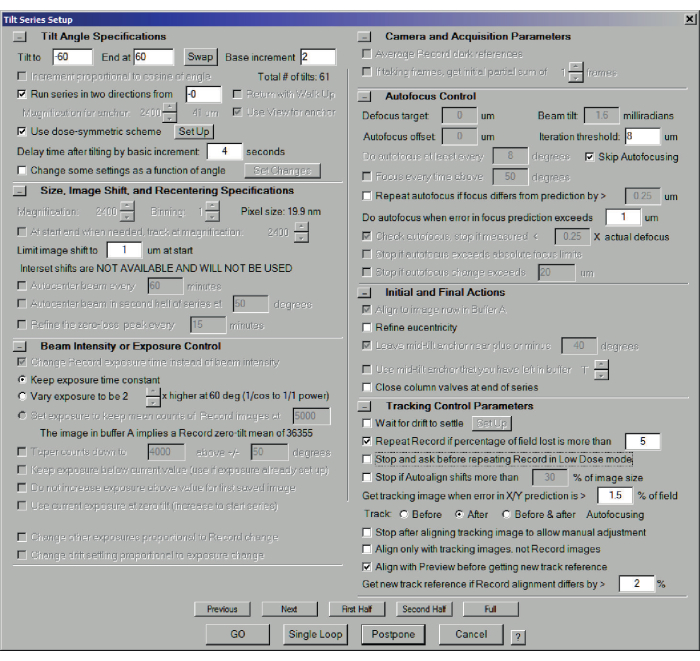
Figura 6: Finestra di dialogo di configurazione della serie Tilt per una serie di inclinazione STEM. Lo schema simmetrico di dose viene utilizzato da -60° a +60° in incrementi di 2°. La messa a fuoco automatica viene saltata a causa dell'elevata profondità di messa a fuoco quando si utilizza un angolo di convergenza basso. Non c'è bisogno di affinare l'eucentrismo come è stato fatto prima. Fare clic qui per visualizzare una versione ingrandita di questa figura.
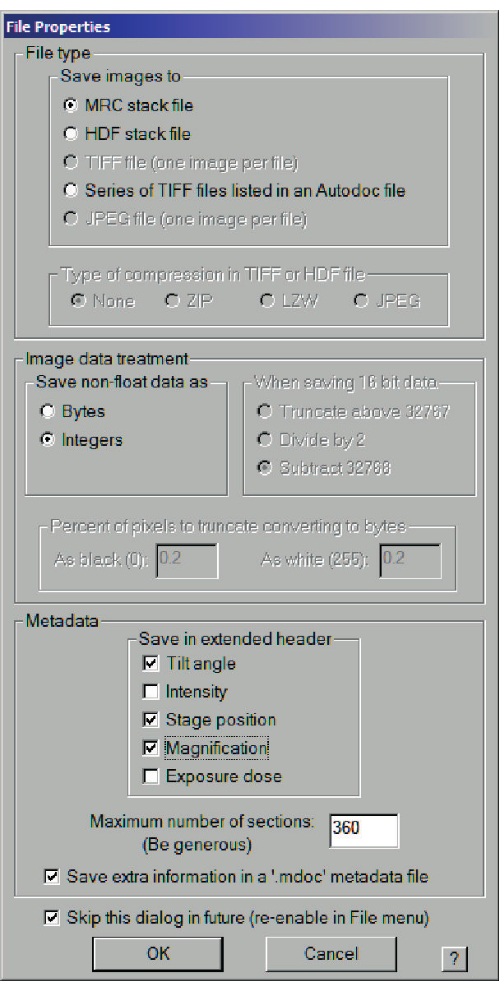
Figura 7: Finestra di dialogo delle proprietà dei file per mappe a media risoluzione. Salvare come file dello stack MRC, scegliere numeri interi e salvare informazioni aggiuntive in un file di metadati con estensione mdoc. Fare clic qui per visualizzare una versione ingrandita di questa figura.
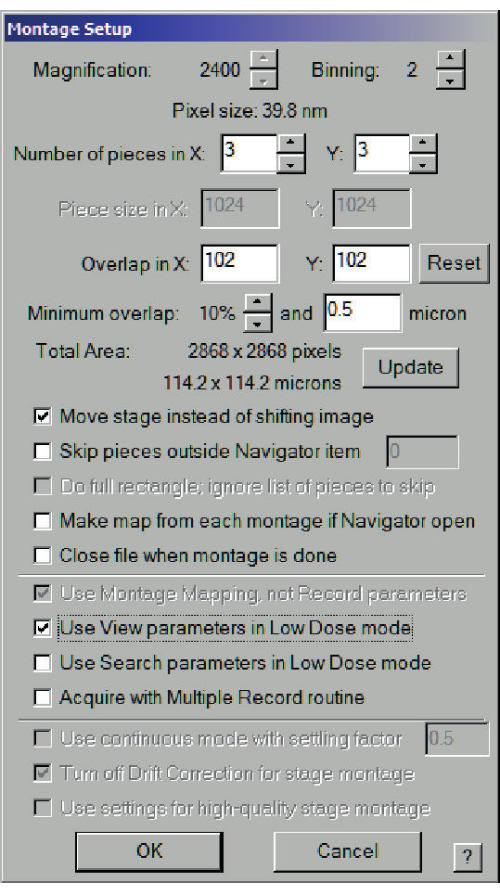
Figura 8: Finestra di dialogo di impostazione del montaggio per il salvataggio di mappe a media risoluzione. Per un quadrato su una griglia a 200 maglie, larga 90 μm, è consigliabile un'area totale di almeno 90 μm x 90 μm. Scegliete Sposta fase invece di spostare l'immagine e Usa i parametri di visualizzazione in modalità Dose bassa. Fare clic qui per visualizzare una versione ingrandita di questa figura.
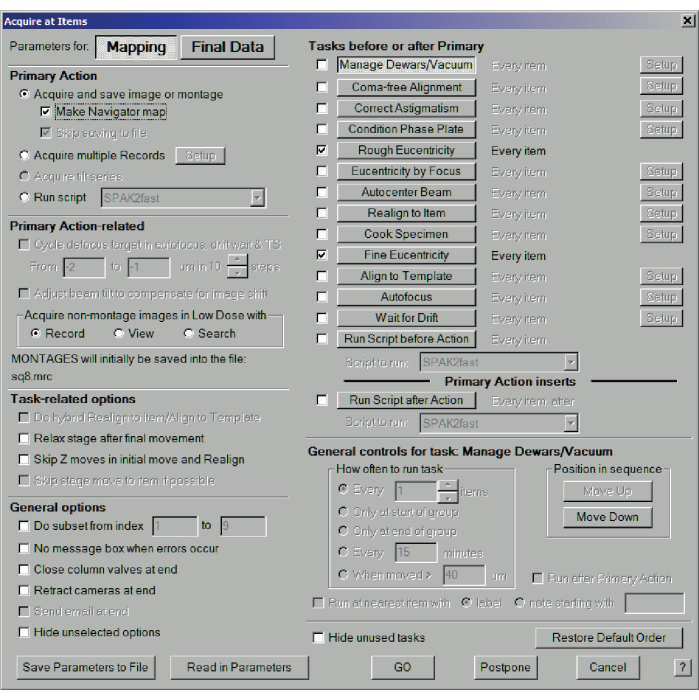
Figura 9: Registrazione della mappa a media risoluzione. Acquisisci nella finestra di dialogo Elementi per la registrazione di mappe a media risoluzione. Scegli Mappatura, Acquisisci e salva immagine o montaggio e seleziona Crea mappa navigatore. Nelle Attività prima o dopo la Primaria, scegli Eucentricità approssimativa e fine. In caso di assenza di assistenza, scegliere facoltativamente Nessuna finestra di messaggio quando si verifica un errore e Chiudi valvole colonna alla fine. Fare clic qui per visualizzare una versione ingrandita di questa figura.

Figura 10: Immagine della mappa a media risoluzione. Mappa a media risoluzione (Square Map) registrata in modalità STEM da un montaggio di 3 x 3. Le due Anchor Map a media risoluzione per la raccolta dei dati sono indicate dai quadrati blu. Barra di scala = 10 μm. Fare clic qui per visualizzare una versione ingrandita di questa figura.
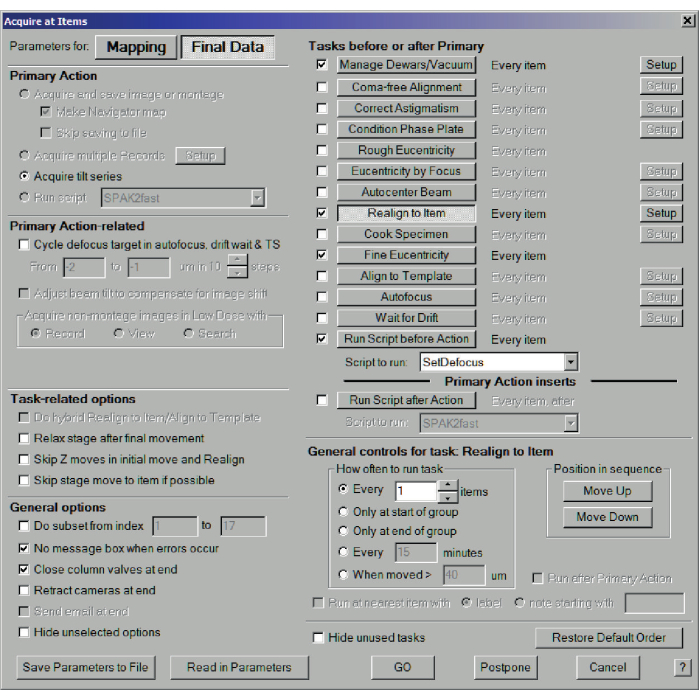
Figura 11: Dati della serie di inclinazione del batch. Finestra di dialogo Acquisisci elementi per la raccolta di serie di inclinazione batch. Per la raccolta dei dati delle serie di inclinazione, scegliere Dati finali e Acquisisci serie di inclinazione. In Attività prima o dopo Primario, selezionare Gestisci dewar/Vuoto (scegliere le impostazioni appropriate nel menu Imposta), Riallinea a Elemento, Eucentricità fine ed Esegui script prima dell'azione. Nelle opzioni Generali, selezionare Nessuna finestra di messaggio quando si verifica un errore e Chiudi valvole di colonna alla fine (vedere 5.14). Fare clic qui per visualizzare una versione ingrandita di questa figura.
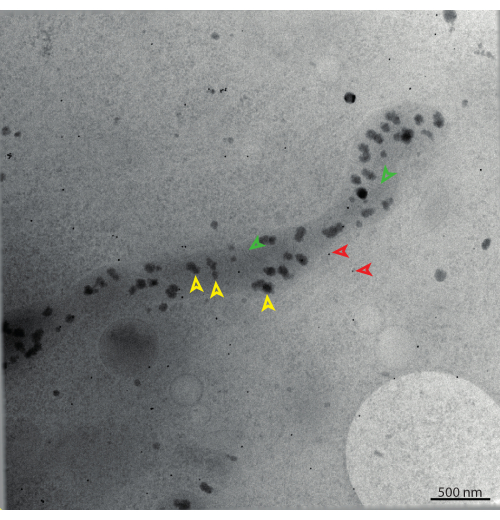
Figura 12: Inclinazione 0° di una serie di inclinazioni STEM di un mitocondrio. Le punte di freccia arancioni indicano depositi di fosfato di calcio (granuli di matrice), punte di freccia verdi a cristae e punte di freccia rosse a marcatori fiduciali d'oro da 15 nm. Barra della scala = 500 nm. Fare clic qui per visualizzare una versione ingrandita di questa figura.
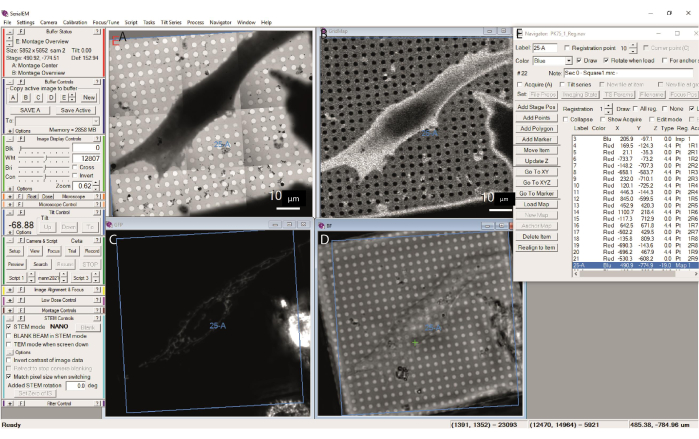
Figura 13: Mappe crio-CLEM registrate. (A) La mappa STEM a media risoluzione (mappa quadrata, 26-A) è registrata sulla (B) mappa STEM a bassa risoluzione, (C) mappa crio-FM del canale BF e (D) mappa crio-FM del canale GFP. I nove punti di registrazione (etichette 4-21) sono indicati nel (E) Navigator. Fare clic qui per visualizzare una versione ingrandita di questa figura.

Figura 14: rendering del volume. Rendering volumetrico di sezioni spesse (A) 60 nm e (B) 40 nm attraverso un tomogramma filtrato simile a SIGT (30 cicli) a diverse profondità. La dimensione dei pixel è di 2,048 nm, il che si traduce in un campo visivo di circa 4 μm. Si noti la densità invertita rispetto alla Figura 12, il che significa che le caratteristiche ad alta intensità sono luminose. Le punte di freccia arancioni, bianche, rosse, verdi e azzurre indicano rispettivamente depositi di fosfato di calcio, ribosomi, microtubuli, membrana mitocondriale interna ed esterna e filamenti di actina. Barra della scala = 500 nm. Fare clic qui per visualizzare una versione ingrandita di questa figura.
Discussione
Il protocollo dovrebbe aiutare i microscopisti delle scienze della vita che sono interessati a ottenere una visione 3D degli organelli intracellulari in regioni della cellula che non sono accessibili alla tomografia TEM convenzionale. Lo stesso protocollo può essere utilizzato anche per la tomografia STEM di sezioni plastiche, con vincoli rilassati sull'esposizione. Il protocollo dovrebbe essere considerato come un punto di partenza piuttosto che un insieme di regole rigide. In effetti, il potere di STEM è la sua flessibilità; Non esiste un modo giusto per gestirlo.
Sottolineiamo che STEM, di per sé, si riferisce solo alla sonda scansionata e non definisce la formazione dell'immagine. Il contrasto dipende principalmente dalla configurazione dei rilevatori. I metodi più semplici impiegano rivelatori con simmetria assiale, un disco centrato sull'asse ottico o un anello che lo circonda. In generale, quando l'illuminazione colpisce direttamente il rivelatore, registriamo un'immagine BF in cui il campione (di diffusione elettronica) appare scuro; al contrario, quando il rivelatore raccoglie solo elettroni sparsi, registriamo un'immagine DF in cui il campione denso appare luminoso. Quando è disponibile l'hardware adatto, SerialEM può acquisire e registrare entrambi questi segnali contemporaneamente. Sono disponibili configurazioni ancora più sofisticate, che vanno dai rilevatori con più segmenti alle telecamere completamente pixelate. L'imaging a contrasto di fase può essere ottenuto, ad esempio, valutando la differenza tra gli elementi rivelatori fuori asse32,33. La raccolta dell'intero modello di scattering 2D (diffrazione) per pixel definisce il metodo noto come 4D STEM34, che consente la ricostruzione di più contrasti di immagine dagli stessi dati originali. Lo STEM quadridimensionale consente la ptychography elettronica, che fornisce un altro mezzo per ottenere il contrasto di fase35.
Ci siamo concentrati sulla particolare modalità STEM che riteniamo più utile per lo studio di organelli e cellule intatte o sezioni cellulari spesse micron. Ciò comporta in particolare l'uso dell'imaging BF con un piccolo angolo di semiconvergenza nell'illuminazione e un angolo di raccolta relativamente ampio sul rivelatore. La piccola convergenza fornisce un'ampia profondità di campo in modo che l'intero campione rimanga a fuoco per tutta la serie di inclinazione19. In pratica, con un buon stadio al microscopio, può anche eliminare la necessità di regolazione della messa a fuoco durante l'acquisizione. Il prezzo è un compromesso nella risoluzione, come descritto nella sezione 3 del protocollo. Abbiamo suggerito immagini 4k con una sonda di ~2 nm, che con una spaziatura dei pixel di 1 nm raggiunge un campo visivo di 4 μm. Tuttavia, il lettore è fortemente incoraggiato a sperimentare. La seconda considerazione è sul lato del rilevatore. Quando il disco di illuminazione riempie il rilevatore BF in asse, il contrasto di fase viene soppresso e domina il contrasto di dispersione (ampiezza); Questa condizione è stata chiamata contrasto di campo luminoso incoerente36. La domanda è da quale frazione riempire e la risposta dipende dal campione. Un campione molto sottile mostrerà il miglior contrasto con il rilevatore completamente riempito (cioè, l'angolo di raccolta corrisponde a quello dell'illuminazione), ma un campione più spesso disperderà tutta l'intensità, lasciando un segnale rumoroso con scarso contrasto. Una regola empirica utile è che più spesso è il campione, maggiore dovrebbe essere l'angolo di taglio esterno del rilevatore BF21. Le dimensioni e la posizione del rivelatore sono ovviamente fisse, quindi la dimensione del disco di diffrazione viene regolata utilizzando la lunghezza della telecamera, come descritto nella sezione 1. Se le impostazioni dell'amplificatore del rilevatore sono tali che il segnale riempie la gamma dinamica ma non satura sotto l'illuminazione diretta (1.12.3), la lunghezza della telecamera può essere aumentata fino a raggiungere un'intensità e un contrasto del segnale ragionevoli. Ancora una volta, il lettore è incoraggiato a sperimentare. L'arte, per così dire, è negli angoli.
Un altro parametro, non discusso nel protocollo, è la tensione di accelerazione del microscopio. L'interazione degli elettroni illuminanti con il campione sarà più forte a una tensione inferiore. A parità di tutto il resto, possiamo aspettarci un contrasto maggiore a una tensione inferiore. D'altra parte, è l'inizio della diffusione multipla all'interno del campione che limita lo spessore del campione utilizzabile, quindi una tensione più elevata consente l'uso di campioni più spessi. Questi effetti sono piuttosto sottili, tuttavia. Le nostre esperienze fino ad oggi con accelerazioni a 200 kV e 300 kV sono simili.
Considerando ciò che ci si può aspettare da STEM in termini di risoluzione, questo dipende ancora una volta dal campione e dalla configurazione del rivelatore. Utilizzando un approccio di analisi a singola particella, gli ioni metallici sulla ferritina potrebbero essere localizzati da STEM in campo oscuro anulare con una precisione di pochi angstrom37. Più recentemente, è stata raggiunta una risoluzione sub-nanometrica per campioni di virus e proteine utilizzando immagini ottenute mediante contrasto di fase differenziale integrato (iDPC) STEM32 e ptychography35. Per oggetti unici in campioni cellulari spessi, e con i metodi descritti qui, una risoluzione così elevata non è realistica. La risoluzione ottimale è il diametro della sonda, che si riferisce all'angolo di semiconvergenza come descritto. Altri fattori degraderanno la risoluzione, in particolare un campionamento di pixel grossolani per raggiungere un ampio campo visivo, disallineamenti nella serie di inclinazione e diffusione del fascio della sonda in trasmissione. Le immagini si confrontano bene con la tomografia a sezione plastica. Con la configurazione qui descritta, ad esempio, dovrebbe essere facile vedere il nucleo cavo dei microtubuli, ma non i singoli protofilamenti.
Per riassumere, i metodi STEM e l'hardware sono entrambi in fase di sviluppo. Possiamo aspettarci che le innovazioni nell'imaging avranno un impatto anche sulla tomografia, portando STEM in domini che non sono stati accessibili da altre tecniche. Ci aspettiamo che una convergenza con l'imaging a fluorescenza criogenica correlativa basata su moderni metodi ottici a super-risoluzione sarà particolarmente fruttuosa. La scala degli organelli, 100-1.000 nm, è un obiettivo ideale per questi metodi emergenti.
Divulgazioni
Gli autori non dichiarano conflitti di interesse.
Riconoscimenti
Siamo estremamente grati per il supporto continuo e costante da parte dell'autore e manutentore del pacchetto software SerialEM, David Mastronade, e Günther Resch. P.K. è stato finanziato dall'Austrian Science Fund (FWF) attraverso una Schrödinger Fellowship J4449-B. Ai fini dell'accesso aperto, gli autori hanno applicato una licenza di copyright pubblico CC-BY a qualsiasi versione del manoscritto accettato dall'autore derivante da questa presentazione. M.E. e S.G.W. riconoscono il finanziamento della Israel Science Foundation, sovvenzione n. 1696/18, e del progetto di gemellaggio Horizon 2020 dell'Unione Europea, ImpaCT (grant no.857203). M.E. riconosce il finanziamento del CER nel progetto CryoSTEM (sovvenzione n. 101055413). M.E. è il titolare della cattedra Sam e Ayala Zacks Professorial e capo del Centro Irving e Cherna Moskowitz per l'imaging nano e bio-nano. Il laboratorio di M.E. ha beneficiato della generosità storica della famiglia Harold Perlman. Riconosciamo anche il finanziamento da parte dell'Unione europea. Le opinioni e le opinioni espresse sono tuttavia esclusivamente quelle dell'autore o degli autori e non riflettono necessariamente quelle dell'Unione europea o dell'Agenzia esecutiva del Consiglio europeo della ricerca. Né l'Unione europea né l'autorità che concede l'aiuto possono essere ritenute responsabili per loro.
Materiali
| Name | Company | Catalog Number | Comments |
| SerialEM | University of Colorado | Veriosn 4.0 | SerialEM is a free software platform for microscope control and data acquisition. |
| STEM-capable transmission electron microscope | The protocol was written based on experience with several microscopes of Thermo Fisher Scientific: Titan Krios, Talos Arctica, and Tecnai T20-F. In principle it should be applicable to other manufacturers as well. |
Riferimenti
- Elbaum, M. Expanding horizons of cryo-tomography to larger volumes. Current Opinion in Microbiology. 43, 155-161 (2018).
- Schertel, A., et al. Cryo FIB-SEM: Volume imaging of cellular ultrastructure in native frozen specimens. Journal of Structural Biology. 184 (2), 355-360 (2013).
- Do, M., Isaacson, S. A., McDermott, G., Le Gros, M. A., Larabell, C. A. Imaging and characterizing cells using tomography. Archives of Biochemistry and Biophysics. 581, 111-121 (2015).
- Groen, J., Conesa, J. J., Valcárcel, R., Pereiro, E. The cellular landscape by cryo soft X-ray tomography. Biophysical Reviews. 11 (4), 611-619 (2019).
- Kapishnikov, S., et al. Oriented nucleation of hemozoin at the digestive vacuole membrane in Plasmodium falciparum. Proceedings of the National Academy of Sciences. 109 (28), 11188-11193 (2012).
- Kapishnikov, S., et al. Unraveling heme detoxification in the malaria parasite by in situ correlative X-ray fluorescence microscopy and soft X-ray tomography. Scientific Reports. 7 (1), (2017).
- Sviben, S., et al. A vacuole-like compartment concentrates a disordered calcium phase in a key coccolithophorid alga. Nature Communications. 7, 11228 (2016).
- Wan, W., Briggs, J. A. Cryo-electron tomography and subtomogram averaging. Methods in Enzymology. , 329-367 (2016).
- Zhang, P. Advances in cryo-electron tomography and subtomogram averaging and classification. Current Opinion in Structural Biology. 58, 249-258 (2019).
- Bäuerlein, F. J. B., Baumeister, W. Towards visual proteomics at high resolution. Journal of Molecular Biology. 433 (20), 167187 (2021).
- Elbaum, M., Seifer, S., Houben, L., Wolf, S. G., Rez, P. Toward compositional contrast by cryo-STEM. Accounts of Chemical Research. 54 (19), 3621-3631 (2021).
- Elbaum, M., Wolf, S. G., Houben, L. Cryo-scanning transmission electron tomography of biological cells. MRS Bulletin. 41 (7), 542-548 (2016).
- Wolf, S. G., Houben, L., Elbaum, M. Cryo-scanning transmission electron tomography of vitrified cells. Nature Methods. 11 (4), 423-428 (2014).
- Houben, L., Weissman, H., Wolf, S. G., Rybtchinski, B. A mechanism of ferritin crystallization revealed by cryo-STEM tomography. Nature. 579 (7800), 540-543 (2020).
- Li, Y., Huang, W., Li, Y., Chiu, W., Cui, Y. Opportunities for cryogenic electron microscopy in materials science and nanoscience. ACS Nano. 14 (8), 9263-9276 (2020).
- Hohmann-Marriott, M. F., et al. Nanoscale 3D cellular imaging by axial scanning transmission electron tomography. Nature Methods. 6 (10), 729-731 (2009).
- Engel, A. Scanning transmission electron microscopy: biological applications. Advances in Imaging and Electron Physics. 159, 357-386 (2009).
- Wolf, S. G., Elbaum, M. CryoSTEM tomography in biology. Methods in Cell Biology. 152, 197-215 (2019).
- Biskupek, J., Leschner, J., Walther, P., Kaiser, U. Optimization of STEM tomography acquisition-A comparison of convergent beam and parallel beam STEM tomography. Ultramicroscopy. 110 (9), 1231-1237 (2010).
- Trepout, S., Messaoudi, C., Perrot, S., Bastin, P., Marco, S. Scanning transmission electron microscopy through-focal tilt-series on biological specimens. Micron. 77, 9-15 (2015).
- Rez, P., Larsen, T., Elbaum, M. Exploring the theoretical basis and limitations of cryo-STEM tomography for thick biological specimens. Journal of Structural Biology. 196 (3), 466-478 (2016).
- Mastronarde, D. N. Automated electron microscope tomography using robust prediction of specimen movements. Journal of Structural Biology. 152 (1), 36-51 (2005).
- Schorb, M., Haberbosch, I., Hagen, W. J. H., Schwab, Y., Mastronarde, D. N. Software tools for automated transmission electron microscopy. Nature Methods. 16 (6), 471-477 (2019).
- Sheng, Y., Morris, K., Radecke, J., Zhang, P. Cryo-electron tomography remote data collection and subtomogram averaging. Journal of Visualized Experiments. (185), e63923 (2022).
- Weis, F., Hagen, W. J. H., Schorb, M., Mattei, S. Strategies for optimization of cryogenic electron tomography data acquisition. Journal of Visualized Experiments. (169), e62383 (2021).
- O'Toole, E., vander Heide, P., McIntosh, J. R., Mastronarde, D. Large-scale electron tomography of cells using SerialEM and IMOD. Cellular Imaging: Electron Tomography and Related Techniques. , 95-116 (2018).
- Zheng, S., et al. AreTOMO: An integrated software package for automated marker-free, motion-corrected cryo-electron tomographic alignment and reconstruction. Journal of Structural Biology. (6), 100068 (2022).
- Seifer, S., Elbaum, M. ClusterAlign: A fiducial tracking and tilt series alignment tool for thick sample tomography. Biological Imaging. 2, 7 (2022).
- Waugh, B., et al. Three-dimensional deconvolution processing for STEM cryotomography. Proceedings of the National Academy of Sciences. 117 (44), 27374-27380 (2020).
- Kirchenbuechler, D., et al. Cryo-STEM tomography of intact vitrified fibroblasts. AIMS Biophysics. 2 (3), 259-273 (2015).
- Schneider, C. A., Rasband, W. S., Eliceiri, K. W. NIH Image to ImageJ: 25 years of image analysis. Nature Methods. 9 (7), 671-675 (2012).
- Lazić, I., et al. Single-particle cryo-EM structures from iDPC-STEM at near-atomic resolution. Nature Methods. 19 (9), 1126-1136 (2022).
- Seifer, S., Houben, L., Elbaum, M. Flexible STEM with simultaneous phase and depth contrast. Microscopy and Microanalysis. , 1-12 (2021).
- Ophus, C. Four-dimensional scanning transmission electron microscopy (4D-STEM): from scanning nanodiffraction to ptychography and beyond. Microscopy and Microanalysis. 25 (3), 563-582 (2019).
- Zhou, L., et al. Low-dose phase retrieval of biological specimens using cryo-electron ptychography. Nature Communications. 11 (1), 2773 (2020).
- Nellist, P. D. The principles of STEM imaging. Scanning Transmission Electron Microscopy: Imaging and Analysis. , 91-115 (2011).
- Elad, N., Bellapadrona, G., Houben, L., Sagi, I., Elbaum, M. Detection of isolated protein-bound metal ions by single-particle cryo-STEM. Proceedings of the National Academy of Sciences. 114 (42), 11139-11144 (2017).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneEsplora altri articoli
This article has been published
Video Coming Soon