このコンテンツを視聴するには、JoVE 購読が必要です。 サインイン又は無料トライアルを申し込む。
Method Article
プロービングDNA安定同位体(DNA - SIP)
要約
DNA安定同位体プロービングには、特定の基質を利用することのできる微生物の活発なコミュニティを識別し、特徴付けるために栽培に依存しない方法です。重い同位体に富む基板の同化は、微生物バイオマスへの標識原子の取り込みにつながる。密度勾配超遠心分離は、下流の分子解析のための標識DNAを取得します。
要約
DNA安定同位体(DNA - SIP)プローブは、細胞バイオマスに特定の炭素基質と栄養素を吸収し、アクティブな微生物を識別するための強力な手法です。このように、この栽培に依存しない手法は、陸域と水域環境の広い範囲に生息する多様なコミュニティへの代謝機能を割り当てるための重要な方法論となっています。安定同位体標識化合物による環境試料のインキュベーションに続いて、抽出した核酸は、異なる密度の核酸を分離する密度勾配超遠心分離とその後の勾配分画に供される。塩化セシウムからのDNAの精製は、その後の分子特性(例えば指紋、マイクロアレイ、クローンライブラリ、メタゲノミクス)のための標識と非標識DNAを取得します。このJoveのビデオプロトコルは密度勾配超遠心分離、勾配分画とラベルされたDNAの回復のためのプロトコルの視覚的なステップバイステップの説明を提供します。プロトコルはまた、サンプルのSIPデータとハイライトの重要なヒントが含まれており、成功したDNA - SIPの分析を確実にするために考慮しなければならないことをご承知おき下さい。
プロトコル
1。試薬の調製
DNA - SIPは、実際の手順の事前に準備すべき試薬を使用する必要があります。各試薬を調製するための方向性は、このセクションにリストされており、以前のSIPプロトコル1から変更されています。
- SIPの勾配を調製するための塩化セシウム(CsCl密度)ソリューション-徐々に500mLの最終容量を蒸留し、脱イオン水に塩化セシウムの603.0グラム(のddH 2 O)を溶解して7.163 M塩化セシウム溶液を調製。 500mlを超えないように注意してください!攪拌しながら少しソリューションを温めては、塩化セシウムのすべてを溶かすのに役立ちます。密封されたアリコートの最終的なソリューション注し。私たちの研究室では、一般的なストレージの練習は、ブチルゴム栓とクリンプシールされています125 - mLの血清バイアル、100 mLのアリコートを準備することです。密閉されたアリコートを室温(20℃)で無期限に保存することができます。シールは、蒸発し、塩化セシウム"クラスト"の形成を防ぐのに役立ちます。三重100μLアリコートを比較検討することによって、または慎重にCsCl密度のソリューションのために校正されたデジタル屈折計(例えばライヘルトAR200)を用いて溶液の密度を決定する。一度成功したキャリブレーション、ライヒェルトAR200は一貫性があり、数年のための正確な測定値を提供します。室温(20℃)で、この溶液の最終濃度は、通常、1.88〜1.89グラムml -1の範囲である。密度はわずかに新しい株が準備されるたびに異なります。
- 無菌のddH 2 Oの水250 mLで塩化セシウム250 gを結合-エチジウムブロマイド(反応ステップ)でグラデーションを調製するための塩化セシウムのソリューション。は1.1で説明されているブチルゴムのシール付きの圧着シールされている別の血清バイアルに分注し、このソリューション。
- 勾配バッファ - 1 Mトリス - 塩酸、3.75グラムのKCl、0.5 M EDTA 1 mlの水400mlに50 mlの結合。 KClを溶解し、500mlにのddH 2 Oを加える。ろ過滅菌し、オートクレーブ。最終的な解決には、0.1Mトリス、0.1MのKClおよび1mMのEDTAです。
- ポリエチレングリコール(PEG)ソリューション- 500mLの(30%PEG、1.6 M NaCl)の合計量に無菌のddH 2 Oの水にポリエチレングリコール6000とNaClの46.8 gの150gを溶解してPEG溶液を調製。オートクレーブ。
注:このソリューションでは、オートクレーブで二相に分離する。これが発生した場合は、このソリューションが適切に混合することができるように、オートクレーブボトル内の攪拌棒が含まれています。 - TEバッファーには、 - 1 Mトリス-塩酸(pH8.0)と0.5 M EDTA(のオートクレーブ原液を使用して、無菌のddH 2 Oの水に10mMトリス- HCl(pH8.0)と1mMのEDTA(pH8.0)を溶液を調製しますpHは8.0)。滅菌とオートクレーブフィルタリング。
- 70%エタノール-滅菌のddH 2 Oを150mlの水と高純度のエタノール350 mlを組み合わせる。
2。サンプルのインキュベーションとDNA抽出
DNA - SIPのインキュベーションのために、サンプルは一般的に重い同位体炭素(13 C)基質とインキュベートされています。潜伏期間と条件(例えば、栄養補給、水分、光)がインキュベートされ、サンプルの種類や基板の性質によって異なります。 DNA - SIPの実験は成功し、単一の炭素化合物2,3、マルチ炭素化合物4,5,6、および標識窒素7,8または酸素9を使用してさまざまな方法を使って行われている。しかし、15 N -または18 O -標識化合物を使用する欠点は、炭素原子の相対DNAとRNAの少ない窒素と酸素原子の存在が主な原因標識核酸の減少物理的に分離する、です。
DNA - SIPの実験のための重要なコントロールは、ネイティブ(例えば12 C)基板との間で確立同一のインキュベーションです。このインキュベーションには、核酸のいずれかの明白なラベリングは超遠心分離または分離10に寄与するDNAのG + C含量の密度の違いのアーティファクトではないことを確認するために、後続の比較を示します。それは"光"と"重い"DNAとの比較のために凍結サンプルの材料を保つことも重要である、とSIPのインキュベーションを通じて背景の人口の変化を評価するために、無基質の制御を含めて価値がある。
- 標識基質(図1)を含む小宇宙で環境サンプルをインキュベートする。我々の経験では、我々は、サンプルのグラム当たりの13 Cの炭素の500から500マイクロモルの間の最小の組み込みが、そのような土壌のサンプル1として高いバイオマスを含むサンプルに適していることを発見した。土壌よりもバイオマスを含む水生サンプルについては、法人の100から100マイクロモルリットルあたり13 Cの炭素が検出可能な重い同位体署名1を生成する可能性があります。炭素の修正量は、バイオマスと同化するための補助栄養素を追加するための要件に組み込ま炭素の割合は、すべての分析される試料の特性に依存し、対象とOになります興味のrganisms。サンプルのインキュベーションのガイドラインの一つのセットは、すべての試料に対しては適用されません。重要なのは、SIPのインキュベーションのために使用される基質の濃度は、理想的には可能な限り近づけ、通常はその場に遭遇する濃度にしてください。実験的なバイアスは、濃縮培養条件10の帰結かもしれない。
- 安定同位体標識基質とサンプルのインキュベーション後、厳格な抽出プロトコール(PCRまたは小さなインサートクローニング用)または高分子量のクローニングのための信頼できる酵素溶解(例えば大規模挿入メタゲノム)を使用して、小宇宙からDNAを抽出する。 RNAの共抽出は一般的にそのRNAを得るだけでなく、DNAのプロトコルを使用することができる、分析には影響しません。抽出されたDNAの超遠心分離は、〜50 kbのペア1より短い断片せん断しません。
- CsCl密度勾配超遠心管のセットアップの前に抽出されたDNAを定量化する。抽出プロトコールはDNAのみを(例えばカラムベースのキット)が得られる場合分光光度計(例えば、光度計2000)を使用してDNAを定量化する。また、アガロースゲル電気泳動を用いて定量化する。
3。超遠心用グラデーションソリューションの準備
この手順では、チューブを遠心するためにDNAを追加する必要があります。正確なプロトコルが異なりますし、メーカーの指示に依存しますので、チューブとローターには複数のタイプがあります。それはそうと、私たちは光と重いDNAの可能な最大の分離を確保するために縦の穴ローターを使用することをお勧めします。我々は、5.1ミリリットルQuickSealのポリアロマーチューブを保持するための16ウェルとBeckman - Coulter社VTI 65.2ローターを使用し、プロトコルは、これらの条件のための手順と考慮事項を提供します。
- 超遠心チューブにDNA5μgの - ステップ2.3で決定されたDNAの濃度を用いて、0.5μgのを提供するために必要とされる抽出されたDNAの必要量を計算する。
- グラデーションのバッファー(ステップ1.3参照)と滅菌済み使い捨て15 mlのチューブに〜6mlの総体積に7.163 M塩化セシウムの4.8 mlを - 抽出されたDNA(5μgの0.5)を組み合わせる。塩化セシウム溶液の密度は(ステップ1.1参照)と同じモル濃度でも変化することに注意してください。次式は、適切な混合比を生成するために必要とされるグラデーションのバッファ/ DNA混合物の体積を決定するために使用することができます。
勾配のバッファとDNA溶液の体積(ml)=(塩化セシウムのストック溶液の密度-所望の最終濃度)のCsClストック溶液のX量は、x 1.52を追加
4.80ミリリットルで塩化セシウムストック溶液の量を指定します。所望の最終濃度は1.725グラムml -1のはずです。ストック溶液の密度は、ステップ1.1で決定した。
CsCl及びグラデーションバッファ/ DNAの相対量が5.1ミリリットルを超えての合計量になることにも注意してください。超遠心チューブ(5.1 ml以上大きい)の最大ボリュームの容量を超えての準備のボリュームは完全に管を埋めるのに十分な解決策があることを保証します。 - 反転10倍で混ぜる。 DNAはCsClで室温で安定です。
4。反応ステップのコントロールのグラデーションを作成(オプション)
流しますがDNAとの複合体は、UV光の下でそれが見えるようにすることインターカレート色素であるため、彼らは前のサンプルチューブ(例えば、図1)の分画に勾配形成の直接の視覚的確認を提供するため、EtBrを含む制御勾配が便利です。流しますと12 C - DNAと13 C - DNA(または14 N - DNAと15 N - DNA)の両方の混合物を含むコントロールチューブを含めると、超遠心分離の完了時にチューブ内のバンドの形成を即座に視覚化できます。超遠心分離または不適切なプログラムの実行条件の間に破裂したチューブが失敗した勾配の形成につながることができるので、これは重要です。 DNAに結合した、反応ステップは、DNAの密度を低下させ、結果として、異なるプロトコルが勾配を準備するために続いている。他の核酸の汚れが代わりに流します11の使用することができるが、プロトコルは、他の蛍光団で最適化が必要になることに注意してください。
- 完全に安定同位体で標識1およびラベルなしのいずれかのコントロールのグラデーションは、ゲノムDNA中の二巻が必要です。我々は一般的に13 C -または弊社のコントロールのような12 C -メタンの存在下で培養した唯一の炭素源、またはMethylococcus capsulatus株のバスとして、13 Cまたは12 C -グルコースを含む培地で培養Sinorhizobium melilotiのいずれかを使用します。
- 12 C - DNAと13使い捨て15 mlのスクリューキャップチューブの1.00 mlの最終体積にグラデーションのバッファを持つC - DNAの両方の5〜10μgの量を組み合わせる。
- 同じチューブに固体塩化セシウム1.00gのを追加。転倒混和します。
- 110μlを加え10 mgのml -1の反応ステップのソリューションとステップ4.2で使用したのと同じスクリューキャップチューブに1グラムml -1の塩化セシウムのストック溶液4.3 mlの。溶液の最終濃度は、そのオリジナルの塩化セシウム原液の近似を務める。
- EtBrを含む追加の"ブランク"のコントロールソリューションは、カウンターバランス、ステップ4.4で作成したソリューションをする必要があります。グラデーションのバッファの1.00 mLを、塩化セシウム1.00gの、10mgのml -1の反応ステップのソリューションとは別15ミリリットルのスクリューキャップ付き遠心管と反転して混合で1グラムml -1の塩化セシウムのストック溶液4.3 mlの110μlを組み合わせる。
5。超遠心分離
- 電球とパスツールピペットを使用して、慎重にステップ3.2(または反応ステップのコントロールのグラデーションを作成した場合4.4ステップ)で調製した勾配のソリューションを超遠心管を埋める。慎重にパスツールピペットを用いてチューブにソリューションを追加します。細かい永久的なマーカーとチューブの肩にチューブにラベルを付けます。注意:チューブは、チューブの首の付け根に正確に入力されていることを確認してください。十分に満たされたチューブは、超遠心分離中に破裂する可能性があります。
- 必要なチューブのすべての試料溶液で満たされている場合、各チューブの正確な質量を記録する。ペアチューブと00から10ミリグラム以内にそれらのバランスをとる。彼らはバランスになるまで分散のために、できるだけチューブのネックのベースに近いようなソリューションのレベルを維持し、ほぼ一致したペアを見つけ、解決策の微量を追加または削除。チューブの重量を量るために、我々はバランスのためのチューブホルダーとして半分にカットされて反転15 mlのスクリューキャップ付きチューブを使用することに注意してください。
- 製造元の指示に従って"チューブトッパー"を使用してチューブをシール。
- 管はそれを反転し、適度な圧力を適用することによって適切に密封されていることを確認してください。彼らはまだ00から10ミリグラム以内のシール後にバランスされていることを確認し、再度チューブを秤量する。
- 井戸を清潔にし、ゴミやほこりのないことを保証するために慎重にもそれぞれローターをチェックして、超遠心分離中に穿刺チューブをかもしれない。
- 互いに反対側のバランスの取れたペアでローターにチューブを挿入します。超遠心分離プロセスはマーカーのラベルが破損または消去される可能性があるため、各サンプルの回転子位置を記録します。慎重に、製造者によって示さローター井戸をシール。
- 超遠心機にローターをロードする。超遠心機のドアを閉じ、真空を適用する。 VTI 65.2ロ ーターを使用して、44100回転(〜177000 XG AV)、20 36〜40時間° C、および超遠心時の温度に回転速度を設定する場合。真空、最大加速度、そして(減速によって中断されない勾配を確保する)ブレーキをオフにする]を選択します。ブレーキをオフにすると、実行時に追加の1〜2時間を追加することに注意してください。また、短い実行時間は、十分なバンドの分解能を達成できないことがあるので注意してください。彼らは異なる核酸のバンドの高い解像度につながるとして、長い超遠心分離法の実行は、推奨されています。
- すぐに超遠心分離の手続の完了時に、慎重にローターを取り外します。任意の傾斜を避けたり、ローターのバンピング、穏やかにチューブ内に勾配を乱すことを避けるためにローターからチューブを取り外します。まれな状況では、チューブは、実行時のバーストになります。その場合、他のチューブに勾配が正しく形成されなかった可能性があります。コントロールのグラデーションが含まれていた場合、勾配の形成を確認するために、UV光の下で慎重にこのチューブを確認してください。勾配は、コントロールのチューブで適切に形成されていない場合、それは5つの手順をすべて繰り返すことが最善です。反応ステップ制御のチューブと、そのブランクコントロールが暗所に保存されていると、六月を限度として再利用できることに注意してください。バーストのチューブが除去されると、製造元の指示に従って慎重にローターをきれいにするように注意してください。ローターウェルに傷を避けるためにローターの井戸を清掃するために金属ブラシや研磨剤入り洗剤は使用しないでください!ローター特有のブラシとクリーニング液をベックマンから購入することができます。
6。勾配分画
分別と針の抽出:現在、超遠心チューブからDNAを回収するために使用される2つの方法があります。このプロトコルは、分画の技法を用いてDNAを抽出するプロセスを説明します。ほとんどのSIPの実験のために、標識したDNAが反応ステップで可視化することはできず、代わりに複数のサンプルチューブから同等の光と重い分画を比較することによって検出される必要があるためです。シリンジポンプは、非常に超遠心管から等しい密度勾配分画を取得することをお勧めします。我々は、BSPモデル注入ポンプ(ブレーントリーサイエンティフィック社製)を使用してください。低流量蠕動ポンプまたはHPLCポンプを使用してもよい。
- 無菌のddH 2 Oは、濃い青色を提供するのに十分なブロモフェノールブルー色素を含有する滅菌60 mlの注射器を埋める。シリンジポンプのローディングアームに注射器を置きます。"針といくつかのddH 2 Oまで、ポンプの電源を入れるが針の最後まで来ている。こののddH 2 Oの供給で、気泡は負の分画プロセスに影響を与えることに注意してください。23ゲージ1を取り付けたポンプチューブを取り付けます
- クランプスタンドに超遠心分離管のいずれかを修正。クランプが十分に変位してからチューブを防止するためにタイトですがチューブを貫通されたときに、チューブに圧力が塩化セシウム溶液の放出を引き起こすようではないことを確認してください。ピアース新鮮な23ゲージ1"針を使用してチューブの継ぎ目に沿ってチューブの最下部。最良の結果を得るために、ピアス、制御された迅速、と自信たっぷりな態度でチューブ。これは、よく行うことは非常に困難であるが、前に数回の練習をこれは最初のサンプルチューブに試行されます。
- 各サンプルについて、サンプル数と分数を(;光に重い1-12)を示すラベルを12滅菌1.5 mlのマイクロチューブを用意する。継ぎ目に沿って、アッパーチューブの肩上のチューブの上部ピアス、ポンプチューブ(ステップ6.1)に接続された針を使用した。マイクロ遠心チューブを使用して勾配溶液を収集する。として迅速かつ制御された方法でチューブはピアス、チューブの底のために行った。事前に練習し、チューブを通過するのと指への強制的な針を防ぐために制御引っ張る動きを使用することは非常に注意してください! 12分(425μlを分-1)で12 × 425μlを分画が得られる以前に校正されたポンプの速度を使用してください。
- 適切な勾配の形成を確認するために、または1つの勾配からの画分の密度を確認するために化学天秤、デジタル屈折計(推奨例ライヘルトAR200)を使用してください。このテスト用のサンプルの〜50μlを使用する必要があります。私たちはしばしば、分別のためのコントロールとして機能し、密度測定のためにこれを使用するために1チューブで純粋培養のDNAを(のような反応ステップの制御の勾配を準備するための説明)が含まれます。密度が〜1.725グラムml -1の中央値は密度で、〜1.690〜1.760グラムml -1の範囲で変動することを期待しています。
7。 DNA沈殿
- 第一沈殿のためのキャリアとしての線形ポリアクリルアミドの20μgを添加することにより、すべての分画からDNAを沈殿させる。転倒混和します。 2 PEG溶液の量(ステップ1を参照)と反転混和する。 (注)降水量(例えばグリコーゲンまたは線形ポリアクリルアミド)のためのキャリアは、勾配の派閥からのDNAの定量的な回復に重要であることが、グリコーゲンは、このプロトコルのための降水量の担体として使用されている場合は注意が必要です。グリコーゲンの準備は簡単にSIPの勾配分画12の解釈を混乱させることができる細菌の核酸や汚れで汚染されることが示されている。
- 沈殿物にDNAを可能にするために室温で2時間チューブを残す。必要に応じて、チューブを室温で一晩放置することができます。
- ローターでの一貫性のあるチューブの方向のための外側を向くチューブのバックで30分間13,000 gで遠心する。慎重に吸引し、上清を捨てる。ペレットは、表示されるはずですが、この段階で参照することは非常に困難です。ペレットを可視化を支援するために明るい光源(例:デスクランプ)の下で働く。
- 70%エタノール500μlでペレットを洗浄します。 10分間13,000 gで遠心する。慎重に吸引し、上清を捨てる。ペレットは、通常、このステップのためのより多く表示されますが、より簡単に管の壁から解離される。
- ペレットを室温で15分間乾燥することができます。
- TE緩衝液50μlに各ペレットを一時停止する(ステップ1.5参照)。標準的なラボのプロトコールにしたがってアガロースゲルに各画分の5μlを実行します。
8。分数のキャラクタリゼーション
SIPのインキュベーションの成功を評価するために勾配分画を特徴付けるために使用されるメソッドは、機器のラボと可用性によって異なります。 16S rRNA遺伝子を標的とするためのフィンガープリント方式を使用すると、共通のアプローチとそのような端末の制限断片長多型(T - RFLP)や変性剤濃度勾配ゲル電気泳動などの方法であること(DGGE)(図1)に適しています。プロトコル上記に続いて、光DNAを分画9-11(〜1.705〜1.720グラムml -1)を、画分5-8(〜1.720〜1.735グラムml -1の中で関連していることが重いのDNA指紋と関連付けられることを期待)。安定同位体インキュベートした試料の分画5-8、ではなく、ネイティブ基板インキュベートしたコントロールに関連付けられた一意のフィンガープリントは、特定の標識基質の代謝に特定の生物を結ぶ強力な証拠を提供しています。不十分な標識DNAはいくつかのアプリケーション(ハイブリダイゼーション、メタゲノム)のために残っている場合は、複数の変位増幅がより大量13-15を生成するために使用されることがありますが、これは増幅されたDNA 14,16にキメラを導入することができます。
9。結果
典型的なDNA - SIPの結果は、超遠心分離によって形成された勾配の標識および非標識DNAの分離のデモンストレーションを行います。理想的には、標識物質からの高分子量の遺伝物質の完全な分解能(例えば13 C、15 N)が達成されます。解像度は、反応ステップの制御管のバンドの形成を観察することによって視覚的に目撃することができます。個々の勾配分画に含まれる取得されたゲノムDNAの濃度はまた、適切な勾配の形成を確認するために使用されることがあります。
このプロトコルのために、我々は2つの純粋培養(図2)から核酸を使用して実行勾配超遠心分離の代表的な結果が含まれています。ここに含まれ画した勾配はSから抽出したゲノムDNAを用いて調製したmeliloti(ATCC 1021)、および13 C -標識M. capsulatus strに 。バス。続いて遠心分離、分画とDNA回収、ラベル付けと異なる密度(図2A)と、それぞれの勾配分画に分離したゲノムDNAを非標識。非標識DNAを分画9-10に高濃度で検出されるのに対し、重同位体標識されたDNAは、フラクション4-5に観察することができます。各分画からDNAを変性剤濃度勾配ゲル電気泳動17と勾配(図2B)に含まれる2つの生物に対応する離散的なバンドパターンを生成したPCR増幅産物で特徴付けられた。画分の密度は〜1.580の範囲であった- 1.759グラムml -1の 、そしてそれらは左から右に密度の降順に表示されます。
純粋な13の分離は、C -および12 C - DNA(図2)と発音することができる、環境サンプルのインキュベーションでは、解釈するより難しいかもしれませんが。例えば、我々は、インキュベーション15℃14日間の期間のいずれかの12 C -または13 C標識グルコース℃でレゾリュートベイ(ヌナブト準州、カナダ)から土壌をツンドラ勾配分画の精製DNAのアガロースゲルは、ゲノムDNAは、12 C -及び13 C -インキュベーション(それぞれ図3Aと3C、)の両方のための7-10分画を越えて"塗った"されることが示された。この場合、特定の微生物の分類群からバイオマスの13 C -濃縮は、16S rRNA遺伝子のDGGEのようなアプローチを決定することができます。 12 C -グルコース培養土壌のDNAは、すべての勾配分画(図3B)間で類似のパターンを生成しますが、13 C -グルコースインキュベートしたサンプルは、一意に分画5-8(図3D)に関連付けられているDGGEフィンガープリントを生成する。特に興味深いのは、矢印で示される保存のバンドです。この支配的な"phylotypeは、"すべての勾配分画が13 C -グルコース培養土壌から得られたDNAのための重い分画へのシフトを通じて一貫しています。このバンドおよび/またはクローンライブラリー解析のシーケンシング、その後のDNAはこの特定の16S rRNA遺伝子の同一性を確認し、その後のメタゲノムや栽培ベースのアプローチを導くでしょう。
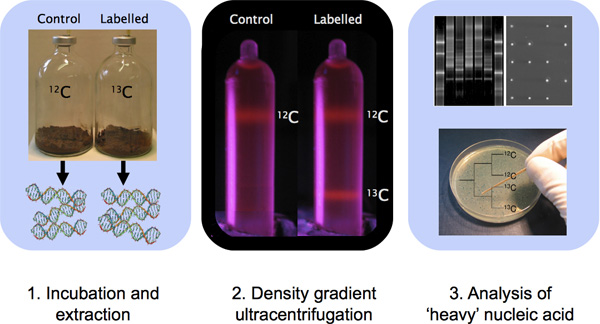
図1サンプルのインキュベーション、DNA抽出、CsCl密度勾配超遠心分離と分子生物学的手法によるDNAの特性評価を含むDNA - SIPの実験の概要。
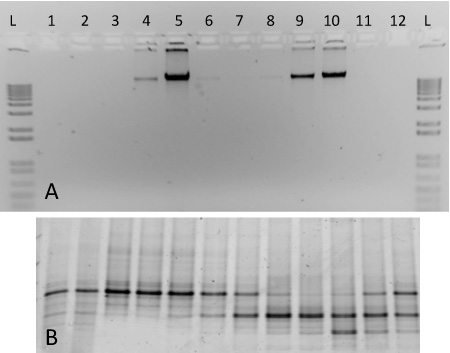
図2。2つの純粋な培養物からDNAを含むSIP勾配分画の結果が予想される。 (A)勾配分画1から12までのDNAのアリコートを、13 C標識Mを含有する勾配から1%アガロースゲル上で実行されたcapsulatusひずみバス(分画4-6)及び12 C -標識S. meliloti(フラクション8-10)。 1 - kbのラダーは、(B)PCR -増幅DNA同じ画分から10%のDGGEゲル上で実行された比較のために含まれています。指紋のパターンは、例えば分画5と9の間の明確な違いを、明らかに。
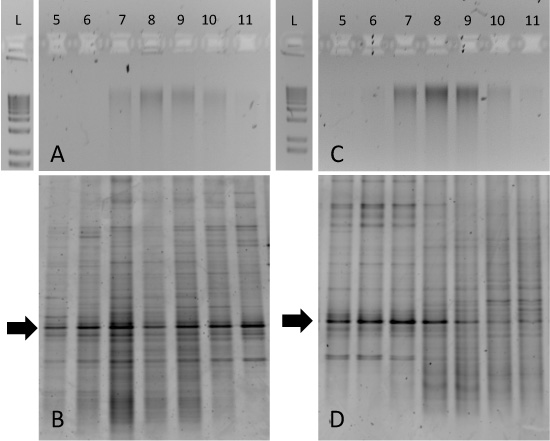
図3。土壌サンプルのインキュベーションからSIP勾配分別の結果を期待される。両方の12 C -グルコース改正土壌(A)及び13 C -グルコース改正土壌(C)から勾配分画のアリコートは、1%アガロースゲル上で実行され、1 kbのラダーは、比較のために含まれています。これらのサンプルのそれぞれに対応するDGGE指紋が(B)と(D)に示されています。分数のフィンガープリントは、フラクション5-8(D)における13 C -グルコース改正サンプルに特定の細菌の分類群の濃縮を明らかにする。
ディスカッション
安定同位体プロービング実験の適切な設計は、バックグラウンドラベルのない社会の上に標識されたDNAを得るために非常に重要である。サンプルのインキュベーション時間、基質濃度、インキュベーションの条件(例えば、栄養素、土壌水分の含有量)に関連した考慮事項、栄養共生とレプリケーションが他の場所で10,18議論と我々は、SIPインキュベーションを設計する際に、読者が...
開示事項
謝辞
この作品は、戦略的プロジェクトとカナダ自然科学工学研究評議会(NSERC)からJDNのディスカバリーの補助金によって支えられている。
資料
| Name | Company | Catalog Number | Comments | |
| Bromophenol Blue | Reagent | Fisher Scientific | BP115-25 | |
| Cesium chloride | Reagent | Fisher Scientific | BP210-500 | |
| Ethanol, reagent grade | Reagent | Sigma-Aldrich | 652261 | |
| Ethidium bromide | Reagent | Sigma-Aldrich | E1510 | |
| Hydrochloric acid | Reagent | Fisher Scientific | 351285212 | |
| Linear polyacrylamide | Reagent | Applichem | A6587 | |
| Polyethylene Glycol 6000 | Reagent | VWR international | CAPX1286L-4 | |
| Potassium Chloride | Reagent | Fisher Scientific | AC42409-0010 | |
| Sodium Chloride | Reagent | Fisher Scientific | S2711 | |
| Sodium Hydroxide pellets | Reagent | Fisher Scientific | S3181 | |
| Tris base | Reagent | Fisher Scientific | BP1521 | |
| Dark Reader | Equipment | Clare Chemical | DR46B | |
| Microcentrifuge | Equipment | Eppendorf | 5424 000.410 | |
| Nanodrop 2000 | Equipment | Fisher Scientific | 361013650 | |
| Infusion pump | Equipment | Braintree Scientific, Inc. | N/A | Model Number: BSP See www.braintreesci.com for ordering details. |
| Tube sealer | Equipment | Beckman Coulter Inc. | 358312 | |
| Ultracentrifuge | Equipment | Beckman Coulter Inc. | ||
| Ultracentrifuge rotor | Equipment | Beckman Coulter Inc. | 362754 | |
| Ultraviolet light source | Equipment | UVP Inc. | 95-0017-09 | Any UV source will suffice |
| Ultraviolet light face shield | Equipment | Fisher Scientific | 114051C | |
| Butyl rubber stoppers, gray | Material | Sigma-Aldrich | 27232 | |
| Centrifuge tubes | Material | Beckman Coulter Inc. | 342412 | |
| Hypodermic needle, 23 gauge, 2” length | Material | BD Biosciences | 305145 | |
| Microfuge tubes, 1.5 mL | Material | DiaMed | AD151-N500 | |
| Open center seals, 20 mm diameter | Material | Sigma-Aldrich | 27230-U | |
| Pasteur pipettes, glass | Material | Fisher Scientific | 13-678-6C | |
| Pipet tips | Material | DiaMed | BPS340-1000 | Catalogue number is for 200 μl tips. 10 or 20 μl tips may be purchased from the same source |
| Pump tubing 1.5 mm bore x 1.5 mm wall | Material | Appleton Woods | ||
| Screw-cap tubes, 15 mL | Material | DiaMed | AD15MLP-S | |
| Serum vials, 125 mL volume | Material | Sigma-Aldrich | Z114014 | |
| Syringe, 60 mL | Material | BD Biosciences | 309653 |
参考文献
- Neufeld, J. D. DNA stable-isotope probing. Nat. Protocols. 2, 860-866 (2007).
- Neufeld, J. D., Boden, R., Moussard, H., Schäfer, H., Murrell, J. C. Substrate-specific clades of active marine methylotrophs associated with a phytoplankton bloom in a temperate coastal environment. Appl. Environ. Microbiol. 74, 7321-7328 (2009).
- Nercessian, O., Noyes, E., Kalyuzhnaya, M. G., Lidstrom, M. E., Chistoserdova, L. Bacterial populations active in metabolism of C1 compounds in the sediment of Lake Washington, a freshwater lake. Appl. Environ. Microbiol. 71, 6885-6899 (2005).
- Padmanabhan, P. Respiration of 13C-labelled substrates added to soil in the field and subsequent 16S rRNA gene analysis of 13C-labelled soil DNA. Appl. Environ. Microbiol. 69, 1614-1622 (2003).
- Bernard, L. Dynamics and identification of soil microbial populations actively assimilating carbon from 13C-labelled wheat residue as estimated by DNA- and RNA-SIP techniques. Environ. Microbiol. 9, 752-764 (2007).
- Haichar, e. l. Z. a. h. a. r., F, . Identification of cellulolytic bacteria in soil by stable isotope probing. Environ. Microbiol. 9, 625-634 (2007).
- Addison, S., McDonald, I., Lloyd-Jones, G. Stable isotope probing: Technical considerations when resolving 15N-labelled RNA in gradients. J. Microbiol. Meth. 80, 70-75 (2009).
- Buckley, D. H., Huangyutitham, V., Hsu, S. -. F., Nelson, T. A. Stable isotope probing with 15N achieved by disentangling the effects of genome G + C content and isotope enrichment on DNA density. Appl. Environ. Microbiol. 73, 3189-3195 (2007).
- Schwartz, E. Characterization of growing microorganisms in soil by stable isotope probing with H218O. Appl. Environ. Microbiol. 73, 2541-2546 (2007).
- Neufeld, J. D., Dumont, M. G., Vohra, J., Murrell, J. C. Methodological considerations for the use of stable isotope probing in microbial ecology. Microb. Ecol. 53, 435-442 (2007).
- Martineau, C., Whyte, L., Greer, C. Development of a SYBR safe technique for the sensitive detection of DNA in cesium chloride density gradients for stable isotope probing assays. J. Microbiol. Meth. 73, 199-202 (2008).
- Bartram, A. K., Poon, C., Neufeld, J. D. Nucleic acid contamination of glycogen used in nucleic acid precipitation and assessment of linear polyacrylamide as an alternative co-precipitant. Biotechniques. 47, 1019-1022 (2009).
- Chen, Y. Revealing the uncultivated majority: combining DNA stable-isotope probing, multiple displacement amplification and metagenomic analyses of uncultivated Methylocystis in acidic peatlands. Environ. Microbiol. 10, 2609-2622 (2008).
- Neufeld, J. D., Chen, Y., Dumont, M. G., Murrell, J. C. Marine methylotrophs revealed by stable-isotope probing, multiple displacement amplification and metagenomics. Environ. Microbiol. 10, 1526-1535 (2008).
- Kalyuzhnaya, M. High-resolution metagenomics targets specific functional types in complex microbial communities. Nat. Biotechnol. 26, 1029-1034 (2008).
- Binga, E. K., Lasken, R. S., Neufeld, J. D. Something from (almost) nothing: the impact of multiple displacement amplification on microbial ecology. ISME J. 2, 233-241 (2008).
- Green, S. J., Leigh, M. B., Neufeld, J. D., Timmis, K. N. . Microbiology of Hydrocarbon and Lipid Microbiology. , 4137-4158 (2010).
- Neufeld, J. D., Wagner, M., Murrell, J. C. Who eats what, where and when? Isotope-labelling experiments are coming of age. ISME J. 1, 103-110 (2007).
- Gallagher, E., McGuinness, L., Phelps, C., Young, L. Y., Kerkhof, L. J. DNA shortens the incubation time needed to detect benzoate-utilizing denitrifying bacteria by stable-isotope probing. Appl. Environ. Microbiol. 71, 5192-5196 .
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請さらに記事を探す
This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved