このコンテンツを視聴するには、JoVE 購読が必要です。 サインイン又は無料トライアルを申し込む。
Method Article
HCVの制限要因を発見するヘテロカリオンの形成の2つの方法
要約
我々は、条件は、次の2つの方法について説明します。トランス-相補性C型肝炎ウイルス(HCV)は、アセンブリとヘテロカリオンの形成に依存している完全なウイルスのライフサイクルの完了。これらの技術は、感染性HCV子孫の生産を妨げる主要な制限因子を発現する細胞系の画面に適しています。
要約
C型肝炎ウイルス(HCV)は、ヒトとチンパンジーに制限宿主域を持つ肝炎ウイルスです。 HCV RNAの複製は、人間の非肝臓およびマウス細胞株で観察されているが、効率が非常に低く、HCVレプリコンを使用して、必要な長期的な選定方法については支配的な抗生物質選択マーカー1-5を発現して構築します。 in vitroでの研究で HCVしたがって、ウイルスのライフサイクルのウイルスのエントリと終了を許容するヒト肝癌細胞株に限定されています。狭い種の指向性をHCVsのため、完全なHCV複製サイクル6-8を維持使用可能な免疫小動物モデルは存在しません。マウス由来の例えば、非ヒト細胞中のHCVの非効率的な複製は、おそらく本質的なホストの依存因子および/または制限因子の発現の遺伝的不和合性の欠如によるものである。
我々はHCVの伝播が支配的な制限FAによって抑制されるかどうかを調べた非肝組織から、またはマウスの肝臓細胞株の由来のヒト細胞株のいずれかでctors。この目的のために、我々は、体細胞融合に頼るつの独立した条件付きのトランス相補方法を開発した。両方のケースでは、ウイルス複製サイクルの完了は、ヘテロカリオンでのみ可能です。その結果、感染性のウイルス子孫の de novoの生産を測定することによって決定される成功したトランス相補性は、支配的な制限がないことを示します。
具体的には、ルシフェラーゼ遺伝子を運ぶサブゲノムHCVレプリコンは非常に寛容なヒト肝癌細胞(ホ-7.5細胞)にトランスフェクトした。その後、これらの細胞は共培養し、HCV構造タンパク質コア、エンベロープ1および2(E1、E2)とアクセサリータンパク質P7とNS2を表現する種々のヒトおよびマウス細胞に融合させた。細胞融合は、ポリエチレングリコール(PEG)で処理することにより開始されている限り、文化は、感染性ウイルスのPAを発表受容体に依存する形でナイーブ細胞に感染rticles。
セルのエントリ、RNAの翻訳、複製およびウイルスのアセンブリを含む完全なウイルスのライフサイクル上の支配的な制限の影響を評価するために、我々は、CD81の内因性発現を欠いているヒト肝細胞株(ホ- 7 Lunet N細胞9)を利用したHCVの本質的な侵入因子。異所的に発現CD81の欠如では、これらの細胞は、HCV感染症の10〜本質的に難治性である。重要なのは、共培養し、少なくとも別の重要な細胞侵入因子(すなわち、SR-BI、CLDN1、OCLN)ヒトCD81が不足して発現する細胞と融合させたときにのみ、結果としてヘテロカリオンは、感染症のために必要なHCVエントリー因子の完全なセットを表示します。したがって、主要な制限因子はHCVの複製サイクルの完了を抑制するかどうかを分析するために、我々は、上記のcritを満たすヒトとマウスの原点からの様々な細胞でLunet N個の細胞を融合ERIA。共培養細胞は、プロトタイプの泡沫状ウイルスの変異性の高い融合ウイルスエンベロープタンパク質(PFV 11)でトランスフェクトし、その後、感染性HCV粒子(HCVcc)でチャレンジしたときに、感染性ウイルスの de novo生産が観察された。これは、HCVが成功し、これらの細胞株における主要な制限因子の発現を除外ヘテロカリオンでのレプリケーションサイクルを完了したことを示します。これらの新しい条件付きトランス相補方法は、HCV特有の主要な制限因子の発現のための細胞株および初代培養細胞の大型パネルをスクリーニングに有用であろう。
プロトコル
PEGによる細胞融合
1。細胞培養
- 文化ホ-7.5、HeLaおよびDMEM完全培地(DMEM CPLT)培地で15cmの細胞培養皿上ナイーブまたはパッケージセルライン12を Hep56.1D、DMEMは1x非必須アミノ酸、100 U /、2 mM L-グルタミンを添加したmLのペニシリン、100μg/ mLのストレプトマイシン、および10%ウシ胎児血清。 表1に示された安定な細胞株に適切な選択を適用します。
2。 HCV RNAのトランスフェクション
- 、リン酸二ホ-7.5細胞(1コンフルエント15cmの皿)(PBS)緩衝生理食塩水洗浄トリプシン処理し、Cytomixで1.5×10 7細胞/ mL(120 mMの塩化カリウム、0.15mMのCaCl 2を 、10mMのKの濃度で再懸濁2 HPO 4 2 mMのATP、5 mMのグルタチオンを含有するpHが7.6]、25mMのHEPES、2mMのEGTA、5mMのMgCl 2、KOHでpH 7.6に調整し、濾過により滅菌)。
- electroporat経由してトランスフェクション0.4センチメートルのギャップ幅と975に設定ジーンパルサーX細胞システム(バイオラッド)をキュベットを使用して10μgのHCV RNA(pFKi389Luc-EI/NS3-3 '_JFH1_dg 13)細胞懸濁液(6×10 6細胞)のイオン400μL μFと270 Vは、以前に14を説明します。
- すぐに10cmの直径の単一の培養皿にDMEM CPLT培地と種子全体の懸濁液10 mLにトランスフェホ-7.5のセルを転送します。 37℃で24時間培養細胞℃、5%CO 2。
3。 PEGによる核融合の誘導
- 収穫の細胞は、2.1で説明DMEM CPLT培地に再懸濁し、5×10 5細胞/ mLに希釈する。
- それぞれ、シングルセルホ-7.5の懸濁液を、HeLa細胞、およびHep56.1Dパッケージング細胞、希に5×10 5細胞/ mL、2×10 5細胞/ mLとし、2×10 5細胞/ mLを生成します。の2の導入後の異所的にそのパッケージング細胞はHCVタンパク質のコア、E1、E2、P7とNS2に注意してください。C-E1とE2-P7-NS2発現カセットを伝達に依存するレンチウイルスベクターは、それぞれ13、15。
- 融合効率のさらなる分析のために、ウェルあたり2から3滅菌ガラスカバースリップを6ウェル培養プレートを準備します。
- 6ウェルプレートの1ウェルにパッケージング細胞、種子の1mLでサブゲノムレプリコンを保有するトランスフェクトされたホ-7.5細胞の1 mLを組み合わせて、37℃、5%CO 2で24時間インキュベートします。
- この時点で、細胞密度は、細胞が融合する細胞膜の近接であることができるようにするために、細胞融合の誘導前にコンフルエント%60から80の間での範囲である必要があります。
- 共培養細胞から培地を吸引除去し、1mLのPBSで1回洗浄し、慎重にヘテロカリオンの形成を誘導するために37℃で5分間制御とインキュベートとして予め温めておいた40パーセントPEG-1500またはPBS 500μLを追加します。
- PEGを吸引除去し、慎重に余分なPEGを除去し、最後に2を追加するには、2 mLのPBSで洗浄ごとに1分(min)の3-5倍を洗う各ウェルにDMEM CPLT培地mLおよび37℃、5%CO 2をインキュベートします。
- 四十八時間融合後、0.45μmフィルターを介して各上清をフィルターを収集します。
- 500無細胞培養液のμLとルシフェラーゼのその後の決定とナイーブホ-7.5細胞(8×10 4細胞/ウェルで12ウェルプレートに播種し、接種前に24時間)の接種による融合ヘテロカリオンの感染性粒子のメジャー生産活動14。
4。融合効率を決定するために免疫蛍光
- 細胞培養上清(3.8節)を収集した後、カバーガラスを含む洗浄井戸は1分間PBS 1mLで一回スリップします。以下のすべてのステップは室温(RT)で行われるべきであり、洗浄は洗浄ごとに1分間1 mLのPBSで行う必要があります。
- 15分間、PBS中3%パラホルムアルデヒド(PFA)600μLで細胞を固定し、PBSで3倍に洗浄する。慎重にカバースリップを転送鉗子を用いて24ウェルプレートに。
- 0.5%の500μLトリトンX-100で5分間PBS中で細胞を透過処理し、その後PBSで3倍に洗浄する。
- 5%ヤギ血清を添加したPBSで一次抗体溶液を調製します。 6.4 ng / mLの濃度で0.5μg/ mLとし、ヒトモノクローナル抗E2抗体(CBH-23)の濃度で抗NS5A抗体(9E10)を使用します。各ウェルに250μLを適用し、45分間室温でインキュベートします。
- PBSで3倍の洗浄およびAlexa-546または弗素を添加したPBSで2μg/ mLの濃度でのAlexa-®488への結合抗マウスまたはヤギ抗ヒトIgG特異的抗体を250μLの二次ヤギでバインドされた一次抗体を検出する5%ヤギ血清。暗所で室温で30分間インキュベートします。
- その後、PBSで1倍を洗い、その後暗所に室温で1分間インキュベートし、PBSで1:3,000に希釈した250μLのDAPI(4 '、6'-ジアミジノ-2 - フェニルインドール二塩酸塩)と細胞核を染色する。
- H 2を含むPBSおよび1xと4xの洗浄 Oしてから、顕微鏡スライド上にFluoromountのドロップの上に逆さまにカバースリップをマウントし、間接蛍光顕微鏡で暗いし、評価サンプルでは、乾燥させます。
プロトタイプ泡沫状ウイルス(PFV)糖タンパク質の一過性トランスフェクションによる融合
5。ウイルス接種の準備
- ホ-7.5細胞へのin vitro転写HCV RNA でトランスフェクトすると、以前に14を説明したように48時間および72時間後に細胞培養上清を採取することにより、高力価のウイルスストックを生成します。希釈アッセイ(TCID 50)13を制限することにより、ウイルス力価を決定します。
6。融合性PFV糖タンパク質のトランスフェクションによって細胞培養、細胞融合
- 適切な選択を含むDMEM CPLT培地中の表1の手順に従って、文化Lunet N、HeLa細胞、およびHep56.1D hCD81細胞。
- 2.1で説明したように細胞を剥がし、DMEM CPLT mで、単一細胞懸濁液を調製edium、共同育成Lunet N当たり1.5×10 5細胞の総細胞密度が得られる適切な比率(Hep56.1D hCD81またはHeLa細胞の1:1例えば1:2)でHeLa細胞またはHep56.1D hCD81の細胞株で細胞12ウェルと37℃で24時間インキュベート℃、5%CO 2。
- 次の日に、一時的に製造元の指示に従って、リポフェクタミン2000を使用して高度に融合PFVエンベロープタンパク質(pczHFVenvEM066 16)で細胞をトランスフェクション。
7。感染アッセイ
- 一晩12時間のウイルス株の350μL(MOI〜2.3)でトランスフェクション後のヘテロカリオン30 hを接種し、PBSで1回洗浄し、ウェルあたりDMEM CPLT培地1 mLを加える。 °C、5%CO 2、37℃で48時間インキュベートする。
- 限界希釈アッセイで、前日(1×10 4細胞/ウェル)を96ウェルプレートに播種し0.45μmのフィルターと接種ホ-7.5細胞を通じて、フィルタヘテロカリオン文化の収穫上清、 13。
- 感染性子孫粒子の生産を定量化するため、72時間後に、HCV特異的抗体(9E10 NS5A)に感染したホ-7.5細胞を染色。
8。細胞融合の可視化
- 融合イベントを可視化するために、無添加DMEM培地でCellTracker染料の5から10μM溶液を調製し、(セクション6.2と6.3で説明)を共培養する前に、6時間以下の手順を実行します。
- 5mlのPBSで一度付着細胞を洗浄し、その後37℃で45分間10cmのディッシュあたり染色溶液4 mLで細胞をインキュベート℃、5%CO 2(Lunet N CellTrackerオレンジCMTMRおよびHeLaまたはHep56.1Dで細胞を染色CellTrackerグリーンCMFDA、別々に)とhCD81細胞。
- 1分間5mlのPBSで一回細胞を洗浄し、DMEM CPLT媒体を追加し、6で37時間℃、5%CO 2でインキュベートします。
- セクション6.2しかしで上記のように染色した細胞の播種とトランスフェクションを準備し、カバースリップを追加する播種前に細胞培養皿。
- 30時間トランスフェクション後、RTで15分間3%PFA 250μLの細胞を固定して1分間PBS 1mLで慎重に細胞を洗浄します。セクション4.6で説明したように、1分間1mLのPBSで一回細胞を洗浄し、1分間DAPIで染色し、(セクション4.7を参照してスライドガラス上にH 2 Oとマウントカバースリップで洗浄を繰り返し、1分間1 mLのPBSで1回洗浄。)蛍光顕微鏡で細胞を分析します。
| 種 | 細胞株 | 起源 | 増殖培地と選択 |
| 人間 | ホ-7.5 | ホ- 7肝癌細胞株17のサブクローン | DMEM CPLT |
| ホ-7.5 [CE1] [E2p7NS2] | 安定したコア、E1、E2、P7とNS2はウイルスタンパク質を発現するレンチウイルス遺伝子tで導入二つの独立した遺伝子カセットの順伝達 | DMEM CPLT +ブラストサイジン5μg/ mLの | |
| ホ- 7 Lunet N | ホ- 7肝癌細胞株9のサブクローン | DMEM CPLT | |
| HeLa細胞 | 子宮頸部腺癌細胞株(ATCC番号:CCL-2) | DMEM CPLT | |
| HeLa細胞[CE1] [E2p7NS2] | 安定したコア、E1、E2、P7とNS2はウイルスタンパク質を発現する、2つの独立した遺伝子カセットのレンチウイルス遺伝子導入による形質導入 | DMEM CPLT + G418 750μg/ mLで、ブラストサイジン5μg/ mLの | |
| マウス | Hep56.1D | 原発性肝細胞癌(J.エンケの親切な贈り物) 大人のC57BL/6Jマウス | DMEM CPLT |
| Hep56.1D [CE1] [E2p7NS2] | 安定EXウイルスタンパク質を押すと、コア、E1、E2、P7とNS2は、2つの独立した遺伝子カセットのレンチウイルス遺伝子導入による形質導入 | DMEM CPLT + G418 750μg/ mLで、ブラストサイジン5μg/ mLの | |
| Hep56.1D hCD81 | 安定的に発現するヒトCD81は、レンチウイルス遺伝子導入による形質導入 | DMEM CPLT +ブラストサイジン5μg/ mLの |
完了すると、G418:ジェネティシン、H:ヒューマン。使用細胞株CPLTの起源と文化の状態については表1の仕様。
9。代表的な結果
本研究では、私たちは、特にヘテロカリオンにおけるHCVの複製サイクルの完了時の制限要因の影響を調査することができます体細胞融合の2つの異なる方法を適用した。注目すべきは、二つの異なる種類の細胞融合時にトランス相補性の条件設計があることを保証唯一のヒト肝細胞と非許容細胞系、ウイルスのアセンブリ(アプローチ1)またはウイルスエントリと完全なHCV複製サイクル(アプローチ2)との間の真のヘテロカリオンで達成されています。
最初のアプローチではHCVのための非常に寛容なホ-7.5細胞は、一過性にサブゲノムHCV RNAの複製(ホ-7.5レプリコン細胞)をサポートしているルシフェラーゼ遺伝子およびHCV非構造蛋白質を発現するサブゲノムレプリコントランスフェクションした。これらの細胞は共培養と、E1、E2と同様に、アクセサリータンパク質のP7、とNS2のHCV構造タンパク質のコアを発現する細胞株と融合させた。図1aは、実験手順の概要を示します。重要なのは、PEG融合後に、粒子の組み立てに必要なすべてのタンパク質は、個々のウイルス蛋白質に対する免疫蛍光法により確認することができます形成されたヘテロカリオン中に存在しなければならない。図1Bは、ホ-7.5レプリコン細胞で発現NS5AとE2の両方の信号の典型的な融合事象を示していますホ-7.5で表される[CE1] [E2p7NS2]パッケージング細胞は、PEG-誘導ヘテロカリオンの形成時に同一の細胞質で検出された。共培養細胞の代わりにPBSで処理した場合、何の融合は、このように、単一の陽性細胞を誘導されなかった、信号のない共局在は認められなかった。さらに、導入遺伝子の発現を制御するために、我々は、コアとE2(以前は詳細15で報告)のために特異的ELISAを行った。 7.5ホ·コントロール·パッケージング細胞株に加えて[CE1] [E2p7NS2]、我々はそれぞれ、ヒト非肝臓およびマウス肝臓細胞の代表としてHeLaおよびHep56.1Dパッケージング細胞を生成します。これらの後者の細胞中のHCV RNAの複製効率が低く、HCVアセンブリであり、リリースが示されていない。図1Cは、融合アッセイの結果を示しています。上清を48時間後の融合誘導で共培養から回収し、その後のルシフェラーゼアッセイのためにナイーブホ-7.5細胞を接種するために使用した。特に、時ホ-7.5レプリコン細胞のいずれかで融合させたコントロールとしてPBSで処理したナイーブ細胞株やは、1感染性は培養液中に検出されなかった。しかし、ホ-7.5レプリコンおよびパッケージング細胞株との間の細胞融合はPEGによって誘導されたときに、レプリコンと恒常的に発現構造のタンパク質間のトランス相補性は、結果のヘテロカリオンで ウイルス産生を救出し、感染性ウイルス粒子は、培養液中に放出された。そこで我々はウイルス産生は、パッケージング細胞株におけるウイルス蛋白質の細胞融合と表現が必要であると結論付けた。驚くべきことに、ホ-7.5ヒト肝癌細胞のヘテロカリオンでなく、人間以外の肝臓(HeLa細胞[CE1] [E2p7NS2])およびマウスの肝臓(Hep56.1D [CE1] [E2p7NS2])細胞が許可されてウイルスのリリースではヘテロカリオンだけでなく、アセンブリとリリースが支配的にこれらの細胞型で可能な制限要因によって阻害されていないことを示しています。パッケージング細胞株と標的細胞への本格的なエントリの構造タンパク質の発現がフレンツェンに示されたら15。
細胞融合の代替方法は、セルのエントリ、RNAの翻訳および複製サイクルのRNA複製の段階に影響を与える可能性の制約を分析し、トランスフェホ7.5レプリコン細胞において高ウイルス負荷が飽和によって制限因子の検出を妨げることを除外するために利用された。この目的のために、我々はHCVccと、これらの細胞におけるde novoのウイルス産生の検出によるヘテロカリオンの接種に基づいて、独立したアッセイを開発しました。図2Aで概説したように、体細胞融合は、細胞の共培養後の融合蛋白質の発現によって誘導された。感度が増加融合の効率化に可能性が増加したため、このメソッドが選ばれました。ヘテロカリオンでは、共培養に先立ってCellTracker色素を持つ別の細胞株を染色により可視化した。示すように細胞質内に均一に分散染料のヘテロカリオンは、2種類の細胞融合を示しています図2Bのd。のみLunet N HeLa細胞またはHep56.1D hCD81細胞と細胞のヘテロカリオンですべてのHCVエントリーの要因は、このように選択的にHCVの細胞のエントリへの影響を受けやすいヘテロカリオンをレンダリング表現されています。 HCVccの課題は、約10倍のアッセイのバックグラウンド(図2C)上記の感染性のウイルス子孫の de novo生産から明らかであるヘテロカリオン中のHCVの完全な複製をもたらした。コントロールとして、Lunet N個の細胞がLunet N細胞と融合させた。重要なのは検出限界に感染性HCVに近いのは非常に低いレベルが観察された。 Lunet N個の細胞がナイーブHep56.1D細胞と融合させた場合、同様の結果が得られた。 HCVは生産ヘテロカリオンに感染しないことができるように両方のケースでは、人間のCD81は不在だった。これら二つの後者のケースで検出されたこの非常に低い感染性はLunet N個の細胞および/または接種から残存感染性ウイルスの入力の低レベルの感染症の低レベルの可能性が起因している。コントラストHeLa細胞と彼のp56.1D hCD81の細胞はこのようにヘテロカリオンで欠けている細胞侵入因子を補完し、HCVセルの入力を可能にして、人間のCD81を発現する。これら2つの細胞株を含むヘテロカリオンの媒体で検出された感染性HCVの10倍に増加したレベルは、したがって、おそらくヘテロカリオンの感染性粒子の de novo生産を反映しています。したがって、我々は、HCVの複製サイクルのその完成は、これらの細胞株における支配的なHCVの制限因子の発現を除く限定されるものではないと結論づけた。
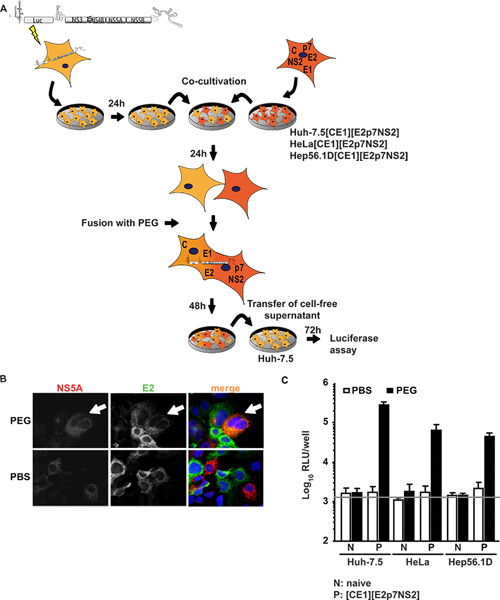
図1:ヘテロカリオンにおけるHCVアセンブリとリリースのトランス相補性。PEG介在細胞融合の実験手順の()回路図の概要。 JFH1ルシフェラーゼレポーターレプリコンリュック-NS3-5Bは、エレクトロポレーションによってナイーブホ-7.5細胞にトランスフェクトした。翌日、細胞が切り離され、ナイーブ細胞や包装と共培養恒常的にHCVコア、E1、E2、P7とNS2を発現する細胞。 24時間後の共培養、ヘテロカリオンの形成は、ネガティブコントロールとしてPBSを用いて40%PEGで処理することにより誘導された。 48時間後に、無細胞上清は、ナイーブホ-7.5細胞を接種するために収穫した。感染性はルシフェラーゼ活性により測定した。 (B)ヘテロカリオンの検出のために、細胞はE2とNS5Aに対するモノクローナル抗体を用いて免疫染色した。細胞内の両方のタンパク質の同時発現は、細胞融合の指標である。 (C)ルシフェラーゼの測定は、ヘテロカリオンから生産、ウイルス感染を定量化するために行われた。 3つの独立した実験手段の標準偏差の平均値が与えられている。水平バーは、感染していないホ-7.5細胞で決定した背景のRLUを表しています。 拡大図を表示するには、ここをクリックしてください 。
9/4029fig2.jpg "/>
図2。ヘテロカリオンの接種後のHCV複製サイクルが完了する。()実験手順の概略。 CD81を欠損ホ - 7 Lunet N個の細胞は、ヒトCD81を欠損または発現している指定された細胞株と共培養した。重要なことは、これらの後者の細胞は、少なくとも一つのHCV侵入因子を欠いているため、生産性に感染することはできません。次の日は、融合がPFVから融合ウイルスエンベロープ蛋白質のトランスフェクションによって開始されました。 30時間後、細胞を感染性HCV粒子でチャレンジした。これらのヘテロカリオンによって支えセルエントリ、RNA複製及びウイルス産生を測定するために、無細胞培養液に放出感染性は48時間後の接種を決定した。この目的のために、ナイーブホ-7.5細胞は、限界希釈アッセイ(TCID 50)を標的細胞として用いた。 (B)ヘテロカリオンの検出のために、細胞は共同cultiv前にCellTrackerグリーンまたはオレンジCellTracker 6時間で染色したationとトランスフェクション。 30時間後、細胞を固定し、DAPIで染色した。 (C)Lunet N個の細胞は、ヒトCD81を発現するHeLa細胞、ナイーブHep56.1DまたはHep56.1D細胞に融合させた。これらの培養物をHCVcc(MOI 2.3)を接種した。培養液は48時間後に採取し、ヘテロカリオンからのde novo粒子の放出はナイーブホ-7.5標的細胞を用いてTCID 50により測定した。 3つの独立した実験の平均値が与えられている。水平バーは、アッセイの検出限界を表しています。 拡大図を表示するには、ここをクリックしてください 。
ディスカッション
我々は、HCVの複製を妨げるドミナントネガティブ制約の分析のために培養細胞におけるヘテロカリオンの形成を誘導する2つのメソッドを紹介します。これらの手順を使用して様々なヒト非肝臓およびマウス肝臓細胞株における支配的な恒常的に発現またはウイルス誘発因子の存在を除外した。制限要因は感染症の子孫のHCVアセンブリと放出を防止する場合、最初のアッセイでは、主に分析し...
開示事項
TPはバイオテストから顧問料を受け取った。
謝辞
我々は、それぞれ、JFH1とJ6CF分離株のTakaji脇田とイェンス·ブフに感謝しています。さらに我々は、チャールズホ-7.5セルのライスと9E10抗体、E2特異的抗体CBH-23のスティーブンFoung、および実験的ウイルス学、有用な提案や議論のためのTwincore学科のすべてのメンバーに感謝します。
資料
| Name | Company | Catalog Number | Comments |
| 試薬の名前 | 会社 | カタログ番号 | |
| DMEM | インビトロジェン社、カールスルーエ、ドイツ | 41965-039 | |
| L-グルタミン酸 | インビトロジェン社、カールスルーエ、ドイツ | 25030-024 | |
| 非必須アミノ酸 | インビトロジェン社、カールスルーエ、ドイツ | 11140-035 | |
| ペニシリン/ストレプトマイシン | インビトロジェン社、カールスルーエ、ドイツ | 15140-122 | |
| ウシ胎仔血清 | PAA、コルベ、ドイツ | A15151 | |
| α-E2(CBH23) | 親切にスティーブンFoung 10が提供する | ||
| ATP | シグマ、シュタインハイム、ドイツ | A2833-106 | |
| グルタチオン | シグマ、シュタインハイム、ドイツ | G4251-1G | |
| ブラストサイジン | Invivoゲン、サンディエゴ、アメリカ合衆国 | ANT-BL-1 | |
| G418(ゲネチシン) | インビトロジェン社、カールスルーエ、ドイツ | 11811-064 | |
| ポリエチレングリコール-1500 | ロシュ、マンハイム、ドイツ | 10783641001 | |
| パラホルムアルデヒド | ロス、カールスルーエ、ドイツ | 0335.3 | |
| トリトンX-100 | ロス、カールスルーエ、ドイツ | 3051.2 | |
| ヤギ血清 | シグマ、シュタインハイム、ドイツ | G9023-5mLの | |
| α-NS5A(9E10) | 親切にチャールズ·ライス7が提供する | ||
| DAPI(4 '、6'-ジアミジノ-2 - フェニルインドール二塩酸塩) | インビトロジェン | D1306 | |
| アレクサ·弗素546 - ヤギ抗ヒトIgG | インビトロジェン社、カールスルーエ、ドイツ | A21089 | |
| アレクサ·弗素488 - ヤギ抗マウスIgG | インビトロジェン社、カールスルーエ、ドイツ | A10680 | |
| リポフェクタミン2000 | インビトロジェン社、カールスルーエ、ドイツ | 11668-019 | |
| CellTracker CMTMR | インビトロジェン社、カールスルーエ、ドイツ | C2927 | |
| CellTracker CMFDA | インビトロジェン社、カールスルーエ、ドイツ | C2925 | |
| Fluoromount | シグマ、シュタインハイム、ドイツ | F4680-25ML | |
| 他のすべての化学物質 | ロス、カールスルーエ、ドイツ | ||
| 細胞培養材料 | ザルスタット、Nümbrecht、ドイツ |
参考文献
- Zhu, Q., Guo, J. T., Seeger, C. Replication of hepatitis C virus subgenomes in nonhepatic epithelial and mouse hepatoma cells. J. Virol. 77, 9204-9210 (2003).
- Kato, T. Nonhepatic cell lines HeLa and 293 support efficient replication of the hepatitis C virus genotype 2a subgenomic replicon. J. Virol. 79, 592-596 (2005).
- Ali, S., Pellerin, C., Lamarre, D., Kukolj, G. Hepatitis C virus subgenomic replicons in the human embryonic kidney 293 cell line. J. Virol. 78, 491-501 (2004).
- Date, T. Genotype 2a hepatitis C virus subgenomic replicon can replicate in HepG2 and IMY-N9 cells. J. Biol. Chem. 279, 22371-22376 (2004).
- Chang, K. S. Replication of hepatitis C virus (HCV) RNA in mouse embryonic fibroblasts: protein kinase R (PKR)-dependent and PKR-independent mechanisms for controlling HCV RNA replication and mediating interferon activities. J. Virol. 80, 7364-7374 (2006).
- Zhong, J. Robust hepatitis C virus infection in vitro. Proc. Natl. Acad. Sci. U.S.A. 102, 9294-9299 (2005).
- Lindenbach, B. D. Complete replication of hepatitis C virus in cell culture. Science. 309, 623-626 (2005).
- Wakita, T. Production of infectious hepatitis C virus in tissue culture from a cloned viral genome. Nat. Med. 11, 791-796 (2005).
- Witteveldt, J. CD81 is dispensable for hepatitis C virus cell-to-cell transmission in hepatoma cells. J. Gen. Virol. 90, 48-58 (2009).
- Bitzegeio, J. Adaptation of hepatitis C virus to mouse CD81 permits infection of mouse cells in the absence of human entry factors. PLoS Pathog. 6, e1000978 (2010).
- Lindemann, D., Goepfert, P. A. The foamy virus envelope glycoproteins. Curr Top Microbiol Immunol. 277, 111-129 (2003).
- Brohm, C. Characterization of determinants important for hepatitis C virus p7 function in morphogenesis by using trans-complementation. J. Virol. 83, 11682-11693 (2009).
- Steinmann, E., Brohm, C., Kallis, S., Bartenschlager, R., Pietschmann, T. Efficient trans-encapsidation of hepatitis C virus RNAs into infectious virus-like particles. J. Virol. 82, 7034-7046 (2008).
- Koutsoudakis, G. Characterization of the early steps of hepatitis C virus infection by using luciferase reporter viruses. J. Virol. 80, 5308-5320 (2006).
- Frentzen, A. Completion of hepatitis C virus replication cycle in heterokaryons excludes dominant restrictions in human non-liver and mouse liver cell lines. PLoS Pathog. 7, e1002029 (2011).
- Lindemann, D. A particle-associated glycoprotein signal peptide essential for virus maturation and infectivity. J. Virol. 75, 5762-5771 (2001).
- Blight, K. J., McKeating, J. A., Rice, C. M. Highly permissive cell lines for subgenomic and genomic hepatitis C virus RNA replication. J. Virol. 76, 13001-13014 (2002).
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請さらに記事を探す
This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved