Method Article
ライブの検出
要約
定量PCRアッセイは、 大腸菌 O157を検出するために開発されました:H7は、固有の遺伝子マーカー、Z3276をターゲットに。定量PCRは、生細胞を検出するためのプロピジウムモノアジド(PMA)処理と合わせた。このプロトコルは、多数のサンプルの容易かつ一貫性のある操作のための96ウェルプレートフォーマットに変更され、適応されてい
要約
ユニークなオープンリーディングフレーム(ORF)を、E. Z3276に特異的な遺伝子マーカーとして同定された大腸菌 O157:H7。定量PCRアッセイは、E.の検出のために開発された大腸菌 O157:ORF Z3276をターゲットにH7。このアッセイでは、我々は、 大腸菌のDNAのゲノムのいくつかのコピーと低く、検出することができます大腸菌 O157:H7。アッセイの感度および特異性は、E.、多数の集中的な検証テストにより確認された大腸菌 O157:H7株た(n = 369)および非O157菌株た(n = 112)。さらに、我々は死細胞から生細胞を選択的に検出するため新たに開発された定量PCRプロトコルでプロピジウムモノアジド(PMA)の手順を組み合わせている。 PMA処理死細胞からのDNAの増幅がほぼ完全にPMA処理生細胞からのDNAのほとんど影響増幅とは対照的に阻害された。さらに、プロトコルは変更され、多数のサンプルの容易かつ一貫性のある操作のための96ウェルプレートフォーマットに適合されている。 T彼の方法は、正確な微生物学および食品安全性、環境光源の疫学的監視に影響を与えることが期待される。
概要
PCRは、E.の検出のための一般的な技術である大腸菌 O157:食品や環境サンプルからH7。 E.のための具体的なバイオマーカーを同定大腸菌 O157:H7はPCRアッセイで成功を検出するための重要な要因の一つである。 E.共通の検出のためのPCRアッセイに用いられるバイオマーカー大腸菌 O157:H7は、志賀毒素(STX 1及びSTX 2)1,2、 のuidA 3,4、EAEA 5,6、rfbE 7、およびはfliC遺伝子8,9が含まれています。これらのバイオマーカーを同定するための変数効率を提供することができるが、バイオマーカーのほとんどは、 大腸菌の検出のためのユニークなバイオマーカーとして使用することができない大腸菌 O157:H7。標的遺伝子の分化能力のこの不備は、E.の同定のためのより特異的かつ強力なバイオマーカー(単数または複数)を要求する大腸菌 O157:H7の10。
Eの遺伝子または仮想的なオープンリーディングフレーム(ORF)のための多数のプローブ大腸菌のO157:H7 11は我々の研究室からのDNAジェノタイピングマイクロアレイから国立バイオテクノロジー情報センターのGenBankデータベース内で検索して手がかりをもとに定量PCRアッセイのために設計されていた。線毛遺伝子11であるZ3276 ORFの配列は、定量PCR分析のために選択した。続いて、定量PCRアッセイは、このようなプライマーおよびプローブの濃度、ならびにアニーリングおよび伸長温度(データは図示せず)のようなPCRパラメータに関する多数の試験により開発された。例えば大腸菌などの参照株に対するインセンティブ包括と排他性試験後大腸菌 O157:H7のqPCRにおいて、非O157や赤痢菌の菌株、ORF Z3276を大腸菌の同定に特異的かつ強力な遺伝子マーカーとして同定された大腸菌 O157:H7。その後、我々はE.を識別するためのユニークなバイオマーカーを使用して、新しい定量PCR法を開発大腸菌 O157:H7。
Eの検出に別の課題大腸菌 O157:H7にfoを汚染ODSと環境サンプルからの生きた細胞の正確な決定である。死細胞およびPCRアッセイで生存能力を差別化することができないことからのDNAの拡張プレゼンス検出の生細胞数の過大評価、その結果、偽陽性測定値を引き起こす可能性があります。その結果、これは食中毒病原体12の正確な検出にPCRの使用量を制限します。死細胞のDNAを区別するための新しいアプローチは、ここ数年13〜15にPCR法でプロピジウムモノアジド(PMA)処理を組み合わせることによって行われています。 PMAは、死細胞の内側に移動して、光にさらされると、DNAにインターカレートすることができますが、それは生きた細胞のDNAと反応させるために生きている細胞の中に入ることはできません。このようにして、生細胞と死細胞のPMA処理混合物中で、死んだ細胞のDNAをPCR 13で増幅することができません。我々は選択的にライブEを検出するために、新たに開発された定量PCRアッセイでPMA処理を組み合わせた大腸菌 O157:H7の細胞。さらに、当社は、PMA-QPCR Aを行ったハイスループット検出が可能ssay。
プロトコル
1。細菌株およびDNAテンプレートの調製
- Eの細菌株を育てる大腸菌 O157:H7、非O157、一晩振とうしながら、このような37℃でのサルモネラや赤痢菌などの他の病原性種、。
- 適切なキット(材料の表を参照)を使用して、メーカーの指示に従って細菌培養物からDNAを抽出する。
- 分光光度計を用いてDNA濃度を測定する光学密度(OD 260)を測定する。
2。定量PCRアッセイ用のプライマーおよびプローブの設計
- 大腸菌 O157:H7配列は、GenBank登録番号AE00517411からのものである。設計プライマーおよび(詳細は材料の表を参照)リアルタイムPCRのためのプライマーおよびプローブ設計のための適切なソフトウェアを用いてプローブ。
プライマーおよびプローブの配列は以下の通りである。Z3276フォワード(F)5'-TATTCCGCGATGCTTGTTTTT-3 '
Z3276-リバース(R)5'-ATTATCTCACCAGCAAACTGGCGG-3 '
Z3276-プローブ、FAM-CCGCAATCTTTCC-MGBNFQ - プライマー作業液として10μMの濃度で、その結果、水によるプライマ粉末を再構成する。
- プローブ作業ソリューションとして10μMのプローブを希釈します。標識プローブは、バッチで効力が異なります。そのため、最適な定量PCRパフォーマンスを使用する前に、標識プローブの各バッチを滴定。
3。定量PCRアッセイ条件の設定
- 2回のリアルタイムPCRマスターミックスの25.0μlを、フォワードプライマー、200nMのリバースプライマー、200 nMの、および100nMのプローブから構成され、反応混合物を調製する。
- 50μlの最終容量に到達するために5サンプルDNA(100 PG)のμLと水の適切な量を追加します。
- 非テンプレートコントロールのために、DNAサンプルを置き換えるために水5μlを使用する。
- 変性のために10秒、60&、95℃の40サイクル、続いて95°CでのTaqManの活性化のため10分間以下のように定量PCR条件を設定する#176;℃アニーリング/伸長のための1分間。
4。定量PCR感度テスト
- (;メッキにより約1.5×10 8 CFU / mlのOD 600 = 0.5)中期指数期の培養物から連続10倍希釈を作る。
- 三連で96ウェルプレートに細胞希釈の100ミリリットルを追加します。
- 10分間2500×gでプレートを遠心分離する。
- 各ウェルにDNA抽出溶液50mlを加える。
- マルチチャンネルピペットで再懸濁細胞ペレット。
- 10分間水浴中でプレートを沸かし、フィルムでプレートをシールし、2分間2500×gで遠心する。
5。 PMA処理のための生死細胞混合物の準備
- Eの10ミリリットルを育てる大腸菌 O157:中期指数期まで37℃でH7および2つのアリコートに文化を分ける。
- 死細胞のための水浴中で10分間の細胞の1アリコートを沸かし、生細胞のための他の分量にしておきます。
- 確認する再LB寒天プレート上に細胞をプレーティングすることによって熱殺菌アリコートからの生細胞ません。
- ライブおよび熱殺菌細胞の2ミリリットルを取り、8の濃度に調整×10 6 CFU / LB培地でミリリットル。 8×10 は0〜8×10 6 CFU / mlの範囲の細胞希釈の4セットを作成。
- PMAまたは未処理で処理された生細胞の細胞希釈液の最初の2セットを使用してください。
- ライブや死細胞の混合物を作るために-細胞希釈の第3および第4のセットに、各細胞希釈(8×10 6 8×10 0)に8×10 6死細胞を追加します。
- PMAで希釈の第三のセットを扱い、制御のための希釈液の第4セットを使用する。
6。 PMAで細胞を処理すること
- 10mMストック溶液と暗所で-20℃で保存を行うためにジメチルスルホキシド(または水)中にPMAを溶解する。
- 生細胞、死細胞、および生細胞と死細胞中の混合物のアリコートを400μl3つの別々のマイクロチューブ。
- 50μMの最終濃度をそれぞれの細胞アリコートに10 mMのPMAの2.0ミリリットルを追加します。
- 穏やかに数回、3秒毎に時間振とう、暗所で5分間、周囲温度でサンプルをインキュベートする。
- 96ウェルプレート上のサンプルを100μlを加える。
- 光学フィルムでプレートをシールし、氷の上にプレートを置く。
- 20センチメートル650 Wのハロゲン光源からのプレートをセットし、露光用2分間さらす。
- 10分間2500×gでプレートを遠心し、静かに上清を廃棄し、慎重に吸収紙の上にプレートを排出。
- DNA抽出液50μlに再懸濁した細胞ペレットにピペッティングすることにより、マルチチャンネルピペットマンで20回ダウン。
- 水浴中で10分間沸騰させ、フィルムでプレートをシールし、2分間2500×gで遠心分離します。プレート内の上澄み液は、PMAおよび定量PCRの準備ができてで処理した細胞からのDNAである。
- descriとして定量PCRを行う上記ベッド、DNAの5μLを使用。
結果
Eの検出大腸菌 O157:ORF Z3276を使用して定量PCRによりH7
このアッセイの重要な要因の一つは、定量PCRに用いられるZ3276プローブの感度および特異性である。それは、 大腸菌のDNAのゲノムのいくつかのコピーと低く、検出することができます大腸菌 O157:H7、図1Aに示すように、 6主要な非O157 STEC株、O104を含め、検討120非O157菌株の表1に示したように、H4株、 サルモネラ 、 赤痢菌株 、交差反応性は、認められなかった。
定量PCRとのPMA処理の組み合わせ
インキュベーションE.大腸菌 O157:H7、暗所で5分間PMA(50 mM)を有する細胞、2分間光に曝すは、PMA処理条件のために選択した。さらに、当社は、PMA処理手順を変更しました。架橋工程において使用される種々の透明なマイクロチューブと比較して、96ウェルプレートを、より効率的であることが見出された最適な架橋効果と、露光プロセス中に試料管を多数取り扱いよりも便利に達する。
定量PCRでのPMA処理生細胞と死細胞からのDNAの増幅
この定量PCRアッセイに死細胞からのDNAの増幅のPMA媒介性阻害の効果は、2つの連続10倍生または死細胞由来のDNAの希釈液を用いて、図1に示した。結果は、PMA(赤い曲線)およびPMAなし(青の曲線)で処理した生細胞から生成された曲線は線形で互いにほぼ同じに見えることを示している。わずかなCT値の差は、PMA処理は、定量PCRにおける生細胞のDNAの増幅にほとんど影響を及ぼさなかったことを示唆し、そしてPMAでPMA( 図1A)なしで処理した生細胞の間に示された。しかし、15 - CT値差(32,000倍)は、PMAで処理された死細胞間およびPMA、デモなしで示されたPMA処理を効率的に死滅した細胞( 図1B)からのDNA増幅を抑制することがnstrating。
定量PCRに住んでいて、死んだ細胞混合物からの生細胞の検出
生細胞希釈(8×10 0から 8×10 6 CFU)の2つのセットは、生/死細胞の混合物から生細胞を検出するために、PMAでまたはPMAなしで処理した。定量PCRの結果( 図2A)は、未処理サンプル(青いバー)ように処理生細胞(紫色のバー)の数に関連するものとしてのCT値の並列逆進歩的な傾向を示した。処理された生細胞は、未処理の生細胞のものよりも幾分高いCT値が得られた。 CT値で同様の逆プログレッシブな傾向は、 図2(b)にも同様のPMA(緑色のバー)で処置した生/死細胞の混合物中の生細胞の実数で観察された。また、CT値のこの減少傾向であった106死細胞の存在にもかかわらず、混合物中の生細胞数で観察した。これらの結果は、死細胞からのDNAの増幅が効率的にPMA処理により抑制されたのに対し、生/死細胞混合物のCT値は、単独で生細胞からDNAを表したことを示した。しかし、PMAで処理された生/死細胞混合物のCT値が変動し、 図2(b)の混合物中の生細胞数(黄色のバー)に反映しなかったし。
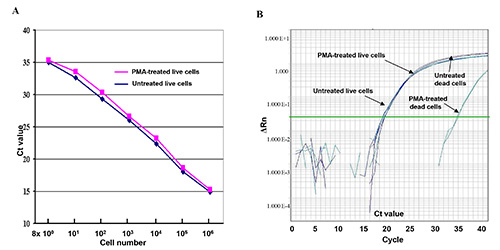
図1。生細胞と死細胞をPMAで、または定量PCRアッセイにおけるPMAなしで処理した。(A)のシリアルは、ライブEを 10倍に希釈した大腸菌 O157:H7の細胞希釈(8×10 0から 8×10 6 CFU)は、PMAでまたはPMAなしで処理した。CT値は、定量PCRアッセイにおける三つ組の平均CT値を表す。 (B)Q比較定量PCRアッセイでPMAでとPMAなしで処理生細胞と死細胞のDNA増幅の比較。 ΔRnのは、強度変化を蛍光に言及。 拡大画像を表示するにはここをクリックしてください。
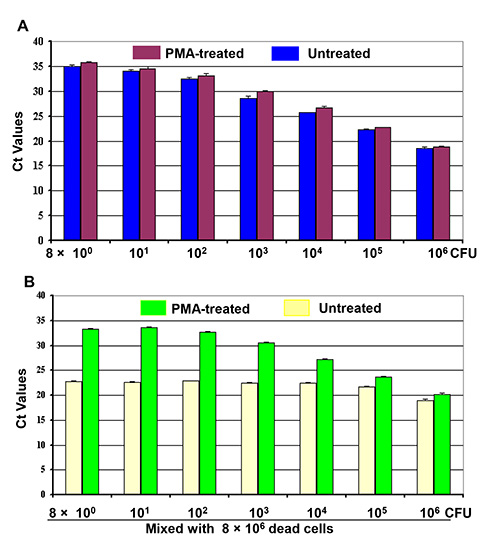
図2。 。PMA-qPCRの細胞培養の10倍希釈液(8×10 0から 8×10 6 CFU)の4組によるライブ/死細胞の混合物からの生細胞の分化が示されているように行われました。第二の細胞希釈DNA抽出の前にあったか、または未処理の細胞希釈物(A)第一セットは、PMAで処理した。(B)8×10 6個の死んだ細胞を示すように生/死細胞混合物の二組を作るために細胞希釈の第3および第4のセットと混合した。細胞混合物の一組は、PMAで処理し、他のセットは、DNA抽出の前に、PMAなしで処理した。各バーは、±SD三連の実験の平均CT値を表す。
| 生体 | 血清型 | 試験した株の数 |
| E.コリ | 2006ほうれん草O157:H7アウトブレイクの分離株 | 186 |
| 2006タコベルO157:H7の分離株 | 58 | |
| 2006タコスジョンO157:H7の分離株 | 11 | |
| 他のO157からDMB株コレクション:H7のアウトブレイク | 112 | |
| O104:H4 | 3 | |
| O26 | 18 | |
| O103 | 13 | |
| O111 | 24 | |
| O121 | 2 | |
| O45 | 5 | |
| O145 | 9 | |
| O113 | 2 | |
| O91 | 1 | |
| O55 | 9 | |
| その他 | 19 | |
| サルモネラ | チフス菌 | 1 |
| ニューポート | 2 | |
| 志賀赤痢菌 | 6 | 2 |
| 赤痢ソンネ | 不明 | 2 |
| 赤痢菌 | 4 | 1 |
| 赤痢菌boydii | 1 | 1 |
| 合計 | 481 | |
TE.同定するためのリアルタイムPCRアッセイで検証するために使用できる1。 株大腸菌 O157:H7。
ディスカッション
研究の主な目的は、EにORF Z3276、独特の使用を含む大腸菌 O157:H7、定量PCRアッセイのための唯一のバイオマーカーとして。現在では、ほとんどのqPCRアッセイはそのようなのStx1、STX2、及びEAEA 1,2,5,6またはそのようにuidA 3,4、rfbEとはfliC遺伝子 の8,9などの共有表現型の遺伝子、病原性遺伝子をターゲットとしている。これらの遺伝子は、異なる種に存在しているかだけでなく、16の株として時折、種または株は間違いなく、このようなSTX 1およびSTX 2遺伝子として、病原性遺伝子(S)によって識別することができません。標的遺伝子についてrfbEとはfliCを使用して、両方の遺伝子は、完全な17の識別のために使用することが必要である。バイオマーカーとしてのORF Z3276を用いて、この定量PCRアッセイは高感度かつ特異的なアッセイ( 表1)であることが実証されている。このアッセイは、O157を含む、ストリンジェントな包括性および排他性試験に供された:H7株(n = 3のサルモネラや赤痢菌など67)、100以上の非O157の菌株、および他の病原性種、。すべてO157:H7株を調べ正に検出され、交差反応性は、ORF Z3276は、E.を検出するための固有かつ強力なバイオマーカーであることを示す、非O157株( 表1)から見られなかった大腸菌 O157:H7。
試験の他の目的は、選択的にDNA抽出の前にPMA処理により死細胞から生細胞を検出することである。 PMAは妥協膜と死細胞に浸透し、DNAに結合し、したがって、PCR 13における死細胞のDNA増幅を阻害することができます。我々は、PMA-処理手順を修飾し、手順96ウェルプレートフォーマットに適応している。露光の右強度は、最適な架橋効果を得ることが不可欠である。以前は、マイクロチューブは、このステップ13〜15で使用されている。しかし、別々のマイクロチューブライトEXで取り扱いが困難であるposure工程、これらのチューブは、最高のクロス好みを得ることは絶対に透過的ではありません。 96ウェルプレートを用いて、PMA-処理の効率を向上させる。これらの変化は、いくつかの方法でアッセイに影響を与える可能性がある:それらは、特に、多数のサンプルを用いて、より簡単に徹底的かつ均一な架橋効果を得ることが、処理された試料は、多数の検出のためのそれを可能にするために別のプレートから移動させることができるサンプルで、彼らは、全体のプロセスのための自動化の可能性と、このPMA-定量PCRアッセイを提供する。このPMA-定量PCRアッセイの制限は、PMA処理が幾分PCRアッセイのコストを増加させ、わずかにPCRアッセイの感度を減少させることである。
開示事項
著者らは、開示することは何もありません。
謝辞
当社は、資金調達の一部を提供するための米国国土安全保障省に感謝します。
資料
| Name | Company | Catalog Number | Comments |
| AB 7900HT Fast Real-time PCR system | ABI | ||
| 2x Universal master mix | ABI | 4304437 | |
| PrepMan Ultra | ABI | 4318930 | |
| Primer Express | ABI | ||
| Probes | ABI | Top of Form | |
| PMA | Biodium | 40013 | |
| Puregen cell and tissue kit | Qiagene | ||
| DU530 spectrophotometer | Beckman | ||
| Spectrophotometer | NanoDrop Technology | ND-1000 | |
| 650 W Halogen light source | Britek | H8061S | |
| Otptical Adhesive film | ABI | 4311971 | |
| Optical 96-well reaction plate | ABI | 4316813 |
参考文献
- Barletta, F., et al. Validation of five-colony pool analysis using multiplex real-time PCR for detection of diarrheagenic Escherichia coli. J. Clin. Microbiol. 47, 1915-1917 (2009).
- Gannon, V. P., King, R. K., Kim, J. Y., Thomas, E. J. Rapid and sensitive method for detection of Shiga-like toxin-producing Escherichia coli in ground beef using the polymerase chain reaction. Appl. Environ. Microbiol. 58, 3809-3815 (1992).
- Cebula, T. A., Payne, W. L., Feng, P. Simultaneous identification of strains of Escherichia coli serotype O157:H7 and their Shiga-like toxin type by mismatch amplification mutation assay-multiplex PCR. J. Clin. Microbiol. 33, 248-250 (1995).
- Li, Y., Mustapha, A. Simultaneous detection of Escherichia coli O157:H7, Salmonella, and Shigella in apple cider and produce by a multiplex PCR. J. Food. Prot. 67, 27-33 (2004).
- Schmidt, H., et al. Differentiation in virulence patterns of Escherichia coli possessing eae genes. Med. Microbiol. Immunol. 183, 23-31 (1994).
- Fratamico, P. M., Sackitey, S. K., Wiedmann, M., Deng, M. Y. Detection of Escherichia coli O157:H7 by multiple PCR. J. Clin. Microbiol. 33, 2188-2191 (1995).
- Desmarchelier, P. M., Bilge, S. S., Fegan, N., Mills, L., Vary, J. C., Tarr, P. I. A PCR specific for Escherichia coli O157 based on the rfb locus encoding O157 lipopolysaccharide. J. Clin. Microbiol. 36, 1801-1804 (1998).
- Fields, P. I., Blom, K., Hughes, H. J., Helsel, L. O., Feng, P., Swaminathan, B. Molecular characterization of the gene encoding H antigen in Escherichia coli and development of a PCR-restriction fragment length polymorphism test for identification of E. coli O157:H7 and O157:NM. J. Clin. Microbiol. 35, 1066-1070 (1997).
- Fricker, M., Messelhäußer, U., Bushch, U., Scherer, S., Ehling-Schulz, M. Diagnostic real-time PCR assays for detection of emetic Bacillus cereus strains in foods and recent food-borne outbreaks. Appl. Environ. Microbiol. 73, 1892-1898 (2007).
- Li, B., Chen, J. Q. Real-time PCR methodology for selective detection of viable Escherichia coli O157:H7 cells by targeting Z3276 as a genetic marker. Appl Environ. Microbiol. 78, 5297-5304 (2012).
- Perna, N. T., et al. Genome sequence of enterohaemorrhagic Escherichia coli O157:H7. Nature. 409, 529-533 (2001).
- Wang, S., Levin, R. E. Discrimination of viable Vibrio vulnificus cells from dead cells in real-time PCR. J. Microbiol. Methods. 64, 1-8 (2006).
- Nocker, A., Cheung, C. Y., Camper, A. K. Comparison of propidium monoazide with ethidium monoazide for differentiation of live vs. dead bacteria by selective removal of DNA from dead cells. J. Microbiol. Methods. 67, 310-320 (2006).
- Nocker, A., Sossa-Fernandez, P., Burr, M. D., Camper, A. K. Use of propidium monoazide for live/dead distinction in microbial ecology. Appl. Environ. Microbiol. 73, 5111-5117 (2007).
- Cawthorn, D. M., Witthuhn, R. C. Selective PCR detection of viable Enterobacter sakazakii cells utilizing propidium monoazide or ethidium bromide monoazide. J. Appl. Microbiol. 105, 1178-1185 (2008).
- Chassagne, L., Pradel, N., Robin, F., Livrelli, V., Bonnet, R., Delmas, J. Detection of stx1, stx2, and eae genes of enterohemorrhagic Escherichia coli using SYBR Green in a real-time polymerase chain reaction. Diagn. Microbiol. Infect. Dis. 64, 98-101 (2009).
- Liu, Y., Gilchrist, A., Zhang, J., Li, X. F. Detection of viable but nonculturable Escherichia coli O157:H7 bacteria in drinking water and river water. Appl. Environ. Microbiol. 74, 1502-1507 (2008).
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請さらに記事を探す
This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved