Method Article
Détection de Live
Dans cet article
Résumé
Une analyse qPCR est développé pour la détection de Escherichia coli O157: H7 de cibler un marqueur génétique unique, Z3276. La qPCR a été combiné avec propidium monoazide (PMA) de traitement pour la détection de cellules vivantes. Ce protocole a été modifié et adapté à un format de plaque à 96 puits pour une manipulation aisée et uniforme de nombreux échantillons
Résumé
Un cadre de lecture ouverte unique (ORF) Z3276 a été identifié comme un marqueur génétique spécifique de E. coli O157: H7. Un essai qPCR a été développé pour la détection de E. coli O157: H7 en ciblant ORF Z3276. Avec ce dosage, on peut détecter aussi peu que quelques copies du génome de l'ADN de E. coli O157: H7. La sensibilité et la spécificité du test ont été confirmés par des tests de validation intensifs avec un grand nombre de E. coli O157: H7 souches (n = 369) et des souches non-O157 (n = 112). En outre, nous avons combiné propidium monoazoture (PMA) avec la procédure de protocole de qPCR nouvellement développé pour la détection sélective de cellules vivantes à partir de cellules mortes. L'amplification de l'ADN à partir de cellules mortes PMA-traitées a été presque complètement inhibée par opposition à l'amplification de l'ADN pratiquement pas affecté par les cellules vivantes traitées au PMA. En outre, le protocole a été modifié et adapté à un format de plaque à 96 puits pour une manipulation facile et uniforme d'un grand nombre d'échantillons. Tsa méthode devrait avoir un impact sur microbiologique précis et la surveillance épidémiologique de la sécurité alimentaire et la source de l'environnement.
Introduction
La PCR est une technique couramment utilisée pour la détection de E. coli O157: H7 dans les échantillons alimentaires et environnementaux. L'identification de biomarqueurs spécifiques pour E. coli O157: H7 est l'un des facteurs essentiels pour la détection de succès dans des essais de PCR. Les biomarqueurs couramment utilisés dans des essais de PCR pour la détection de E. coli O157: H7 comprennent des toxines Shiga (stx 1 et stx 2) 1,2, 3,4 uidA, eaeA 5,6, rfbE 7, et fliC gènes 8,9. Ces marqueurs peuvent fournir une efficacité variable pour l'identification, mais la plupart des marqueurs biologiques ne peuvent être utilisés en tant que biomarqueur unique pour la détection de E. coli O157: H7. Cette insuffisance de la puissance de la différenciation des gènes cibles nécessite biomarqueur (s) plus précis et plus fort pour l'identification de E. coli O157: H7 10.
De nombreuses sondes pour les gènes hypothétiques ou des cadres de lecture ouverts (ORF) dans E. coliO157: H7 11 ont été conçus pour qPCR analyse sur la base des indices de puces ADN de génotypage de notre laboratoire et par la recherche dans la base de données GenBank du Centre national d'information sur la biotechnologie. La séquence de l'ORF Z3276, qui est un gène fimbriae 11, a été choisi pour l'analyse qPCR. Par la suite, un test PCR quantitative a été développé par le biais de nombreux essais sur les paramètres de la PCR tels que les concentrations des amorces et des sondes, ainsi que les températures d'hybridation et d'extension (données non présentées). Après inclusivité d'incitation et d'exclusivité des tests sur les souches de référence tels que E. coli O157: H7, non-O157 et Shigella dans la qPCR, l'ORF Z3276 a été identifié comme un marqueur génétique spécifique et forte pour l'identification de E. coli O157: H7. Ensuite, nous développons une nouvelle méthode qPCR utilisant ce biomarqueur unique pour l'identification de E. coli O157: H7.
Un autre défi pour la détection de E. coli O157: H7 dans contaminé foods et de l'environnement est une détermination précise des cellules vivantes à partir d'échantillons. La présence prolongée de l'ADN des cellules mortes et l'incapacité de différencier viabilité en PCR pourrait causer des lectures faussement positifs, à une surestimation du nombre de cellules vivantes dans la détection. Par conséquent, ce qui limite l'utilisation de PCR dans la détection précise de pathogènes d'origine alimentaire 12. Une nouvelle approche pour différencier l'ADN des cellules mortes a été pris en combinant propidium monoazide (PMA) un traitement à la procédure PCR au cours des dernières années 13-15. La PMA est capable d'aller à l'intérieur des cellules mortes, et s'intercaler dans l'ADN lors de l'exposition à la lumière, mais elle ne peut pas aller à l'intérieur de cellules vivantes à réagir avec l'ADN des cellules vivantes. Ainsi, dans des mélanges PMA-traités de cellules vivantes et mortes, l'ADN des cellules mortes ne peut pas être amplifiée par PCR en 13. Nous avons combiné le traitement PMA avec le test qPCR nouvellement développé pour détecter sélectivement en direct E. coli O157: H7 cellules. De plus, nous avons fait le PMA-qPCR uneéssayé possible pour la détection à haut débit.
Protocole
Une. Souches bactériennes et Modèle Préparation ADN
- Cultiver des souches bactériennes de E. coli O157: H7, non-O157, et d'autres espèces pathogènes comme Salmonella et Shigella, à 37 ° C pendant une nuit sous agitation.
- Extraire l'ADN à partir de cultures bactériennes en utilisant le kit approprié (voir tableau des matériaux) et en suivant les instructions du fabricant.
- Mesurer la densité optique (OD 260) pour déterminer les concentrations d'ADN à l'aide d'un spectrophotomètre.
2. Primer et conception de la sonde pour le test qPCR
- E. coli O157: H7 sont des séquences de numéro d'accession GenBank AE00517411. amorces de conception et sonde en utilisant un logiciel approprié pour l'amorce et la conception de la sonde pour la PCR en temps réel (voir le tableau des matériaux pour plus de détails).
Les séquences des amorces et la sonde sont les suivantes: Z3276-avant (F) 5'-TATTCCGCGATGCTTGTTTTT-3 '
Z3276-marche arrière (R) 5'-ATTATCTCACCAGCAAACTGGCGG-3 '
Z3276-Probe, FAM-CCGCAATCTTTCC-MGBNFQ - Reconstituer la poudre d'apprêt à l'eau, résultant en une concentration de 10 uM en tant que solution de travail de l'amorce.
- Diluer les sondes à 10 uM en tant que solutions de travail de la sonde. Les sondes marquées varient en puissance avec des lots. Par conséquent, titrer chaque lot de sondes marquées avant d'utiliser pour des performances optimales qPCR.
3. Réglage des qPCR Conditions de l'essai
- Préparer le mélange de réaction, qui se compose de 25,0 pi de 2x en temps réel par PCR Master Mix, 200 nM d'amorce directe, 200 nM d'amorce inverse, et 100 nM de sonde.
- Ajouter 5 ul de l'ADN de l'échantillon (100 pg) et un volume approprié d'eau pour atteindre un volume final de 50 ul.
- Pour un contrôle sans modèle, utiliser 5 pi d'eau pour remplacer l'échantillon d'ADN.
- Définir les conditions de qPCR comme suit: 95 ° C pendant 10 minutes pour l'activation de TaqMan, suivie de 40 cycles de 95 ° C pendant 10 secondes pour la dénaturation et 60 et# 176; C pendant 1 minute pour l'hybridation / extension.
4. qPCR test de sensibilité
- Faire une série de dilution de 10 fois à partir d'une culture de milieu de la phase exponentielle (DO600 = 0,5; environ 1,5 x 10 8 CFU / ml par placage).
- Ajouter 100 ml de dilutions de cellules sur une plaque à 96 puits en triple.
- Centrifuger la plaque à 2500 g pendant 10 min.
- Ajouter 50 ml de solution d'extraction de l'ADN dans chaque puits.
- culots cellulaires d'Remettre en suspension avec une pipette à canaux multiples.
- Sceller la plaque avec un film, faire bouillir la plaque dans un bain d'eau pendant 10 min, et centrifuger à 2500 g pendant 2 min.
5. Préparation des mélanges vivants et morts cellulaires pour le traitement de PMA
- Développez 10 ml de E. coli O157: H7 à 37 ° C jusqu'à la phase semi-exponentielle de la culture et de diviser en deux parties aliquotes.
- Faire bouillir une fraction aliquote de cellules pendant 10 min dans un bain d'eau pour les cellules mortes et laisser l'autre portion aliquote de cellules vivantes.
- Vérifiez lare n'y a pas de cellules vivantes de l'aliquote tué par la chaleur par l'étalement des cellules sur des plaques d'agar LB.
- Prendre 2 ml des cellules vivantes et tuées par la chaleur et d'ajuster la concentration de 8 x 10 6 UFC / ml avec du milieu LB. Créer quatre séries de dilutions de cellules allant de 8 x 10 0 à 8 x 10 6 UFC / ml.
- Utilisez les deux premières séries de dilutions de cellules pour des cellules vivantes traitées avec de la PMA ou non traitée.
- Pour les troisième et quatrième séries de dilutions de cellules, ajouter 8 x 10 6 cellules mortes pour chaque dilution de la cellule (8 x 10 0 - 8 x 10 6) de faire des mélanges de cellules vivantes et mortes.
- Traiter la troisième série de dilution avec du PMA et utiliser la quatrième série de dilution pour le contrôle.
6. Le traitement des cellules avec de la PMA
- Dissoudre PMA dans le diméthylsulfoxyde (ou l'eau) pour obtenir une solution 10 mM d'actions et stockés à -20 ° C dans l'obscurité.
- Aliquote de 400 ul de cellules vivantes, les cellules mortes, et le mélange de cellules vivantes et mortes danstrois microtubes séparés.
- Ajouter 2,0 ml de PMA 10 mM à chaque aliquote de cellules à une concentration finale de 50 uM.
- Incuber les échantillons à température ambiante pendant 5 min dans l'obscurité, en secouant doucement plusieurs fois, trois secondes à chaque fois.
- Ajouter 100 ul d'échantillons d'une plaque à 96 puits.
- Sceller la plaque avec un film optique et mettre la plaque sur la glace.
- Placer la plaque de 20 cm d'une source de lumière W 650 d'halogène et exposer pendant 2 min pour exposition à la lumière.
- Centrifuger la plaque à 2500 g pendant 10 min, jetez doucement le surnageant et égoutter la plaque sur un morceau de papier absorbant soigneusement.
- culots cellulaires d'Remettre en suspension dans 50 ul de solution d'extraction de l'ADN par pipetage de haut en bas vingt fois avec un multi-canal Pipetman.
- Sceller la plaque avec un film, faire bouillir pendant 10 min dans un bain d'eau, et centrifuger à 2500 g pendant 2 min. Le surnageant de la plaque est de l'ADN à partir de cellules traitées avec du PMA et prêt pour la qPCR.
- Effectuer qPCR que descrilit au-dessus, en utilisant 5 ul d'ADN.
Résultats
La détection de E. coli O157: H7 par qPCR utilisant ORF Z3276
L'un des facteurs clés de cette analyse est la sensibilité et la spécificité de la sonde Z3276 utilisé dans la qPCR. Il peut détecter aussi peu que quelques copies du génome de l'ADN de E. coli O157: H7 comme représenté sur la Figure 1A. Sur les 120 souches non-O157 examinés, y compris les six grandes souches non-O157 STEC O104: souches, H4, Salmonella et souches de Shigella, aucune réactivité croisée n'a été trouvée, comme indiqué dans le tableau 1.
Combinaison du traitement PMA avec qPCR
Incubation E. coli O157: H7 des cellules avec PMA (50 mM) pendant 5 minutes à l'obscurité et l'exposition à la lumière pendant 2 min ont été choisies pour des conditions de traitement au PMA. En outre, nous avons modifié PMA procédure de traitement. Par rapport aux différents micro-tubes transparents utilisés dans l'étape de réticulation, une plaque de 96 puits a été trouvé pour être plus efficace d'atteindre l'effet de reticulation optimal et plus pratique que la manipulation d'un grand nombre de tubes d'échantillons au cours du processus d'exposition à la lumière.
Amplification de l'ADN à partir de cellules vivantes et mortes PMA-traités dans qPCR
Les effets de l'inhibition médiée par le PMA de l'amplification d'ADN à partir de cellules mortes à ce dosage qPCR ont été présentés dans la figure 1 à l'aide d'ADN provenant de deux dilutions de 10 fois de cellules vivantes ou mortes série. Les résultats montrent que les courbes produites par les cellules vivantes traitées avec du PMA (courbe rouge) et sans PMA (courbe bleu) semblent linéaire et presque identique à une autre. Différences de valeurs de CT légères ont été montrées entre les cellules vivantes traitées avec du PMA et sans PMA (Figure 1A), ce qui suggère que le traitement de PMA a peu d'effet sur l'amplification de l'ADN des cellules vivantes dans la qPCR. Mais un 15 - différence de valeur de CT (32000 fois) a été montré entre les cellules mortes traitées avec du PMA et sans PMA, démonstrating que le traitement PMA supprimée efficacement l'amplification d'ADN à partir des cellules mortes (figure 1B).
Détection de cellules vivantes à partir de mélanges de cellules vivantes et mortes dans qPCR
Deux séries de dilutions de cellules vivantes (8 x 10 0 - 8 x 6 10 UFC) ont été traités avec de la PMA ou sans PMA pour détecter les cellules vivantes de morts mélanges live / cellulaires. Les résultats de qPCR (figure 2A) ont démontré une tendance progressive inverse parallèle de valeurs CT comme liée au nombre de cellules vivantes traitées (barres pourpres) aux échantillons non traités (barres bleues). Les cellules vivantes traitées ont donné des valeurs de CT d'un peu plus élevées que celles de cellules vivantes non traités. Une tendance progressive inverse similaire dans les valeurs CT a été observée avec le nombre réel de cellules vivantes dans les mélanges morts vivants / cellulaires traitées avec de la PMA (barres vertes) ainsi que dans la figure 2B. En outre, cette tendance à la baisse des valeurs de CT étaitobservé avec le nombre de cellules vivantes dans les mélanges en dépit de la présence de 106 cellules mortes. Ces résultats ont montré que les valeurs de CT des mélanges vivants / morts cellulaires représentent l'ADN des cellules vivantes seulement, alors que l'amplification de l'ADN des cellules mortes a été efficacement supprimée par traitement au PMA. Mais, les valeurs de CT de mélanges vivants / morts cellulaires traitées avec du PMA ont fluctué et n'ont pas reflété les nombres de cellules vivantes dans les mélanges de la figure 2B (barres jaunes).
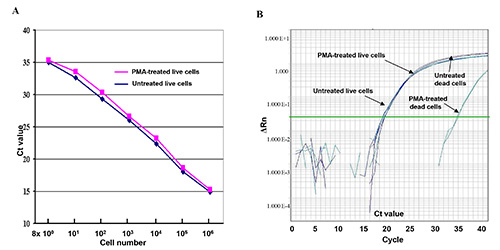
Figure 1. Vivent et les cellules mortes traitées avec de la PMA ou sans PMA dans l'essai qPCR. (A) Une série de 10 fois dilué direct E. coli O157: H7 dilutions cellulaires (8 x 10 0 - 8 x 10 6 UFC) ont été traités avec de la PMA ou sans PMA.Les valeurs CT représentent les valeurs CT moyennes des trois exemplaires dans l'essai qPCR. (B) Comparaison de l'amplification d'ADN de cellules vivantes et les cellules mortes traitées avec du PMA et sans PMA dans la comparaison de q essai qPCR. ARn se réfère à la fluorescence changement d'intensité. Cliquez ici pour agrandir l'image.
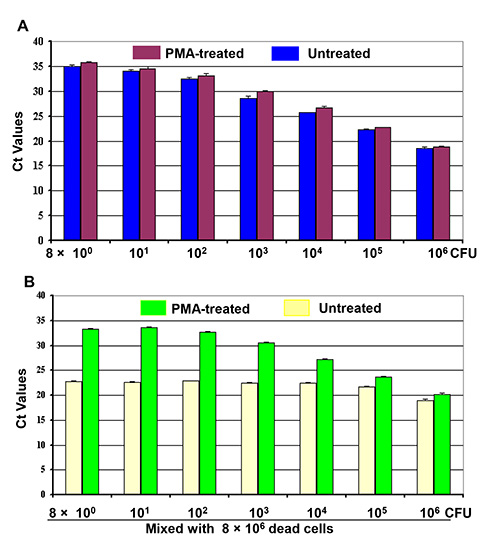
Figure 2. La différenciation des cellules vivantes à partir de mélanges vivants / morts cellulaires par PMA-qPCR Quatre séries de dilutions de 10 fois de cultures de cellules (8 x 10 0-8 x 10 6 CFU). Ont été effectués comme indiqué. (A) Première série de dilution des cellules a été traitée avec du PMA pendant la deuxième dilution cellulaire était ou non traitée avant l'extraction de l'ADN.(B) 8 x 10 6 cellules mortes ont été mélangés avec les troisième et quatrième séries de dilutions de cellules pour effectuer deux séries de mélanges vivants / morts cellulaires comme indiqué. Une série de mélanges de cellules a été traitée avec du PMA et de l'autre ensemble a été traité sans PMA avant l'extraction de l'ADN. Chaque barre représente les valeurs de CT moyen d'une expérience triple ± SD.
| Organisme | Sérotype | Nombre de souches testées |
| E. coli | 2006 épinards O157: H7 isolats de | 186 |
| 2006 Taco Bell O157: H7 isolats | 58 | |
| 2006 Taco John O157: H7 isolats | 11 | |
| collections de souches de DMB de l'autre O157: H7 | 112 | |
| O104: H4 | 3 | |
| O26 | 18 | |
| O103 | 13 | |
| O111 | 24 | |
| O121 | 2 | |
| O45 | 5 | |
| O145 | 9 | |
| O113 | 2 | |
| O91 | 1 | |
| O55 | 9 | |
| Autre | 19 | |
| Salmonella | Typhi | 1 |
| Newport | 2 | |
| Shigella dysenteriae | 6 | 2 |
| Shigella sonnei | Inconnu | 2 |
| Shigella flexneri | 4 | 1 |
| Shigella boydii | 1 | 1 |
| Total | 481 | |
Tmesure 1. souches utilisées pour la validation de PCR en temps réel pour l'identification de E. coli O157: H7.
Discussion
Un objectif principal de l'étude implique l'utilisation d'un Z3276 ORF, un unique E. coli O157: H7, en tant que biomarqueur pour une seule analyse qPCR. À l'heure actuelle, la plupart des analyses qPCR ciblent des gènes de virulence, comme stx1, stx2, et eaeA 1,2,5,6 ou gènes phénotypiques communs, comme uid 3,4, rfbE et gènes fliC 8,9. De temps en temps, une espèce ou une souche ne peuvent pas être identifiés définitivement par le gène (s) de virulence, tels que stx 1 et stx deux gènes, comme les gènes sont présents dans différentes espèces ou souches 16 ainsi. Utilisation rfbE et fliC de gènes cibles, les deux gènes sont nécessaires pour être utilisé pour l'identification complète 17. Utilisation ORF Z3276 en tant que biomarqueur, ce dosage qPCR a été démontrée comme étant dosage sensible et spécifique (tableau 1). Ce test a été soumis à inclusivité et l'exclusivité des tests rigoureux, y compris O157: H7 (n = 367), plus de 100 souches non-O157, et d'autres espèces pathogènes comme Salmonella et Shigella. Tous O157: H7 ont été examinés de façon positive détectés, et pas de réactivité croisée n'a été trouvée à partir des souches non-O157 (tableau 1), ce qui indique que l'ORF Z3276 est un biomarqueur unique et forte pour la détection de E. coli O157: H7.
L'autre objectif de l'étude est de détecter sélectivement les cellules vivantes des cellules mortes par traitement au PMA avant extraction de l'ADN. PMA peut pénétrer dans les cellules mortes de la membrane est compromise et la lier à l'ADN et inhibe ainsi l'amplification de l'ADN des cellules mortes dans les 13 PCR. Nous avons modifié la procédure PMA-traitement, et adapté à un format de plaque à 96 puits pour la procédure. L'intensité droit de exposition à la lumière est essentielle pour obtenir un effet optimal réticulation. Auparavant, les microtubes ont été utilisés dans cette étape de 13 à 15. Cependant, microtubes séparés sont difficiles à manipuler dans l'ex lumièrel'étape de Posure, et ces tubes ne sont pas absolument transparent pour obtenir le meilleur goût transversale. En utilisant une plaque de 96 puits, nous avons amélioré l'efficacité de la PMA-traitement. Ces modifications peuvent influer sur le dosage de plusieurs façons: ils rendent plus facile d'obtenir un effet de réticulation complète et uniforme, en particulier avec de nombreux échantillons, les échantillons traités peuvent être déplacés d'une plaque à l'autre pour rendre possible la détection d'un grand nombre d'échantillons, et ils offrent ce test PMA-qPCR avec un potentiel d'automatisation pour l'ensemble du processus. La limitation de ce dosage PMA-qPCR est que le traitement augmente le coût du PMA de PCR et légèrement réduit quelque peu la sensibilité du dosage par PCR.
Déclarations de divulgation
Les auteurs n'ont rien à révéler.
Remerciements
Nous remercions le ministère de la Sécurité intérieure des États-Unis pour fournir une partie du financement.
matériels
| Name | Company | Catalog Number | Comments |
| AB 7900HT Fast Real-time PCR system | ABI | ||
| 2x Universal master mix | ABI | 4304437 | |
| PrepMan Ultra | ABI | 4318930 | |
| Primer Express | ABI | ||
| Probes | ABI | Top of Form | |
| PMA | Biodium | 40013 | |
| Puregen cell and tissue kit | Qiagene | ||
| DU530 spectrophotometer | Beckman | ||
| Spectrophotometer | NanoDrop Technology | ND-1000 | |
| 650 W Halogen light source | Britek | H8061S | |
| Otptical Adhesive film | ABI | 4311971 | |
| Optical 96-well reaction plate | ABI | 4316813 |
Références
- Barletta, F., et al. Validation of five-colony pool analysis using multiplex real-time PCR for detection of diarrheagenic Escherichia coli. J. Clin. Microbiol. 47, 1915-1917 (2009).
- Gannon, V. P., King, R. K., Kim, J. Y., Thomas, E. J. Rapid and sensitive method for detection of Shiga-like toxin-producing Escherichia coli in ground beef using the polymerase chain reaction. Appl. Environ. Microbiol. 58, 3809-3815 (1992).
- Cebula, T. A., Payne, W. L., Feng, P. Simultaneous identification of strains of Escherichia coli serotype O157:H7 and their Shiga-like toxin type by mismatch amplification mutation assay-multiplex PCR. J. Clin. Microbiol. 33, 248-250 (1995).
- Li, Y., Mustapha, A. Simultaneous detection of Escherichia coli O157:H7, Salmonella, and Shigella in apple cider and produce by a multiplex PCR. J. Food. Prot. 67, 27-33 (2004).
- Schmidt, H., et al. Differentiation in virulence patterns of Escherichia coli possessing eae genes. Med. Microbiol. Immunol. 183, 23-31 (1994).
- Fratamico, P. M., Sackitey, S. K., Wiedmann, M., Deng, M. Y. Detection of Escherichia coli O157:H7 by multiple PCR. J. Clin. Microbiol. 33, 2188-2191 (1995).
- Desmarchelier, P. M., Bilge, S. S., Fegan, N., Mills, L., Vary, J. C., Tarr, P. I. A PCR specific for Escherichia coli O157 based on the rfb locus encoding O157 lipopolysaccharide. J. Clin. Microbiol. 36, 1801-1804 (1998).
- Fields, P. I., Blom, K., Hughes, H. J., Helsel, L. O., Feng, P., Swaminathan, B. Molecular characterization of the gene encoding H antigen in Escherichia coli and development of a PCR-restriction fragment length polymorphism test for identification of E. coli O157:H7 and O157:NM. J. Clin. Microbiol. 35, 1066-1070 (1997).
- Fricker, M., Messelhäußer, U., Bushch, U., Scherer, S., Ehling-Schulz, M. Diagnostic real-time PCR assays for detection of emetic Bacillus cereus strains in foods and recent food-borne outbreaks. Appl. Environ. Microbiol. 73, 1892-1898 (2007).
- Li, B., Chen, J. Q. Real-time PCR methodology for selective detection of viable Escherichia coli O157:H7 cells by targeting Z3276 as a genetic marker. Appl Environ. Microbiol. 78, 5297-5304 (2012).
- Perna, N. T., et al. Genome sequence of enterohaemorrhagic Escherichia coli O157:H7. Nature. 409, 529-533 (2001).
- Wang, S., Levin, R. E. Discrimination of viable Vibrio vulnificus cells from dead cells in real-time PCR. J. Microbiol. Methods. 64, 1-8 (2006).
- Nocker, A., Cheung, C. Y., Camper, A. K. Comparison of propidium monoazide with ethidium monoazide for differentiation of live vs. dead bacteria by selective removal of DNA from dead cells. J. Microbiol. Methods. 67, 310-320 (2006).
- Nocker, A., Sossa-Fernandez, P., Burr, M. D., Camper, A. K. Use of propidium monoazide for live/dead distinction in microbial ecology. Appl. Environ. Microbiol. 73, 5111-5117 (2007).
- Cawthorn, D. M., Witthuhn, R. C. Selective PCR detection of viable Enterobacter sakazakii cells utilizing propidium monoazide or ethidium bromide monoazide. J. Appl. Microbiol. 105, 1178-1185 (2008).
- Chassagne, L., Pradel, N., Robin, F., Livrelli, V., Bonnet, R., Delmas, J. Detection of stx1, stx2, and eae genes of enterohemorrhagic Escherichia coli using SYBR Green in a real-time polymerase chain reaction. Diagn. Microbiol. Infect. Dis. 64, 98-101 (2009).
- Liu, Y., Gilchrist, A., Zhang, J., Li, X. F. Detection of viable but nonculturable Escherichia coli O157:H7 bacteria in drinking water and river water. Appl. Environ. Microbiol. 74, 1502-1507 (2008).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon