Method Article
Обнаружение эфир
В этой статье
Резюме
КПЦР был разработан анализ для выявления кишечной палочки O157: H7 таргетирования уникальный генетический маркер, Z3276. КПЦР сочеталась с пропидий моноазид (PMA) лечения для обнаружения живых клеток. Этот протокол был изменен и приспособлен к формату 96-луночного планшета для легкой и последовательной обработке многочисленных проб
Аннотация
Уникальная открытая рамка считывания (ORF) Z3276 был идентифицирован как специфический генетический маркер для Е. палочки O157: H7. КПЦР был разработан анализ для обнаружения E. палочки O157: H7, направляя ORF Z3276. С помощью этого анализа, мы можем обнаружить по цене от нескольких копий генома ДНК E. палочки O157: H7. Чувствительность и специфичность анализа были подтверждены интенсивных оценочных испытаний с большим количеством Е. палочки O157: H7 (п = 369) и не-O157 штаммы (п = 112). Кроме того, мы объединили процедуру пропидий моноазид (PMA) с вновь разработанной протокола КПЦР для селективного детектирования живых клеток от мертвых клеток. Амплификация ДНК из обработанных РМА-мертвых клеток была почти полностью ингибируется в отличие от практически не зависит амплификации ДНК от PMA-обработанных живых клеток. Кроме того, протокол был изменен и приспособлен к формату 96-луночного планшета для легкой и последовательной обработки большого количества образцов. Тего метод, как ожидается, оказывают влияние на точной микробиологических и эпидемиологического мониторинга безопасности пищевых продуктов и природного источника.
Введение
ПЦР является распространенный метод для обнаружения E. палочки O157: H7 из продуктов питания и проб окружающей среды. Определение конкретных биомаркеров для Е. палочки O157: H7 является одним из ключевых факторов для успешного обнаружения в ПЦР-анализов. В биомаркеры, обычно используемые в ПЦР для выявления E. палочки O157: H7 включают Shiga токсины (STX 1 и STX 2) 1,2, uidA 3,4, EAEA 5,6, rfbE 7, и Флик гены 8,9. Эти биомаркеры могут обеспечить переменную для идентификации эффективность, однако большинство биомаркеров не может быть использована в качестве уникального биомаркера для определения E. палочки O157: H7. Эта неадекватность в дифференциации власти генов-мишеней требует более конкретного и сильного биомаркеров (ы) для идентификации E. палочки O157: H7 10.
Многочисленные датчики для генов или гипотетических открытых рамок считывания (ORFs) в E. палочкаO157: H7 11 были разработаны для КПЦР анализа, основанного на ключи от генотипирования ДНК микрочипов из нашей лаборатории и путем поиска в базе данных GenBank Национального центра биотехнологической информации. Последовательность Z3276 ORF, который представляет собой ген фимбриальной 11, был выбран для КПЦР анализа. Впоследствии анализ КПЦР была разработана на основе многочисленных испытаний на параметры, такие как ПЦР концентраций праймеров и зондов, а также отжига и расширение температурах (данные не показаны). После стимулом всеохватности и исключительных испытаний на контрольных штаммов, таких как E. палочки O157: H7, не O157 и Shigella напряжения в кПЦР, ORF Z3276 был идентифицирован как специфического и устойчивого генетического маркера для идентификации E. палочки O157: H7. Тогда мы разработать новый метод КПЦР, используя эту уникальную биомаркеров для идентификации E. палочки O157: H7.
Еще одной проблемой в выявлении E. палочки O157: H7 в загрязненных FOОРВ и среда точное определение живых клеток из образцов. Продлен наличие ДНК от мертвых клеток и неспособности различать жизнеспособность в ПЦР-анализе может привести ложноположительные показания, в результате чего завышению цифр живых клеток в обнаружении. Следовательно, это ограничивает использование ПЦР в точного обнаружения пищевых патогенов 12. Новый подход к дифференциации ДНК мертвых клеток были получены путем объединения пропидий моноазид (PMA) обработку методом ПЦР в последние годы 13-15. PMA может войти внутрь от мертвых клеток, и интеркаляции в ДНК при воздействии света, но она не может войти внутрь живых клеток реагировать с ДНК живых клеток. Таким образом, в PMA-обработанных смесей живых и мертвых клеток, ДНК мертвых клеток не может быть усилен в ПЦР 13. Мы комбинированное лечение PMA с недавно разработанной КПЦР анализа выборочно обнаружить живой E. палочки O157: H7 клетки. Кроме того, мы сделали PMA-КПЦР AДни рождения Ssay возможно для обнаружения высокой пропускной способности.
протокол
1. Штаммы бактерий и ДНК Подготовка шаблона
- Расти бактериальных штаммов E. палочки O157: H7, не O157 и другие патогенные виды, такие как сальмонелла и Shigella, при 37 ° С в течение ночи при встряхивании.
- Извлечение ДНК из бактериальных культур с использованием соответствующего набора (см. таблицу материалов) и в соответствии с инструкциями изготовителя.
- Измерение оптической плотности (OD 260) для определения концентрации ДНК с помощью спектрофотометра.
2. Грунтовка и зонд Дизайн для анализа КПЦР
- Е. палочки O157: H7 последовательности взяты из GenBank номер доступа AE00517411. Дизайн праймеры и зонд с помощью соответствующего программного обеспечения для грунтовки и дизайна зонда для ПЦР в реальном времени (см. таблицу материалов для деталей).
Последовательности праймеров и зонда являются: Z3276-Forward (F) 5'-TATTCCGCGATGCTTGTTTTT-3 '
Z3276-Reverse (R) 5'-ATTATCTCACCAGCAAACTGGCGG-3 '
Z3276-зонд, FAM-CCGCAATCTTTCC-MGBNFQ - Развести грунтовочный порошок с водой, в результате чего концентрации 10 мкМ в виде раствора праймера работы.
- Развести зондов до 10 мкм в качестве зонда рабочих решений. Меченых зондов варьироваться в потенции с партиями. Поэтому титрование каждую партию меченых зондов, прежде чем использовать для достижения оптимальной производительности КПЦР.
3. Установка КПЦР Условия анализа
- Подготовка реакционной смеси, которая состоит из 25,0 мкл 2х ПЦР в реальном времени мастер-смеси, 200 нМ прямого праймера, 200 нМ обратного праймера и 100 нМ зонда.
- Добавьте 5 мкл образца ДНК (100 мкг) и соответствующий объем воды до конечного объема 50 мкл.
- Для nontemplate контроля, использовать 5 мкл водой заменить образец ДНК.
- Установите условия КПЦР следующим образом: 95 ° C в течение 10 мин для активации TaqMan; с последующими 40 циклами 95 ° С в течение 10 сек для денатурации и 60 и# 176; C в течение 1 мин для отжига / продление.
4. КПЦР Чувствительность теста
- Сделать серийный 10-кратное разбавление от культуры середины экспоненциальной фазы (OD 600 = 0,5; около 1,5 х 10 8 КОЕ / мл при посеве).
- Добавить 100 мл разведений клеток на 96-луночный планшет трижды.
- Центрифуга пластину в 2500 х г в течение 10 мин.
- Добавьте 50 мл экстракции ДНК раствора в каждую лунку.
- Ресуспендируют осадок клеток с пипеткой многоканальной.
- Уплотнение пластину с пленкой, кипятить тарелку на водяной бане в течение 10 мин, и центрифуге при 2500 мкг в течение 2 мин.
5. Подготовка живых и мертвых Смеси Сотовые для PMA Лечение
- Grow 10 мл Е. палочки O157: H7 при 37 ° С до середины экспоненциальной фазы и делить культуру на две аликвоты.
- Варить одну аликвоту клеток в течение 10 мин на водяной бане в течение мертвых клеток и оставить другую аликвоту для живых клеток.
- Убедитесь, чтоповторно не живые клетки от убитых нагреванием аликвоты путем посева клеток на чашках с агаром LB.
- Принимать по 2 мл живых и убитых нагреванием клеток и регулировать концентрацию до 8 х 10 6 КОЕ / мл с LB среды. Создание четыре набора клеток разведений, начиная с 8 х 10 0 до 8 х 10 6 КОЕ / мл.
- Используйте первые два набора клеток разведений для живых клеток, обработанных РМА или лечить.
- Для третьего и четвертого наборов клеток разведений, добавьте 8 × 10 6 мертвые клетки в каждого разведения клеток (8 × 10 0 - 8 × 10 6), чтобы сделать живых и мертвых клеток смесей.
- Treat третий набор разбавления РМА и использовать четвертый набор разбавления для контроля.
6. Обработки клеток ФМА
- Растворить PMA в диметилсульфоксиде (или воды), чтобы сделать 10 мМ маточного раствора и хранили при -20 ° С в темноте.
- Аликвоты 400 мкл живых клеток, мертвые клетки, и смеси живых и мертвых клеток втри отдельных пробирок.
- Добавить 2,0 мл 10 мМ РМА к каждой аликвоте клеток в конечной концентрации 50 мкМ.
- Инкубируйте образцы при комнатной температуре в течение 5 мин в темноте, дрожа мягко несколько раз, 3 сек каждый раз.
- Добавить 100 мкл образцов на 96-луночный планшет.
- Уплотнение тарелку с оптическим пленкой и поставить тарелку на льду.
- Установите пластину 20 см от 650 Вт галогенные источника света и выставить в течение 2 мин для освещенности.
- Центрифуга пластину на 2500 мкг в течение 10 мин, осторожно отбросить супернатант и осторожно слить пластину на листе абсорбирующей бумаги.
- Ресуспендируют клеточный осадок в 50 мкл выделения ДНК решения с помощью пипетки вверх и вниз двадцать раз с многоканальным Pipetman в.
- Уплотнение пластину с пленкой, кипятить в течение 10 мин на водяной бане, и центрифуге при 2500 мкг в течение 2 мин. Супернатант в пластине является ДНК из клеток, обработанных РМА и готов к количественной ПЦР.
- Выполните КПЦР как descriкровать выше, с использованием 5 мкл ДНК.
Результаты
Обнаружение E. палочки O157: H7 КПЦР использованием ORF Z3276
Одним из ключевых факторов этого анализа является чувствительность и специфичность зонда Z3276 используется в кПЦР. Он может обнаружить по цене от нескольких копий генома ДНК E. палочки O157: H7, как показано на рисунке 1а. Из 120 не-O157 штаммов, в том числе исследованных шести основных штаммов не-O157 Стек, O104: H4 штаммов, Salmonella, Shigella и штаммов, не перекрестной реактивности не было найдено, как показано в таблице 1.
Сочетание лечения РМА с кПЦР
Инкубация Е. палочки O157: H7 клетки с PMA (50 мм) в течение 5 мин в темноте и подвергая воздействию света в течение 2 мин были отобраны для условий обработки PMA. Кроме того, мы изменили процедуру обработки PMA. По сравнению с различных прозрачных микропробирок, используемых в сшивающего шаге 96-луночный планшет было установлено, что более эффективнымдостичь оптимального сшивающий эффект и более удобны, чем обработки большого количества пробирок в процессе освещенности.
Амплификация ДНК из обработанных РМА-живых и мертвых клеток в кПЦР
Эффекты PMA-опосредованного ингибирования амплификации ДНК от мертвых клеток на этой КПЦР анализа были показаны на рисунке 1, с использованием ДНК, полученной из двух последовательных 10-кратных живые или мертвые клетки разведений. Результаты показывают, что кривые, полученные из живых клеток, обработанных РМА (красная кривая) и без PMA (синий кривая), кажется линейные и почти идентичны друг другу. Различия значений CT Небольшие были показаны между живых клетках, обработанных РМА и без PMA (рис. 1А), предполагая, что обработка ФМА оказывает незначительное влияние на амплификации ДНК живых клеток в количественной ПЦР. Но в 15 - разница КТ значение (32000 раза) было показано, между мертвыми клетками, обработанными PMA и без PMA, демоnstrating что лечение PMA эффективно подавляется амплификации ДНК из мертвых клеток (рис. 1б).
Обнаружение живых клеток из живых и мертвых смесей клеток в кПЦР
Два комплекта живых клеток разведений (8 х 10 0 - 8 × 10 6 КОЕ) обрабатывали ФМА или без PMA для обнаружения живых клеток живых / мертвых клеток смесей. Результаты КПЦР (рис. 2а) продемонстрировали параллельную обратную прогрессивную тенденцию значений КТ по сравнению с цифрами обработанных живых клеток (фиолетовый баров) в необработанных образцов (синие полосы). Обработанные живые клетки дали несколько более высокие значения CT, чем те, необработанных живых клеток. Аналогичный обратная прогрессивная тенденция в значениях КТ наблюдалось с реальным количеством живых клеток в живых / мертвых смесей клеток, обработанных РМА (зеленый бар), а на рисунке 2B. Кроме того, эта тенденция значений КТ былонаблюдается с числом живых клеток в смесях, несмотря на присутствие 106 мертвых клеток. Эти результаты показали, что значения КТ живых / мертвых клеток смесей представлены ДНК из одних живых клеток, тогда как амплификации ДНК из мертвых клеток эффективно подавлено путем обработки РМА. Но, значения CT живых / мертвых смесей клеток, обработанных РМА были колебались и не отражали живые числа клеток в смесях на рисунке 2b (желтые бар).
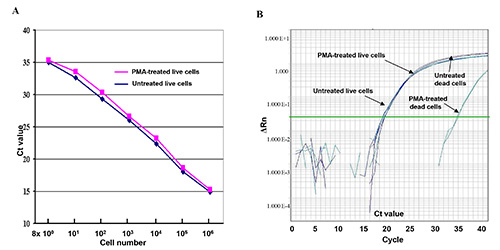
Рисунок 1. Живых и мертвых клеток обрабатывали PMA или без PMA в КПЦР анализа. (А) сериал 10-кратное разбавленный живая E. палочки O157: H7 клеток разведения (8 х 10 0 - 8 × 10 6 КОЕ) обрабатывали ФМА или без PMA.Значения CT представляют средние значения КТ трижды в анализе КПЦР. (B) Сравнение амплификации ДНК живых клеток и мертвых клеток, обработанных РМА и без PMA в сравнении д КПЦР анализа. ΔRn относится к флуоресценции изменение интенсивности. Кликните здесь, чтобы посмотреть увеличенное изображение.
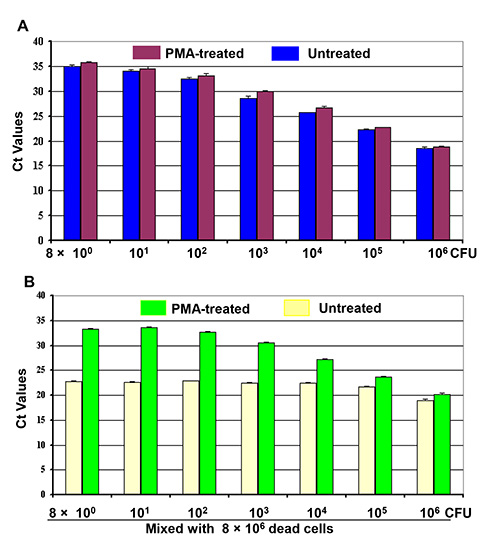
Рисунок 2. Дифференциация живых клеток у живых / мертвых клеток смесей по PMA-кПЦР четыре комплекта 10-кратных разведений клеточных культур (8 х 10 0 - 8 х 10 6 КОЕ). Были сделаны, как указано. (А)-й сет разбавления клеток обрабатывали PMA в то время как разведение вторая ячейка была или не лечить до выделения ДНК.(B) 8 × 10 6 мертвые клетки смешивали с третьим и четвертым множествами разведений клеток, чтобы сделать два набора живых / мертвых клеток смесей, как указано. Один набор клеточных смесей обрабатывают PMA и другой набор лечился без PMA до выделения ДНК. Каждый столбец представляет средние значения КТ в трех экземплярах эксперимента ± SD.
| Организм | Серологический тип | Кол-во штаммов |
| Кишечная палочка | 2006 шпинат O157: H7 вспышка изоляты | 186 |
| 2006 Taco Bell O157: H7 изоляты | 58 | |
| 2006 Taco John O157: H7 изоляты | 11 | |
| Коллекции деформации DMB от другой O157: H7 вспышек | 112 | |
| O104: H4 | 3 | |
| О26 | 18 | |
| O103 | 13 | |
| O111 | 24 | |
| Ø121 | 2 | |
| O45 | 5 | |
| O145 | 9 | |
| O113 | 2 | |
| O91 | 1 | |
| O55 | 9 | |
| Другой | 19 | |
| Сальмонелла | Typhi | 1 |
| Ньюпорт | 2 | |
| Шигеллами дизентерии | 6 | 2 |
| Shigella Зонне | Неизвестный | 2 |
| Shigella Флекснера | 4 | 1 |
| Shigella boydii | 1 | 1 |
| Общий | 481 | |
Тв состоянии 1. Штаммы, используемые для проверки в реальном времени ПЦР для идентификации E. палочки O157: H7.
Обсуждение
Основная цель исследования предполагает использование в ORF Z3276, уникальный Е. палочки O157: H7, в качестве единственного биомаркеров для КПЦР анализа. В настоящее время большинство КПЦР анализы ориентируетесь гены вирулентности, таких как stx1, Stx2 и EAEA 1,2,5,6 или общих фенотипических генов, таких как uidA 3,4, rfbE и Флик генов 8,9. Иногда, вид или штамм не может быть точно идентифицирован ген (ы) вирулентности, таких как STX 1 и 2 STX генов, так как эти гены присутствуют у разных видов или штаммов 16, а также. Использование rfbE и FLIC для генов-мишеней, оба гена необходимо использовать для полной идентификации 17. Использование ORF Z3276 в качестве биомаркера, это КПЦР анализ было продемонстрировано, чтобы быть чувствительный и специфичный анализ (табл. 1). Этот анализ был подвергнут жестким всеохватности и исключительных испытаний, в том числе O157: H7 (N = 367), более 100 не-O157 штаммы и другие патогенные виды, такие как сальмонелла и Shigella. Все O157: H7 штаммов рассмотрены положительно обнаружены, и не перекрестной реактивности не было найдено от не-O157 штаммов (табл. 1), что указывает на ORF Z3276 является уникальным и сильным биомаркеров для обнаружения E. палочки O157: H7.
Другой целью исследования является выборочно обнаружить живые клетки от мертвых клеток путем обработки PMA до выделения ДНК. PMA может проникать в мертвых клеток с нарушенной мембраной и связываются с ДНК, и таким образом подавляет амплификации ДНК мертвых клеток в ПЦР 13. Мы модифицировали процедуру PMA-лечения, и адаптировали его к формату пластины 96-а для процедуры. Право интенсивности освещенности имеет важное значение для получения оптимального эффекта сшивки. Ранее микропробирки были использованы на этой стадии, 13-15. Тем не менее, отдельные пробирок с ними трудно работать в легкой эксэкспонирования шагом, и эти трубы не абсолютно прозрачны для получения наилучшего перекрестного симпатию. Использование 96-луночный планшет с, мы повысили эффективность PMA-лечения. Эти изменения могут повлиять на анализ несколькими способами: они легче получить полное и равномерное сшивающий эффект, особенно с многочисленными образцов; Обработанные образцы могут быть перемещены из одной пластины к другой, чтобы сделать возможным для обнаружения большого числа образцов, и они предоставляют эту PMA-КПЦР анализа с потенциалом автоматизации для всего процесса. Ограничение этого PMA-КПЦР анализа является то, что лечение PMA увеличивает стоимость анализа ПЦР несколько и несколько снижена чувствительность ПЦР.
Раскрытие информации
Авторы не имеют ничего раскрывать.
Благодарности
Мы благодарим Департамент внутренней безопасности США за предоставление часть финансирования.
Материалы
| Name | Company | Catalog Number | Comments |
| AB 7900HT Fast Real-time PCR system | ABI | ||
| 2x Universal master mix | ABI | 4304437 | |
| PrepMan Ultra | ABI | 4318930 | |
| Primer Express | ABI | ||
| Probes | ABI | Top of Form | |
| PMA | Biodium | 40013 | |
| Puregen cell and tissue kit | Qiagene | ||
| DU530 spectrophotometer | Beckman | ||
| Spectrophotometer | NanoDrop Technology | ND-1000 | |
| 650 W Halogen light source | Britek | H8061S | |
| Otptical Adhesive film | ABI | 4311971 | |
| Optical 96-well reaction plate | ABI | 4316813 |
Ссылки
- Barletta, F., et al. Validation of five-colony pool analysis using multiplex real-time PCR for detection of diarrheagenic Escherichia coli. J. Clin. Microbiol. 47, 1915-1917 (2009).
- Gannon, V. P., King, R. K., Kim, J. Y., Thomas, E. J. Rapid and sensitive method for detection of Shiga-like toxin-producing Escherichia coli in ground beef using the polymerase chain reaction. Appl. Environ. Microbiol. 58, 3809-3815 (1992).
- Cebula, T. A., Payne, W. L., Feng, P. Simultaneous identification of strains of Escherichia coli serotype O157:H7 and their Shiga-like toxin type by mismatch amplification mutation assay-multiplex PCR. J. Clin. Microbiol. 33, 248-250 (1995).
- Li, Y., Mustapha, A. Simultaneous detection of Escherichia coli O157:H7, Salmonella, and Shigella in apple cider and produce by a multiplex PCR. J. Food. Prot. 67, 27-33 (2004).
- Schmidt, H., et al. Differentiation in virulence patterns of Escherichia coli possessing eae genes. Med. Microbiol. Immunol. 183, 23-31 (1994).
- Fratamico, P. M., Sackitey, S. K., Wiedmann, M., Deng, M. Y. Detection of Escherichia coli O157:H7 by multiple PCR. J. Clin. Microbiol. 33, 2188-2191 (1995).
- Desmarchelier, P. M., Bilge, S. S., Fegan, N., Mills, L., Vary, J. C., Tarr, P. I. A PCR specific for Escherichia coli O157 based on the rfb locus encoding O157 lipopolysaccharide. J. Clin. Microbiol. 36, 1801-1804 (1998).
- Fields, P. I., Blom, K., Hughes, H. J., Helsel, L. O., Feng, P., Swaminathan, B. Molecular characterization of the gene encoding H antigen in Escherichia coli and development of a PCR-restriction fragment length polymorphism test for identification of E. coli O157:H7 and O157:NM. J. Clin. Microbiol. 35, 1066-1070 (1997).
- Fricker, M., Messelhäußer, U., Bushch, U., Scherer, S., Ehling-Schulz, M. Diagnostic real-time PCR assays for detection of emetic Bacillus cereus strains in foods and recent food-borne outbreaks. Appl. Environ. Microbiol. 73, 1892-1898 (2007).
- Li, B., Chen, J. Q. Real-time PCR methodology for selective detection of viable Escherichia coli O157:H7 cells by targeting Z3276 as a genetic marker. Appl Environ. Microbiol. 78, 5297-5304 (2012).
- Perna, N. T., et al. Genome sequence of enterohaemorrhagic Escherichia coli O157:H7. Nature. 409, 529-533 (2001).
- Wang, S., Levin, R. E. Discrimination of viable Vibrio vulnificus cells from dead cells in real-time PCR. J. Microbiol. Methods. 64, 1-8 (2006).
- Nocker, A., Cheung, C. Y., Camper, A. K. Comparison of propidium monoazide with ethidium monoazide for differentiation of live vs. dead bacteria by selective removal of DNA from dead cells. J. Microbiol. Methods. 67, 310-320 (2006).
- Nocker, A., Sossa-Fernandez, P., Burr, M. D., Camper, A. K. Use of propidium monoazide for live/dead distinction in microbial ecology. Appl. Environ. Microbiol. 73, 5111-5117 (2007).
- Cawthorn, D. M., Witthuhn, R. C. Selective PCR detection of viable Enterobacter sakazakii cells utilizing propidium monoazide or ethidium bromide monoazide. J. Appl. Microbiol. 105, 1178-1185 (2008).
- Chassagne, L., Pradel, N., Robin, F., Livrelli, V., Bonnet, R., Delmas, J. Detection of stx1, stx2, and eae genes of enterohemorrhagic Escherichia coli using SYBR Green in a real-time polymerase chain reaction. Diagn. Microbiol. Infect. Dis. 64, 98-101 (2009).
- Liu, Y., Gilchrist, A., Zhang, J., Li, X. F. Detection of viable but nonculturable Escherichia coli O157:H7 bacteria in drinking water and river water. Appl. Environ. Microbiol. 74, 1502-1507 (2008).
Перепечатки и разрешения
Запросить разрешение на использование текста или рисунков этого JoVE статьи
Запросить разрешениеThis article has been published
Video Coming Soon
Авторские права © 2025 MyJoVE Corporation. Все права защищены