Method Article
Erkennung von Live-
In diesem Artikel
Zusammenfassung
H7 gezielt eine einzigartige genetische Marker, Z3276: Ein qPCR-Assay wurde für den Nachweis von Escherichia coli O157 entwickelt. Die qPCR wurde mit Propidium Monoazid (PMA)-Behandlung für Lebendzellerkennung kombiniert. Dieses Protokoll wurde modifiziert und auf eine 96-Well-Plattenformat für die einfache und einheitliche Behandlung einer Vielzahl von Proben geeignet ist
Zusammenfassung
Eine einzigartige offene Leserahmen (ORF) Z3276 wurde als ein spezifischer genetischer Marker für E. identifiziert coli O157: H7. Ein qPCR-Assay wurde für den Nachweis von E. entwickelt coli O157: H7, indem sie auf ORF-Z3276. Mit diesem Test können wir so niedrig wie ein paar Kopien des Genoms von DNA von E. erkennen coli O157: H7. Die Empfindlichkeit und Spezifität des Assays wurden durch intensive Validierungstests mit einer großen Anzahl von E. bestätigt coli O157: H7-Stämme (n = 369) und nicht-O157-Stämme (n = 112). Darüber hinaus haben wir Propidium Monoazid (PMA) Verfahren mit den neu entwickelten qPCR-Protokoll für den selektiven Nachweis von lebenden Zellen von toten Zellen kombiniert. Die Amplifikation von DNA aus PMA-behandelten toten Zellen wurde fast vollständig im Gegensatz zu nahezu unbeeinflusst Amplifikation von DNA aus PMA-behandelten lebenden Zellen gehemmt. Außerdem wird das Protokoll geändert wurde und auf eine 96-Well-Plattenformat für eine einfache und einheitliche Handhabung einer großen Anzahl von Proben geeignet. Tseine Methode wird erwartet, dass die Auswirkungen auf genaue mikrobiologische und epidemiologische Überwachung der Lebensmittelsicherheit und Umwelt Quelle haben.
Einleitung
PCR ist eine übliche Technik zum Nachweis von E. coli O157: von Lebensmittel-und Umweltproben H7. Beschreibung spezifischer Biomarker für E. coli O157: H7 ist einer der Schlüsselfaktoren für die erfolgreiche Detektion in PCR-Assays. Die üblicherweise in PCR-Assays für den Nachweis von E. verwendet Biomarker coli O157: H7 gehören Shiga-Toxine (STX 1 und stx 2) 1,2, 3,4 uidA, eaeA 5,6, rfbE 7 und 8,9 fliC Gene. Diese Biomarker können variable Effizienz zur Identifizierung bereitzustellen, aber die meisten der Biomarker kann nicht als eine einzigartige Biomarker zum Nachweis von E. verwendet werden coli O157: H7. Diese Unzulänglichkeit in der Differenzierung von Zielgenen Leistung fordert mehr und stärker spezifische Biomarker (s) zur Identifizierung von E. coli O157: H7 10.
Zahlreiche Sonden für Gene oder hypothetischen offenen Leserahmen (ORFs) in E. coliO157: H7 11 wurden für die qPCR-Assay auf der Grundlage der Hinweise von DNA-Microarrays Genotypisierung aus unserem Labor und durch die Suche in der Datenbank GenBank des National Center for Biotechnology Information ausgelegt. Die Reihenfolge der Z3276 ORF, der ein Gen Fimbrien-11 wurde für die qPCR-Assay ausgewählt. Anschließend wurde eine qPCR Assay durch zahlreiche Studien auf PCR-Parameter wie Konzentrationen von Primer und Sonden sowie Rungs-und Verlängerungstemperaturen (Daten nicht gezeigt) entwickelt. Nach Anreiz Inklusivität und Exklusivität Tests an Referenzstämme wie E. coli O157: H7, nicht-O157-und Shigella-Stämme in der qPCR wurde die ORF-Z3276 als eine spezifische und starke genetische Marker zur Identifizierung von E. identifiziert coli O157: H7. Dann entwickeln wir eine neue qPCR-Methode mit dieser einzigartigen Biomarker für die Identifizierung von E. coli O157: H7.
Eine weitere Herausforderung bei der Detektion von E. coli O157: H7 in für kontaminierteMethoden und Umwelt ist eine genaue Bestimmung der lebenden Zellen aus Proben. Die erweiterte Präsenz von DNA aus toten Zellen und die Unfähigkeit zur Lebensfähigkeit in PCR-Assays unterscheiden könnte falsch-positive Werte verursachen, was zu einer Überschätzung von Live-Zahlen Zelle in Erkennung. Folglich schränkt dies die PCR-Nutzung in präzise Erkennung von Krankheitserregern 12. Ein neuer Ansatz zur DNA der toten Zellen differenzieren, wurde durch die Kombination von Propidium Monoazid (PMA) der Behandlung mit PCR-Verfahren in den letzten Jahren 13 bis 15 übernommen. PMA ist in der Lage, innerhalb der toten Zellen zu gehen, und die in die DNA interkalieren, wenn sie Licht ausgesetzt wird, aber es kann nicht innerhalb von lebenden Zellen gehen, um mit der DNA der lebenden Zellen reagieren. Somit wird in PMA-behandelten Mischungen von lebenden und toten Zellen, die DNA aus toten Zellen können in PCR 13 verstärkt werden. Wir kombinierten PMA-Behandlung mit dem neu entwickelten qPCR Assay zur selektiven Nachweis Live E. coli O157: H7-Zellen. Darüber hinaus haben wir die PMA-qPCR eine gemacht habenssay für Hochdurchsatz-Erkennung möglich.
Protokoll
1. Bakterienstämme und DNA-Template Vorbereitung
- Wachsen Bakterienstämme von E. coli O157: H7, O157 nicht, und anderen pathogenen Arten, wie Salmonella und Shigella, bei 37 ° C über Nacht unter Schütteln inkubiert.
- Extrahieren von DNA aus Bakterienkulturen mit dem entsprechenden Kit (siehe Tabelle der Werkstoffe) und nach Anweisungen des Herstellers.
- Die optische Dichte (OD 260), um die DNA-Konzentration unter Verwendung eines Spektrophotometers zu bestimmen.
2. Primer-und Sonden-Design für die qPCR Assay
- E. coli O157: H7-Sequenzen sind von GenBank Zugangsnummer AE00517411. Design-Primer und Sonde mit der entsprechenden Software für die Primer-und Sondendesign für Echtzeit-PCR (siehe Materialtabelle für Details).
Die Sequenzen von Primern und Sonden sind wie folgt: Z3276-Vorwärts (F) 5'-TATTCCGCGATGCTTGTTTTT-3 '
Z3276-Rücklauf (R)-5'-ATTATCTCACCAGCAAACTGGCGG-3 '
Z3276-Probe, FAM-CCGCAATCTTTCC-MGBNFQ - Rekonstitution des Primers Pulver mit Wasser, was zu einer Konzentration von 10 uM als Primer Arbeitslösung.
- Verdünnen Sie die Sonden bis 10 um als Sonde Arbeitslösungen. Markierte Sonden unterscheiden sich in Potenz mit Chargen. Daher titriert jede Partie markierten Sonden, bevor Sie für eine optimale Leistung qPCR.
3. Einstellen der qPCR Assay AGB
- Bereiten Reaktionsmischung, die von 25,0 ul 2x Echtzeit-PCR Master Mix, 200 nM von Vorwärts-Primer, 200 nM von Reverse-Primer und 100 nM Sonde besteht.
- Mit 5 ul Probe DNA (100 pg) und einem geeigneten Volumen an Wasser, um ein Endvolumen von 50 &mgr; l zu erreichen.
- Für nontemplate Kontrolle sind 5 ul Wasser zu DNA-Probe zu ersetzen.
- Festlegen der qPCR Bedingungen wie folgt: 95 ° C für 10 Minuten zur Aktivierung des TaqMan, gefolgt von 40 Zyklen von 95 ° C für 10 sec und 60 für die Denaturierung undNr. 176, C 1 min Annealing / Extension.
4. qPCR Empfindlichkeitstest
- Einen seriellen 10-fachen Verdünnung von einer Kultur der mittleren exponentiellen Phase (OD 600 = 0,5, etwa 1,5 x 10 8 CFU / ml durch Plattieren).
- 100 ml der Zellverdünnungen auf eine 96-Well-Platte in dreifacher Ausfertigung.
- Zentrifugieren Sie die Platte bei 2500 g für 10 min.
- 50 ml DNA-Extraktionslösung in jede Vertiefung.
- Resuspendieren Zellpellets mit einer Mehrkanalpipette.
- Verschließen Sie die Platte mit einem Film, kochen Sie die Platte in einem Wasserbad für 10 min und zentrifugiert bei 2500 g für 2 min.
5. Vorbereiten Live und Dead Cell Mischungen für PMA-Behandlung
- Wachsen 10 ml E. coli O157: H7 bei 37 ° C bis zur mittleren exponentiellen Phase und teilen die Kultur in zwei Aliquots.
- Kochen Sie ein Aliquot der Zellen für 10 min in einem Wasserbad für toten Zellen und lassen Sie die anderen aliquoten für lebende Zellen.
- Überprüfen Sie, ob dieWieder sind keine lebenden Zellen aus der Hitze abgetöteten Aliquot durch Ausplattieren der Zellen auf LB-Agar-Platten.
- Nehmen Sie 2 ml der Live-und Hitze abgetöteten Zellen und stellen Sie die Konzentration auf 8 x 10 6 KBE / ml mit LB-Medium. Erstellen vier Sätze von Zellverdünnungen im Bereich von 8 x 10 0 bis 8 × 10 6 CFU / ml.
- Mit den ersten beiden Sätzen von Zellverdünnungen für lebende Zellen mit PMA behandelt oder unbehandelt.
- Um den dritten und vierten Satz von Zellverdünnungen, fügen 8 x 10 6 toten Zellen zu jeder Zelle Verdünnung (8 x 10 0 - 8 x 10 6) an lebenden und toten Zellgemischen zu machen.
- Behandeln Sie den dritten Satz der Verdünnung mit PMA und verwenden Sie den vierten Satz der Verdünnung für die Steuerung.
6. Die Behandlung von Zellen mit PMA
- Lösen PMA in Dimethylsulfoxid (oder Wasser) auf 10 mM Stammlösung und bei -20 ° C im Dunkeln gelagert zu machen.
- 400 ul Aliquot der lebenden Zellen, toten Zellen, und die Mischung von lebenden und toten Zellen indrei separate Mikroröhrchen.
- 2,0 ml von 10 mM PMA jeder Zelle Aliquot auf eine Endkonzentration von 50 uM.
- Die Proben inkubiert bei Raumtemperatur für 5 min in der Dunkelheit sanft ein paar Mal, 3 Sek. jedes Mal zittern.
- 100 ul-Proben auf einer Platte mit 96 Vertiefungen.
- Verschließen Sie die Platte mit einem optischen Film und stellte den Teller auf Eis.
- Stellen Sie die Platte 20 cm von einer 650 W Halogen-Lichtquelle aussetzen und für 2 min für Belichtung.
- Zentrifugieren Sie die Platte bei 2500 g für 10 min, vorsichtig den Überstand verwerfen und sorgfältig abtropfen lassen die Platte auf ein Stück Papier zu absorbieren.
- Resuspendieren Zellpellets in 50 ul DNA-Extraktionslösung durch Auf-und Abpipettieren zwanzigmal mit einem Mehrkanal-Pipetman.
- Verschließen Sie die Platte mit einem Film, kochen Sie für 10 Minuten in ein Wasserbad, und Zentrifuge bei 2500 g für 2 min. Der Überstand in der Platte ist die DNA aus Zellen mit PMA und bereit für qPCR behandelt.
- Führen qPCR wie beschrieBett über, unter Verwendung von 5 ul der DNA.
Ergebnisse
Nachweis von E. coli O157: von qPCR mit ORF Z3276 H7
Einer der Schlüsselfaktoren für diesen Test ist die Sensitivität und Spezifität des Z3276 Sonde in der qPCR eingesetzt. Es kann als nur einige wenige Kopien des Genoms von DNA von E. erkennen coli O157: H7, wie in 1A gezeigt. Der 120 nicht-O157-Stämme untersucht, einschließlich der sechs großen nicht-O157 STEC-Stämme O104: H4-Stämme, Salmonella und Shigella-Stämme wurde keine Kreuzreaktivität festgestellt, wie in Tabelle 1 gezeigt.
Die Kombination der Behandlung mit PMA qPCR
Ausbrüten E. coli O157: H7-Zellen mit PMA (50 mM) für 5 min im Dunkeln und Belichten für 2 min wurden PMA Behandlungsbedingungen ausgewählt. Zusätzlich modifiziert wir PMA Behandlungsverfahren. Vergleich verschiedener transparenter Mikroröhrchen in dem Vernetzungsschritt verwendet wurde, wurde eine Platte mit 96 Vertiefungen gefunden, um effizienter zuerreichen die optimale Vernetzungseffekt und bequemer Handhabung einer großen Anzahl von Probenröhrchen während der Belichtungsprozess.
Die Amplifikation von DNA aus PMA-behandelten lebenden und toten Zellen in qPCR
Die Wirkungen von PMA-vermittelte Hemmung der Amplifikation von DNA aus toten Zellen in diesem Assay wurden qPCR in Fig. 1 durch die Verwendung von zwei seriellen 10-fachen lebenden oder toten Zellverdünnungen abgeleitete DNA gezeigt. Die Ergebnisse zeigen, dass die aus den lebenden Zellen mit PMA (rote Kurve) und ohne PMA (blaue Kurve) behandelt erzeugt Kurven scheinen linearen und nahezu identisch zueinander. Leichte CT Wertdifferenzen zwischen den lebenden Zellen, die mit PMA und ohne PMA (1A) behandelt, die vermuten lassen, dass PMA-Behandlung hatte wenig Einfluss auf die Amplifikation der DNA der lebenden Zellen in der qPCR. Aber ein 15 - CT-Wert-Differenz (32.000-fach) wurde zwischen der toten Zellen mit PMA behandelt und ohne PMA, Demo gezeigtnstrating dass PMA-Behandlung effizient unterdrückt DNA-Amplifikation von den toten Zellen (Abbildung 1B).
Nachweis von lebenden Zellen von lebenden und toten Zellgemischen in qPCR
Zwei Sätze von lebenden Zellen Verdünnungen (8 x 10 0 - 8 x 10 6 CFU) wurden mit oder ohne PMA PMA behandelt, um lebende Zellen von Live / Dead-Zellgemischen zu erkennen. Die Ergebnisse qPCR (2A) gezeigt, eine parallel inverse progressive Entwicklung der CT-Werte, um die Zahl der lebenden Zellen behandelt (lila Balken) auf die unbehandelten Proben (blaue Balken) bezogen. Die behandelten lebenden Zellen ergab eine etwas höhere CT-Werte als die der unbehandelten lebenden Zellen. Eine ähnliche inverse progressive Tendenz in CT-Werte wurde mit der realen Zahl der lebenden Zellen in den mit PMA (grüne Balken) in 2B behandelt sowie Live / Dead-Zellgemischen beobachtet. Zusätzlich sind Abwärtstrend der CT-Werte wartrotz der Anwesenheit von 106 toten Zellen mit der Anzahl lebender Zellen in der Mischung beobachtet. Diese Ergebnisse zeigten, daß die CT-Werte der lebenden / toten Zellgemischen dargestellt DNA aus den lebenden Zellen alleine, während der DNA-Amplifikation aus den toten Zellen effizient durch PMA-Behandlung unterdrückt. Aber die CT-Werte von Live mit PMA behandelt / tote Zelle Mischungen wurden schwankte und es versäumt, reflektiert die Live-Zahlen Zelle in den Mischungen in 2B (gelbe Balken).
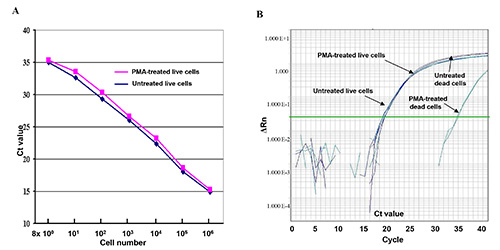
Fig. 1 ist. Lebenden und toten Zellen mit PMA oder ohne PMA in qPCR-Assay behandelt. (A) Eine Serie von 10-fach verdünnte Live E. coli O157: H7 Zelle Verdünnungen (8 x 10 0 - 8 x 10 6 CFU) wurden mit PMA oder ohne PMA behandelt.Die CT-Werte stellen die Mittelwerte der CT dreifach in der qPCR-Assays. (B) Vergleich der DNA-Amplifikation von lebenden und toten Zellen mit PMA und ohne PMA in der q Vergleich qPCR-Assay behandelt. ΔRn bezieht sich auf Intensitätsänderung der Fluoreszenz. Klicken Sie hier für eine größere Ansicht.
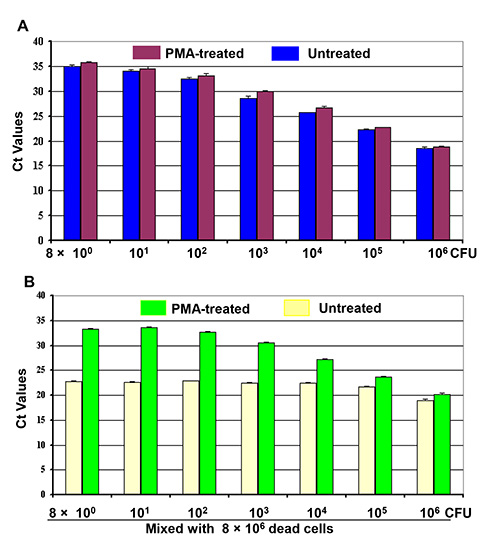
2. Differenzierung von lebenden Zellen von lebenden / toten Zellgemischen durch PMA-qPCR Vier Sätze von 10-fachen Verdünnungen von Zellkulturen (8 x 10 0 - 8 x 10 6 CFU). Wurden wie angegeben. (A) Erste Reihe von Zellverdünnung wurde mit PMA behandelt, während die zweite Zelle Verdünnung war oder unbehandelt, bevor die DNA-Extraktion.(B) 8 x 10 6 Tote Zellen wurden mit den dritten und vierten Sätzen von Zellen Verdünnungen vermischt, um zwei Sätze von lebenden / toten Zellgemische stellen, wie angegeben. Ein Satz der Zellgemische wurden mit PMA behandelt und die andere Gruppe wurde ohne PMA vor der DNA-Extraktion behandelt. Jeder Balken stellt den Mittelwert CT-Werte eines Dreifachversuch ± SD.
| Organismus | Serotyp | Anzahl der getesteten Stämme |
| E. coli | 2006 Spinat O157: H7 Ausbruch Isolate | 186 |
| 2006 Taco Bell O157: H7-Isolate | 58 | |
| 2006 Taco John O157: H7-Isolate | 11 | |
| DMB Stammsammlungen von anderen O157: H7 Ausbrüche | 112 | |
| O104: H4 | 3 | |
| O26 | 18 | |
| O103 | 13 | |
| O111 | 24 | |
| O121 | 2 | |
| O45 | 5 | |
| O145 | 9 | |
| O113 | 2 | |
| O91 | 1 | |
| O55 | 9 | |
| Andere | 19 | |
| Salmonellen | Typhi | 1 |
| Newport | 2 | |
| Shigella dysenteriae | 6 | 2 |
| Shigella sonnei | Unbekannt | 2 |
| Shigella flexneri | 4 | 1 |
| Shigella boydii | 1 | 1 |
| Gesamt | 481 | |
TLage ein. Stämme für die Validierung in Echtzeit PCR-Test für die Identifizierung von E. verwendet coli O157: H7.
Diskussion
Eine primäre Ziel der Studie beinhaltet die Verwendung von einem ORF Z3276, eine einzigartige E. coli O157: H7, als alleinige Biomarker für qPCR-Assay. Derzeit sind die meisten qPCR-Assays Targeting Virulenz-Gene, wie stx1, stx2 und eaeA 1,2,5,6 oder gemeinsamen phänotypischen Gene, wie uidA 3,4, rfbE und fliC Gene 8,9. Gelegentlich Spezies oder Stamm kann nicht definitiv von Virulenz-Gen (e) identifiziert werden, wie stx 1 und 2 Gene stx, wie diese Gene bei verschiedenen Spezies oder Stämme 16 und vorhanden sind. Verwendung rfbE und fliC für Zielgene, beide Gene benötigt werden, um für eine vollständige Identifizierung 17 verwendet werden. Verwendung ORF Z3276 als Biomarker, hat qPCR-Assay wurde gezeigt, empfindlichen und spezifischen Test (Tabelle 1). H7-Stämme (n = 3: Dieser Test hat den strengen Inklusivität und Exklusivität Tests, einschließlich O157 zogen wurde67), über 100 nicht-O157-Stämme und andere pathogene Arten, wie Salmonella und Shigella. Alle O157: H7-Stämme untersucht wurden positiv detektiert und keine Kreuzreaktivität wurde von den nicht-O157-Stämme (Tabelle 1) gefunden, was anzeigt, dass der ORF Z3276 ist ein einzigartiges und starkes Biomarker zum Nachweis von E. coli O157: H7.
Das andere Ziel der Studie ist es, lebende Zellen vor DNA-Extraktion selektiven Nachweis von toten Zellen durch PMA-Behandlung. PMA kann in der toten Zellen mit beeinträchtigter Membran durchdringen und an die DNA binden und hemmt so die DNA-Amplifikation von toten Zellen in 13 PCR. Wir haben die PMA-Behandlung Verfahren modifiziert und angepasst ist, um eine 96-Well-Plattenformat für das Verfahren. Die richtige Intensität der Belichtung notwendig, um die optimale Vernetzung Wirkung zu erzielen. Früher wurden Röhrchen aus diesem Schritt 13-15 verwendet. Allerdings sind separate Mikroröhrchen schwierig zu handhaben in Licht abBelichtungsschritt und diese Röhren nicht absolut transparent für die besten Cross-Geschmack zu erhalten. Verwendung eines 96-Well-Platte, konnten wir die Effizienz der PMA-Behandlung. Diese Änderungen können die Assays auf mehrere Arten beeinflussen: Sie machen es einfacher, eine gründliche und gleichmäßige Vernetzungseffekt zu erzielen, insbesondere bei einer Vielzahl von Proben, die behandelten Proben können von einer Platte zur anderen bewegt werden, um es möglich zu machen Detektion Vielzahl Proben, und sie liefern diese PMA-qPCR-Assay mit Automatisierungspotential für den gesamten Prozess. Die Einschränkung dieser PMA-qPCR-Assay ist, dass PMA-Behandlung erhöht die Kosten etwas PCR und die Empfindlichkeit der PCR-Assay leicht reduziert.
Offenlegungen
Die Autoren haben nichts zu offenbaren.
Danksagungen
Wir danken dem US-Department of Homeland Security für die Bereitstellung Teil der Finanzierung.
Materialien
| Name | Company | Catalog Number | Comments |
| AB 7900HT Fast Real-time PCR system | ABI | ||
| 2x Universal master mix | ABI | 4304437 | |
| PrepMan Ultra | ABI | 4318930 | |
| Primer Express | ABI | ||
| Probes | ABI | Top of Form | |
| PMA | Biodium | 40013 | |
| Puregen cell and tissue kit | Qiagene | ||
| DU530 spectrophotometer | Beckman | ||
| Spectrophotometer | NanoDrop Technology | ND-1000 | |
| 650 W Halogen light source | Britek | H8061S | |
| Otptical Adhesive film | ABI | 4311971 | |
| Optical 96-well reaction plate | ABI | 4316813 |
Referenzen
- Barletta, F., et al. Validation of five-colony pool analysis using multiplex real-time PCR for detection of diarrheagenic Escherichia coli. J. Clin. Microbiol. 47, 1915-1917 (2009).
- Gannon, V. P., King, R. K., Kim, J. Y., Thomas, E. J. Rapid and sensitive method for detection of Shiga-like toxin-producing Escherichia coli in ground beef using the polymerase chain reaction. Appl. Environ. Microbiol. 58, 3809-3815 (1992).
- Cebula, T. A., Payne, W. L., Feng, P. Simultaneous identification of strains of Escherichia coli serotype O157:H7 and their Shiga-like toxin type by mismatch amplification mutation assay-multiplex PCR. J. Clin. Microbiol. 33, 248-250 (1995).
- Li, Y., Mustapha, A. Simultaneous detection of Escherichia coli O157:H7, Salmonella, and Shigella in apple cider and produce by a multiplex PCR. J. Food. Prot. 67, 27-33 (2004).
- Schmidt, H., et al. Differentiation in virulence patterns of Escherichia coli possessing eae genes. Med. Microbiol. Immunol. 183, 23-31 (1994).
- Fratamico, P. M., Sackitey, S. K., Wiedmann, M., Deng, M. Y. Detection of Escherichia coli O157:H7 by multiple PCR. J. Clin. Microbiol. 33, 2188-2191 (1995).
- Desmarchelier, P. M., Bilge, S. S., Fegan, N., Mills, L., Vary, J. C., Tarr, P. I. A PCR specific for Escherichia coli O157 based on the rfb locus encoding O157 lipopolysaccharide. J. Clin. Microbiol. 36, 1801-1804 (1998).
- Fields, P. I., Blom, K., Hughes, H. J., Helsel, L. O., Feng, P., Swaminathan, B. Molecular characterization of the gene encoding H antigen in Escherichia coli and development of a PCR-restriction fragment length polymorphism test for identification of E. coli O157:H7 and O157:NM. J. Clin. Microbiol. 35, 1066-1070 (1997).
- Fricker, M., Messelhäußer, U., Bushch, U., Scherer, S., Ehling-Schulz, M. Diagnostic real-time PCR assays for detection of emetic Bacillus cereus strains in foods and recent food-borne outbreaks. Appl. Environ. Microbiol. 73, 1892-1898 (2007).
- Li, B., Chen, J. Q. Real-time PCR methodology for selective detection of viable Escherichia coli O157:H7 cells by targeting Z3276 as a genetic marker. Appl Environ. Microbiol. 78, 5297-5304 (2012).
- Perna, N. T., et al. Genome sequence of enterohaemorrhagic Escherichia coli O157:H7. Nature. 409, 529-533 (2001).
- Wang, S., Levin, R. E. Discrimination of viable Vibrio vulnificus cells from dead cells in real-time PCR. J. Microbiol. Methods. 64, 1-8 (2006).
- Nocker, A., Cheung, C. Y., Camper, A. K. Comparison of propidium monoazide with ethidium monoazide for differentiation of live vs. dead bacteria by selective removal of DNA from dead cells. J. Microbiol. Methods. 67, 310-320 (2006).
- Nocker, A., Sossa-Fernandez, P., Burr, M. D., Camper, A. K. Use of propidium monoazide for live/dead distinction in microbial ecology. Appl. Environ. Microbiol. 73, 5111-5117 (2007).
- Cawthorn, D. M., Witthuhn, R. C. Selective PCR detection of viable Enterobacter sakazakii cells utilizing propidium monoazide or ethidium bromide monoazide. J. Appl. Microbiol. 105, 1178-1185 (2008).
- Chassagne, L., Pradel, N., Robin, F., Livrelli, V., Bonnet, R., Delmas, J. Detection of stx1, stx2, and eae genes of enterohemorrhagic Escherichia coli using SYBR Green in a real-time polymerase chain reaction. Diagn. Microbiol. Infect. Dis. 64, 98-101 (2009).
- Liu, Y., Gilchrist, A., Zhang, J., Li, X. F. Detection of viable but nonculturable Escherichia coli O157:H7 bacteria in drinking water and river water. Appl. Environ. Microbiol. 74, 1502-1507 (2008).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten