このコンテンツを視聴するには、JoVE 購読が必要です。 サインイン又は無料トライアルを申し込む。
Method Article
遺伝性トランスサイレチン Ala97Ser の遺伝子解析関連アミロイド症
要約
ここでは、例として Ala97Ser、台湾では、最も一般的な風土病突然変異を使用して遺伝性トランスサイレチンのアミロイド症の診断のための点突然変異の存在を確認するためのプロトコルを提示します。
要約
遺伝学的検査は遺伝性トランスサイレチンの最も信頼性の高いテスト関連のアミロイドーシスとトランスサイレチン アミロイドーシス (ATTR) のほとんどのケースで実行する必要があります。ATTR が異種表現型; でまれな、致命的な病気したがって、診断が遅れることもあります。関心と新たな治療法と同様、ATTR の初期症状の広範な認識を増やすこと、適切な診断の研究、トランスサイレチン (TTR) 遺伝子を含むテスト、種類を確認して ATTR の亜種が基本的な予後の改善に。ポリメラーゼの連鎖反応 (PCR) 方法と遺伝学的解析がより迅速に TTR ポイント突然変異の存在を確認、南しみなどの従来の方法よりも安全です。ここで、ATTR Ala97Ser 突然変異、台湾で最も一般的な風土病突然変異の遺伝学的確認を示します。プロトコルには 4 つの主要なステップが装備されている: 収集全血試料、DNA の抽出、PCR とすべての 4 つ TTR エクソンの遺伝学的解析および DNA の配列。
概要
トランスサイレチン (TTR) アミロイドーシス (ATTR) 遺伝性の全身性アミロイドーシスの1、最も一般的な形式し、トランスサイレチン (TTR) 遺伝子2常染色体優性遺伝の突然変異によって引き起こされることができます。TTR 変異 4 量体タンパク質の構造を不安定にし、アミロイド線維2に再アセンブル単量体への解離に 。以上 100 アミロイド TTR の突然変異は報告されている世界的な1をされています。ポリメラーゼの連鎖反応 (PCR) 方法と遺伝学的解析は TTR ポイント突然変異の存在を確認、南しみ3と比較して放射能によって分類されたプローブの処理を回避するなどの利点があります。PCR は、現代の科学4における多数のフィールドに適用されている高速、簡単、安価で、信頼性の高い手法です。
その表現型の多様性を与えられて、この進行と致命的な病気の早期診断は困難です。注意と新興治療5と同様、ATTR の初期症状の広範な認識に伴い、TTR 遺伝子検査を含む適切な診断学的意義、予後を改善するために非常に基本的なしたがって。さらに、異なる突然変異形質の異なる浸透度、発症年齢、進行疾患の重症度、生存期間中央値、肝移植や TTR スタビライザー2,6の有効性のパターンに関連付けられているし、遺伝カウンセリング7,8,9に大きな影響を与える神経と心の関与の可変度。その上、高精度の遺伝子検査は ATTR の 2 つのタイプを区別する唯一のツール: 遺伝性 (突然変異) と野生型 (非突然変異体の形態、老人性全身性アミロイドーシス、SSA)7。2に治療法が異なるため、属性の種類を確認することが不可欠です。したがって、TTR 遺伝子検査の段階的なプロトコルを記述する必要性がますますあります。
突然変異を検出する分子のアプローチは、例として Ala97Ser、台湾では、最も一般的な風土病突然変異を使用して説明されます。DNA 抽出手順の変更は使用される 3 つのソリューションの量を減らすし、DNA の十分な量が得られます。このプロトコルでは 5' 上流、3' 下流、プロモーター、イントロンを含む地域中のすべての 4 つの TTR エクソンが行ったし、非翻訳領域 (UTR) クローニングされたないです。
プロトコル
臨床研究室改善改正 (CLIA) 1988 年の規制、制度検討委員会の長庚記念病院による承認の要件に従って行われた実験室で行ったテストと(ライセンス番号 100 - 4470A3 ・ 104 2462A3) の大学。すべての患者からインフォームド コンセントを得た。
1. 血液検体採取
- 市販の EDTA 処理管に全血を収集します。穏やかに混合し、処理まで 4 ° c の血液サンプルを格納します。
2. 末梢血から DNA 抽出法
ゲノム DNA の抽出の DNA 抽出キットを使用 (材料の表を参照してください)。
- ソリューション 1 の 10 mL を 4.0 mL の血液サンプルに追加します。10 分間氷のサンプルをインキュベートします。
- 4 ° C で 5 分間 2,000 x g でサンプルを遠心分離します。削除し、上澄みを慎重に廃棄します。ソリューション 1 の 3 mL にペレットを再懸濁します。
- 再び 4 ° C で 5 分間 2,000 x g でサンプルを遠心し、上清を捨てます。ソリューション 2 の 3 mL にペレットを再懸濁します。
- 最終濃度 100 μ G/ml を取得してよく混ぜるプロナーゼ原液 (225 mg/mL) の 10 μ L を追加します。37 ° C で一晩インキュベートします。
- 10 分追加 0.8 mL ソリューション 3 のサンプルに氷の上管を冷やすし、反転の 3 - 5 回。5 分間氷の上にサンプルを配置します。
- 4 ° C で 15 分間 3,000 x g でサンプルを遠心分離します。慎重に 15 mL 滅菌円錐形遠心チューブに上清を転送します。
- 最終濃度 20 μ G/ml を得るため RNase 原液 (10 mg/mL) の 6 μ L を追加します。37 ° c 15 分間インキュベートします。
- DNA を沈殿させるために数回を軽く反転して徹底的にイソプロピル アルコールとミックスの 2.5 mL を追加 (白、綿状の素材の鎖となる)。大口径ピペットを使用して、新しい 1.5 mL 遠心チューブに DNA 鉛塩法を転送します。
- 4 ° C で 3 分間、12,000 × g で遠心分離慎重に上清を捨てます。70% エタノール 500 μ L を追加することによって洗浄の DNA の餌。
- 1 分間 12,000 × g で遠心分離し、上澄みを廃棄します。空気常温乾燥 DNA の餌。DdH2O の 100 μ L を追加し、DNA を再懸濁しますに 15 分の 65 ° C で孵化させなさい。
3. ゲノム DNA の定量化
分光光度計を使用して (材料の表を参照) ゲノム DNA の質と量を検出します。
- 分光光度計のマシンに接続されているコンピューターにログインします。ソフトウェアを開きます。
- 分光光度計マシンを初期化します。
- 分光光度計ソフトウェアを「核酸」をクリックします。
- 使い捨ての紙ワイプと精製水で台座をクリーンアップします。
- 空白の分光光度計。
- 台座に精製水 2 μ L をロードします。
- 分光光度計のアッパー アームを下げ、それを校正するための「空白」をクリックします。
- それが終わったら、アッパー アームを持ち上げて拭くと台座を乾燥します。
- このサンプルを次のように測定します。
- サンプルの種類に「DNA 50」をクリックします。
- 2 μ L の台座に DNA のサンプルをロードします。
- アッパー アームを下げ、A260/A280 比とサンプルの濃度を測定する「測定」をクリックします。
- ワイプと精製水とそれぞれの実行の間に台座をクリーンアップします。
- データを収集する「印刷画面」をクリックします。
4. 遺伝の突然変異解析
PCR4ターゲット DNA を増幅します。25 μ L 反応で PCR を行う DNA ポリメラーゼを用いた混合キット (材料の表を参照してください)。TTR 遺伝子のイントロンのプライマーを表 1に示します。
| 遺伝子 | プライマー シーケンス |
| Exon1 | F: 5'-TCAGATTGGCAGGGATAAGC-3' R: 5'-GCAAAGCTGGAAGGAGTCAC-3' |
| Exon2 | F: 5'-TCTTGTTTCGCTCCAGATTTC-3' R: 5'-TCTACCAAGTGAGGGGCAAA-3' |
| Exon3 | F: 5'-GTGTTAGTTGGTGGGGGTGT-3' R: 5'-TGAGTAAAACTGTGCATTTCCTG-3' |
| Exon4 | F: 5'-GACTTCCGGTGGTCAGTCAT-3' R: 5'-GCGTTCTGCCCAGATACTTT-3' |
テーブル 1。TTR 遺伝子イントロン プライマー 。(NCBI 参照シーケンス: NC_000018.10)
- 0.2 mL PCR チューブに表 2に試薬を追加します。
| コンポーネント | (Μ l) | 最終濃度 |
| 10 x バッファー | 2.5 | 1 x |
| MgCl2 (25 mM) | 1.5 | 1.5 mM |
| 360 GC エンハンサー | 1 | - |
| 360 DNA ポリメラーゼ | 0.2 | 2 U/反応 |
| dNTP ミックス (25 mM) | 2 | 2 mM |
| プライマー F (10 μ M) | 1 | 0.4 Μ M |
| プライマー R (10 μ M) | 1 | 0.4 Μ M |
| DNA (100 ng) | 2 | |
| PCR グレード水 | 13.8 | - |
| 総容積 | 25 | - |
表 2。PCR 反応条件。
- 穏やかに混合、ベンチトップ遠心管の底にすべてのコンポーネントを収集するために簡単に遠心力場します。たちにチューブを置きます。
- 30 サイクルの PCR を実行します。各サイクルは 30 の変性のステップで構成されています 94 ° c、プライマーアニー リングの 30 s 58 ° C、および 45 のポリメラーゼ拡張子 s 72 ° c s (表 3参照)。
| ステップ | 温度 (° C) | 時間 | サイクル |
| 初期変性 | 95 | 5 分 | 1 |
| DNA の変性のステップ | 94 | 30 s | 30 サイクル |
| プライマーのアニーリング ステップ | 58 | 30 s | 30 サイクル |
| ポリメラーゼ拡張ステップ | 72 | 45 s | 30 サイクル |
| ポスト延長のステップ | 72 | 10 分 | 1 |
| PCR のサイクリング | 4 | 無期限 |
表 3。PCR の製品を増幅するための条件をサイクリングします。
- ゲル電気泳動法に可視化して PCR の製品を検証します。
- 1.2% の agarose のゲルを準備します。
- アガロース粉末 1.2 g を混ぜて 0.5 100 mL 電子レンジのフラスコの中の x TBE。電子レンジ 2 分、アガロースを完全に溶解するまで。55 ° C に agarose の混合物を冷却します。
- Agarose の混合物にエチジウム ブロマイド溶液 (10 mg/mL) の 2 μ L を加え、軽く混ぜます。注ぎ agarose ゲル トレイ 5-7 mm 程度にし、場所に、comb(s) を追加します。Comb(s) を慎重に取り外します (30 分以上) を固めることができます。
- サンプルをロードし、agarose のゲルを実行します。
- 電気泳動セルにゲルとトレイを配置します。電気泳動セルに 0.5 を入力 x TBE ゲルが覆われるまで。
- 慎重に積載 2 μ L 100 bp の DNA の梯子分子量マーカーとゲルの 1 つの車線に (材料の表を参照してください)。負荷に追加車線の染料/サンプル混合物 (1 μ L 6 × 読み込み染料でに PCR プロダクト ミックスの 5 μ L)。
- 電源をオンにします。100 V で 25 分のためのゲルを実行します。
- 電気泳動セルからゲルを削除します。イメージング システムでの紫外光下で DNA のフラグメントを視覚化します。
- 1.2% の agarose のゲルを準備します。
5. DNA の配列
- 市販のキットを使用して PCR の製品の浄化し、商業的または自動シーケンサーのシーケンス (材料の表を参照してください)。
- 塩基配列を塩基配列データベース10ヌクレオチド クエリを比較する NCBI BLAST サーバーに送信します。結果が表示されたら、配列アラインメントを実行し、予想される突然変異を識別します。
結果
アガロース電気泳動の 2 つの患者と (図 1) TTR 遺伝子の exon 4 の 454 bp の PCR の製品を含む、予想される規模の個々 の健康的な 1 つ明らかにバンド。
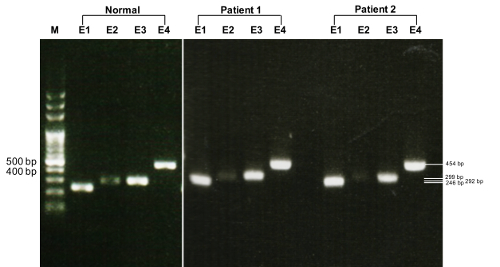
図 1.PCR を描いた電気泳動増幅 TTR 遺伝子。通常: 健康?...
ディスカッション
プロトコル内で 2 つの重要な手順があります。まず、白血球細胞の十分な数を持つために hemodiluted 試験片は回避11をする必要があります。第二に、適切な PCR のプライマーの使用は信頼性の高い結果12を取得する基本的です。プライマー ブラスト web ツールを使用してプライマー4,13; を設計するには4 つの TTR エク...
開示事項
著者が明らかに何もありません。
謝辞
我々 は実験にミス新楽しい呉を感謝したいと思います。本研究は、チャン庚医学研究プログラム (CMRPG3C0371、CMRPG3C0372、CMRPG3C0373) および IRB 100-4470A3、104-2462A3、台湾からの助成金によって支えられました。
資料
| Name | Company | Catalog Number | Comments |
| EDTA-treated tubes | BD | ||
| DNA Extraction Kit | Stratagen | 200600 | |
| NanoDrop ND2000 spectrophotometer | Thermo Fisher Scientific | NanoDrop 2000 | |
| Delicate Task Wipers | Kimberly-Clark | Kimtech Science Kimwipes | |
| AmpliTaq Gold 360 DNA Polymerase kit | Applied Biosystems | 4398823 | |
| TTR gene intronic primers | Exon1 | F: 5’-TCAGATTGGCAGGGATAAGC-3’ | |
| Exon1 | R: 5’-GCAAAGCTGGAAGGAGTCAC-3’ | ||
| Exon2 | F: 5’-TCTTGTTTCGCTCCAGATTTC-3’ | ||
| Exon2 | R: 5’-TCTACCAAGTGAGGGGCAAA-3’ | ||
| Exon3 | F: 5’-GTGTTAGTTGGTGGGGGTGT-3’ | ||
| Exon3 | R: 5’-TGAGTAAAACTGTGCATTTCCTG-3’ | ||
| Exon4 | F: 5’-GACTTCCGGTGGTCAGTCAT-3’ | ||
| Exon4 | R: 5’-GCGTTCTGCCCAGATACTTT-3’ | ||
| thermocycler | Applied Biosystems | GeneAmp PCR System 9700 | |
| electrophoresis cell | ADVANCE | Mupid-2plus | |
| DNA ladder | Protech | PT-M1-100 | |
| dye | BioLabs | B7021 | |
| AlphaImager EC | Alpha Innotech | AlphaImager EC | |
| automatic sequencer | Applied Biosystems | 3730xl DNA Analyzer |
参考文献
- Planté-Bordeneuve, V., Said, G. Familial amyloid polyneuropathy. The Lancet Neurology. 10 (12), 1086-1097 (2011).
- Planté-Bordeneuve, V., et al. Long-term treatment of transthyretin familial amyloid polyneuropathy with tafamidis: a clinical and neurophysiological study. Journal of Neurology. 264 (2), 268-276 (2017).
- Nichols, W. C., Benson, M. D. Hereditary amyloidosis: detection of variant prealbumin genes by restriction enzyme analysis of amplified genomic DNA sequences. Clinical Genetics. 37 (1), 44-53 (1990).
- Lorenz, T. C. Polymerase Chain Reaction: Basic Protocol Plus Troubleshooting and Optimization Strategies. Journal of Visualized Experiments. (63), e3998 (2012).
- Hsu, H. C., et al. Phenotypic expressions of hereditary Transthyretin Ala97Ser related Amyloidosis (ATTR) in Taiwanese. BMC Neurology. 17 (1), 178 (2017).
- Barroso, F. A., et al. Long-term safety and efficacy of tafamidis for the treatment of hereditary transthyretin amyloid polyneuropathy: results up to 6 years. Amyloid. 24 (3), 194-204 (2017).
- Rapezzi, C., et al. Disease profile and differential diagnosis of hereditary transthyretin-related amyloidosis with exclusively cardiac phenotype: an Italian perspective. European Heart Journal. 34 (7), 520-528 (2013).
- Mariani, L. L., et al. Genotype-phenotype correlation and course of transthyretin familial amyloid polyneuropathies in France. Annals of Neurology. 78 (6), 901-916 (2015).
- Hammarström, P., Jiang, X., Hurshman, A. R., Powers, E. T., Kelly, J. W. Sequence-dependent denaturation energetics: A major determinant in amyloid disease diversity. Proceedings of the National Academy of Sciences of the United States of America. 99, 16427-16432 (2002).
- Johnson, M., Zaretskaya, I., Raytselis, Y., Merezhuk, Y., McGinnis, S., Madden, T. L. NCBI BLAST: a better web interface. Nucleic Acids Research. 36, 5-9 (2008).
- Fox, A. J., et al. Next generation sequencing for the detection of actionable mutations in solid and liquid tumors. Journal of Visualized Experiments. (115), (2016).
- Date, Y., Nakazato, M., Kangawa, K., Shirieda, K., Fujimoto, T., Matsukura, S. Detection of three transthyretin gene mutations in familial amyloidotic polyneuropathy by analysis of DNA extracted from formalin-fixed and paraffin-embedded tissues. Journal of the Neurological Sciences. 150 (2), 143-148 (1997).
- Ye, J., Coulouris, G., Zaretskaya, I., Cutcutache, I., Rozen, S., Madden, T. L. Primer-BLAST: a tool to design target-specific primers for polymerase chain reaction. BMC Bioinformatics. 13, 134 (2012).
- Green, M. R., Sambrook, J. . Molecular cloning: A laboratory manual. , (2012).
- Buckingham, L., Flaws, M. L. . Molecular diagnostics: Fundamentals, methods, and clinical applications. , (2012).
- Tan, S. C., Yiap, B. C. DNA, RNA, and Protein Extraction: The Past and The Present. Journal of Biomedicine and Biotechnology. 2009, (2009).
- Rapezzi, C., et al. Systemic cardiac amyloidoses: disease profiles and clinical courses of the 3 main types. Circulation. 120 (13), 1203-1212 (2009).
- Gertz, M. A., Dispenzieri, A., Sher, T. Pathophysiology and treatment of cardiac amyloidosis. Nature Reviews Cardiology. 12 (2), 91-102 (2015).
- Vrana, J. A., et al. Clinical diagnosis and typing of systemic amyloidosis in subcutaneous fat aspirates by mass spectrometry-based proteomics. Haematologica. 99 (7), 1239-1247 (2014).
- Nakamura, M., et al. Identification of a new transthyretin variant (Ile49) in familial amyloidotic polyneuropathy using electrospray ionization mass spectrometry and nonisotopic RNase cleavage assay. Human Heredity. 49 (4), 186-189 (1999).
- Ueda, M., Ando, Y. Recent advances in transthyretin amyloidosis therapy. Translational Neurodegeneration. 3, 19 (2014).
- Brown, E. E., et al. Genetic testing improves identification of transthyretin amyloid (ATTR) subtype in cardiac amyloidosis. Amyloid. 24 (2), 92-95 (2017).
- Tasaki, M., et al. Rapid detection of wild-type and mutated transthyretins. Annals of Clinical Biochemistry. 53 (4), 508-510 (2015).
- Plante-Bordeneuve, V., et al. Diagnostic pitfalls in sporadic transthyretin familial amyloid polyneuropathy (TTR-FAP). Neurology. 69 (7), 693-698 (2007).
- Hsu, J. L., et al. A prospective, observational study of patients with uncommon distal symmetric painful small-fiber neuropathy. PLoS ONE. 12 (9), 0183948 (2017).
- Salvi, F., Pastorelli, F., Plasmati, R., Bartolomei, I., Dall'Osso, D., Rapezzi, C. Genotypic and phenotypic correlation in an Italian population of hereditary amyloidosis TTR-related (HA-TTR): Clinical and neurophysiological aids to diagnosis and some reflections on misdiagnosis. Amyloid. 19, 58-60 (2012).
- Suhr, O. B., Lundgren, E., Westermark, P. One mutation, two distinct disease variants: Unravelling the impact of transthyretin amyloid fibril composition. Journal of Internal Medicine. 281 (4), 337-347 (2017).
- Vilà-Rico, M., et al. Quantitative analysis of post-translational modifications in human serum transthyretin associated with familial amyloidotic polyneuropathy by targeted LC-MS and intact protein MS. Journal of Proteomics. 127, 234-246 (2015).
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請さらに記事を探す
This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved