É necessária uma assinatura da JoVE para visualizar este conteúdo. Faça login ou comece sua avaliação gratuita.
Method Article
Análise genética de hereditária transtirretina Ala97Ser relacionados a amiloidose
Neste Artigo
Resumo
Aqui, apresentamos um protocolo para confirmar a presença da mutação de ponto para o diagnóstico de amiloidose hereditária transtirretina, usando Ala97Ser, a mutação mais comum endémica em Taiwan, por exemplo.
Resumo
Testes genéticos são o teste mais confiável para transtirretina hereditária relacionadas com amiloidose e deve ser realizado na maioria dos casos de amiloidose transtirretina (ATTR). ATTR é uma doença rara mas fatal com fenótipos heterogêneos; Portanto, o diagnóstico é por vezes adiado. Com o aumento da atenção e reconhecimento mais amplo sobre as manifestações iniciais de ATTR, bem como tratamentos emergentes, estudos de diagnósticos adequados, incluindo a transtirretina (TTR) genética teste, para confirmar os tipos e variantes de ATTR são, portanto fundamental para melhorar o prognóstico. Análises genéticas com métodos de reação em cadeia (PCR) polimerase confirmam a presença de mutações pontuais TTR muito mais rápido e mais seguro do que métodos convencionais tais como borrão do Sul. Aqui, demonstramos a confirmação genética da ATTR Ala97Ser mutação, a mutação mais comum endémica em Taiwan. O protocolo é composto por quatro etapas principais: coleta de amostra de sangue total, a extração de DNA, a análise genética de todos os quatro exões TTR com PCR e sequenciamento de DNA.
Introdução
Amiloidose transtirretina (TTR) (ATTR) é a forma mais comum de amiloidose sistêmica hereditária1e pode ser causada por uma mutação autossômica dominante herdada no gene transtirretina (TTR)2. Mutações de TTR desestabilizam a estrutura da proteína tetramérica e levam a sua dissociação em monômeros que reagrupa em amiloides2. Mais de 100 mutações de TTR amiloidogénicos foram relatados em todo o mundo1. Análises genéticas com métodos de reação em cadeia (PCR) polimerase confirmam a presença da mutação de ponto TTR e têm vantagens, incluindo evitar a manipulação de sondas radioactivamente etiquetadas em comparação com o borrão do Sul3. PCR é uma técnica rápida, fácil, barata e confiável que tem sido aplicada a inúmeros campos em ciências modernas4.
O diagnóstico precoce desta doença progressiva e fatal é um desafio, dada a sua heterogeneidade fenotípica. Com o aumento da atenção e reconhecimento mais amplo sobre as manifestações iniciais de ATTR, bem como as emergentes de tratamentos5, estudos de diagnóstico apropriados, incluindo teste genético de TTR, portanto, são criticamente fundamentais para melhorar o prognóstico. Além disso, diferentes mutações estão associados com penetrância diferente do traço, idade de início, os padrões de progressão, severidade da doença, a sobrevida mediana, eficácia do transplante de fígado, ou TTR estabilizadores2,6, e graus variáveis de envolvimento neurológico e cardiológico, que têm grandes implicações para o aconselhamento genético7,8,9. Além disso, um teste genético altamente preciso é a única ferramenta que diferencia os dois tipos distintos de ATTR: hereditária (mutante) e tipo selvagem (formulário não mutantes, amiloidose sistêmica senil, SSA)7. É imperativo para confirmar os tipos de ATTR, porque as terapias variam amplamente2. Portanto, há uma necessidade crescente para descrever o protocolo gradual do teste genético de TTR.
A abordagem molecular para detectar a mutação será ilustrada usando o Ala97Ser, a mutação mais comum endémica em Taiwan, por exemplo. Modificações na etapa de extração de DNA, reduzir a quantidade das três soluções usadas e produz uma quantidade suficiente de DNA. Neste protocolo, todos os quatro exões TTR foram analisados, enquanto regiões incluindo 5' montante 3' a jusante, os promotores, os intrões, e regiões untranslated (UTR) não foram sequenciadas.
Protocolo
Os testes realizados em laboratório foi realizado em conformidade com os requisitos do clínico laboratório melhoria alterações (CLIA), de 1988, os regulamentos aprovados pela institucional Review Board de Chang Gung Memorial Hospital e Universidade (licença n º 100 - 4470A3 e 2462A3-104). Foi obtido o consentimento de todos os pacientes.
1. coleta de amostras de sangue
- Colete sangue total em tubos de EDTA-Tratado comercialmente disponíveis. Misture delicadamente e armazenar a amostra de sangue a 4 ° C até o processamento.
2. o DNA extraído do sangue periférico
Usar um Kit de extração de DNA para a extração de DNA genômica (ver Tabela de materiais).
- Adicione 10 mL de solução 1 para a amostra de sangue de 4,0 mL. Incube a amostra no gelo por 10 min.
- Centrifugar a amostra a 2.000 x g por 5 min a 4 ° C. Remover e descartar o sobrenadante cuidadosamente. Resuspenda o pellet em 3 mL de solução 1.
- Centrifugar a amostra a 2.000 x g por 5 min a 4 ° C novamente e descartar o sobrenadante. Resuspenda o pellet em 3 mL de solução 2.
- Adicione 10 µ l de solução-mãe pronase (225 mg/mL) para obter uma concentração final de 100 µ g/mL e misture bem. Incube a 37 ° C durante a noite.
- Refrigere o tubo no gelo durante 10 min. Adicionar 0,8 mL de solução 3 para a amostra e inverter a 3 - 5 vezes. Coloque a amostra no gelo por 5 min.
- Centrifugar a amostra a 3.000 x g por 15 min a 4 ° C. Transferi com cuidado o sobrenadante para um tubo de centrifuga conico estéril 15 mL.
- Adicione 6 µ l da solução estoque do RNase (10 mg/mL) para obter uma concentração final de 20 µ g/mL. Incube a 37 ° C por 15 min.
- Adicione 2,5 mL de álcool isopropílico e misturar completamente suavemente várias vezes por inversão para precipitar o DNA (fios de material branco, floculante formarão). Usando uma pipeta de grande diâmetro, transferi o dessensibilizador de DNA para um novo tubo de centrifugação de 1,5 mL.
- Centrifugar a 12.000 x g por 3 min a 4 ° C. Desprezar o sobrenadante cuidadosamente. Pellet de DNA lave adicionando 500 µ l de etanol 70%.
- Centrifugar a 12.000 x g por 1 min e descartar o sobrenadante. Ar seca pelota do ADN à temperatura ambiente. Adicione 100 µ l de DDQ2O e incubar a 65 ° C por 15 min Ressuspender o DNA.
3. quantificação de DNA genômica
Use um espectrofotômetro (consulte a Tabela de materiais) para detectar a quantidade e a qualidade do DNA genômico.
- Faça logon no computador ligado à máquina Espectrofotômetro. Abra o software.
- Inicialize a máquina espectrofotômetro:
- Clique em "Ácidos nucleicos" sobre o software de Espectrofotômetro.
- Limpe o pedestal com uma toalhita de papel descartáveis e água purificada.
- Branco o espectrofotômetro:
- Carrega 2 µ l de água filtrada no pedestal.
- Abaixe o braço superior do espectrofotómetro e clique "Em branco" para calibrá-lo.
- Quando estiver pronto, levante o braço e o pedestal com uma limpeza a seco.
- Medida da amostra:
- Clique em "DNA-50" no tipo de amostra.
- Carrega 2 µ l de amostra de DNA no pedestal.
- Abaixe o braço superior e clique em "Medida" para medir a relação A260/A280 e a concentração da amostra.
- Limpe o pedestal entre cada execução com uma limpeza e água purificada.
- Clique em "Print Screen" para coletar os dados.
4. genéticas análises de mutações
Amplifica o ADN do alvo com a PCR4. Realizar a PCR em uma reação de 25 µ l kit de mistura usando uma DNA polimerase (ver Tabela de materiais). A tabela 1 mostra o gene TTR intrônicas cartilhas.
| Gene | Sequência da primeira demão |
| Exon1 | F: 5'-TCAGATTGGCAGGGATAAGC-3' R: 5'-GCAAAGCTGGAAGGAGTCAC-3' |
| Exon2 | F: 5'-TCTTGTTTCGCTCCAGATTTC-3' R: 5'-TCTACCAAGTGAGGGGCAAA-3' |
| Exon3 | F: 5'-GTGTTAGTTGGTGGGGGTGT-3' R: 5'-TGAGTAAAACTGTGCATTTCCTG-3' |
| Exon4 | F: 5'-GACTTCCGGTGGTCAGTCAT-3' R: 5'-GCGTTCTGCCCAGATACTTT-3' |
Tabela 1. As primeiras demão intrônicas de gene TTR. (Sequência de referência NCBI: NC_000018.10)
- Adicione os reagentes na tabela 2 , a um tubo PCR de 0,2 mL.
| Componentes | Volume (µ l) | Concentração final |
| 10 x Buffer | 2.5 | 1 x |
| MgCl2 (25 mM) | 1.5 | 1.5 mM |
| 360 GC Enhancer | 1 | - |
| 360 da DNA polimerase | 0.2 | 2 U/reação |
| dNTP Mix (25 mM cada) | 2 | 2 mM cada |
| Primeira demão F (10 µM) | 1 | 0,4 ΜM |
| Primeira demão R (10 µM) | 1 | 0,4 ΜM |
| DNA (100 ng) | 2 | |
| Água da PCR-classe | 13,8 | - |
| Volume total | 25 | - |
Tabela 2. Condições de reação de PCR.
- Misture delicadamente e centrifugue brevemente em uma centrífuga de bancada para coletar todos os componentes para o fundo do tubo. Colocar o tubo em um thermocycler.
- Realize o PCR para 30 ciclos. Cada ciclo consiste de uma etapa de desnaturação por 30 s a 94 ° C, primeira demão recozimento por 30 s a 58 ° C e extensão do polymerase para 45 s a 72 ° C (ver quadro 3).
| Passo | Temperatura (° C) | Tempo | ciclo de |
| Desnaturação inicial | 95 | 5 min | 1 |
| Passo de desnaturação do DNA | 94 | 30 s | 30 ciclos |
| Passo de recozimento da primeira demão | 58 | 30 s | 30 ciclos |
| Etapa da extensão do polymerase | 72 | 45 s | 30 ciclos |
| Etapa de alongamento do post | 72 | 10 min | 1 |
| Final do PCR ciclismo | 4 | Por tempo indeterminado |
Tabela 3. Condições de ciclismo para amplificar produtos do PCR.
- Valide o produto do PCR visualizando com eletroforese em gel:
- Prepare um gel de agarose 1,2%:
- Misturar 1,2 g de agarose em pó com 100 mL de 0.5 x TBE num balão microwavable. Microondas por 2 min até a agarose é completamente dissolvido. Arrefecer a mistura de agarose a 55 ° C.
- Adicionar 2 µ l de uma solução de brometo de etídio (10 mg/mL) para a mistura de agarose e misture delicadamente. Despeje a mistura de agarose em uma bandeja de gel para cerca de 5 a 7 mm, em seguida, adicione o comb(s) no lugar. Deixe-o solidificar (pelo menos 30 min) e remova cuidadosamente comb(s).
- Carregar amostras e executar um gel de agarose:
- Coloque o gel e bandeja para a célula de electroforese. Preencher a célula de eletroforese com 0,5 x TBE até que o gel é coberto.
- Cuidadosamente, carregar 2 µ l de uma 100 bp DNA ladder (ver Tabela de materiais) para a um faixa do gel como marcador de tamanho molecular. Mistura de corante/amostra de carga (5 µ l do mix de produtos PCR com 1 µ l 6 x tintura de carregamento) em pistas adicionais.
- Ligue a fonte de alimentação. Funcione o gel para 25 min a 100 V.
- Remova o gel da célula de electroforese. Visualize os fragmentos de DNA sob UV luz em um sistema da imagem latente.
- Prepare um gel de agarose 1,2%:
5. sequenciamento de DNA
- Purificar os produtos de PCR usando um kit comercial e sequenciar comercialmente ou com um sequenciador automático (consulte a Tabela de materiais).
- Envie sequências de nucleotídeos para o servidor de NCBI BLAST para comparar a consulta de bancos de dados o nucleotídeo10nucleotídeo. Depois que os resultados são exibidos, realizar alinhamentos de sequência e identificar a mutação esperada.
Resultados
Electroforese do gel de agarose de dois pacientes e uma saudável individual revelou bandas dos tamanhos esperados, incluindo um produto PCR bp 454 para exon 4 do gene TTR (Figura 1).
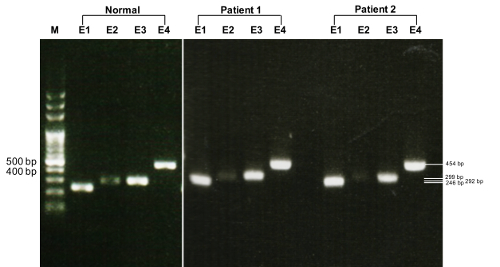
Figura 1 . Eletroforese em gel retratando PCR amplificados gene TTR. Normal: um indiví...
Discussão
Há duas etapas críticas dentro do protocolo. Em primeiro lugar, a fim de ter o número suficiente de células brancas do sangue, um espécime de hemodiluted deve ser evitada11. Em segundo lugar, o uso de primers PCR apropriados é fundamental para obter resultados fiáveis12. Usamos a ferramenta web de Primer-BLAST para projetar as primeiras demão4,13; um mínimo de 40 pares de base em cada lado dos exões TTR qu...
Divulgações
Os autores não têm nada para divulgar.
Agradecimentos
Gostaríamos de agradecer a Srta Wu Shin-diversão pela ajuda nos experimentos. Este estudo foi suportado por uma concessão do Chang Gung Medical Research Program (CMRPG3C0371, CMRPG3C0372, CMRPG3C0373) e IRB 100-4470A3, 104-2462A3, Taiwan.
Materiais
| Name | Company | Catalog Number | Comments |
| EDTA-treated tubes | BD | ||
| DNA Extraction Kit | Stratagen | 200600 | |
| NanoDrop ND2000 spectrophotometer | Thermo Fisher Scientific | NanoDrop 2000 | |
| Delicate Task Wipers | Kimberly-Clark | Kimtech Science Kimwipes | |
| AmpliTaq Gold 360 DNA Polymerase kit | Applied Biosystems | 4398823 | |
| TTR gene intronic primers | Exon1 | F: 5’-TCAGATTGGCAGGGATAAGC-3’ | |
| Exon1 | R: 5’-GCAAAGCTGGAAGGAGTCAC-3’ | ||
| Exon2 | F: 5’-TCTTGTTTCGCTCCAGATTTC-3’ | ||
| Exon2 | R: 5’-TCTACCAAGTGAGGGGCAAA-3’ | ||
| Exon3 | F: 5’-GTGTTAGTTGGTGGGGGTGT-3’ | ||
| Exon3 | R: 5’-TGAGTAAAACTGTGCATTTCCTG-3’ | ||
| Exon4 | F: 5’-GACTTCCGGTGGTCAGTCAT-3’ | ||
| Exon4 | R: 5’-GCGTTCTGCCCAGATACTTT-3’ | ||
| thermocycler | Applied Biosystems | GeneAmp PCR System 9700 | |
| electrophoresis cell | ADVANCE | Mupid-2plus | |
| DNA ladder | Protech | PT-M1-100 | |
| dye | BioLabs | B7021 | |
| AlphaImager EC | Alpha Innotech | AlphaImager EC | |
| automatic sequencer | Applied Biosystems | 3730xl DNA Analyzer |
Referências
- Planté-Bordeneuve, V., Said, G. Familial amyloid polyneuropathy. The Lancet Neurology. 10 (12), 1086-1097 (2011).
- Planté-Bordeneuve, V., et al. Long-term treatment of transthyretin familial amyloid polyneuropathy with tafamidis: a clinical and neurophysiological study. Journal of Neurology. 264 (2), 268-276 (2017).
- Nichols, W. C., Benson, M. D. Hereditary amyloidosis: detection of variant prealbumin genes by restriction enzyme analysis of amplified genomic DNA sequences. Clinical Genetics. 37 (1), 44-53 (1990).
- Lorenz, T. C. Polymerase Chain Reaction: Basic Protocol Plus Troubleshooting and Optimization Strategies. Journal of Visualized Experiments. (63), e3998 (2012).
- Hsu, H. C., et al. Phenotypic expressions of hereditary Transthyretin Ala97Ser related Amyloidosis (ATTR) in Taiwanese. BMC Neurology. 17 (1), 178 (2017).
- Barroso, F. A., et al. Long-term safety and efficacy of tafamidis for the treatment of hereditary transthyretin amyloid polyneuropathy: results up to 6 years. Amyloid. 24 (3), 194-204 (2017).
- Rapezzi, C., et al. Disease profile and differential diagnosis of hereditary transthyretin-related amyloidosis with exclusively cardiac phenotype: an Italian perspective. European Heart Journal. 34 (7), 520-528 (2013).
- Mariani, L. L., et al. Genotype-phenotype correlation and course of transthyretin familial amyloid polyneuropathies in France. Annals of Neurology. 78 (6), 901-916 (2015).
- Hammarström, P., Jiang, X., Hurshman, A. R., Powers, E. T., Kelly, J. W. Sequence-dependent denaturation energetics: A major determinant in amyloid disease diversity. Proceedings of the National Academy of Sciences of the United States of America. 99, 16427-16432 (2002).
- Johnson, M., Zaretskaya, I., Raytselis, Y., Merezhuk, Y., McGinnis, S., Madden, T. L. NCBI BLAST: a better web interface. Nucleic Acids Research. 36, 5-9 (2008).
- Fox, A. J., et al. Next generation sequencing for the detection of actionable mutations in solid and liquid tumors. Journal of Visualized Experiments. (115), (2016).
- Date, Y., Nakazato, M., Kangawa, K., Shirieda, K., Fujimoto, T., Matsukura, S. Detection of three transthyretin gene mutations in familial amyloidotic polyneuropathy by analysis of DNA extracted from formalin-fixed and paraffin-embedded tissues. Journal of the Neurological Sciences. 150 (2), 143-148 (1997).
- Ye, J., Coulouris, G., Zaretskaya, I., Cutcutache, I., Rozen, S., Madden, T. L. Primer-BLAST: a tool to design target-specific primers for polymerase chain reaction. BMC Bioinformatics. 13, 134 (2012).
- Green, M. R., Sambrook, J. . Molecular cloning: A laboratory manual. , (2012).
- Buckingham, L., Flaws, M. L. . Molecular diagnostics: Fundamentals, methods, and clinical applications. , (2012).
- Tan, S. C., Yiap, B. C. DNA, RNA, and Protein Extraction: The Past and The Present. Journal of Biomedicine and Biotechnology. 2009, (2009).
- Rapezzi, C., et al. Systemic cardiac amyloidoses: disease profiles and clinical courses of the 3 main types. Circulation. 120 (13), 1203-1212 (2009).
- Gertz, M. A., Dispenzieri, A., Sher, T. Pathophysiology and treatment of cardiac amyloidosis. Nature Reviews Cardiology. 12 (2), 91-102 (2015).
- Vrana, J. A., et al. Clinical diagnosis and typing of systemic amyloidosis in subcutaneous fat aspirates by mass spectrometry-based proteomics. Haematologica. 99 (7), 1239-1247 (2014).
- Nakamura, M., et al. Identification of a new transthyretin variant (Ile49) in familial amyloidotic polyneuropathy using electrospray ionization mass spectrometry and nonisotopic RNase cleavage assay. Human Heredity. 49 (4), 186-189 (1999).
- Ueda, M., Ando, Y. Recent advances in transthyretin amyloidosis therapy. Translational Neurodegeneration. 3, 19 (2014).
- Brown, E. E., et al. Genetic testing improves identification of transthyretin amyloid (ATTR) subtype in cardiac amyloidosis. Amyloid. 24 (2), 92-95 (2017).
- Tasaki, M., et al. Rapid detection of wild-type and mutated transthyretins. Annals of Clinical Biochemistry. 53 (4), 508-510 (2015).
- Plante-Bordeneuve, V., et al. Diagnostic pitfalls in sporadic transthyretin familial amyloid polyneuropathy (TTR-FAP). Neurology. 69 (7), 693-698 (2007).
- Hsu, J. L., et al. A prospective, observational study of patients with uncommon distal symmetric painful small-fiber neuropathy. PLoS ONE. 12 (9), 0183948 (2017).
- Salvi, F., Pastorelli, F., Plasmati, R., Bartolomei, I., Dall'Osso, D., Rapezzi, C. Genotypic and phenotypic correlation in an Italian population of hereditary amyloidosis TTR-related (HA-TTR): Clinical and neurophysiological aids to diagnosis and some reflections on misdiagnosis. Amyloid. 19, 58-60 (2012).
- Suhr, O. B., Lundgren, E., Westermark, P. One mutation, two distinct disease variants: Unravelling the impact of transthyretin amyloid fibril composition. Journal of Internal Medicine. 281 (4), 337-347 (2017).
- Vilà-Rico, M., et al. Quantitative analysis of post-translational modifications in human serum transthyretin associated with familial amyloidotic polyneuropathy by targeted LC-MS and intact protein MS. Journal of Proteomics. 127, 234-246 (2015).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados