Для просмотра этого контента требуется подписка на Jove Войдите в систему или начните бесплатную пробную версию.
Method Article
Генетический анализ наследственных Транстиретин Ala97Ser связанных амилоидоза
В этой статье
Резюме
Здесь мы представляем протокол для подтверждения наличия Точечная мутация для диагностики наследственных Транстиретин амилоидоза, используя Ala97Ser, наиболее распространенных эндемических мутации в Тайване, в качестве примера.
Аннотация
Генетическое тестирование является наиболее надежный тест для наследственных Транстиретин связанных амилоидоза и должна быть выполнена в большинстве случаев Транстиретин амилоидоз (ATTR). ATTR является редким, но роковой болезни с гетерогенными фенотипов; Следовательно диагноз иногда задерживается. С увеличением внимания и более широкого признания на ранних проявлений ATTR, а также новые процедуры, соответствующие диагностические исследования, включая Транстиретин (TTR) генетический тест, чтобы подтвердить типы и варианты ATTR поэтому фундаментальное значение для улучшения прогноза. Генетический анализ с помощью полимеразной цепной реакции (ПЦР) методов подтвердить наличие TTR точечные мутации гораздо быстрее и безопаснее, чем традиционные методы, такие как Южная помарка. Здесь мы демонстрируем Генетическое подтверждение ATTR Ala97Ser мутации, наиболее распространенных эндемических мутации в Тайване. Протокол включает четыре основных этапа: сбор образцов цельной крови, экстракции ДНК, генетический анализ всех четырех TTR экзонов с PCR и секвенирования ДНК.
Введение
Амилоидоз Транстиретин (TTR) (ATTR) является наиболее распространенной формой наследственной амилоидоз системный1и может быть вызвана аутосомно-преимущественно унаследованные мутации в ген Транстиретин (TTR)2. TTR мутации дестабилизировать структуры tetrameric белков и привести к ее диссоциации в мономеров, которые собирает в амилоидных фибрилл2. Более чем 100 amyloidogenic TTR мутации были сообщалось во всем мире1. Генетический анализ с помощью полимеразной цепной реакции (ПЦР) методов подтвердить наличие TTR Точечная мутация и имеют преимущества, включая избежать обработки радиоактивно помечены зонды по сравнению с Южная помарка3. ПЦР является быстрый, простой, дешевый и надежный метод, который был применен к многочисленным полям в современных наук4.
Ранняя диагностика этого прогрессивного и смертельной болезнью является сложной задачей, учитывая его фенотипической неоднородности. С увеличением внимания и более широкого признания на ранних проявлений ATTR, а также новых методов лечения5, соответствующие диагностические исследования, включая TTR генетический тест поэтому критически важное значение для улучшить прогноз. Кроме того, различные мутации связаны с различными пенетрантностью черта, возраст начала, узоры прогрессии, тяжести заболевания, медиана выживаемости, эффективность трансплантации печени, или TTR стабилизаторы2,6, и переменной степени неврологических и кардиологических участия, которые имеют большие последствия для генетическое консультирование7,8,9. Кроме того, высокоточные генетический тест является единственным инструментом, который отличает два различных вида ATTR: наследственные (мутанты) и дикого типа (форма не мутант, сенильных системных амилоидоза, SSA)7. Важно подтвердить типы ATTR, потому что лечения различаются2. Таким образом существует растущее необходимость описания ступенчатой протокол TTR генетического теста.
Молекулярный подход для выявления мутации будут проиллюстрированы с помощью Ala97Ser, наиболее распространенных эндемических мутации в Тайване, в качестве примера. Изменения в шаге экстракции ДНК уменьшить количество три решения, используемые и дает достаточное количество ДНК. В этом протоколе были проанализированы все четыре TTR экзонов, а регионы, в том числе 5' вверх по течению, 3' вниз по течению, промоутеров, интронов, и непереведенные регионов (утр) не были упорядочены.
протокол
Испытаний, проведенных в лаборатории была проведена в соответствии с требованиями из клинических лабораторных улучшение поправки (CLIA) 1988 года, положения, утвержденного институционального обзора Совет из Чанг Gung Мемориальный госпиталь и Университет (лицензия № 100 - 4470A3 и 104-2462A3). Информированное согласие было получено от всех пациентов.
1. кровь образец коллекции
- Сбор цельной крови в коммерчески доступных лечение ЭДТА трубы. Осторожно перемешать и хранить пробы крови при температуре 4 ° C до обработки.
2. ДНК извлечение из периферической крови
Используйте комплект экстракции ДНК для извлечения геномной ДНК (см. Таблицу материалы).
- Добавьте 10 мл раствора 1 4.0 мл крови. Проинкубируйте образцы на льду за 10 мин.
- Центрифугуйте образцы на 2000 x g 5 мин при 4 ° C. Снимите и выбросьте супернатант тщательно. Ресуспензируйте гранулы в 3 мл раствора 1.
- Центрифугуйте образцы на 2000 x g 5 минут при температуре 4 ° C снова и удалить супернатант. Ресуспензируйте гранулы в 3 мл раствора 2.
- 10 мкл раствора Проназа штока (225 мг/мл) для получения конечной концентрации 100 мкг/мл и хорошо перемешайте. Инкубируйте при 37 ° C на ночь.
- Chill трубку на льду для 10 минут добавить 0,8 мл раствора 3 образца и инвертировать 3 - 5 раз. Пример места на льду на 5 мин.
- Центрифугуйте образцы на 3000 x g 15 мин при 4 ° C. Тщательно передать супернатант 15 мл стерильного конические пластиковых пробирок.
- 6 мкл раствора РНКазы акций (10 мг/мл) для получения конечной концентрации 20 мкг/мл. Инкубируйте при 37 ° C 15 мин.
- Добавить 2,5 мл изопропилового спирта и смесь тщательно аккуратно переворачивать несколько раз, чтобы осадить ДНК (нити белого, хлопьевидный материал будет форма). С помощью пипетки крупнотоннажных, ДНК осадителя передать новых пластиковых пробирок 1.5 мл.
- Центрифуга на 12000 g x 3 мин при 4 ° C. Тщательно удалить супернатант. Вымойте ДНК Пелле, добавив 500 мкл на 70% спирте.
- Центрифуга на 12000 x г за 1 мин и удалить супернатант. Воздух сухой Пелле ДНК при комнатной температуре. 100 мкл ddH2O и Инкубируйте на 65 ° C на 15 минут, чтобы Ресуспензируйте ДНК.
3. геномной ДНК количественная оценка
Использовать спектрофотометр (см. Таблицу материалы), чтобы определить количество и качество геномной ДНК.
- Войдите в компьютер, подключенный к машине спектрофотометр. Открытое программное обеспечение.
- Инициализируйте спектрофотометр машины:
- Нажмите «Нуклеиновых кислот» на спектрофотометре программного обеспечения.
- Очистите пьедестал с одноразовые бумажные стирание и очищенной воды.
- Пустой спектрофотометра:
- Загрузка 2 мкл очищенной воды на пьедестал.
- Опустите верхнюю руку спектрофотометра и нажмите кнопку «Пустой» для калибровки его.
- Когда это делается, поднять руки и сухой пьедестал салфеткой.
- Мера образца:
- Нажмите на образец типа «ДНК-50».
- Загрузка 2 мкл образца ДНК на пьедестал.
- Опустите верхнюю руку и нажмите кнопку «Мера» измерить коэффициент A260/A280 и концентрация образца.
- Очистите пьедестал между каждого запуска с очистки и очищенной воды.
- Нажмите кнопку «Print Screen» для сбора данных.
4. генетический анализ мутаций
Усиливает ДНАО цели с ПЦР4. Выполнять ПЦР в 25 мкл реакции смесь с помощью ДНК-полимеразы комплекта (см. Таблицу материалы). Таблица 1 показывает TTR гена intronic грунтовки.
| Джин | Грунтовка последовательность |
| Exon1 | F: 5'-TCAGATTGGCAGGGATAAGC-3' R: 5'-GCAAAGCTGGAAGGAGTCAC-3' |
| Exon2 | F: 5'-TCTTGTTTCGCTCCAGATTTC-3' R: 5'-TCTACCAAGTGAGGGGCAAA-3' |
| Exon3 | F: 5'-GTGTTAGTTGGTGGGGGTGT-3' R: 5'-TGAGTAAAACTGTGCATTTCCTG-3' |
| Exon4 | F: 5'-GACTTCCGGTGGTCAGTCAT-3' R: 5'-GCGTTCTGCCCAGATACTTT-3' |
Таблицы 1. TTR гена intronic грунты. (NCBI ссылка последовательности: NC_000018.10)
- Добавьте реагентов в таблице 2 0.2 мл ПЦР-пробирку.
| Компоненты | Объем (мкл) | Конечная концентрация |
| 10 x буфер | 2.5 | 1 x |
| MgCl2 (25 мм) | 1.5 | 1,5 мм |
| 360 ГК Enhancer | 1 | - |
| 360 ДНК-полимераза | 0.2 | 2 U/реакция |
| dNTP смесь (по 25 мм) | 2 | 2 мм |
| Праймера F (10 мкм) | 1 | 0,4 МКМ |
| Грунтовка R (10 мкм) | 1 | 0,4 МКМ |
| ДНК (100 нг) | 2 | |
| ПЦР класс воды | 13,8 | - |
| Общий объем | 25 | - |
Таблица 2. Условия реакции PCR.
- Осторожно перемешать и кратко центрифуга настольная центрифуга для сбора всех компонентов в нижней части трубки. Место труб в Термоциклер.
- Выполните ПЦР для 30 циклов. Каждый цикл состоит из шага denaturation 30 s на 94 ° C, грунтовка, отжиг для 30 s на 58 ° C и расширения полимеразы 45 s при 72 ° C (см. таблицу 3).
| Шаг | Температура (° C) | Время | цикл |
| Первоначальный денатурации | 95 | 5 мин | 1 |
| Шага denaturation дна | 94 | 30 s | 30 циклов |
| Шаг отжига праймера | 58 | 30 s | 30 циклов |
| Полимеразы расширение шаг | 72 | 45 s | 30 циклов |
| Пост удлинение шаг | 72 | 10 мин | 1 |
| Конец ПЦР Велоспорт | 4 | Неопределенный |
Таблица 3. Велоспорт условия для усиления продукты PCR.
- Визуализация с электрофорезом геля для проверки продукта PCR:
- Подготовьте 1,2% агарозном геле:
- Смешать 1.2 g агарозы порошка с 100 мл 0,5 x TBE в микроволновой печи колбе. Микроволновая печь за 2 мин до тех пор, пока полностью не растворится агарозы. Охладьте вниз агарозы смесь до 55 ° C.
- Добавить 2 мкл раствора бромид ethidium (10 мг/мл) в смеси агарозы и осторожно перемешать. Вылейте смесь агарозы в каппу примерно 5-7 мм, а затем добавить comb(s) на месте. Позвольте ему затвердеть (по крайней мере 30 минут) и тщательно удалить comb(s).
- Загрузить образцы и запустить геля агарозы:
- Поместите гель и лоток в ячейку электрофореза. Заполните ячейки электрофорез с 0,5 x TBE до тех пор, пока гель покрыта.
- Тщательно нагрузки 2 мкл 100 bp ДНК лестница (см. Таблицу материалы) в одной майну гель как молекулярный размер маркера. Нагрузки краска/образец смесь (5 мкл номенклатура продуктов ПЦР с 1 мкл 6 x краска загрузки) в дополнительные полосы.
- Включите источник питания. Побегите гель на 25 мин на 100 V.
- Удалите гель из клетки электрофореза. Визуализируйте фрагментов ДНК под ультрафиолетовым светом в систему обработки изображений.
- Подготовьте 1,2% агарозном геле:
5. ДНК-последовательности
- Очистить продукты PCR, используя набор коммерческих и последовательность коммерчески или с автоматической секвенсор (см. Таблицу материалы).
- Отправьте нуклеотидные последовательности взрыва NCBI сервер сравнить запрос нуклеотидов в нуклеотидных баз данных10. После того, как результаты отображаются, выполняют выравниваний последовательности и определить ожидаемые мутации.
Результаты
Электрофорез геля агарозы двух пациентов и один здоровый отдельных выявленных полос ожидаемых размеров, в том числе 454 продукт PCR bp для экзона 4 TTR гена (рис. 1).
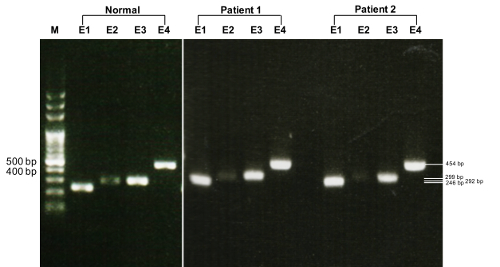
Рису...
Обсуждение
Существует два важных шага в рамках протокола. Во-первых для того чтобы иметь достаточное количество белых кровяных клеток, hemodiluted образец следует избегать11. Во-вторых использование соответствующих праймеры PCR имеет основополагающее значение для получения надежных резул?...
Раскрытие информации
Авторы не имеют ничего сообщать.
Благодарности
Мы хотим поблагодарить Мисс Ву Син-удовольствие за ее помощь в экспериментах. Это исследование было поддержано грант от Чанг Gung программа медицинских исследований (CMRPG3C0371, CMRPG3C0372, CMRPG3C0373) и IRB 100-4470A3, 104-2462A3, Тайвань.
Материалы
| Name | Company | Catalog Number | Comments |
| EDTA-treated tubes | BD | ||
| DNA Extraction Kit | Stratagen | 200600 | |
| NanoDrop ND2000 spectrophotometer | Thermo Fisher Scientific | NanoDrop 2000 | |
| Delicate Task Wipers | Kimberly-Clark | Kimtech Science Kimwipes | |
| AmpliTaq Gold 360 DNA Polymerase kit | Applied Biosystems | 4398823 | |
| TTR gene intronic primers | Exon1 | F: 5’-TCAGATTGGCAGGGATAAGC-3’ | |
| Exon1 | R: 5’-GCAAAGCTGGAAGGAGTCAC-3’ | ||
| Exon2 | F: 5’-TCTTGTTTCGCTCCAGATTTC-3’ | ||
| Exon2 | R: 5’-TCTACCAAGTGAGGGGCAAA-3’ | ||
| Exon3 | F: 5’-GTGTTAGTTGGTGGGGGTGT-3’ | ||
| Exon3 | R: 5’-TGAGTAAAACTGTGCATTTCCTG-3’ | ||
| Exon4 | F: 5’-GACTTCCGGTGGTCAGTCAT-3’ | ||
| Exon4 | R: 5’-GCGTTCTGCCCAGATACTTT-3’ | ||
| thermocycler | Applied Biosystems | GeneAmp PCR System 9700 | |
| electrophoresis cell | ADVANCE | Mupid-2plus | |
| DNA ladder | Protech | PT-M1-100 | |
| dye | BioLabs | B7021 | |
| AlphaImager EC | Alpha Innotech | AlphaImager EC | |
| automatic sequencer | Applied Biosystems | 3730xl DNA Analyzer |
Ссылки
- Planté-Bordeneuve, V., Said, G. Familial amyloid polyneuropathy. The Lancet Neurology. 10 (12), 1086-1097 (2011).
- Planté-Bordeneuve, V., et al. Long-term treatment of transthyretin familial amyloid polyneuropathy with tafamidis: a clinical and neurophysiological study. Journal of Neurology. 264 (2), 268-276 (2017).
- Nichols, W. C., Benson, M. D. Hereditary amyloidosis: detection of variant prealbumin genes by restriction enzyme analysis of amplified genomic DNA sequences. Clinical Genetics. 37 (1), 44-53 (1990).
- Lorenz, T. C. Polymerase Chain Reaction: Basic Protocol Plus Troubleshooting and Optimization Strategies. Journal of Visualized Experiments. (63), e3998 (2012).
- Hsu, H. C., et al. Phenotypic expressions of hereditary Transthyretin Ala97Ser related Amyloidosis (ATTR) in Taiwanese. BMC Neurology. 17 (1), 178 (2017).
- Barroso, F. A., et al. Long-term safety and efficacy of tafamidis for the treatment of hereditary transthyretin amyloid polyneuropathy: results up to 6 years. Amyloid. 24 (3), 194-204 (2017).
- Rapezzi, C., et al. Disease profile and differential diagnosis of hereditary transthyretin-related amyloidosis with exclusively cardiac phenotype: an Italian perspective. European Heart Journal. 34 (7), 520-528 (2013).
- Mariani, L. L., et al. Genotype-phenotype correlation and course of transthyretin familial amyloid polyneuropathies in France. Annals of Neurology. 78 (6), 901-916 (2015).
- Hammarström, P., Jiang, X., Hurshman, A. R., Powers, E. T., Kelly, J. W. Sequence-dependent denaturation energetics: A major determinant in amyloid disease diversity. Proceedings of the National Academy of Sciences of the United States of America. 99, 16427-16432 (2002).
- Johnson, M., Zaretskaya, I., Raytselis, Y., Merezhuk, Y., McGinnis, S., Madden, T. L. NCBI BLAST: a better web interface. Nucleic Acids Research. 36, 5-9 (2008).
- Fox, A. J., et al. Next generation sequencing for the detection of actionable mutations in solid and liquid tumors. Journal of Visualized Experiments. (115), (2016).
- Date, Y., Nakazato, M., Kangawa, K., Shirieda, K., Fujimoto, T., Matsukura, S. Detection of three transthyretin gene mutations in familial amyloidotic polyneuropathy by analysis of DNA extracted from formalin-fixed and paraffin-embedded tissues. Journal of the Neurological Sciences. 150 (2), 143-148 (1997).
- Ye, J., Coulouris, G., Zaretskaya, I., Cutcutache, I., Rozen, S., Madden, T. L. Primer-BLAST: a tool to design target-specific primers for polymerase chain reaction. BMC Bioinformatics. 13, 134 (2012).
- Green, M. R., Sambrook, J. . Molecular cloning: A laboratory manual. , (2012).
- Buckingham, L., Flaws, M. L. . Molecular diagnostics: Fundamentals, methods, and clinical applications. , (2012).
- Tan, S. C., Yiap, B. C. DNA, RNA, and Protein Extraction: The Past and The Present. Journal of Biomedicine and Biotechnology. 2009, (2009).
- Rapezzi, C., et al. Systemic cardiac amyloidoses: disease profiles and clinical courses of the 3 main types. Circulation. 120 (13), 1203-1212 (2009).
- Gertz, M. A., Dispenzieri, A., Sher, T. Pathophysiology and treatment of cardiac amyloidosis. Nature Reviews Cardiology. 12 (2), 91-102 (2015).
- Vrana, J. A., et al. Clinical diagnosis and typing of systemic amyloidosis in subcutaneous fat aspirates by mass spectrometry-based proteomics. Haematologica. 99 (7), 1239-1247 (2014).
- Nakamura, M., et al. Identification of a new transthyretin variant (Ile49) in familial amyloidotic polyneuropathy using electrospray ionization mass spectrometry and nonisotopic RNase cleavage assay. Human Heredity. 49 (4), 186-189 (1999).
- Ueda, M., Ando, Y. Recent advances in transthyretin amyloidosis therapy. Translational Neurodegeneration. 3, 19 (2014).
- Brown, E. E., et al. Genetic testing improves identification of transthyretin amyloid (ATTR) subtype in cardiac amyloidosis. Amyloid. 24 (2), 92-95 (2017).
- Tasaki, M., et al. Rapid detection of wild-type and mutated transthyretins. Annals of Clinical Biochemistry. 53 (4), 508-510 (2015).
- Plante-Bordeneuve, V., et al. Diagnostic pitfalls in sporadic transthyretin familial amyloid polyneuropathy (TTR-FAP). Neurology. 69 (7), 693-698 (2007).
- Hsu, J. L., et al. A prospective, observational study of patients with uncommon distal symmetric painful small-fiber neuropathy. PLoS ONE. 12 (9), 0183948 (2017).
- Salvi, F., Pastorelli, F., Plasmati, R., Bartolomei, I., Dall'Osso, D., Rapezzi, C. Genotypic and phenotypic correlation in an Italian population of hereditary amyloidosis TTR-related (HA-TTR): Clinical and neurophysiological aids to diagnosis and some reflections on misdiagnosis. Amyloid. 19, 58-60 (2012).
- Suhr, O. B., Lundgren, E., Westermark, P. One mutation, two distinct disease variants: Unravelling the impact of transthyretin amyloid fibril composition. Journal of Internal Medicine. 281 (4), 337-347 (2017).
- Vilà-Rico, M., et al. Quantitative analysis of post-translational modifications in human serum transthyretin associated with familial amyloidotic polyneuropathy by targeted LC-MS and intact protein MS. Journal of Proteomics. 127, 234-246 (2015).
Перепечатки и разрешения
Запросить разрешение на использование текста или рисунков этого JoVE статьи
Запросить разрешениеСмотреть дополнительные статьи
This article has been published
Video Coming Soon
Авторские права © 2025 MyJoVE Corporation. Все права защищены