このコンテンツを視聴するには、JoVE 購読が必要です。 サインイン又は無料トライアルを申し込む。
Method Article
細胞間の接触でタンパク質間相互作用の分析計蛍光の変動
要約
このプロトコルでは、直接細胞内の細胞接合における局在蛋白質すなわち細胞間相互作用を仲介する蛋白質間の相互作用を調査する変動蛍光分光法に基づくアプローチについて説明します。測定器の校正、データ集録および解析、修正可能なアーチファクトのソースを含む詳細なガイドラインを提供します。
要約
さまざまな生物学的プロセスには、通常隣接セル界面における相互作用するタンパク質を介した細胞間相互作用が含まれます。関心のだけほとんど試金は、具体的には徹底的な細胞に直接そのような相互作用が可能 します。細胞間の接触で、隣接する細胞の表面に発現するタンパク質の結合を測定するアッセイを紹介します。この試金は 2 つの手順で構成されています: 共焦点レーザー走査型顕微鏡を用いた細胞間の接触で蛍光揺らぎ分光測定が続く異なる蛍光タンパク質を融合した興味の蛋白質を発現する細胞の混合します。我々 は細胞間でアミロイドの前駆物質のような蛋白質 1 (APLP1) の相互作用を測定することによって生物学的文脈でこのアッセイの可能性を実証します。(走査蛍光相互相関分光法、相関数と明るさ解析) 蛍光ベースの技術と計測器の校正を使用してデータ集録の詳細なプロトコルを提供します。さらに、データ分析および識別して退色や細胞の動きのためのそれらなどの外部のスプリアス信号のバリエーションを修正する方法で重要な手順をについて説明します。
一般に、提示されたアッセイはホモ - または同一または異なる種類の細胞間に細胞間の接触、有性タンパク質間相互作用に適用され、商業共焦点レーザー走査型顕微鏡に実装することができます。重要な要件は、いくつかの分以上興味の蛋白質の拡散の動力学を調査するための十分なする必要がありますシステムの安定性です。
概要
多くの生物学的プロセスは、細胞間相互作用、例えば細胞間接着1,の2,3細胞融合4細胞認識5のサイトで発生します。このようなイベントは、多細胞生物の細胞間コミュニケーション、例えば、免疫反応時の開発時に特に重要です。これらのプロセスは通常表面、すなわち、隣接する細胞の膜 (PM) にローカライズされてで規制された空間と時間が正確に特定細胞間接触の相互作用を受ける蛋白質によって仲介されます。多くの場合、これらの相互作用は直接ホモ - または血液タンパク質トランス相互作用がイオンまたは細胞外リンカー1として配位子を伴うことがあります。基本的な重要性のプロービング生きている細胞のネイティブ環境で直接これらの特定タンパク質の相互作用アッセイの欠如があります。多くのメソッドいずれか必要セル中断 (co 免疫沈降6など生化学的アッセイなど)、固定 (例えば、いくつかの超解像光学顕微鏡検査の技術や細胞の電子顕微鏡観察7の連絡先)、または、非特定の例えば、集約接着アッセイ8,9/。この問題を克服するためには、蛍光共鳴エネルギー移動 (FRET)10または蛍光補完11に基づく蛍光技術を実施しています。ただし、fluorophores が付いて十分に小さい距離を達成するためにこれらのメソッド、蛋白質10、トランス相互作用と干渉する可能性がある細胞側の蛍光ラベルが必要です。
細胞間の接触でタンパク質間相互作用の代替蛍光を用いた測定を紹介します。このアプローチを組み合わせた蛍光相互相関方法 (スキャン蛍光相互相関分光法 (sFCCS)、相関数と明るさ (ccN と B)) との蛋白質の融合コンストラクトを発現する細胞の混合例えば、接着受容体、関心。2 つの相互作用する細胞の調査受容体が細胞内から 2 つのスペクトル分離蛍光タンパク質 (FPs) が付いています (図 1 aを参照)。
採用方法は、共焦点レーザー走査型顕微鏡の焦点音量を介し蛍光融合タンパク質の拡散運動による蛍光揺らぎの統計分析に基づいています。詳しくは、アッセイは、細胞間の接触の両方の近隣の PMs の興味の蛋白質の共同の拡散をプローブします。タンパク質は、トランス相互作用を受ける、これらトランス錯体は蛍光蛋白質両方のスペクトル チャネルに発光、両エミッタの蛍光相関変動の原因を運ぶ。その一方で、バインディングが発生しない PMs が直面しているタンパク質の数値変動は独立して相関の変動を引き起こしてないになります。買収は、2 つの方法で実行できます: 1) sFCCS 細胞間接触の間でライン状のスキャンに基づいており、プローブ接触領域にあるスポットでの相互作用を効果的に。蛍光変動の時空間分析を通じて sFCCS 情報を提供もダイナミクスすなわち、蛋白質複合体; の拡散係数2) ccN & B は、細胞間の接触領域で取得されるイメージのシーケンスの画素単位の分析に基づいています。プローブする能力を持ってする、ダイナミクスに関する情報は提供されません全体に沿ってマップ相互作用領域 (1 つの焦点面を使用) にお問い合わせください。両方の方法は、分子の明るさ、すなわち、平均蛍光信号は時間単位で単一拡散蛋白質複合体によって放出され、したがって、タンパク質複合体の化学量論の見積もりを提供の分析と組み合わせること細胞間の接触。
この記事では、試料調製、測定器の校正、データ集録および解析商業共焦点レーザー走査型顕微鏡の提示の分析を実行するための詳細なプロトコルを提供します。実験は、フォトンカウンティングまたはアナログ検出器、高開口数と目的を備えてあらゆる楽器で実行できます。さらに、プロトコルの重要な手順について説明し、artefactual 信号の変動、例えば、検出器雑音、退色や細胞の動きを引き起こしているいくつかのプロセスの補正方式を用意しています。もともと付着性のセル間の相互作用をプローブを開発し、アッセイ懸濁細胞の変更可能性がありますまたは適応モデル膜システム、例えば、巨大リポソーム (Guv) または巨大なプラズマ膜小胞 (GPMVs) できるように、異なる脂質環境や組織の骨格12,13の不在には相互作用の定量化。
走査蛍光相互相関分光蛍光相互相関分光法14の修正版は、脂質膜15の遅い拡散ダイナミクスを調べるために設計されました。それは興味の蛍光蛋白質を含んでいる午後に垂直なライン スキャン取得に基づいています。2 つの異なる分類された蛋白質種の相互作用を調査するには、買収はスペクトル分離 fluorophores のレーザーの 2 の行と 2 つの検出ウィンドウを使用して 2 つのスペクトル チャネルで実行されます。PM の蛋白質の遅い拡散ダイナミクスによる (D≤ ~ 1 μ m2/s)、15の行に行から励起方式を交互にクロス話無料測定を実行できます。分析から始まります: block-wise ~ 1000 行、2) 最大蛍光信号、すなわち、 PM 位置各ブロック内の位置の測定の平均と 3) シフトに基づく水平細胞運動のためのアルゴリズムの修正を配置 1)ための共通の起源12,15、各チャンネルで個別にすべてのブロック。その後、PM に対応するピクセルの自動選択は、すべて整列ライン (すなわちセンター ± 2.5σ) の合計のガウス近似から中央の領域を選択するによって実行されます。各ラインの信号の統合により、各チャンネルの膜の蛍光時間シリーズF(t) (g = グリーン チャンネル、r = 赤のチャネル)。ピクセル サイズが十分に小さくなど注意してください < 200 nm のポイントの形状広がり関数と PM の位置に対応する、その中心を見つけます。実質的な退色の存在下で各チャンネルの蛍光時間シリーズが二重指数関数でモデル化し、し、次の数式を修正:16
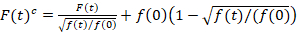 . (1)
. (1)
この数式は、振幅とF(t)c、裸眼F(t)から得られるパラメーターの推定値と比較しての相関分析から得られた拡散時の両方に効果的に補正に注意してくださいすることが重要です。自動・相互相関関数、(各種/艦砲射撃) 蛍光性の信号が計算されます。
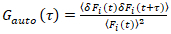 、(2)
、(2)
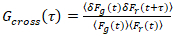 、(3)
、(3)
どこ δFは私私F(t) = 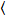 Fは私(t)
Fは私(t)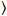 と私= g、r。
と私= g、r。
二次元拡散モデルは、すべての相関関数 (CFs) を装着されています。
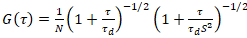 . (4)
. (4)
ここで、 Nは、各チャネルの拡散時間観測量とτd蛍光タンパク質の数を表します。このモデルは、記述されている実験の設定で PM におけるタンパク質の拡散は x-z 平面で発生した蛍光相関の一般的に使用される構成とは対照的分光法 (FCS) 実験膜プロービング考慮共焦点容積17の x y 平面に拡散。腰 w0と構造因子 S、伸長を記述する wz z、S の焦点の容積の wz/w0を =、スペクトルと同様の染料と同じ光設定実行ポイント FCS 校正測定から得られる拡散係数の既に使用可能な値を使用してD染料:
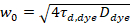 、(5)。
、(5)。
τd、染料がデータを 3次元拡散モデルをフィッティングから得られる色素の測定平均拡散時間を考慮する分数すべてN分子のTのアカウント切り替え、時定数と三重項状態ττ:
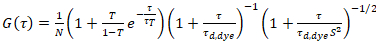 . (6)
. (6)
最後に、拡散係数 (D)、分子の明るさの値 (ε) と sFCCS データ (rel.cc.) の相対的な相互相関は次のように計算されます。
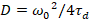 、(7)
、(7)
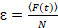 、(8)
、(8)
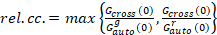 、(9)
、(9)
相互相関関数の振幅Gクロス(0) と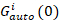 は、 i番目のチャネル チャネルの自己相関関数の振幅。
は、 i番目のチャネル チャネルの自己相関関数の振幅。
この定義の相対の相関、すなわち方程式 9 で意味の代わりにmaxを使用して、異なる濃度で現在 2 つの蛋白質種の複合体の最大数が制限されている考慮、種は低い数値で提示します。
相関数と明るさは、瞬間の分析に基づいてサンプルでは、固定位置で時間をかけて得た画像のスタックの各ピクセルの蛍光強度通常 100 〜 200 から成るフレーム、スペクトルの 2 つのチャンネル (g = グリーン チャンネル、r = 赤のチャネル)。時間平均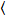 私
私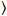 私と分散
私と分散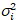 、各ピクセルのスペクトル チャネルで分子明るさ εiと番号の私n の計算 (私= g、 r)18。
、各ピクセルのスペクトル チャネルで分子明るさ εiと番号の私n の計算 (私= g、 r)18。
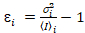 、(10)
、(10)
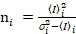 .(11)
.(11)
真フォトンカウンティング検出器の理想的な場合に与えられた方程式が適用されることに注意してくださいすることが重要です。アナログ検出システムでは、次の方程式は19,20を適用されます。
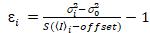 、(12)
、(12)
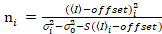 . (13)
. (13)
ここで、S は検出された光子と記録されたデジタル カウントの変換係数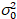 読み出しノイズは、オフセット検出器強度オフセットを参照します。一般に、これらの量は、安定した照明19、例えば、反射金属表面または乾燥色素溶液の強度の関数として検出器分散を測定に基づいて、任意の検出器型の校正する必要があります。オフセットは、励起光のないサンプルの計数率を測定することによって決定できます。検出器関連する差異の線形回帰を実行することによって
読み出しノイズは、オフセット検出器強度オフセットを参照します。一般に、これらの量は、安定した照明19、例えば、反射金属表面または乾燥色素溶液の強度の関数として検出器分散を測定に基づいて、任意の検出器型の校正する必要があります。オフセットは、励起光のないサンプルの計数率を測定することによって決定できます。検出器関連する差異の線形回帰を実行することによって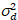 対強度 (私) プロット、S と
対強度 (私) プロット、S と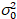 19決定をすることができます。
19決定をすることができます。
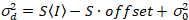 . (14)
. (14)
最後に、相互相関明るさは、各ピクセルの計算し、21として一般的に定義されます。
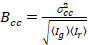 、(15)
、(15)
どこ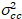 クロス分散です
クロス分散です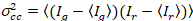 。
。
長寿命の変動をフィルターするためにすべての ccN & B 計算次のフィルタ リングされていない、独立して各ピクセル22有蓋車されます。簡単に言えば、 niε は私(私= g、 r) Bccは 8-15 フレームなどのセグメントをスライド式で計算されます。こうして得られた値を平均して最終的なピクセルの数と明るさの値を取得できます。
化学量論の分析
細胞間の接触でタンパク質複合体の化学量論組成を推定するために、sFCCS や ccN & B データの各スペクトル チャネルで分子の明るさを別々 に分析できます。SFCCS、各チャネルの測定あたり 1 つの輝度値を取得します。CcN & B は、細胞間の接触に対応するすべてのピクセルの輝度のヒストグラムが得られるし、測定の代表的な明るさとして平均 (または中央値) の値を使用できます。単量体の参照について同じ分析を実行する直接検出された蛋白質複合体の平均のオリゴマーの状態を取得するすべての明るさの値を正規化することができます。この時点で、オリゴマーの状態を過小評価可能性があります非蛍光性 FPs の存在を修正することが重要です。これは通常 1 色 sFCS または番号を使用して参照のホモ二量体蛋白質23,24の明るさと明るさ (N & B) を測定することによって実行されます。
Access restricted. Please log in or start a trial to view this content.
プロトコル
1. 前処理: 細胞混合アッセイ
注: 次のプロトコルは、付着性のセルのための混合の手順を説明します。それは懸濁液で培養された細胞に変更することがあります。
- 6 ウェル プレート、(カウント部屋ノイバウアーとカウント) 800,000 HEK 293 t 細胞などトランスフェクション前日細胞の適切な数を種子します。数は、播種、トランスフェクションの間の時間に応じて変更あり、他の細胞型の調整できます。基礎実験 (すなわち、利子および陰性対照の蛋白質) を実行するために少なくとも 4 井戸を準備します。37 ° c、5% CO2ダルベッコ変更イーグル培地 (DMEM) 培地での培養細胞は、ウシ胎児血清 (10%) と L-グルタミン (1%) 補われます。
- 製造元の指示に従ってセルを transfect (材料の表を参照してください)。
- 基本的な実験を実行するには、transfect 個別井戸、融合した (例えば、単量体強化された緑の蛍光蛋白質 (mEGFP)、または黄色い蛍光蛋白質 (mEYFP)) ' グリーン' または 'レッド' (例えばmCherry、興味の蛋白質のためのプラスミドまたは mCardinal) 蛍光タンパク質。
注: このプロトコルで我々 は APLP1 mEYFP と APLP1 mCardinal12, と対応する否定的な制御などにおける-パルミトイル-mEYFP (myr-ヤシ-mEYFP) - mCardinal (myr-ヤシ-mCardinal)12の焦点します。一般的には、200 ng の - 1 μ g のプラスミド DNA で十分です。高いトランスフェクション効率は、接触の '赤' と '緑' のセルを検索する機会を増やします。トランスフェクション効率を最適化するためにプラスミッド、トランスフェクション試薬の量を変更します。重要な:細胞株と細胞密度は約 70% でする必要があります。細胞は過剰合流、トランスフェクション効率が低下します。細胞が十分に合流、トランスフェクションと混合可能性がありますストレスを誘発して混合した後適切な添付ファイルから多くの細胞を防ぐ。
- 基本的な実験を実行するには、transfect 個別井戸、融合した (例えば、単量体強化された緑の蛍光蛋白質 (mEGFP)、または黄色い蛍光蛋白質 (mEYFP)) ' グリーン' または 'レッド' (例えばmCherry、興味の蛋白質のためのプラスミドまたは mCardinal) 蛍光タンパク質。
- トランスフェクション後~4 ± 2 h を混合セルを実行します。
- 成長培地を外し、各 1 mL の Mg2 +や Ca2 +を添加した PBS で優しく洗います。PBS を削除します。(重要です)洗濯中に細胞の剥離を防ぐためにもエッジに PBS をドロップします。
- 細胞の剥離を容易にするために各ウェルに drop-wise ~ 50 μ L トリプシン エチレンジアミン四酢酸 (EDTA) 溶液を追加します。その後、37 ° C で 2 分間インキュベート、セルをデタッチするのには横方向に 6 ウェル プレートをゆっくりと振る。
注: 拡張インキュベーション時間は細胞の種類によって要求されるかもしれない。 - 各ウェルに培 950 μ l 添加しそれにより井戸の底からすべてのセルをデタッチ上下に数回をピペッティングにより細胞を再懸濁します。(重要です)セルが正しく再停止して、再懸濁後大規模な細胞凝集塊の有無を調べるのため視覚的にチェックすることによって互いから切り離されたを確認します。それ以外の場合混合後多くの '赤'-'赤' または '緑'-'緑' 連絡先を取得します。
- 1 つの井戸 (ネガティブ コントロールや興味の蛋白質) の携帯ソリューションに転送への対応も、すなわち、 '赤' (例えば、 APLP1 mCardinal トランスフェクション) 'グリーン' (例えば、 APLP1 mEYFP トランスフェクション) 細胞。優しく上下に数回をピペッティングで混ぜます。、種子 35 mm ガラス底培養皿 (一皿、混合セル溶液 1 mL) プラス成長培地 1 mL と文化混合細胞を播種した細胞 37 ° C、5% CO2で別の日。

図 1.実験的ワークフローおよび走査蛍光相互相関分光法と細胞間の接触で相関数と明るさ解析の概略図。(A) 試料作製法: トランスフェクション後 2 つのスペクトルの異なる蛍光蛋白質 (例えば、 mEYFP および mCardinal) に溶ける (例えばAPLP1) の興味の蛋白質をトランスフェクトした 2 つの細胞集団を混在しています。顕微鏡実験で異なる transfected セルの連絡先が選択されます。細胞結合ドメインとの干渉を避けるためには、興味の蛋白質の細胞内の末端を蛍光タンパク質を融合する必要があります。(B) スキャン FCCS (sFCCS) の測定は、2 つのスペクトル チャンネル (チャンネル 1、緑と赤、2) 細胞間接触に垂直に実行されます。(Kymographs として表されます) のスキャン ラインに並べ、膜ピクセルの合計します。その後、各種と艦砲射撃がF私(t)強度の痕跡から計算されます。各種は、赤と緑で表されます。CCF は青で表されます。(C) 相関 N & B (ccN
2. 試料調製: 相関実験と明るさ解析のホモ二量体構造の肯定的な制御
- 35 mm ガラス底培養皿トランスフェクション前日に商工会議所を数えるセルとカウント、600,000 の HEK 293 t 細胞を播きます。37 ° c、5% CO2完全 DMEM 培地で細胞の培養 (手順 1.1 参照) 別の日。
- 〜 250 とセルを transfect の製造元の指示に従ってプラスミッド DNA の ng。肯定的な相互相関制御膜アンカー型蛍光タンパク質のヘテロは、ダイマーは、例えばmyr-ヤシ-mCherry-mEGFP または myr-ヤシ-mCardinal-mEYFP12興味の蛋白質の FPs に対応するエンコーディング プラスミドを使用します。輝度校正用膜アンカー型 FP モノマーと両方ホモ ダイマー myr-ヤシ-mEYFP-mEYFP、例えば、 myr パーム mEYFP 興味の蛋白質に溶ける FPs に対応するプラスミドを使用の明度分析を調整APLP1 mEYFP12。
- 37 ° c、5% CO2完全 DMEM 培地での細胞の培養 (手順 1.1 参照) 別の日。
3. 共焦点レーザー走査型顕微鏡: セットアップと焦点のボリューム調整
メモ: 次のプロトコルは、mEGFP/mEYFP、mCherry/mCardinal 走査共焦点顕微鏡の本研究で使用されるレーザーに関する実験の書き込まれます。光の設定、ソフトウェア設定 (レーザー ライン、ダイクロイック ミラー、フィルター) および校正染料の選択は、他の FPs と顕微鏡のセットアップの変更可能性があります。
- レーザーの安定性と温度の平衡を確認する実験の前に時間、少なくとも顕微鏡とレーザを入れます。
- 水または濃度が 10-50 nM の範囲で焦点のボリュームを調整する PBS の適切な水溶性蛍光染料ソリューション (例のためのテーブルの材料を参照) の 100-200 μ L を準備します。
- 色素溶液にすなわち、 0.16 0.19 の厚さを有するクリーンな 35 ミリメートルのガラス底皿 #1.5 mm。
注: は、理想的には、低の厚さの許容差、例えば0.170 ± 0.005 ミリメートル、最適な首輪リング補正 (ステップ 3.6) を許可を有する高性能カバー ガラスと料理を使用します。料理として次の実験の後の同じタイプを使用することが重要です。 - ソリューションに焦点を確保する目的 (好ましくは、水浸、NA 1.2) に直接染料の解決を含んでいる皿を配置します。また、サンプル ホルダーおよびフォーカスのサンプル (例えば10-20 皿の底の上の μ m) に料理を配置します。
注: 我々 は水溶液にディープに着目したとき、貧しい信号による石油目的を使用しては勧めません。 - 1 風通しの良い単位 (AU) のピンホール サイズ検出ウィンドウ 499 552 nm、488/561 nm ダイクロイック ミラー 488 nm レーザーを選択励起と放射の path を設定など。ピンホールの大きさが相関測定で使用されるものと同じであることを確認します。
- ピンホール位置 (ピンホール調整) とカウント率を最大限に客観的首輪リングを調整します。この目的には、最大カウント率が検出されるまで首輪リングを回します。
注: 首輪リング補正は使用されるカバーのガラスの厚さを占めています。すなわち計数率を最大化する。 できるだけ分子当たり多くの光子を収集、測定の信号対雑音比 (SNR) を最大化する非常に重要です。 - 相互相関で使われている同じレーザー パワーで一連のポイント FCS 測定 (例えば、 6 10 s、すなわち、 2.5 分合計時間、サンプリング滞留時間が 1 μ s 以下の 15 の繰り返しから成る別の場所で計測) を実行します。(通常、~ 1%、すなわち1 〜 2 μ) が測定できます。
- データへのトリプレット貢献 (方程式 6) を含む三次元拡散モデルに適合します。
注: 通常、得られた拡散時間が約 30 μ s と構造因子の周りは 4-8。 - 腰を計算する測定の平均拡散時間と 5 方程式によると室温25で使用される染料の拡散係数の公開された値から w0 。典型的な値は、200-250 nm です。
- 必要に応じて、2 番目の検出チャネルの異なる蛍光色素を校正ルーチン (ステップ 3.4 3.9) を繰り返します (例えば、 561 nm 励起と 570 の間検出 nm と 695 nm)。最初の検出チャンネルに設定されたピンホールの位置とサイズを維持します。
- 校正測定から分子の明るさ (方程式 8) を計算し、得られた値を格納します。
注: セットアップの使用の典型的な値は、 ~8-10 kHz/1.8 μ 488 nm 励起電源の分子 (MOL)。目的、セットアップまたは減らされたレーザー出力のミスアラインメントの汚れを示す可能性があります通常の価値よりも低い。チェックし、定期的にパワーメータを使用目的でレーザー出力を格納します。さまざまな設定の比較、励起レーザー出力で規格化した分子の明るさは顕微鏡の性能を評価する最もわかりやすいパラメーターです。
4. スキャン蛍光相互相関分光法: 買収
メモ: 次のプロトコルは、mEGFP/mEYFP (緑) と実験と mCherry/mCardinal ('赤') レーザー走査型共焦点顕微鏡に本研究で使用される書き込まれます。光のセットアップやソフトウェアの設定 (フィルター、ダイクロイック ミラー レーザー ライン) は、他の FPs または顕微鏡のセットアップの異なる場合があります。
- 光パス、例えば、 488 nm と 561 nm 励起 488/561 nm ダイクロイック ミラー、1 にピンホールを設定 488 nm 励起の AU。スペクトルのクロストークを避けるためには、興奮し、mEGFP/mEYFP (488 nm 励起、緑のチャネル) と mCherry を検出する 2 つの別々 のトラックを選択/mCardinal (561 nm 励起、赤のチャネル) 順番にスイッチ トラックすべてのラインを選択。検出のためには、チャネル、緑のチャネルで例えば499-552 nm と 570 695 nm の赤のチャネルの両方適切なフィルターを使用します。
- 交互の励起が可能でない場合はスペクトルのクロストークを最小限に抑える赤のチャンネルに適切なフィルター設定を使用 (すなわち600 以下 mCherry/mCardinal 蛍光を検出 nm)。これは赤のチャンネルで検出される光子の量を減らす、SNR を減らす可能性があります。
- サンプル ホルダーに混合セルを含んでいる皿を配置します。温度平衡を確認し、フォーカスずれを抑える少なくとも 10 分を待ちます。
- 検索メニューの光伝送を使用して細胞に焦点を当てます。
- '赤' と '緑' のセルが互いに接触のペアを検索します。明るさコントロール (セクション 2 を参照してください) にはホモ二量体または正の相関、午後チャネルまたはそれぞれのホモ二量体信号の両方で蛍光を発光孤立セルを検索します。
注: (重大)最小化サンプル露出済み漂白を避けるために細胞を捜している間相互相関26を減らすこと。したがって、最速のスキャン速度と低レーザーでスキャンします。強く発現細胞イメージングしながら検出器の飽和を避けるためには、統合モードで検索します。ただし、露出を最小限に抑えるために低いレーザーでスキャンするは、フォトンカウンティングモードで可能です。 - スキャン パス垂直細胞接触 (または肯定的な相互相関またはホモ二量体の明るさコントロールを 1 つのセルの午後) を選択、図 1 b 2 aで示されている、[トリミング] ボタンを使用します。
注: いくつかの古い顕微鏡のスキャン、任意の方向は認めません。この場合、細胞間の接触スキャン方向に垂直な向きで配置する必要。 -
50-200 nm とスキャン モードでの行を選択のピクセル サイズを達成するためにズームをします。128 × 1 ピクセルのフレーム サイズを設定します。
注: 一般的なピクセル サイズは 160 nm、約 20 μ m のスキャンの長さに対応します。 - スキャン速度に設定最大許容値、例えば、ラインごとに 472.73 μ 秒。
注: 代替励起方式のこれは 954.45 μ 秒スキャン時間、すなわち〜 1000 スキャン/秒使用される設定に対応します。興味の蛋白質の拡散係数に応じて、スキャンの速度を調整すること。膜蛋白質のため周辺に典型的な拡散時間 10-20 さんスキャン時間は少なくとも 10 倍未満の拡散時間する必要があります。下のスキャン速度が強く退色を誘導し、低い照明力を必要とします。また、一時停止などを課す 1 つ時系列サブメニューで間隔を利用した非常にゆっくりと拡散の複合の各スキャンの間に、5 ms。 - 適切なレーザー、488 の例えば1 〜 2 μ 選択 nm と 5 ~ 10 561 nm 励起 μ。
メモ: 高いレーザー、SNR の向上が退色を増やします。したがって、レーザーが退色が初期カウント率の 50% 未満になるように選択されます。 - 100,000 - 500, 000サイクルを設定します。
注: 番号スキャンのすなわち測定の期間が異なる場合があります: ただし、セルと退色の運動制限最大測定測定時間が長く SNR を向上させるし、ゆっくりと拡散分子のより適切な場合があります、時間。ここで表示されるデータが 3 〜 6 の買収された日常的に分、すなわち、 200,000 400,000 ライン スキャンします。 - 検出器をフォトンカウンティングモードに設定します。実験の開始、集録を開始するを押してください。別のセルを測定する 4.5 4.11 の手順を繰り返します。
注: 異なる表現のレベルでサンプルごとの 10-15 セルを測定することをお勧めします。(重要です)高式レベル検出器の飽和を避けるため。最大カウント率 〜 1 MHz を超えてはいけません。 - オリゴマーの状態を確認するのに明るさ解析が実施変更手順 4.1 4.11 に従ってホモ二量体輝度校正測定を実行: 別途 (隔離されたセル作製には各蛍光タンパク質ホモ ダイマーを測定プロトコル セクション 2)、スペクトルの 1 つのチャネルでのみ計測を実施します。
5. スキャン蛍光相互相関分光法: データ分析
注: 次のプロトコルで以前の記事12,15詳しく解析手順の実装に従います。ソフトウェア コードの作者にリクエスト可能です。
- 生データの形式で RGB TIFF イメージの生データ (例えばCZI) ファイルをエクスポートします。このファイルは緑と kymograph を含み、赤のチャネル データ チャネルと呼ばれる G および R のイメージのそれぞれ。
- 適切な分析ソフトウェアで TIFF ファイルをインポートして、分析を実行に進みます。
注: 次の手順 (手順 5.3 5.7) は、各チャンネルに別々 に適用されます。 - 500-1000 行ブロックをリフティングスキームまたは移動の時間平均を実行することによって行を合わせます。膜の位置、すなわち、最大カウント率は、各ブロック内のピクセル位置を決定します。すべてのブロックを同じ水平位置にシフトします。この手順は、細胞接触、例えば、細胞運動のための変形を修正します。
- 時間軸に沿って一直線に並べられたすべての行を合計し、ガウス関数を使用して平均強度プロファイルに適合します。重要な細胞内の背景の存在下でガウス分布に加えて、シグモイド関数を使用します。膜の位置と各時間ポイントを (すなわち、各ライン スキャンで) 単一蛍光信号値を取得する、各行のこれらのピクセルの強度を合計の ±2.5σ 内のすべてのピクセルと膜に対応するピクセルを定義します。
- 必要な場合 (例えば、背景 > 膜信号の 10%)、細胞質膜の蛍光は、1000 行のブロックから (ピクセル単位) で 2.5σ を乗じた平均画素値を減算することによってバック グラウンド補正を適用。背景のピクセルを選択するときは、明るい細胞内小胞を避けてください。
- 退色が見られる場合は、漂白の補正を適用します。したがって、二重指数関数を用いた膜蛍光時間シリーズに適合し、適切な補正式は、式 116を適用します。
注: あるいは、フーリエ スペクトル補正方式があります適用27。(重要です)CFs がひどく乱れるし、パラメーター推定値が偏って退色がないため修正が存在する場合 (例えば図 5Eを参照)。 - 各種、方程式 2 と 3 を使用すると、例えば、複数タウ アルゴリズム28によると艦砲射撃を計算します。分析の信頼性を向上させると工芸品を避けるために、10-20 全測定の等しいセグメントの計算を実行します。各セグメントの蛍光時間シリーズと CFs を検査し、明らかに歪んだセグメント (図 4 a- 4 Dの例を参照してください) を削除します。すべての歪みのないセグメントを平均します。
注: この手順は、データ29に主観的なバイアスを避けるために自動化できます。非常に不安定な測定の多くの短いセグメントを持っていることは、役に立つかもしれません。ただし、セグメントの長さはまだ上記の拡散を避けるために、少なくとも 3 桁をする必要がありますエラー29,30,17アンダー サンプリング統計。 - 二次元拡散モデル、得られた cfs の方程式 4 に適合します。したがって、校正測定 (プロトコル セクション 3) で得られた値に構造因子を修正します。フィットの精度は、各データ ポイントを取得して複数のタウ アルゴリズムから統計的重み係数を用いた重み付き近似を実行することで改善できます。
- 式 7 によると校正の腰を用いた拡散係数を計算します。
- 対応する方程式 8 粒子数で各チャンネルの平均蛍光強度を割ることによって分子の明るさを計算します。アカウントの非蛍光性の FPs23考慮オリゴマーの状態を取得する対応する単量体参照の平均の明るさによって各チャンネルの断固とした明るさの値を正規化します。この目的に非蛍光性 FPs23割合の計算に 1 色分析から平均ホモ二量体の明るさの値を決定します。
- 方程式 9 によると相対的な相互相関を計算します。
6. 相関数と明るさ: 検出器校正
注: 次のプロトコルは検出システムを調整する方法に関する一般的なガイドラインを提供します。この手順はアナログ検出システムに義務的ですが、真光子計数器を使用する場合厳密に必要はありません。
- 35 ミリメートルのガラス底皿の上適切な水溶性色素溶液 (例については材料の表を参照) を乾燥させます。光パスを設定する、すなわち、 488 561 nm 励起や 499 552 nm または 570 695 nm で検出それぞれ。
注: また、反射金属面使えます乾燥色素溶液の代わりに目的の上に直接金属部分を配置することによって。 - 地域別の色素濃度、異なるレーザーで 1 色 N & B 測定を実行します。したがって、300 のピクセル サイズを達成するためにズームを使用してスキャン速度を適切なピクセル滞留時間、例えば、 25 μ s、100-200 フレームにサイクルを設定する nm。
- フォトンカウンティング(またはアナログ モードアナログ検出と測定が実行される場合) に検出器を設定し、集録を開始する開始実験を押します。強度のオフセットを決定するゼロの励振電力で計測を実行します。
- ピクセルの分散を測定のすべてのピクセルのピクセルの輝度の関数としてプロットし、これらのデータの線形フィットを実行します。特定の直線近似の傾斜。S と方程式 14 に従って決められた強度オフセットを使用して、y 切片から読み出しノイズを計算します。
7. 相関数と明るさ: 買収
- SFCCS 獲得プロトコルの手順 4.1 4.4.
- 作物を使用して細胞間接触 (またはホモ二量体の明るさコントロールの分離の PM) 512 × 128 ピクセルのフレームを選択しますとズームを 50 ~ 100 nm のピクセル サイズを達成するために。
- スキャン速度を使用して、適切なピクセル滞留時間、例えば、 6.3 μ s を設定します。
注: N & B、画素のドウェル時間が興味の蛋白質の拡散時間よりもはるかに小さいでする必要があります。代替励起方式を選択した場合は、すべての行をトラック切り替えなど、 2 つのトラック間の時間は興味の蛋白質の拡散時間よりも小さくなければなりません。そうでなければ検出相関が低下します。 - 100-200 フレームにサイクルを設定します。
注: 高いフレーム番号は、sn 比が改善されます、ただし、細胞運動の総測定時間を制限可能性があります。フレームあたりのスキャン時間は興味の蛋白質の拡散時間よりもはるかに高いはずです。それ以外の場合明白な明るさが減少、すなわち粒子がモバイルに表示されます。非常にゆっくりと拡散の複合体を課す一時停止、例えば2 秒間に時系列サブメニューの間隔を使用してフレーム。 - レーザーを適切な値に設定 (典型的な値は、1 〜 2 μ 488 の nm と 5 ~ 10 561 nm 励起 μ)。
注: ハイパワー レーザーは、高輝度と改良された SNR も強化された退色につながります。レーザーは 〜 1 kHz 以上の検出された明るさを達成するために十分に高くする必要があります/MOL が以上の 10-20% の退色を避けるために十分に低い保った。10% 未満の mEGFP/mEYFP、mCherry/mCardinal、退色が取得されます通常。 - 検出器をフォトンカウンティング(またはアナログ モードアナログ検出と測定が実行される場合) に設定します。実験の開始、集録を開始するを押してください。
- 光子計数率を評価します。細胞接触ピクセルのカウント率は 1 MHz を超えている場合を減らすレーザー力または低発現レベルを持つセルを選択します。7.2 7.7 の手順を繰り返します。次のセルの組み合わせを測定します。異なった表現のレベルで実験ごとの 10-15 セルを測定することをお勧めします。
- 重合を定量化する明るさ解析を実行する場合は、変更された手順 7.1 7.7 に従ってホモ二量体明るさ校正計測を実行: 別途 (隔離されたセル作製には各蛍光タンパク質ホモ ダイマーを測定プロトコル セクション 2)、スペクトルの 1 つのチャネルでのみ計測を実施します。
8. 相関数と明るさ: データ分析
注:次のプロトコルは、前述の分析手順12,31従います。ソフトウェア コード リクエスト著者から可能です。
- Raw データをインポート (例えば、 CZI ファイルをインポートすることができます Bioformats32パッケージを使用して)。すべてのフレームを平均し、細胞間接触回避領域 (ROI) を選択します。
- 画像位置合わせアルゴリズム33、例えば、両方のチャネルを平均した任意の横方向翻訳のため後続のフレーム ・ ロワの空間相関を最大化して実行します。この手順は、セルの横方向の動きを修正します。
- 残留細胞運動などまたは背景を漂白から余分なの長期変動を減らすために有蓋車フィルター22を適用します。また、退色34を修正する detrending メソッドに適用できます。
注: 場合ないリフティングスキーム分析やトレンド除去が適用されます、明白な明るさは大きく過大評価される可能性があります。- など8 に 15 フレーム (例えばフレーム 1 に 8 2 9 など) のスライド式セグメントを定義し、方程式 10、11、15 の各セグメントで画素単位によるとチャネルとの相関の明るさの値を計算します。探知機でない真のフォトンカウンティング検出器、明るさ、すなわち、使用方程式 12 および 13 を代わりに計算するとき考慮校正器パラメーターを取る。
注: は、絶対的な明るさの 10-20% 過小評価と粒子数の 10-20% 過大評価につながる 8 に 15 フレームのセグメントで明るさの値を計算します。それにもかかわらず、明るさ比 (例えばモノマーの明るさにダイマー) は影響ない限り、セグメントの長さを分析 (データは示されていない) 全体で一定に保たれます。指定されたセグメントの長さの誤差はシミュレーションにより決定され、従って修正します。 - すべてのセグメント上得られた明るさを画素単位平均します。この手順では平均からセグメントの明るさの値の最大値と最小 5% を削除するか細胞内小胞または集合体がこれらの一時的存在、強さのために、例えば、明確な歪みを示すセグメントを除外することがあります 1 つピクセル。
- など8 に 15 フレーム (例えばフレーム 1 に 8 2 9 など) のスライド式セグメントを定義し、方程式 10、11、15 の各セグメントで画素単位によるとチャネルとの相関の明るさの値を計算します。探知機でない真のフォトンカウンティング検出器、明るさ、すなわち、使用方程式 12 および 13 を代わりに計算するとき考慮校正器パラメーターを取る。
- ピクセルの明るさの値をピクセル強度の関数としてプロットし、細胞間接触に対応するピクセル数を選択します。背景のピクセルは非常に低強度の値を持ちます。この時点で、最大カウント率を再評価します。パイルアップを防ぐために 1 MHz を超えるカウント レートを持つピクセルを除外します。
- 連絡先ピクセルの選択したセルのチャネルおよび相互相関の輝度ヒストグラムを作成し、投資収益率平均輝度値を取得します。アカウントの非蛍光性の FPs23考慮オリゴマーの状態を取得する対応する単量体参照の平均の明るさによって平均チャネルの輝度値を正規化します。したがって、非蛍光性 FPs23割合の計算に 1 色分析から平均ホモ二量体の明るさの値を決定します。
- 図では、チャネルおよび相互相関の明るさマップをプロットします。
Access restricted. Please log in or start a trial to view this content.
結果
最初のテスト蛋白質蛋白質の相互作用アッセイのすなわちsFCCS/ccN & B 測定 (図 1)、続いてスペクトルの異なる蛍光タンパク質を発現する細胞の混合で実行されるがタンパク質細胞細胞の接触 (すなわち、ネガティブ コントロール) で対話します。したがって、HEK 293 t 細胞における-パルミトイル-mEYFP (myr-ヤシ-mEYFP) または - mCardinal を?...
Access restricted. Please log in or start a trial to view this content.
ディスカッション
ここで説明した実験手順により、蛋白質蛋白質の調査は細胞間の接触、蛍光揺らぎ分光技術、すなわち sFCCS、ccN & B でトランス相互作用これらのメソッドは、それぞれ 1 つまたは他の融合蛋白質を表現する 2 つの隣接セルとの接触で興味の蛋白融合した 2 つのスペクトル分離 FPs によって放射される蛍光変動の統計的分析を含みます。トランス錯体の存在は、近隣の PMs のタンパ?...
Access restricted. Please log in or start a trial to view this content.
開示事項
著者が明らかに何もありません。
謝辞
この作品は、ドイツ研究振興協会 (DFG) によってを部分的に支持された 254850309 を付与します。著者は、論文の批判的な読みの Madlen リュクネールをありがとうございます。
Access restricted. Please log in or start a trial to view this content.
資料
| Name | Company | Catalog Number | Comments |
| DMEM growth medium | PAN-Biotech | P04-01548 | |
| DPBS w/o: Ca2+ and Mg2+ | PAN-Biotech | P04-36500 | |
| DPBS w: Ca2+ and Mg2+ | PAN-Biotech | P04-35500 | |
| Trypsin EDTA | PAN-Biotech | P10-023100 | |
| TurboFect Transfection Reagent | Thermo Fisher Scientific | R0531 | |
| HEK 293T cells | DSMZ | ACC 635 | |
| Alexa Fluor 488 NHS Ester | Thermo Fisher Scientific | A20000 | |
| Rhodamine B | Sigma-Alderich | 83689-1G | |
| Plasmid DNA | Addgene | NA | See reference 12 (Dunsing et. al., MBoC 2017),for a detailed description of all plasmids |
| 6-well plate | Starlab | CC7672-7506 | |
| 35-mm glass bottom dishes | CellVis | D35-14-1.5-N | |
| Zeiss LSM780 confocal | Carl Zeiss | NA | |
| MATLAB software package | MathWorks | 2015b | |
| Neubauer cell counting chamber | Marienfeld | 640110 |
参考文献
- Alberts, B., Johnson, A., Lewis, J., Raff, M., Roberts, K., Walter, P. Molecular biology of the cell. , Garland Science. (2002).
- Tepass, U., Truong, K., Godt, D., Ikura, M., Peifer, M. Cadherins in embryonic and neural morphogenesis. Nature Reviews Molecular Cell Biology. 1 (2), 91-100 (2000).
- Harris, T. J. C., Tepass, U. Adherens junctions: from molecules to morphogenesis. Nature Reviews Molecular Cell Biology. 11 (7), 502-514 (2010).
- Hernández, J. M., Podbilewicz, B. The hallmarks of cell-cell fusion. Development. 144 (24), Cambridge, England. 4481-4495 (2017).
- Huppa, J. B., Davis, M. M. T-cell-antigen recognition and the immunological synapse. Nature Reviews Immunology. 3 (12), 973-983 (2003).
- Kaden, D., Voigt, P., Munter, L. -M., Bobowski, K. D., Schaefer, M., Multhaup, G. Subcellular localization and dimerization of APLP1 are strikingly different from APP and APLP2. Journal of cell science. 122, Pt 3 368-377 (2009).
- Yap, A. S., Michael, M., Parton, R. G. Seeing and believing: recent advances in imaging cell-cell interactions. F1000Research. 4, F1000 Faculty Rev 273(2015).
- Kashef, J., Franz, C. M. Quantitative methods for analyzing cell-cell adhesion in development. Developmental Biology. 401 (1), 165-174 (2015).
- Soba, P., et al. Homo- and heterodimerization of APP family members promotes intercellular adhesion. The EMBO Journal. 24 (20), 3624-3634 (2005).
- Kim, S. A., Tai, C. -Y., Mok, L. -P., Mosser, E. A., Schuman, E. M. Calcium-dependent dynamics of cadherin interactions at cell-cell junctions. Proceedings of the National Academy of Sciences of the United States of America. 108 (24), 9857-9862 (2011).
- Feinberg, E. H., et al. GFP Reconstitution Across Synaptic Partners (GRASP) Defines Cell Contacts and Synapses in Living Nervous Systems. Neuron. 57 (3), 353-363 (2008).
- Dunsing, V., Mayer, M., Liebsch, F., Multhaup, G., Chiantia, S. Direct evidence of amyloid precursor-like protein 1 trans interactions in cell-cell adhesion platforms investigated via fluorescence fluctuation spectroscopy. Molecular biology of the cell. 28 (25), 3609-3620 (2017).
- Schneider, F., et al. Diffusion of lipids and GPI-anchored proteins in actin-free plasma membrane vesicles measured by STED-FCS. Molecular Biology of the Cell. 28 (11), 1507-1518 (2017).
- Bacia, K., Kim, S. A., Schwille, P. Fluorescence cross-correlation spectroscopy in living cells. Nature methods. 3 (2), 83-89 (2006).
- Ries, J., Schwille, P. Studying Slow Membrane Dynamics with Continuous Wave Scanning Fluorescence Correlation Spectroscopy. Biophysical Journal. 91 (5), 1915-1924 (2006).
- Ries, J., Chiantia, S., Schwille, P. Accurate Determination of Membrane Dynamics with Line-Scan FCS. Biophysical Journal. 96 (5), 1999-2008 (2009).
- Chiantia, S., Ries, J., Schwille, P. Fluorescence correlation spectroscopy in membrane structure elucidation. Biochimica et Biophysica Acta (BBA) - Biomembranes. 1788 (1), 225-233 (2009).
- Digman, M. A., Dalal, R., Horwitz, A. F., Gratton, E. Mapping the number of molecules and brightness in the laser scanning microscope. Biophysical journal. 94 (6), 2320-2332 (2008).
- Dalal, R. B., Digman, M. A., Horwitz, A. F., Vetri, V., Gratton, E. Determination of particle number and brightness using a laser scanning confocal microscope operating in the analog mode. Microscopy research and technique. 71 (1), 69-81 (2008).
- Unruh, J. R., Gratton, E. Analysis of Molecular Concentration and Brightness from Fluorescence Fluctuation Data with an Electron Multiplied CCD Camera. Biophysical Journal. 95 (11), 5385-5398 (2008).
- Digman, M. A., Wiseman, P. W., Choi, C., Horwitz, A. R., Gratton, E. Stoichiometry of molecular complexes at adhesions in living cells. Proceedings of the National Academy of Sciences of the United States of America. 106 (7), 2170-2175 (2009).
- Hellriegel, C., Caiolfa, V. R., Corti, V., Sidenius, N., Zamai, M. Number and brightness image analysis reveals ATF-induced dimerization kinetics of uPAR in the cell membrane. The FASEB journal official publication of the Federation of American Societies for Experimental Biology. 25 (9), 2883-2897 (2011).
- Dunsing, V., Luckner, M., Zühlke, B., Petazzi, R. A., Herrmann, A., Chiantia, S. Optimal fluorescent protein tags for quantifying protein oligomerization in living cells. Scientific Reports. 8 (1), 10634(2018).
- Chen, Y., Johnson, J., Macdonald, P., Wu, B., Mueller, J. D. Observing Protein Interactions and Their Stoichiometry in Living Cells by Brightness Analysis of Fluorescence Fluctuation Experiments. Methods in enzymology. 472, 345-363 (2010).
- Kapusta, P. Absolute Diffusion Coefficients: Compilation of Reference Data for FCS Calibration. , Available from: https://www.picoquant.com/images/uploads/page/files/7353/appnote_diffusioncoeffients.pdf (2010).
- Foo, Y. H., Naredi-Rainer, N., Lamb, D. C., Ahmed, S., Wohland, T. Factors affecting the quantification of biomolecular interactions by fluorescence cross-correlation spectroscopy. Biophysical journal. 102 (5), 1174-1183 (2012).
- Baum, M., Erdel, F., Wachsmuth, M., Rippe, K. Retrieving the intracellular topology from multi-scale protein mobility mapping in living cells. Nature Communications. 5, 4494(2014).
- Wohland, T., Rigler, R., Vogel, H. The standard deviation in fluorescence correlation spectroscopy. Biophysical journal. 80 (6), 2987-2999 (2001).
- Ries, J., et al. Automated suppression of sample-related artifacts in Fluorescence Correlation Spectroscopy. Optics Express. 18 (11), 11073(2010).
- Ries, J., Schwille, P. New concepts for fluorescence correlation spectroscopy on membranes. Physical Chemistry Chemical Physics. 10 (24), 3487(2008).
- Mayer, M. C., et al. Amyloid precursor-like protein 1 (APLP1) exhibits stronger zinc-dependent neuronal adhesion than amyloid precursor protein and APLP2. Journal of Neurochemistry. 137 (2), 266-276 (2016).
- Linkert, M., et al. Metadata matters: access to image data in the real world. The Journal of Cell Biology. 189 (5), 777-782 (2010).
- Trullo, A., Corti, V., Arza, E., Caiolfa, V. R., Zamai, M. Application limits and data correction in number of molecules and brightness analysis. Microscopy Research and Technique. 76 (11), 1135-1146 (2013).
- Nolan, R., et al. nandb-number and brightness in R with a novel automatic detrending algorithm. Bioinformatics. 33 (21), Oxford, England. 3508-3510 (2017).
- Hammond, G. R. V., Sim, Y., Lagnado, L., Irvine, R. F. Reversible binding and rapid diffusion of proteins in complex with inositol lipids serves to coordinate free movement with spatial information. The Journal of cell biology. 184 (2), 297-308 (2009).
- Hendrix, J., Dekens, T., Schrimpf, W., Lamb, D. C. Arbitrary-Region Raster Image Correlation Spectroscopy. Biophysical journal. 111 (8), 1785-1796 (2016).
- Hendrix, J., et al. Live-cell observation of cytosolic HIV-1 assembly onset reveals RNA-interacting Gag oligomers. The Journal of cell biology. 210 (4), 629-646 (2015).
- Hendrix, J., Schrimpf, W., Höller, M., Lamb, D. C. Pulsed Interleaved Excitation Fluctuation Imaging. Biophysical Journal. 105 (4), 848-861 (2013).
- Honigmann, A., et al. Scanning STED-FCS reveals spatiotemporal heterogeneity of lipid interaction in the plasma membrane of living cells. Nature Communications. 5 (1), 5412(2014).
- Chojnacki, J., et al. Envelope glycoprotein mobility on HIV-1 particles depends on the virus maturation state. Nature Communications. 8 (1), 545(2017).
- Godin, A. G., et al. Revealing protein oligomerization and densities in situ using spatial intensity distribution analysis. Proceedings of the National Academy of Sciences of the United States of America. 108 (17), 7010-7015 (2011).
- Müller, J. D., Chen, Y., Gratton, E. Resolving Heterogeneity on the Single Molecular Level with the Photon-Counting Histogram. Biophysical Journal. 78 (1), 474-486 (2000).
- Kim, S. A., Heinze, K. G., Bacia, K., Waxham, M. N., Schwille, P. Two-Photon Cross-Correlation Analysis of Intracellular Reactions with Variable Stoichiometry. Biophysical Journal. 88 (6), 4319-4336 (2005).
- Jenkins, E., et al. Reconstitution of immune cell interactions in free-standing membranes. Journal of cell science. 132 (4), (2018).
Access restricted. Please log in or start a trial to view this content.
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請さらに記事を探す
This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved