È necessario avere un abbonamento a JoVE per visualizzare questo. Accedi o inizia la tua prova gratuita.
Method Article
Un'analisi di spettroscopia di fluorescenza fluttuazione delle interazioni proteina-proteina a contatti cellula-cellula
In questo articolo
Riepilogo
Questo protocollo descrive un approccio di basato su spettroscopia di fluorescenza fluttuazione per indagare le interazioni tra proteine che mediano interazioni cellula-cellula, cioè proteine localizzate nelle giunzioni delle cellule, direttamente nelle cellule viventi. Forniamo le linee guida dettagliate sulla calibrazione dello strumento, acquisizione dati e analisi, comprese le correzioni al fonti possibili manufatto.
Abstract
Una varietà di processi biologici coinvolge interazioni cellula-cellula, in genere mediati dalle proteine che interagiscono a livello di interfaccia tra le cellule vicine. Di interesse, solo pochi saggi sono in grado di sondare specificamente tali interazioni direttamente nelle cellule viventi. Qui, presentiamo un test per misurare l'associazione di proteine espresse sulle superfici delle cellule vicine, a contatti cellula-cellula. Questo test è costituito da due passaggi: miscelazione delle cellule che esprimono le proteine di interesse fusa a diverse proteine fluorescenti, seguita da misure di spettroscopia di fluorescenza fluttuazione a contatti cellula-cellula utilizzando un microscopio a scansione confocale del laser. Dimostriamo la fattibilità di questo test in un contesto biologicamente rilevante misurando le interazioni della proteina amiloide precursore-come 1 (APLP1) attraverso giunzioni della cellula-cellula. Forniamo protocolli dettagliati sull'acquisizione dati utilizzando tecniche basate sulla fluorescenza (scansione di spettroscopia di correlazione di fluorescenza, il numero di cross-correlazione e analisi di luminosità) e la taratura dello strumento richiesto. Ulteriormente, discutiamo passaggi critici nell'analisi dei dati e come identificare e correggere le variazioni di segnale esterno, spurie, come quelle dovute a movimenti photobleaching o cella.
In generale, il saggio presentato è applicabile a qualsiasi omo - o interazioni eterotipiche proteina-proteina a contatti cellula-cellula, fra le cellule dei tipi uguali o diversi e può essere implementato su un commerciale scansione microscopio confocale del laser. Un requisito importante è la stabilità del sistema, che deve essere sufficiente per sondare dinamica diffusiva delle proteine di interesse per diversi minuti.
Introduzione
Molti processi biologici verificano presso i siti delle interazioni cellula-cellula, per esempio, cellula-cellula adesione1,2,3, cellula-cellula fusion4 e riconoscimento cellulare5. Tali eventi sono particolarmente importanti durante lo sviluppo degli organismi multicellulari e per la comunicazione cellula-cellula, per esempio, durante le risposte immunitarie. Questi processi sono in genere mediati dalle proteine che sono localizzate alla superficie, cioè, alla membrana del plasma (PM) delle cellule vicine e sottoposti a specifiche interazioni al contatto cellula-cellula che sono precisamente regolamentato in spazio e tempo. In molti casi, queste interazioni sono diretto omo - o eterotipiche interazioni della proteina-proteina trans , ma possono anche coinvolgere gli ioni o ligandi che agiscono come linker extracellulare1. Sebbene di fondamentale importanza, c'è una mancanza di saggi sondando queste interazioni proteina-proteina specifica direttamente nell'ambiente nativo di cellule viventi. Molti metodi di distruzione cellulare (ad es., analisi biochimiche come co-immunoprecipitazione6), richiedono la fissazione (ad es., alcune delle tecniche di Super-risoluzione microscopia ottica e microscopia elettronica delle cellule contatti7), o sono non-specifici, ad esempio, aggregazione / adesione saggi8,9. Per ovviare a questo problema, sono state implementate tecniche di fluorescenza basata sulla fluorescenza resonance energy transfer (FRET)10 o fluorescenza complementazione11. Tuttavia, per raggiungere distanze sufficientemente piccole tra fluorofori, questi metodi richiedono etichette fluorescenti sul lato extracellulare delle proteine10, potenzialmente interferenti con interazioni di trans .
Qui, presentiamo un'analisi di fluorescenza-basata alternativa per le interazioni proteina-proteina a contatti cellula-cellula. Questo approccio combina approcci di cross-correlazione di fluorescenza (scansione spettroscopia di correlazione di fluorescenza (sFCCS), numero di cross-correlazione e luminosità (ccN & B)) e la miscelazione delle cellule che esprimono un costrutto di fusione della proteina di interesse, ad esempio, un recettore di adesione. I recettori studiati nelle due cellule interagenti sono etichettati con due proteine fluorescenti spettralmente separate (FPs), dall'intracellulare (Vedi Figura 1A).
I metodi impiegati sono basati sull'analisi statistica delle fluttuazioni di fluorescenza indotta dal movimento diffusivo delle proteine di fusione fluorescenti attraverso il volume focale di un microscopio a scansione confocale del laser. Più in dettaglio, il test sonde co-diffusione delle proteine di interesse in entrambi vicini PMs a contatti cellula-cellula. Se le proteine subiscono interazioni trans , questi complessi trans porterà proteine fluorescenti che emettono in entrambi i canali spettrali, causando fluttuazioni di fluorescenza correlata di entrambi gli emettitori. D'altra parte, se si verifica alcuna associazione, le fluttuazioni numero di proteine nell'affrontare PMs sarà indipendente, non causando fluttuazioni correlate. L'acquisizione può essere eseguita in due modi: 1) sFCCS si basa su una scansione a forma di linea attraverso il contatto cellula-cellula e sonde in modo efficace le interazioni in un punto situato nella regione del Contatta. Attraverso un'analisi temporale delle fluttuazioni di fluorescenza, sFCCS fornisce anche informazioni dinamiche, cioè, i coefficenti di diffusione dei complessi della proteina; 2) ccN & B si basa su un'analisi di immagine di una sequenza di immagini acquisite presso le regioni di contatto cellula-cellula. Esso ha la capacità di sondare e mappa interazioni lungo tutta la regione (in un piano focale) di contatto, ma non fornisce informazioni sulle dinamiche. Entrambi i metodi possono essere combinati con un'analisi della luminosità molecolare, cioè, il segnale di fluorescenza media emessa nell'unità di tempo da complessi proteici diffusore singolo e, quindi, fornire stime della stechiometria dei complessi della proteina a contatti cellula-cellula.
In questo articolo, forniamo dettagliati protocolli per la preparazione del campione, calibrazione dello strumento, acquisizione dati e analisi ad eseguire l'analisi presentata su un commerciale scansione microscopio confocale del laser. Gli esperimenti possono essere eseguiti su qualsiasi strumento dotato di conteggio di fotoni o rivelatori analogici e un obiettivo con apertura numerica elevata. Abbiamo ulteriormente discutere passaggi critici del protocollo e forniscono schemi di correzione per diversi processi che causano fluttuazioni artefactual segnale, ad esempio, rivelatore rumore, movimento photobleaching o cella. Originariamente sviluppato per sondare le interazioni tra cellule aderenti, il dosaggio può essere modificato per cellule in sospensione, o adattati ai sistemi di membrane modello, ad esempio, le vescicole unilamellari gigante (GUV) o plasma gigante vescicole di membrana (GPMVs), che consente la quantificazione delle interazioni in lipidi diversi ambienti o in assenza di un citoscheletro organizzato12,13.
Spettroscopia di correlazione di fluorescenza di scansione è una versione modificata di spettroscopia di correlazione di fluorescenza14 ed è stato specificamente progettato per sonda dinamica diffusiva lenta del lipido membrane15. Si basa su un'acquisizione di scansione linea perpendicolare al PM contenenti le proteine fluorescenti di interesse. Per sondare le interazioni delle due specie di proteina marcata in modo diverso, l'acquisizione viene eseguita in due canali spettrali utilizzando due linee laser e due finestre di rilevamento per fluorofori spettralmente separati. A causa della dinamica lenta diffusione di proteine nel PM (D≤ ~ 1 µm2/s), una misura di cross-talk-free possa essere eseguita alternando lo schema di eccitazione da linea a linea15. L'analisi inizia con: 1) un algoritmo di allineamento correggere per movimento laterale cellulare basato su block-wise con una media di ~ 1000 righe, 2) determinazione della posizione con fluorescenza massimo segnale, cioè, il PM posizione ogni blocco e 3) mutevole di tutti i blocchi per una comune origine12,15, separatamente in ciascun canale. Quindi, una selezione automatica dei pixel corrispondenti al PM viene eseguita selezionando la regione centrale da una vestibilità gaussiana della somma di tutte le linee allineate (cioè, centro ± 2.5σ). Integrazione del segnale in ogni linea produce la serie temporale fluorescenza a membrana f (t) in ciascun canale (g = verde canale, r = canale rosso). Nota che la dimensione in pixel deve essere abbastanza piccola, ad esempio, < 200 nm, per ricostruire la forma del punto di diffondere funzione e trovare il suo centro, corrispondente alla posizione del PM. In presenza di sostanziale photobleaching, le serie temporali di fluorescenza in ciascun canale possono essere modellate con una funzione di doppio-esponenziale e poi corretto con la seguente formula:16
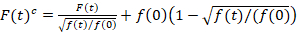 . (1)
. (1)
È importante notare che questa formula corregge efficacemente sia le ampiezze e tempi di diffusione ottenuti da analisi di correlazione di f (t)c, rispetto alle stime dei parametri che si otterrebbe dalla non corretta f (t). Quindi, le funzioni di auto - e cross-correlazione (ACFs / CCFs) della fluorescenza segnali vengono calcolati:
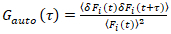 , (2).
, (2).
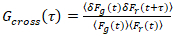 , (3).
, (3).
dove δFho = Fio(t) - 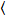 Fio(t)
Fio(t)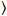 ed io = g, r.
ed io = g, r.
Un modello bidimensionale diffusione è poi montato su tutte le funzioni di correlazione (CFs):
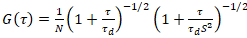 . (4)
. (4)
Qui, N indica il numero di proteine fluorescenti nel volume di osservazione e τd il tempo di diffusione per ogni canale. Questo modello prende in considerazione che in ambito sperimentale descritto, diffusione delle proteine nel PM si verifica nel piano x-z, in contrasto con la configurazione comunemente usata di correlazione di fluorescenza spettroscopia (FCS) esperimenti sulle membrane di sondaggio diffusione nel piano x-y del confocale volume17. La vita w0 e il fattore di struttura S, descrivendo l'allungamento wz del volume focale in z, S = wz/w0, sono ottenuti da una misura di calibrazione punto FCS effettuata con coloranti spettralmente simili e stesse impostazioni ottiche utilizzando valori già disponibili per il coefficente di diffusione Dtintura:
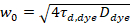 , (5).
, (5).
dove τd, colorante è il tempo di diffusione medio misurato delle molecole della tintura, ottenuta da un modello tridimensionale diffusione ai dati, di montaggio tenendo le transizioni di conto di una frazione T di tutte le molecole di N per un stato di tripletto con una costante di tempo ττ:
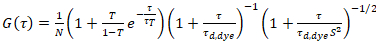 . (6)
. (6)
Infine, i coefficenti di diffusione (D), i valori di luminosità molecolare (ε) e la relativa correlazione incrociata dei dati sFCCS (rel.cc.) sono calcolati come segue:
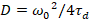 , (7).
, (7).
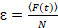 , (8).
, (8).
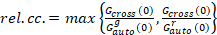 , (9).
, (9).
dove Gcross(0) è l'ampiezza della funzione di correlazione incrociata e 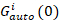 è l'ampiezza della funzione di autocorrelazione nel canale dei -esima.
è l'ampiezza della funzione di autocorrelazione nel canale dei -esima.
Questa definizione della relativa correlazione incrociata, cioè utilizzando max invece di dire all'equazione 9, tiene conto del fatto che il numero massimo dei complessi di due specie proteiche presenti alle concentrazioni differenti è limitato per il specie presenti in un numero inferiore.
Luminosità e il numero di Cross-correlazione si basa su un'analisi del momento dell'intensità di fluorescenza per ogni pixel di una serie di immagini acquisite nel tempo ad un in posizione fisso nel campione, costituiti in genere da ~ 100-200 fotogrammi, con due spettrali canali ( g = verde canale, r = canale rosso). Dalla media temporale 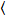 ho
ho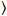 e la varianza
e la varianza 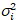 , la luminosità molecolare εi e numero nmi vengono calcolate in ogni pixel e canale spettrale (io = g, r)18:
, la luminosità molecolare εi e numero nmi vengono calcolate in ogni pixel e canale spettrale (io = g, r)18:
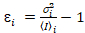 , (10).
, (10).
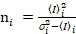 . (11)
. (11)
È importante notare che le equazioni determinate si applicano al caso ideale di un vero rilevatore photon-counting. Per sistemi di rivelazione analogica, le seguenti equazioni applicano19,20:
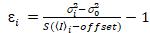 , (12).
, (12).
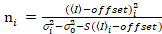 . (13)
. (13)
Qui, S è il fattore di conversione tra fotoni rilevati ed i conteggi digitali registrati, 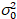 è il rumore di lettura e offset si riferisce all'offset di intensità del rivelatore. Generalmente, queste quantità devono essere calibrate, per qualsiasi tipo di rivelatore, basato su misura la varianza del rivelatore in funzione dell'intensità per illuminazione costante19, ad esempio, una superficie di metallo riflettente o soluzione colorante secchi. L' offset può essere determinato misurando la velocità di conteggio per un campione senza luce di eccitazione. Eseguendo una regressione lineare della varianza rivelatore-associated
è il rumore di lettura e offset si riferisce all'offset di intensità del rivelatore. Generalmente, queste quantità devono essere calibrate, per qualsiasi tipo di rivelatore, basato su misura la varianza del rivelatore in funzione dell'intensità per illuminazione costante19, ad esempio, una superficie di metallo riflettente o soluzione colorante secchi. L' offset può essere determinato misurando la velocità di conteggio per un campione senza luce di eccitazione. Eseguendo una regressione lineare della varianza rivelatore-associated 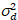 versus trama di intensità (mi), S e
versus trama di intensità (mi), S e 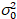 può essere determinato19:
può essere determinato19:
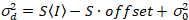 . (14)
. (14)
Infine, la luminosità di cross-correlazione è calcolata in ogni pixel ed è definita in generale come21
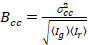 , (15).
, (15).
dove 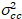 è la croce-varianza
è la croce-varianza 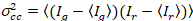 .
.
Per filtrare le fluttuazioni longeve, tutti ccN & calcoli B vengono eseguiti seguendo un vagone coperto del filtro, in modo indipendente per ogni pixel22. Brevemente, ni, εho (ho = g, r) e Bcc sono calcolati in segmenti di ad esempio, 8-15 infissi scorrevoli. I valori così ottenuti possono essere fatte la media poi per ottenere il pixel finale numero e la luminosità.
Analisi di stechiometria
Al fine di stimare la stechiometria dei complessi della proteina a contatti cellula-cellula, la luminosità molecolare possa essere analizzata separatamente in ciascun canale spettrale per il sFCCS o ccN & dati B. In sFCCS, si ottiene un valore di luminosità per misura in ciascun canale. In ccN & B, si ottiene un istogramma di luminosità di tutti i pixel corrispondenti al contatto cellula-cellula e il valore medio (o mediano) può essere utilizzato come luminosità rappresentativo per la misurazione. Eseguendo la stessa analisi su un riferimento monomerico, tutti i valori di luminosità possono essere normalizzati per ottenere direttamente lo stato oligomerico medio dei complessi proteici rilevati. A questo punto, è importante correggere per la presenza di FPs non fluorescente che può comportare una sottovalutazione dello stato oligomerico. Questo viene in genere eseguito misurando la luminosità di un homo-dimerica riferimento proteina23,24 utilizzando SFC un colore o numero e luminosità (N & B).
Protocollo
1. preparazione del campione: Cella di miscelazione dosaggio
Nota: Il seguente protocollo descrive la procedura di miscelazione per le celle aderenti. Può essere modificato per le cellule coltivate in sospensione.
- Un numero adeguato di cellule su una piastra a 6 pozzetti, per esempio, 800.000 cellule HEK 293T (contati con un Neubauer camera di conteggio), un giorno prima di transfezione del seme. Il numero può essere modificato a seconda del periodo tra la semina e la transfezione e regolato per altri tipi di cellule. Per eseguire un esperimento di base (cioè, proteine di interesse e di controllo negativo), preparare almeno 4 pozzi. Cellule di coltura a 37 ° C, 5% CO2 in mezzo di Eagle Medium per volta (DMEM) di Dulbecco, completati con siero bovino fetale (10%) e L-Glutammina (1%).
- Transfect celle secondo le istruzioni del produttore (Vedi la Tabella materiali).
- Per eseguire un esperimento di base, transfect, in pozzetti separati, plasmidi per la proteina di interesse fuso a un 'verde' (ad es., monomerico avanzata proteina fluorescente verde (mEGFP), o la proteina fluorescente gialla (mEYFP)) o il 'rosso' (ad es., mCherry, o mCardinal) proteina fluorescente.
Nota: In questo protocollo, ci concentriamo su APLP-1-mEYFP e APLP1-mCardinal12e il controllo negativo corrispondente, ad esempio, miristoilate-palmitoilata-mEYFP (myr-palm-mEYFP) e - mCardinal (myr-palm-mCardinal)12. In genere, 200 ng - 1 µ g di DNA plasmidico sono sufficienti. Efficienza di trasfezione alta aumenta la possibilità di trovare le celle 'rosse' e 'verde' in contatto. Modificare la quantità di plasmide e transfezione reagente per ottimizzare l'efficienza di trasfezione. Critico: Confluenza delle cellule dovrebbe essere di circa il 70% quando la trasfezione le cellule. Se le cellule sono sovra-confluenti, farà diminuire l'efficienza di trasfezione. Se le cellule non sono abbastanza, confluente transfezione e miscelazione può indurre stress e prevenire molte cellule da un collegamento corretto dopo la miscelazione.
- Per eseguire un esperimento di base, transfect, in pozzetti separati, plasmidi per la proteina di interesse fuso a un 'verde' (ad es., monomerico avanzata proteina fluorescente verde (mEGFP), o la proteina fluorescente gialla (mEYFP)) o il 'rosso' (ad es., mCherry, o mCardinal) proteina fluorescente.
- Eseguire la miscelazione ~4 ± 2 ore dopo la trasfezione delle cellule.
- Rimuovere il mezzo di crescita e lavare ogni bene delicatamente con 1 mL di PBS completati con Mg2 + e Ca2 +. Quindi, rimuovere il PBS. (Critico) Goccia PBS sul bordo bene per evitare il distacco delle cellule durante il lavaggio.
- Aggiungere la soluzione di ~ 50 µ l della tripsina Etilendiamminotetraacetico acido (EDTA) drop-wise ad ogni pozzetto per facilitare il distacco delle cellule. Incubare a 37 ° C per 2 min in seguito, lentamente agitare la piastra 6 pozzetti lateralmente per staccare le cellule.
Nota: Tempi di incubazione prolungato potrebbero essere necessari per alcuni tipi di cellule. - Aggiungere 950 µ l di terreno di coltura ad ogni pozzetto e risospendere le cellule pipettando un paio di volte su e giù, quindi staccare tutte le celle dalla parte inferiore di ben. (Critico) Garantire che le cellule sono risospese correttamente e staccate da altro controllando visivamente per l'assenza di aggregati di grandi cellule dopo la risospensione. In caso contrario si otterranno molti contatti 'verde' 'Rossi'-'Rossi' o 'verde' - dopo la miscelazione.
- Trasferire la soluzione di cella di un pozzo (proteina di interesse o di controllo negativo) al corrispondente, vale a dire, 'rosso' (ad es., APLP1-mCardinal transfettate) 'verde' (ad es., APLP1-mEYFP transfettate) cellule. Mescolare pipettando delicatamente un paio di volte su e giù. Quindi, seme le cellule miste su piatti di fondo di vetro 35mm (1 mL di soluzione di cellule miste per ogni piatto), oltre a 1 mL di terreno di coltura e cultura seminato cellule per un altro giorno a 37 ° C, 5% CO2.

Figura 1 . Flusso di lavoro sperimentale e rappresentazione schematica della scansione di spettroscopia di correlazione di fluorescenza e analisi di numero e luminosità di cross-correlazione a contatti cellula-cellula. (A) schema di preparazione del campione: due popolazioni di cellule trasfettate con la proteina di interesse (ad es., APLP1) fusa a due proteine fluorescenti spettralmente distinte (ad es., mEYFP e mCardinal) sono mescolate dopo trasfezione. Contatti delle cellule transfettate in modo diverso sono selezionati negli esperimenti di microscopia. Per evitare interferenze con domini di legame extracellulare, la proteina fluorescente dovrebbe essere fusa al capolinea intracellulare della proteina di interesse. (B) misure di scansione FCCS (sFCCS) sono eseguiti perpendicolarmente al contatto cellula-cellula in due canali spettrali (canali 1, verde e rosso, 2). Linee di scansione (rappresentati come chimografi) sono allineati e pixel di membrana sommati. Quindi, ACFs e CCFs sono calcolati dalle tracce intensità Fio(t). ACFs sono rappresentati in rosso e verde. CCF è rappresentato in blu. (C) Cross-correlazione N & B (ccN & B) acquisizione si traduce in una pila tridimensionale immagine (x-y-time). Un ROI è selezionato intorno al contatto cellula-cellula. Quindi luminosità canale e cross-correlazione (ε1, ε2e Bcc) i valori sono calcolati in ciascun pixel di contatto cellula-cellula. I risultati vengono quindi visualizzati come istogrammi, pool di tutti i pixel selezionati. Clicca qui per visualizzare una versione più grande di questa figura.
2. preparazione del campione: Controllo positivo per esperimenti di Cross-correlazione e Homo-dimero costrutto per l'analisi di luminosità
- 600.000 cellule HEK 293T, conteggiate con una cella camera, su piatti di 35 mm vetro inferiore un giorno prima di transfezione di conteggio del seme. Le cellule a 37 ° C, 5% CO2 in terreno DMEM completo della coltura (Vedi punto 1.1) per un altro giorno.
- Transfect cellule con ~ 250 ng di DNA plasmidico secondo le istruzioni del produttore. Per il controllo positivo di cross-correlazione, utilizzare un plasmide che codifica un proteina fluorescente ancorata alla membrana etero-dimero, per esempio, myr-palm-mCherry-mEGFP o myr-palm-mCardinal-mEYFP12 corrispondenti alla FPs della proteina di interesse. Per la calibrazione della luminosità, utilizzare plasmidi codifica sia un ancorata alla membrana FP monomero e homo-dimero corrispondente alla FPs fusa per la proteina di interesse, ad esempio, myr-palm-mEYFP e myr-palm-mEYFP-mEYFP per calibrare l'analisi di luminosità di APLP1-mEYFP12.
- Coltura di cellule a 37 ° C, 5% CO2 in terreno DMEM completo (Vedi punto 1.1) per un altro giorno.
3. confocale microscopia: Installazione e calibrazione Volume focale
Nota: Il seguente protocollo è scritto per gli esperimenti condotti con mEGFP/mEYFP e mCherry/mCardinal sul microscopio confocale utilizzato in questo studio di scansione laser. La configurazione ottica, le impostazioni del software (linee laser, specchi dicroici, filtri) e la scelta di calibrazione coloranti possono essere modificati per altre configurazioni di FPs e microscopio.
- Accendere il microscopio e il laser almeno un'ora prima dell'esperimento per garantire stabilità di laser e l'equilibratura della temperatura.
- Preparare 100-200 µ l di soluzioni appropriate idrosolubile colorante fluorescente (Vedi la Tabella materiali per esempi) in acqua o in PBS per calibrare il volume focale, con le concentrazioni nell'intervallo 10-50 nM.
- Posizionare le soluzioni di tintura su un piatto da fondo pulito 35 mm vetro #1.5, cioè, avere uno spessore di 0.16-0.19 mm.
Nota: Idealmente, usare piatti con copertura in vetro ad alte prestazioni con una tolleranza di basso spessore, ad esempio, 0,170 ± 0,005 mm, che permette un'ottimale collo anello correzione (punto 3.6). È importante utilizzare lo stesso tipo di piatto come utilizzato in seguito per i seguenti esperimenti. - Mettere il recipiente contenente la soluzione di colorante direttamente sull'obiettivo (preferibilmente, l'immersione in acqua, con NA 1.2) per garantire la messa a fuoco nella soluzione. In alternativa, posizionare il piatto sulla porta campione e messa a fuoco nel campione (ad es., 10-20 µm sopra il fondo del piatto).
Nota: Si sconsiglia di utilizzare obiettivi di olio dovuto il povero segnale ottenuto quando concentrandosi profondo in campioni acquosi. - Impostare il percorso di eccitazione e di emissione, per esempio, scegliere la 488 nm del laser, uno specchio dicroico di 488/561 nm, rilevamento finestra 499-552 nm e una dimensione di foro di spillo di 1 unità arioso (AU). Assicurarsi che la dimensione del foro stenopeico è identico a quello che verrà utilizzato nelle misure di correlazione incrociata.
- Regolare la posizione di foro stenopeico (regolazione pinhole) e l'anello del collare oggettivi per massimizzare il tasso di conteggio. A questo scopo, è necessario girare dell'anello del collare fino a quando non viene rilevato il tasso di conteggio massimo.
Nota: La correzione di anello collare rappresenta lo spessore specifico del vetro di copertura utilizzato. Massimizzare il tasso di conteggio, vale a dire., raccogliendo come molti fotoni per molecola come possibile, è fondamentale per massimizzare il rapporto segnale-rumore (SNR) delle misurazioni. - Eseguire una serie di misure punto FCS (ad es., 6 misure alle posizioni differenti, ciascuno composto da 15 ripetizioni di 10 s, vale a dire 2,5 min tempo totale, campionata con tempo di permanenza di 1 µs o meno) a parità di potenza laser utilizzato in cross-correlazione misure (in genere ~ 1%, cioè, ~ 1-2 µW).
- Montare un modello tridimensionale diffusione compreso un contributo di tripletto (equazione 6) ai dati.
Nota: In genere, i tempi di diffusione ottenuti sono circa 30 µs e il fattore di struttura è di circa 4-8. - Calcolare la vita w0 dal tempo di diffusione medio misurato e valori pubblicati per il coefficente di diffusione del colorante usato a temperatura ambiente25 secondo l'equazione 5. I valori tipici sono 200-250 nm.
- Ripetere la procedura di calibrazione (passaggi 3.4-3.9) con un colorante fluorescente differente per un secondo canale di rilevamento se necessario (ad es., 561 nm eccitazione e rilevamento tra 570 nm e 695 nm). Mantenere la posizione foro stenopeico e dimensioni come è stato fissato per il primo canale di rilevamento.
- Calcolare la luminosità molecolare (equazione 8) dalle misurazioni di calibrazione e memorizzare i valori ottenuti.
Nota: I valori tipici per il setup usato sono ~8-10 kHz/molecola (MOL) per 1,8 µW 488 nm il potere di eccitazione. Inferiori ai valori usuali potrebbero indicare lo sporco sull'obiettivo, il disallineamento di un uscita ridotta laser o l'installazione. Controllare e memorizzare potenze laser all'obiettivo regolarmente utilizzando un misuratore di potenza. Per il confronto di diverse configurazioni, luminosità molecolare normalizzata per la potenza del laser di eccitazione è il parametro più significativo per valutare la performance del microscopio.
4. spettroscopia di correlazione di fluorescenza di scansione: acquisizione
Nota: Il seguente protocollo è scritto per gli esperimenti condotti con mEGFP/mEYFP ('verde') e mCherry/mCardinal ('rosso') sul microscopio confocale a scansione laser utilizzati in questo studio. L'installazione di ottica e le impostazioni del software (linee laser, specchi dicroici, filtri) possono essere diverse per altre configurazioni di FPs o microscopio.
- Impostare il percorso ottico, ad es., 488 nm e 561 eccitazione di nanometro e 488/561 nm specchio dicroico, pinhole 1 AU per l'eccitazione di 488 nm. Per evitare interferenze spettrali, selezionare due tracce separate per eccitare e rilevare mEGFP/mEYFP (488 nm eccitazione, canale verde) e mCherry / mCardinal (561 nm eccitazione, canale rosso) in sequenza e selezionare interruttore tiene traccia di ogni linea. Per il rilevamento, è possibile utilizzare filtri appropriati per entrambi i canali, per esempio, 499-552 nm nel verde del canale e 570-695 nm nel canale del rosso.
- Se eccitazione alternati non è possibile, utilizzare le impostazioni di filtro appropriato per il canale rosso per minimizzare il colloquio trasversale spettrale (cioè rilevare mCherry/mCardinal fluorescenza non inferiore a 600 nm). Questo può ridurre la quantità di fotoni rilevati nel canale del rosso e quindi ridurre il SNR.
- Mettere il piatto contenente le celle miste il supporto del campione. Attendere almeno 10 minuti per garantire l'equilibrio di temperatura e per ridurre la deriva di messa a fuoco.
- Concentrarsi sulle cellule mediante trasmissione luce nel menu Locate .
- Ricerca di un paio di un 'rosso' e una cella 'verde' a contatto con l'altro. Per la cross-correlazione positiva o homo-dimero controllo luminosità (Vedi paragrafo 2), cercare una cella isolata che emette fluorescenza in entrambi i canali o il segnale di rispettivi homo-dimero al PM.
Nota: (Critico) Riduci a icona campione esposizione durante la ricerca di cellule evitare pre-sbiancamento, che può ridurre la cross-correlazione26. Pertanto, esegue la scansione alla velocità di scansione più veloce e poteri di laser a basso. Per evitare la saturazione del rivelatore, formazione immagine fortemente che esprimono le cellule, ricerca in modalità di integrazione. Tuttavia, per ridurre al minimo l'esposizione, scansione a potenze inferiori laser è possibile in modalità photon counting . - Selezionare una perpendicolare di percorso di scansione a contatto cellula-cellula (o a PM di una singola cella per il controllo di luminosità di cross-correlazione o homo-dimero positivo) utilizzando il pulsante Ritaglia come rappresentato nelle figure 1B e 2A.
Nota: Alcuni più vecchi microscopi non consentono le direzioni arbitrarie scansione. In questo caso, contatti cellula-cellula con un orientamento perpendicolare alla direzione di scansione devono essere posizionato. -
Zoom per raggiungere una dimensione di pixel di 50-200 nm e selezionare riga in Modalità di scansione. Impostare dimensioni fotogramma su 128 × 1 pixel.
Nota: Dimensione dei pixel tipico è di 160 nm, corrispondente ad una lunghezza di scansione di circa 20 µm. - Impostare velocità di scansione per il valore massimo consentito, ad esempio, 472,73 µs per ogni riga.
Nota: Per un regime alternativo di eccitazione, questo corrisponde al tempo di scansione 954,45 µs, cioè ~ 1000 scansioni/s sulla configurazione utilizzata. La velocità di scansione può essere regolata secondo il coefficiente di diffusione della proteina di interesse. Per le proteine di membrana ancorate, tempi di diffusione tipici sono intorno 10-20 ms il tempo di scansione dovrebbe essere almeno dieci volte più piccolo rispetto ai tempi di diffusione. Velocità di scansione più basse può indurre photobleaching più forte e richiedono potenze inferiori di illuminazione. In alternativa, si può imporre una pausa, per esempio, 5 ms, tra ogni scansione per molto lentamente diffondente complessi utilizzando l'intervallo nel sottomenu Time Series . - Scegliere le potenze laser appropriato, ad esempio, ~ 1-2 µW per 488 nm e ~ 5-10 µW per eccitazione di 561 nm.
Nota: Potenze laser superiori migliorano SNR, ma aumentano photobleaching. Di conseguenza, laser poteri dovrebbero essere scelti tali che photobleaching è inferiore al 50% del tasso di conteggio iniziale. - Impostare cicli a 100.000-500, 000.
Nota: Il numero di scansioni, vale a dire, durata della misurazione, può variare: tempi più lunghi di misura miglioreranno SNR e potrebbero essere più appropriati per diffondersi lentamente molecole, tuttavia, il movimento delle cellule e photobleaching limitare la misura massima tempo. I dati qui presentati sono stati acquisiti ordinariamente per ~ 3-6 min, vale a dire, 200.000-400.000 linea scansioni. - Impostare modalità Photon counting rivelatori. Premere Avvia esperimento per avviare l'acquisizione. Ripetere i passaggi da 4.5-4.11 per misurare un'altra cella.
Nota: Si consiglia di misurare 10-15 cellule per esempio ai livelli di espressione diversa. (Critico) Evitare la saturazione del rivelatore a livelli di alta espressione. Il tasso di conteggio massimo non dovrebbe superare ~ 1 MHz. - Se luminosità analisi sono effettuata per determinare stati oligomerici, eseguire misurazioni di calibrazione di homo-dimero luminosità secondo modificati passaggi 4.1-4.11: misurare ogni proteina fluorescente homo-dimero separatamente (in cellule isolate, preparati utilizzando protocollo sezione 2) ed esegue misure solo in un canale spettrale.
5. scansione spettroscopia di correlazione di fluorescenza: Analisi dei dati
Nota: Il seguente protocollo segue un'implementazione della procedura analisi descritta in dettaglio nel precedente articoli12,15. Il codice del software è disponibile su richiesta agli autori.
- Esportare i file di dati non elaborati (ad esempio, GFS185051) in un'immagine TIFF RGB in formato di dati grezzi. Questo file conterrà un kymograph con il verde e i dati del canale rosso, nel canale denominato G e R dell'immagine, rispettivamente.
- Importare il file TIFF con il software di analisi appropriato e procedere per eseguire l'analisi.
Nota: La seguente procedura (passaggi 5,3-5,7) vengono applicata separatamente per ogni canale: - Allineare le linee eseguendo una media di tempo segment-wise o in movimento con blocchi di linee di 500-1000. Determinare la posizione della membrana, cioè, la posizione del pixel con il tasso di conteggio massimo, in ogni blocco. Spostare tutti i blocchi sulla stessa posizione laterale. Questa procedura consente di correggere per spostamento laterale del contatto cellula-cellula, per esempio, a causa di movimento cellulare.
- Riassumere tutte le linee allineate lungo l'asse del tempo e misura il profilo di intensità media utilizzando una funzione gaussiana. In presenza di significativo sfondo intracellulare, utilizzare una gaussiana plus una funzione sigmoidale. Definire i pixel corrispondenti alla membrana come tutti i pixel all'interno di ±2.5σ della membrana posizione e riassumere l'intensità di questi pixel in ogni riga, ottenendo un valore di segnale di fluorescenza singola per ogni punto di tempo (cioè, per ogni riga di scansione).
- Se necessario (ad es., sfondo > 10% del segnale di membrana), applicare una correzione del fondo sottraendo l'intensità di pixel medi nel citoplasma moltiplicato per 2.5σ (in unità di pixel) rispetto alla fluorescenza di membrana, in blocchi di 1000 righe. Evitare di vescicole intracellulari brillante quando si seleziona pixel dello sfondo.
- Se si osserva il photobleaching, applicare una correzione di imbianchimento. Di conseguenza, adatta le serie temporali di fluorescenza della membrana con una funzione di doppio-esponenziale e applicare la correzione appropriata formula, equazione 116.
Nota: In alternativa, Fourier spettro basato su schemi di correzione possono essere applicata27. (Critico) Se photobleaching è presente ma non corretto per, il CFs può essere gravemente distorta e le stime dei parametri possono essere influenzate fortemente (per esempio, vedere la Figura 5E). - Calcolare la ACFs e CCFs secondo equazioni 2 e 3 utilizzando, ad esempio, un algoritmo di multiplo-tau28. Per migliorare l'affidabilità dell'analisi ed evitare artefatti, eseguire i calcoli per 10-20 segmenti uguali della misurazione totale. Ispezionare la serie temporale di fluorescenza e CFs in ogni segmento e rimuovere segmenti chiaramente distorti (Vedi esempi in Figura 4A- 4D). Media di tutti i segmenti non distorta.
Nota: Questa procedura può essere automatizzata per evitare una distorsione soggettiva ai dati29. Per misurazioni molto instabile può essere utile avere molti brevi segmenti. Tuttavia, la lunghezza di un segmento dovrebbe essere ancora almeno tre ordini di grandezza sopra il tempo di diffusione per evitare statistica sottocampionamento di errori29,30,17. - Montare un modello bidimensionale diffusione, equazione 4, al CFs ottenuti. Pertanto, correggere il fattore di struttura al valore ottenuto nella misurazione di taratura (protocollo sezione 3). La precisione della misura può essere migliorata mediante l'esecuzione di una misura ponderata utilizzando i pesi statistici di ogni punto di dati ottenuti dall'algoritmo più di tau.
- Calcolare il coefficiente di diffusione utilizzando la vita calibrata secondo l'equazione 7.
- Calcolare la luminosità molecolare dividendo l'intensità di fluorescenza media in ogni canale per il corrispondente numero di particelle, equazione 8. Normalizzare il valore di luminosità determinata in ciascun canale di luminosità media del corrispondente riferimento monomerico per ottenere lo stato oligomerico, tenendo conto non fluorescente FPs23. A questo scopo, determinare i valori di luminosità media homo-dimero dall'analisi di un solo colore per calcolare la frazione di non fluorescente FPs23.
- Calcolare la relativa correlazione secondo equazione 9.
6. Cross-correlazione numero e luminosità: calibrazione del rivelatore
Nota: Il seguente protocollo fornisce una linea guida generale per quanto riguarda come calibrare il sistema di rilevamento. Questa procedura è obbligatoria per sistemi di rivelazione analogica, ma non è strettamente necessaria quando vengono utilizzati rivelatori di conteggio di fotoni vero.
- A secco di soluzioni appropriate tintura solubile in acqua (Vedi Tabella materiali per esempi) su un piatto fondo di vetro 35mm. Impostare il percorso ottico di conseguenza, cioè, 488 o 561 nm eccitazione e rilevazione in 499-552 nm o 570-695 nm, rispettivamente.
Nota: In alternativa, una superficie riflettente in metallo utilizzabile invece di soluzione verniciante essiccato inserendo il pezzo di metallo direttamente sopra l'obiettivo. - Effettuare misurazioni di N & B monocolore in regioni con concentrazioni differenti della tintura o alle potenze laser diversi. Di conseguenza, usare lo Zoom per ottenere una dimensione di pixel di 300 nm, velocità di scansione per impostare il tempo di sosta appropriato pixel, ad esempio, 25 µs e cicli su 100-200 fotogrammi.
- Impostare rivelatori a conteggio di fotoni (o modalità analogica se le misure sono eseguite con rilevamento analogico) e premere Avvia esperimento per avviare l'acquisizione. Eseguire la misurazione a zero il potere di eccitazione per determinare l'offset di intensità.
- Trama di varianza pixel in funzione dell'intensità di pixel per pixel misurati tutti ed eseguire una misura lineare di questi dati. Determinare S come il pendio del fit lineare. Calcolare il rumore di lettura dall'intercetta y, utilizzando S e l'offset di determinata intensità secondo equazione 14.
7. Cross-correlazione numero e luminosità: acquisizione
- Procedura 4.1-4.4 del protocollo di acquisizione sFCCS.
- Utilizzare Ritaglia per selezionare un fotogramma di 512 × 128 pixel intorno un contatto cellula-cellula (o PM isolato per controllo di luminosità di homo-dimero) e Zoom per raggiungere una dimensione di pixel di 50-100 nm.
- Utilizzare la velocità di scansione per impostare tempo di sosta appropriato pixel, ad esempio, 6,3 µs.
Nota: In N & B, il tempo di permanenza di pixel deve essere molto più piccolo rispetto al tempo di diffusione della proteina di interesse. Se viene scelto un regime alternativo di eccitazione, per esempio, commutazione tiene traccia di ogni linea, il tempo tra le due tracce dovrebbe essere più piccolo rispetto al tempo di diffusione della proteina di interesse. In caso contrario la cross-correlazione rilevabile è ridotto. - Impostare cicli di 100-200 fotogrammi.
Nota: Un numero di telaio superiore migliorerà il SNR, tuttavia, il movimento cellulare può limitare il tempo di misurazione totale. Il tempo di scansione per ogni frame deve essere molto più elevato rispetto al tempo di diffusione della proteina di interesse. In caso contrario la luminosità apparente è ridotto, cioè particelle sembrano essere immobile. Per i complessi molto lentamente diffondente, imporre una pausa, per esempio, 2 s, tra fotogrammi utilizzando l'intervallo nel sottomenu Time Series . - Impostare valori appropriati poteri laser (i valori tipici sono ~ 1-2 µW per 488 nm e ~ 5-10 µW per eccitazione di 561 nm).
Nota: Aumento della potenza laser porta a maggiore luminosità e SNR migliorata, ma anche rafforzata photobleaching. Poteri del laser dovrebbero essere sufficiente per raggiungere una luminosità rilevati almeno ~ 1 kHz/MOL, ma tenuto abbastanza basso per evitare più di 10-20% photobleaching. Per mEGFP/mEYFP o mCherry/mCardinal, a meno di 10% photobleaching sono ottenuti solitamente. - Impostare rivelatori a conteggio di fotoni (o modalità analogica se le misure sono eseguite con rilevamento analogico). Premere Avvia esperimento per avviare l'acquisizione.
- Valutare il tasso di conteggio del fotone. Se i tassi di conteggio in pixel contatto cellula-cellula superare 1 MHz, ridurre la potenza del laser o selezionare le celle con più bassi livelli di espressione. Ripetere i passaggi 7.2-7.7. per misurare la coppia successiva delle cellule. Si raccomanda di misurare 10-15 cellule per esperimento a livelli di espressione diversa.
- Se analisi di luminosità viene eseguita per quantificare oligomerizzazione, eseguire misurazioni di calibrazione di homo-dimero luminosità secondo modificati passaggi 7.1-7.7: misurare ogni proteina fluorescente homo-dimero separatamente (in cellule isolate, preparati utilizzando protocollo sezione 2) ed esegue misure solo in un canale spettrale.
8. Cross-correlazione numero e luminosità: analisi dei dati
Nota: Il seguente protocollo segue un analisi precedentemente descritta procedura12,31. Il codice del software è disponibile da parte degli autori su richiesta.
- Importare i dati grezzi (ad es., GFS185051 file possono essere importati utilizzando il pacchetto di32 Bioformats). Media di tutti i fotogrammi e selezionare una regione di interesse (ROI) intorno al contatto cellula-cellula.
- Eseguire un immagine allineamento algoritmo33, ad es., massimizzando la correlazione spaziale tra ROIs in frame successivi per arbitrarie traduzioni laterale, calcolato in media su entrambi i canali. Questa procedura verrà corretto per movimento laterale delle cellule.
- Applicare un filtro di boxcar22 per ridurre le fluttuazioni longeve estranee, provenienti da, per esempio, il movimento cellulare residua o al candeggio di sfondo. In alternativa, un metodo detrending può essere applicato per risolvere per photobleaching34.
Nota: Se nessun analisi segment-wise o detrending viene applicato, la luminosità apparente può essere largamente sopravvalutata.- Definire i segmenti scorrevole di, per esempio, 8 a 15 fotogrammi (ad es., da 1 a 8, 2 a 9 e così via) e calcolare i valori di luminosità di canale e cross-correlazione secondo equazioni 10, 11 e 15 immagine in ogni segmento. Se i rivelatori non sono vero fotone conteggio rivelatori, prendere i parametri calibrati rivelatore in considerazione quando si calcola la luminosità, cioè, utilizzare equazioni 12 e 13.
Nota: Il calcolo dei valori di luminosità in segmenti di 8 a 15 fotogrammi conduce ad una sottovalutazione di 10-20% della luminosità assoluta e una sopravvalutazione di 10-20% dei numeri di particelle. Tuttavia, i rapporti di luminosità (per esempio, dimero di luminosità di monomero) non sono interessati, purché la lunghezza del segmento viene mantenuta costante durante l'analisi (dati non mostrati). L'errore statistico per una lunghezza di segmento specifico può essere determinato tramite simulazioni e così corretto per. - Media dei valori ottenuti luminosità pixel-wise sopra tutti i segmenti. In questo passaggio, uno può rimuovere il 5% più alto e più basso dei valori di luminosità di segmento dalla media o escludere segmenti che mostrano una chiara distorsione nell'intensità, a causa, ad esempio, una vescicola intracellulare o aggregato transitoriamente presente in questi pixel.
- Definire i segmenti scorrevole di, per esempio, 8 a 15 fotogrammi (ad es., da 1 a 8, 2 a 9 e così via) e calcolare i valori di luminosità di canale e cross-correlazione secondo equazioni 10, 11 e 15 immagine in ogni segmento. Se i rivelatori non sono vero fotone conteggio rivelatori, prendere i parametri calibrati rivelatore in considerazione quando si calcola la luminosità, cioè, utilizzare equazioni 12 e 13.
- Tracciare i valori di luminosità del pixel in funzione dell'intensità di pixel e selezionare la popolazione di pixel corrispondente al contatto cellula-cellula. Pixel di sfondo avrà valori di intensità molto bassa. A questo punto, ri-valutare il tasso di conteggio massimo. Escludere i pixel con tariffe totali sopra 1MHz per evitare effetti di pile-up.
- Creare istogrammi di luminosità per canale e cross-correlazione della cella-cella selezionata contatto pixel e ottenere i valori di luminosità media di ROI. Normalizzare il valore di luminosità del canale medio di luminosità media del corrispondente riferimento monomerico per ottenere lo stato oligomerico, tenendo conto non fluorescente FPs23. Di conseguenza, determinare i valori di luminosità media homo-dimero dall'analisi di un solo colore per calcolare la frazione di non fluorescente FPs23.
- Per l'illustrazione, tracciare mappe luminosità canale e cross-correlazione.
Risultati
Un primo test per il dosaggio di interazione proteina-proteina, cioè, miscelazione delle cellule che esprimono le proteine fluorescenti spettralmente distinte seguite da sFCCS/ccN & misure B (Figura 1), deve essere eseguita sulle proteine che non sono tenute a interagire al contatto cellula-cellula (cioè, un controllo negativo). Di conseguenza, le cellule HEK 293T che esprimono miristoilate-palmitoilata-mEYFP (myr-palm-mEYFP) o - mCardinal...
Discussione
La procedura sperimentale descritta qui permette l'indagine su proteine trans interazioni a contatti cellula-cellula, che impiegano tecniche di spettroscopia di fluorescenza fluttuazione, vale a dire sFCCS e ccN & B. Questi metodi implicano un'analisi statistica delle fluttuazioni di fluorescenza emessa da FPs spettralmente separati due fusi per la proteina di interesse a un contatto di due cellule vicine, ognuno esprimendo la una o l'altra proteina di fusione. La presenza di complessi trans è quantifi...
Divulgazioni
Gli autori non hanno nulla a rivelare.
Riconoscimenti
Questo lavoro è stato parzialmente supportato dalla Deutsche Forschungsgemeinschaft (DFG) concedere 254850309. Gli autori ringraziano Madlen Luckner per lettura critica del manoscritto.
Materiali
| Name | Company | Catalog Number | Comments |
| DMEM growth medium | PAN-Biotech | P04-01548 | |
| DPBS w/o: Ca2+ and Mg2+ | PAN-Biotech | P04-36500 | |
| DPBS w: Ca2+ and Mg2+ | PAN-Biotech | P04-35500 | |
| Trypsin EDTA | PAN-Biotech | P10-023100 | |
| TurboFect Transfection Reagent | Thermo Fisher Scientific | R0531 | |
| HEK 293T cells | DSMZ | ACC 635 | |
| Alexa Fluor 488 NHS Ester | Thermo Fisher Scientific | A20000 | |
| Rhodamine B | Sigma-Alderich | 83689-1G | |
| Plasmid DNA | Addgene | NA | See reference 12 (Dunsing et. al., MBoC 2017),for a detailed description of all plasmids |
| 6-well plate | Starlab | CC7672-7506 | |
| 35-mm glass bottom dishes | CellVis | D35-14-1.5-N | |
| Zeiss LSM780 confocal | Carl Zeiss | NA | |
| MATLAB software package | MathWorks | 2015b | |
| Neubauer cell counting chamber | Marienfeld | 640110 |
Riferimenti
- Alberts, B., Johnson, A., Lewis, J., Raff, M., Roberts, K., Walter, P. . Molecular biology of the cell. , (2002).
- Tepass, U., Truong, K., Godt, D., Ikura, M., Peifer, M. Cadherins in embryonic and neural morphogenesis. Nature Reviews Molecular Cell Biology. 1 (2), 91-100 (2000).
- Harris, T. J. C., Tepass, U. Adherens junctions: from molecules to morphogenesis. Nature Reviews Molecular Cell Biology. 11 (7), 502-514 (2010).
- Hernández, J. M., Podbilewicz, B. The hallmarks of cell-cell fusion. Development. 144 (24), 4481-4495 (2017).
- Huppa, J. B., Davis, M. M. T-cell-antigen recognition and the immunological synapse. Nature Reviews Immunology. 3 (12), 973-983 (2003).
- Kaden, D., Voigt, P., Munter, L. -. M., Bobowski, K. D., Schaefer, M., Multhaup, G. Subcellular localization and dimerization of APLP1 are strikingly different from APP and APLP2. Journal of cell science. 122, 368-377 (2009).
- Yap, A. S., Michael, M., Parton, R. G. Seeing and believing: recent advances in imaging cell-cell interactions. F1000Research. 4, 273 (2015).
- Kashef, J., Franz, C. M. Quantitative methods for analyzing cell-cell adhesion in development. Developmental Biology. 401 (1), 165-174 (2015).
- Soba, P., et al. Homo- and heterodimerization of APP family members promotes intercellular adhesion. The EMBO Journal. 24 (20), 3624-3634 (2005).
- Kim, S. A., Tai, C. -. Y., Mok, L. -. P., Mosser, E. A., Schuman, E. M. Calcium-dependent dynamics of cadherin interactions at cell-cell junctions. Proceedings of the National Academy of Sciences of the United States of America. 108 (24), 9857-9862 (2011).
- Feinberg, E. H., et al. GFP Reconstitution Across Synaptic Partners (GRASP) Defines Cell Contacts and Synapses in Living Nervous Systems. Neuron. 57 (3), 353-363 (2008).
- Dunsing, V., Mayer, M., Liebsch, F., Multhaup, G., Chiantia, S. Direct evidence of amyloid precursor-like protein 1 trans interactions in cell-cell adhesion platforms investigated via fluorescence fluctuation spectroscopy. Molecular biology of the cell. 28 (25), 3609-3620 (2017).
- Schneider, F., et al. Diffusion of lipids and GPI-anchored proteins in actin-free plasma membrane vesicles measured by STED-FCS. Molecular Biology of the Cell. 28 (11), 1507-1518 (2017).
- Bacia, K., Kim, S. A., Schwille, P. Fluorescence cross-correlation spectroscopy in living cells. Nature methods. 3 (2), 83-89 (2006).
- Ries, J., Schwille, P. Studying Slow Membrane Dynamics with Continuous Wave Scanning Fluorescence Correlation Spectroscopy. Biophysical Journal. 91 (5), 1915-1924 (2006).
- Ries, J., Chiantia, S., Schwille, P. Accurate Determination of Membrane Dynamics with Line-Scan FCS. Biophysical Journal. 96 (5), 1999-2008 (2009).
- Chiantia, S., Ries, J., Schwille, P. Fluorescence correlation spectroscopy in membrane structure elucidation. Biochimica et Biophysica Acta (BBA) - Biomembranes. 1788 (1), 225-233 (2009).
- Digman, M. A., Dalal, R., Horwitz, A. F., Gratton, E. Mapping the number of molecules and brightness in the laser scanning microscope. Biophysical journal. 94 (6), 2320-2332 (2008).
- Dalal, R. B., Digman, M. A., Horwitz, A. F., Vetri, V., Gratton, E. Determination of particle number and brightness using a laser scanning confocal microscope operating in the analog mode. Microscopy research and technique. 71 (1), 69-81 (2008).
- Unruh, J. R., Gratton, E. Analysis of Molecular Concentration and Brightness from Fluorescence Fluctuation Data with an Electron Multiplied CCD Camera. Biophysical Journal. 95 (11), 5385-5398 (2008).
- Digman, M. A., Wiseman, P. W., Choi, C., Horwitz, A. R., Gratton, E. Stoichiometry of molecular complexes at adhesions in living cells. Proceedings of the National Academy of Sciences of the United States of America. 106 (7), 2170-2175 (2009).
- Hellriegel, C., Caiolfa, V. R., Corti, V., Sidenius, N., Zamai, M. Number and brightness image analysis reveals ATF-induced dimerization kinetics of uPAR in the cell membrane. The FASEB journal official publication of the Federation of American Societies for Experimental Biology. 25 (9), 2883-2897 (2011).
- Dunsing, V., Luckner, M., Zühlke, B., Petazzi, R. A., Herrmann, A., Chiantia, S. Optimal fluorescent protein tags for quantifying protein oligomerization in living cells. Scientific Reports. 8 (1), 10634 (2018).
- Chen, Y., Johnson, J., Macdonald, P., Wu, B., Mueller, J. D. Observing Protein Interactions and Their Stoichiometry in Living Cells by Brightness Analysis of Fluorescence Fluctuation Experiments. Methods in enzymology. 472, 345-363 (2010).
- . Absolute Diffusion Coefficients: Compilation of Reference Data for FCS Calibration Available from: https://www.picoquant.com/images/uploads/page/files/7353/appnote_diffusioncoeffients.pdf (2010)
- Foo, Y. H., Naredi-Rainer, N., Lamb, D. C., Ahmed, S., Wohland, T. Factors affecting the quantification of biomolecular interactions by fluorescence cross-correlation spectroscopy. Biophysical journal. 102 (5), 1174-1183 (2012).
- Baum, M., Erdel, F., Wachsmuth, M., Rippe, K. Retrieving the intracellular topology from multi-scale protein mobility mapping in living cells. Nature Communications. 5, 4494 (2014).
- Wohland, T., Rigler, R., Vogel, H. The standard deviation in fluorescence correlation spectroscopy. Biophysical journal. 80 (6), 2987-2999 (2001).
- Ries, J., et al. Automated suppression of sample-related artifacts in Fluorescence Correlation Spectroscopy. Optics Express. 18 (11), 11073 (2010).
- Ries, J., Schwille, P. New concepts for fluorescence correlation spectroscopy on membranes. Physical Chemistry Chemical Physics. 10 (24), 3487 (2008).
- Mayer, M. C., et al. Amyloid precursor-like protein 1 (APLP1) exhibits stronger zinc-dependent neuronal adhesion than amyloid precursor protein and APLP2. Journal of Neurochemistry. 137 (2), 266-276 (2016).
- Linkert, M., et al. Metadata matters: access to image data in the real world. The Journal of Cell Biology. 189 (5), 777-782 (2010).
- Trullo, A., Corti, V., Arza, E., Caiolfa, V. R., Zamai, M. Application limits and data correction in number of molecules and brightness analysis. Microscopy Research and Technique. 76 (11), 1135-1146 (2013).
- Nolan, R., et al. nandb-number and brightness in R with a novel automatic detrending algorithm. Bioinformatics. 33 (21), 3508-3510 (2017).
- Hammond, G. R. V., Sim, Y., Lagnado, L., Irvine, R. F. Reversible binding and rapid diffusion of proteins in complex with inositol lipids serves to coordinate free movement with spatial information. The Journal of cell biology. 184 (2), 297-308 (2009).
- Hendrix, J., Dekens, T., Schrimpf, W., Lamb, D. C. Arbitrary-Region Raster Image Correlation Spectroscopy. Biophysical journal. 111 (8), 1785-1796 (2016).
- Hendrix, J., et al. Live-cell observation of cytosolic HIV-1 assembly onset reveals RNA-interacting Gag oligomers. The Journal of cell biology. 210 (4), 629-646 (2015).
- Hendrix, J., Schrimpf, W., Höller, M., Lamb, D. C. Pulsed Interleaved Excitation Fluctuation Imaging. Biophysical Journal. 105 (4), 848-861 (2013).
- Honigmann, A., et al. Scanning STED-FCS reveals spatiotemporal heterogeneity of lipid interaction in the plasma membrane of living cells. Nature Communications. 5 (1), 5412 (2014).
- Chojnacki, J., et al. Envelope glycoprotein mobility on HIV-1 particles depends on the virus maturation state. Nature Communications. 8 (1), 545 (2017).
- Godin, A. G., et al. Revealing protein oligomerization and densities in situ using spatial intensity distribution analysis. Proceedings of the National Academy of Sciences of the United States of America. 108 (17), 7010-7015 (2011).
- Müller, J. D., Chen, Y., Gratton, E. Resolving Heterogeneity on the Single Molecular Level with the Photon-Counting Histogram. Biophysical Journal. 78 (1), 474-486 (2000).
- Kim, S. A., Heinze, K. G., Bacia, K., Waxham, M. N., Schwille, P. Two-Photon Cross-Correlation Analysis of Intracellular Reactions with Variable Stoichiometry. Biophysical Journal. 88 (6), 4319-4336 (2005).
- Jenkins, E., et al. Reconstitution of immune cell interactions in free-standing membranes. Journal of cell science. 132 (4), (2018).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon