É necessária uma assinatura da JoVE para visualizar este conteúdo. Faça login ou comece sua avaliação gratuita.
Method Article
Um ensaio de espectroscopia de flutuação de fluorescência das interações da proteína-proteína em contatos do celular
Neste Artigo
Resumo
Este protocolo descreve uma abordagem de baseados em espectroscopia de flutuação de fluorescência para investigar interações entre proteínas mediando as interações célula-célula, ou seja, as proteínas localizadas em cruzamentos de célula, diretamente em células vivas. Nós fornecemos orientações detalhadas na calibração de instrumentos, aquisição de dados e análise, incluindo correcções de fontes possível artefato.
Resumo
Uma variedade de processos biológicos envolve interações célula-célula, normalmente mediadas por proteínas que interagem na interface entre células vizinhas. De interesse, apenas alguns ensaios são capazes de sondagem especificamente tais interações diretamente em células vivas. Aqui, apresentamos um ensaio para medir a ligação de proteínas expressado nas superfícies das células vizinhas, em contatos do celular. Este ensaio consiste em duas etapas: mistura de celulas que expressam as proteínas de interesse fundido a diferentes proteínas fluorescentes, seguido por medições de espectroscopia de flutuação de fluorescência em contatos de celular usando um laser confocal microscópio. Podemos demonstrar a viabilidade deste teste em um contexto biologicamente relevante medindo as interações da proteína amiloide precursora, como 1 (APLP1) através de junções célula-célula. Nós fornecemos protocolos detalhados sobre a aquisição de dados usando técnicas baseadas em fluorescência (varredura de espectroscopia de correlação cruzada de fluorescência, número de correlação cruzada e análise de brilho) e as calibrações de instrumentos necessários. Além disso, discutimos os passos críticos na análise dos dados e como identificar e corrigir as variações de sinal externo, espúrios, tais como as causadas por movimento fotobranqueamento ou célula.
Em geral, o ensaio apresentado é aplicável a qualquer homo - ou interação da proteína-proteína heterotypic em contatos de celular, entre as células dos tipos iguais ou diferentes e pode ser implementado em um comercial laser confocal microscópio. Um requisito importante é a estabilidade do sistema, que deve ser suficiente para sondar difusiva dinâmica das proteínas de interesse ao longo de vários minutos.
Introdução
Muitos processos biológicos ocorrem aos sítios de interações célula-célula, por exemplo, célula-célula adesão1,2,3, celular fusão4 e reconhecimento celular5. Tais eventos são particularmente importantes durante o desenvolvimento de organismos multicelulares e para a comunicação célula-célula, por exemplo, durante as respostas imunes. Esses processos são normalmente mediados por proteínas que são localizadas na superfície, ou seja, na membrana plasmática (PM) de vizinhos células e passam por interações específicas no contato célula-célula que são precisamente regulamentados no espaço e no tempo. Em muitos casos, essas interações são homo direto - ou heterotypic-interações da proteína proteína trans , mas também podem envolver íons ou ligantes atuando como extracelular linkers1. Apesar de fundamental importância, há uma falta de ensaios de sondagem essas interações proteína-proteína específica diretamente no ambiente nativo de células vivas. Muitos métodos exigem também o rompimento da pilha (por exemplo, ensaios bioquímicos como co-imunoprecipitação6), fixação (por exemplo, algumas das técnicas de microscopia óptica de super-resolução e microscopia eletrônica de célula-célula entra em contato com7), ou são não-específicos, por exemplo, agregação /8,9ensaios de aderência. Para superar esse problema, técnicas de fluorescência foram implementadas com base na ressonância de fluorescência a energia transferência (FRET)10 ou fluorescência complementação11. No entanto, para atingir suficientemente pequenas distâncias entre fluorophores, esses métodos exigem etiquetas fluorescentes no lado extracelular das proteínas10, potencialmente interferir com interações trans .
Aqui, apresentamos um ensaio alternativo baseado em fluorescência para interações da proteína-proteína em contatos do celular. Esta abordagem combina abordagens de correlação cruzada de fluorescência (espectroscopia de correlação cruzada de fluorescência digitalização (sFCCS), número de correlação cruzada e brilho (ccN & B)) e a mistura de células expressando um construto de fusão da proteína de interesse, por exemplo, um receptor de adesão. Os receptores investigados nas duas células interagindo são rotulados com duas proteínas fluorescentes espectralmente separadas (FPs), do intracelular lateral (veja figura 1A).
Os métodos empregados são baseados na análise estatística das flutuações da fluorescência induzida pelo movimento difusiva de proteínas fluorescentes fusão através do volume focal de um laser confocal microscópio. Mais detalhadamente, o ensaio de sondas a difusão co das proteínas de interesse em ambos os PMs vizinhas em contatos do celular. Se as proteínas sofrem interações trans , estes complexos trans vão levar proteínas fluorescentes emitem em ambos os canais espectrais, causando a fluorescência correlacionados flutuações de ambos os emissores. Por outro lado, se nenhuma ligação ocorre, as flutuações números de proteínas para enfrentar a TPM será independentes, causando flutuações não correlacionadas. A aquisição pode ser realizada de duas maneiras: 1) sFCCS é baseado em uma varredura em forma de linha através do contato célula-célula e sondas efetivamente as interações em um ponto localizado na região de contato. Através de uma análise temporal das flutuações de fluorescência, sFCCS fornece também informação dinâmica, ou seja, os coeficientes de difusão de complexos de proteínas; 2) ccN & B baseia-se numa análise pixel-wise de uma sequência de imagens adquiridas nas regiões de contato célula-célula. Tem a capacidade de sondar e interações do mapa ao longo de toda a região (em um plano focal) entre em contato com, mas não fornece informações sobre a dinâmica. Ambos os métodos podem ser combinados com uma análise do brilho molecular, ou seja, o sinal de média fluorescência emitida na unidade de tempo por simples difusão complexos de proteínas e, assim, proporcionar estimativas a estequiometria de complexos de proteínas no contatos do celular.
Neste artigo, nós fornecemos protocolos detalhados para preparação de amostras, calibração de instrumentos, aquisição de dados e análise realizar o ensaio apresentado em um comercial laser confocal microscópio. As experiências podem ser executadas em qualquer instrumento equipado com contagem de fótons ou detectores analógicos e um objectivo com abertura numérica elevada. Mais discutimos os passos críticos do protocolo e fornecem esquemas de correção para vários processos, causando flutuações artefactual sinal, por exemplo, de detector de ruído, movimento fotobranqueamento ou célula. Originalmente desenvolvida para sonda interações entre as células aderentes, o ensaio pode ser modificado para células de suspensão, ou adaptados para sistemas de membrana modelo, por exemplo, vesículas de gigante unilamellar (GUVs) ou plasma gigante vesículas de membrana (GPMVs), permitindo que o quantificação de interações em ambientes diferentes lipídios ou, na falta de um citoesqueleto organizado12,13.
Espectroscopia de fluorescência correlação cruzada de digitalização é uma versão modificada da fluorescência espectroscopia de correlação cruzada14 e foi projetado especificamente para sondar a dinâmica difusiva lenta em lipídios membranas15. É baseado na aquisição de digitalizar linha perpendicular ao PM contendo as proteínas fluorescentes de interesse. Para sondar as interações de duas espécies de proteína rotulada de forma diferente, a aquisição é realizada em dois canais espectrais usando duas linhas de laser e duas janelas de deteção para fluorophores espectralmente separados. Devido a dinâmica de difusão lenta de proteínas no PM (D≤ ~ 1 µm2/s), uma cruz-conversa livre de medição pode ser realizada alternando-se o esquema de excitação de linha a linha15. A análise começa com: 1) é um algoritmo de alinhamento corrigindo para o movimento lateral da célula com base na block-wise uma média de ~ 1000 linhas, 2) determinação da posição com fluorescência máximo sinalou seja, a PM posição, em cada bloco e 3) deslocando de todos os blocos para uma comum origem12,15, separadamente em cada canal. Em seguida, uma seleção automática de pixels correspondentes ao PM é executada selecionando a região central de um ajuste Gaussian da soma de todas as linhas alinhadas (i.e., centro ± 2.5σ). Integração do sinal em cada linha produz a série de tempo de fluorescência do membrana f (t) em cada canal (g = green channel, r = canal vermelho). Nota que o tamanho do pixel deve ser pequeno o suficiente, por exemplo, < 200 nm, para reconstruir a forma do ponto de espalhar a função e encontrar o seu centro, correspondente à posição da PM. Na presença de fotobranqueamento substancial, a série de tempo de fluorescência em cada canal pode ser modelada com uma função dupla-exponencial e então corrigida com a seguinte fórmula:16
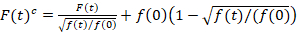 . (1)
. (1)
É importante notar que esta fórmula efetivamente corrige o amplitudes e tempos de difusão resultantes da análise de correlação de f (t)c, em comparação com as estimativas do parâmetro que seria obtidas da não corrigida f (t). Então, as funções de autoe correlação cruzada (ACFs / CCFs) da fluorescência, calculam-se os sinais:
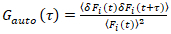 , (2).
, (2).
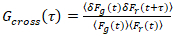 , (3).
, (3).
onde δFeu = Feu(t) - 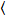 Feu(t)
Feu(t)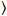 e eu = g, r.
e eu = g, r.
Um modelo de difusão bidimensional então é equipado para todas as funções de correlação (CFs):
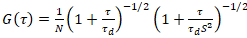 . (4)
. (4)
Aqui, N denota o número de proteínas fluorescentes no volume de observação e τd o tempo de difusão para cada canal. Este modelo leva em conta que no cenário experimental descrito, difusão de proteínas no PM ocorre no plano x-z, em contraste com a configuração comumente usada de correlação de fluorescência espectroscopia (FCS) experiências com membranas de sondagem difusão no plano x-y do confocal volume17. A cintura w0 e o fator de estrutura S, descrevendo o alongamento wz do volume focal em z, S = wz/w0, são obtidos a partir de uma medição de calibração de ponto FCS realizada com corantes espectralmente similares e mesmas configurações ópticas usando valores já está disponíveis para o coeficiente de difusão Dtintura:
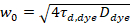 , (5).
, (5).
onde τd, a tintura é o tempo medido difusão média das moléculas do corante, obtidos a partir da montagem de um modelo para difusão tridimensional para os dados, tendo em transições de conta de uma fração T de todas as moléculas de N para um Estado de trio com uma constante de tempo ττ:
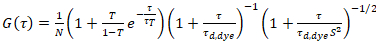 . (6)
. (6)
Finalmente, coeficientes de difusão (D), valores de brilho molecular (ε) e a correlação cruzada relativa dos dados de sFCCS (rel.cc.) são calculados como segue:
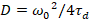 , (7).
, (7).
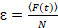 , (8).
, (8).
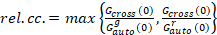 , (9).
, (9).
onde GCruz(0) é a amplitude da função de correlação cruzada e 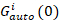 é a amplitude da função de autocorrelação no canal eu-th.
é a amplitude da função de autocorrelação no canal eu-th.
Esta definição da relativa correlação cruzada, ou seja, usando o máximo em vez de dizer na equação 9, leva em conta que o número máximo de complexos de duas espécies de proteínas presentes em diferentes concentrações é limitado pelo espécies presentes em um número menor.
Brilho e número de correlação cruzada é baseado em uma análise do momento da intensidade da fluorescência para cada pixel de uma pilha de imagem adquirida ao longo do tempo em uma posição fixa na amostra, geralmente constituídos por ~ 100-200 quadros, com espectral dois canais ( g = verde canal, r = canal vermelho). Da média temporal 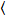 eu
eu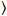 eu e a variância
eu e a variância 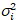 , o brilho molecular εi e número n são calculados em cada pixel e canal espectral (eu = g, r)18:
, o brilho molecular εi e número n são calculados em cada pixel e canal espectral (eu = g, r)18:
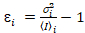 , (10).
, (10).
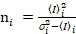 . (11)
. (11)
É importante notar que as equações determinadas se aplicam para o caso ideal de um verdadeiro detector de contagem de fótons. Para sistemas de detecção analógica, as equações seguintes aplicam-se a19,20:
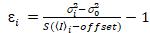 , (12).
, (12).
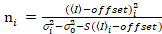 . (13)
. (13)
Aqui, S é o fator de conversão entre fótons detectados e as contagens de Digitas gravadas, 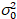 é o ruído de leitura e deslocamento refere-se ao deslocamento de intensidade do detector. Geralmente, essas quantidades devem ser calibradas, para qualquer tipo de detector, com base na variação do detector de medição em função da intensidade de iluminação constante19, por exemplo, uma superfície reflexiva de metal ou solução corante seco. O deslocamento pode ser determinada medindo-se a taxa de contagem para uma amostra sem luz de excitação. Realizando-se uma regressão linear da variância associada detector
é o ruído de leitura e deslocamento refere-se ao deslocamento de intensidade do detector. Geralmente, essas quantidades devem ser calibradas, para qualquer tipo de detector, com base na variação do detector de medição em função da intensidade de iluminação constante19, por exemplo, uma superfície reflexiva de metal ou solução corante seco. O deslocamento pode ser determinada medindo-se a taxa de contagem para uma amostra sem luz de excitação. Realizando-se uma regressão linear da variância associada detector 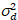 contra o enredo de intensidade (eu), S e
contra o enredo de intensidade (eu), S e 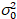 pode ser determinado19:
pode ser determinado19:
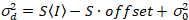 . (14)
. (14)
Finalmente, o brilho de correlação cruzada é calculado em cada pixel e é definido em geral como21
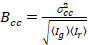 , (15).
, (15).
onde 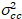 é a Cruz-variância
é a Cruz-variância 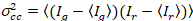 .
.
Para filtrar as flutuações long-lived, todos ccN & cálculos B são executados seguindo um vagão de filtragem, de forma independente para cada pixel de22. Brevemente, ni, εeu (eu = g, r) e Bcc são calculados em deslizamento segmentos de , por exemplo, 8-15 frames. Assim obtidos podem ser então a média dos valores para obter o último pixel valores número e brilho.
Análise de estequiometria
A fim de estimar a estequiometria de complexos de proteínas em contatos do celular, o brilho molecular pode ser analisado separadamente em cada canal espectral para o sFCCS ou ccN & B dados. No sFCCS, um valor de brilho é obtido por medição em cada canal. Em ccN & B, um histograma de brilho de todos os pixels correspondentes para o contato célula-célula é obtido e o valor médio (ou mediano) pode ser usado como brilho representativo para a medição. Realizando a mesma análise em uma referência monomérica, todos os valores de brilho podem ser normalizados para obter diretamente o estado oligoméricos médio dos complexos proteína detectada. Neste ponto, é importante corrigir a presença de FPs não-fluorescente, que pode resultar em uma subestimação do estado oligoméricas. Isto normalmente é realizado medindo-se o brilho de uma proteína dimérica homo referência do23,24 usando uma cor sFCS ou número e brilho (N & B).
Protocolo
1. amostra preparação: Celular ensaio de mistura
Nota: O seguinte protocolo descreve o procedimento de misturando de células aderentes. Pode ser modificado para células cultivadas em suspensão.
- Sementes de um número adequado de células em uma placa de 6, por exemplo, 800.000 células HEK 293T (contadas com uma contagem câmara de Neubauer), um dia antes do transfection. O número pode ser modificado dependendo do tempo entre a semeadura e transfecção e ajustado para outros tipos de células. Para executar um experimento básico (ou seja, as proteínas de interesse e controle negativo), prepare pelo menos 4 poços. Células de cultura a 37 ° C, 5% CO2 médio de Dulbecco e modificado águia médio (DMEM), suplementado com soro fetal bovino (10%) e L-glutamina (1%).
- Transfect células de acordo com as instruções do fabricante (ver a Tabela de materiais).
- Para executar um experimento básico, transfect, em separado wells, plasmídeos da proteína de interesse fundido a um 'verde' (por exemplo, monomérica avançada proteína verde fluorescente (mEGFP), ou proteína fluorescente amarela (mEYFP)) ou 'vermelho' (por exemplo, mCherry, ou mCardinal) proteína fluorescente.
Nota: No presente protocolo, focamos em APLP1-mEYFP e APLP1-mCardinal12e o correspondente controlo negativo, por exemplo, myristoylated-palmitoylated-mEYFP (myr-palm-mEYFP) e - mCardinal (myr-palm-mCardinal)12. Geralmente, 200 ng - 1 µ g de DNA de plasmídeo são suficientes. Eficiência de transfeccao alta aumenta a chance de encontrar células 'vermelhas' e 'verdes' em contato. Modifica a quantidade de reagente do plasmídeo e do transfection para otimizar a eficiência do transfection. Crítica: Confluência de célula deve ser ao redor 70% quando transfecting as células. Se as células são excesso confluente, irá diminuir a eficiência do transfection. Se as células não são suficientemente confluentes, transfection e mistura podem induzir stress e prevenir muitas células de fixação adequada após a mistura.
- Para executar um experimento básico, transfect, em separado wells, plasmídeos da proteína de interesse fundido a um 'verde' (por exemplo, monomérica avançada proteína verde fluorescente (mEGFP), ou proteína fluorescente amarela (mEYFP)) ou 'vermelho' (por exemplo, mCherry, ou mCardinal) proteína fluorescente.
- Execute célula mistura ± ~4 2 h depois do transfection.
- Retire o meio de crescimento e lave cada um bem delicadamente com 1 mL de PBS suplementado com Mg2 + e Ca2 +. Em seguida, remova a PBS. (Crítica) Largue o PBS na borda bem para evitar o desprendimento de células durante a lavagem.
- Adicione ~ 50 µ l do trypsin ácido etilenodiaminotetracético (EDTA) solução de ácido drop-wise a cada poço a fim de facilitar o desprendimento de células. Incubar a 37 ° C por 2 min. depois, lentamente, agitar a placa de 6 lateralmente para desprender as células.
Nota: Tempos de incubação prolongado podem ser necessários para alguns tipos de células. - Adicione 950 µ l do meio de crescimento a cada poço e ressuspender as células pipetando algumas vezes e para baixo, destacando, assim, todas as células do fundo bem. (Crítica) Certifique-se que as células são resuspended corretamente e desanexadas do outro, verificando visualmente a ausência de agregados de células grandes após ressuspensão. Caso contrário 'vermelhos' contatos 'verdes'-'vermelhos' ou 'verdes' - serão obtidos após a mistura.
- Transferir a solução de célula de um poço (proteína de interesse ou controlo negativo) para a correspondente, ou seja, 'vermelho' (por exemplo, APLP1-mCardinal transfectada) 'verde' (por exemplo, APLP1-mEYFP transfectada) células. Misture pipetando suavemente várias vezes para cima e para baixo. Então, semente as células mistas em pratos de fundo de vidro 35mm (1 mL da solução de mistura de células por prato), além de 1 mL de meio de crescimento e cultura semeado células para outro dia a 37 ° C, 5% de CO2.

Figura 1 . Fluxo de trabalho experimental e representação esquemática de varredura de espectroscopia de correlação cruzada de fluorescência e análise de correlação cruzada número e brilho em contatos celular. (A) esquema de preparação da amostra: duas populações de células transfectadas com a proteína de interesse (por exemplo, APLP1) fundida a duas proteínas fluorescentes espectralmente distintas (por exemplo, mEYFP e mCardinal) são misturadas depois do transfection. Contatos de células transfectadas diferente são selecionados nas experiências de microscopia. Para evitar interferência com domínios de ligação extracelular, a proteína fluorescente deve ser fundida ao terminus intracelular da proteína de interesse. (B) medições de digitalização FCCS (sFCCS) são realizada perpendicular ao contato célula-célula em dois canais espectrais (canal 1, verde e canal 2, vermelho). Linhas de varredura (representadas como kymographs) são alinhadas e pixels de membrana somados. Em seguida, ACFs e CCFs são calculados a partir de vestígios de intensidade Feu(t). ACFs são representados em vermelho e verde. CCF é representado em azul. (C) correlação cruzada N & B (ccN & B) aquisição resulta em uma pilha de imagem tridimensional (x-y-tempo). Um ROI é selecionado em torno do contato célula-célula. Em seguida, brilho de canal e correlação cruzada (ε1, ε2e Bcc) os valores são calculados em cada pixel de contato célula-célula. Em seguida, os resultados são visualizados como histogramas, reunindo todos os pixels selecionados. Clique aqui para ver uma versão maior desta figura.
2. amostra preparação: Controle positivo para experimentos de correlação cruzada e Homo-dímero construção para análise de brilho
- Sementes de 600.000 células HEK 293T, contadas com uma célula contando a câmara, em pratos de fundo de vidro 35mm um dia antes do transfection. Cultura de células a 37 ° C, 5% de CO2 em meio DMEM completo (consulte a etapa 1.1) para outro dia.
- Transfect células com ~ 250 ng de DNA de plasmídeo de acordo com as instruções do fabricante. Para o controlo positivo de correlação cruzada, use um plasmídeo codificação de um proteína fluorescente ancorado a membrana hétero-dímero, por exemplo, myr-palm-mCherry-mEGFP ou myr-palm-mCardinal-mEYFP12 correspondente para o FPs da proteína de interesse. Para a calibração de brilho, use plasmídeos codificação uma membrana-ancorado monômero FP e homo-dímero correspondente para o FPs fundido à proteína de interesse, por exemplo, myr-palma-mEYFP e myr-palm-mEYFP-mEYFP para calibrar a análise de brilho da APLP1-mEYFP12.
- Cultura de células, a 37 ° C, 5% de CO2 em meio DMEM completo (consulte a etapa 1.1) para outro dia.
3. confocal Laser microscopia de varredura: Configuração e calibração de Volume Focal
Nota: O seguinte protocolo escrito para experimentos realizados com mEGFP/mEYFP e mCherry/mCardinal do laser confocal microscópio utilizado neste estudo. A configuração óptica, as configurações de software (linhas de laser, espelhos dicroicos, filtros) e escolha de corantes de calibração podem ser modificados para outras configurações de FPs e microscópio.
- Ligue o microscópio e lasers pelo menos uma hora antes do experimento para garantir a estabilidade do laser e o equilíbrio da temperatura.
- Prepare 100-200 µ l de soluções tintura fluorescente hidrossolúvel apropriado (consulte a Tabela de materiais para obter exemplos) na água ou PBS para calibrar o volume focal, com concentrações na faixa de 10-50 nM.
- Coloque as soluções do corante em um vidro limpo 35-mm inferior prato #1.5, ou seja, com uma espessura de 0,16 0,19 mm.
Nota: Idealmente, use pratos com vidro de tampa de alto desempenho com uma tolerância de baixa espessura, por exemplo, 0,170 ± 0,005 mm, permitindo uma correção do anel de colarinho ideal (passo 3.6). É importante usar o mesmo tipo de prato como usado mais tarde para os experimentos seguintes. - Coloque o prato que contém a solução de tintura diretamente sobre o objetivo (de preferência, imersão de água, com at 1.2) para garantir foco na solução. Como alternativa, coloque o prato no porta-amostras e foco para a amostra (por exemplo, 10-20 µm acima do fundo do prato).
Nota: Não recomendamos utilizar objectivos de óleo devido o pobre sinal obtido quando o foco profundo em amostras aquosas. - Configurar o caminho da excitação e emissão, por exemplo, escolher o 488 nm do laser, espelho dicroico 561/488 nm, deteção janela 499-552 nm e um tamanho de furo de 1 unidade arejado (AU). Certifique-se que o tamanho do furo de pino é o mesmo que aquele que será usado em medições de correlação cruzada.
- Ajuste a posição do furo de pino (ajuste de pinhole) e o anel de coleira objetiva maximizar taxa de contagem. Para esse fim, rode o anel de coleira até taxa de contagem máxima é detectada.
Nota: A correção do anel colar representa a espessura específica do vidro tampa usado. Maximizar a taxa de contagem, i. e., coletando como muitos fótons por molécula quanto possível, é crucial para maximizar a relação sinal-ruído (SNR) das medições. - Realizar uma série de medições ponto FCS (por exemplo, 6 medições em locais diferentes, cada um composto por 15 repetições de 10 s, ou seja, 2,5 min tempo total, amostradas com tempo de interrupção de 1 µs ou menos) para a mesma potência do laser usado em correlação cruzada medições (tipicamente ~ 1%, ou seja, ~ 1-2 µW).
- Ajuste um modelo tridimensional de difusão incluindo uma contribuição triplet (equação 6) para os dados.
Nota: Normalmente, os tempos de difusão obtidos são cerca de 30 µs e o fator de estrutura é em torno de 4-8. - Calcular a cintura w0 do tempo de difusão média medido e publicado os valores para o coeficiente de difusão do corante usado em temperatura ambiente25 de acordo com a equação 5. Valores típicos são 200-250 nm.
- Repita a rotina de calibração (etapas 3.4-3,9) com uma tintura fluorescente diferente para um segundo canal de deteção, se necessário (por exemplo, 561-nm excitação e deteção entre 570 nm e 695 nm). Manter a posição de pinhole e tamanho como foi definido para o primeiro canal de deteção.
- Calcular o brilho molecular (equação 8) das medidas de calibração e armazenar os valores obtidos.
Nota: Valores típicos para a configuração usado são ~8-10 kHz/molécula (MOL) para 1,8 potência de excitação nm 488 µW. Mais baixo do que o habituais valores podem indicar a sujeira sobre o objectivo, desalinhamento de configuração ou uma saída reduzida do laser. Verifique e armazenar poderes de saída do laser no objetivo regularmente usando um medidor de energia. Para comparação de configurações diferentes, brilho molecular normalizado pelo poder da excitação do laser é o parâmetro mais importante para avaliar o desempenho do microscópio.
4. espectroscopia de correlação cruzada de fluorescência de digitalização: aquisição
Nota: O seguinte protocolo escrito para experimentos realizados com mEGFP/mEYFP ('verde') e mCherry/mCardinal ('vermelho') sobre o microscópio confocal digitalização laser utilizado neste estudo. A configuração óptica e os ajustes do software (linhas de laser, espelhos dicroicos, filtros) podem ser diferentes para outras configurações de FPs ou microscópio.
- Configurar o caminho óptico, por exemplo, 488 nm e 561 excitação nm e um espelho dicroico nm 488/561, pinhole em 1 AU para 488 excitação nm. Para evitar conversas cruzadas-espectral, selecione duas faixas separadas para excitar e detectar mEGFP/mEYFP (488 nm de excitação, canal verde) e mCherry / mCardinal (561 nm de excitação, canal vermelho) sequencialmente e selecione interruptor controla cada linha. Para a deteção, use filtros adequados para ambos os canais, por exemplo, 499-552 nm no canal verde e 695-570 nm no canal vermelho.
- Se excitação alternada não for possível, use as configurações de filtro apropriado para o canal vermelho para minimizar espectral-conversas cruzadas (ou seja, detectar a fluorescência de mCherry/mCardinal não abaixo de 600 nm). Isto pode reduzir a quantidade de fótons detectado no canal vermelho e assim reduzir o SNR.
- Coloque o prato que contém as células mistas no porta-amostra. Espere pelo menos 10 min para garantir equilíbrio de temperatura e reduzir o desvio de foco.
- Concentre-se nas células usando a transmissão de luz no menu Localizar .
- Busca por um par de um 'vermelho' e uma 'verde' célula em contato com os outros. Para o Cruz-correlação positiva ou homo-dímero controle de brilho (ver secção 2), uma busca por uma célula isolada emitindo fluorescência em ambos os canais ou o sinal de homo-dímero respectivos para a PM.
Nota: (Crítica) minimizar amostra exposição enquanto busca de células evitar pre-branqueamento, que pode reduzir a correlação cruzada de26. Portanto, digitalizar na mais rápida velocidade de digitalização e poderes do laser de baixa. Para evitar a saturação do detector enquanto imagem latente expressando fortemente as células, busca no modo de integração. No entanto, para minimizar a exposição, digitalização em baixas potências do laser é possível no modo de contagem de fótons . - Selecione uma perpendicular de caminho de varredura para contato célula-célula (ou PM de uma única célula para o controle de brilho de correlação cruzada ou homo-dímero positivo) usando o botão recortar conforme ilustrado nas Figuras 1B e 2A.
Nota: Alguns microscópios mais antigos não permitem digitalizar arbitrário direções. Neste caso, contatos do celular com uma orientação perpendicular à direção de varredura tem que ser localizado. -
Zoom para atingir um tamanho de pixel de 50-200 nm e selecione linha no Modo de varredura. Definir o tamanho do quadro para 128 × 1 pixels.
Nota: O tamanho do pixel típico é 160 nm, correspondente a um comprimento de varredura de cerca de 20 µm. - Defina velocidade de digitalização para o valor máximo permitido, por exemplo, 472,73 µs por linha.
Nota: Para um regime alternativo de excitação, isto corresponde a 954,45 µs tempo de verificação, ou seja, ~ 1000 varreduras/s na configuração do usado. A velocidade de varredura pode ser ajustada dependendo do coeficiente de difusão da proteína de interesse. Para proteínas de membrana-ancorado, tempos típicos de difusão são em torno de 10-20 ms. O tempo de verificação deve ser pelo menos dez vezes menor do que os tempos de difusão. Baixas velocidades de varredura podem induzir fotobranqueamento mais forte e exigem mais baixo poder de iluminação. Alternativamente, um pode impor uma pausa, por exemplo, 5 ms, entre cada verificação para complexos de difusão muito lentamente usando intervalo no submenu de Séries temporais . - Escolha o poderes do laser adequado, por exemplo, ~ 1-2 µW para 488 nm e ~ 5-10 µW para a excitação de nm 561.
Nota: Maiores potências do laser melhorar SNR, mas aumentam fotobranqueamento. Por conseguinte, poderes do laser devem ser escolhidas fotobranqueamento é inferior a 50% da taxa inicial de contagem. - Conjunto de ciclos para 100.000-500, 000.
Nota: O número de exames, ou seja, a duração da medição, podem variar: tempos de medição mais melhorarão SNR e podem ser mais adequados para difundir lentamente moléculas, no entanto, o movimento das células e fotobranqueamento limitar a medida máxima está na hora. Os dados apresentados aqui foram rotineiramente adquiridos para ~ 3-6 min, ou seja, a linha de 200.000-400.000 varreduras. - Conjunto de detectores para modo de contagem de fótons . Pressione Iniciar o experimento para iniciar a aquisição. Repita os passos 4,5-4.11 para medir outra célula.
Nota: Recomenda-se medir de 10-15 células por amostra em níveis diferentes de expressão. (Crítica) Evite a saturação do detector em níveis de alta expressão. A taxa de contagem máxima não deve exceder ~ 1 MHz. - Se a análise de brilho é executada para determinar os Estados oligoméricos, realizar medições de calibração de brilho de homo-dímero de acordo com passos modificados 4.1-4.11: medir cada proteína fluorescente homo-dímero separadamente (em células isoladas, preparada com protocolo, seção 2) e realizar medições apenas em um canal espectral.
5. espectroscopia de correlação cruzada de fluorescência digitalização: Análise de dados
Nota: O seguinte protocolo segue uma implementação do procedimento de análise descrito detalhadamente em anteriores artigos12,15. O código do software está disponível mediante solicitação aos autores.
- Exporte os arquivos de dados brutos (por exemplo, CZI) para uma imagem TIFF RGB no formato de dados brutos. Este arquivo irá conter um kymograph com o verde e dados de canal vermelho, no canal denominado G e R da imagem, respectivamente.
- Importar o arquivo TIFF com o software de análise adequada e proceder para realizar a análise.
Nota: Os seguintes passos (5.3-5,7) são aplicados separadamente para cada canal: - Alinhe as linhas, realizando uma média de tempo de segment-wise ou em movimento com blocos de linhas 500-1000. Determine a posição de membrana, ou seja, a posição do pixel com a taxa máxima de contagem, em cada bloco. Deslocar todos os blocos para a mesma posição lateral. Este procedimento corrige para deslocamento lateral do contato célula-célula, por exemplo, devido ao movimento da célula.
- Resumir todas as linhas alinhadas ao longo do eixo do tempo e se encaixa no perfil de intensidade média, usando uma função gaussiana. Na presença de fundo intracelular significativo, use um Gaussian mais uma função sigmoide. Defina os pixels correspondentes à membrana como todos os pixels dentro de ±2.5σ da posição de membrana e resumir a intensidade desses pixels em cada linha, obtendo um valor de sinal de fluorescência único para cada ponto de tempo (ou seja, para cada verificação de linha).
- Se necessário (por exemplo, plano de fundo > 10% do sinal da membrana), aplicar uma correção de fundo, subtraindo-se a intensidade do pixel média no citoplasma multiplicado pelo 2.5σ (em unidades de pixel) de fluorescência da membrana, em blocos de 1000 linhas. Evite vesículas intracelulares brilhantes quando a seleção de pixels do fundo.
- Se for observado fotobranqueamento, aplica uma correção de branqueamento. Portanto, cabe a série de tempo de fluorescência de membrana com uma função dupla-exponencial e aplicar a correção adequada fórmula, equação 116.
Nota: Como alternativa, Fourier espectro com base em esquemas de correção podem ser aplicada27. (Crítica) Se fotobranqueamento está presente mas não corrigido para, o CFs podem ser severamente distorcidos e as estimativas do parâmetro podem ser fortemente tendencioso (por exemplo, veja a Figura 5E). - Calcule o ACFs e CCFs de acordo com as equações 2 e 3 usando, por exemplo, um algoritmo de múltiplo-tau28. Para melhorar a confiabilidade da análise e evitar artefatos, executar os cálculos para 10-20 segmentos iguais da medição total. Inspecione as séries temporais de fluorescência e CFs em cada segmento e remover segmentos claramente distorcidos (ver exemplos na Figura 4A- 4D). Média de todos os segmentos sem distorção.
Nota: Este procedimento pode ser automatizado para evitar um viés subjetivo para os dados de29. Para medições muito instáveis, ter muitos segmentos curtos pode ser útil. No entanto, o comprimento de um segmento ainda deve estar a pelo menos três ordens de magnitude acima o tempo de difusão para evitar estatística undersampling erros29,30,17. - Ajuste um modelo de difusão bidimensional, equação 4, para o CFs obtidos. Portanto, corrigi o fator de estrutura para o valor obtido na medição da calibração (protocolo seção 3). A precisão do ajuste pode ser melhorada através da realização de um ajuste ponderado usando os pesos estatísticos de cada ponto de dados obtidos de algoritmo múltiplo de tau.
- Calcule o coeficiente de difusão usando a cintura calibrada de acordo com a equação 7.
- Calcule o brilho molecular, dividindo-se a intensidade de fluorescência média em cada canal pelo correspondente número de partículas, a equação 8. Normalize o valor determinado brilho em cada canal pelo brilho médio da referência monomérico correspondente para obter o estado oligoméricos, tendo em conta não-fluorescente FPs23. Para este objectivo, determine os valores de brilho médio homo-dímero da análise de uma cor para calcular a fração de não-fluorescente FPs23.
- Calcule a correlação cruzada relativa de acordo com a equação 9.
6. correlação cruzada número e brilho: calibração do Detector
Nota: O seguinte protocolo fornece uma orientação geral sobre como calibrar o sistema de detecção. Este procedimento é obrigatório para os sistemas de deteção analógico, mas não é estritamente necessário quando são utilizados detectores de contagem de fótons verdadeiro.
- Soluções de corante hidrossolúvel apropriado (consulte a Tabela de materiais para obter exemplos) em um prato fundo de vidro 35mm a seco. Definir o caminho óptico em conformidade, ou seja, 488 ou 561 nm excitação e deteção em 499-552 nm ou 695-570 nm, respectivamente.
Nota: Como alternativa, uma superfície reflexiva de metal pode ser usada em vez de soluções do corante seco colocando a peça de metal diretamente sobre o objetivo. - Realize medições de N & B de uma cor em regiões com concentrações diferentes de tintura ou poderes diferentes do laser. Portanto, use o Zoom para atingir um tamanho de pixel de 300 nm, velocidade de digitalização para definir o tempo de permanência de pixel apropriado, por exemplo, 25 µs e definir ciclos de 100-200 quadros.
- Detectores para contagem de fótons (ou modo analógico se as medições são realizadas com deteção analógica) e pressionar o Experimento iniciar para iniciar a aquisição. Realize a medição no zero energia de excitação para determinar o deslocamento de intensidade.
- Plotagem de variância pixel em função da intensidade de pixel para todos os pixels medidos e realizar um ajuste linear destes dados. Determinar S como a inclinação do ajuste linear. Calcule o ruído de leitura da intercepção de y, usando o S e o deslocamento de intensidade determinada de acordo com a equação 14.
7. correlação cruzada número e brilho: aquisição
- Siga os passos 4,1-4,4 do protocolo de aquisição de sFCCS.
- Use o Crop para selecionar um quadro de 512 × 128 pixels, ao redor de um contato célula-célula (ou PM isolado para controle de brilho de homo-dímero) e Zoom para atingir um tamanho de pixel de 50-100 nm.
- Use a velocidade de digitalização para definir o tempo de permanência de pixel apropriado, por exemplo, 6,3 µs.
Nota: Em N & B, o tempo de permanência do pixel deve ser muito menor do que o tempo de difusão da proteína de interesse. Se for escolhido um regime alternativo de excitação, por exemplo, alternar faixas cada linha, o tempo entre as duas faixas deve ser menor do que o tempo de difusão da proteína de interesse. Caso contrário, a correlação cruzada detectável é reduzida. - Definir ciclos de 100-200 quadros.
Nota: Um número maior de quadro irá melhorar o SNR, no entanto, o movimento celular pode limitar o tempo de medição total. O tempo de verificação por quadro deveria ser muito maior do que o tempo de difusão da proteína de interesse. Caso contrário, o brilho aparente é reduzido, ou seja, as partículas parecem estar imóvel. Para complexos de difusão muito lentamente, impor uma pausa, por exemplo, 2 s, entre quadros usando o intervalo no submenu de Séries temporais . - Conjunto de poderes do laser com valores apropriados (valores típicos são ~ 1-2 µW para 488 nm e ~ 5-10 µW para 561 excitação nm).
Nota: Maior potência do laser leva à maior brilho e SNR melhorada, mas também fotobranqueamento reforçado. Poderes do laser devem ser altas o suficiente para alcançar um brilho detectado pelo menos ~ 1 kHz/MOL, mas mantido baixo o suficiente para evitar mais do que 10-20% fotobranqueamento. Para fotobranqueamento mEGFP/mEYFP ou mCherry/mCardinal, a menos de 10% são normalmente obtidos. - Como detectores de fóton contando (ou modo analógico se as medições são realizadas com deteção analógica). Pressione Iniciar o experimento para iniciar a aquisição.
- Avalie a taxa de contagem de fótons. Se taxas de contagem em pixels contato célula-célula excederem 1 MHz, reduza a potência do laser ou selecione células com menores níveis de expressão. Repita as etapas de 7,2-7,7. para medir o próximo par de células. Recomenda-se medir de 10-15 células por experiência em níveis diferentes de expressão.
- Se a análise de brilho é realizada para quantificar oligomerização, realizar medições de calibração de brilho de homo-dímero de acordo com passos modificados 7.1-7.7: medir cada proteína fluorescente homo-dímero separadamente (em células isoladas, preparada com protocolo, seção 2) e realizar medições apenas em um canal espectral.
8. correlação cruzada número e brilho: análise de dados
Nota: O seguinte protocolo segue um procedimento de análise descritas anteriormente12,31. O código do software está disponível a partir dos autores mediante pedido.
- Importar os dados crus (por exemplo, arquivos de CZI podem ser importados usando o pacote de32 Bioformats). Média de todos os quadros e selecione uma região de interesse (ROI) em torno do contato célula-célula.
- Execute uma imagem alinhamento algoritmo33, por exemplo, maximizando a correlação espacial entre ROIs em quadros subsequentes para traduções laterais arbitrários, em média, ambos os canais. Este procedimento irá corrigir para o movimento lateral das células.
- Aplica um filtro de boxcar22 para reduzir flutuações long-lived estranhas, provenientes, por exemplo, movimento de célula residual ou fundo de clareamento. Alternativamente, pode ser aplicado um método detrending para corrigir para fotobranqueamento34.
Nota: Se nenhuma análise segment-wise ou detrending é aplicado, o brilho aparente pode ser superestimado em grande parte.- Definir segmentos deslizantes de, por exemplo, 8 a 15 quadros (por exemplo, quadros 1 a 8, 2 a 9 e assim por diante) e calcular os valores de brilho de canal e correlação cruzada de acordo com as equações 10, 11 e 15 pixel-wise em cada segmento. Se detectores não são verdade fóton contando detectores, levar os parâmetros de detector calibrado em consideração ao calcular o brilho, ou seja, usar equações 12 e 13, em vez disso.
Nota: Cálculo dos valores de brilho em segmentos de 8 a 15 quadros leva a uma subestimação de 10-20% do brilho absoluto e uma superestimação de 10-20% de números de partículas. Não obstante, rácios de brilho (por exemplo, dímero de brilho de monômero) não são afetados, enquanto o comprimento do segmento é mantido constante em toda a análise (dados não mostrados). O erro estatístico para um comprimento dado segmento pode ser determinado através de simulações e, portanto, corrigido para. - Os brilho obtidos os valores médios pixel-wise sobre todos os segmentos. Nesta etapa, pode-se remover a maior e menor de 5% dos valores de brilho do segmento a partir da média ou excluir segmentos que mostram uma clara distorção na intensidade, devido, por exemplo, uma vesícula intracelular ou agregado transitoriamente presente nestes pixéis.
- Definir segmentos deslizantes de, por exemplo, 8 a 15 quadros (por exemplo, quadros 1 a 8, 2 a 9 e assim por diante) e calcular os valores de brilho de canal e correlação cruzada de acordo com as equações 10, 11 e 15 pixel-wise em cada segmento. Se detectores não são verdade fóton contando detectores, levar os parâmetros de detector calibrado em consideração ao calcular o brilho, ou seja, usar equações 12 e 13, em vez disso.
- Plotar os valores de brilho do pixel em função da intensidade do pixel e selecionar a população de pixels que corresponde ao contato célula-célula. Pixels do fundo terão valores de intensidade muito baixa. Neste ponto, re-avalie a taxa de contagem máxima. Exclua pixels com taxas de contagem acima de 1 MHz para evitar efeitos de engavetamento.
- Criar canal e correlação cruzada histogramas de brilho de célula-célula selecionada contatos pixels e obter os valores de brilho ROI-média. Normalize o valor de brilho médio canal pelo brilho médio da referência monomérico correspondente para obter o estado oligoméricos, tendo em conta não-fluorescente FPs23. Portanto, determine os valores de brilho médio homo-dímero da análise de uma cor para calcular a fração de não-fluorescente FPs23.
- Para ilustração, traça mapas de brilho de canal e correlação cruzada.
Resultados
Um primeiro teste para o ensaio de interação da proteína-proteína, ou seja, mistura de celulas que expressam proteínas fluorescentes espectralmente distintas, seguidas por sFCCS/ccN & B as medições (Figura 1), deve ser realizada em proteínas que não são esperadas para interagir no contato célula-célula (ou seja, um controle negativo). Portanto, misturavam-se células HEK 293T expressando myristoylated-palmitoylated-mEYFP (myr-pal...
Discussão
O procedimento experimental descrito aqui permite a investigação da proteína-proteína trans interações em contatos de celular, empregando técnicas de espectroscopia de flutuação de fluorescência, ou seja, sFCCS e ccN & B. Esses métodos envolvem uma análise estatística das flutuações de fluorescência emitida por dois espectralmente separados FPs fundido para as somáticas de interesse em um contato de duas células vizinhas, cada uma expressando uma ou a outra proteína de fusão. A presença de c...
Divulgações
Os autores não têm nada para divulgar.
Agradecimentos
Este trabalho foi parcialmente financiado pela Deutsche Forschungsgemeinschaft (DFG) conceder 254850309. Os autores agradecer Madlen Luckner leitura crítica do manuscrito.
Materiais
| Name | Company | Catalog Number | Comments |
| DMEM growth medium | PAN-Biotech | P04-01548 | |
| DPBS w/o: Ca2+ and Mg2+ | PAN-Biotech | P04-36500 | |
| DPBS w: Ca2+ and Mg2+ | PAN-Biotech | P04-35500 | |
| Trypsin EDTA | PAN-Biotech | P10-023100 | |
| TurboFect Transfection Reagent | Thermo Fisher Scientific | R0531 | |
| HEK 293T cells | DSMZ | ACC 635 | |
| Alexa Fluor 488 NHS Ester | Thermo Fisher Scientific | A20000 | |
| Rhodamine B | Sigma-Alderich | 83689-1G | |
| Plasmid DNA | Addgene | NA | See reference 12 (Dunsing et. al., MBoC 2017),for a detailed description of all plasmids |
| 6-well plate | Starlab | CC7672-7506 | |
| 35-mm glass bottom dishes | CellVis | D35-14-1.5-N | |
| Zeiss LSM780 confocal | Carl Zeiss | NA | |
| MATLAB software package | MathWorks | 2015b | |
| Neubauer cell counting chamber | Marienfeld | 640110 |
Referências
- Alberts, B., Johnson, A., Lewis, J., Raff, M., Roberts, K., Walter, P. . Molecular biology of the cell. , (2002).
- Tepass, U., Truong, K., Godt, D., Ikura, M., Peifer, M. Cadherins in embryonic and neural morphogenesis. Nature Reviews Molecular Cell Biology. 1 (2), 91-100 (2000).
- Harris, T. J. C., Tepass, U. Adherens junctions: from molecules to morphogenesis. Nature Reviews Molecular Cell Biology. 11 (7), 502-514 (2010).
- Hernández, J. M., Podbilewicz, B. The hallmarks of cell-cell fusion. Development. 144 (24), 4481-4495 (2017).
- Huppa, J. B., Davis, M. M. T-cell-antigen recognition and the immunological synapse. Nature Reviews Immunology. 3 (12), 973-983 (2003).
- Kaden, D., Voigt, P., Munter, L. -. M., Bobowski, K. D., Schaefer, M., Multhaup, G. Subcellular localization and dimerization of APLP1 are strikingly different from APP and APLP2. Journal of cell science. 122, 368-377 (2009).
- Yap, A. S., Michael, M., Parton, R. G. Seeing and believing: recent advances in imaging cell-cell interactions. F1000Research. 4, 273 (2015).
- Kashef, J., Franz, C. M. Quantitative methods for analyzing cell-cell adhesion in development. Developmental Biology. 401 (1), 165-174 (2015).
- Soba, P., et al. Homo- and heterodimerization of APP family members promotes intercellular adhesion. The EMBO Journal. 24 (20), 3624-3634 (2005).
- Kim, S. A., Tai, C. -. Y., Mok, L. -. P., Mosser, E. A., Schuman, E. M. Calcium-dependent dynamics of cadherin interactions at cell-cell junctions. Proceedings of the National Academy of Sciences of the United States of America. 108 (24), 9857-9862 (2011).
- Feinberg, E. H., et al. GFP Reconstitution Across Synaptic Partners (GRASP) Defines Cell Contacts and Synapses in Living Nervous Systems. Neuron. 57 (3), 353-363 (2008).
- Dunsing, V., Mayer, M., Liebsch, F., Multhaup, G., Chiantia, S. Direct evidence of amyloid precursor-like protein 1 trans interactions in cell-cell adhesion platforms investigated via fluorescence fluctuation spectroscopy. Molecular biology of the cell. 28 (25), 3609-3620 (2017).
- Schneider, F., et al. Diffusion of lipids and GPI-anchored proteins in actin-free plasma membrane vesicles measured by STED-FCS. Molecular Biology of the Cell. 28 (11), 1507-1518 (2017).
- Bacia, K., Kim, S. A., Schwille, P. Fluorescence cross-correlation spectroscopy in living cells. Nature methods. 3 (2), 83-89 (2006).
- Ries, J., Schwille, P. Studying Slow Membrane Dynamics with Continuous Wave Scanning Fluorescence Correlation Spectroscopy. Biophysical Journal. 91 (5), 1915-1924 (2006).
- Ries, J., Chiantia, S., Schwille, P. Accurate Determination of Membrane Dynamics with Line-Scan FCS. Biophysical Journal. 96 (5), 1999-2008 (2009).
- Chiantia, S., Ries, J., Schwille, P. Fluorescence correlation spectroscopy in membrane structure elucidation. Biochimica et Biophysica Acta (BBA) - Biomembranes. 1788 (1), 225-233 (2009).
- Digman, M. A., Dalal, R., Horwitz, A. F., Gratton, E. Mapping the number of molecules and brightness in the laser scanning microscope. Biophysical journal. 94 (6), 2320-2332 (2008).
- Dalal, R. B., Digman, M. A., Horwitz, A. F., Vetri, V., Gratton, E. Determination of particle number and brightness using a laser scanning confocal microscope operating in the analog mode. Microscopy research and technique. 71 (1), 69-81 (2008).
- Unruh, J. R., Gratton, E. Analysis of Molecular Concentration and Brightness from Fluorescence Fluctuation Data with an Electron Multiplied CCD Camera. Biophysical Journal. 95 (11), 5385-5398 (2008).
- Digman, M. A., Wiseman, P. W., Choi, C., Horwitz, A. R., Gratton, E. Stoichiometry of molecular complexes at adhesions in living cells. Proceedings of the National Academy of Sciences of the United States of America. 106 (7), 2170-2175 (2009).
- Hellriegel, C., Caiolfa, V. R., Corti, V., Sidenius, N., Zamai, M. Number and brightness image analysis reveals ATF-induced dimerization kinetics of uPAR in the cell membrane. The FASEB journal official publication of the Federation of American Societies for Experimental Biology. 25 (9), 2883-2897 (2011).
- Dunsing, V., Luckner, M., Zühlke, B., Petazzi, R. A., Herrmann, A., Chiantia, S. Optimal fluorescent protein tags for quantifying protein oligomerization in living cells. Scientific Reports. 8 (1), 10634 (2018).
- Chen, Y., Johnson, J., Macdonald, P., Wu, B., Mueller, J. D. Observing Protein Interactions and Their Stoichiometry in Living Cells by Brightness Analysis of Fluorescence Fluctuation Experiments. Methods in enzymology. 472, 345-363 (2010).
- . Absolute Diffusion Coefficients: Compilation of Reference Data for FCS Calibration Available from: https://www.picoquant.com/images/uploads/page/files/7353/appnote_diffusioncoeffients.pdf (2010)
- Foo, Y. H., Naredi-Rainer, N., Lamb, D. C., Ahmed, S., Wohland, T. Factors affecting the quantification of biomolecular interactions by fluorescence cross-correlation spectroscopy. Biophysical journal. 102 (5), 1174-1183 (2012).
- Baum, M., Erdel, F., Wachsmuth, M., Rippe, K. Retrieving the intracellular topology from multi-scale protein mobility mapping in living cells. Nature Communications. 5, 4494 (2014).
- Wohland, T., Rigler, R., Vogel, H. The standard deviation in fluorescence correlation spectroscopy. Biophysical journal. 80 (6), 2987-2999 (2001).
- Ries, J., et al. Automated suppression of sample-related artifacts in Fluorescence Correlation Spectroscopy. Optics Express. 18 (11), 11073 (2010).
- Ries, J., Schwille, P. New concepts for fluorescence correlation spectroscopy on membranes. Physical Chemistry Chemical Physics. 10 (24), 3487 (2008).
- Mayer, M. C., et al. Amyloid precursor-like protein 1 (APLP1) exhibits stronger zinc-dependent neuronal adhesion than amyloid precursor protein and APLP2. Journal of Neurochemistry. 137 (2), 266-276 (2016).
- Linkert, M., et al. Metadata matters: access to image data in the real world. The Journal of Cell Biology. 189 (5), 777-782 (2010).
- Trullo, A., Corti, V., Arza, E., Caiolfa, V. R., Zamai, M. Application limits and data correction in number of molecules and brightness analysis. Microscopy Research and Technique. 76 (11), 1135-1146 (2013).
- Nolan, R., et al. nandb-number and brightness in R with a novel automatic detrending algorithm. Bioinformatics. 33 (21), 3508-3510 (2017).
- Hammond, G. R. V., Sim, Y., Lagnado, L., Irvine, R. F. Reversible binding and rapid diffusion of proteins in complex with inositol lipids serves to coordinate free movement with spatial information. The Journal of cell biology. 184 (2), 297-308 (2009).
- Hendrix, J., Dekens, T., Schrimpf, W., Lamb, D. C. Arbitrary-Region Raster Image Correlation Spectroscopy. Biophysical journal. 111 (8), 1785-1796 (2016).
- Hendrix, J., et al. Live-cell observation of cytosolic HIV-1 assembly onset reveals RNA-interacting Gag oligomers. The Journal of cell biology. 210 (4), 629-646 (2015).
- Hendrix, J., Schrimpf, W., Höller, M., Lamb, D. C. Pulsed Interleaved Excitation Fluctuation Imaging. Biophysical Journal. 105 (4), 848-861 (2013).
- Honigmann, A., et al. Scanning STED-FCS reveals spatiotemporal heterogeneity of lipid interaction in the plasma membrane of living cells. Nature Communications. 5 (1), 5412 (2014).
- Chojnacki, J., et al. Envelope glycoprotein mobility on HIV-1 particles depends on the virus maturation state. Nature Communications. 8 (1), 545 (2017).
- Godin, A. G., et al. Revealing protein oligomerization and densities in situ using spatial intensity distribution analysis. Proceedings of the National Academy of Sciences of the United States of America. 108 (17), 7010-7015 (2011).
- Müller, J. D., Chen, Y., Gratton, E. Resolving Heterogeneity on the Single Molecular Level with the Photon-Counting Histogram. Biophysical Journal. 78 (1), 474-486 (2000).
- Kim, S. A., Heinze, K. G., Bacia, K., Waxham, M. N., Schwille, P. Two-Photon Cross-Correlation Analysis of Intracellular Reactions with Variable Stoichiometry. Biophysical Journal. 88 (6), 4319-4336 (2005).
- Jenkins, E., et al. Reconstitution of immune cell interactions in free-standing membranes. Journal of cell science. 132 (4), (2018).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados