Zum Anzeigen dieser Inhalte ist ein JoVE-Abonnement erforderlich. Melden Sie sich an oder starten Sie Ihre kostenlose Testversion.
Method Article
Eine Fluoreszenz Fluktuation Spektroskopie Assay von Protein-Protein-Wechselwirkungen an den Zell-Zell-Kontakten
In diesem Artikel
Zusammenfassung
Dieses Protokoll beschreibt einen Fluoreszenz Fluktuation Spektroskopie basierende Ansatz zur Untersuchung von Interaktionen zwischen den Proteinen, die Vermittlung von Zell-Zell-Interaktionen, d.h. Proteine in der Zelle Verzweigungen direkt in lebenden Zellen lokalisiert. Wir bieten detaillierte Richtlinien auf Kalibrierung, Datenerfassung und -Analyse, einschließlich Korrekturen möglich Artefakt Quellen.
Zusammenfassung
Eine Vielzahl von biologischen Prozessen umfasst Zell-Zell-Interaktionen, in der Regel vermittelt durch Proteine, die an der Schnittstelle zwischen benachbarten Zellen interagieren. Von Interesse sind nur einige Proben in der Lage, speziell solche Interaktionen direkt in lebenden Zellen zu sondieren. Hier präsentieren wir Ihnen einen Test um die Bindung der Proteine an den Oberflächen von benachbarten Zellen, Zell-Zell Kontakte zu messen. Dieser Test besteht aus zwei Schritten: Mischen von Zellen mit dem Ausdruck der Proteine des Interesses mit verschiedenen fluoreszierenden Proteinen verschmolzen, gefolgt von Fluoreszenz Fluktuation Spektroskopie Messungen bei Zell-Zell-Kontakte mit einem konfokalen Laser-scanning-Mikroskop. Wir zeigen die Machbarkeit des Assays in einem biologisch relevanten Kontext durch die Messung der Wechselwirkungen von Amyloid Vorläufer-Like Protein 1 (APLP1) über Zell-Zell-Verbindungen. Wir bieten detaillierte Protokolle über die Datenerfassung mit Fluoreszenz-basierte Techniken (Scannen Kreuzkorrelation Fluoreszenzspektroskopie, Kreuzkorrelation Anzahl und Helligkeit Analyse) und den gewünschten Gerätes Kalibrierungen. Darüber hinaus diskutieren wir wichtige Schritte in der Datenanalyse und Gewusst wie: identifizieren und beheben Sie externe, falsche Signal Variationen, wie Sie durch Immunofluoreszenz oder Zelle Bewegung.
Im Allgemeinen der vorgestellte Test gilt für alle Homo- oder Zellkulturmodells Proteinprotein Interaktion auf Zell-Zell Kontakte zwischen den Zellen der gleichen oder unterschiedlichen Typen und kann auf eine kommerzielle konfokale Laser-scanning-Mikroskop implementiert werden. Eine wichtige Voraussetzung ist die Stabilität des Systems, das muss ausreichen, um die Sonde diffusiven Dynamik der interessierenden Proteine über mehrere Minuten.
Einleitung
Viele biologische Prozesse auftreten, an den Standorten der Zell-Zell-Interaktionen, z.B. Zellezelle Adhäsion1,2,3, Zell-Zell-Fusion-4 und zelluläre Anerkennung5. Solche Ereignisse sind besonders wichtig bei der Entwicklung von mehrzelligen Lebewesen und für die Zell-Zell-Kommunikation, z. B. während Immunantworten. Diese Prozesse werden in der Regel durch Proteine vermittelt, die an der Oberfläche, d. h. an der Plasmamembran (PM) der benachbarten Zellen lokalisiert sind und spezifische Wechselwirkungen, die auf der Zell-Zell-Kontakt genau geregelten in Raum und Zeit sind zu unterziehen. In vielen Fällen diese Interaktionen sind direkte Homo- oder Zellkulturmodells Protein-Protein- Trans -Wechselwirkungen, aber können auch beinhalten, Ionen oder Liganden als extrazelluläre linker1. Obwohl von grundlegender Bedeutung, fehlt von Assays sondieren dieser spezifischen Protein-Protein-Interaktionen direkt in der natürlichen Umgebung von lebenden Zellen. Viele Methoden erfordern entweder Zellaufschluss (z. B. biochemische Tests wie z. B. co-Immunopräzipitation6), Fixierung (z. B. einige der Höchstauflösung optische Mikroskopie-Techniken und der Zell-Zell-Elektronen-Mikroskopie 7Kontakte), oder sind unspezifisch, z. B. Aggregation / Adhäsion assays8,9. Um dieses Problem zu überwinden, wurden Fluoreszenztechniken umgesetzt, basierend auf Fluoreszenz Resonanz Energie Transfer (FRET)10 oder Fluoreszenz Ergänzung11. Um ausreichend geringe Abstände zwischen Fluorophore zu erreichen, erfordern diese Methoden jedoch Fluoreszenzmarkierungen auf der extrazellulären Seite der Proteine10, potenziell Trans Interaktionen behinderen.
Hier präsentieren wir Ihnen eine alternative Fluoreszenz basierende Assays für Protein-Protein-Wechselwirkungen an den Zell-Zell-Kontakten. Dieser Ansatz verbindet Fluoreszenz Kreuzkorrelation Ansätze (Fluoreszenzspektroskopie Kreuzkorrelation Scan (sFCCS), Kreuzkorrelation Anzahl und Helligkeit (CcN & B)) und Mischen von Zellen mit dem Ausdruck einer Fusion-Konstrukt des Proteins Interesse, z. B. eine Adhäsion-Rezeptor. Die untersuchten Rezeptoren in den zwei interagierenden Zellen mit zwei spektral getrennte fluoreszierende Proteine (FPs), von der intrazellulären gekennzeichnet sind (siehe Abbildung 1A).
Die eingesetzten Methoden basieren auf der statistischen Auswertung der Fluoreszenz Schwankungen induziert durch die diffusive Bewegung von fluoreszierenden Fusionsproteinen durch die fokalen Volumen von einem konfokalen Laser-scanning-Mikroskop. Mehr im Detail Sonden der Assay die Co Diffusion der Proteine des Interesses an beiden benachbarten PMs an den Zell-Zell-Kontakten. Wenn die Proteine Trans Interaktionen durchmachen, tragen diese Trans -komplexe fluoreszierende Proteine in beiden spektralen Kanälen ausstrahlen, wodurch korrelierte Fluoreszenz Schwankungen der beiden Strahler. Auf der anderen Seite, wenn keine Bindung auftritt, werden die Nummer Schwankungen der Proteine in PMs vor, verursacht keine korrelierten Schwankungen unabhängig. Die Übernahme kann auf zwei Arten durchgeführt werden: 1) sFCCS basiert auf einer linienförmigen Scan über die Zell-Zell-Kontakt und effektiv Sonden die Interaktionen in einem Ort befindet sich im Kontaktbereich. Durch eine zeitliche Analyse der Fluoreszenz Schwankungen informiert sFCCS auch Dynamik, d. h. die Diffusionskoeffizienten von Proteinkomplexen; (2) CcN & B basiert auf eine pixelweise Analyse einer Sequenz von Bildern bei der Zell-Zell-Kontaktbereichen erworben. Es hat Fähigkeit, Sonde und Karte Interaktionen entlang der gesamten Region (in einer Bildebene) zu kontaktieren, aber liefert keine Informationen auf Dynamik. Beide Methoden sind kombinierbar mit einer Analyse der molekularen Helligkeit, d. h. die durchschnittliche Fluoreszenzsignal einzelne diffundierende Proteinkomplexe in der Zeiteinheit abgegebene und geben damit Schätzungen der Stöchiometrie von Proteinkomplexen im Zell-Zell-Kontakte.
In diesem Artikel bieten wir ausführliche Protokolle zur Probenvorbereitung, Kalibrierung, Datenerfassung und Analyse durchführen den vorgestellten Test auf eine kommerzielle konfokale Laser-scanning-Mikroskop. Die Experimente können auf jedem Instrument ausgestattet mit Photon counting oder analogen Detektoren und einem Objektiv mit hoher numerischer Apertur durchgeführt werden. Weiter wichtige Schritte des Protokolls zu diskutieren und mehrere Prozesse verursacht artifizielle Signalschwankungen, z. B. Detektor Lärm, Immunofluoreszenz oder Zelle Bewegung Korrektur Regelungen vorsehen. Entwickelt um die Wechselwirkungen zwischen adhärente Zellen Sonde, Assays für Aussetzung Zellen geändert werden kann, oder angepasst Modell Membran-Systeme, z. B. giant Unilamellar Vesikeln (GUVs) oder riesigen Plasma Membranvesikel (GPMVs), so dass die Quantifizierung von Interaktionen in verschiedenen Lipid-Umgebungen oder in Ermangelung einer organisierten Zytoskelett12,13.
Scannen Kreuzkorrelation Fluoreszenzspektroskopie ist eine modifizierte Version der Fluoreszenz-Spektroskopie Kreuzkorrelation14 und wurde speziell für die Sonde langsam diffusiven Dynamik in Lipid-Membranen-15. Es basiert auf einer Linie Scan Akquisition senkrecht zur PM mit fluoreszierenden Proteinen von Interesse. Um Wechselwirkungen zwischen zwei unterschiedlich beschriftete Protein Arten Sonde, erfolgt die Übernahme in zwei Spektralkanäle mit zwei Laser-Linien und zwei Erkennung Fenster für die spektral getrennte Fluorophore. Durch die langsame Diffusion Dynamik von Proteinen in der PM (D≤ ~ 1 µm2/s), eine Cross-Talk-freie Messung kann durch den Wechsel der Erregung System von Linie auf15Linie durchgeführt werden. Die Analyse beginnt mit: (1) eine Ausrichtung Algorithmus Korrektur der seitlichen Zellbewegung basierend auf abschnittsweise durchschnittlich ~ 1000 Zeilen, (2) die Bestimmung der Position mit maximale Fluoreszenz-Signald.h. die Uhr Position, in jedem Block und (3) verschieben alle Blöcke bis hin zum gemeinsamen Ursprung12,15, in jeden Kanal separat. Anschließend wird eine automatische Auswahl der Pixel entspricht der PM durchgeführt, indem der Zentralregion aus einem "glockenförmig" Anfall von der Summe aller ausgerichteten Linien (z. B. Zentrum ± 2.5σ). Integration des Signals in jeder Zeile ergibt die Membran Fluoreszenz Zeitreihen F(t) in jedem Kanal (g = Grün-Kanal, R = Rot-Kanal). Beachten Sie, dass die Pixelgröße klein genug, z. B. muss < 200 nm, die Form des Punktes zu rekonstruieren Funktion zu verbreiten und finden Sie ihre Mitte, entsprechend der Position des Premierministers. In Gegenwart von erheblichen Immunofluoreszenz Fluoreszenz Zeitreihen in jedem Kanal können mit einer Doppel-exponential-Funktion modelliert und dann mit der folgenden Formel korrigiert:16
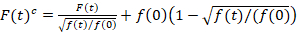 . (1)
. (1)
Es ist wichtig zu beachten, dass diese Formel effektiv korrigiert die Amplituden und Diffusionszeiten aus der Korrelationsanalyse von F(t)c, im Vergleich zu Parameterschätzungen, die sich aus der unkorrigierte F(t)würde. Dann das Auto und Kreuzkorrelation Funktionen (ACFs / CCFs) der Fluoreszenz Signale werden berechnet:
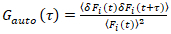 , (2).
, (2).
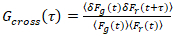 , (3).
, (3).
wo δFich = Fich(t) - 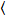 Fich(t)
Fich(t)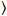 und ich = g, R.
und ich = g, R.
Eine zweidimensionale Diffusion Modell ist dann auf alle Korrelation Funktionen (CFs) ausgestattet:
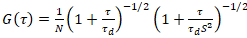 . (4)
. (4)
Dabei bezeichnet N die Anzahl der fluoreszierende Proteine in der Beobachtung Volumen und τd Diffusionszeit für jeden Kanal. Dieses Modell berücksichtigt, dass in der beschriebenen experimentellen Einstellung Diffusion von Proteinen in der Uhr tritt in der X-Z-Ebene, im Gegensatz zu der häufig verwendete Konfiguration der Fluoreszenz Korrelation Spektroskopie (FCS) auf Membranen sondieren Experimente Verbreitung in der X-y-Ebene die konfokale Volumen17. Die Taille w0 und der Struktur Faktor S, beschreibt die Dehnung von fokalen Volumen in Z, S wZ = wZ/w0, stammen aus einer Punkt-FCS Kalibrierung Messung mit spektral ähnliche Farbstoffe und gleichen optischen Einstellungen durchgeführt mit bereits vorhandenen Werten für den Diffusionskoeffizienten DFarbstoff:
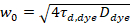 , (5).
, (5).
wo τd, Farbstoff die gemessene durchschnittliche Verbreitung der Farbstoffmoleküle anpassen eines Modells zur dreidimensionalen Diffusion an den Daten entnommen ist Berücksichtigung Konto Übergänge von einem Bruchteil T aller N Moleküle zu einem Triplett-Zustand mit einer Zeitkonstante ττ:
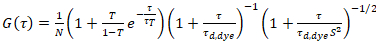 . (6)
. (6)
Schließlich sind die Diffusionskoeffizienten (D), molekulare Helligkeitswerte (ε) und die relative Kreuzkorrelation von sFCCS Daten (rel.cc.) wie folgt berechnet:
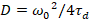 , (7).
, (7).
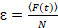 , (8).
, (8).
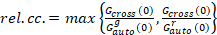 , (9).
, (9).
Wo befindet sich die Amplitude der Kreuzkorrelation Funktion Güberqueren(0) und 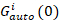 ist die Amplitude der Autokorrelationsfunktion in der ich-th Kanal.
ist die Amplitude der Autokorrelationsfunktion in der ich-th Kanal.
Diese Definition der relativen Kreuzkorrelation, d. h. mit max statt in der Gleichung 9 bedeuten berücksichtigt, die die maximale Anzahl von komplexen zwei Protein-Arten in unterschiedlichen Konzentrationen von begrenzt ist die in einer geringeren Anzahl vorkommenden Arten.
Kreuzkorrelation Anzahl und Helligkeit basiert auf einen Augenblick Analyse der Fluoreszenzintensität für jeden Bildpunkt ein Bildstapel erwarb im Laufe der Zeit an einer festen Position in der Probe in der Regel bestehend aus ca. 100-200 Bildern, mit zwei spektralen Kanäle () g = Grün-Kanal, R = Rot-Kanal). Aus dem zeitlichen Mittelwert 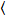 ich
ich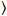 ich und Varianz
ich und Varianz 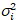 , die molekulare Helligkeit εi und Nummer nich werden in jedem Pixel und Spektralkanal berechnet (Ich = g, R)18:
, die molekulare Helligkeit εi und Nummer nich werden in jedem Pixel und Spektralkanal berechnet (Ich = g, R)18:
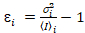 , (10).
, (10).
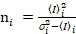 . (11)
. (11)
Es ist wichtig zu beachten, dass im Idealfall eine wahre Photon counting Detektor Formeln zuweisen. Für analoge Systeme gelten die folgenden Gleichungen19,20:
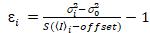 , (12).
, (12).
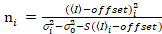 . (13)
. (13)
Hier, S ist der Umrechnungsfaktor zwischen erkannten Photonen und die aufgezeichneten digitalen Grafen 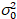 ist das Auslesen Rauschen und Offset bezieht sich auf den Detektor Intensität Offset. Im Allgemeinen sollten diese Mengen für jeden Melder, basierend auf der Messung der Detektor Varianz als eine Funktion der Intensität für stetigen Beleuchtung19, z. B. einer reflektierenden Metalloberfläche oder getrocknete Farbstofflösung kalibriert werden. Der Versatz kann durch Messung der Pulsrate für eine Probe ohne Anregungslicht bestimmt werden. Durch die Durchführung einer linearen Regression der Detektor-assoziierten Varianz
ist das Auslesen Rauschen und Offset bezieht sich auf den Detektor Intensität Offset. Im Allgemeinen sollten diese Mengen für jeden Melder, basierend auf der Messung der Detektor Varianz als eine Funktion der Intensität für stetigen Beleuchtung19, z. B. einer reflektierenden Metalloberfläche oder getrocknete Farbstofflösung kalibriert werden. Der Versatz kann durch Messung der Pulsrate für eine Probe ohne Anregungslicht bestimmt werden. Durch die Durchführung einer linearen Regression der Detektor-assoziierten Varianz 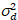 versus Intensität (ich) Grundstück, S und
versus Intensität (ich) Grundstück, S und 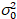 ermittelten19werden können:
ermittelten19werden können:
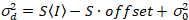 . (14)
. (14)
Zu guter Letzt die Kreuzkorrelation Helligkeit wird in jedem Pixel berechnet und versteht man im Allgemeinen als21
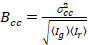 , (15).
, (15).
wo 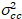 ist die Kreuz-Varianz
ist die Kreuz-Varianz 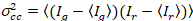 .
.
Zur langlebige Schwankungen zu filtern, werden alle CcN & B Berechnungen durchgeführt nach einem Güterwagen, Filterung, unabhängig für jedes Pixel-22. Kurz, ni, εich (Ich = g, f) und Bcc in Schiebe-Segmente von z. B. 8-15 Frames berechnet. Die so erhaltenen Werte können dann gemittelt werden, um das letzte Pixel Anzahl und Helligkeit Werte zu erhalten.
Stöchiometrie-Analyse
Um die Stöchiometrie der Proteinkomplexe an den Zell-Zell-Kontakten abschätzen zu können, kann die molekulare Helligkeit separat in jedem spektralen Kanal für die sFCCS oder CcN & B Daten analysiert werden. In sFCCS erhält man einen Helligkeitswert pro Messung in jedem Kanal. In CcN & B erhält man ein Histogramm Helligkeit alle Pixel, die die Zell-Zell-Kontakt und der durchschnittliche (oder mittlere) Wert kann als repräsentative Helligkeit für die Messung verwendet werden. Durch die Ausführung der gleichen Analyse auf eine Monomere Referenz können alle Helligkeitswerte normalisiert werden, um den durchschnittlichen Oligomere Zustand der erkannten Proteinkomplexe direkt zu erhalten. An dieser Stelle ist es wichtig, auf das Vorhandensein von nicht-fluoreszierende FPs zu korrigieren, die zu einer Unterschätzung der oligomere Zustand führen kann. Dies erfolgt in der Regel durch die Messung der Helligkeit von einem Homo-dimeres Referenz Protein23,24 mit einfarbigen sFCS oder Nummer und Helligkeit (N & B).
Protokoll
1. Probenvorbereitung: Zell-Zell-mischen-Assay
Hinweis: Das folgende Protokoll beschreibt den Mischvorgang für adhärente Zellen. Er kann für Zellen in Suspension kultiviert geändert werden.
- Samen Sie eine entsprechende Anzahl von Zellen auf einem 6-Well-Platte, z. B. 800.000 HEK 293T Zellen (mit einem Neubauer Kammer zählen gezählt), einen Tag vor Transfektion. Die Anzahl kann je nach Jahreszeit zwischen Aussaat und Transfektion geändert und bereinigt um andere Zelltypen. Um ein grundlegendes Experiment (z.B. Proteine des Interesses und Negativkontrolle) durchführen, bereiten Sie mindestens 4 Brunnen. Zellkulturen bei 37 ° C, 5 % CO2 in Dulbeccos geändert Eagle Medium (DMEM) Mittel, ergänzt mit fetalen bovine Serum (10 %) und L-Glutamin (1 %).
- Transfizieren Zellen entsprechend den Anweisungen des Herstellers (siehe die Tabelle der Materialien).
- Um ein grundlegendes Experiment durchführen, transfizieren Sie, in separaten Brunnen, Plasmide für das Protein des Interesses verschmolzen zu einem "grünen" (z. B. Monomeren verbesserte grün fluoreszierendes Protein (mEGFP) oder gelb fluoreszierende Protein (mEYFP)) oder 'rot' (z. B. mCherry, oder mCardinal) fluoreszierendes Protein.
Hinweis: In diesem Protokoll konzentrieren wir uns auf APLP1-mEYFP und APLP1-mCardinal12, und die entsprechende negativ-Kontrolle, z. B. Myristoylated-Palmitoylated-mEYFP (Myr-Palm-mEYFP) und -mCardinal (Myr-Palm-mCardinal)12. In der Regel 200 ng - 1 µg Plasmid DNA sind ausreichend. Hohen Transfektion Effizienz erhöht die Chance, "rote" und "grüne" Zellen in Kontakt zu finden. Ändern Sie die Menge des Plasmids und Transfection Reagens Transfektion Effizienz zu optimieren. Kritische: Zelle Konfluenz sollte ca. 70 %, wenn die Zellen transfecting. Wenn Zellen über Zusammenfluss sind, verringert sich die Transfektion Effizienz. Wenn Zellen nicht genug, konfluierende Transfektion sind und mischen kann Stress auslösen und zu verhindern, dass viele Zellen aus richtigen Anlage nach dem mischen.
- Um ein grundlegendes Experiment durchführen, transfizieren Sie, in separaten Brunnen, Plasmide für das Protein des Interesses verschmolzen zu einem "grünen" (z. B. Monomeren verbesserte grün fluoreszierendes Protein (mEGFP) oder gelb fluoreszierende Protein (mEYFP)) oder 'rot' (z. B. mCherry, oder mCardinal) fluoreszierendes Protein.
- Mischen ~4 ± 2 h nach Transfektion Zelle führen.
- Entfernen Sie Wachstumsmedium zu und waschen Sie auch sanft mit 1 mL PBS ergänzt mit Mg2 + und Ca2 +. Dann entfernen Sie die PBS. (Kritische) Fallen Sie PBS auch hochkant, Ablösung der Zellen während des Waschens zu verhindern.
- Fügen Sie ~ 50 µL Trypsin Ethylenediaminetetraacetic (EDTA) Säurelösung tropfenweise in jede Vertiefung, Ablösung der Zellen zu erleichtern. Inkubation bei 37 ° C für 2 Minuten danach, schütteln langsam 6-Well-Platte seitlich um die Zellen zu lösen.
Hinweis: Längere Inkubationszeiten möglicherweise für einige Zelltypen erforderlich. - Jedes gut 950 µL Wachstumsmedium hinzu und Aufschwemmen Sie Zellen durch Pipettieren ein paar Mal rauf und runter, damit alle Zellen von gut unten abnehmen. (Kritische) Sicherstellen Sie, dass Zellen sind richtig Nukleinsäuretablette und indem visuell auf das Fehlen von großzellige Aggregate nach Wiederfreisetzung voneinander getrennt. Ansonsten erhält man viele "rote"-"rot" oder "grün"-"grüne" Kontakte nach dem mischen.
- Übertragen die Zelle Lösung einen Brunnen (Protein des Interesses oder negativ-Kontrolle) mit den entsprechenden gut, d. h. 'rot' (z. B. APLP1-mCardinal-transfizierten) auf "grün" (z. B. APLP1-mEYFP-transfizierten) Zellen. Mischen von Pipettieren sanft ein paar Mal rauf und runter. Dann ausgesät Samen die gemischten Zellen auf 35mm unten Glasschalen (1 mL gemischte Zelle Lösung pro Schale) plus 1 mL Wachstumsmedium und Kultur Zellen für einen weiteren Tag bei 37 ° C, 5 % CO2.

Abbildung 1 . Experimentelle Workflow und schematische Darstellung des Scannens Kreuzkorrelation Fluoreszenzspektroskopie und Kreuzkorrelation Anzahl und Helligkeit Analysen bei Zell-Zell Kontakte. (A) Regelung der Probenvorbereitung: zwei Zellpopulationen transfiziert mit dem Protein des Interesses (z.B. APLP1) mit zwei spektral unterschiedliche fluoreszierende Proteine (z. B. mEYFP und mCardinal) verschmolzen werden nach Transfektion gemischt. Kontakte von anders transfizierten Zellen sind in der Mikroskopie-Experimente ausgewählt. Um Interferenzen mit extrazellulären Bindung Domains zu vermeiden, sollte das fluoreszierende Protein an den intrazellulären Terminus des Proteins des Interesses fusioniert werden. (B) Scannen FCC (sFCCS) Messungen sind durchgeführten senkrecht an den Zell-Zell-Kontakt in zwei spektralen Kanälen (Channel 1, grün und Kanal 2, rot). Scanlinien (dargestellt als Kymographs) ausgerichtet und Membran Pixel zusammengefasst. Dann werden ACFs und CCFs die Intensität Spuren Fich(t)berechnet. Prävalenz werden in rot und grün dargestellt. CCF ist in blau dargestellt. (C) Kreuzkorrelation N & B (CcN & B) Erwerb führt zu einer dreidimensionalen Bildstapel (X-y-Time). Ein ROI ist rund um die Zell-Zell-Kontakt ausgewählt. Dann Kanal und Kreuzkorrelation Helligkeit (ε1, ε-2und Bcc) Werte sind in jedem Zellezelle Kontakt Pixel berechnet. Die Ergebnisse werden dann als Histogramme, Bündelung aller ausgewählten Pixel visualisiert. Bitte klicken Sie hier für eine größere Version dieser Figur.
2. Probenvorbereitung: Positivkontrolle für Kreuzkorrelation Experimente und Homo-Dimer Konstrukt für Helligkeit Analyse
- Samen Sie 600.000 HEK 293T Zellen, die mit einer Zelle zählen Kammer, auf 35-mm-Glas unteren Gerichte einen Tag vor Transfektion gezählt. Kultur der Zellen bei 37 ° C, 5 % CO2 im kompletten DMEM Medium (siehe Punkt 1.1) für einen anderen Tag.
- Transfizieren Zellen mit ~ 250 ng Plasmid DNA nach Anweisungen des Herstellers. Verwenden Sie für die Positivkontrolle Kreuzkorrelation ein Plasmid Codierung eine Membran verankerte fluoreszierenden Proteins Hetero-Dimer, z. B. Myr-Palm-mCherry-mEGFP oder Myr-Palm-mCardinal-mEYFP12 entspricht der FPs des Proteins des Interesses. Verwenden Sie für die Kalibrierung von Helligkeit Plasmide, die sowohl eine Membran verankerte FP Monomer und Homo-Dimer entspricht die FPs verschmolzen mit dem Protein des Interesses, z. B. Myr Palm mEYFP und Myr-Palm-mEYFP-mEYFP-Codierung, um die Helligkeit Analyse der zu kalibrieren APLP1-mEYFP-12.
- Kulturzellen bei 37 ° C, 5 % CO2 im kompletten DMEM Medium (siehe Punkt 1.1) für einen anderen Tag.
3. konfokale Laser Scanning Microscopy: Setup und Kalibrierung der fokalen Volumen
Hinweis: Das folgende Protokoll ist für Experimente mit mEGFP/mEYFP und mCherry/mCardinal auf die Laser-scanning-confocal Mikroskop in dieser Studie verwendeten geschrieben. Der optische Aufbau, der Software-Einstellungen (Laserlinien, dichroitischen Spiegel, Filter) und Auswahl der Kalibrierung Farbstoffe können für andere FPs und Mikroskop Setups geändert werden.
- Schalten Sie das Mikroskop und Laser mindestens eine Stunde vor dem Experiment Laser Stabilität und Gleichgewicht der Temperatur zu gewährleisten.
- Bereiten Sie 100-200 µL geeignete wasserlösliche Fluoreszenzfarbstoff Lösungen (siehe die Tabelle der Materialien für Beispiele) in Wasser oder PBS fokale Volumen, mit Konzentrationen im Bereich von 10-50 nM zu kalibrieren.
- Legen Sie die Farbstofflösungen auf eine saubere 35 mm unten Glasschale #1.5, d. h. mit einer Dicke von 0,16 0,19 mm.
Hinweis: Verwenden Sie idealerweise Gerichte mit Hochleistungs-Deckglas mit einem niedrigen Dickentoleranz, z. B. 0.170 ± 0,005 mm, so dass eine optimale Halsband Ring Korrektur (Schritt 3,6). Es ist wichtig, verwenden Sie die gleiche Art von Gericht, wie später für die folgenden Experimente verwendet. - Legen Sie die Schale mit der Farbstofflösung direkt auf das Ziel (vorzugsweise Wasser eintauchen, mit NA 1.2) um Konzentration in der Lösung zu gewährleisten. Alternativ legen Sie die Schüssel auf Probenhalter und Konzentration in der Probe (z. B. 10-20 µm über den Boden der Schale).
Hinweis: Wir empfehlen nicht mit Öl Ziele durch das schlechte Signal erhält man, wenn tief in wässrigen Proben zu konzentrieren. - Richten Sie die Anregung und Emission Pfad, z. B. wählen, 488 nm Laser, 488/561 nm dichroitischen Spiegel, Erkennung Fenster 499-552 nm und eine Lochkamera Größe 1 luftig Einheit (AU). Stellen Sie sicher, dass die Lochkamera Größe ist identisch mit der, die Kreuzkorrelation Messungen verwendet werden.
- Passen Sie Lochkamera Position (Pinhole-Anpassung) und den objektiven Halsband Ring Zählrate zu maximieren. Drehen Sie zu diesem Zweck Halsband Ring bis maximale Pulsrate erkannt wird.
Hinweis: Die Halsband Ring Korrektur entfallen auf die spezifische Stärke der Glasabdeckung verwendet. Maximierung der Pulsrate, dh., wie viele Photonen pro Molekül wie möglich zu sammeln, ist entscheidend für das Signal-Rausch-Verhältnis (SNR) der Messungen zu maximieren. - Führen Sie eine Reihe von Punkt FCS Messungen (z. B. 6 Messungen an verschiedenen Standorten, jeweils bestehend aus 15 Wiederholungen von 10 s, d. h. 2,5 min Gesamtzeit, abgetastet mit 1 µs Verweilzeit oder weniger) bei gleicher Laserleistung wie in Kreuzkorrelation Messungen (in der Regel ~ 1 %, d. h. ~ 1-2 µW).
- Passen Sie einen dreidimensionalen Diffusion Modell einschließlich einen Triplett-Beitrag (Gleichung 6) auf die Daten.
Hinweis: In der Regel die erhaltenen Diffusionszeiten sind rund 30 µs und die Struktur-Faktor ist um 4-8. - Berechnen Sie die Taille w0 aus dem gemessenen durchschnittlichen Diffusionszeit und veröffentlichten Werte für den Diffusionskoeffizienten des verwendeten Farbstoffs bei Raumtemperatur25 nach Gleichung 5. Typische Werte sind 200-250 nm.
- Die Kalibrierroutine (Schritte 3,4-3,9) mit einem anderen Fluoreszenzfarbstoff für einen zweiten Erkennung Kanal gegebenenfalls wiederholen (z. B. 561 nm Anregung und Erkennung zwischen 570 nm und 695 nm). Halten Sie die Lochkamera Position und Größe, wie es für den ersten Kanal der Erkennung festgelegt wurde.
- Berechnen Sie die molekulare Helligkeit (Gleichung 8) aus der Kalibrierung Messungen und speichern Sie die erhaltenen Werte.
Hinweis: Typische Werte für das Setup verwendet werden ~8 bis 10 kHz/Molekül (MOL) für 1.8 µW 488 nm Erregerleistung. Niedriger als die übliche Werten Schmutz auf das Ziel, die Schiefstellung des Setup oder ein reduzierter Laserleistung hinweisen könnten. Prüfen Sie und speichern Sie Laser Ausgangsleistungen bei dem Ziel, die regelmäßig mit einem Leistungsmesser. Zum Vergleich von verschiedenen Setups ist molekulare Helligkeit durch Laser Erregerleistung normiert der sinnvollste Parameter Mikroskop Leistung bewerten.
4. Scannen Kreuzkorrelation Fluoreszenzspektroskopie: Übernahme
Hinweis: Das folgende Protokoll ist für Experimente mit mEGFP/mEYFP ("grün") und mCherry/mCardinal ("rot") auf der konfokalen Laser-Rastermikroskop in dieser Studie verwendeten geschrieben. Der optische Aufbau und Einstellungen der Software (Laserlinien, dichroitischen Spiegel, Filter) können für andere FPs oder Mikroskop Setups abweichen.
- Richten Sie den Strahlengang, z. B. 488 nm und 561 nm Anregung und 488/561 nm dichroitischen Spiegel, Lochkamera auf 1 AU für 488 nm Anregung. Um spektrale Übersprechen zu vermeiden, wählen Sie zwei separate Spuren zu begeistern und zu erkennen, mEGFP/mEYFP (488 nm Anregung, Grün-Kanal) und mCherry / mCardinal (561 nm Anregung, Rot-Kanal) gegenüber dem Vorquartal und Schalter verfolgt jede Zeileauswählen. Verwenden Sie für den Nachweis geeigneter Filter für beide Kanäle, z. B. 499-552 nm in den Grün-Kanal und 570-695 nm im roten Kanal.
- Wenn abwechselnd Erregung nicht möglich ist, verwenden Sie geeignete Filter-Einstellungen für den Rot-Kanal spektrale Nebensprechen zu minimieren (d.h. erkennen mCherry/mCardinal Fluoreszenz nicht unter 600 nm). Dies kann reduzieren die Menge der Photonen, die im roten Kanal erkannt und reduzieren damit die SNR.
- Legen Sie die Schale mit der gemischten Zellen auf dem Probenhalter. Warten Sie mindestens 10 min Temperatur Gleichgewichtherstellung zu gewährleisten und Fokus Drift zu reduzieren.
- Konzentrieren Sie sich auf die Zellen mit Licht im Menü " Suchen ".
- Suchen Sie nach ein paar "rot" und eine "grüne" Zelle miteinander in Kontakt treten. Für die positive Kreuzkorrelation oder Homo-Dimer Helligkeitsregelung (siehe Abschnitt 2), suchen Sie nach einer isolierten Zelle emittiert Fluoreszenz in Kanälen oder der jeweiligen Homo-Dimer-Signal um die Uhr.
Hinweis: (Kritische) minimieren Probe Ausstellung, bei der Suche nach Zellen zu vermeiden vorher Bleichen, die Kreuzkorrelation26reduzieren kann. Deshalb Scannen Sie am schnellsten Scan-Geschwindigkeit und geringen Laserleistungen. Zur Vermeidung von Detektor Sättigung bei der Bildgebung mit dem stark Ausdruck Zellen in Integrationsmodussuchen. Jedoch ist um zu minimieren, Scannen am unteren Laserleistungen in Photon counting -Modus möglich. - Wählen Sie einen Scan Weg senkrecht auf Zell-Zell-Kontakt (oder PM aus einer einzigen Zelle für die Kreuzkorrelation oder Homo-Dimer Helligkeit Positivkontrolle) verwenden die Schaltfläche Zuschneiden , wie dargestellt in Zahlen 1 b und 2A.
Hinweis: Einige ältere Mikroskope erlauben keine beliebigen Scan-Richtungen. In diesem Fall müssen Zell-Zell Kontakte mit Ausrichtung senkrecht zur Scan-Richtung gefunden werden. -
Zoom auf eine Pixelgröße von 50-200 nm und select- Line im Scan-Moduszu erreichen. Frame-Größe auf 128 × 1 Pixel festgelegt.
Hinweis: Typische Pixelgröße beträgt 160 nm, entspricht einer Scan-Länge von ca. 20 µm. - Legen Sie Scan-Geschwindigkeit auf den zulässigen Maximalwert, z. B. 472,73 µs pro Zeile.
Hinweis: Für eine Alternative Erregung Schema entspricht dies 954,45 µs Scan-Zeit, d.h. ~ 1000 Scans/s auf das Setup verwendet. Die Scan-Geschwindigkeit kann je nach den Diffusionskoeffizienten des Proteins des Interesses eingestellt werden. Für Membran verankerte Proteine sind typische Diffusionszeiten um 10-20 ms die Scan-Zeit sollte mindestens zehn Mal kleiner als die Diffusionszeiten. Unteren Scangeschwindigkeiten können induzieren stärker Immunofluoreszenz und geringer Beleuchtung Befugnisse erfordern. Alternativ kann man eine Pause, z.B. 5 ms, zwischen jedem Scan sehr langsam diffundierende komplexe mit Intervall im Untermenü Time Series verhängen. - Wählen Sie die entsprechende Laserleistungen, z. B. ~ 1-2 µW für 488 nm und ~ 5-10 µW 561 nm Erregung.
Hinweis: Höhere Laserleistungen SNR verbessern, aber Immunofluoreszenz zu erhöhen. Daher sollten Laserleistungen werden so gewählt, dass Immunofluoreszenz weniger als 50 % der ursprünglichen Zählrate ist. - 100.000-500.000 Zyklen soll.
Hinweis: Die Anzahl der Scans, d. h. die Dauer der Messung, variieren: längere Messzeiten SNR verbessert werden und möglicherweise besser geeignet für langsam diffundierende Moleküle, aber Bewegung der Zellen und Immunofluoreszenz begrenzen die maximale Messung Zeit. Die hier vorgestellte Daten wurden routinemäßig erworben für ~ 3-6 min, d. h. 200.000-400.000 Linie scannt. - Detektoren auf Photon counting -Modus einstellen Drücken Sie Starten Experiment um die Übernahme zu starten. Wiederholen Sie die Schritte 4,5-4.11 zu einer anderen Zelle zu messen.
Hinweis: Es wird empfohlen, 10-15 Zellen pro Probe anderer Ausdruck Ebene zu messen. (Kritische) Vermeiden Sie Detektor Sättigung auf hohe Expression Ebenen. Die maximale Zählrate überschreiten ~ 1 MHz nicht. - Wenn Helligkeit Analyse festzustellen, Oligomere Staaten durchgeführt wird, führen Sie Homo-Dimer Helligkeitsmessungen Kalibrierung entsprechend modifizierten Schritte 4.1-4.11: Messen Sie jeden fluoreszierenden Proteins Homo-Dimer separat (in isolierten Zellen, zubereitet Protokoll Abschnitt 2) und führen Messungen nur in einem spektralen Kanal.
5. Scannen Kreuzkorrelation Fluoreszenzspektroskopie: Datenanalyse
Hinweis: Das folgende Protokoll folgt eine Implementierung von Analyseverfahren, die in vorherigen Artikeln12,15ausführlich beschrieben. Der Softwarecode ist auf Anfrage bei den Autoren.
- Exportieren Sie die raw-Daten (z. B. CZI) Dateien in ein RGB-TIFF-Bild in Rohdaten-Format. Diese Datei enthält eine Glomeruli mit grünen und roten Kanaldaten im Kanal bezeichnet bzw. G und R des Bildes.
- Importieren Sie die TIFF-Datei mit der entsprechenden Analysesoftware und fahren Sie mit der Analyse durchführen.
Hinweis: Die folgenden Schritte (Steps 5,3-5,7) gelten für jeden Kanal separat: - Richten Sie die Linien durch die Durchführung eines segment-wise oder sich bewegenden Zeit Durchschnitts mit Blöcken von 500-1000 Linien. Bestimmen Sie die Membran Position, d. h. die Pixelposition mit der maximalen Pulsrate in jedem Block. Verschieben Sie alle Blöcke auf der gleichen Seitenlage. Dieses Verfahren behebt für seitliche Verschiebung der Zell-Zell-Kontakt, z. B. durch Zelle Bewegung.
- Zusammenfassend alle ausgerichteten Linien entlang der Zeitachse und die durchschnittliche Intensität Profil mithilfe einer Gaußschen Funktion passen. In Anwesenheit von bedeutenden intrazellulären Hintergrund verwenden einer Gaußschen plus eine sigmoid Funktion. Definieren Sie die Pixel, die die Membran als alle Pixel im ±2.5σ der Membran und fassen die Intensität dieser Pixel in jeder Zeile, Erhalt einer einzigen Fluoreszenz Signalwert für jeden Zeitpunkt (d. h. für jeden Line-Scan).
- Bei Bedarf (z. B. Hintergrund > 10 % von der Membran-Signal), eine Hintergrundkorrektur durch Subtraktion der durchschnittliche Pixel Intensität im Zytoplasma multipliziert mit 2.5σ (in Pixeleinheiten) von der Membran-Fluoreszenz in Blöcke von 1000 Zeilen anwenden. Vermeiden Sie helle intrazelluläre Vesikeln, bei der Auswahl der Hintergrundpixel.
- Wenden Sie Immunofluoreszenz beobachtet wird, eine Bleiche Korrektur. Daher passen Sie die Membran Fluoreszenz Zeitreihen mit einer Doppel-exponential-Funktion und gelten Sie die entsprechende Korrektur Formel, Formel 116.
Hinweis: Alternativ können Fourier Spektrum basierte Korrektur Systeme angewandte27sein. (Kritische) Immunofluoreszenz vorhanden, aber nicht korrigierten für CFs kann stark verzerrt und Parameterschätzungen stark beeinflusst werden können (z.B., siehe Abbildung 5E). - Berechnen Sie die Prävalenz und CCFs nach den Gleichungen 2 und 3 verwenden, z. B. ein Multiple-Tau-Algorithmus-28. Zur Verbesserung der Zuverlässigkeit der Analyse und Artefakte zu vermeiden, Berechnungen Sie die für 10-20 gleich große Segmente der gesamten Messung. Kontrollieren Sie der Fluoreszenz-Zeitreihen und CFs in jedem Segment und entfernen Sie deutlich verzerrt Segmente (siehe Beispiel in Abbildung 4A- 4D). Durchschnittlich alle unverfälschten Segmente.
Hinweis: Dieses Verfahren kann automatisiert werden, um eine subjektive Verzerrungen auf der Daten-29zu vermeiden. Für sehr instabile Messungen kann es hilfreich sein, mit vielen kurzen Segmenten. Die Länge eines Segments sollte jedoch noch mindestens drei Größenordnungen über der Diffusionszeit zu statistischen undersampling Fehler29,30,17. - Eine zweidimensionale Diffusion Modell, Gleichung 4, um die erhaltenen CFs angepasst. Daher fix der Struktur-Faktors, um den Wert in Kalibriermessung (Protokoll Abschnitt 3). Die Genauigkeit der Passung kann verbessert werden, indem Sie ausführen eine gewichtete Passform mit Hilfe der statistischen Gewichte der einzelnen Datenpunkte aus der mehrere Tau-Algorithmus gewonnen.
- Berechnen Sie die Diffusionskoeffizienten mit der kalibrierten Taille nach Gleichung 7.
- Berechnen Sie die molekulare Helligkeit durch Division der durchschnittliche Fluoreszenzintensität in jedem Kanal durch die entsprechende Anzahl von Partikeln, Gleichung 8. Normalisieren Sie die ermittelten Helligkeitswert in jedem Kanal durch die durchschnittliche Helligkeit des entsprechenden Monomeren Verweises auf die Oligomeren Zustand, unter Berücksichtigung Konto nicht fluoreszierenden FPs23erhalten. Bestimmen Sie zu diesem Zweck, durchschnittliche Homo-Dimer Helligkeitswerte von einer Farbanalyse, den Anteil der nicht-fluoreszierende FPs23zu berechnen.
- Berechnen Sie die relative Kreuzkorrelation nach Gleichung 9.
6. Kreuzkorrelation Anzahl und Helligkeit: Detektor-Kalibrierung
Hinweis: Das folgende Protokoll sieht eine allgemeine Richtlinie betreffend, wie man das Erkennungssystem kalibrieren. Dieses Verfahren ist obligatorisch für analoge Systeme, aber ist nicht zwingend erforderlich, wenn wahre Photon counting Detektoren verwendet werden.
- Trocknen Sie geeignete wasserlösliche Farbstofflösungen (siehe Tabelle der Materialien für Beispiele) auf einer 35 mm unten Glasschale. Legen Sie den optischen Pfad entsprechend, d. h. 488 oder 561 nm Anregung und Erkennung bei 499-552 nm oder 570-695 nm, beziehungsweise.
Hinweis: Alternativ kann eine reflektierende metallische Oberfläche anstelle von getrockneten Farbstofflösungen verwendet werden indem man das Metallstück direkt auf das Ziel. - Führen Sie eine N & B Farbmessungen in Regionen mit unterschiedlichen Farbstoff Konzentrationen oder bei verschiedenen Laserleistungen. Daher mit dem Zoom zu einer Pixelgröße von 300 nm, Scangeschwindigkeit zu legen entsprechende Pixel Verweilzeit, z. B. 25 µs und Zyklen auf 100-200 Bilder.
- Detektoren auf Photon counting (oder analog Modus wenn Messungen mit analogen Erkennung durchgeführt werden) und drücken Sie Experiment starten um die Übernahme zu starten. Führen Sie die Messung auf Null Erregerleistung bestimmen die Intensität Offset.
- Als Funktion der Pixel-Intensität für alle gemessenen Pixel Pixel Abweichung des Grundstückes und führen eine lineare Anpassung dieser Daten. Bestimmen S wie die Steigung der linearen Fit. Berechnen Sie den Auslesen Lärm von der y-Achsenabschnitt, mit S und der ermittelten Intensität Offset nach Gleichung 14.
(7) Kreuzkorrelation Anzahl und Helligkeit: Übernahme
- Führen Sie die Schritte 4.1-4.4 sFCCS-Übernahme-Protokoll.
- Ernte zu verwenden, um einen Rahmen von 512 × 128 Pixel um eine Zell-Zell-Kontakt (oder isolierte PM für Homo-Dimer Helligkeitsregelung) wählen und vergrößern , um eine Pixelgröße von 50-100 nm zu erreichen.
- Verwenden Sie Scan-Geschwindigkeit , um entsprechende Pixel Verweilzeit, z. B. 6,3 µs festzulegen.
Hinweis: In N & B, sollte die Verweilzeit Pixel viel kleiner als die Diffusionszeit des Proteins des Interesses sein. Wenn eine Alternative Erregung Schema ausgewählt ist, z. B. Wechsel jede Linie verfolgt, die Zeit zwischen den beiden Spuren sollte kleiner sein als die Diffusionszeit des Proteins des Interesses. Ansonsten ist die nachweisbare Kreuzkorrelation reduziert. - Eingestellten Zyklen auf 100-200 Frames.
Hinweis: Eine höhere Frame-Nummer verbessert die SNR, jedoch Zelle Bewegung kann die gesamte Messzeit zu begrenzen. Die Scanzeit pro Bild sollte viel höher als die Diffusionszeit des Proteins von Interesse sein. Ansonsten die scheinbare Helligkeit wird reduziert, d. h. Partikel erscheinen unbeweglich. Verhängen Sie für sehr langsam diffundierende komplexe eine Pause, z. B. 2 s zwischen Frames mit Intervall im Untermenü " Time Series ". - Laserleistungen auf geeignete Werte eingestellt (typische Werte sind ~ 1-2 µW für 488 nm und ~ 5-10 µW 561 nm Anregung).
Hinweis: Höhere Laserleistung führt zu höhere Helligkeit und bessere SNR, sondern auch verbesserte Immunofluoreszenz. Laserleistungen sollte hoch genug sein, eine erkannten Helligkeit von mindestens ~ 1 kHz zu erreichen/MOL behielt aber niedrig genug, um mehr als 10 %-20 % Immunofluoreszenz zu vermeiden. Für mEGFP/mEYFP oder mCherry/mCardinal, weniger als 10 % Immunofluoreszenz sind in der Regel erhalten. - Detektoren auf Photon counting (oder analog Modus wenn Messungen mit analogen Erkennung durchgeführt werden) festgelegt. Drücken Sie Starten Experiment um die Übernahme zu starten.
- Die Photon Zählrate zu bewerten. Wenn Zählraten in Zellezelle Kontakt Pixel 1 MHz überschreiten, reduzieren Sie die Laserleistung oder wählen Sie Zellen mit geringerer Ausdruck. Wiederholen Sie die Schritte 7.2-7.7. um das nächste paar von Zellen zu messen. Es wird empfohlen, 10-15 Zellen pro Experiment verschiedene Ausdruck Ebene zu messen.
- Wenn Helligkeit Analyse zur Oligomerisierung Quantifizierung durchgeführt wird, führen Sie Homo-Dimer Helligkeitsmessungen Kalibrierung entsprechend modifizierten Schritte 7,1-7,7: Messen Sie jeden fluoreszierenden Proteins Homo-Dimer separat (in isolierten Zellen, zubereitet Protokoll Abschnitt 2) und führen Messungen nur in einem spektralen Kanal.
8. Kreuzkorrelation Anzahl und Helligkeit: Datenanalyse
Hinweis: Das folgende Protokoll folgt eine Analyse der zuvor beschriebenen Verfahren12,31. Der Softwarecode ist von den Autoren auf Anfrage zur Verfügung.
- Importieren Sie die raw-Daten (z. B. CZI Dateien können importiert werden mit dem Bioformats-32 -Paket). Im Durchschnitt alle Frames und wählen Sie eine Region of Interest (ROI) um die Zell-Zell-Kontakt.
- Führen Sie ein Bild Ausrichtung Algorithmus33, z. B. durch die Maximierung der räumlichen Korrelation zwischen ROIs in unmittelbar aufeinander folgenden Einstellungen für beliebige seitliche Übersetzungen, gemittelt über beide Kanäle. Dieses Verfahren wird für die seitliche Bewegung der Zellen korrigiert.
- Beantragen Sie, dass ein Boxcar Filter22 reduzieren überflüssige langlebige Schwankungen aus, z. B. verbleibende Zelle Bewegung oder Hintergrund zu bleichen. Alternativ kann eine detrending Methode angewendet werden, um für Immunofluoreszenz34zu korrigieren.
Hinweis: Wenn keine segment-wise Analyse oder detrending angewendet wird, die scheinbare Helligkeit kann weitgehend überschätzt.- Definieren Sie verschiebbare Segmente, z.B. 8 bis 15 Bilder (z. B. Bilder 1 bis 8, 2, 9 und so weiter) zu und berechnen Sie die Kanal und Kreuzkorrelation Helligkeitswerte nach Gleichungen 10, 11 und 15 pixelweise in jedem Segment. Wenn Detektoren nicht wahr Photon counting Detektoren sind, berücksichtigen Sie die kalibrierten Detektor-Parameter bei der Berechnung der Helligkeit, d. h. Verwendung Gleichungen 12 und 13 statt.
Hinweis: Berechnen die Helligkeitswerte in Segmenten von 8 bis 15 Frames führt zu einer Unterschätzung von 10-20 % von der absoluten Helligkeit und eine 10-20 % Überschätzung der Partikelanzahl. Helligkeit-Verhältnisse (z. B. Dimer Monomer Helligkeit) sind jedoch nicht betroffen, solange die Segmentlänge während der Analyse (Daten nicht gezeigt) konstant gehalten wird. Der statistische Fehler für ein bestimmtes Segmentlänge werden über Simulationen ermittelt und somit für korrigiert. - Durchschnitt der erzielten Helligkeitswerte pixelweise über alle Segmente. Man kann in diesem Schritt entfernen Sie die höchste und niedrigste 5 % Segment Helligkeitswerte vom Durchschnitt oder ohne Segmente, die einer klare Verzerrung in der Intensität, z. B. durch eine intrazelluläre Vesikel oder Aggregat vorübergehend vorhanden in diesen zeigen Pixel.
- Definieren Sie verschiebbare Segmente, z.B. 8 bis 15 Bilder (z. B. Bilder 1 bis 8, 2, 9 und so weiter) zu und berechnen Sie die Kanal und Kreuzkorrelation Helligkeitswerte nach Gleichungen 10, 11 und 15 pixelweise in jedem Segment. Wenn Detektoren nicht wahr Photon counting Detektoren sind, berücksichtigen Sie die kalibrierten Detektor-Parameter bei der Berechnung der Helligkeit, d. h. Verwendung Gleichungen 12 und 13 statt.
- Die Helligkeitswerte der Pixel in Abhängigkeit von der Intensität der Pixel des Grundstückes und wählen Sie die Bevölkerung von Pixeln, die die Zell-Zell-Kontakt entspricht. Hintergrundpixel haben sehr geringe Intensitätswerte. An dieser Stelle neu zu bewerten, die maximale Zählrate. Schließen Sie Pixel mit Zählraten oberhalb von 1 MHz, Pile-Up Effekte zu verhindern.
- Schaffen Sie Kanal und Kreuzkorrelation Helligkeit die Histogramme der ausgewählten Zellezelle Kontakt Pixel zu und zu erhalten Sie die ROI gemittelte Helligkeitswerte. Normalisieren Sie die durchschnittliche Helligkeit Kanalwert, indem die durchschnittliche Helligkeit des entsprechenden Monomeren Verweises auf die Oligomeren Zustand, unter Berücksichtigung Konto nicht fluoreszierenden FPs23erhalten. Daher ermitteln Sie, durchschnittliche Homo-Dimer Helligkeitswerte von einer Farbanalyse, den Anteil der nicht-fluoreszierende FPs23zu berechnen.
- Zur Veranschaulichung Grundstück Kanal und Kreuzkorrelation Helligkeit Karten.
Ergebnisse
Ein erster Test für die Protein-Protein-Interaktion-Assay von Zellen mit dem Ausdruck spektral unterschiedliche fluoreszierende Proteine, gefolgt von sFCCS/CcN & B Messungen (Abbildung 1), d. h. mischen durchgeführt werden auf Proteine, die nicht zu erwarten ist bei der Zell-Zell-Kontakt (d. h. eine Negativkontrolle) interagieren. Daher HEK 293T Zellen mit dem Ausdruck Myristoylated-Palmitoylated-mEYFP (Myr-Palm-mEYFP) oder -mCardinal wurd...
Diskussion
Die hier beschriebene experimentelle Vorgehensweise ermöglicht die Untersuchung von Protein-Protein Trans Interaktionen bei Zell-Zell-Kontakten, Fluoreszenz Fluktuation Spektroskopie Techniken, nämlich sFCCS und CcN & B. Diese Methoden beinhalten eine statistische Analyse der Fluoreszenz-Schwankungen, die durch zwei spektral getrennte FPs verschmolzen, benutzt von Interesse an einen Kontakt von zwei benachbarten Zellen, jeweils mit dem Ausdruck einer oder der andere Fusionsproteins emittiert. Das Vorhandensein...
Offenlegungen
Die Autoren haben nichts preisgeben.
Danksagungen
Diese Arbeit wurde teilweise unterstützt von der Deutschen Forschungsgemeinschaft (DFG) 254850309 zu gewähren. Die Autoren danken Madlen Luckner für kritische Lektüre des Manuskripts.
Materialien
| Name | Company | Catalog Number | Comments |
| DMEM growth medium | PAN-Biotech | P04-01548 | |
| DPBS w/o: Ca2+ and Mg2+ | PAN-Biotech | P04-36500 | |
| DPBS w: Ca2+ and Mg2+ | PAN-Biotech | P04-35500 | |
| Trypsin EDTA | PAN-Biotech | P10-023100 | |
| TurboFect Transfection Reagent | Thermo Fisher Scientific | R0531 | |
| HEK 293T cells | DSMZ | ACC 635 | |
| Alexa Fluor 488 NHS Ester | Thermo Fisher Scientific | A20000 | |
| Rhodamine B | Sigma-Alderich | 83689-1G | |
| Plasmid DNA | Addgene | NA | See reference 12 (Dunsing et. al., MBoC 2017),for a detailed description of all plasmids |
| 6-well plate | Starlab | CC7672-7506 | |
| 35-mm glass bottom dishes | CellVis | D35-14-1.5-N | |
| Zeiss LSM780 confocal | Carl Zeiss | NA | |
| MATLAB software package | MathWorks | 2015b | |
| Neubauer cell counting chamber | Marienfeld | 640110 |
Referenzen
- Alberts, B., Johnson, A., Lewis, J., Raff, M., Roberts, K., Walter, P. . Molecular biology of the cell. , (2002).
- Tepass, U., Truong, K., Godt, D., Ikura, M., Peifer, M. Cadherins in embryonic and neural morphogenesis. Nature Reviews Molecular Cell Biology. 1 (2), 91-100 (2000).
- Harris, T. J. C., Tepass, U. Adherens junctions: from molecules to morphogenesis. Nature Reviews Molecular Cell Biology. 11 (7), 502-514 (2010).
- Hernández, J. M., Podbilewicz, B. The hallmarks of cell-cell fusion. Development. 144 (24), 4481-4495 (2017).
- Huppa, J. B., Davis, M. M. T-cell-antigen recognition and the immunological synapse. Nature Reviews Immunology. 3 (12), 973-983 (2003).
- Kaden, D., Voigt, P., Munter, L. -. M., Bobowski, K. D., Schaefer, M., Multhaup, G. Subcellular localization and dimerization of APLP1 are strikingly different from APP and APLP2. Journal of cell science. 122, 368-377 (2009).
- Yap, A. S., Michael, M., Parton, R. G. Seeing and believing: recent advances in imaging cell-cell interactions. F1000Research. 4, 273 (2015).
- Kashef, J., Franz, C. M. Quantitative methods for analyzing cell-cell adhesion in development. Developmental Biology. 401 (1), 165-174 (2015).
- Soba, P., et al. Homo- and heterodimerization of APP family members promotes intercellular adhesion. The EMBO Journal. 24 (20), 3624-3634 (2005).
- Kim, S. A., Tai, C. -. Y., Mok, L. -. P., Mosser, E. A., Schuman, E. M. Calcium-dependent dynamics of cadherin interactions at cell-cell junctions. Proceedings of the National Academy of Sciences of the United States of America. 108 (24), 9857-9862 (2011).
- Feinberg, E. H., et al. GFP Reconstitution Across Synaptic Partners (GRASP) Defines Cell Contacts and Synapses in Living Nervous Systems. Neuron. 57 (3), 353-363 (2008).
- Dunsing, V., Mayer, M., Liebsch, F., Multhaup, G., Chiantia, S. Direct evidence of amyloid precursor-like protein 1 trans interactions in cell-cell adhesion platforms investigated via fluorescence fluctuation spectroscopy. Molecular biology of the cell. 28 (25), 3609-3620 (2017).
- Schneider, F., et al. Diffusion of lipids and GPI-anchored proteins in actin-free plasma membrane vesicles measured by STED-FCS. Molecular Biology of the Cell. 28 (11), 1507-1518 (2017).
- Bacia, K., Kim, S. A., Schwille, P. Fluorescence cross-correlation spectroscopy in living cells. Nature methods. 3 (2), 83-89 (2006).
- Ries, J., Schwille, P. Studying Slow Membrane Dynamics with Continuous Wave Scanning Fluorescence Correlation Spectroscopy. Biophysical Journal. 91 (5), 1915-1924 (2006).
- Ries, J., Chiantia, S., Schwille, P. Accurate Determination of Membrane Dynamics with Line-Scan FCS. Biophysical Journal. 96 (5), 1999-2008 (2009).
- Chiantia, S., Ries, J., Schwille, P. Fluorescence correlation spectroscopy in membrane structure elucidation. Biochimica et Biophysica Acta (BBA) - Biomembranes. 1788 (1), 225-233 (2009).
- Digman, M. A., Dalal, R., Horwitz, A. F., Gratton, E. Mapping the number of molecules and brightness in the laser scanning microscope. Biophysical journal. 94 (6), 2320-2332 (2008).
- Dalal, R. B., Digman, M. A., Horwitz, A. F., Vetri, V., Gratton, E. Determination of particle number and brightness using a laser scanning confocal microscope operating in the analog mode. Microscopy research and technique. 71 (1), 69-81 (2008).
- Unruh, J. R., Gratton, E. Analysis of Molecular Concentration and Brightness from Fluorescence Fluctuation Data with an Electron Multiplied CCD Camera. Biophysical Journal. 95 (11), 5385-5398 (2008).
- Digman, M. A., Wiseman, P. W., Choi, C., Horwitz, A. R., Gratton, E. Stoichiometry of molecular complexes at adhesions in living cells. Proceedings of the National Academy of Sciences of the United States of America. 106 (7), 2170-2175 (2009).
- Hellriegel, C., Caiolfa, V. R., Corti, V., Sidenius, N., Zamai, M. Number and brightness image analysis reveals ATF-induced dimerization kinetics of uPAR in the cell membrane. The FASEB journal official publication of the Federation of American Societies for Experimental Biology. 25 (9), 2883-2897 (2011).
- Dunsing, V., Luckner, M., Zühlke, B., Petazzi, R. A., Herrmann, A., Chiantia, S. Optimal fluorescent protein tags for quantifying protein oligomerization in living cells. Scientific Reports. 8 (1), 10634 (2018).
- Chen, Y., Johnson, J., Macdonald, P., Wu, B., Mueller, J. D. Observing Protein Interactions and Their Stoichiometry in Living Cells by Brightness Analysis of Fluorescence Fluctuation Experiments. Methods in enzymology. 472, 345-363 (2010).
- . Absolute Diffusion Coefficients: Compilation of Reference Data for FCS Calibration Available from: https://www.picoquant.com/images/uploads/page/files/7353/appnote_diffusioncoeffients.pdf (2010)
- Foo, Y. H., Naredi-Rainer, N., Lamb, D. C., Ahmed, S., Wohland, T. Factors affecting the quantification of biomolecular interactions by fluorescence cross-correlation spectroscopy. Biophysical journal. 102 (5), 1174-1183 (2012).
- Baum, M., Erdel, F., Wachsmuth, M., Rippe, K. Retrieving the intracellular topology from multi-scale protein mobility mapping in living cells. Nature Communications. 5, 4494 (2014).
- Wohland, T., Rigler, R., Vogel, H. The standard deviation in fluorescence correlation spectroscopy. Biophysical journal. 80 (6), 2987-2999 (2001).
- Ries, J., et al. Automated suppression of sample-related artifacts in Fluorescence Correlation Spectroscopy. Optics Express. 18 (11), 11073 (2010).
- Ries, J., Schwille, P. New concepts for fluorescence correlation spectroscopy on membranes. Physical Chemistry Chemical Physics. 10 (24), 3487 (2008).
- Mayer, M. C., et al. Amyloid precursor-like protein 1 (APLP1) exhibits stronger zinc-dependent neuronal adhesion than amyloid precursor protein and APLP2. Journal of Neurochemistry. 137 (2), 266-276 (2016).
- Linkert, M., et al. Metadata matters: access to image data in the real world. The Journal of Cell Biology. 189 (5), 777-782 (2010).
- Trullo, A., Corti, V., Arza, E., Caiolfa, V. R., Zamai, M. Application limits and data correction in number of molecules and brightness analysis. Microscopy Research and Technique. 76 (11), 1135-1146 (2013).
- Nolan, R., et al. nandb-number and brightness in R with a novel automatic detrending algorithm. Bioinformatics. 33 (21), 3508-3510 (2017).
- Hammond, G. R. V., Sim, Y., Lagnado, L., Irvine, R. F. Reversible binding and rapid diffusion of proteins in complex with inositol lipids serves to coordinate free movement with spatial information. The Journal of cell biology. 184 (2), 297-308 (2009).
- Hendrix, J., Dekens, T., Schrimpf, W., Lamb, D. C. Arbitrary-Region Raster Image Correlation Spectroscopy. Biophysical journal. 111 (8), 1785-1796 (2016).
- Hendrix, J., et al. Live-cell observation of cytosolic HIV-1 assembly onset reveals RNA-interacting Gag oligomers. The Journal of cell biology. 210 (4), 629-646 (2015).
- Hendrix, J., Schrimpf, W., Höller, M., Lamb, D. C. Pulsed Interleaved Excitation Fluctuation Imaging. Biophysical Journal. 105 (4), 848-861 (2013).
- Honigmann, A., et al. Scanning STED-FCS reveals spatiotemporal heterogeneity of lipid interaction in the plasma membrane of living cells. Nature Communications. 5 (1), 5412 (2014).
- Chojnacki, J., et al. Envelope glycoprotein mobility on HIV-1 particles depends on the virus maturation state. Nature Communications. 8 (1), 545 (2017).
- Godin, A. G., et al. Revealing protein oligomerization and densities in situ using spatial intensity distribution analysis. Proceedings of the National Academy of Sciences of the United States of America. 108 (17), 7010-7015 (2011).
- Müller, J. D., Chen, Y., Gratton, E. Resolving Heterogeneity on the Single Molecular Level with the Photon-Counting Histogram. Biophysical Journal. 78 (1), 474-486 (2000).
- Kim, S. A., Heinze, K. G., Bacia, K., Waxham, M. N., Schwille, P. Two-Photon Cross-Correlation Analysis of Intracellular Reactions with Variable Stoichiometry. Biophysical Journal. 88 (6), 4319-4336 (2005).
- Jenkins, E., et al. Reconstitution of immune cell interactions in free-standing membranes. Journal of cell science. 132 (4), (2018).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten