Un abonnement à JoVE est nécessaire pour voir ce contenu. Connectez-vous ou commencez votre essai gratuit.
Method Article
Un dosage de spectroscopie par Fluorescence Fluctuation d’Interactions protéine-protéine à Contacts cellule-cellule
Dans cet article
Résumé
Ce protocole décrit une approche d’axée sur la spectroscopie de fluorescence fluctuation pour étudier les interactions entre protéines médiant les interactions cellule-cellule, c'est-à-dire des protéines localisées dans les jonctions cellulaires, directement dans les cellules vivantes. Nous fournissons des lignes directrices détaillées sur l’étalonnage de l’instrument, d’acquisition de données et l’analyse, y compris les corrections apportées aux sources de l’artefact possible.
Résumé
Une variété de processus biologiques implique des interactions cellule-cellule, généralement véhiculées par les protéines qui interagissent à l’interface entre les cellules voisines. D’intérêt, seulement quelques essais sont capables de sonder précisément ces interactions directement dans les cellules vivantes. Nous présentons ici un test pour mesurer la liaison des protéines exprimées à la surface des cellules voisines, aux contacts de cellules. Ce test comprend deux étapes : mélange des cellules exprimant les protéines d’intérêt fusionné à différentes protéines fluorescentes, suivie de mesures de spectroscopie de fluorescence fluctuation aux contacts de cellules à l’aide d’un laser confocal microscope à balayage. Nous démontrer la faisabilité de ce test dans un contexte biologiquement pertinent en mesurant les interactions de la protéine amyloïde de précurseurs comme 1 (APLP1) jonctions cellule-cellule. Nous fournissons des protocoles détaillés sur l’acquisition de données à l’aide de techniques axées sur la fluorescence (analyse spectroscopie de corrélation de fluorescence, nombre de corrélation croisée et l’analyse de la luminosité) et l’étalonnage de l’instrument requis. De plus, nous discutons des étapes essentielles dans l’analyse des données et la façon d’identifier et de corriger les variations de signal externe, parasites, telles que celles dues à un mouvement de photoblanchiment ou cellule.
En général, l’analyse présentée est applicable à tout homo - ou d’une interaction de protéines hétérotypiques à contacts de cellule-cellule, entre les cellules des types identiques ou différents et peut être implémentée sur un laser confocal commercial microscope à balayage. Une exigence importante est la stabilité du système, qui doit être suffisante pour sonder par diffusion dynamique des protéines d’intérêt pendant plusieurs minutes.
Introduction
Beaucoup de processus biologiques se produire sur les sites des interactions cellule-cellule, par exemple, cellules adhérence1,2,3, cellule-cellule fusion4 et reconnaissance cellulaire5. Ces événements sont particulièrement importants lors du développement des organismes pluricellulaires et pour la communication de cellule-cellule, par exemple, au cours de la réponse immunitaire. Ces processus sont généralement véhiculées par des protéines qui sont localisés à la surface, c'est-à-dire à la membrane plasmique (PM) des cellules voisines et subissent des interactions spécifiques au contact cellule-cellule qui sont précisément réglementées dans l’espace et le temps. Dans de nombreux cas, ces interactions sont directement homo - ou des interactions protéine-protéine hétérotypiques trans , mais peuvent aussi comporter des ions ou des ligands agissant comme linkers extracellulaire1. Bien que d’une importance fondamentale, il y a un manque d’essais probing ces interactions protéine-protéine spécifique directement dans l’environnement natif des cellules vivantes. Plusieurs méthodes nécessitent soit désorganisation cellulaire (par exemple, des tests biochimiques comme co-immunoprécipitation6), la fixation (par exemple, certaines des techniques de microscopie optique de Super-résolution et microscopie électronique de cellules contacts7), ou sont non spécifiques, par exemple, agrégation / adhérence dosages8,9. Pour résoudre ce problème, techniques de fluorescence ont été appliquées à l’issu de la fluorescence resonance energy transfert (FRET)10 ou fluorescence complémentation11. Toutefois, pour atteindre suffisamment petites distances entre les fluorophores, ces méthodes nécessitent des étiquettes fluorescentes du côté extracellulaire des protéines10, potentiellement interférer avec des interactions de trans .
Nous présentons ici une analyse alternative de basés sur la fluorescence pour interactions protéine-protéine à contacts cellule-cellule. Cette approche combine des approches de corrélation de fluorescence (balayage spectroscopie de corrélation de fluorescence (sFCCS), nombre de corrélation croisée et la luminosité (ccN & B)) et le mélange des cellules exprimant une construction de fusion de la protéine de intérêt, par exemple, un récepteur d’adhésion. Les récepteurs étudiés dans les deux cellules qui interagissent sont étiquetés avec deux protéines fluorescentes spectralement séparés (FPs), de l’intracellulaire (voir Figure 1 a).
Les méthodes employés reposent sur l’analyse statistique des variations de fluorescence induite par le mouvement diffusif fluorescent des protéines de fusion par le biais du volume focal d’un confocal laser scanning microscope. Plus en détail, le test des sondes la diffusion conjointe des protéines d’intérêt dans les deux voisins PMs à contacts cellule-cellule. Si les protéines subissent des interactions de trans , ces complexes trans portera des protéines fluorescentes émettant dans les deux canaux spectraux, provoquant des fluctuations de corrélation de fluorescence des deux émetteurs. En revanche, si aucune liaison se produit, les fluctuations des protéines numéros face à PMs sera indépendantes, ne causant aucune fluctuation corrélées. L’acquisition peut être effectuée de deux façons : 1) sFCCS repose sur une analyse en forme de ligne au travers le contact de cellule-cellule et sondes efficacement les interactions dans un endroit situé dans la région de contact. Grâce à une analyse temporelle des variations de fluorescence, sFCCS renseigne aussi dynamique, c'est-à-dire, les coefficients de diffusion des complexes protéiques ; 2) ccN & B repose sur une analyse d’un d’une séquence d’images acquises dans les régions de contact cellule-cellule. Il a une capacité à sonder et le long de l’ensemble des interactions de carte contact région (dans un plan focal), mais ne fournit pas d’informations sur la dynamique. Les deux méthodes peuvent être combinées avec une analyse de la luminosité moléculaire, c'est-à-dire le signal de fluorescence moyenne émise dans l’unité de temps par simple diffusion complexes de protéines et, ainsi, fournir des estimations de la stoechiométrie des complexes protéiques au contacts de cellule-cellule.
Dans cet article, nous fournissons des protocoles détaillés pour la préparation des échantillons, d’étalonnage des instruments, d’acquisition de données et d’analyse effectuer l’essai présenté sur un laser confocal commercial microscope à balayage. Les expériences peuvent être effectuées sur n’importe quel instrument équipé de comptage de photons ou détecteurs analogiques et un objectif à grande ouverture numérique. Plus loin, nous discutons des étapes critiques du protocole et fournissent des systèmes de correction pour plusieurs processus causant des fluctuations artéfactuelles signal, par exemple, bruit de détecteur, photoblanchiment ou cellule mouvement. Développé à l’origine pour sonder les interactions entre cellules adhérentes, l’essai peut être modifié pour les cellules en suspension, ou adaptée aux systèmes à membrane modèle, par exemple, les vésicules unilamellaires géant (GUVs) ou le plasma géant vésicules de la membrane (GPMVs), ce qui permet la quantification des interactions dans des environnements différents lipides ou en l’absence d’un cytosquelette organisé12,13.
Spectroscopie de corrélation de fluorescence de balayage est une version modifiée de la spectroscopie de corrélation de fluorescence14 et a été spécialement conçu pour sonder la lente par diffusion dynamique de membranes lipidiques15. Il repose sur une acquisition de balayage ligne perpendiculaire à la PM contenant les protéines fluorescentes d’intérêt. Pour sonder les interactions entre les deux espèces de protéines marquées différemment, l’acquisition est effectuée en deux canaux spectraux en utilisant deux lignes laser et deux fenêtres de détection de fluorophores spectralement séparés. En raison de la dynamique de diffusion lente des protéines dans la MP (D≤ ~ 1 µm2/s), une mesure cross-talk-gratuit peut être effectuée en alternance le régime de l’excitation de la ligne à la ligne15. L’analyse commence par : 1) l’alignement algorithme correction de mouvement latéral cellulaire basé sur block-wise avec une moyenne de ~ 1000 lignes, 2) Détermination de la position avec fluorescence maximum signal, c'est-à-dire la PM position dans chaque bloc et 3) shifting de tous les blocs d’une commune origine12,15, séparément pour chaque voie. Puis, une sélection automatique des pixels correspondant à la PM est exécutée en sélectionnant la zone centrale d’un ajustement gaussien de la somme de toutes les lignes alignées (c.-à-d., Centre ± 2.5σ). Intégration du signal de chaque ligne donne la série temporelle de fluorescence membranaire f (t) dans chaque canal (g = vert canal, r = canal rouge). Notez que la taille du pixel doit être assez petit par exemple, < 200 nm, pour reconstruire la forme du point de fonction d’étalement et de trouver son centre, correspondant à la position de la MP. En présence de photoblanchiment substantiel, les séries chronologiques de fluorescence dans chaque canal peuvent être modélisés par une fonction exponentielle double et puis corrigés avec la formule suivante :16
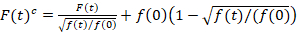 . (1)
. (1)
Il est important de noter que cette formule corrige efficacement tant les amplitudes et les heures de diffusion obtenues par l’analyse de corrélation de f (t)c, par rapport aux estimations de paramètre qui seraient obtenues à partir du non corrigée f (t). Ensuite, les fonctions auto - et la corrélation croisée (ACFs / CCP) de la fluorescence des signaux sont calculées :
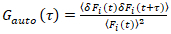 , (2).
, (2).
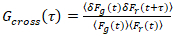 , (3).
, (3).
où δFj’ai = Fi(t) - 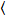 Fi(t)
Fi(t)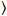 et i = g, r.
et i = g, r.
Un modèle de diffusion bidimensionnelle est ensuite ajusté à toutes les fonctions de corrélation (CFs) :
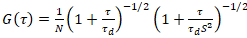 . (4)
. (4)
Ici, N désigne le nombre de protéines fluorescentes dans le volume de l’observation et τd le temps de diffusion pour chaque canal. Ce modèle prend en compte que dans le cadre expérimental décrit, diffusion des protéines dans la MP se produit dans le plan x-z, contrairement à la configuration courante de corrélation de fluorescence spectroscopy (FCS) expériences sur les membranes de palpage diffusion dans le plan x-y du confocal volume17. La taille w0 et le facteur de structure S, décrivant l’allongement wz du volume focal en z, S = wz/w0, sont obtenues à partir d’une mesure de calibration de FCS point effectuée avec colorants spectralement similaires et les mêmes paramètres optiques à l’aide de valeurs déjà disponibles pour le coefficient de diffusion Dcolorant:
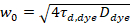 , (5).
, (5).
où τd, colorant est le temps de diffusion moyenne mesurée des molécules de colorant, obtenu à partir d’un modèle pour la diffusion en trois dimensions des données, compte tenu des transitions de compte d’une fraction T de toutes les molécules de N à un état triplet avec une constante de temps ττ:
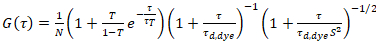 . (6)
. (6)
Enfin, les coefficients de diffusion (D), les valeurs de luminosité moléculaire (ε) et la corrélation croisée relative des données sFCCS (rel.cc.) sont calculées comme suit :
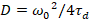 , (7).
, (7).
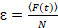 , (8).
, (8).
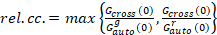 , (9).
, (9).
où GCroix(0) est l’amplitude de la fonction de corrélation croisée et 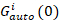 est l’amplitude de la fonction d’autocorrélation dans le canal du je-th.
est l’amplitude de la fonction d’autocorrélation dans le canal du je-th.
Cette définition de la relative corrélation croisée, c'est-à-dire à l’aide de max au lieu de signifier l’équation 9, tient compte du fait que le nombre maximal de complexes des deux espèces de protéines présents à des concentrations différentes est limité par la espèces présentes dans un nombre inférieur.
Luminosité et nombre de corrélation croisée repose sur une analyse de l’instant de l’intensité de fluorescence pour chaque pixel d’une image de pile a acquis au fil du temps à une position fixe dans l’échantillon, généralement composé de ~ 100-200 cadres, avec deux spectrale des canaux () g = vert canal, r = canal rouge). D’après la moyenne temporelle 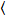 je
je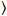 et la variance
et la variance 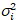 , la luminosité moléculaire εi et numéro nj’ai sont calculées dans chaque pixel et de canaux spectraux (j’ai = g, r)18:
, la luminosité moléculaire εi et numéro nj’ai sont calculées dans chaque pixel et de canaux spectraux (j’ai = g, r)18:
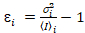 , (10).
, (10).
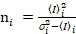 . (11)
. (11)
Il est important de noter que les équations données s’appliquent pour le cas idéal d’un vrai détecteur de comptage de photons. Pour les systèmes de détection analogique, les équations suivantes s’appliquent à19,20:
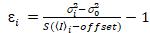 , (12).
, (12).
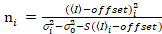 . (13)
. (13)
Ici, S est le facteur de conversion entre les photons détectés et les comtes numériques enregistrées, 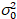 est le bruit de lecture et offset fait référence à l’offset d’intensité de détecteur. Généralement, ces quantités doivent être étalonnées, pour n’importe quel type de détecteur, basée sur la mesure de la variance du détecteur en fonction de l’intensité d’éclairage constant19, par exemple, une surface métallique réfléchissante ou solution de colorant séchées. Le décalage peut être déterminé en mesurant le taux de comptage pour un échantillon sans lumière d’excitation. En effectuant une régression linéaire de la variance associée à détecteur
est le bruit de lecture et offset fait référence à l’offset d’intensité de détecteur. Généralement, ces quantités doivent être étalonnées, pour n’importe quel type de détecteur, basée sur la mesure de la variance du détecteur en fonction de l’intensité d’éclairage constant19, par exemple, une surface métallique réfléchissante ou solution de colorant séchées. Le décalage peut être déterminé en mesurant le taux de comptage pour un échantillon sans lumière d’excitation. En effectuant une régression linéaire de la variance associée à détecteur 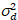 contre le tracé de l’intensité (I), S et
contre le tracé de l’intensité (I), S et 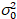 peut être déterminée19:
peut être déterminée19:
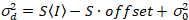 . (14)
. (14)
Enfin, la luminosité de la corrélation croisée est calculée dans chaque pixel et est définie en général comme21
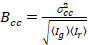 , (15).
, (15).
où 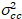 est la Croix et la variance
est la Croix et la variance 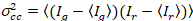 .
.
Pour filtrer les fluctuations à long terme, toutes les ccN & calculs B sont effectuées suivant un wagon couvert, filtrage, indépendamment pour chaque pixel22. En bref, ni, εj’ai (j’ai = g, r) et Bcc sont calculées en glissant des segments de p. ex. cadres de 8-15. Les valeurs ainsi obtenues peuvent être ensuite la moyenne pour obtenir le pixel final les valeurs de nombre et de la luminosité.
Analyse de stoechiométrie
Afin d’estimer la stoechiométrie des complexes protéiques à contacts cellule-cellule, la luminosité moléculaire peut être analysée séparément pour chaque voie spectral pour le sFCCS ou le ccN & B data. Dans sFCCS, une valeur de luminosité est obtenue par la mesure de chaque canal. Dans la ccN & B, on obtient un histogramme de luminosité des pixels situés correspondant au contact cellule-cellule et la valeur moyenne (ou médiane) peut servir de luminosité représentative pour la mesure. En effectuant la même analyse sur une référence monomère, toutes les valeurs de luminosité peuvent être normalisées pour obtenir directement l’État oligomère moyenne des complexes protéine détectée. À ce stade, il est important de corriger la présence de FPs non fluorescent pouvant entraîner une sous-estimation de l’État oligomère. Ceci est généralement effectuée par la mesure de la luminosité d’un homo-dimère référence protéine23,24 utilisant une couleur SCTS-m ou le numéro et la luminosité (N & B).
Protocole
1. préparation : Cellules mélange dosage
Remarque : Le protocole suivant décrit la procédure de mélange pour les cellules adhérentes. Il peut être modifié pour les cellules cultivées en suspension.
- Un nombre adéquat de cellules sur une plaque 6 puits, par exemple, 800 000 cellules HEK 293 t (comptées avec un Neubauer chambre de comptage), un jour avant la transfection des semences. Le nombre peut être modifié selon le délai entre l’amorçage et de transfection et ajusté pour les autres types de cellules. Pour réaliser une expérience de base (c.-à-d., les protéines d’intérêt et de contrôle négatif), préparer au moins 4 puits. Des cellules de culture à 37 ° C, 5 % de CO2 dans un milieu Eagle modifié (DMEM) de Dulbecco, additionné de sérum de veau fœtal (10 %) et de la L-glutamine (1 %).
- Transfecter cellules selon les instructions du fabricant (voir la Table des matières).
- Pour réaliser une expérience de base, transfecter, en puits séparés, plasmides pour la protéine d’intérêt fondue à un « vert » (p. ex., monomère meilleure protéine fluorescente verte (mEGFP), ou une protéine fluorescente jaune (mEYFP)) ou « rouge » (p. ex., mCherry, ou mCardinal) protéine fluorescente.
Remarque : Dans ce protocole, nous nous concentrons sur APLP1-mEYFP et APLP1-mCardinal12et le contrôle négatif correspondant, par exemple, myristoylated-palmitoylées-mEYFP (myr-palm-mEYFP) et - mCardinal (myr-palm-mCardinal)12. En général, de 200 ng - 1 µg d’ADN plasmidique suffisent. Efficacité de transfection élevé augmente les chances de trouver des cellules « rouges » et « verts » en contact. Modifier la quantité de réactif de plasmides et de transfection pour optimiser l’efficacité de transfection. Critique : Confluence de la cellule devrait être d’environ 70 % lorsque transfectants des cellules. Si les cellules sont trop anastomosé, diminue l’efficacité de transfection. Si les cellules ne sont pas assez confluentes, transfection et le mélange peuvent induire des contraintes et prévenir plusieurs cellules d’une bonne fixation après mélange.
- Pour réaliser une expérience de base, transfecter, en puits séparés, plasmides pour la protéine d’intérêt fondue à un « vert » (p. ex., monomère meilleure protéine fluorescente verte (mEGFP), ou une protéine fluorescente jaune (mEYFP)) ou « rouge » (p. ex., mCherry, ou mCardinal) protéine fluorescente.
- Effectuer le mélange ~4 ± 2 h après la transfection de cellules.
- Enlever le milieu de croissance et laver chaque bien doucement avec 1 mL de PBS additionné de Mg2 + et Ca2 +. Ensuite, retirez le PBS. (Critique) Drop PBS sur bord bien pour éviter le détachement des cellules pendant le lavage.
- Ajouter goutte-à-goutte ~ 50 µL trypsine tétraacétique (EDTA) une solution d’acide dans chaque puits pour faciliter le détachement des cellules. Incuber à 37 ° C pendant 2 min. par la suite, secouez doucement la plaque 6 puits latéralement pour détacher les cellules.
Remarque : Les durées d’incubation prolongée peuvent être requises pour certains types de cellules. - Ajouter 950 µL de milieu de culture dans chaque puits et remettre en suspension les cellules en pipettant également, quelques fois monter et descendre, détachant ainsi toutes les cellules du fond du puits. (Critique) S’assurer que les cellules sont correctement remis en suspension et détachés de l’autre en vérifiant visuellement l’absence d’agrégats de grandes cellules après la remise en suspension. Dans le cas contraire nombreux contacts « rouges »-« rouges » ou « verts »-« vertes » seront obtenus après mélange.
- Transférer la solution de la cellule d’un puits (protéine d’intérêt ou de témoin négatif) dans le correspondant, c'est-à-dire, « rouge » (par exemple, APLP1-mCardinal transfectée) « vert » (par exemple, APLP1-mEYFP transfectée) des cellules. Mélanger doucement pipetage quelques fois de haut en bas. Puis, les cellules mixtes sur plats en bas 35 mm verre (1 mL de solution de cellules mixtes par plat), ainsi que 1 mL de milieu de culture et de la culture de semences graines cellules pour une autre journée à 37 ° C, 5 % de CO2.

Figure 1 . Des flux de travail expérimental et représentation schématique du balayage, spectroscopie de corrélation de fluorescence et d’analyse de nombre et de la luminosité de la corrélation croisée à contacts cellule-cellule. (A) schéma de préparation des échantillons : deux populations de cellules transfectées par la protéine d’intérêt (p. ex., APLP1), fusionnée à deux protéines fluorescentes spectralement distinctes (par exemple, mEYFP et mCardinal) sont mélangées après la transfection. Contacts des cellules transfectées différemment sont sélectionnés dans les expériences de microscopie. Pour éviter toute interférence avec les domaines de liaison extracellulaire, la protéine fluorescente doit être fondue jusqu’au terminus intracellulaire de la protéine d’intérêt. (B) mesures de balayage SCCC (sFCCS) sont effectué perpendiculairement au contact cellule-cellule dans deux canaux spectraux (canal 1, verte et le canal 2, rouge). Lignes de balayage (représentées par kymographs) sont alignés et additionnés de pixels de la membrane. Puis, ACFs CCFs sont calculés et les traces d’intensité Fi(t). ACFs sont représentés en rouge et vert. CCF est représentée en bleu. (C) la corrélation croisée N & B (ccN & B) acquisition résulte dans une pile d’image en trois dimensions (x-y-temps). Un retour sur investissement est sélectionné autour du contact cellule-cellule. Puis le canal et la corrélation croisée de luminosité (ε1, ε2et Bcc) les valeurs sont calculées à chaque pixel de contact cellule-cellule. Les résultats sont ensuite visualisées comme les histogrammes, mise en commun de tous les pixels sélectionnés. S’il vous plaît cliquez ici pour visionner une version agrandie de cette figure.
2. préparation de l’échantillon : Contrôle positif pour des expériences de corrélation croisée et la construction de l’Homo-dimère pour l’analyse de la luminosité
- 600 000 cellules HEK 293 t, a compté avec une cellule comptant chambre, sur les plats en bas 35 mm verre un jour avant la transfection des semences. Les cellules à 37 ° C, 5 % de CO2 dans le milieu DMEM complet de la culture (voir étape 1.1) pour un autre jour.
- Transfecter cellules avec ~ 250 ng d’ADN plasmidique conformément aux instructions du fabricant. Pour le contrôle positif de corrélation croisée, utilisez un plasmide codant une protéine fluorescente ancrées à membrane hétéro-dimères, par exemple, myr-palm-mCherry-mEGFP ou myr-palm-mCardinal-mEYFP12 correspondant à la FPF de la protéine d’intérêt. Pour l’étalonnage de la luminosité, utiliser des plasmides codant une membrane ancrées monomère FP et homo-dimère correspondant à la FPF fondue à la protéine d’intérêt, par exemple, myr-palm-mEYFP et myr-palm-mEYFP-mEYFP pour calibrer l’analyse de la luminosité de APLP1-mEYFP12.
- La culture de cellules à 37 ° C, 5 % de CO2 dans le milieu DMEM complet (Voir l’étape 1.1) pour un autre jour.
3. confocal Laser Scanning Microscopy : Configuration et calibrage Volume Focal
Remarque : Le protocole suivant est écrit pour les expériences réalisées avec mEGFP/mEYFP et mCherry/mCardinal sur le laser scanning microscope confocal utilisée dans cette étude. La configuration optique, les paramètres du logiciel (lignes laser, miroirs dichroïques, filtres) et choix de colorants d’étalonnage peuvent être modifiées pour les autres configurations FPs et microscope.
- Allumez le microscope et le laser au moins une heure avant l’expérience pour assurer la stabilité de laser et d’équilibrage de la température.
- Préparer 100-200 µL des solutions appropriées colorant fluorescent soluble dans l’eau (voir la Table des matières pour des exemples) dans l’eau ou PBS pour calibrer le volume focal, avec des concentrations dans la gamme de 10 à 50 nM.
- Placer les solutions de teinture sur un plat de fond verre propre 35 mm #1.5, c'est-à-dire ayant une épaisseur de 0,16-0,19 mm.
NOTE : Idéalement, utilisez des plats avec couvercle en verre haute performance ayant une tolérance de faible épaisseur, par exemple, 0,170 ± 0,005 mm, ce qui permet une correction de bague collier optimal (étape 3.6). Il est important d’utiliser le même type de plat tel qu’utilisé plus tard pour les expériences suivantes. - Placer la capsule contenant la solution de colorant directement sur l’objectif (de préférence, immersion dans l’eau, avec NA 1.2) afin d’assurer la mise au point dans la solution. Vous pouvez également placer la capsule sur le porte-échantillon et le focus dans l’échantillon (par exemple, 10 à 20 µm au-dessus du fond du plat).
Remarque : Nous ne recommandons pas l’utilisation d’objectifs de pétrole en raison du mauvais signal obtenu lorsque nous nous concentrons profond dans des échantillons aqueux. - Le chemin d’accès d’excitation et d’émission, par exemple, de choisir le laser à 488 nm, un miroir dichroïque de 488/561 nm, détection fenêtre 499-552 nm et une taille de trou d’épingle de 1 unité aérée (UA). Assurez-vous que la taille du trou d’épingle est le même que celui qui sera utilisé dans les mesures de corrélation croisée.
- Ajustez la position de trou d’épingle (ajustement de trou d’épingle) et la bague collier objective afin de maximiser les taux de comptage. Dans ce but, tournez la bague collier jusqu'à ce que le taux de comptage maximale est détecté.
NOTE : La correction de bague collier représente l’épaisseur spécifique du couvercle verre utilisé. Maximiser le taux de comptage, i.e., recueillant le plus grand nombre de photons par molécule que possible, est crucial pour maximiser le rapport signal-bruit (RSB) des mesures. - Effectuer une série de mesures (par exemple, 6 mesures à différents endroits, composées chacune de 15 répétitions de 10 s, c'est-à-dire 2,5 min Durée totale, échantillonné avec temps de pause de 1 µs ou moins) de la FCS point à la même puissance de laser que celui utilisé dans la corrélation croisée mesures (généralement ~ 1 %, c'est-à-dire, ~ 1-2 µW).
- Ajuster un modèle de diffusion en trois dimensions dont une contribution de triplet (équation 6) aux données.
Remarque : En règle générale, les temps de diffusion obtenues sont environ 30 µs et le facteur de structure est d’environ 4-8. - Calculer la taille w0 du temps de diffusion moyenne mesurées et les valeurs publiées pour le coefficient de diffusion du colorant utilisé à température ambiante25 selon l’équation 5. Valeurs typiques sont de 200 à 250 nm.
- Répétez la routine de calibration (étapes 3,4-3,9) avec un colorant fluorescent différent pour un deuxième canal de détection si nécessaire (par exemple, 561 nm excitation et détection entre 570 nm et 695 nm). Gardez la position du trou d’épingle et la taille telle qu’elle a été définie pour la première chaîne de détection.
- Calculer la luminosité moléculaire (équation 8) par les mesures d’étalonnage et stocker les valeurs obtenues.
Remarque : Les valeurs typiques pour le programme d’installation utilisé sont ~8-10 kHz/molécule (MOL) pour 1,8 puissance d’excitation de µW à 488 nm. Plus faibles que les valeurs usuelles pouvant indiquer la poussière sur l’objectif, le désalignement de la configuration ou une sortie laser réduit. Vérifier et stocker les puissances de sortie du laser à l’objectif régulièrement à l’aide d’un wattmètre. Pour comparaison des configurations différentes, luminosité moléculaire normalisée par la puissance de laser d’excitation est le paramètre plus significatif pour évaluer la performance de microscope.
4. analyse spectroscopie de corrélation de Fluorescence : Acquisition
Remarque : Le protocole suivant est écrit pour les expériences réalisées avec mEGFP/mEYFP (« vert ») et mCherry/mCardinal (« rouge ») sur le microscope confocal à balayage laser utilisé dans cette étude. La configuration optique et les paramètres du logiciel (lignes laser, miroirs dichroïques, filtres) peuvent être différents pour les autres configurations FPs ou microscope.
- Mettre en place le chemin optique, par exemple, 488 nm et une excitation 561 nm et un miroir dichroïque de 488/561 nm, sténopé sur 1 pour une excitation 488 nm. Pour éviter l’interférence spectrale, sélectionnez deux pistes séparées pour exciter et de détecter les mEGFP/mEYFP (488 excitation nm, canal vert) et mCherry / mCardinal (561 excitation nm, canal rouge) dans l’ordre et sélectionnez le commutateur suit chaque ligne. Pour la détection, utilisez les filtres appropriés pour les deux canaux, par exemple, 499-552 nm dans le canal vert et 570-695 nm dans le canal rouge.
- Si excitation alternée n’est pas possible, utilisez les paramètres de filtre approprié pour le canal rouge afin de minimiser la diaphonie spectrale (c'est-à-dire détecter fluorescence mCherry/mCardinal pas en dessous de 600 nm). Cela peut réduire la quantité de photons détectés dans le canal rouge et ainsi réduire la SNR.
- Placer la capsule contenant les cellules mixtes sur le porte-échantillon. Attendre au moins 10 min pour assurer l’équilibrage de la température et pour réduire la dérive de la mise au point.
- Mettre l’accent sur les cellules à l’aide de transmission lumineuse dans le menu Rechercher .
- Vous recherchez une paire de « rouge » et une cellule « verte » en contact avec l’autre. Pour la corrélation croisée positive ou homo-dimère, réglage de la luminosité (voir chapitre 2), recherche sur une cellule isolée émettant de fluorescence dans les deux canaux ou le signal de l’homo-dimère respectifs PM.
NOTE : (Critique) Minimisez échantillon l’exposition tout en cherchant des cellules éviter le blanchiment préalable, qui peut réduire la corrélation croisée26. Par conséquent, scan à la vitesse de balayage plus rapide et les pouvoirs de laser faible. Pour éviter la saturation du détecteur tout en imagerie exprimant fortement les cellules, une recherche en mode d’intégration. Toutefois, pour minimiser l’exposition, la numérisation à plus faibles puissances de laser est possible en mode comptage de photons . - Sélectionnez une perpendiculaire de chemin d’accès de scan de contact cellule-cellule (ou heures d’une seule cellule pour le contrôle de luminosité de corrélation croisée ou homo-dimère positif) en utilisant le bouton rogner , comme illustré dans les Figures 1 b et 2 a.
Remarque : Certains microscopes anciens ne permettent pas de scan arbitraire directions. Dans ce cas, des contacts de cellules avec une orientation perpendiculaire à la direction de l’analyse doivent être situé. -
Zoom pour atteindre une taille de pixel de 50-200 nm et sélectionnez ligne en Mode Scan. Taille de l’image la valeur 128 × 1 pixel.
Remarque : Taille de pixel typique est de 160 nm, correspondant à une longueur de balayage d’environ 20 µm. - La valeur vitesse de numérisation jusqu'à la valeur maximale autorisée, par exemple, 472,73 µs par ligne.
Remarque : Pour un mécanisme de rechange excitation, cela correspond à 954,45 µs de temps de scan, soit ~ 1000 scans/s sur la configuration utilisée. La vitesse de balayage peut être ajustée selon le coefficient de diffusion de la protéine d’intérêt. Pour les protéines ancrées à membrane, diffusion typique temps est autour de 10-20 m, le temps d’acquisition doit être au moins dix fois plus petit que les temps de diffusion. À basse vitesse de balayage peut induire photoblanchiment plus fort et nécessitent des puissances d’éclairage inférieurs. Alternativement, on peut imposer une pause, par exemple, 5 ms, entre chaque balayage diffusant très lentement complexes à l’aide de délai dans le sous-menu de Séries chronologiques . - Choisissez les puissances de laser approprié, par exemple, ~ 1-2 µW à 488 nm et ~ 5-10 µW pour une excitation 561 nm.
Remarque : Des puissances supérieures de laser améliorer SNR, mais augmentent le photoblanchiment. Par conséquent, puissances de laser doivent être choisis tels que photoblanchiment est inférieure à 50 % du tarif compteur initial. - Sur les 100 000-500 000 Cycles .
Remarque : Le nombre de balayages, savoir, la durée de la mesure, peuvent varier : mesure plus longs amélioreront SNR et peuvent être plus appropriés pour diffuser lentement les molécules, cependant, le mouvement des cellules et le photoblanchiment limitent la mesure maximale temps. Les données présentées ici ont été régulièrement acquises pour ~ 3-6 min, soit 200 000-400 000 ligne scans. - La valeur des détecteurs mode de comptage de photons . Appuyez sur Expérience de démarrage pour démarrer l’acquisition. Répétez les étapes 4,5-4.11 pour mesurer une autre cellule.
Remarque : Il est recommandé de mesurer les 10-15 cellules par exemple à des niveaux d’expression différents. (Critique) Éviter la saturation de détecteur à des niveaux de haute expression. Le taux de comptage maximale ne doit pas dépasser environ 1 MHz. - Si l’analyse de luminosité est effectuée afin de déterminer les États oligomériques, effectuer des homo-dimère luminosité étalonnage mesures suivant étapes mis à jour le 4.1-4.11 : mesurer chaque protéine fluorescente homo-dimère séparément (dans les cellules isolées, établie à l’aide Protocole 2 de l’article) et effectuer des mesures que dans un seul canal spectral.
5. analyse spectroscopie de corrélation croisée de Fluorescence : Analyse des données
Remarque : Le protocole suivant fait suite à une mise en œuvre de la procédure d’analyse décrite en détail dans précédents articles12,15. Le code du logiciel est disponible sur demande aux auteurs.
- Exporter les fichiers de données brutes (p. ex., CZI) vers une image RVB TIFF au format de données brutes. Ce fichier contiendra une kymographes avec le vert et les données des canaux rouge, dans le chenal appelé G et R de l’image, respectivement.
- Importer le fichier TIFF avec le logiciel d’analyse appropriée et procéder à l’analyse.
Remarque : Les étapes suivantes (étapes de 5,3 à 5,7) sont appliquées séparément pour chaque canal : - Aligner les lignes en effectuant une moyenne de temps segment-wise ou mobiles avec des blocs de lignes 500-1000. Déterminer la position de la membrane, c'est-à-dire la position de pixel avec le taux de comptage maximale, dans chaque bloc. Mettez tous les blocs à la même position latérale. Cette procédure corrige pour un déplacement latéral du contact cellule-cellule, par exemple, en raison du mouvement de la cellule.
- Résumer toutes les lignes alignées le long de l’axe du temps et correspondait au profil d’intensité moyenne à l’aide d’une fonction gaussienne. En présence d’importants antécédents intracellulaire, utiliser une gaussienne, plus une fonction sigmoïde. Définir les pixels correspondant à la membrane comme tous les pixels dans la ±2.5σ de la position de la membrane et les résumer l’intensité de ces pixels dans chaque ligne, l’obtention d’une valeur de signal de fluorescence unique pour chaque point dans le temps (c'est-à-dire, pour chaque analyse de ligne).
- Le cas échéant (p. ex., fond > 10 % du signal membrane), appliquer une correction de fond en soustrayant l’intensité moyenne de pixel dans le cytoplasme multiplié par 2.5σ (en unités de pixels) de la fluorescence de la membrane, en blocs de 1000 lignes. Éviter des vésicules intracellulaires lumineuses lors de la sélection des pixels de l’arrière-plan.
- Si on observe le photoblanchiment, appliquer une correction de blanchiment. Par conséquent, adapter la série de temps de fluorescence membranaire avec une fonction exponentielle double et applique la correction appropriée de l’équation 1 formule,16.
Remarque : Vous pouvez également Fourier spectre basé correction régimes peuvent être appliquée27. (Critique) Si photoblanchiment est présent mais pas corrigée pour, les SADC peuvent être sévèrement déformée et estimations des paramètres peuvent être fortement biaisées (par exemple, voir la Figure 5E). - Calculer l’ACFs et cadres de coopération selon les équations 2 et 3 en utilisant, par exemple, un algorithme de multiple-tau28. Pour améliorer la fiabilité de l’analyse et éviter les artéfacts, effectuer les calculs pour les 10-20 segments égaux de la mesure totale. Inspecter les séries chronologiques de fluorescence et le CFs dans chaque segment et supprimer des segments clairement déformées (voir exemples dans la Figure 4 a- 4D). Moyenne de tous les segments sans distorsion.
Remarque : Cette procédure peut être automatisée pour éviter un biais subjectif pour les données de29. Pour les mesures très instables, ayant beaucoup de courts segments peut être utile. Toutefois, la longueur d’un segment soit encore au moins trois ordres de grandeur au-dessus de la durée de diffusion pour éviter statistique sous-échantillonnage erreurs29,30,17. - Ajuster un modèle de diffusion bidimensionnelle, équation 4, aux SADC obtenu. Par conséquent, fixer le facteur de structure à la valeur obtenue en mesure de calibration (protocole, article 3). La précision de l’ajustement peut être améliorée en effectuant un ajustement pondéré en utilisant les poids statistiques de chaque point de données obtenues de l’algorithme de tau multiples.
- Calculer le coefficient de diffusion à l’aide de la taille calibrée selon l’équation 7.
- Calculer la luminosité moléculaire en divisant l’intensité de fluorescence moyenne dans chaque canal par le nombre de particules, équation 8 correspondant. Normaliser la valeur de luminosité déterminé dans chaque canal par la luminosité moyenne de la référence monomère correspondante pour obtenir l’État oligomère, prenant en compte non fluorescent FPs23. À cette fin, déterminer les valeurs de luminosité moyenne homo-dimère unicolore analyse pour calculer la fraction non fluorescent FPs23.
- Calculer la corrélation croisée relative selon l’équation 9.
6. corrélation croisée nombre et luminosité : étalonnage du détecteur
Remarque : Le protocole suivant fournit une orientation générale concernant comment calibrer le système de détection. Cette procédure est obligatoire pour les systèmes de détection analogique, mais il n’est pas strictement nécessaire lorsqu’on utilise des détecteurs de comptage de photons vrai.
- Faire sécher des solutions appropriées de colorant soluble dans l’eau (voir Table des matières par exemple) sur un plat de fond de verre de 35 mm. Définissez le chemin optique en conséquence, c'est-à-dire, 488 ou 561 nm excitation et détection à 499-552 nm ou 570-695 nm, respectivement.
NOTE : En tant qu’alternativement, une surface métallique réfléchissante peut être utilisée au lieu de solutions de colorant séchées en plaçant le morceau de métal directement sur le dessus de l’objectif. - Mesurer une couleur N & B dans les régions avec des concentrations de teintes différentes ou à des puissances différentes. Par conséquent, utiliser Zoom pour atteindre une taille de pixel de 300 nm, vitesse de numérisation de définir temporisation pixel appropriées, par exemple, 25 µs et Cycles à 100-200 cadres.
- Set de détecteurs de comptage de photons (ou mode analogique si les mesures sont réalisées avec détection analogique) et appuyez sur Expérience de démarrage pour démarrer l’acquisition. Effectuer la mesure à zéro puissance d’excitation afin de déterminer le décalage de l’intensité.
- Tracer la variance pixel en fonction de l’intensité du pixel pour tous les pixels mesurées et effectuer un ajustement linéaire de ces données. Déterminer S comme la pente de l’ajustement linéaire. Calculer le bruit de lecture de l’ordonnée à l’origine, à l’aide de S et le décalage d’intensité déterminée selon l’équation 14.
7. la corrélation croisée nombre et luminosité : Acquisition
- Suivez les étapes 4.1 à 4.4 du protocole d’acquisition de la sFCCS.
- Permet de sélectionner une image de 512 × 128 pixels autour d’un contact de cellule-cellule (ou isolés PM pour contrôle de luminosité de homo-dimère) récolte et Zoom pour atteindre une taille de pixel de 50 à 100 nm.
- Utilisez la vitesse de numérisation pour définir temporisation pixel appropriées, par exemple, 6,3 µs.
Remarque : En N & B, le temps de pause de pixel devrait être beaucoup plus petit que le temps de diffusion de la protéine d’intérêt. Si un schéma d’excitation remplaçant est choisi, par ex., changement des pistes chaque ligne, le temps entre les deux pistes devrait être inférieur à l’heure de diffusion de la protéine d’intérêt. Dans le cas contraire la corrélation croisée détectable est réduite. - La valeur de Cycles à 100-200 cadres.
Remarque : Un nombre plus élevé de cadre améliorera le SNR, cependant, les mouvement cellulaire peut limiter la durée de la mesure totale. Le temps de scan par image devrait être beaucoup plus élevé que le temps de diffusion de la protéine d’intérêt. Sinon, la luminosité apparente est réduite, c'est-à-dire, les particules semblent être immobile. Complexes de diffus très lentement, impose une pause, par exemple, 2 s, entre les deux images à l’aide d’intervalle dans le sous-menu de Séries chronologiques . - Définir les puissances de laser des valeurs appropriées (valeurs typiques sont ~ 1-2 µW à 488 nm et ~ 5-10 µW pour une excitation 561 nm).
Remarque : La cheftaine laser conduit à luminosité plus élevée et SNR amélioré, mais aussi amélioré photoblanchiment. Puissances de laser devraient être assez élevés pour atteindre une luminosité détectée d’au moins 1 ~ kHz/MOL mais maintenu assez bas pour éviter plus de photoblanchiment de 10 à 20 %. Pour mEGFP/mEYFP ou mCherry/mCardinal, à moins de 10 % le photoblanchiment sont généralement obtenus. - La valeur de détecteurs de comptage de photons (ou mode analogique si les mesures sont réalisées avec détection analogique). Appuyez sur Expérience de démarrage pour démarrer l’acquisition.
- Évaluer le taux de comptage de photons. Si les taux de comptage en pixels contact cellule-cellule dépassent 1 MHz, réduire la puissance du laser ou sélectionner des cellules avec des niveaux d’expression. Répétez les étapes 7.2 à 7.7. pour mesurer la prochaine paire de cellules. Il est recommandé de mesurer les 10-15 cellules par expérience à des niveaux d’expression différents.
- Si l’analyse de luminosité est réalisée afin de quantifier l’oligomérisation, effectuer des homo-dimère luminosité étalonnage mesures suivant étapes mis à jour le 7.1 à 7.7 : mesurer chaque protéine fluorescente homo-dimère séparément (dans les cellules isolées, établie à l’aide Protocole 2 de l’article) et effectuer des mesures que dans un seul canal spectral.
8. corrélation croisée nombre et luminosité : analyse des données
Remarque : Le protocole suivant fait suite à une analyse décrite précédemment procédure12,31. Le code du logiciel est disponible auprès des auteurs sur demande.
- Importer les données brutes (par exemple, les fichiers CZI peuvent être importés en utilisant le paquet de32 Bioformats). Moyenne de tous les cadres et sélectionnez une région d’intérêt (ROI) autour du contact cellule-cellule.
- Effectuez une image alignement algorithme33, par exemple, en maximisant la corrélation spatiale entre les ROIs dans les trames suivantes pour des traductions arbitraires latérales, en moyenne sur les deux canaux. Cette procédure permettra de corriger pour le mouvement latéral des cellules.
- Appliquer un filtre de boxcar22 afin de réduire les fluctuations de longue durée de vie étrangères, provenant, par exemple, le mouvement cellulaire résiduel ou de blanchiment de fond. Par ailleurs, une méthode de redressement peut être appliquée pour corriger la photobleaching34.
Remarque : Si aucune analyse segment-wise ou dissociation est appliqué, l’éclat apparent peut être largement surestimé.- Définir des segments coulissants de, par exemple, 8 et 15 cadres (par exemple, 1 à 8, 2 à 9 et ainsi de suite) et calculer les valeurs de luminosité de canal et de la corrélation croisée selon les équations 10, 11 et 15 un dans chaque segment. Si les détecteurs ne sont pas vrai photon comptant des détecteurs, prendre les paramètres du détecteur étalonné en compte lors du calcul de la luminosité, c'est-à-dire utilisation équations 12 et 13 au lieu de cela.
Remarque : Calcul des valeurs de luminosité dans les segments de 8 à 15 images conduit à une sous-estimation de 10 à 20 % de la luminosité absolue et une surestimation de 10 à 20 % du nombre de particules. Néanmoins, les ratios de luminosité (par exemple, le dimère de luminosité monomère) ne sont pas affectés, aussi longtemps que la longueur du segment est maintenue constante tout au long de l’analyse (données non présentées). L’erreur statistique pour une longueur de segment donné peut être déterminée au moyen de simulations et ainsi corrigé. - Moyenne des valeurs de luminosité obtenue pixel-wise sur tous les segments. Dans cette étape, on peut retirer le maximum et minimum 5 % des valeurs de luminosité de segment par rapport à la moyenne ou exclure segments qui montrent une déformation claire dans l’intensité, en raison, par exemple, une vésicule intracellulaire ou agrégat transitoirement présent dans ces pixels.
- Définir des segments coulissants de, par exemple, 8 et 15 cadres (par exemple, 1 à 8, 2 à 9 et ainsi de suite) et calculer les valeurs de luminosité de canal et de la corrélation croisée selon les équations 10, 11 et 15 un dans chaque segment. Si les détecteurs ne sont pas vrai photon comptant des détecteurs, prendre les paramètres du détecteur étalonné en compte lors du calcul de la luminosité, c'est-à-dire utilisation équations 12 et 13 au lieu de cela.
- Tracer les valeurs de luminosité des pixels en fonction de l’intensité du pixel, puis sélectionnez la population de pixels qui correspond au contact cellule-cellule. Pixels de fond auront des valeurs de très faible intensité. À ce stade, ré-évaluer le taux de comptage maximale. Exclure les pixels avec le taux de comptage au-dessus de 1 MHz à prévenir les effets de carambolage.
- Créer des histogrammes de luminosité channel et la corrélation croisée de cellule-cellule sélectionnée pixels contact et obtenir les valeurs de luminosité moyenne ROI. Normaliser la valeur de luminosité du canal moyen par la luminosité moyenne de la référence monomère correspondante pour obtenir l’État oligomère, prenant en compte non fluorescent FPs23. Par conséquent, déterminer des valeurs de luminosité moyenne homo-dimère d’unicolore analyse pour calculer la fraction non fluorescent FPs23.
- À titre d’illustration, tracer des cartes de luminosité de canal et de la corrélation croisée.
Résultats
Un premier test pour le test d’interaction protéine-protéine, c.-à-d., mélange de cellules exprimant des protéines fluorescentes spectralement distinctes suivies sFCCS/ccN & mesures B (Figure 1), doit être effectué sur les protéines qui ne sont pas censés interagir au contact cellule-cellule (par exemple, un contrôle négatif). Par conséquent, les cellules HEK 293 t exprimant myristoylated-palmitoylées-mEYFP (myr-palm-mEYFP) ou...
Discussion
La procédure expérimentale décrite ici permet l’étude des protéines des interactions trans à contacts de cellule-cellule, employant des techniques de spectroscopie par fluorescence fluctuation, à savoir sFCCS et ccN & B. Ces méthodes impliquent une analyse statistique des variations de fluorescence émise par deux FPs spectralement séparés fusionnés pour les protéines d’intérêt au contact de deux cellules voisines, chacune exprimant l’un ou l’autre protéine de fusion. La présence de compl...
Déclarations de divulgation
Les auteurs n’ont rien à divulguer.
Remerciements
Ce travail a été partiellement soutenu par la Deutsche Forschungsgemeinschaft (DFG) accorder 254850309. Les auteurs remercient Madlen Luckner pour une lecture critique du manuscrit.
matériels
| Name | Company | Catalog Number | Comments |
| DMEM growth medium | PAN-Biotech | P04-01548 | |
| DPBS w/o: Ca2+ and Mg2+ | PAN-Biotech | P04-36500 | |
| DPBS w: Ca2+ and Mg2+ | PAN-Biotech | P04-35500 | |
| Trypsin EDTA | PAN-Biotech | P10-023100 | |
| TurboFect Transfection Reagent | Thermo Fisher Scientific | R0531 | |
| HEK 293T cells | DSMZ | ACC 635 | |
| Alexa Fluor 488 NHS Ester | Thermo Fisher Scientific | A20000 | |
| Rhodamine B | Sigma-Alderich | 83689-1G | |
| Plasmid DNA | Addgene | NA | See reference 12 (Dunsing et. al., MBoC 2017),for a detailed description of all plasmids |
| 6-well plate | Starlab | CC7672-7506 | |
| 35-mm glass bottom dishes | CellVis | D35-14-1.5-N | |
| Zeiss LSM780 confocal | Carl Zeiss | NA | |
| MATLAB software package | MathWorks | 2015b | |
| Neubauer cell counting chamber | Marienfeld | 640110 |
Références
- Alberts, B., Johnson, A., Lewis, J., Raff, M., Roberts, K., Walter, P. . Molecular biology of the cell. , (2002).
- Tepass, U., Truong, K., Godt, D., Ikura, M., Peifer, M. Cadherins in embryonic and neural morphogenesis. Nature Reviews Molecular Cell Biology. 1 (2), 91-100 (2000).
- Harris, T. J. C., Tepass, U. Adherens junctions: from molecules to morphogenesis. Nature Reviews Molecular Cell Biology. 11 (7), 502-514 (2010).
- Hernández, J. M., Podbilewicz, B. The hallmarks of cell-cell fusion. Development. 144 (24), 4481-4495 (2017).
- Huppa, J. B., Davis, M. M. T-cell-antigen recognition and the immunological synapse. Nature Reviews Immunology. 3 (12), 973-983 (2003).
- Kaden, D., Voigt, P., Munter, L. -. M., Bobowski, K. D., Schaefer, M., Multhaup, G. Subcellular localization and dimerization of APLP1 are strikingly different from APP and APLP2. Journal of cell science. 122, 368-377 (2009).
- Yap, A. S., Michael, M., Parton, R. G. Seeing and believing: recent advances in imaging cell-cell interactions. F1000Research. 4, 273 (2015).
- Kashef, J., Franz, C. M. Quantitative methods for analyzing cell-cell adhesion in development. Developmental Biology. 401 (1), 165-174 (2015).
- Soba, P., et al. Homo- and heterodimerization of APP family members promotes intercellular adhesion. The EMBO Journal. 24 (20), 3624-3634 (2005).
- Kim, S. A., Tai, C. -. Y., Mok, L. -. P., Mosser, E. A., Schuman, E. M. Calcium-dependent dynamics of cadherin interactions at cell-cell junctions. Proceedings of the National Academy of Sciences of the United States of America. 108 (24), 9857-9862 (2011).
- Feinberg, E. H., et al. GFP Reconstitution Across Synaptic Partners (GRASP) Defines Cell Contacts and Synapses in Living Nervous Systems. Neuron. 57 (3), 353-363 (2008).
- Dunsing, V., Mayer, M., Liebsch, F., Multhaup, G., Chiantia, S. Direct evidence of amyloid precursor-like protein 1 trans interactions in cell-cell adhesion platforms investigated via fluorescence fluctuation spectroscopy. Molecular biology of the cell. 28 (25), 3609-3620 (2017).
- Schneider, F., et al. Diffusion of lipids and GPI-anchored proteins in actin-free plasma membrane vesicles measured by STED-FCS. Molecular Biology of the Cell. 28 (11), 1507-1518 (2017).
- Bacia, K., Kim, S. A., Schwille, P. Fluorescence cross-correlation spectroscopy in living cells. Nature methods. 3 (2), 83-89 (2006).
- Ries, J., Schwille, P. Studying Slow Membrane Dynamics with Continuous Wave Scanning Fluorescence Correlation Spectroscopy. Biophysical Journal. 91 (5), 1915-1924 (2006).
- Ries, J., Chiantia, S., Schwille, P. Accurate Determination of Membrane Dynamics with Line-Scan FCS. Biophysical Journal. 96 (5), 1999-2008 (2009).
- Chiantia, S., Ries, J., Schwille, P. Fluorescence correlation spectroscopy in membrane structure elucidation. Biochimica et Biophysica Acta (BBA) - Biomembranes. 1788 (1), 225-233 (2009).
- Digman, M. A., Dalal, R., Horwitz, A. F., Gratton, E. Mapping the number of molecules and brightness in the laser scanning microscope. Biophysical journal. 94 (6), 2320-2332 (2008).
- Dalal, R. B., Digman, M. A., Horwitz, A. F., Vetri, V., Gratton, E. Determination of particle number and brightness using a laser scanning confocal microscope operating in the analog mode. Microscopy research and technique. 71 (1), 69-81 (2008).
- Unruh, J. R., Gratton, E. Analysis of Molecular Concentration and Brightness from Fluorescence Fluctuation Data with an Electron Multiplied CCD Camera. Biophysical Journal. 95 (11), 5385-5398 (2008).
- Digman, M. A., Wiseman, P. W., Choi, C., Horwitz, A. R., Gratton, E. Stoichiometry of molecular complexes at adhesions in living cells. Proceedings of the National Academy of Sciences of the United States of America. 106 (7), 2170-2175 (2009).
- Hellriegel, C., Caiolfa, V. R., Corti, V., Sidenius, N., Zamai, M. Number and brightness image analysis reveals ATF-induced dimerization kinetics of uPAR in the cell membrane. The FASEB journal official publication of the Federation of American Societies for Experimental Biology. 25 (9), 2883-2897 (2011).
- Dunsing, V., Luckner, M., Zühlke, B., Petazzi, R. A., Herrmann, A., Chiantia, S. Optimal fluorescent protein tags for quantifying protein oligomerization in living cells. Scientific Reports. 8 (1), 10634 (2018).
- Chen, Y., Johnson, J., Macdonald, P., Wu, B., Mueller, J. D. Observing Protein Interactions and Their Stoichiometry in Living Cells by Brightness Analysis of Fluorescence Fluctuation Experiments. Methods in enzymology. 472, 345-363 (2010).
- . Absolute Diffusion Coefficients: Compilation of Reference Data for FCS Calibration Available from: https://www.picoquant.com/images/uploads/page/files/7353/appnote_diffusioncoeffients.pdf (2010)
- Foo, Y. H., Naredi-Rainer, N., Lamb, D. C., Ahmed, S., Wohland, T. Factors affecting the quantification of biomolecular interactions by fluorescence cross-correlation spectroscopy. Biophysical journal. 102 (5), 1174-1183 (2012).
- Baum, M., Erdel, F., Wachsmuth, M., Rippe, K. Retrieving the intracellular topology from multi-scale protein mobility mapping in living cells. Nature Communications. 5, 4494 (2014).
- Wohland, T., Rigler, R., Vogel, H. The standard deviation in fluorescence correlation spectroscopy. Biophysical journal. 80 (6), 2987-2999 (2001).
- Ries, J., et al. Automated suppression of sample-related artifacts in Fluorescence Correlation Spectroscopy. Optics Express. 18 (11), 11073 (2010).
- Ries, J., Schwille, P. New concepts for fluorescence correlation spectroscopy on membranes. Physical Chemistry Chemical Physics. 10 (24), 3487 (2008).
- Mayer, M. C., et al. Amyloid precursor-like protein 1 (APLP1) exhibits stronger zinc-dependent neuronal adhesion than amyloid precursor protein and APLP2. Journal of Neurochemistry. 137 (2), 266-276 (2016).
- Linkert, M., et al. Metadata matters: access to image data in the real world. The Journal of Cell Biology. 189 (5), 777-782 (2010).
- Trullo, A., Corti, V., Arza, E., Caiolfa, V. R., Zamai, M. Application limits and data correction in number of molecules and brightness analysis. Microscopy Research and Technique. 76 (11), 1135-1146 (2013).
- Nolan, R., et al. nandb-number and brightness in R with a novel automatic detrending algorithm. Bioinformatics. 33 (21), 3508-3510 (2017).
- Hammond, G. R. V., Sim, Y., Lagnado, L., Irvine, R. F. Reversible binding and rapid diffusion of proteins in complex with inositol lipids serves to coordinate free movement with spatial information. The Journal of cell biology. 184 (2), 297-308 (2009).
- Hendrix, J., Dekens, T., Schrimpf, W., Lamb, D. C. Arbitrary-Region Raster Image Correlation Spectroscopy. Biophysical journal. 111 (8), 1785-1796 (2016).
- Hendrix, J., et al. Live-cell observation of cytosolic HIV-1 assembly onset reveals RNA-interacting Gag oligomers. The Journal of cell biology. 210 (4), 629-646 (2015).
- Hendrix, J., Schrimpf, W., Höller, M., Lamb, D. C. Pulsed Interleaved Excitation Fluctuation Imaging. Biophysical Journal. 105 (4), 848-861 (2013).
- Honigmann, A., et al. Scanning STED-FCS reveals spatiotemporal heterogeneity of lipid interaction in the plasma membrane of living cells. Nature Communications. 5 (1), 5412 (2014).
- Chojnacki, J., et al. Envelope glycoprotein mobility on HIV-1 particles depends on the virus maturation state. Nature Communications. 8 (1), 545 (2017).
- Godin, A. G., et al. Revealing protein oligomerization and densities in situ using spatial intensity distribution analysis. Proceedings of the National Academy of Sciences of the United States of America. 108 (17), 7010-7015 (2011).
- Müller, J. D., Chen, Y., Gratton, E. Resolving Heterogeneity on the Single Molecular Level with the Photon-Counting Histogram. Biophysical Journal. 78 (1), 474-486 (2000).
- Kim, S. A., Heinze, K. G., Bacia, K., Waxham, M. N., Schwille, P. Two-Photon Cross-Correlation Analysis of Intracellular Reactions with Variable Stoichiometry. Biophysical Journal. 88 (6), 4319-4336 (2005).
- Jenkins, E., et al. Reconstitution of immune cell interactions in free-standing membranes. Journal of cell science. 132 (4), (2018).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon