Method Article
結合されたヌクレオチドおよび線虫のタンパク質抽出
要約
変動が減少し、再現性を向上させるための解釈を容易にするための努力で同じサンプルからの蛋白質、DNA、RNA の隔離のためのプロトコルを紹介します。
要約
単一の生物的サンプルについては、の茄多を保持して、今同時に分子の処理と異なる条件の間の変更の複数のレベルの全体像をキャプチャするいくつかの高分子を調査するの一般的。このプロトコルは、DNA、RNA を分離する手法を提案して線虫導入されたときこれらの biomolecules の複製から切り離されているバリエーションを削除する線虫の同じサンプルからの蛋白質が、最終的に同様に扱われる異なるサンプル。核酸とタンパク質は、その後沈殿物、洗濯、それぞれの可溶化と酸のためチオシアン酸-フェノール-クロロホルム抽出法を使用して線虫から抽出されます。マツ材線虫病と大人の動物に良いタンパク質分離結果での HeLa 細胞の 3 系統から 1 つのサンプルからの蛋白質、DNA、RNA の分離を成功させるを紹介します。また、線虫からためチオシアン酸-フェノール-クロロホルム抽出タンパク質はタンパク質の従来の RIPA の抽出と比較して免疫ブロットによって観測された拡張の検出可能なレベルでの大きい蛋白質の解像度を向上します。
ここに示すメソッドは、プロテオーム、トランスクリプトームの探査用 multiomic アプローチを使用してサンプルを調査するときに便利です。分子シグナリングは、基になる複雑な生物学的現象が相補的なレベルで発生すると考えられるために、multiomics を同時に評価する技術が訴えています。しかし、ますますの mRNA のレベルの変更を行うタンパク質レベルで同じ変更を常に反映しないことと、コレクションの時間が概日リズムの規則のコンテキストで関連しているを参照してくださいに共通となっています。(Intrasample) 同じサンプル内で異なる内容を試金するときにこのメソッドがビジュアルサーボイングのバリエーションすべてを削除します
概要
疾患特性1、大きなデータ セットを処理するとき、Multiomics、ゲノム、プロテオーム、トランスクリプトーム、エピゲノム、マイクロバイ、または lipidome などのオミックスの組み合わせを使用して分析のアプローチはますます人気となっています。 2。証拠は、シングル「青梅」へのアプローチを制限する (Rotroff と Motsinger reif によって1によって再検討されました) 不完全な分子分析を提供することを示しています。ハイスループット技術を使用して画面を実行するときに特に大きなデータ セットが生成されるが、完全な絵を描くために、または最も関連性の高いターゲットを識別するために、multiomic のアプローチをお勧め。しかし、multiomics のアプローチを使って、mRNA および蛋白質レベル3,4,5,6間の不一致の頻繁な観察があります。特に、mRNA および蛋白質 - サイド バイ サイド トランスクリプトームおよびプロテオーム解析 RNA シーケンス (RNAseq) と液体クロマトグラフィー-タンデム質量分析法 (MS/LC-MS)、それぞれ、使用は頻繁にから同様に扱われたサンプルから得られる異なる複製3,4,5,6の条件可能性があります同じ間の変化を紹介します。Harvald ら実行エレガントな長寿の重要な転写因子を持たないhlh 30変異ワームのワームの比較トランスクリプトーム ・ プロテオーム解析 (WT) を野生型のc. の elegans飢餓時間コースの研究7. ノートの RNA および蛋白質は、同じサンプルからそうではない、同じ条件複製から収穫されました。彼らの調査結果を示す mRNA のレベルと各時点でのタンパク質レベルの低相関 (r = 0.628 に 0.559)。実際には、彼らのヒートマップ形成 4 つのクラスター: mRNA の大きな減少をしていたクラスター レベル対応するタンパク質レベルのほとんど、あるいは全くの減少が, クラスター II は、ほとんど、あるいは全く増加 mRNA レベルが蛋白質のレベルの増加で, クラスター III は、mRNA の増加レベルが蛋白質のレベル、およびクラスター IV の減少の mRNA のレベルの増加、タンパク質レベル4のだけ微妙な変化があった。さらに、同じ条件のサンプルを正確に同じ時間で収集されませんの場合このビジュアルサーボイングのバリエーションを導入する可能性があります。たとえば、mRNA および蛋白質概日周期、9の8,日までの時間や、光9; にc. の elegansのより具体的には、露出によって変動します。これらの体内のタンパク質の発現は、遺伝子発現誘導10後 8 h まで遅れる可能性があります。それにもかかわらず、この観察の有病率は限りませんが間違っています。実際、これは有益であることを証明するかもしれない。タンパクと mRNA の形成と分解の間一定の動的状態では。さらに、安定性を高めるか、劣化11を誘導するために、蛋白質 posttranslationally しばしば変更されます。例えば、そのユビキチン化状態は活性化やプロテアソームまたは劣化12リソソームにターゲットのいずれか可能性があります。また、Rna は、転写、転写後のステージ13で遺伝子発現の調節に重要な役割を再生します。したがって、問題は、これら線虫研究において矛盾が本当であることを確認するための変数を制限する方法です。
ここでは、同じサンプルから異なる高分子の解析によりビジュアルサーボイングの変数を削除する方法を提案する.このプロトコルの目的は、一貫して再現性を向上させる変化を削減する努力の DNA、RNA および蛋白質c. の elegans (ワームとも呼ばれる) の 1 つのサンプルからを分離する方法を提供し、解釈を容易にすることです。同じサンプルを使用しての追加の利点があります貴重な限られたサンプル、成長し、維持、しにくい菌株を得るなどの横断的分析を促進するサンプル収集中に時間とリソースの節約intrasample mRNA および蛋白質レベル変動による高分子の差動制御に洞察力。
このメソッドは、遺伝子発現と蛋白分子処理の複数のレベルのより包括的な評価を可能にするワームの 1 つのサンプルからの評価に適しています。ためチオシアン酸-フェノール-クロロホルム (GTCp) 試薬14RNA を分離する一般的に使用される化学物質は、その後沈殿物、洗濯、それぞれの可溶化と、ワームから核酸やタンパク質の抽出に使用されます。このプロトコルは、さまざまなプロトコル15,16 線虫に焦点を当てて設計のマイナーな変更とのコンパイル、次の HeLa 細胞のペレットから DNA、タンパク質、RNA よりも正常に分離した、同じ手順を実行します。ここでテストしていませんが、このプロトコルは17,18組織と同様に動作する可能性があります。
プロトコル
注:各高分子の沈殿物のステップは同時に行う洗浄に続いて順次実行されます。ただし、それは RNA を隔離推奨それは本質的に安定していないので、最初。
1. サンプル コレクション
- 適切な成長条件1910 cm プレートあたり 1,000 のワームの卵をシードします。72 h 20 ° C で孵化させなさい。
注:上記の19として卵を収集するために卵を持った大人を漂白剤します。 - 約 5 mL の M9 バッファーを洗浄してチューブに 1,000 大人のワームを収集します。
注:M9 バッファーは、滅菌水19の硫酸マグネシウム 1 mM 22 mM リン酸-、カリウム、塩化ナトリウム 102 mM 35 mM ナトリウム リン酸水素で構成されます。 - 1 分、1,000 x gでワームを遠心分離機、上澄みを廃棄し、M9 バッファーの 1 つの mL を小球形にされたワームを 1.5 mL 遠心チューブに転送します。
- 再び 1 分 845 × gで遠心、上清のほとんどを破棄します。4 h の最小-80 ° C で小球形にされたワームを格納します。
注:冷凍餌を使用して新鮮なペレットよりも高い利回りを生成します。ワーム キューティクルをクラックするドライアイスの液体窒素または 95% エタノールを使用して凍結/解凍サイクルのシリーズをお勧めします新鮮なペレットを使用している場合。ペレットに M9 の少量を残して凍結時にキューティクルを破るのに役立ちます。
2. ヌクレオチドおよび蛋白質の隔離
- 解凍のペレットから任意の上清を除去し、冷たい GTCp 試薬 1 mL を追加します。上下にピペッティングでよく混ぜます。10 分間氷の上、サンプルを置き、それを逆さま回すことによって時折まぜます。
注意:フェノールは神経毒性、腐食性、毒性の高いを化学火傷や失明を引き起こす可能性があります。
注:報告されたボリュームを出発原料として最大 5,000 の成虫を使いました。各ボリュームは、GTCp 試薬 1 mL の使用に基づいています。大きいサンプルを使用する場合は、スケール アップする必要があります。 - ワームや GTCp のソリューション、冷たいクロロホルム 200 μ L を追加します。指の間チューブを保持、積極的に振る 15 s. 休暇のそれ 3 分の室温 (RT) で。
注意:クロロホルムの毒性刺激は、皮膚の傷と、他の器官をターゲットとした害を引き起こす可能性があります、可能な発癌物質。 - 4 ° C で 15 分間 13,500 x gでチューブを遠心分離します。
注:このスピンした後 3 つの層で形成: 脂質や DNA を含む小さな、曇りの相間、ピンクの有機相下部クリア水相トップ。 - 新しい RNase フリー 1.5 mL の微量遠心チューブ (管 A) (として 2.5 の手順で詳細な) アルコール沈殿によって RNA を分離し新しいチューブ (残りのペレットからピンク層 (有機相) を移動する明確なトップ層 (水相) マイクロ ピペットを使用して、移動します。チューブ B) と氷の上に置きます。
注:ピンク有機相は、DNA および蛋白質はこのサンプルからの分離まで-80 ° C で凍結することができます。大きいサンプル水溶液と有機相の間の厚い白い層が生成されます。この画分には DNA が含まれています。このレイヤーは、他の段階を乱すことがなく削除することができます、そう、別の管 (管 B2) でそれを置きます。 -
とおりの RNA を隔離します。
- チューブ A でクリア水相からの 100% イソプロピル アルコール液 500 μ L の RNA の沈殿物します。室温 10 分間インキュベートします。4 ° C で 10 分間 13,500 × gで遠心管 A、
注:RNA の小さな白いペレットが管の下部に表示されます。 - 上清の大半をデカントします。針で 1 mL 注射器で残りの部分を削除し、上澄みを廃棄します。
注:針のサイズが重要でないです。ただし、大きい針のゲージはペレットを邪魔しないようにより詳細に制御を提供します。マイクロ ピペットは、針が利用できない場合に使用できます。 - 75% エタノール 1 mL をチューブ、ペレットを洗浄する A に追加します。4 ° C で 5 分間 5,300 × gでチューブをスピンします。
- 2.5.2 の手順で説明されているようにデカントして針で 1 mL 注射器を使用ペレットから上澄みを除去し、それを破棄します。
- ペレットを聞かせて 5-10 分の風乾が overdry それをさせてください。RNase フリー水の 50 μ L を使用して、それは完全に透明になる前に RNA のペレットを再構築します。
注:これは 1,000-3,000 のワームのための適切な開始ボリュームが、それはどのくらいの原料が使用されたに応じて調整する必要があります。 - 55-60 ° c 10 分で溶解するペレットを孵化させなさい。
- 濃度と純度分光光度計を使用して測定します。260 で吸光度を記録 RNA 濃度、不純物を識別するために 230 280 nm nm。
- 汚染物を除去、フェノール残基や DNA などカラム精製またはその後エタノールの洗浄および DNase 処理する RNA をクリーンアップします。
注:この時点で、RNA RNAseq 分析のために送信する準備ができているまたは RT qPCR (詳細についてはセクション 3.1 を参照) に使用する cDNA を作るに使用することができます。RNA は、さらに使用するまで-80 ° C で保存できます。
- チューブ A でクリア水相からの 100% イソプロピル アルコール液 500 μ L の RNA の沈殿物します。室温 10 分間インキュベートします。4 ° C で 10 分間 13,500 × gで遠心管 A、
-
とおり DNA を分離します。
- チューブ B のピンクの有機相及び管内 B2、サンプルからある場合はインバージョンによる 100% のエタノールとミックスの 300 μ L を追加することによって、DNA を沈殿させます。2-3 分間常温、尿管を残します。
- 管 B、B2 375 x gペレット DNA への 4 ° C で 5 分間遠心します。
- 新しい 2 mL 管 (管 C) にそれを注ぐことによってチューブ B、B2 から上澄みを移動し、後続の蛋白質の隔離のための氷にそれを残します。チューブ B または B2 1 mL の 0.1 M クエン酸ナトリウム 4 ° C で 5 分間 375 x gで 30 分遠沈管 B、B2 の 10% エタノールによる DNA の餌を洗う
注:ナトリウムは、クエン酸ナトリウム、エタノール、低速遠心分離によって除去される不純物を許可する DNA をより容易に沈殿物作る DNA のバックボーンにバインドします。 - 2.6.3 の手順で説明するように、洗浄手順を繰り返します。75% のエタノールの 1.5 mL にペレットを再懸濁し、upending によって時折混合 20 分間常温放置します。
- 4 ° C で 5 分間 375 × gで遠心チューブ B と B2
- 上澄みを廃棄し、5-10 分間乾燥すること。
- 8 mM 水酸化ナトリウムを 150 μ L でペレットを溶解します。必要に応じて、HEPES を目的の pH に調整します。4 ° C で 5 分間 375 x gでサンプルをスピンします。
- マイクロ ピペットを使用して、新しいチューブに上清 (DNA) を移動します。濃度を測定し、分光光度計を使用して純度を決定します。260 で吸光度を記録 DNA 濃度の不純物を識別するために 230 と 280 nm nm。
-
とおりタンパク質を分離します。
- 蛋白質の沈殿物にチューブ C、数回反転によるミックスのピンクの上澄みに 100% イソプロパノールの最大 1.5 mL を追加し、室温で 10 分間インキュベートします。
- 4 ° C で 10 分間 13,500 x gで C 管を遠心分離します。
- 上澄みを廃棄し、4 ° C で 5 分間 5,300 × gで RT。 遠心管 C で 20 分の 95% エタノールで 0.3 M グアニジン塩酸 2 mL にペレットを洗う
- 2.7.3 の手順で説明するように洗浄手順を繰り返します 2 倍。
- 蛋白ペレットを新しい 1.5 mL チューブ (管 C2) に移動し、95% のエタノールの最大 1.5 mL を追加します。渦と RT で 20 分待ちます。
- チューブ C2 4 ° C で 5 分間 5,300 × gで遠心分離します。上澄みを廃棄し、乾燥したディゾルブで 10 分の 60 分の 50 ° c 5 %sds の 300 μ L でペレット ペレット。
注: 長いインキュベーション時間は蛋白ペレットを完全に溶解する必要があります。過去には、ペレットは、品質を損なうことがなく 6 h までの培養されています。 - チューブ C2 13,500 x g 17 ° C で 10 分間遠心します。上清を新しいチューブに移動します。
- 洗剤と互換性のある好ましい蛋白質定量アッセイを使用して濃度を測定します。
注:蛋白質は SDS のページおよび西部にしみが付くことで使用する準備ができて。それは、将来の使用のための-20 ° C で保存できます。LC ・ MS/MS などの他の技術の洗剤を透析する必要があります。
3. mRNA および蛋白質レベルの評価
-
RT qPCR
- 1 の逆のトランスクリプションによって cDNA を準備次たちのプロトコルを使用して RNA の ng: 逆のトランスクリプション (46 ° C で 20 分)、そして RT 不活性化 (95 ° c 1 分) (25 ° c 5 分) をプライミングします。
- 96 ウェル プレートの個々 の井戸への各 cDNA サンプルの 1: 100 希釈液 100 μ L の追加によって cDNA のストック プレートを作る。ないのテンプレート/水専用の井戸、希釈したサンプル 1:25 から 1: 400 (すべての分析サンプルからプールされた cdna 由来) などの適切なコントロールは、標準曲線分析各遺伝子のプライマー効率を確立するを許可します。
注:この形式は、96 ウェル microdispenser、ウェル ウェル分注バリエーションの軽減、RT qPCR プレートにプレートのすべてのサンプルを転送するを可能にすると使用できます。マルチ チャンネル ピペットを適切なマスター ミックス (例えば、SYBR + プライマー) それぞれにもその後、追加できます。全体的にみて、このアプローチは手ピペッティング各サンプルでは、このようにさらに可変性を減らすときの誤差を低減します。 - ストック プレートから RT qPCR プレートの井戸に cDNA の 3 μ L を追加します。
- 転送し、逆にプライマー、サンプルあたり最大 7 μ L を水 1 x スーパーミックス DNA 間をインターカ レート シアニン色素の 5 μ を含むを含むプライマーの各セットのマスター ミックスを作る。RT qPCR 板の適切な井戸を 7 μ L を追加し、ミックスに軽く揺り動かします。
注:20の結果を正規化するための参照として使用するハウスキーピング遺伝子を測定するサンプルが含まれます。 - 使用されているプライマーに適した Rt-qpcr プロトコルを使用してプレートを実行します。
-
西部のしみ
注:西部のしみの詳細については、マフムードと陽の21を参照してください。- 2 倍の塩化物イオンとサンプル バッファーと 95 ° C, 10 分で沸騰の等量と蛋白質の 20 μ g を組み合わせることにより SDS ページのサンプルを準備します。
- 4-15% トリス-グリシンのゲルにサンプルをロードし、150 V、1 時間または染料前ゲルの下端に到達するまでにそれらを実行します。
- セミドライの転送装置で 30 分間 25 V でニトロセルロース膜にゲルからタンパク質を転送します。
- Ponceau S 染色で膜の汚損によって適切な転送を確認します。
- トリス緩衝生理食塩水 (TBS) 0.01% ポリソルベート 20 (TBS-T) と膜から汚れを徹底的に洗います。
- 室温 1 時間 TBS t 5% 乳脂肪の膜をブロックします。
- 供給者の勧告に従って適切な希釈で一次抗体で膜を孵化させなさい。4 ° C でロッカーに一晩置いておきます。
- 洗浄、3 x、5 分ごとに、TBS"T"で
- 供給者の勧告に従って適切な希釈で適切な二次抗体の膜を孵化させなさい。室温 1 時間ロッカーにそれを残す
- 洗浄、3 x、5 分ごとに、TBS"T"で
- イメージして推奨される方法を使用して西部のしみを定量化します。
結果
RNA、DNA および蛋白質のサンプルを分析し、RNA および蛋白質の分離を使用して代表的な結果はここに表示されます。さらに、我々 は収穫共通 RIPA 溶解法22GTCp メソッドを使用したタンパク質サンプルを比較します。私たちの実験では、各 WT (N2) ワームと 2 つの長寿命変異体ワーム、食べる 2 林産物 123,24の 4 つの独立した複製を使用しました。
RNA と DNA の量と質
RNA 水の 50 μ L で再停止されるときの濃度範囲 0.2-2 μ g/μ L、約 3,000 の大人を使用してワームの原料として。純度を示す吸光度の比率は、汚染せず GTCp コンポーネント、フェノールまたはグアニジン塩酸などに、RNA を正しく抽出を示す A260/A280 の 2.2、A260/A230 (表 1) に 2.5 1.9 2.0 からであった。
DNA の抽出に成功しましたが、品質は変数。DNA 濃度 150 μ L の水酸化ナトリウム 0.04 〜 1.1 μ g/μ L で再停止されるとき。DNA を正しく抽出を示す 〜 1.8 2.1 A260/A280 と 0.9 〜 1.6 A260/A230 (表 1) の吸光度比ただし、いくつかのサンプルは GTCp コンポーネント、フェノールまたはグアニジン塩酸塩のように汚染されていた。DNA の隔離が必要な場合、中間層 GTCp 相分離から分離するときに注意が必要し、より多くの洗浄必要があります。
選択したターゲットの RT qPCR
各ワームのひずみの 4 つの独立したサンプルからの RNA の隔離は、成功したし、その後 RT qPCR シアニン系感光色素を含むスーパー ミックスを使用して実行で RNAseq 分析のため送信されました。RT qPCR による分析の目標は、WT ワームと比較して両方の突然変異体の一般的な最も特異的に調整された遺伝子に基づく大規模な RNAseq データセットから選ばれました。F07C4.1225、相同物人間ニューロリギン 3、アイソ フォーム b に選択した共有誘導目標であった。我々 はまた両方の突然変異体、 mrp 126、食べる 2ワームで亢進、林産物 1ワームでダウンレギュ レートが追加の分析のための特異的に調整された遺伝子が含まれています。Rt-qpcr; を介してmrp 1の発現変動が確認されました。しかし、林産物 1ワーム RNAseq 解析による予測で F07C4.12 アップレギュレーション Rt-qpcr (図 1) で見られなかった。
ワームの検証抗体と追加のターゲットをも調べた。Hadwiger ら27で特徴づけられるいくつかのオルガネラ マーカーが含まれます。これらのマーカーの数いた突然変異体のワームの mRNA レベルで発現します。図 1からわかるように、 lmp 128 dlg 129いた食べる 2ワーム; で亢進hsp 4lmp 1されhsp 7030、 30林産物 1ワーム; で亢進hsp 6031とpas 732いた食べる 2ワームにダウンレギュ レートhsp 60 ・ tac 133は林産物 1ワームでダウンレギュ レートだった
同時対 RIPA 蛋白質の準備
GTCp 方法前述のように効率とタンパク質の品質を確認するには、我々 は RIPA 溶解22のより伝統的な方法を使用して収集したタンパク質を比較しました。開始材料は、すなわち、大人のみ (約 10,000 ワーム) または成虫、幼虫および卵 (約 130,000 ワーム) の混合された人口の同量を使用して GTCp で収集された全面的な量少なかったイコール エンド ボリューム (テーブル 1 で再停止されるとき);ただし、大人専用の母集団から抽出された混合された人口からよりも成功。さらに、蛋白質の品質は同様に、GTCp 抽出タンパク質は、Coomassie 染色図2 で示すように、総蛋白の等しい読み込み時に大きい蛋白質のより良い解決を示したにもかかわらず。確かに、図 3の西部のしみを用いてターゲットは大抵の同じようなレベルを示したしかし、75 kDa より大きい蛋白質 GTCp 抽出タンパク質 (図 3 aは、右車線に比べて RIPA 抽出タンパク質の低レベルを示した図 3 b、黄色バー)。
ここに示す抽出法も、哺乳類セルライン hela 細胞で評価した.参照、RIPA 600 万 HeLa 細胞 (〜 25 μ L ペレット) の小型ペレットから抽出したタンパク質の量はワーム (、~ 75 μ L 小型ペレット) の 130,000 の混合された人口から抽出に匹敵するまたは 10,000 から抽出されるものの約半分表 1に見られるように大人のワーム (、~ 100 μ L 重力定住ペレット)。(表 1) の蛋白質の抽出の効率と大きい蛋白質 (図 2) の解像度 GTCp HeLa 細胞でのタンパク質の RIPA 抽出と比較してこの手法はの大人の人口でより良い動作を示唆で減少していたことを示すワーム。GTCp 抽出 hela 細胞蛋白質から蛋白質ペレットの不完全な可溶化哺乳類細胞におけるこのメソッドの制限があります。
RT qPCR 用いてイムノブロッティング ターゲット
次に、mRNA のレベルが蛋白質のレベルと相関かどうかを決定する RT qPCR によるテスト遺伝子産物のタンパク質レベルを調べた。多くのターゲットの変更平均の mRNA 発現と蛋白質の平均発現変動の相関図 3からわかるように、ただし、いくつかのタンパク質レベルは、mRNA のレベルの変更を反映しなかった。多くの場合、食べる 2ワームでの mRNA のレベルは他の系統よりも高かったが、蛋白質レベルが同じかまたは同じターゲットへの他の系統よりも低い。最も顕著な観測されたdlg 1の非常に高いレベルを食べる 2ワームより多くのタンパク質に翻訳しなかった mRNA実際には、他の系統 (図 3) と比較して低いdlg 1蛋白質レベルがあった。
最後に、GTCp 抽出タンパク質、RIPA 抽出タンパク質レベルは、顕著な違いを示した。RNA は、両方の同じ方法で抽出されました。ただし、著しく示した RIPA 溶解によって収集された似ていますが、別のサンプルは 75 kDa (図 3 a、右車線より大きいそれらのために特にタンパク質レベルを低下図 3 b、黄色バー)。
MRNA および蛋白質レベルの intrasample の比較
このプロトコルの目標の 1 つは mRNA で見た変化とタンパク質レベルは、本物だった場合、またはビジュアルサーボイング変化の成果物ができるかを判断します。図 4ターゲットのサブセット内で単一のサンプルとを比較しました。それぞれの個々 のサンプルは、同じ日陰とサンプル 1 (食べる 2)、灰色 (WT)、青の明るい色または赤 (林産物 1) として暗い陰とサンプル 4 をされていると、ドットの色として表示されます。ほとんどのサンプルの内で西部のしみの半定量分析の潜在的な欠陥、タンパク質レベルで大きい可変性と mRNA のレベルの低い変動があった。各 mRNA および蛋白質のペアの間の色のドットの位置を見ると、最高・最低の順序よく一致しませんでした。たとえば、WT、 hsp 60 mRNA 順序だったサンプルの 4、3、2、1、しかし蛋白質のレベルが 1、2、3、4。したがって、サンプル内の mRNA および蛋白質のレベルの違いは確かに存在するが、本手法により得られた差の可能なソースとしてコレクションの時間を削除するユーザー。
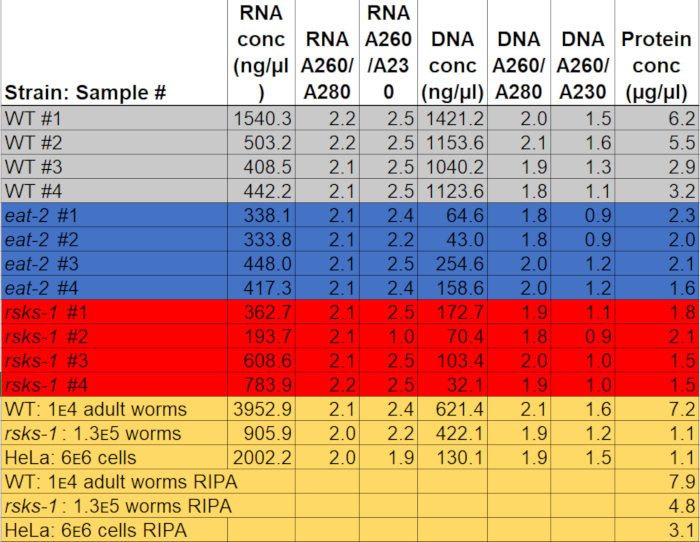
表 1: 濃度と吸収比率。RNA と DNA の濃度と純度比率を分光光度計で測定しました。蛋白質の比色定量アッセイを用いたタンパク質濃度を調べた。グレー、青と赤は、RT qPCR と西部のしみのための試料を強調表示します。黄色は、RIPA 蛋白質の抽出、hela 細胞の蛋白質にワーム タンパク質 GTCp を比較するサンプルを示します。このファイルをダウンロードするここをクリックしてください。
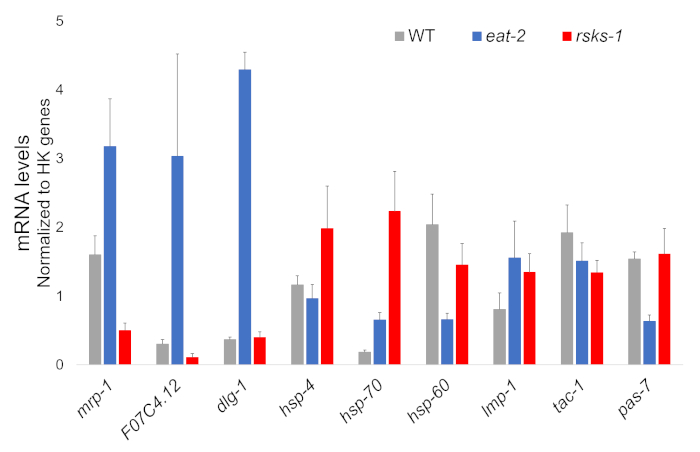
図 1: RT qPCR による遺伝子発現します。RT qPCR RNAseq データからとオルガネラ マーカー遺伝子の特定のターゲットを確認するこのメソッドによって得られる mRNA の解析。誤差範囲を表す標準偏差;n = 4。MRNA の濃度は、プライマーの各セットの標準曲線に対して定義されました。すべての mRNA のレベルは、一連の遺伝子、法 1、 cdc 42海部 1、 nhr 23、 pmp 3 cyn 1などとして使用される六つのハウスキーピング遺伝子のなかに正規化されました。この図の拡大版を表示するのにはここをクリックしてください。
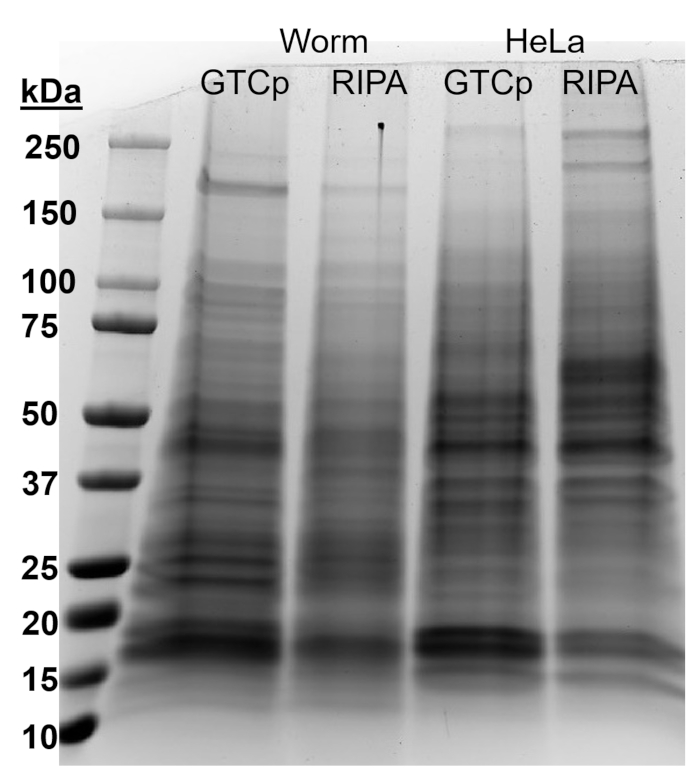
図 2: 比較 GTCp-ワームおよび HeLa 細胞でのタンパク質の RIPA 抽出と。野生型 (WT) ワームや SDS ページで区切られ、ブルーに染まった GTCp または RIPA の換散方法で収穫された HeLa 細胞の蛋白。各レーンには、総蛋白の 25 μ g が含まれています。この図の拡大版を表示するのにはここをクリックしてください。
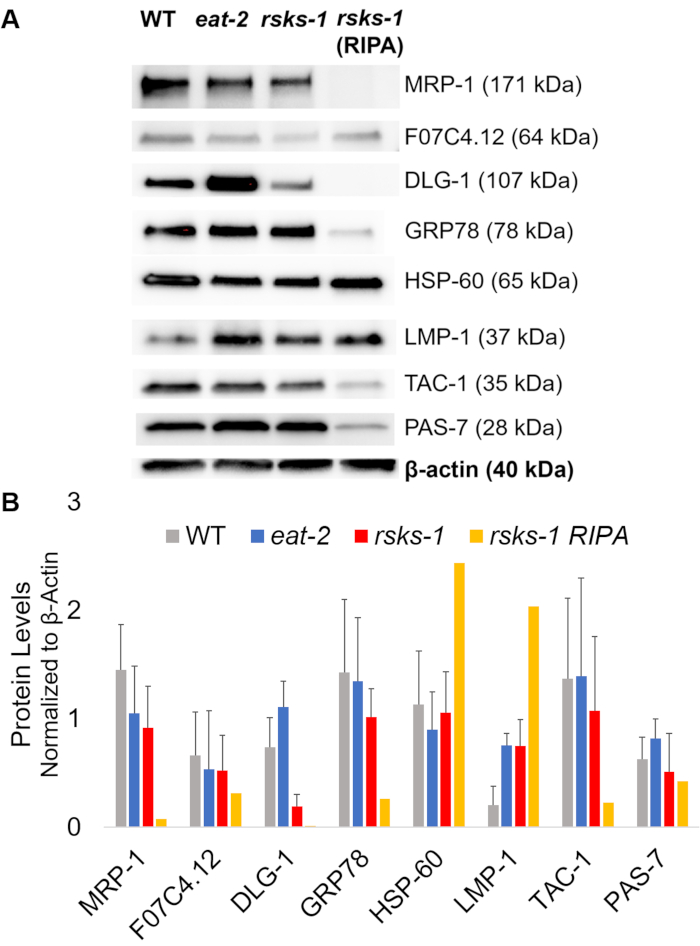
図 3: ワームのタンパク質レベル。(A) 信号強度の RT qPCR と (B) のデンシトメトリーの分析を用いてターゲットの西部のしみ。各レーンでは、野生型 (WT)、食べる 2、または林産物 1突然変異体ワームから合計 GTCp 抽出蛋白質の 20 μ g 含まれています。示されているイメージは、ワームひずみあたり 4 つの独立した複製の表現です。Β-アクチン (下) は、各ターゲットの等しい荷重の制御に使用しました。1 つだけの代表的なセットが表示されます。右の車線には、林産物 1突然変異体ワームから合計 RIPA 抽出蛋白質の 20 μ g が含まれています。(B) 対応する信号強度は ImageJ を用いた定量化されました。誤差範囲を表す標準偏差;n = 4。この図の拡大版を表示するのにはここをクリックしてください。
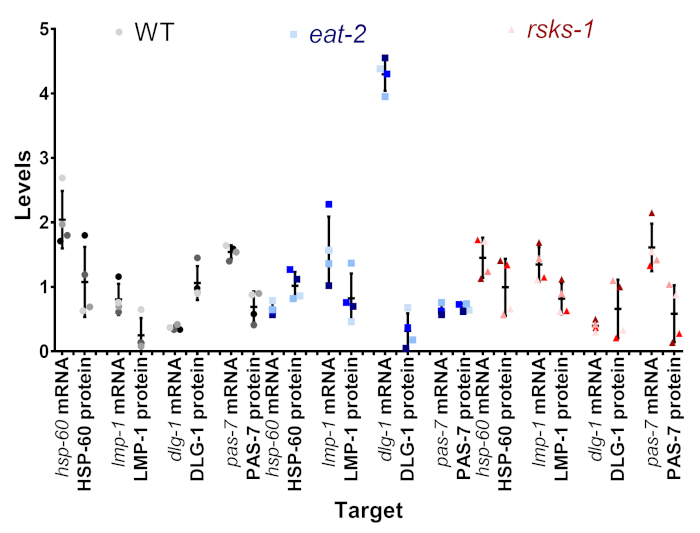
図 4: Intrasample mRNA および蛋白質レベル比較します。各緊張に対象のサブセットから個々 のサンプルから mRNA および蛋白質のレベルを配置します。色は表 1の代表です。WT は、グレー/ブラック、ブルーは食べる 2、そして赤は林産物 1。mRNA と同じターゲットへのタンパク質は隣同士にペアし、線虫のひずみで区切られました。この図の拡大版を表示するのにはここをクリックしてください。
ディスカッション
DNA、RNA、タンパク質などの生体分子分離法多くの場合技術的な重複有無の組み合わせ最適化されています。これは、特に不利なサンプルは、入手が困難な異なる時間に同じ条件の下でサンプルの収穫につながる可能性があります。携帯電話の経路によって複製を別々 に収集はバリエーションを生成するかもしれません。この原稿は、同時分離およびサンプルの収穫のタイミングのワームは、さまざまな分離技術によって生じるばらつきを減らす同じサンプルから各生体分子の精製を有効にして、このハードルを回避するための手順を提供していますまたは等しくない収穫します。これらの変数を制御するだけでなく、時間とリソースを節約、また再現性が容易になります。ここでは、DNA と変数の結果を伴い妥協の RNA とタンパク質の品質を回避する組合せアプローチを示しています。準備は、DNA クリーニング手順を使用して、さらに最適化できます。線虫C. elegans HeLa 細胞から材料を使用してアプローチを行った。
前作トランスクリプトーム ・ プロテオーム解析 N2 WT 動植物を食べる 2 林産物 1突然変異体の探索は、寿命4,34,35 を拡張するメカニズムを含む様々 な経路に洞察力を提供しています。 ,36。世界のタンパク質合成の全体的な低下がある安定同位体標識アミノ酸細胞培養 (SILAC) 解析が見つかりましたその食べる 2ワームに寿命を拡張することでカロリー制限のメカニズムを調査する努力、36します。 ここに表示されるデータは、一貫性がこの発見も同じターゲットの mRNA のレベルを大幅に増加。別のグループは S6K を介した長寿のエフェクタを特定することを目的し、したがって、林産物 1ワーム34のプロテオームの画面を実行します。現在の研究で見つかった RNAseq データから我々 はこの画面から特定の蛋白質を裏付ける、少なくとも 3 つの遺伝子を同定しました。MRP 1 と公認会計士とニューロリギン (F07C4.12) のホモログは、WT N234と比べて林産物 1ワームに発現されているとして発見されました。
このメソッドを使用して生成されたデータは、以前の multiomic の調査と一致。9 ターゲットの mRNA のレベルは、各サンプルの蛋白質のレベルを予測する使用されました。これらのターゲットの多くは mRNA のレベルに基づいて予測可能なタンパク質レベルを持っていた。それにもかかわらず、mRNA および蛋白質レベルの間顕著な不一致があった。重要なは、ここで提示されたプロトコルは科学者が自信を持って評価し、同じサンプルから mRNA および蛋白質を集めることによってビジュアルサーボイングの変動を除去することによってこれらの違いを解釈することができます。さらに、蛋白質のレベルは、同じサンプルから収集またはリッパな収穫、似ているが異なるサンプル採集に mRNA のレベルを比較しました。我々 はリパ抽出サンプル中のタンパク質の多くの低レベルがあったターゲット数を示した。ビジュアルサーボイングのバリエーションを制御する、なければないこの違いは mRNA および蛋白質の差動規則による場合を知ることは不可能。
これらの異なる高分子用に最適化されたプロトコルがあることを留意することが重要ですので、横断的分析は実験の最終目標ではない場合、は、代わりにこれらのメソッドを採用する適切な。GTCp を使用して DNA やタンパク質を分離すると難溶、NaOH などの弱い塩基の DNA の再構成を必要として不完全なの危険を冒して、暖房装置を持つ洗剤の高濃度バッファーでタンパク質を可になること可溶化します。また、GTCp のため、プロテアーゼなどの酵素を不活性化、酸性のフェノールが含まれている、場合を除き、冷凍に時間をかけてゆっくりと蛋白質を低下します。それは、これらの制限の回避の価値があるかどうかを決定する研究員の裁量になります。
重要なは、RNA、生殖不能の技術、特に断らない限り、氷のサンプルを維持する、市販の除染の使用を使用するときは、RNA をそのまま維持する試薬を実行することをお勧めします。特に、大きいサンプル必要がありますもっと GTCp から始まるため、各抽出溶媒がスケール アップする必要があります。GTCp の適切な量を使用する場合、このプロトコルはワームまたはセルのサンプルの任意の手動の均質化は不要です。DNA の隔離の中で、利回りは有機 (ピンク) 層を回復する能力に大きく依存です。最後に、分離中にタンパク質の可溶化を向上させるため、resolubilization バッファーのボリュームを増加または SDS のほかに他の洗剤を追加する必要があります。確かに、成虫、幼虫および卵の混合物ではなくワームの大人専用人口からの蛋白質は resolubilize やすいです。
全体的に、生体分子分離する統合的なアプローチを提供していますこのプロトコルを使用して、異なる生体分子を別々 に収穫から生じる mRNA および蛋白質のレベルの間の相関関係、またはその欠如の解釈を容易にサンプル。このメソッドを使用してさまざまな条件下での転写と翻訳後調節機構のより深い調査につながることができます蛋白質の mRNA の翻訳を相関ではない場合を正しく識別する科学者を助けることができます。
開示事項
著者が明らかに何もありません。
謝辞
L.R.L. 資金が供給された付与による NIH/ニア (R00 AG042494 および R01 AG051810) から高齢化研究のグレン財団法人研究の生物学的老化のメカニズムとアメリカ連合から中学校教員補助医療研究賞。著者は、この原稿の執筆に彼らの役に立つフィードバックのアニタの Kumar は、市泉ウォンを感謝したいです。
資料
| Name | Company | Catalog Number | Comments |
| 4–15% Mini-PROTEAN® TGX Stain-Free™ Protein Gels | BIORAD | 4568084 | |
| Antibodies: | |||
| CePAS7-s | DHSB- U of Iowa | ||
| CeTAC1-s | DHSB- U of Iowa | ||
| DLG1-s | DHSB- U of Iowa | ||
| GRP78 | Novus Bio. | NBp1-06274 | |
| HRP Goat Anti-Mouse | Li-Cor | 926-80010 | |
| HRP Goat Anti-Rabbit | Li-Cor | 92680011 | |
| HSP60-s | DHSB- U of Iowa | ||
| LMP1-s | DHSB- U of Iowa | ||
| MRP-1 | Abcam | ab24102 | |
| Neuroligin 3 | Abcam | ab172798 | |
| β-actin | Millipore | MAB1501R | |
| ChemiDoc MP Imaging System | BIORAD | ||
| Chloroform | Fisher | C298-500 | Health hazard, Irritant, Toxic |
| Coomassie Brilliant Blue | ThermoSci | 20279 | |
| DC Protein assay | BIORAD | 500-0116 | |
| Epoch 2 microplate reader | BioTek | ||
| Ethanol (200 proof) | Fisher | 04-355-223 | Flammable, health hazard |
| HeLa cells | ATCC | CCL-2 | BSL2 |
| iScript Reverse Transcription Supermix | BIORAD | 1708840 | |
| Hydra microdispenser: Matrix Hydra | Robbins/ThermoFisher | ||
| Isopropanol | Fisher | A516-4 | Flammable, health hazard |
| M9 buffer: 3 g KH2PO4 6 g Na2HPO4 5 g NaCl Add H2O to 1 liter. Sterilize by autoclaving. After solution cools down, add 1 mL sterile 1 M MgSO4 | |||
| Laemmli Sample Buffer (2x) | BIORAD | 161-0737 | |
| NanoDrop One | ThermoSci | ||
| PAGE apparatus | BIORAD | ||
| Ponceau S | Alfa Aesar | J60744 | |
| Primers | IDT | 25 nmole DNA Oligo with Standard Desalting | |
| dlg-1: F (5'-GGTCCTACCA GGCAGTTGAG-3') R (5'-CACGTCCGTT AACCTCTCCC-3') | |||
| hsp-4: F (5'-AGAGGGCTTT GTCAACCCAG-3') R (5'-TCGTCAGGGT TGATTCCACG-3') | |||
| hsp-70: F (5'-CGGCATGTGA ACGTGCTAAG-3') R (5'-GAGCAGTTGA GGTCCTTCCC-3') | |||
| hsp-60: F (5'-ATTGAGCAAT CGACGAGCGA-3') R (5'-CAACACCTCC TCCTGGAACG-3') | |||
| lmp-1: F (5'-ACAACAACAC CGGACTCACG-3') R (5'-ATCGAGCTCC CACTCTTTGG-3') | |||
| tac-1: F (5'-AGTGGCAGGC AAAGTTCCTC-3') R (5'-TGAGCACCTT GATCTCGTCG-3') | |||
| pas-7: F (5'-GTACGCTCAA AAGGCTGTCG-3') R (5'-CTGAATCGGC ATTGGCTCAC-3') | |||
| mrp-1: F (5'-TTTGCCTTGC GCTTGTTCTG-3') R (5'-AGTTCCAGTG CGGAGCATAC-3') | |||
| F07C4.12: F (5'-TGCTGAGCAT GAAGGACTGT-3') R (5'-TGGCAATAGC TCCTCCGTTG-3') | |||
| HK Actin: F (5'-CTACGAACTT CCTGACGGACAAG-3') R (5'-CCGGCGGACT CCATACC-3') | |||
| HK cyn-1: F (5'-GTGTCACCAT GGAGTTGTTC-3') R (5'-TCCGTAGATT GATTCACCAC-3') | |||
| HK nhr-23: F (5'-CAGAAACACT GAAGAACGCG-3') R (5'-CGATCTGCAG TGAATAGCTC-3') | |||
| HK ama-1: F (5'-TGGAACTCTG GAGTCACACC-3') R (5'-CATCCTCCTT CATTGAACGG-3') | |||
| HK cdc-42: F (5'-CTGCTGGACA GGAAGATTACG-3') R (5'-CTCGGACATT CTCGAATGAAG-3') | |||
| HK pmp-3: F (5'-GTTCCCGTGT TCATCACTCAT-3') R (5'-ACACCGTCGA GAAGCTGTAGA-3') | |||
| LightCycler 96 qPCR machine | Roche | ||
| RIPA buffer: 10 mM Tris-Cl (pH 8.0) 1 mM EDTA 1% Triton X-100 0.2% SDS 140 mM NaCl 1 tablet of Roche protease inhibitor per 20 mL | |||
| SsoAdvanced Universal SYBR Supermix | BIORAD | 1725274 | |
| SuperSignal West Femto Max Sens Substrate | ThermoSci | 34095 | |
| Trans-Blot Transfer apparatus | BIORAD | ||
| Trans-Blot Turbo Transfer Pack | BIORAD | 170-4159 | |
| TRIzol reagent | Invitrogen | 15596026 | Health hazard (skin, eyes) |
| Worm strains: | Caenorhabditis Genetics Center (CGC) | ||
| N2 (wild type) | Caenorhabditis Genetics Center (CGC) | ||
| eat-2 (MAH95) | Caenorhabditis Genetics Center (CGC) | ||
| rsks-1 (VB633) | Caenorhabditis Genetics Center (CGC) |
参考文献
- Rotroff, D. M., Motsinger-Reif, A. A. Embracing Integrative Multiomics Approaches. International Journal of Genomics. 2016, 1715985 (2016).
- Chen, R., Snyder, M. Promise of personalized omics to precision medicine. Wiley Interdisciplinary Reviews: System Biology and Medicine. 5 (1), 73-82 (2013).
- Anderson, L., Seilhamer, J. A comparison of selected mRNA and protein abundances in human liver. Electrophoresis. 18 (3-4), 533-537 (1997).
- Harvald, E. B., et al. Multi-omics Analyses of Starvation Responses Reveal a Central Role for Lipoprotein Metabolism in Acute Starvation Survival in C. elegans. Cell Systems. 5 (1), (2017).
- Griffin, T. J., et al. Complementary profiling of gene expression at the transcriptome and proteome levels in Saccharomyces cerevisiae. Molecular and Cell Proteomics. 1 (4), 323-333 (2002).
- Greenbaum, D., Colangelo, C., Williams, K., Gerstein, M. Comparing protein abundance and mRNA expression levels on a genomic scale. Genome Biology. 4 (9), 117 (2003).
- Lapierre, L. R., et al. The TFEB orthologue HLH-30 regulates autophagy and modulates longevity in Caenorhabditis elegans. Nature Communications. 4, 2267 (2013).
- Doherty, C. J., Kay, S. A. Circadian control of global gene expression patterns. Annual Reviews Genetics. 44, 419-444 (2010).
- Goya, M. E., Romanowski, A., Caldart, C. S., Benard, C. Y., Golombek, D. A. Circadian rhythms identified in Caenorhabditis elegans by in vivo long-term monitoring of a bioluminescent reporter. Proceedings of the National Academy of Sciences USA. 113 (48), E7837-E7845 (2016).
- Dalvin, L. A., Fautsch, M. P. Analysis of Circadian Rhythm Gene Expression With Reference to Diurnal Pattern of Intraocular Pressure in Mice. Investigative Ophthalmology and Visual Science. 56 (4), 2657-2663 (2015).
- Narayanan, A., Jacobson, M. P. Computational studies of protein regulation by post-translational phosphorylation. Current Opinion in Structural Biology. 19 (2), 156-163 (2009).
- Swatek, K. N., Komander, D. Ubiquitin modifications. Cell Research. 26 (4), 399-422 (2016).
- Cech, T. R., Steitz, J. A. The noncoding RNA revolution-trashing old rules to forge new ones. Cell. 157 (1), 77-94 (2014).
- Chomczynski, P., Sacchi, N. Single-step method of RNA isolation by acid guanidinium thiocyanate-phenol-chloroform extraction. Analytical Biochemistry. 162 (1), 156-159 (1987).
- Triant, D. A., Whitehead, A. Simultaneous Extraction of High-Quality RNA and DNA from Small Tissue Samples. Journal of Heredity. 100 (2), 246-250 (2009).
- Liu, X., Harada, S. RNA Isolation from Mammalian Samples. Current Protocols in Molecular Biology. 103 (1), 11-14 (2013).
- Hummon, A. B., Lim, S. R., Difilippantonio, M. J., Ried, T. Isolation and solubilization of proteins after TRIzol extraction of RNA and DNA from patient material following prolonged storage. Biotechniques. 42 (4), 467-470 (2007).
- Kopec, A. M., Rivera, P. D., Lacagnina, M. J., Hanamsagar, R., Bilbo, S. D. Optimized solubilization of TRIzol-precipitated protein permits Western blotting analysis to maximize data available from brain tissue. Journal of Neuroscience Methods. 280, 64-76 (2017).
- Stiernagle, T. Maintenance of C. elegans. WormBook. , (2006).
- Hoogewijs, D., Houthoofd, K., Matthijssens, F., Vandesompele, J., Vanfleteren, J. R. Selection and validation of a set of reliable reference genes for quantitative sod gene expression analysis in C. elegans. BMC Molecular Biology. 9 (1), 9 (2008).
- Mahmood, T., Yang, P. -. C. Western blot: technique, theory, and trouble shooting. North American Journal of Medical Sciences. 4 (9), 429-434 (2012).
- Peach, M., Marsh, N., MacPhee, D. J., Kurien, B. T., Scofield, R. H. Protein solubilization: Attend to the choice of lysis buffer. Protein Electrophoresis: Methods and Protocols. , 37-47 (2012).
- Lakowski, B., Hekimi, S. The genetics of caloric restriction in Caenorhabditis elegans. Proceedings of the National Academy of Science USA. 95 (22), 13091-13096 (1998).
- Pan, K. Z., et al. Inhibition of mRNA translation extends lifespan in Caenorhabditis elegans. Aging Cell. 6 (1), 111-119 (2007).
- Gaudet, P., Livstone, M. S., Lewis, S. E., Thomas, P. D. Phylogenetic-based propagation of functional annotations within the Gene Ontology consortium. Briefings in Bioinformatics. 12 (5), 449-462 (2011).
- Broeks, A., Gerrard, B., Allikmets, R., Dean, M., Plasterk, R. H. Homologues of the human multidrug resistance genes MRP and MDR contribute to heavy metal resistance in the soil nematode Caenorhabditis elegans. The EMBO Journal. 15 (22), 6132-6143 (1996).
- Hadwiger, G., Dour, S., Arur, S., Fox, P., Nonet, M. L. A Monoclonal Antibody Toolkit for C. elegans. PLoS One. 5 (4), e10161 (2010).
- Kostich, M., Fire, A., Fambrough, D. M. Identification and molecular-genetic characterization of a LAMP/CD68-like protein from Caenorhabditis elegans. Journal of Cell Science. 113 (14), 2595 (2000).
- Firestein, B. L., Rongo, C. DLG-1 is a MAGUK similar to SAP97 and is required for adherens junction formation. Molecular Biology of the Cell. 12 (11), 3465-3475 (2001).
- Heschl, M. F., Baillie, D. L. Characterization of the hsp70 multigene family of Caenorhabditis elegans. DNA. 8 (4), 233-243 (1989).
- Yoneda, T., et al. Compartment-specific perturbation of protein handling activates genes encoding mitochondrial chaperones. Journal of Cell Science. 117 (18), 4055 (2004).
- Takahashi, M., Iwasaki, H., Inoue, H., Takahashi, K. Reverse Genetic Analysis of the Caenorhabditis elegans 26S Proteasome Subunits by RNA Interference. Biological Chemistry. 383, 1263 (2002).
- Le Bot, N., Tsai, M. C., Andrews, R. K., Ahringer, J. TAC-1, a regulator of microtubule length in the C. elegans embryo. Current Biology. 13 (17), 1499-1505 (2003).
- McQuary, P. R., et al. C. elegans S6K Mutants Require a Creatine-Kinase-like Effector for Lifespan Extension. Cell Reports. 14 (9), 2059-2067 (2016).
- Larance, M., et al. Global Proteomics Analysis of the Response to Starvation in C. elegans . Molecular and Cell Proteomics. 14 (7), 1989-2001 (2015).
- Yuan, Y., et al. Enhanced energy metabolism contributes to the extended life span of calorie-restricted Caenorhabditis elegans. Journal of Biological Chemistry. 287 (37), 31414-31426 (2012).
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved