Method Article
Combinado nucleotídeos e proteínas extrações em Caenorhabditis elegans
Neste Artigo
Resumo
Aqui, apresentamos um protocolo para a isolação do RNA, DNA e proteínas da mesma amostra, em um esforço para reduzir a variação, melhorar a reprodutibilidade e facilitar interpretações.
Resumo
Uma única amostra biológica contém uma infinidade de informações, e agora é prática comum para investigar simultaneamente várias macromoléculas para capturar uma imagem completa dos vários níveis de processamento molecular e alterações entre diferentes condições. Este protocolo apresenta o método de isolar o DNA, RNA, e proteína da mesma amostra do nemátodo Caenorhabditis elegans , para retirar a variação introduzida quando estas biomoléculas são isoladas de repetições de tratamento similar mas finalmente amostras diferentes. Ácidos nucleicos e proteínas são extraídos o nematódeo usando o método de extração de tiocianato-fenol-clorofórmio guanidínio ácido, com posterior precipitação, lavagem e solubilização de cada um. Nós mostramos o sucesso isolamento de RNA, DNA e proteínas de uma única amostra de três estirpes de nematódeo e células HeLa, com melhores resultados de isolamento da proteína em animais adultos. Além disso, tiocianato-fenol-clorofórmio-extraído de guanidínio proteína de nematoides melhora a resolução de proteínas maiores, com níveis detectáveis reforçadas como observado pelo immunoblotting, comparado com a extração de RIPA tradicional de proteína.
O método apresentado aqui é útil quando investigando amostras usando uma abordagem multiomic, especificamente para a exploração do transcriptoma e proteoma. Técnicas que simultaneamente avaliar multiomics são atraentes porque moleculares sinalização subjacentes complexos fenômenos biológicos é pensado para ocorrer em níveis complementares; no entanto, tornou-se cada vez mais comum ver que mudanças nos níveis de RNAm não reflectem sempre a mesma alteração dos níveis de proteína e que o tempo da coleção é relevante no contexto dos regulamentos circadianos. Esse método remove qualquer variação intersample quando a análise do conteúdo diferente dentro da mesma amostra (intrasample).
Introdução
Multiomics, a abordagem analítica que usa uma combinação de omics, tais como o genoma, proteoma, transcriptoma, Epigenoma, microbiome ou lipidome, tornou-se cada vez mais popular ao processar grandes conjuntos de dados para caracterização de doença1, 2. Evidências tem mostrado que limitar abordagens para um único "ome" fornece uma análise molecular incompleta (revista por Rotroff e Motsinger-Reif1). Grandes conjuntos de dados são gerados, particularmente ao executar telas usando técnicas de alta produtividade, mas para pintar um quadro completo ou para identificar os alvos mais relevantes, multiomic abordagens são preferíveis. Com a utilização de abordagens de multiomics, no entanto, há a observação frequente das discrepâncias entre o mRNA e proteína níveis3,4,5,6. Notavelmente, mRNA e proteína usado para transcriptomic de side-by-side e proteomic análises com a sequenciação do ARN (RNAseq) e líquida espectrometria de massa em tandem-cromatografia (LC-MS/MS), respectivamente, são muitas vezes obtidas amostras tratadas da mesma forma de réplicas diferentes, introduzir potencialmente variação entre os mesmos condições3,4,5,6. Harvald et al. realizaram um elegante estudo de tempo-curso de fome de c. elegans que comparou o transcriptoma e proteoma do selvagem-tipo (WT) vermes ao de hlh-30 vermes mutantes que não possuem um fator de transcrição importante na longevidade 7. da nota, o RNA e proteína foram colhidas as mesma condição repetições, então não a partir da mesma amostra. Suas conclusões mostram uma baixa correlação entre os níveis de proteína em cada ponto de tempo e os níveis do mRNA (r = 0.559 para 0.628). Na verdade, seu heatmap formados quatro grupos: Cluster, tive uma grande diminuição no mRNA níveis mas pouca ou nenhuma diminuição nos níveis de proteína correspondente, Cluster II tiveram pouco ou nenhum aumento nos níveis de mRNA, mas um aumento nos níveis de proteína, Cluster III tiveram um aumento no mRNA níveis, mas uma diminuição nos níveis de proteína e Cluster IV teve um aumento nos níveis de mRNA, mas somente uma mudança sutil em níveis de proteína4. Além disso, esta variação de intersample pode ser introduzida em casos onde as amostras da mesma condição não são coletadas ao mesmo tempo. Por exemplo, o mRNA e proteínas regulamentadas pelo ciclo circadiano flutuarem dependendo do tempo de dia8,9, ou, mais especificamente, a exposição de c. elegans a luz9; expressão destas proteínas circadiano pode ser atrasada até 8 h após de indução de expressão de gene10. Não obstante, a prevalência desta observação não significa necessariamente que é errado; na verdade, isso pode vir a ser informativo. Proteína e mRNA estão em um constante estado de dinâmico entre formação e degradação. Além disso, as proteínas posttranslationally frequentemente são modificadas para aumentar a estabilidade ou para induzir a sua degradação11. Por exemplo, seu status da ubiquitination pode levar à ativação ou direcionamento para o proteossomo ou lisossoma para degradação12. Além disso, o RNA não-codificante desempenham um papel importante na regulação da expressão gênica em estágios transcriptional e posttranscriptional13. Assim, a questão é como limitar as variáveis para confirmar que as discrepâncias que observamos nestes estudos nematoides são reais.
Aqui, propomos um método que remove a variável intersample, permitindo que análises de macromoléculas diferentes da mesma amostra. O objetivo do presente protocolo é oferecer um método para isolar consistentemente o DNA, RNA e proteína de uma única amostra de c. elegans (também referido como vermes) em um esforço para reduzir a variação, melhorar a reprodutibilidade e facilitar a interpretações. Benefícios adicionais de usando a mesma amostra incluem a economia de tempo e recursos durante a coleta de amostra, facilitando a análise transversal de valiosas e limitadas de amostras, incluindo cepas que são difíceis de cultivar e manter e ganhando insights sobre a regulação diferencial de macromoléculas com base em intrasample variações nos níveis de mRNA e proteína.
Este método é adequado para avaliar expressões do gene e os níveis de proteína de uma única amostra de vermes, permitindo uma avaliação mais abrangente de vários níveis de processamento molecular. Guanidínio tiocianato-fenol-clorofórmio (GTCp) reagente14, um produto químico comumente usado para isolar o RNA, é usado para a extração de ácidos nucleicos e proteínas do vermes, com posterior precipitação, lavagem e solubilização de cada um. Este protocolo é uma compilação de vários protocolos15,16 com pequenas modificações, projetado com foco em c. elegans, mas também com sucesso isolamos RNA, proteínas e DNA de um pellet de células HeLa seguindo o mesmas etapas. Embora não testado aqui, este protocolo é provável trabalhar em tecidos também17,18.
Protocolo
Nota: Cada etapa de precipitação da macromolécula é realizada em sequência, seguido de lavagens feitas simultaneamente; no entanto, é recomendável para completar o isolamento de RNA primeiro como é intrinsecamente instável.
1. coleta de amostra
- Sementes de 1.000 ovos de verme por placa de 10 cm com crescimento adequado condições19. Incube a 20 ° C por 72 h.
Nota: Descorante do ovo-rolamento adultos a fim de recolher os ovos, como anteriormente descreveram19. - Lavar a placa com cerca de 5 mL de tampão de M9 e recolher 1.000 vermes adultos em um tubo.
Nota: M9 reserva é composta por 35 milímetros fosfato de sódio dibásico, cloreto de sódio de 102 mM, 22 milímetros de fosfato de potássio monobásico e 1mm de sulfato de magnésio em água estéril19. - Centrifugue os vermes a 1.000 x g por 1 min, desprezar o sobrenadante e transferir os vermes peletizados com 1 mL de tampão de M9 para um tubo de microcentrifugadora de 1,5 mL.
- Centrifugar novamente a 845 x g por 1 min e descartar a maioria do líquido sobrenadante. Armazene os vermes peletizados a-80 ° C por um período mínimo de 4h.
Nota: Usar um sedimento congelado produz um rendimento mais elevado do que um fresco da pelota. Recomenda-se uma série de ciclos de congelamento/descongelamento usando líquido nitrogênio ou 95% etanol em gelo seco para rachar a cutícula do verme se usando uma fresco da pelota. Deixar uma pequena quantidade de M9 sobre a pelota vai ajudar a quebrar a cutícula ao congelar.
2. nucleotídeo e isolamento de proteínas
- Remova qualquer sobrenadante a pelota descongelada e adicione 1 mL do reagente de GTCp frio. Misture bem pipetando para cima e para baixo. Coloque a amostra no gelo por 10 min e misturá-lo ocasionalmente, virando-o de cabeça para baixo.
Atenção: Fenol é altamente tóxico, corrosivo e neurotóxicos e pode causar queimaduras e cegueira.
Nota: Usamos até 5.000 vermes adultos como matérias-primas com os volumes relatados; cada volume é baseado na utilização de 1 mL do reagente de GTCp. Quando se utiliza uma amostra maior, pode ser necessário para scale-up. - Para a solução de vermes e GTCp, adicione 200 µ l de clorofórmio frio. Segure o tubo entre os dedos e agitar vigorosamente para deixar s. 15 à temperatura ambiente (RT) por 3 min.
Atenção: Clorofórmio é um irritante de tóxico, pode causar úlceras de pele e outros danos em órgãos-alvo e é um possível carcinógeno. - Centrifugar o tubo a 13.500 x g durante 15 min a 4 ° C.
Nota: Três camadas são formadas após este spin: cima, fase aquosa clara, fundo, rosa fase orgânica e a interfase pequena, nublada que contém lipídios e DNA. - Utilizando uma micropipeta, mover a camada transparente superior (fase aquosa) para um novo livre de RNase 1,5 mL microcentrifuga tubo (tubo A) isolar o RNA através de precipitação do álcool (conforme detalhado na etapa 2.5) e mova a camada rosa (fase orgânica) de remanescente para um novo tubo ( tubo B) e coloque-o no gelo.
Nota: A fase orgânica-de-rosa pode ser congelada a-80 ° C até o isolamento de DNA e proteína desta amostra. Amostras maiores produzirá uma espessa camada branca entre a fase aquosa e orgânica. Esta fração contém DNA. Se esta camada pode ser removida sem perturbar as outras fases, fazê-lo e colocá-lo em um separado tubo (B2). -
Isole o RNA como segue.
- Da fase aquosa clara no tubo A, precipitar o RNA com 500 µ l de isopropanol 100%. Incubar durante 10 minutos a RT. Em seguida, centrifugar o tubo à 13.500 x g durante 10 minutos a 4 ° C.
Nota: Um pequeno sedimento branco do RNA deve ser visível na parte inferior do tubo. - Decante a maior parte do sobrenadante. Remover o restante com uma seringa de 1 mL com agulha e descartar o sobrenadante.
Nota: O tamanho da agulha não é importante; no entanto, um maior calibre da agulha vai oferecer mais controle para não perturbar a pelota. Uma micropipeta pode ser usada se as agulhas não estão disponíveis. - Adicione 1 mL de etanol a 75% para o tubo A lavar o sedimento. Girar o tubo a 5.300 x g por 5 min a 4 ° C.
- Retirar o sobrenadante a pelota por decantação e utilizando uma seringa de 1 mL com agulha, conforme descrito na etapa 2.5.2 e descartá-lo.
- Deixar o sedimento secar por 5-10 min, mas não deixe ressecar. Use 50 µ l de água livre de RNase para reconstituir a pelota do RNA antes que se torne completamente transparente.
Nota: Este é um volume adequado de partida para 1.000-3.000 vermes, mas talvez precise ser ajustada dependendo da quantidade inicial de material foi usado. - Incube a pelota a 55 – 60 ° C por 10 min para dissolvê-lo.
- Medir a concentração e a pureza usando um espectrofotômetro. Gravar as absorvâncias em 260 nm para a concentração de RNA e a 230 e 280 nm para identificar quaisquer impurezas.
- Limpe o RNA para remover quaisquer contaminantes, tais como resíduos de fenol ou DNA, com a purificação da coluna ou com lavagens subsequentes etanol e tratamento de DNase.
Nota: Neste ponto, o RNA está pronto para ser enviada para análise RNAseq ou pode ser usado para fazer o cDNA usar para RT-qPCR (ver secção 3.1 para obter detalhes). RNA pode ser armazenado a-80 ° C até utilização posterior.
- Da fase aquosa clara no tubo A, precipitar o RNA com 500 µ l de isopropanol 100%. Incubar durante 10 minutos a RT. Em seguida, centrifugar o tubo à 13.500 x g durante 10 minutos a 4 ° C.
-
Isole o DNA como segue.
- Da fase orgânica rosa no tubo B e a amostra no tubo B2, se for o caso, precipitar o DNA adicionando 300 µ l de etanol 100% e misture por inversão. Deixe os tubos em RT para 2-3 min.
- Centrifuga tubos B e B2 a 375 x g por 5 min a 4 ° C para DNA de Pelotas.
- Mova o sobrenadante de tubos B e B2 coloca-lo em um novo tubo de 2 mL (tubo C) e deixá-lo em gelo para o isolamento de proteínas subsequentes. Lavar o sedimento de DNA em tubo B ou B2 com 1 mL de citrato de sódio 0,1 M em 10% de etanol para tubos de centrífuga de 30 min. B e B2 a 375 x g por 5 min a 4 ° C.
Nota: Sódio vincula-se a espinha dorsal de DNA, fazer DNA precipitado mais prontamente em citrato de sódio e etanol, permitindo que as impurezas sejam removidos por centrifugação de baixa velocidade. - Repita a etapa de lavagem, conforme descrito na etapa 2.6.3. Resuspenda o pellet em 1,5 mL de etanol 75% e deixá-lo em RT por 20 min, com ocasionais de mistura por erigir.
- Tubo de centrífuga B e B2 a 375 x g por 5 min a 4 ° C.
- Desprezar o sobrenadante e deixe-a secar por 5-10 min.
- Dissolva a pelota em 150 µ l de hidróxido de sódio de 8 mM. Ajuste o pH desejado com HEPES, se necessário. Girar a amostra a 375 x g por 5 min a 4 ° C.
- Utilizando uma micropipeta, mova o sobrenadante (DNA) para um tubo novo. Medir a concentração e determinar a pureza usando um espectrofotômetro. Gravar as absorvâncias em 260 nm para a concentração de DNA e a 230 e 280 nm para identificar quaisquer impurezas.
-
Isole a proteína como segue.
- Para precipitar a proteína, adicionar até 1,5 mL de isopropanol 100% para o rosa sobrenadante no tubo C, mistura várias vezes, por inversão e incubar a RT por 10 min.
- Centrifugar o tubo C a 13.500 x g durante 10 minutos a 4 ° C.
- Desprezar o sobrenadante e lavar o sedimento com 2 mL de cloridrato de guanidina 0,3 M em etanol a 95% por 20 min no tubo de centrífuga de RT. C a 5.300 x g por 5 min a 4 ° C.
- Repita a etapa de lavagem, conforme descrito na etapa 2.7.3 2x.
- Mover a pelota de proteína para um novo tubo de 1,5 mL (tubo C2) e adicionar a 1,5 mL de etanol a 95%. Vórtice e deixá-lo sentar na RT por 20 min.
- Centrifugar o tubo C2 a 5.300 x g por 5 min a 4 ° C. Desprezar o sobrenadante e deixar o sedimento secar por 10 min a RT. dissolver a pelota em 300 µ l de SDS de 5% a 50 ° C por 60 min.
Nota: Um tempo de incubação pode ser necessário para dissolver totalmente a pelota de proteína. No passado, a pelota tem sido incubada por até 6 h sem sacrificar a qualidade. - Centrifugar o tubo C2 a 13.500 x g durante 10 minutos a 17 ° C. Mova o sobrenadante para um tubo novo.
- Medir a concentração usando um ensaio de quantificação de proteína preferencial que seja compatível com detergente.
Nota: A proteína está pronta para ser usado em SDS-PAGE e mancha ocidental. Pode ser armazenado a-20 ° C para uso futuro. Para outras técnicas, tais como LC-MS/MS, o detergente precisará ser diálise fora.
3. avaliação do mRNA e níveis de proteína
-
RT-qPCR
- Preparar o cDNA por transcrição reversa com 1 ng do RNA, usando o seguinte protocolo de thermocycler: Primer (5 min a 25 ° C), a transcrição reversa (20 min a 46 ° C) e inativação de RT (1 min a 95 ° C).
- Fazer um prato estoque de cDNA adicionando-se 100 µ l de diluições de 1: 100 de cada amostra de cDNA individuais aos poços de uma placa de 96 poços. Incluem controles apropriados, como não somente água/modelo wells e amostras diluídas em série de 01:25 a 1: 400 (originou-se com em pool do cDNA de todas as amostras analisadas) para permitir uma curva padrão estabelecer a eficiência da primeira demão para cada gene analisado.
Nota: Este formato pode ser usado com o microdispenser de 96 poços, que permite a transferência de todas as amostras em uma placa para uma placa de RT-qPCR e para a mitigação de pipetagem variações a bem. A combinação adequada de mestre (por exemplo, SYBR + primeiras demão) pode ser adicionada a cada bem, daí em diante, com uma pipeta multicanal. Em geral, esta abordagem reduz erros introduzidos quando mão-pipetagem de cada amostra, reduzindo ainda mais a variabilidade. - Adicione 3 µ l de cDNA da placa de estoque nos poços da placa de RT-qPCR.
- Faça uma mistura de mestre para cada conjunto de primers que inclui 1 supermix de x, contendo um corante cianina intercalante de DNA, 5 µM cada de encaminhar e reverter as primeiras demão e água até 7 µ l por amostra. Adicionar 7 µ l para os apropriado poços de uma placa de RT-qPCR e agitar levemente para misturar.
Nota: Inclui amostras para medir os genes das tarefas domésticas para usar como uma referência para normalizar os resultados de20. - Verifique a placa usando um protocolo de RT-qPCR que é apropriado para os primers sendo usados.
-
Borrão ocidental
Nota: Para obter informações detalhadas sobre borrões ocidentais, por favor consulte Marques e Yang21.- Prepare amostras para SDS-PAGE, combinando a 20 µ g de proteína com um volume igual de 2x Laemmli Sample Buffer e ferver a 95 ° C por 10 min.
- Carregar as amostras em um gel de Tris-glicina 4 – 15% e executá-las em 150 V por 1h, ou até a frente de corante atinge o fundo do gel.
- Transferir as proteínas do gel para uma membrana de nitrocelulose em 25 V por 30 min em um aparelho de transferência semidry.
- Confirme a transferência adequada pela coloração da membrana com mancha de Ponceau S.
- Lave a mancha da membrana com salino Tris (TBS) com 0.01% polissorbato 20 (TBS-T).
- Bloquear a membrana com 5% de leite desnatado em TBS-T por 1h no RT
- Incube a membrana no anticorpo primário na diluição apropriada de acordo com as recomendações do fornecedor. Deixá-lo no roqueiro a 4 ° C durante a noite.
- Lavar a membrana 3 x, por 5 min cada, com TBS-T.
- Incube membrana no anticorpo secundário apropriado na diluição apropriada de acordo com as recomendações do fornecedor. Deixá-lo ligado o roqueiro por 1h no RT
- Lavar a membrana 3 x, por 5 min cada, com TBS-T.
- Imagem e quantificar o borrão ocidental, usando métodos preferenciais.
Resultados
Analisaram-se as amostras de DNA, RNA e proteína, e usando os RNA e proteína isolados de resultados representativos são apresentados aqui. Além disso, vamos comparar a amostra de proteína colhida usando o método GTCp para o comum de método de Lise RIPA22. Para nosso experimento, foi utilizado quatro repetições independentes de cada vermes WT (N2) e dois vermes mutantes long-lived, comer-2 e riscos-123,24.
Qualidade e quantidade de RNA e DNA
A concentração do RNA quando resuspended em 50 µ l de água variou de 0.2-2 µ g / µ l, usar cerca de 3.000 adultos vermes como matéria-prima. Os rácios de absorvância, que indicam a pureza, variaram de 2.0 para 2.2 para A260/A280 e 1,9 a 2,5 para A260/A230 (tabela 1), indicando a sucesso extração de RNA sem contaminação com GTCp componentes, tais como fenol ou guanidina cloridrato.
A extração de DNA foi bem-sucedida, mas a qualidade era variável. A concentração de DNA quando resuspended em 150 µ l de NaOH variaram de 0,04 a 1.1 µ g / µ l. Os rácios de absorvância variou entre 1.8 e 2.1 para A260/A280 e 0,9 a 1,6 para A260/A230 (tabela 1), indicando a sucesso extração de DNA; no entanto, algumas das amostras foram contaminadas com componentes GTCp, como cloridrato de fenol ou guanidina. Se o isolamento do DNA é necessário, cuidado deve ser tomado quando separa a separação de fases GTCp camada média, e mais lavagens podem ser necessárias.
RT-qPCR de alvos selecionados
O isolamento de RNA de quatro amostras independentes de cada estirpe worm foi um sucesso e foi enviado para análise de RNAseq, com subsequente RT-qPCR executada usando um supermix contendo um corante cianina. Metas analisadas por RT-qPCR foram selecionadas de um conjunto de dados grande RNAseq baseado os genes regulados diferencialmente mais comuns em ambos os mutantes em comparação com os vermes do WT. F07C4.1225, um homólogo de humano neuroligin 3, isoforma b, era o alvo selecionado upregulated compartilhado. Incluímos também um gene diferencialmente regulado entre ambos os mutantes, mrp-1,26, para análise adicional, que foi upregulated em comer-2 worms mas ativador em vermes de riscos-1 . Alterações de expressão do mrp-1 foram confirmadas através de RT-qPCR; no entanto, o upregulation F07C4.12 em vermes de riscos-1 previsto pela análise RNAseq não foi visto por RT-qPCR (Figura 1).
Metas adicionais com anticorpos validados para vermes foram investigadas também. Estes incluíram vários marcadores de organela caracterizados no Hadwiger et al.27. Um número destes marcadores foram diferencialmente expressos no nível de mRNA nos vermes mutantes. Como pode ser visto na Figura 1, o lmp-128 e dlg-129 foram upregulated em comer-2 vermes; HSP-4 30, hsp-7030e lmp-1 foram upregulated em vermes de riscos-1 ; HSP-60 31 e pas-732 foram ativador em comer-2 vermes; HSP-60 e tac-133 foram ativador em vermes de riscos-1 .
Preparação de proteína simultânea vs RIPA
Para afirmar a eficiência e a qualidade da proteína com o método GTCp como descrito acima, comparamos a proteína coletada usando o método mais tradicional de RIPA lise22. Usando a mesma quantidade de material, ou seja apenas adultos (cerca de 10.000 vermes) ou uma população mista de adultos, larvas e ovos (cerca de 130.000 vermes), começar a quantidade global coletada com GTCp foi menor quando resuspended em volumes iguais de fim (tabela 1 ); no entanto, a extração de uma população apenas para adultos foi mais bem sucedida do que de uma população mista. Além disso, a qualidade das proteínas era semelhante, embora a proteína GTCp-extraído mostrou uma melhor resolução de proteínas maiores em cima de igual carga de proteína total, conforme mostrado pelo Coomassie mancha na Figura 2. Com efeito, alvos avaliados pelo borrão ocidental na Figura 3 mostraram níveis semelhantes na maioria dos casos; no entanto, as proteínas maiores que 75 kDa mostraram níveis mais baixos da proteína extraída de RIPA em comparação com a proteína GTCp-extraído (Figura 3A, faixa da direita; Figura 3B, amarelo bar).
O método de extração aqui apresentado também foi avaliado na linha de mamíferos células HeLa. Para referência, a quantidade de proteína extraída com RIPA um sedimento compacto de células HeLa 6 milhões (uma pelota de ~ 25 µ l) foi comparável àquela extraída de uma população mista 130.000 de vermes (um sedimento compacto ~ 75 µ l), ou cerca de metade do que é extraído de 10.000 vermes adultos (um ~ 100 µ l gravidade liquidados pellet), como pode ser visto na tabela 1. Mostramos que a eficiência de extração de proteínas (tabela 1) e a resolução de proteínas maiores (Figura 2) foi diminuídas em GTCp comparado à proteína extraída de RIPA em células HeLa, sugerindo que esta técnica funciona melhor em uma população adulta de vermes. A solubilização incompleta da pelota de proteína de HeLa GTCp-extraído da proteína pode ser uma limitação deste método em células de mamíferos.
Alvos de immunoblotting, analisados por RT-qPCR
Em seguida, investigamos os níveis de proteína dos produtos gene testados por RT-qPCR para determinar se os níveis do mRNA correlacionaram com os níveis de proteína. Como pode ser visto na Figura 3, as alterações de média expressão da proteína correlacionaram com as expressões de mRNA média mudadas para muitos destinos; no entanto, alguns níveis de proteína não reflectia as alterações nos níveis de RNAm. Muitas vezes, os níveis do mRNA em comer-2 worms foram maiores do que aqueles em outras cepas, mas os níveis de proteína foram ambos o mesmo ou menor do que as outras cepas do mesmo destino. A observação mais marcante foi os níveis muito elevados de dlg-1 mRNA em comer-2 worms, que não se traduz em mais proteína; na verdade, havia mais baixos níveis de proteína dlg-1 quando comparado com as outras cepas (Figura 3).
Finalmente, os níveis da proteína GTCp-extraído e a proteína extraída de RIPA mostraram uma diferença marcante. O RNA foi extraído da mesma forma para os dois; no entanto, a amostra semelhante, mas separada coletada por lise RIPA mostrou-se marcadamente reduzido os níveis de proteína, particularmente para aqueles maiores que 75 kDa (Figura 3A, faixa da direita; Figura 3B, amarelo bar).
Intrasample comparação dos níveis de mRNA e proteína
Um dos objetivos do presente protocolo foi determinar se a variação que vimos no mRNA e o nível de proteína verdadeiro, ou se pode ser um artefato de variação intersample. Na Figura 4, um subconjunto de metas foi comparado dentro das amostras simples. Cada amostra individual é mostrada como a mesma sombra e cor do ponto, com amostra 1, sendo a sombra mais escura e a amostra 4 como a mais leve sombra de cinza (WT), azul (comer-2), ou vermelho (riscos-1). Na maioria das amostras, houve uma baixa variabilidade dos níveis de mRNA, com maior variabilidade no nível de proteína, uma falha potencial da análise semiquantitativa de western blots. Quando se olha para a posição de cada um dos pontos coloridos entre os pares de mRNA e proteína, a ordem do maior para o menor, muitas vezes não corresponde; por exemplo, em WT, a ordem de mRNA hsp-60 foi a amostra 4, 3, 2, 1, mas os níveis de proteína foram 1, 2, 3, 4. Assim, certamente existem diferenças entre níveis de mRNA e proteína em uma amostra, mas o método apresentado permite que usuários removam o tempo da coleção como uma fonte possível da diferença observada.
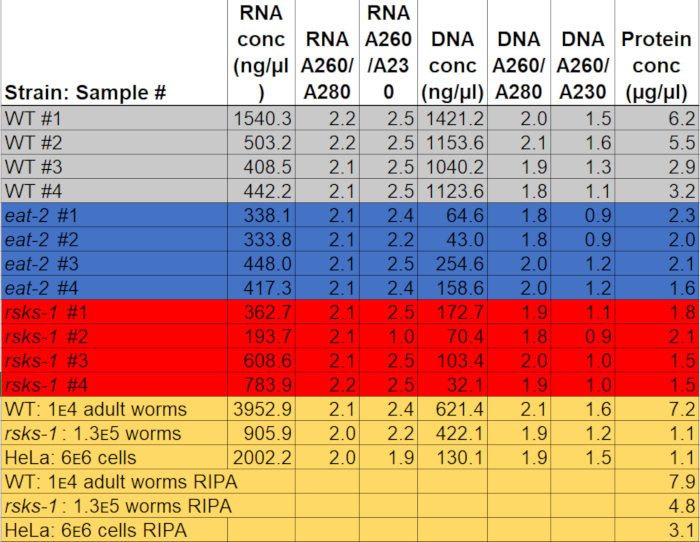
Tabela 1: concentrações e absorção rácios. Rácios de pureza e concentrações de RNA e DNA foram medidos em um espectrofotômetro. As concentrações de proteína foram determinadas utilizando um ensaio de quantificação de proteína colorimétrico. O cinza, azul e vermelho destacam as amostras utilizadas para RT-qPCR e borrão ocidental. O amarelo indica amostras utilizadas para comparar GTCp para extração de proteína RIPA ou worm proteína a proteína HeLa. Clique aqui para baixar este arquivo.
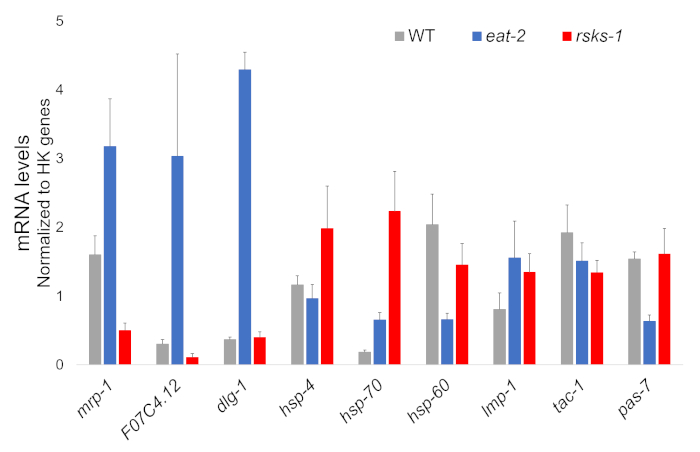
Figura 1: expressão gênica por RT-qPCR. Análise de RT-qPCR para o mRNA obtidas por esse método, confirmando alvos identificados de dados RNAseq e de genes marcadores de organela. As barras de erro representam o desvio padrão; n = 4. A concentração de mRNA foi definida em relação a uma curva padrão para cada conjunto de primers. Todos os níveis do mRNA foram normalizados para a média de um conjunto de seis genes de limpeza usado como genes de referência, que incluem act-1, cdc-42, ama-1, nhr-23, pmp-3e cyn-1. Clique aqui para ver uma versão maior desta figura.
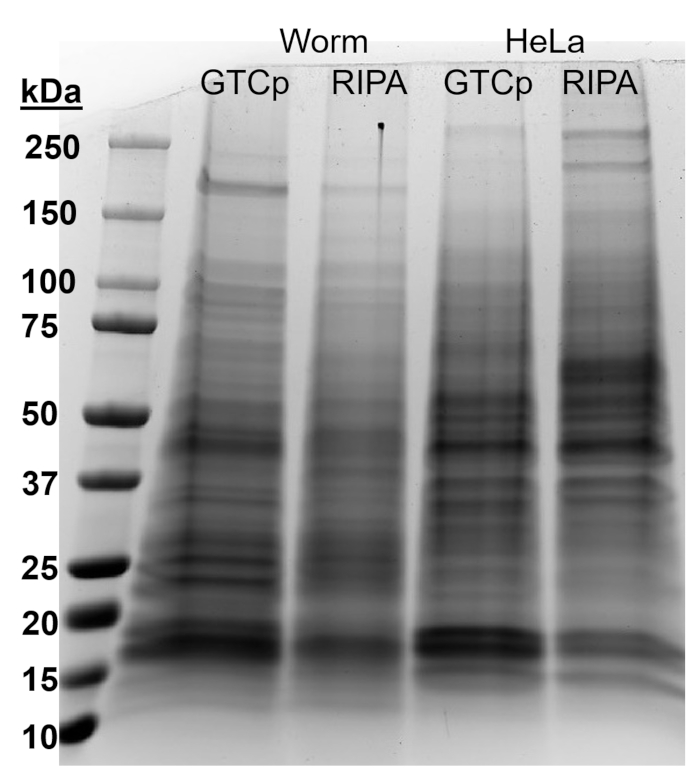
Figura 2: comparação de GTCp-versus proteína extraída de RIPA em worms e células HeLa. Proteínas totais do selvagem-tipo (WT) vermes ou células HeLa colhidas via GTCp ou RIPA método de Lise, separados por SDS-PAGE e corados com azul de Coomassie. Cada pista contém 25 µ g da proteína total. Clique aqui para ver uma versão maior desta figura.
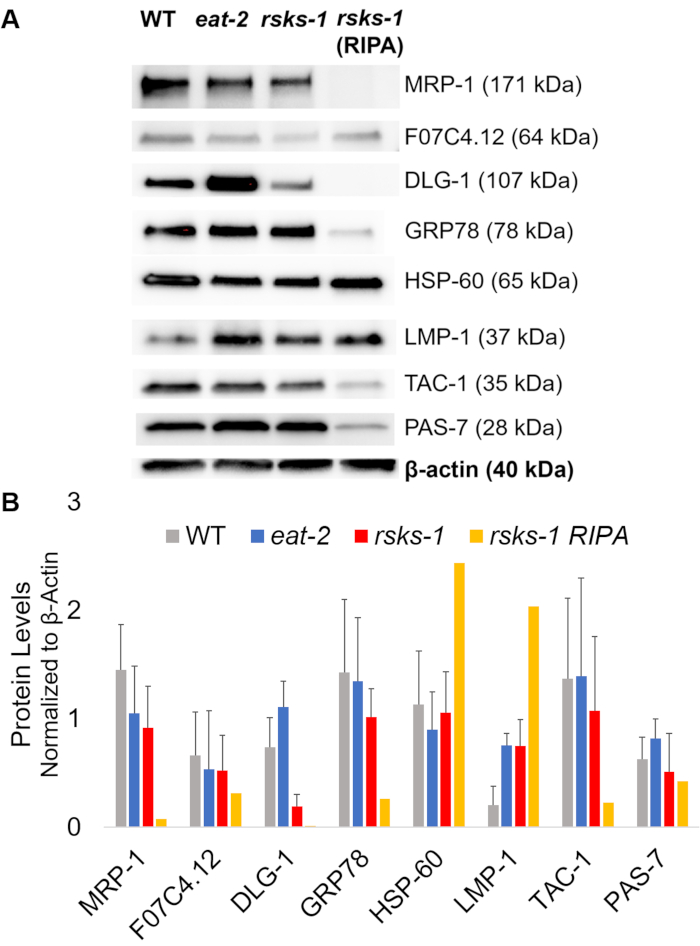
Figura 3: níveis de proteína em worms. (A) ocidental do borrão de alvos investigados por RT-qPCR e (B) análise de densitometria de intensidade do sinal. Cada pista contém 20 µ g da proteína total GTCp-extraído do selvagem-tipo (WT), comer-2ou vermes mutantes de riscos-1 . A imagem mostrada é uma representação de quatro repetições independentes por estirpe de verme. Β-actin (inferior) foi usado para controlar para carregamento igual para cada destino; apenas um conjunto representativo é mostrado. A faixa da direita contém 20 µ g da proteína total de RIPA-extraído do vermes mutantes de riscos-1 . (B) as intensidades de sinal correspondente foram quantificadas usando o ImageJ. As barras de erro representam o desvio padrão; n = 4. Clique aqui para ver uma versão maior desta figura.
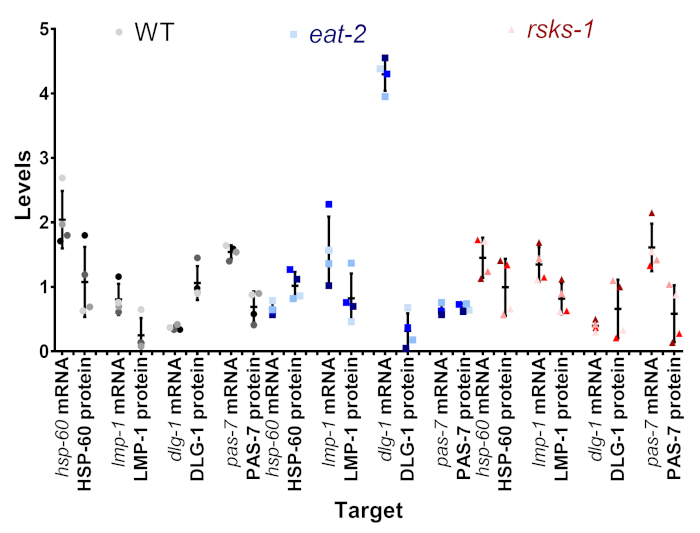
Figura 4: comparação de nível Intrasample mRNA e proteína. Os níveis do mRNA e proteínas de amostras individuais de um subconjunto de metas para cada estirpe estão alinhados. A cor é o representante da tabela 1. O cinza/preto é WT, o azul é comer-2, e o vermelho é riscos-1. mRNA e proteína do alvo mesmo são emparelhados lado a lado e separados por nematoide estirpe. Clique aqui para ver uma versão maior desta figura.
Discussão
Métodos para isolamentos biomolécula, como DNA, RNA e proteínas, muitas vezes são otimizados sem sobreposição técnica ou combinações. Isto é particularmente desfavorável quando amostras são difíceis de obter, que poderia levar a colheita de amostras nas mesmas condições em momentos diferentes. Dependendo dos caminhos celulares, repetições coletadas em momentos diferentes podem gerar variação. Este manuscrito oferece um procedimento para contornar este obstáculo, permitindo que o concorrente isolamento e purificação de cada biomolécula da mesma amostra de vermes, reduzindo as variações introduzidas através de técnicas de isolamento diferente, o momento da colheita da amostra, ou colheita desiguais. Controlar essas variáveis não só economiza tempo e recursos, mas também facilita a reprodutibilidade. Aqui, vamos demonstrar uma abordagem combinatória que evita RNA comprometedora e qualidade de proteína, embora com resultados variáveis com DNA. Preparações podem ser ainda mais otimizadas, usando os procedimentos de limpeza de DNA. Demonstramos a abordagem usando material de pilhas do nemátodo c. elegans e HeLa.
Trabalho anterior, explorando o transcriptoma e proteoma de N2 WT animais e comer-2 e riscos-1 mutantes têm oferecido insight sobre vários caminhos, incluindo mecanismos que estendem a vida útil de4,34,35 ,36. Em um esforço para investigar o mecanismo de restrição calórica em estendendo a vida, um isótopo estável de rotulagem por/com aminoácidos na célula cultura (SILAC) análise descobriu que comer-2 vermes têm uma downregulation global da síntese proteica global 36. os dados aqui apresentados são consistentes com esta conclusão, mesmo que os níveis do mRNA dos mesmos alvos são grandemente aumentados. Outro grupo objetivou identificar efetores da longevidade S6K-mediada e, assim, realizou uma tela de proteomic de riscos-1 vermes34. A partir dos dados de RNAseq encontrados-se com o estudo atual, nós identificamos pelo menos três genes que corroborar com proteínas identificadas a partir desta tela; MRP-1 e homologs do CPA e neuroligin (F07C4.12) foram descobertos como sendo diferencialmente expressos em vermes de riscos-1 em comparação com WT N234.
Os dados gerados usando esse método são consistentes com inquéritos anteriores multiomic. Os níveis do mRNA de nove destinos foram utilizados para prever os níveis de proteína em cada amostra. Dessas metas, muitos tinham níveis de proteína previsível com base nos níveis de RNAm. No entanto, havia discrepâncias notáveis entre os níveis do mRNA e proteínas. Importante, o protocolo aqui apresentado permite aos cientistas com confiança, avaliar e interpretar essas diferenças, removendo variabilidade intersample a recolha de mRNA e proteína pela mesma amostra. Além disso, comparamos os níveis de mRNA para os níveis de proteína recolhidos da mesma amostra ou coletados de uma amostra semelhante, mas diferente, colhida com RIPA. Mostramos que para um número de alvos, havia níveis muito baixos de proteína na amostra extraída de RIPA. Sem controle de variação intersample, seria impossível saber se essa diferença foi devido ao regulamento de diferencial de mRNA e proteína.
É importante ter em mente que existem protocolos otimizados especificamente para estas macromoléculas diferentes, então se uma análise transversal não é o objetivo final do experimento, então seria pertinente a empregar esses métodos em vez disso. Usando GTCp para isolar o DNA e proteína faz com que eles se tornem menos solúvel, que exigem a reconstituição do DNA em uma base fraca, como o NaOH e solubilizing a proteína em um buffer com uma alta concentração de detergente com aquecimento, correndo o risco de incompleto solubilização. Além disso, GTCp contém de guanidinium e ácido fenol, que inactiva enzimas como proteases, mas lentamente irá degradar proteína ao longo do tempo a não ser congelado. Será ao critério do pesquisador para decidir se a evasão dessas limitações vai valer a pena.
Importante, ao trabalhar com o RNA, uma técnica estéril, mantendo a amostra no gelo, a menos que indicado o contrário e o uso de descontaminação comercialmente disponível reagente é recomendado para manter o RNA intacto. Notavelmente, amostras maiores precisará mais GTCp para começar com, no qual cada solvente de extração também terão de ser ampliados. Este protocolo não requer qualquer manual homogeneização das amostras worm ou célula quando a quantidade correta de GTCp é usada. No contexto de isolamento de DNA, o rendimento é altamente dependente da proficiência para recuperar a camada orgânica (rosa). Finalmente, para melhorar a solubilização de proteínas durante o isolamento, aumentar o volume do buffer resolubilization ou adicionando outros detergentes além de SDS pode ser necessário. Na verdade, as proteínas de uma população apenas para adultos de vermes em vez de uma mistura de ovos, larvas e adultos são mais fáceis de dissolvem.
Em geral, usando este protocolo fornece uma abordagem integrada para os isolamentos biomolécula e facilita a interpretação de correlações, ou falta dela, entre os níveis do mRNA e da proteína que podem decorrer da colheita separadamente biomoléculas de diferentes amostras. Usar este método pode ajudar os cientistas a identificar corretamente os casos onde a tradução de mRNA, a proteína não é correlativo e pode levar a mais profunda investigação de mecanismos de regulação pós-traducional e posttranscriptional em diversas condições.
Divulgações
Os autores não têm nada para divulgar.
Agradecimentos
L.R.L foi financiado por concessões do NIH/NIA (R00 AG042494 e R01 AG051810), um Glenn Foundation para prêmio de pesquisa médica para pesquisa em mecanismos biológicos do envelhecimento e uma bolsa de faculdade Júnior da Federação Americana para pesquisa do envelhecimento. Os autores gostaria de agradecer seus comentários úteis na escrita deste manuscrito Anita Kumar e Shi Quan Wong.
Materiais
| Name | Company | Catalog Number | Comments |
| 4–15% Mini-PROTEAN® TGX Stain-Free™ Protein Gels | BIORAD | 4568084 | |
| Antibodies: | |||
| CePAS7-s | DHSB- U of Iowa | ||
| CeTAC1-s | DHSB- U of Iowa | ||
| DLG1-s | DHSB- U of Iowa | ||
| GRP78 | Novus Bio. | NBp1-06274 | |
| HRP Goat Anti-Mouse | Li-Cor | 926-80010 | |
| HRP Goat Anti-Rabbit | Li-Cor | 92680011 | |
| HSP60-s | DHSB- U of Iowa | ||
| LMP1-s | DHSB- U of Iowa | ||
| MRP-1 | Abcam | ab24102 | |
| Neuroligin 3 | Abcam | ab172798 | |
| β-actin | Millipore | MAB1501R | |
| ChemiDoc MP Imaging System | BIORAD | ||
| Chloroform | Fisher | C298-500 | Health hazard, Irritant, Toxic |
| Coomassie Brilliant Blue | ThermoSci | 20279 | |
| DC Protein assay | BIORAD | 500-0116 | |
| Epoch 2 microplate reader | BioTek | ||
| Ethanol (200 proof) | Fisher | 04-355-223 | Flammable, health hazard |
| HeLa cells | ATCC | CCL-2 | BSL2 |
| iScript Reverse Transcription Supermix | BIORAD | 1708840 | |
| Hydra microdispenser: Matrix Hydra | Robbins/ThermoFisher | ||
| Isopropanol | Fisher | A516-4 | Flammable, health hazard |
| M9 buffer: 3 g KH2PO4 6 g Na2HPO4 5 g NaCl Add H2O to 1 liter. Sterilize by autoclaving. After solution cools down, add 1 mL sterile 1 M MgSO4 | |||
| Laemmli Sample Buffer (2x) | BIORAD | 161-0737 | |
| NanoDrop One | ThermoSci | ||
| PAGE apparatus | BIORAD | ||
| Ponceau S | Alfa Aesar | J60744 | |
| Primers | IDT | 25 nmole DNA Oligo with Standard Desalting | |
| dlg-1: F (5'-GGTCCTACCA GGCAGTTGAG-3') R (5'-CACGTCCGTT AACCTCTCCC-3') | |||
| hsp-4: F (5'-AGAGGGCTTT GTCAACCCAG-3') R (5'-TCGTCAGGGT TGATTCCACG-3') | |||
| hsp-70: F (5'-CGGCATGTGA ACGTGCTAAG-3') R (5'-GAGCAGTTGA GGTCCTTCCC-3') | |||
| hsp-60: F (5'-ATTGAGCAAT CGACGAGCGA-3') R (5'-CAACACCTCC TCCTGGAACG-3') | |||
| lmp-1: F (5'-ACAACAACAC CGGACTCACG-3') R (5'-ATCGAGCTCC CACTCTTTGG-3') | |||
| tac-1: F (5'-AGTGGCAGGC AAAGTTCCTC-3') R (5'-TGAGCACCTT GATCTCGTCG-3') | |||
| pas-7: F (5'-GTACGCTCAA AAGGCTGTCG-3') R (5'-CTGAATCGGC ATTGGCTCAC-3') | |||
| mrp-1: F (5'-TTTGCCTTGC GCTTGTTCTG-3') R (5'-AGTTCCAGTG CGGAGCATAC-3') | |||
| F07C4.12: F (5'-TGCTGAGCAT GAAGGACTGT-3') R (5'-TGGCAATAGC TCCTCCGTTG-3') | |||
| HK Actin: F (5'-CTACGAACTT CCTGACGGACAAG-3') R (5'-CCGGCGGACT CCATACC-3') | |||
| HK cyn-1: F (5'-GTGTCACCAT GGAGTTGTTC-3') R (5'-TCCGTAGATT GATTCACCAC-3') | |||
| HK nhr-23: F (5'-CAGAAACACT GAAGAACGCG-3') R (5'-CGATCTGCAG TGAATAGCTC-3') | |||
| HK ama-1: F (5'-TGGAACTCTG GAGTCACACC-3') R (5'-CATCCTCCTT CATTGAACGG-3') | |||
| HK cdc-42: F (5'-CTGCTGGACA GGAAGATTACG-3') R (5'-CTCGGACATT CTCGAATGAAG-3') | |||
| HK pmp-3: F (5'-GTTCCCGTGT TCATCACTCAT-3') R (5'-ACACCGTCGA GAAGCTGTAGA-3') | |||
| LightCycler 96 qPCR machine | Roche | ||
| RIPA buffer: 10 mM Tris-Cl (pH 8.0) 1 mM EDTA 1% Triton X-100 0.2% SDS 140 mM NaCl 1 tablet of Roche protease inhibitor per 20 mL | |||
| SsoAdvanced Universal SYBR Supermix | BIORAD | 1725274 | |
| SuperSignal West Femto Max Sens Substrate | ThermoSci | 34095 | |
| Trans-Blot Transfer apparatus | BIORAD | ||
| Trans-Blot Turbo Transfer Pack | BIORAD | 170-4159 | |
| TRIzol reagent | Invitrogen | 15596026 | Health hazard (skin, eyes) |
| Worm strains: | Caenorhabditis Genetics Center (CGC) | ||
| N2 (wild type) | Caenorhabditis Genetics Center (CGC) | ||
| eat-2 (MAH95) | Caenorhabditis Genetics Center (CGC) | ||
| rsks-1 (VB633) | Caenorhabditis Genetics Center (CGC) |
Referências
- Rotroff, D. M., Motsinger-Reif, A. A. Embracing Integrative Multiomics Approaches. International Journal of Genomics. 2016, 1715985 (2016).
- Chen, R., Snyder, M. Promise of personalized omics to precision medicine. Wiley Interdisciplinary Reviews: System Biology and Medicine. 5 (1), 73-82 (2013).
- Anderson, L., Seilhamer, J. A comparison of selected mRNA and protein abundances in human liver. Electrophoresis. 18 (3-4), 533-537 (1997).
- Harvald, E. B., et al. Multi-omics Analyses of Starvation Responses Reveal a Central Role for Lipoprotein Metabolism in Acute Starvation Survival in C. elegans. Cell Systems. 5 (1), (2017).
- Griffin, T. J., et al. Complementary profiling of gene expression at the transcriptome and proteome levels in Saccharomyces cerevisiae. Molecular and Cell Proteomics. 1 (4), 323-333 (2002).
- Greenbaum, D., Colangelo, C., Williams, K., Gerstein, M. Comparing protein abundance and mRNA expression levels on a genomic scale. Genome Biology. 4 (9), 117 (2003).
- Lapierre, L. R., et al. The TFEB orthologue HLH-30 regulates autophagy and modulates longevity in Caenorhabditis elegans. Nature Communications. 4, 2267 (2013).
- Doherty, C. J., Kay, S. A. Circadian control of global gene expression patterns. Annual Reviews Genetics. 44, 419-444 (2010).
- Goya, M. E., Romanowski, A., Caldart, C. S., Benard, C. Y., Golombek, D. A. Circadian rhythms identified in Caenorhabditis elegans by in vivo long-term monitoring of a bioluminescent reporter. Proceedings of the National Academy of Sciences USA. 113 (48), E7837-E7845 (2016).
- Dalvin, L. A., Fautsch, M. P. Analysis of Circadian Rhythm Gene Expression With Reference to Diurnal Pattern of Intraocular Pressure in Mice. Investigative Ophthalmology and Visual Science. 56 (4), 2657-2663 (2015).
- Narayanan, A., Jacobson, M. P. Computational studies of protein regulation by post-translational phosphorylation. Current Opinion in Structural Biology. 19 (2), 156-163 (2009).
- Swatek, K. N., Komander, D. Ubiquitin modifications. Cell Research. 26 (4), 399-422 (2016).
- Cech, T. R., Steitz, J. A. The noncoding RNA revolution-trashing old rules to forge new ones. Cell. 157 (1), 77-94 (2014).
- Chomczynski, P., Sacchi, N. Single-step method of RNA isolation by acid guanidinium thiocyanate-phenol-chloroform extraction. Analytical Biochemistry. 162 (1), 156-159 (1987).
- Triant, D. A., Whitehead, A. Simultaneous Extraction of High-Quality RNA and DNA from Small Tissue Samples. Journal of Heredity. 100 (2), 246-250 (2009).
- Liu, X., Harada, S. RNA Isolation from Mammalian Samples. Current Protocols in Molecular Biology. 103 (1), 11-14 (2013).
- Hummon, A. B., Lim, S. R., Difilippantonio, M. J., Ried, T. Isolation and solubilization of proteins after TRIzol extraction of RNA and DNA from patient material following prolonged storage. Biotechniques. 42 (4), 467-470 (2007).
- Kopec, A. M., Rivera, P. D., Lacagnina, M. J., Hanamsagar, R., Bilbo, S. D. Optimized solubilization of TRIzol-precipitated protein permits Western blotting analysis to maximize data available from brain tissue. Journal of Neuroscience Methods. 280, 64-76 (2017).
- Stiernagle, T. Maintenance of C. elegans. WormBook. , (2006).
- Hoogewijs, D., Houthoofd, K., Matthijssens, F., Vandesompele, J., Vanfleteren, J. R. Selection and validation of a set of reliable reference genes for quantitative sod gene expression analysis in C. elegans. BMC Molecular Biology. 9 (1), 9 (2008).
- Mahmood, T., Yang, P. -. C. Western blot: technique, theory, and trouble shooting. North American Journal of Medical Sciences. 4 (9), 429-434 (2012).
- Peach, M., Marsh, N., MacPhee, D. J., Kurien, B. T., Scofield, R. H. Protein solubilization: Attend to the choice of lysis buffer. Protein Electrophoresis: Methods and Protocols. , 37-47 (2012).
- Lakowski, B., Hekimi, S. The genetics of caloric restriction in Caenorhabditis elegans. Proceedings of the National Academy of Science USA. 95 (22), 13091-13096 (1998).
- Pan, K. Z., et al. Inhibition of mRNA translation extends lifespan in Caenorhabditis elegans. Aging Cell. 6 (1), 111-119 (2007).
- Gaudet, P., Livstone, M. S., Lewis, S. E., Thomas, P. D. Phylogenetic-based propagation of functional annotations within the Gene Ontology consortium. Briefings in Bioinformatics. 12 (5), 449-462 (2011).
- Broeks, A., Gerrard, B., Allikmets, R., Dean, M., Plasterk, R. H. Homologues of the human multidrug resistance genes MRP and MDR contribute to heavy metal resistance in the soil nematode Caenorhabditis elegans. The EMBO Journal. 15 (22), 6132-6143 (1996).
- Hadwiger, G., Dour, S., Arur, S., Fox, P., Nonet, M. L. A Monoclonal Antibody Toolkit for C. elegans. PLoS One. 5 (4), e10161 (2010).
- Kostich, M., Fire, A., Fambrough, D. M. Identification and molecular-genetic characterization of a LAMP/CD68-like protein from Caenorhabditis elegans. Journal of Cell Science. 113 (14), 2595 (2000).
- Firestein, B. L., Rongo, C. DLG-1 is a MAGUK similar to SAP97 and is required for adherens junction formation. Molecular Biology of the Cell. 12 (11), 3465-3475 (2001).
- Heschl, M. F., Baillie, D. L. Characterization of the hsp70 multigene family of Caenorhabditis elegans. DNA. 8 (4), 233-243 (1989).
- Yoneda, T., et al. Compartment-specific perturbation of protein handling activates genes encoding mitochondrial chaperones. Journal of Cell Science. 117 (18), 4055 (2004).
- Takahashi, M., Iwasaki, H., Inoue, H., Takahashi, K. Reverse Genetic Analysis of the Caenorhabditis elegans 26S Proteasome Subunits by RNA Interference. Biological Chemistry. 383, 1263 (2002).
- Le Bot, N., Tsai, M. C., Andrews, R. K., Ahringer, J. TAC-1, a regulator of microtubule length in the C. elegans embryo. Current Biology. 13 (17), 1499-1505 (2003).
- McQuary, P. R., et al. C. elegans S6K Mutants Require a Creatine-Kinase-like Effector for Lifespan Extension. Cell Reports. 14 (9), 2059-2067 (2016).
- Larance, M., et al. Global Proteomics Analysis of the Response to Starvation in C. elegans . Molecular and Cell Proteomics. 14 (7), 1989-2001 (2015).
- Yuan, Y., et al. Enhanced energy metabolism contributes to the extended life span of calorie-restricted Caenorhabditis elegans. Journal of Biological Chemistry. 287 (37), 31414-31426 (2012).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados