このコンテンツを視聴するには、JoVE 購読が必要です。 サインイン又は無料トライアルを申し込む。
Method Article
核酸プローブを用いたヌクレアーゼ活性の運動スクリーニング
* これらの著者は同等に貢献しました
要約
変化したヌクレアーゼ活性は、バイオマーカーとしての可能性の根底にある異なるヒト条件に関連している。本論文で提示されるモジュラーで実施しやすいスクリーニング方法論により、疾患のバイオマーカーとしてヌクレアーゼ活性を利用するための特異的核酸プローブを選択することができる。
要約
ヌクレアーゼは、リボース糖を連結するホスホジエステル結合の加水分解を触媒することによって核酸を分解する酵素のクラスです。ヌクレアーゼは、ゲノム安定性の維持から病原体に対する保護の提供に至るまで、原核生物および真核生物において様々な重要な生理学的役割を果たします。変化したヌクレアーゼ活性は、細菌感染および癌を含むいくつかの病理学的状態に関連している。この結果、ヌクレアーゼ活性は、特定のバイオマーカーとして利用される大きな可能性を示している。しかしながら、この活性に基づく堅牢で再現性の高いスクリーニング方法は依然として極有である。
本明細書では、核酸プローブを基質として用いたヌクレアーゼ活性のスクリーニングを可能にする方法を紹介し、病理学的状態と健全な状態を区別する範囲を有する。この方法は、特異性を高め、反復的な方法で新しいプローブライブラリを設計する可能性を提供します。したがって、化学的に修飾された核酸の入手可能性を利用して、プローブの設計を強化された機能で改良するためには、複数のスクリーニングが必要です。提案された技術のかなりの可能性は、その柔軟性、高い再現性、および疾患状態に関連するヌクレアーゼ活性のスクリーニングのための汎用性にあります。この技術は、クリニックで大きな可能性を秘めた有望な診断ツールの開発を可能にすることが期待されます。
概要
ヌクレアーゼは、核酸分子の骨格構造を形成するホスホジエステル結合を切断することができる酵素のクラスです。ヌクレアーゼの膨大な多様性は、その分類をかなり困難にします。しかしながら、基質嗜好(デオキシリボ核酸(DNA)またはリボ核酸(RNA))、切断部位(エンドヌクレアーゼまたはエキソヌクレアーゼ)、または金属イオン依存性、とりわけ1のようなヌクレアーゼを記述するために使用されるいくつかの一般的な基準がある。ヌクレアーゼは、原核生物と真核生物の両方で基本的な役割を果たす高度に保存された触媒酵素であり、遺伝子編集ツール2として使用され続けている。また、DNAの維持と複製の基本的なアクターであり、ゲノムの安定性を維持し、校正プロセス3に参加するのに役立ちます。細菌において、例えば、ヌクレアーゼは重要な毒性因子として同定されており、宿主の免疫系4、5、6、7の有効性を低下させることにより細菌生存率を促進することができる、8.哺乳動物において、ヌクレアーゼは、アポトーシス9、ミトコンドリア生体形成および維持10および抗菌および抗ウイルス性自然免疫応答11の調停に関与することが示唆されている。当然のことながら、ヌクレアーゼ活性変化は、増強または欠如のいずれであっても、広範囲のヒト疾患に関与している。これらの疾患は、多種多様な癌12、13から心臓肥大10または自己免疫疾患14まで及んでいる。したがって、ヌクレアーゼは、ヒト状態の異種群のバイオマーカーとして興味深い候補となっている。実際、ヌクレアーゼは、黄色ブドウ球菌や大腸菌15などの特定の細菌剤によって引き起こされる感染症の検出に成功した診断ツールとしての可能性を既に示しています。16.多くの癌タイプにおいて、ブドウ球菌ヌクレアーゼドメイン含有タンパク質1(SND1)リボヌクレアーゼの発現は予後不良を示す17.膵臓癌患者において、リボヌクレアーゼI(RNaseI)血清レベルの上昇が報告されており、癌細胞表現型19に関連することが提案されている。心筋梗塞や不安定狭心症などの虚血性心状態において、デオキシリボヌクレアーゼI(DNaseI)血清レベルは、有効な診断マーカー20、21であることが示されている。
ヌクレアーゼ活性の世界的な青写真は、健康と疾患の状態で異なる可能性があると仮定されています。実際、最近の報告では、ヌクレアーゼ活性の違いを用いて、健康な表現型と癌性表現型22を区別したり、種特異的な方法で病原性細菌感染を同定したりしている。これらの知見は、ヌクレアーゼを疾患のバイオマーカーとして使用するための新しい道を開いた。従って、ヌクレアーゼ活性における疾患関連の相違を系統的に同定できる総合的なスクリーニング法の開発には必要性があり、これは新しい診断ツールの開発において重要な役割を有する可能性がある。
本明細書では、健康および不健康、または細胞または細菌の種類に特異的な活性を区別することができる感受性および特異的プローブを同定するための新しいin vitroスクリーニングアプローチ(図1)を導入し、説明する。核酸のモジュール性を利用して、異なる配列と化学の包括的なセットからなるクエンチされた蛍光オリゴヌクレオチドプローブの初期ライブラリを設計し、ライブラリ設計の重要なパラメータとして設計しました。これらのオリゴヌクレオチドプローブは、フルオロフォア(フルオレセインアミド、FAM)とクエンチャー(タイドクエンチャー2、TQ2)によってそれぞれ5'と3'末端に隣接している(表1)。この蛍光共鳴エネルギー移動(FRET)ベースの蛍光測定アッセイを用いて酵素分解の動態を測定することで、ヌクレアーゼ活性の微分パターンを区別する可能性のある候補プローブを同定することができた。健康または病気の状態に関連付けられています。.最適な候補プローブに基づいて新しいライブラリが作成される反復プロセスを設計し、その後のスクリーニングステップでより具体的な候補プローブを識別できるようにしました。さらに、このアプローチは、感受性を高めるためにヌクレアーゼの触媒性を利用する。これは、レポータープローブの活性化可能な性質と、代替抗体または低分子ベースのスクリーニング方法に対する重要な利点を表す、基質分子を継続的に処理するヌクレアーゼの能力を利用することによって達成される。
このアプローチは、健康と疾患の状態を区別することができる特定の核酸プローブを識別するための非常にモジュール的で柔軟で簡単なスクリーニングツールを提供し、新しい開発のための優れたプラットフォームを提供します将来の臨床適用のために適応することができる診断用具。このように、このアプローチは、この細菌の特異的同定のためにサルモネラ・チピムリウム(本明細書ではサルモネラ菌と呼ばれる)に由来するヌクレアーゼ活性を同定するために用いられた。以下のプロトコルでは、運動分析を用いて細菌ヌクレアーゼ活性をスクリーニングする方法について報告する。
プロトコル
1. オリゴヌクレオチドライブラリーの設計と調製
- 図書館設計
- 以下の基準に基づいて、全てのプローブに対して同じ長さで、二次構造形成を回避するために、8〜12-mer長さの間の急行蛍光オリゴヌクレオチドプローブのライブラリーを設計する。
- アデニン(A)、グアニン(G)、シトシン(C)およびチミン(T)/ウラシル(U)(DNAおよびRNAプローブを表1に含む)の組み合わせを含む少なくとも1つのDNAおよび1つのRNAランダム配列を含む。
注:表1に記載のDNAおよびRNA配列は、DNaseまたはRNaseのいずれかの未知のタイプのヌクレアーゼ活性を分類するための初期基質として有用であった。これらの2つの配列は、細菌および組織サンプルにおけるヌクレアーゼ活性プロファイルを検出する広い能力を示しているので、任意のスクリーニングの出発点として推奨される。これらのDNAおよびRNA配列でヌクレアーゼ活性が観察されない場合、追加のオリゴヌクレオチドの設計が必要である。 - 所定のヌクレアーゼ活性に対する配列依存性を決定する単一タイプのヌクレオチドからなるポリヌクレオチド配列を含む(DNA-PolyA、DNA-ポリT、DNA-PolyC、DNA-ポリG、RNA-ポリA、RNA-Poly U、RNA-ポリCおよびRNA-ポリGを表1に)
- 初期DNAおよびRNA配列と同じヌクレオチド残基の配列を使用して、2'-Fluooおよび2'-O-メチルのようなリボース糖の2'位で化学修飾を有するヌクレオシド類似体を含む配列を、ストリンジェントステップとして含むヌクレアーゼの選択性を高める。
- 完全に2'-フルオロヌクレオシド類似体または2'-O-メチルヌクレオシド類似体からなる完全に修飾された配列を含む(All 2'-FおよびAll 2'-OMe、表1)
- 未修飾天然プリンおよび2'-フルオロピリミジンヌクレオシド類似体からなるキメラ配列を含む(RNAPyr-2'F,表1)。
- 未修飾天然プリンおよび2'-O-メチルピリミジンヌクレオシド類似体からなるキメラ配列を含む(RNAPyr-2'OMe,表1)。
- 未修飾天然ピリミジンおよび2'-フルオロプリンヌクレオシド類似体からなるキメラ配列を含む(RNA Pur-2'F,表1)。
- 未修飾天然ピリミジンおよび2'-O-メチルプリンヌクレオシド類似体からなるキメラ配列を含む(RNA Pur-2'OMe,表1)。
- アデニン(A)、グアニン(G)、シトシン(C)およびチミン(T)/ウラシル(U)(DNAおよびRNAプローブを表1に含む)の組み合わせを含む少なくとも1つのDNAおよび1つのRNAランダム配列を含む。
- 以下の基準に基づいて、全てのプローブに対して同じ長さで、二次構造形成を回避するために、8〜12-mer長さの間の急行蛍光オリゴヌクレオチドプローブのライブラリーを設計する。
- オリゴヌクレオチドプローブの調製と貯蔵
注:オリゴヌクレオチドは、ホスホタミダイト法により合成される。合成後、オリゴヌクレオチドを高速液体クロマトグラフィー(HPLC)を用いて精製し、質量分析法で質量測定します。- 遠心分離機を用いて凍結乾燥オリゴヌクレオチドプローブをスピンダウンし、トリスEDTA(TE)緩衝液で希釈してヌクレアーゼの分解を防ぎます。各プローブの収率に応じて希釈量を決定し、500 pmol/μL濃度のストック溶液をレンダリングします。
- 凍結乾燥したオリゴヌクレオチドを4°Cまたは室温で短期間保存します。長期保存(数ヶ月から数年)の場合は、-20°Cで凍結乾燥オリゴヌクレオチドを保存します。TEの凍結乾燥オリゴヌクレオチドの懸濁液の懸濁時には、ストック溶液を-20°Cまたは、好ましくは-80°Cで保存してください。
2. 細菌培養
- 滅菌条件下で極低温貯蔵バイアルから1つの多孔質ガラスビーズを取ります。
- 個々の細菌コロニー(サルモネラ菌と大腸菌)を分離するには、除細動羊を添加したトリプティック・ソイ・アガー(TSA)培地を含むペトリ皿にビーズを直接ストリーキング/ローリングすることにより、象限法24を使用します。血液。
- 培養物を37°Cで24時間インキュベートする。
3. 上清準備
- 固体媒体から50 mLトリプティック・ソイ・ブロス(TSB)に単一のコロニーを移し、200 rpmで24時間37°Cでインキュベートする。
- 培養物(1:500)をTSB(サブカルチャー)で希釈し、37°Cで24時間200rpmで振とうインキュベーターでインキュベートします。
注:24時間のインキュベーション後、細菌培養物は109 CFU/mLより高い値に達する静止相にあることが予想されます。 - インキュベーション期間後、4,500 x gで30分間、培養物をキャップ付き滅菌チューブと遠心分離機に移します。
- 上清を収集し、すぐにそれを使用してください。または、上清を4°Cまたは-20°Cで保存してください。
4. ヌクレアーゼ活性アッセイ
- 作業ソリューションの準備
- 50 pmol/μLの最終作業濃度を希釈(1:10比)して、1.5 mLヌクレアーゼフリーマイクロ遠心管にプローブごとに20μLの作業溶液を調製します。そのために、MgCl2とCaCl2を含むリン酸緩衝液(PBS)の18μLをプローブ原液の2μLと混合します。
注:作動溶液では、オリゴヌクレオチドはヌクレアーゼ分解に対してより脆弱であるため、反応を設定する直前に作業溶液を調製することをお勧めします。 - フッ素を扱う際は、準備中に直接光の接触を避け、暗い(箔で包まれた)ままにしてください。
- 50 pmol/μLの最終作業濃度を希釈(1:10比)して、1.5 mLヌクレアーゼフリーマイクロ遠心管にプローブごとに20μLの作業溶液を調製します。そのために、MgCl2とCaCl2を含むリン酸緩衝液(PBS)の18μLをプローブ原液の2μLと混合します。
- 反応設定
- フッ素計(例えばサイテーション1)を37°Cに事前に温める。
- 各サンプル(TSB、大腸菌、サルモネラ菌)に対して1.5 mLヌクレアーゼフリーマイクロ遠心管のセット(1チューブ/プローブ)を準備し、それに応じてラベルを付けます。
- TSB滅菌培養媒体、サルモネラ上清または大腸菌上清(ステップ3.4から)の96μLを慎重に添加します。その後、それに応じてプローブ作業溶液の4 μL/チューブを追加します。室温でこの手順を実行します。
注:スクリーニングの第1ラウンドには10個のプローブが使用され、2回目は6つのプローブが使用されました。 - 上下にピペットを組んで十分に混ぜ合わせ、均質な溶液を得る。混合中にサンプルに気泡を導入することは避けてください。
- 各溶液(プローブ+上清または培養媒体)の95 μLを黒底部、非処理96ウェルプレートの別々のウェルにロードします。井戸の壁に近い先端で慎重に分配することにより、ローディング時に井戸内の気泡の形成を最小限に抑えます。
- プレートを蓋で覆います。蓋を目視で検査し、測定アーティファクトを引き起引き起ける可能性のあるペンマーキングやほこり粒子の蓄積を確認します。その場合は、新しいふたを交換してください。
- 測定設定
- ソフトウェアのショートカット アイコン (図 S1A)をクリックして、取得ソフトウェア (Gen5 3.05) を開きます。
- タスク マネージャ ウィンドウで [今すぐ読み取り]を選択し、[新規作成... ] を選択してキネティック測定プロトコルを作成します (図 S1B)。
- [プロシージャ]というタイトルのダイアログ ウィンドウで[温度を設定]をクリックし、[37°C]を選択します。[OK](図S1C)を押して設定を確認し、保存します。
- [プロシージャ]というタイトルのダイアログ ウィンドウで[開始 Kinetics]をクリックします。次に、ポップアップ ダイアログ ウィンドウの [キネティック ステップ]で、[実行時間]入力ボックスで [2 時間] を選択し、[間隔]入力ボックスで 2 分を選択します。[OK](図S1D)を押して設定を確認し、保存します。
- [プロシージャ]というタイトルのダイアログ ウィンドウで[読み取り]をクリックします。次に、ポップアップ ダイアログ ウィンドウの [読み取り方法]で、[検出方法] として[蛍光強度]、[読み取りタイプ]、[光学式]の [フィルタ]を選択します。[OK](図 S1E)を押して設定を確認し、保存します。
- [ステップの読み取り (Kinetic)]というタイトルのポップアップ ダイアログ ウィンドウで、[フィルタ セット]から[緑]を選択します。[OK]をクリックして設定を確認し、保存します (図 S2A)。
- [プロシージャ] というタイトルのダイアログ ウィンドウで、[ lid を使用する ] を選択し、[検証]をクリックして、作成されたプロトコルが有効であることを確認します。これはポップアップ ダイアログ ウィンドウで確認されます (図 S2B)。
- メニューバーでプロトコルを選択し、プロシージャを選択します(図S2C)。
- [プロシージャ]というタイトルのダイアログ ウィンドウで、測定するウェルを定義します (図 S2D)。
- ファイル名入力ボックスに実験の名前を入力します (図 S2E)。
- 蓋をしたプレートをプレートリーダーにロードします。プレートが正しい向きであることを確認します。
- ツールバーの「新規読み取り」ボタンをクリックして取得を開始します (図 S2F)。
- データ分析
- Plate 1 (図 S3A)という名前のダイアログ ウィンドウで、測定されたウェルのいずれかをクリックします。
- [井戸を選択]をクリックし、ウェル選択ダイアログウィンドウにすべての測定されたウェルを含めます。[OK]をクリックして設定を確認し、保存します。(図 S3B)
- [プレート 1]という名前のダイアログ ウィンドウでデータを選択し、集計結果を視覚化します (図 S3C)。
- コンテキスト メニューからクイック エクスポートを選択して、データをスプレッド シートにエクスポートします (図 S3C)。
- スプレッドシートで、各サンプルおよびプローブに応じてデータ列にラベルを付けます。
- 相対蛍光単位(RFU)対時間(x軸:反応の年表とy軸:RfUs)をプロットすることにより、マーカースタイルの線を使用して運動グラフを生成します。
注: グラフの生成に関する説明は、スプレッドシート プログラム (Excel など) を使用する場合に適用できます。ただし、他のプログラムを使用して、RFU と時間をプロットすることで、取得した生データからグラフを生成できます。 - 下記式25:rate=
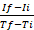 に従って各測定区間の酵素反応の速度を算出し、ここで最大間隔時間時点でRFU、Iiが最小間隔時間点でRFUである場合、 TfとTiは、それぞれ最大間隔の時間ポイントです。
に従って各測定区間の酵素反応の速度を算出し、ここで最大間隔時間時点でRFU、Iiが最小間隔時間点でRFUである場合、 TfとTiは、それぞれ最大間隔の時間ポイントです。 - 各カーブの最大値(Rmax)のレートを選択します。サンプルごとに複数のRmaxがある場合は、最も早い測定時間で発生するRを選択します。
- Rmaxと Rmaxが発生する時間間隔の平均値との比率を計算します。
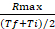
- 各プローブのサルモネラと大腸菌の速度係数の折り目差(FD)を計算します。
- 3 より大きい折り分け差の値は有意であると考えてください。
- 候補プローブとして、および次のスクリーニングラウンドでのライブラリ設計の基礎として、最も高い折り分差値(FD)を持つ有意なプローブを考慮してください。
5. ラウンドの選択基準(図1)
- 第1回審査
- DNA、RNAおよびポリヌクレオチドプローブを用いてヌクレアーゼ活性アッセイ(セクション4で説明)を行う。
- DNA候補プローブとRNA候補プローブの数と性能(FD値)を比較して、DNA化学またはRNA化学の好みを評価します。
- 図 1に示すように、候補プローブの最大数をレンダリングする核酸化学のタイプを選択します。
- 第2回審査
- 第1のスクリーニングラウンドで選択した核酸基質に基づいて設計された完全に修飾されたプローブおよびキメラプローブを用いて、ヌクレアーゼ活性アッセイ(セクション4で説明)を行う。
- 2'-フルオロおよび2'-O-メチルヌクレオシド類似体を含む配列に対する好みを評価し、2'-フルオロおよび2'-O-メチル修飾候補プローブについて得られた結果を比較します。
- 図 1に示すように、候補プローブの最大数をレンダリングするヌクレオシドアナログ変更のタイプを選択します。
結果
図 1は、この方法論の作業フローを 2 つのスクリーニングラウンドに分けて示しています。スクリーニングの第1ラウンドでは、5つのDNAプローブ(DNA、DNAポリA、DNAポリT、DNAポリCおよびDNAポリG)と5つのRNAプローブ(RNA、RNAポリA、RNAポリURNAポリCおよびRNAポリG)を使用しました。このスクリーニングラウンドの生データは補足表1に記載されている。第2ラウンドで?...
ディスカッション
ヌクレアーゼ活性の変化は、異なるタイプの癌および細菌感染を含む多種多様な疾患表現型に関連している。これらの改変は、条件14の原因物質であることが提案されるが、他の場合には有害な生理学的事象20または病原性薬剤16、26の結果である。当然のことながら、診断バイオマーカーとしてヌクレアーゼおよ...
開示事項
著者たちは何も開示する必要はない。
謝辞
著者たちは、原稿の慎重な改訂と貴重なアドバイスについて、ルイザ・I・ヘルナンデス(リンシェーピング大学)を認めたい。この研究は、クヌート・アンド・アリス・ワレンバーグ財団とスウェーデン政府のリンシェーピング大学先端機能材料に関する材料科学戦略研究領域(教員助成金SFO-Mat-LiU No. 2009-00971)によって支援されました。
資料
| Name | Company | Catalog Number | Comments |
| Black bottom, non-treated 96 well plate | Fisher Scientific | 10000631 | |
| Cytation1 | BioTek | CYT1FAV | |
| Eppendorf tubes | Thermofisher | 11926955 | |
| Escherichia coli | ATCC | 25922 | |
| Microbank cryogenic storage vial containing beads | Pro-Lab Diagnostics | 22-286-155 | |
| Nucleic acid probes | Biomers.net | # | |
| Phosphate Buffer Saline containing MgCl2 and CaCl2 | Gibco™ | 14040117 | |
| Salmonella enterica subs. Enterica | ATCC | 14028 | |
| Tris-EDTA | Fisher Scientific | 10647633 | |
| Tryptone Soya Agar with defibrinated sheep blood | Thermo Fisher Scientific | 10362223 | |
| Tryptic Soy Broth | Sigma Aldrich | 22092 |
参考文献
- Yang, W. Nucleases: diversity of structure, function and mechanism. Quarterly Reviews of Biophysics. 44 (1), 1-93 (2011).
- Adli, M. The CRISPR tool kit for genome editing and beyond. Nature Communication. 9 (1), 1911 (2018).
- Mason, P. A., Cox, L. S. The role of DNA exonucleases in protecting genome stability and their impact on ageing. Age (Dordr). 34 (6), 1317-1340 (2012).
- Berends, E. T., et al. Nuclease expression by Staphylococcus aureus facilitates escape from neutrophil extracellular traps. Journal of Innate Immunity. 2 (6), 576-586 (2010).
- Kiedrowski, M. R., et al. Staphylococcus aureus Nuc2 is a functional, surface-attached extracellular nuclease. PLoS One. 9 (4), 95574 (2014).
- Morita, C., et al. Cell wall-anchored nuclease of Streptococcus sanguinis contributes to escape from neutrophil extracellular trap-mediated bacteriocidal activity. PLoS One. 9 (8), 103125 (2014).
- Hasegawa, T., et al. Characterization of a virulence-associated and cell-wall-located DNase of Streptococcus pyogenes. Microbiology. 156, 184-190 (2010).
- Moon, A. F., et al. Structural insights into catalytic and substrate binding mechanisms of the strategic EndA nuclease from Streptococcus pneumoniae. Nucleic Acids Research. 39 (7), 2943-2953 (2011).
- Li, L. Y., Luo, X., Wang, X. Endonuclease G is an apoptotic DNase when released from mitochondria. Nature. 412 (6842), 95-99 (2001).
- McDermott-Roe, C., et al. Endonuclease G is a novel determinant of cardiac hypertrophy and mitochondrial function. Nature. 478 (7367), 114-118 (2011).
- Wang, Y. T., et al. A link between adipogenesis and innate immunity: RNase-L promotes 3T3-L1 adipogenesis by destabilizing Pref-1 mRNA. Cell Death & Disease. 7 (11), 2458 (2016).
- Li, C. L., Yang, W. Z., Shi, Z., Yuan, H. S. Tudor staphylococcal nuclease is a structure-specific ribonuclease that degrades RNA at unstructured regions during microRNA decay. RNA. 24 (5), 739-748 (2018).
- Wan, L., et al. MTDH-SND1 interaction is crucial for expansion and activity of tumor-initiating cells in diverse oncogene- and carcinogen-induced mammary tumors. Cancer Cell. 26 (1), 92-105 (2014).
- Keyel, P. A. Dnases in health and disease. Developmental Biology. 429 (1), 1-11 (2017).
- Hernandez, F. J., et al. Noninvasive imaging of Staphylococcus aureus infections with a nuclease-activated probe. Nature Medicine. 20 (3), 301-306 (2014).
- Flenker, K. S., et al. Rapid Detection of Urinary Tract Infections via Bacterial Nuclease Activity. Molecular Therapy. 25 (6), 1353-1362 (2017).
- Kannan, N., Eaves, C. J. Tipping the balance: MTDH-SND1 curbs oncogene-induced apoptosis and promotes tumorigenesis. Cell Stem Cell. 15 (2), 118-120 (2014).
- Kemmer, T. P., Malfertheiner, P., Buchler, M., Kemmer, M. L., Ditschuneit, H. Serum ribonuclease activity in the diagnosis of pancreatic disease. International Journal of Pancreatology. 8 (1), 23-33 (1991).
- Fernandez-Salas, E., Peracaula, R., Frazier, M. L., de Llorens, R. Ribonucleases expressed by human pancreatic adenocarcinoma cell lines. European Journal of Biochemistry. 267 (5), 1484-1494 (2000).
- Fujibayashi, K., et al. Serum deoxyribonuclease I activity can be a useful diagnostic marker for the early diagnosis of unstable angina pectoris or non-ST-segment elevation myocardial infarction. Journal of Cardiology. 59 (3), 258-265 (2012).
- Kawai, Y., et al. Diagnostic use of serum deoxyribonuclease I activity as a novel early-phase marker in acute myocardial infarction. Circulation. 109 (20), 2398-2400 (2004).
- Hernandez, L. I., Ozalp, V. C., Hernandez, F. J. Nuclease activity as a specific biomarker for breast cancer. Chemical Communication (Cambridge). 52 (83), 12346-12349 (2016).
- Machado, I., et al. Rapid and specific detection of Salmonella infections using chemically modified nucleic acid probes. Analytica Chimica Acta. 1054, 157-166 (2019).
- Sanders, E. R. Aseptic laboratory techniques: plating methods. Journal of Visualized Experiment. (63), e3064 (2012).
- Worthington Biochemical Corporation. . Manual of Clinical Enzyme Measurements. , (1972).
- Burghardt, E. L., et al. Rapid, Culture-Free Detection of Staphylococcus aureus Bacteremia. PLoS One. 11 (6), 0157234 (2016).
- Bibette, J. Gaining confidence in high-throughput screening. Proceedings of the National Academy of Science U. S. A. 109 (3), 649-650 (2012).
- Hernandez, L. I., Machado, I., Schafer, T., Hernandez, F. J. Aptamers overview: selection, features and applications. Current Topics in Medicinal Chemistry. 15 (12), 1066-1081 (2015).
- Pande, J., Szewczyk, M. M., Grover, A. K. Phage display: concept, innovations, applications and future. Biotechnology Advances. 28 (6), 849-858 (2010).
- Skerra, A. Alternative binding proteins: anticalins - harnessing the structural plasticity of the lipocalin ligand pocket to engineer novel binding activities. FEBS Journal. 275 (11), 2677-2683 (2008).
- Ku, T. H., et al. Nucleic Acid Aptamers: An Emerging Tool for Biotechnology and Biomedical Sensing. Sensors (Basel). 15 (7), 16281-16313 (2015).
- Jayasena, S. D. Aptamers: an emerging class of molecules that rival antibodies in diagnostics. Clinical Chemistry. 45 (9), 1628-1650 (1999).
- Gray, B. P., Brown, K. C. Combinatorial peptide libraries: mining for cell-binding peptides. Chemical Reviews. 114 (2), 1020-1081 (2014).
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請さらに記事を探す
This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved