È necessario avere un abbonamento a JoVE per visualizzare questo. Accedi o inizia la tua prova gratuita.
Method Article
Screening cinetico dell'attività nucleana utilizzando sonde con acido Nucleic
* Questi autori hanno contribuito in egual misura
In questo articolo
Riepilogo
L'attività di nucleasi alterata è stata associata a diverse condizioni umane, alla base del suo potenziale come biomarcatore. La metodologia di screening modulare e facile da implementare presentata in questo documento consente di selezionare specifiche sonde di acido nucleico per sfruttare l'attività nuclease come biomarcatore della malattia.
Abstract
Le nucleasi sono una classe di enzimi che scompongono gli acidi nucleici catalizzare l'idrolisi dei legami di fosforestro che legano gli zuccheri ribosio. Le nucleasi mostrano una varietà di ruoli fisiologici vitali negli organismi procariotici ed eucarioti, che vanno dal mantenimento della stabilità del genoma alla protezione contro gli agenti patogeni. L'attività di nucleasi alterata è stata associata a diverse condizioni patologiche, tra cui infezioni batteriche e cancro. A tal fine, l'attività di nucleasi ha mostrato un grande potenziale per essere sfruttata come un biomarcatore specifico. Tuttavia, un metodo di screening robusto e riproducibile basato su questa attività rimane altamente auspicabile.
Qui, introduciamo un metodo che consente lo screening per l'attività nuclease utilizzando sonde di acido nucleico come substrati, con l'ambito di differenziare tra condizioni patologiche e sane. Questo metodo offre la possibilità di progettare nuove librerie di probe, con crescente specificità, in modo iterativo. Pertanto, sono necessari più cicli di screening per affinare il design delle sonde con caratteristiche migliorate, sfruttando la disponibilità di acidi nucleici modificati chimicamente. Il notevole potenziale della tecnologia proposta risiede nella sua flessibilità, alta riproducibilità e versatilità per lo screening dell'attività di nucleasi associata alle condizioni della malattia. Si prevede che questa tecnologia consentirà lo sviluppo di promettenti strumenti diagnostici con un grande potenziale nella clinica.
Introduzione
Le nucleasi sono una classe di enzimi in grado di fendere i legami di fosforestro che formano la struttura della spina dorsale delle molecole di acido nucleico. La grande diversità di nucleasi rende la loro classificazione piuttosto difficile. Tuttavia, ci sono alcuni criteri comuni utilizzati per descrivere le nucleasi, come la preferenza del substrato (acido deossiribonucleico (DNA) o acido ribonucleico (RNA), sito di scissione (endonucleases o esonucleasi), o dipendenza da ioni onani metallici, tra gli altri1. Le nucleasi sono enzimi catalitici altamente conservati che hanno ruoli fondamentali in entrambi gli organismi procariotici ed eucarioti e sono stati utilizzati, e continuano ad essere utilizzati, come strumenti di editing genico2. Sono anche attori fondamentali nella manutenzione e replicazione del DNA, contribuendo a mantenere la stabilità del genoma e partecipando ai processi di proof-reading3. Nei batteri, ad esempio, le nucleasi sono state identificate come importanti fattori di virulenza, in grado di promuovere la sopravvivenza batterica riducendo l'efficacia del sistema immunitario dell'ospite4,5,6,7 ,8. Nei mammiferi, le nucleasi sono state suggerite per essere implicate nell'apoptosi9, biogenesi mitocondriale e manutenzione10 e mediazione delle risposte immunitarie innate antibatteriche e antivirali11. Non sorprende che le alterazioni dell'attività di nucleamento, sia in miglioramento che in mancanza, siano state implicate in una vasta gamma di malattie umane. Queste malattie vanno da un'ampia varietà di tumori12,13 a ipertrofia cardiaca10 o malattie autoimmuni14. Pertanto, le nucleasi sono diventate candidati interessanti come biomarcatori per un gruppo eterogeneo di condizioni umane. Infatti, le nucleasi hanno già mostrato il loro potenziale come strumenti diagnostici di successo per il rilevamento di infezioni causate da specifici agenti batterici, come Staphylococcus aureus o Escherichia coli15, 16.In molti tipi di cancro, l'espressione della proteina ribonuclease nuclease nuclease stafilocca del dominio di nuclease 1 (SND1) è indicativa di prognosi infausta17. Nei pazienti affetti da cancro al pancreas, sono stati riportati18 livelli elevati di siero ribonuclease I (RNase I) e proposti di essere associati a fenotipi cellulari cancerosi19. In condizioni cardiache ischemiche, come l'infarto miocardico o l'angina pectoris instabile, deoxyribonuclease I (DNase I) i livelli del siero sono stati indicati come un valido marcatore diagnostico20,21.
È stato ipotizzato che il progetto globale dell'attività nuclease potrebbe essere diverso negli stati sani e malati. Infatti, recenti rapporti hanno utilizzato differenze nell'attività nuclease per distinguere tra fenotipi sani e cancerosi22 o per identificare le infezioni batteriche patogene in modo specifico per specie15,23. Questi risultati hanno aperto una nuova strada per l'uso di nucleasi come biomarcatori della malattia. Pertanto, esiste la necessità di sviluppo di un metodo di screening completo in grado di identificare sistematicamente le differenze associate alle malattie nell'attività nucleana, che possono essere di fondamentale importanza nello sviluppo di nuovi strumenti diagnostici.
In questo articolo, introduciamo e descriviamo un nuovo approccio di screening in vitro (Figura 1) per identificare sonde sensibili e specifiche in grado di discriminare l'attività nucleasea in sano e malsano, o un'attività specifica di un tipo di cellula o batteri. Approfittando della modularità degli acidi nucleici, abbiamo progettato una libreria iniziale di sonde oligonucleotidi fluorescenti quesinella costituita da un insieme completo di sequenze e chimiche diverse, entrambe parametri importanti per la progettazione della biblioteca. Queste sonde oligonucleotide sono affiancate da un fluoroforo (fluorescein amidite, FAM) e da un quencher (quencher di marea 2, TQ2) rispettivamente alle estremità 5' e 3'(tabella 1). Utilizzando questo saggio fluorometrico basato sul trasferimento di energia a risonanza fluorescente (FRET) per misurare la cinetica della degradazione enzima, siamo stati in grado di identificare le sonde candidate con il potenziale di discriminare i modelli differenziali di attività nucleana associati a stati sani o malati. Abbiamo progettato un processo iterativo, in cui vengono create nuove librerie sulla base delle migliori sonde candidate, che consente l'identificazione di sonde candidate sempre più specifiche nelle successive fasi di screening. Inoltre, questo approccio sfrutta la natura catalitica delle nucleasi per aumentare la sensibilità. Ciò si ottiene sfruttando la natura attivabile delle sonde reporter e la capacità delle nuclesie di elaborare continuamente le molecole di substrato, entrambi rappresentando vantaggi chiave rispetto agli anticorpi alternativi o ai metodi di screening a base di piccole molecole.
Questo approccio offre uno strumento di screening altamente modulare, flessibile e facile da implementare per l'identificazione di specifiche sonde di acido nucleico in grado di discriminare gli stati sani e le malattie, e un'eccellente piattaforma per lo sviluppo di nuovi strumenti diagnostici che possono essere adattati per future applicazioni cliniche. Come tale, questo approccio è stato utilizzato per identificare l'attività di nuclease derivata da Salmonella Typhimurium (qui chiamata Salmonella) per l'identificazione specifica di questo batterio. Nel seguente protocollo, riportiamo un metodo per lo screening dell'attività nucleale batterica utilizzando l'analisi cinetica.
Protocollo
1. Progettazione e preparazione della libreria Oligonucleotide
- Progettazione della libreria
- Sulla base dei seguenti criteri, progettare una libreria di sonde oligonucleotide dissedate tra 8 e 12-mer lunghe per evitare la formazione secondaria della struttura, con uguale lunghezza per tutte le sonde.
- Includere almeno un DNA e una sequenza casuale di RNA contenente una combinazione di adenina (A), guanina (G), citosina (C) e timina (T)/uracil (U) (sondedi DNA e RNA nella tabella 1 ).
NOTA: Le sequenze di DNA e RNA descritte nella Tabella 1 sono state utili come substrati iniziali per classificare un tipo sconosciuto di attività nucleana, DNase o RNase. Queste due sequenze sono raccomandate come punto di partenza per qualsiasi screening, in quanto hanno dimostrato un'ampia capacità di rilevare i profili di attività nuclease nei campioni di batteri e tessuti. Se l'attività nucleata non viene osservata con queste sequenze di DNA e RNA, è necessaria la progettazione di oligonucleotidi aggiuntivi. - Includere sequenze polinucleotidiche costituite da un singolo tipo di nucleotide per determinare la dipendenza della sequenza per una data attività nucleale (DNA-Poly A, DNA-Poly T, DNA-Poly C, DNA-Poly G, RNA-Poly A, RNA-Poly U, RNA-Poly C e RNA-Poly G nella tabella 1).
- Utilizzando la stessa sequenza di residui nucleotidi delle sequenze iniziali di DNA e RNA, includere sequenze che contengono analoghi nucleoside che ospitano modifiche chimiche alla posizione 2' dello zucchero ribosio, come 2'-Fluoro e 2'-O-Methyl, come passo rigoroso per aumentare la selettività delle nucleasi.
- Includere sequenze completamente modificate costituite interamente da analoghi nucleoside 2'-Fluoro o analoghi nucleoside 2'-O-Methyl (Tutti i 2'-F e Tutti i 2'-OMe, Tabella 1)
- Includere sequenze chihimericicostituite da purine naturali non modificate e analoghi 2'-Fluoro pyrimidine nucleoside (RNA Pyr-2'F, Tabella 1).
- Includere sequenze chihimerici costituite da purine naturali non modificate e analoghi nucleoside 2'-O-Methyl (RNA Pyr-2'OMe, Tabella 1).
- Includere sequenze chihimerici costituite da pirimidine naturali non modificate e analoghi di 2'-Fluoro purine (RNA Pur-2'F, Tabella 1).
- Includere sequenze chihimerici costituite da pirimidine naturali non modificate e analoghi di 2'-O-Methyl purina (RNA Pur-2'OMe, Tabella 1).
- Includere almeno un DNA e una sequenza casuale di RNA contenente una combinazione di adenina (A), guanina (G), citosina (C) e timina (T)/uracil (U) (sondedi DNA e RNA nella tabella 1 ).
- Sulla base dei seguenti criteri, progettare una libreria di sonde oligonucleotide dissedate tra 8 e 12-mer lunghe per evitare la formazione secondaria della struttura, con uguale lunghezza per tutte le sonde.
- Preparazione e stoccaggio della sonda Oligonucleotide
NOTA: Gli oligonucleotidi sono sintetizzati dal metodo fosforamidite. Dopo la sintesi, gli oligonucleotidi vengono purificati utilizzando la cromatografia liquida ad alte prestazioni (HPLC) e la massa viene misurata dalla spettrometria di massa.- Abbassare le sonde oligonucleotidi loofilizzate usando una centrifuga e diluirle nel buffer Tris-EDTA (TE) per prevenire la degradazione della nucleasi. Determinare il volume di diluizione in base alla resa di ogni sonda per eseguire il rendering di una soluzione di stock con una concentrazione di 500 pmol/l.
- Conservare oligonucleotidi linfofiloti a 4 gradi centigradi o a temperatura ambiente per un breve periodo. Per lo stoccaggio a lungo termine (da mesi ad anni), conservare gli oligonucleotidi liofilizzati a -20 gradi centigradi. Dopo la sospensione degli oligonucleotidi liofilizzati in TE, conservare le soluzioni azionarie a -20 gradi centigradi o, preferibilmente, a -80 gradi centigradi.
2. Cultura batterica
- Prendere una perla di vetro poroso dalla fiala di stoccaggio criogenico in condizioni sterili.
- Per isolare singole colonie batteriche (Salmonella ed E. coli), utilizzare il metodo quadrante24 striature / rotolamento del tallone direttamente su un piatto Petri contenente Tryptic Soy Agar (TSA) medium integrato con pecora defibrinated sangue.
- Incubare la coltura a 37 gradi centigradi per 24 ore.
3. Preparazione supernatante
- Trasferire una singola colonia da supporti solidi a 50 mL Tryptic Soy Broth (TSB) e incubare a 37 s per 24 h a 200 rpm.
- Diluire la coltura (1:500) in TSB (sottocoltura) e incubare a 37 gradi centigradi per 24 h a 200 giri in un'incubatrice tremante.
NOTA: Dopo 24 h di incubazione le colture batteriche sono in fase stazionaria raggiungendo valori superiori a 109 CFU/mL. - Dopo il periodo di incubazione, trasferire le colture a tubi sterili con tappo e centrifugare a 4.500 x g per 30 min.
- Raccogliere il supernatante e usarlo immediatamente. In alternativa, conservare i supernatanti a 4 o -20 gradi centigradi.
4. Saggio sull'attività di Nuclease
- Preparazione di soluzioni di lavoro
- Preparare una soluzione di lavoro di 20 l l per ogni sonda in tubi di microcentrifuga liberi da 1,5 ml di diluindo (1:10 rapporto) la soluzione di stock (500 pmol/L) per una concentrazione di lavoro finale di 50 pmol / l. A tale scopo, mescolare 18 L di Phosphate Buffer Saline (PBS) contenente MgCl2 e CaCl2 con 2 L di soluzione di sonda.
NOTA: Essere consapevoli del fatto che nella soluzione di lavoro, gli oligonucleotidi sono più vulnerabili alla degradazione di nucleato, quindi, si consiglia di preparare le soluzioni di lavoro appena prima di impostare la reazione. - Evitare il contatto diretto della luce durante la preparazione e tenere al buio (avvolto in un foglio), quando si tratta di fluorofori.
- Preparare una soluzione di lavoro di 20 l l per ogni sonda in tubi di microcentrifuga liberi da 1,5 ml di diluindo (1:10 rapporto) la soluzione di stock (500 pmol/L) per una concentrazione di lavoro finale di 50 pmol / l. A tale scopo, mescolare 18 L di Phosphate Buffer Saline (PBS) contenente MgCl2 e CaCl2 con 2 L di soluzione di sonda.
- Impostazione della reazione
- Preriscaldare il fluorometro (ad es. Citazione 1) a 37 gradi centigradi.
- Preparare un set (un tubo/sonda) di 1,5 mL di tubi di microcentrifuga liberi da nucleato per ogni campione (TSB, E. coli e Salmonella)ed etichettare di conseguenza.
- Aggiungere con cura 96 -L di TSB sterile culture media, Salmonella supernatant o E. coli supernatant (dal passo 3.4.). Successivamente, aggiungere di conseguenza 4 soluzione di lavoro sonda/tubo della sonda. Eseguire questo passaggio a temperatura ambiente.
NOTA: sono state utilizzate 10 sonde per il primo round dello screening e 6 sonde per il secondo round. - Mescolare accuratamente pipetting su e giù per ottenere una soluzione omogenea. Evitare di introdurre bolle d'aria nei campioni durante la miscelazione.
- Caricare 95 l di ogni soluzione (sonda - supernatante o supporto di coltura) in un pozzo separato di un fondo nero, non trattato 96 pozzetto. Ridurre al minimo la formazione di bolle nei pozzi al momento del carico erogando con attenzione con la punta vicino alla parete del pozzo.
- Coprire la piastra con il coperchio. Ispezionare visivamente il coperchio e verificare la presenza di segni della penna o dell'accumulo di particelle di polvere che possono introdurre artefatti di misurazione. Sostituire il coperchio per uno nuovo, se questo è il caso.
- Configurazione della misurazione
- Aprire il software di acquisizione (Gen5 3.05) facendo clic sull'icona del collegamento software (Figura S1A).
- Selezionare Leggi ora nella finestra di gestione delle attività e scegliere Nuovo... per creare il protocollo di misurazione cinetica (Figura S1B).
- Fare clic su Imposta temperatura nella finestra di dialogo intitolata Procedura e selezionare 37 . Confermare e salvare le impostazioni premendo OK (Figura S1C).
- Fare clic su Avvia Kinetics nella finestra di dialogo intitolata Procedura. Quindi, nella finestra di dialogo popup, intitolata Passo Kinetico, selezionare 2 h nella casella di input Tempo di esecuzione e 2 min nella casella di input Intervallo. Confermare e salvare le impostazioni premendo OK (Figura S1D).
- Fare clic su Lettura nella finestra di dialogo intitolata Procedura. Quindi nella finestra di dialogo pop-up, intitolata Metododi lettura , selezionare Intensità di fluorescenza come metodo di rilevamento, Endpoint/Kinetic come tipo di lettura e Filtri come tipo di ottica. Confermare e salvare le impostazioni premendo OK (Figura S1E).
- Nella finestra di dialogo a comparsa intitolata Read Step (Kinetic), selezionare Verde dal gruppo difiltri . Confermare e salvare le impostazioni premendo OK (Figura S2A).
- Nella finestra di dialogo intitolata Procedura, selezionare Usa coperchio e fare clic su Convalida per assicurarsi che il protocollo creato sia valido. Ciò è confermato da una finestra di dialogo popup (Figura S2B).
- Selezionare protocollo nella barra dei menu e scegliere la procedura (Figura S2C).
- Nella finestra di dialogo denominata Procedura, definire i pozzi da misurare (Figura S2D).
- Immettere il nome dell'esperimento nella casella di input del nome file (Figura S2E).
- Caricare la piastra con il coperchio nel lettore di piastre. Assicurarsi che la piastra sia nel giusto orientamento.
- Avviare l'acquisizione facendo clic sul pulsante Leggi nuovo nella barra degli strumenti (Figura S2F).
- Analisi dei dati
- Fare clic su uno dei pozze misurati nella finestra di dialogo denominata Plate 1 (Figura S3A).
- Fare clic su Seleziona pozzi e includere tutti i pozzi misurati nella finestra di dialogo di selezione dei pozzi. Confermare e salvare le impostazioni premendo OK. (Figura S3B).
- Selezionare i dati nella finestra di dialogo denominata Plate 1 per visualizzare i risultati visualizzati (Figura S3C).
- Esportare i dati in un foglio di pagine affiancate selezionando Esportazione rapida dal menu di scelta rapida (Figura S3C).
- Nel foglio di diffusione, etichettare le colonne di dati di conseguenza per ogni campione e sonda.
- Generare grafici cinetici, utilizzando la linea con lo stile dei marcatori, tracciando le unità di fluorescenza relative (RFU) rispetto al tempo (asse x: sequenza temporale della reazione e dell'asse y: RRU).
NOTA: la descrizione della generazione di un grafico è applicabile quando si utilizzano programmi per fogli di calcolo (ad esempio Excel). Tuttavia, qualsiasi altro programma può essere utilizzato per generare grafici dai dati grezzi ottenuti, tracciando RFU rispetto al tempo. - Calcolare la velocità della reazione ezimatica per ogni intervallo
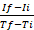 misurato in base alla seguente formula25: rate , dove If è RFU al punto di tempo intervallo massimo e Ii è RFU al punto di tempo intervallo minimo, e Tf e Ti sono rispettivamente i punti di tempo a intervalli massimi e minimi.
misurato in base alla seguente formula25: rate , dove If è RFU al punto di tempo intervallo massimo e Ii è RFU al punto di tempo intervallo minimo, e Tf e Ti sono rispettivamente i punti di tempo a intervalli massimi e minimi. - Selezionare la velocità con il valore più alto (Rmax) per ogni curva. Se è presente più di unR max per campione, selezionare quello che si verifica al primo punto del tempo di misurazione.
- Calcolare il rapporto tra Rmax e il valore medio dell'intervallo di tempo in cui si verifica Rmax: coefficiente di tasso
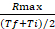
- Calcolare la differenza di piegatura (FD) tra il coefficiente di tasso di Salmonella e E. coli per ogni sonda.
- Considerare significativo un valore di differenza di piegatura superiore a 3.
- Considerare le sonde significative con i valori di differenza di piegatura più alti (FD) come sonde candidate e come base per la progettazione della libreria nel successivo ciclo di screening.
5. Screening dei criteri di selezione dei round(Figura 1)
- Primo ciclo di screening
- Eseguire il saggio di attività nuclease (come descritto nella sezione 4) utilizzando sonde DNA, RNA e polinucleotide.
- Valutare la preferenza per la chimica del DNA o della chimica dell'RNA confrontando il numero e le prestazioni (valore FD) delle sonde candidate al DNA e all'RNA.
- Selezionare il tipo di chimica dell'acido nucleico che rende il maggior numero di sonde candidate, come illustrato nella Figura 1.
- Secondo ciclo di screening
- Eseguire il saggio di attività nuclease (come descritto nella sezione 4) utilizzando sonde completamente modificate e sonde chimeriche progettate in base al substrato acido nucleico selezionato nel primo ciclo di screening.
- Valutare la preferenza verso sequenze contenenti 2'-Fluoro e 2'-O-Methyl nucleoside analogici confrontando i risultati ottenuti per le sonde candidate modificate 2'-Fluoro e 2'-O-Methyl.
- Selezionare il tipo di modifica analogica nucleoside per il rendering del maggior numero di sonde candidate, come illustrato nella Figura 1.
Risultati
La figura 1 mostra il flusso di lavoro di questa metodologia, suddivisa in due cicli di screening. Nel primo ciclo di screening, abbiamo utilizzato 5 sonde di DNA (DNA, DNA Poly A, DNA Poly T, DNA Poly C e DNA Poly G) e anche 5 sonde di RNA (RNA, RNA Poly A, RNA Poly U RNA Poly C e RNA Poly G). I dati grezzi di questo ciclo di screening sono riportati nella tabella supplementari 1. Nel secondo round, le sonde modificate chimicamente sono state sintetizzate sostituendo la seq...
Discussione
Le alterazioni dell'attività di nuclease sono state associate a un'ampia varietà di fenotipi della malattia, tra cui diversi tipi di cancro e infezioni batteriche. Queste alterazioni sono proposte come l'agente causale di una condizione14, mentre in altri casi sono la conseguenza di un evento fisiologico dannoso20 o agente patogeno16,26. Non sorprende che i tentativi di utilizzare nucleasi e attività di nucleasi ...
Divulgazioni
Gli autori non hanno nulla da rivelare.
Riconoscimenti
Gli autori desiderano riconoscere Luiza I. Hernandez (Università di Linkaping) per la sua attenta revisione del manoscritto e preziosi consigli. Questo lavoro è stato sostenuto dalla Fondazione Knut and Alice Wallenberg e dalla Swedish Government Strategic Research Area in Materials Science on Advanced Functional Materials presso l'Università di Link-ping (Faculty Grant SFO-Mat-LiU n. 2009-00971).
Materiali
| Name | Company | Catalog Number | Comments |
| Black bottom, non-treated 96 well plate | Fisher Scientific | 10000631 | |
| Cytation1 | BioTek | CYT1FAV | |
| Eppendorf tubes | Thermofisher | 11926955 | |
| Escherichia coli | ATCC | 25922 | |
| Microbank cryogenic storage vial containing beads | Pro-Lab Diagnostics | 22-286-155 | |
| Nucleic acid probes | Biomers.net | # | |
| Phosphate Buffer Saline containing MgCl2 and CaCl2 | Gibco™ | 14040117 | |
| Salmonella enterica subs. Enterica | ATCC | 14028 | |
| Tris-EDTA | Fisher Scientific | 10647633 | |
| Tryptone Soya Agar with defibrinated sheep blood | Thermo Fisher Scientific | 10362223 | |
| Tryptic Soy Broth | Sigma Aldrich | 22092 |
Riferimenti
- Yang, W. Nucleases: diversity of structure, function and mechanism. Quarterly Reviews of Biophysics. 44 (1), 1-93 (2011).
- Adli, M. The CRISPR tool kit for genome editing and beyond. Nature Communication. 9 (1), 1911 (2018).
- Mason, P. A., Cox, L. S. The role of DNA exonucleases in protecting genome stability and their impact on ageing. Age (Dordr). 34 (6), 1317-1340 (2012).
- Berends, E. T., et al. Nuclease expression by Staphylococcus aureus facilitates escape from neutrophil extracellular traps. Journal of Innate Immunity. 2 (6), 576-586 (2010).
- Kiedrowski, M. R., et al. Staphylococcus aureus Nuc2 is a functional, surface-attached extracellular nuclease. PLoS One. 9 (4), 95574 (2014).
- Morita, C., et al. Cell wall-anchored nuclease of Streptococcus sanguinis contributes to escape from neutrophil extracellular trap-mediated bacteriocidal activity. PLoS One. 9 (8), 103125 (2014).
- Hasegawa, T., et al. Characterization of a virulence-associated and cell-wall-located DNase of Streptococcus pyogenes. Microbiology. 156, 184-190 (2010).
- Moon, A. F., et al. Structural insights into catalytic and substrate binding mechanisms of the strategic EndA nuclease from Streptococcus pneumoniae. Nucleic Acids Research. 39 (7), 2943-2953 (2011).
- Li, L. Y., Luo, X., Wang, X. Endonuclease G is an apoptotic DNase when released from mitochondria. Nature. 412 (6842), 95-99 (2001).
- McDermott-Roe, C., et al. Endonuclease G is a novel determinant of cardiac hypertrophy and mitochondrial function. Nature. 478 (7367), 114-118 (2011).
- Wang, Y. T., et al. A link between adipogenesis and innate immunity: RNase-L promotes 3T3-L1 adipogenesis by destabilizing Pref-1 mRNA. Cell Death & Disease. 7 (11), 2458 (2016).
- Li, C. L., Yang, W. Z., Shi, Z., Yuan, H. S. Tudor staphylococcal nuclease is a structure-specific ribonuclease that degrades RNA at unstructured regions during microRNA decay. RNA. 24 (5), 739-748 (2018).
- Wan, L., et al. MTDH-SND1 interaction is crucial for expansion and activity of tumor-initiating cells in diverse oncogene- and carcinogen-induced mammary tumors. Cancer Cell. 26 (1), 92-105 (2014).
- Keyel, P. A. Dnases in health and disease. Developmental Biology. 429 (1), 1-11 (2017).
- Hernandez, F. J., et al. Noninvasive imaging of Staphylococcus aureus infections with a nuclease-activated probe. Nature Medicine. 20 (3), 301-306 (2014).
- Flenker, K. S., et al. Rapid Detection of Urinary Tract Infections via Bacterial Nuclease Activity. Molecular Therapy. 25 (6), 1353-1362 (2017).
- Kannan, N., Eaves, C. J. Tipping the balance: MTDH-SND1 curbs oncogene-induced apoptosis and promotes tumorigenesis. Cell Stem Cell. 15 (2), 118-120 (2014).
- Kemmer, T. P., Malfertheiner, P., Buchler, M., Kemmer, M. L., Ditschuneit, H. Serum ribonuclease activity in the diagnosis of pancreatic disease. International Journal of Pancreatology. 8 (1), 23-33 (1991).
- Fernandez-Salas, E., Peracaula, R., Frazier, M. L., de Llorens, R. Ribonucleases expressed by human pancreatic adenocarcinoma cell lines. European Journal of Biochemistry. 267 (5), 1484-1494 (2000).
- Fujibayashi, K., et al. Serum deoxyribonuclease I activity can be a useful diagnostic marker for the early diagnosis of unstable angina pectoris or non-ST-segment elevation myocardial infarction. Journal of Cardiology. 59 (3), 258-265 (2012).
- Kawai, Y., et al. Diagnostic use of serum deoxyribonuclease I activity as a novel early-phase marker in acute myocardial infarction. Circulation. 109 (20), 2398-2400 (2004).
- Hernandez, L. I., Ozalp, V. C., Hernandez, F. J. Nuclease activity as a specific biomarker for breast cancer. Chemical Communication (Cambridge). 52 (83), 12346-12349 (2016).
- Machado, I., et al. Rapid and specific detection of Salmonella infections using chemically modified nucleic acid probes. Analytica Chimica Acta. 1054, 157-166 (2019).
- Sanders, E. R. Aseptic laboratory techniques: plating methods. Journal of Visualized Experiment. (63), e3064 (2012).
- Worthington Biochemical Corporation. . Manual of Clinical Enzyme Measurements. , (1972).
- Burghardt, E. L., et al. Rapid, Culture-Free Detection of Staphylococcus aureus Bacteremia. PLoS One. 11 (6), 0157234 (2016).
- Bibette, J. Gaining confidence in high-throughput screening. Proceedings of the National Academy of Science U. S. A. 109 (3), 649-650 (2012).
- Hernandez, L. I., Machado, I., Schafer, T., Hernandez, F. J. Aptamers overview: selection, features and applications. Current Topics in Medicinal Chemistry. 15 (12), 1066-1081 (2015).
- Pande, J., Szewczyk, M. M., Grover, A. K. Phage display: concept, innovations, applications and future. Biotechnology Advances. 28 (6), 849-858 (2010).
- Skerra, A. Alternative binding proteins: anticalins - harnessing the structural plasticity of the lipocalin ligand pocket to engineer novel binding activities. FEBS Journal. 275 (11), 2677-2683 (2008).
- Ku, T. H., et al. Nucleic Acid Aptamers: An Emerging Tool for Biotechnology and Biomedical Sensing. Sensors (Basel). 15 (7), 16281-16313 (2015).
- Jayasena, S. D. Aptamers: an emerging class of molecules that rival antibodies in diagnostics. Clinical Chemistry. 45 (9), 1628-1650 (1999).
- Gray, B. P., Brown, K. C. Combinatorial peptide libraries: mining for cell-binding peptides. Chemical Reviews. 114 (2), 1020-1081 (2014).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon