Bu içeriği görüntülemek için JoVE aboneliği gereklidir. Oturum açın veya ücretsiz deneme sürümünü başlatın.
Method Article
Nükleik Asit Probları Kullanılarak Nükleaz Aktivitesinin Kinetik Taraması
* Bu yazarlar eşit katkıda bulunmuştur
Bu Makalede
Özet
Değiştirilmiş nükleaz aktivitesi farklı insan koşulları ile ilişkili olmuştur, bir biyomarker olarak potansiyelinin altında yatan. Bu makalede sunulan modüler ve kolay tarama metodolojisi, hastalığın biyobelirteci olarak nükleaz aktivitesinden yararlanmak için spesifik nükleik asit problarının seçilmesine olanak sağlar.
Özet
Nükleazlar, riboz şekerlerini birbirine bağlayan fosfodiester bağlarının hidrolizini katalize ederek nükleik asitleri parçalayan enzimler sınıfıdır. Nükleazlar prokaryotik ve ökaryotik organizmalarda genom stabilitesinin korunmasından patojenlere karşı koruma sağlamaya kadar çeşitli hayati fizyolojik roller sergilerler. Değiştirilmiş nükleaz aktivitesi bakteriyel enfeksiyonlar ve kanser de dahil olmak üzere çeşitli patolojik koşullar ile ilişkili olmuştur. Bu amaçla, nükleaz aktivitesi belirli bir biyomarker olarak istismar edilmesi için büyük bir potansiyel göstermiştir. Ancak, bu aktiviteye dayalı sağlam ve tekrarlanabilir bir tarama yöntemi son derece arzu edilir.
Burada, patolojik ve sağlıklı koşullar arasında ayrım kapsamı ile, substrat olarak nükleik asit probları kullanarak nükleaz aktivitesi için tarama sağlayan bir yöntem sunuyoruz. Bu yöntem, yinelemeli bir şekilde, artan özgüllük ile, yeni sonda kitaplıkları tasarlama olanağı sunar. Bu nedenle, kimyasal olarak değiştirilmiş nükleik asitlerin kullanılabilirliğinden yararlanarak, probların tasarımını gelişmiş özelliklerle hassaslaştırmak için birden fazla tarama turu gereklidir. Önerilen teknolojinin önemli potansiyeli, hastalık koşullarıyla ilişkili nükleaz aktivitesinin taranması için esnekliği, yüksek tekrarlanabilirliği ve çok yönlülüğünde yatmaktadır. Bu teknolojinin klinikte büyük bir potansiyele sahip umut verici tanı araçlarının geliştirilmesine olanak sağlaması beklenmektedir.
Giriş
Nükleazlar, nükleik asit moleküllerinin omurga yapısını oluşturan fosfodiester bağlarını kırpabilen bir enzim sınıfıdır. Çekirdeklerin büyük çeşitliliği sınıflandırmalarını oldukça zorlaştırır. Ancak, substrat tercihi (deoksiribonükleik asit (DNA) veya ribonükleik asit (RNA)), dekolte alanı (ensonükleazlar veya eksikonklezler) veya metal iyon bağımlılığı gibi nükleazları tanımlamak için kullanılan bazı ortak kriterlervardır. Nükleazlar, hem prokaryotik hem de ökaryotik organizmalarda temel rolleri olan ve gen düzenleme araçları2olarak kullanılan ve kullanılmaya devam eden katalitik enzimlerdir. Onlar da DNA bakım ve çoğaltma temel aktörler, genom istikrarı tutmak için yardımcı ve prova okuma süreçlerine katılan3. Bakterilerde, örneğin, nükleazlar önemli virülans faktörleri olarak tespit edilmiştir, konak bağışıklık sisteminin etkinliğini azaltarak bakteri sağkalımını teşvik edebiliyoruz4,5,6,7 ,8. Memelilerde, nükleazlar apoptoz 9, mitokondriyalbiyogenez ve bakım10 ve antibakteriyel ve antiviral doğuştan gelen bağışıklık yanıtlarının arabuluculuk karıştığı ileri sürülmüştür11. Beklendiği gibi, nükleaz aktivitesi değişiklikleri, geliştirme veya eksikliği olsun, insan hastalıklarının geniş bir yelpazede karıştığı olmuştur. Bu hastalıklar kanserlerin geniş bir yelpazede değişir12,13 kardiyak hipertrofi10 veya otoimmün hastalıklar14. Bu nedenle, nükleazlar insan koşullarının heterojen bir grup için biyobelirteçler olarak ilginç adaylar haline gelmiştir. Aslında, nükleazlar zaten belirli bakteriyel ajanların neden olduğu enfeksiyonların tespiti için başarılı tanı araçları olarak potansiyellerini göstermiştir, Staphylococcus aureus veya Escherichia coli15gibi, 16. Birçok kanser türünde, stafilokokal nükleaz etki alanı içeren protein 1 (SND1) ribonükleaz ekspresyonu kötü prognoz un göstergesidir17. Pankreas kanseri hastalarında, yüksek ribonükleaz I (RNase I) serum düzeyleri bildirilmiştir18 ve kanserli hücrefenottipleri ile ilişkili olduğu önerilmiştir19. Iskemik kalp koşullarında, miyokard enfarktüsü veya kararsız anjina pektoris gibi, deoksiribonuklez i (DNase I) serum düzeyleri geçerli bir tanı belirteci 20 olduğu gösterilmiştir20,21.
Bu nükleaz aktivitesinin küresel planı sağlıklı ve hastalık durumlarında farklı olabilir hipotez edilmiştir. Aslında, son raporlar sağlıklı ve kanserli fenotipler22 ayırt etmek veya bir türe özgü bir şekilde patojenik bakteriyel enfeksiyonları belirlemek için nükleaz aktivitesi farklılıkları kullandık15,23. Bu bulgular, hastalığın biyobelirteçleri olarak nükleazların kullanımı için yeni bir yol açmıştır. Bu nedenle, yeni tanı araçlarının geliştirilmesinde önemli olabilecek nükleaz aktivitesindeki hastalıkla ilişkili farklılıkları sistematik olarak belirleyebilen kapsamlı bir tarama yönteminin geliştirilmesi için bir gereklilik vardır.
Burada, sağlıklı ve sağlıksız olarak nükleaz aktivitesi veya bir hücre veya bakteri türüne özgü aktivite arasında ayrım yapabilen hassas ve spesifik probları tanımlamak için yeni bir in vitro tarama yaklaşımı(Şekil 1)tanıtıyoruz ve tanımlıyoruz. Nükleik asitlerin modülerliğinden yararlanarak, kütüphane tasarımı için önemli parametreler olan, her ikisi de farklı diziler ve kimyalardan oluşan, söndürülmüş floresan oligonükleotid problardan oluşan bir başlangıç kütüphanesi tasarladık. Bu oligonükleotid problar sırasıyla 5' ve 3' uçlarda bir florofor (floresan amidit, FAM) ve bir sorgulayıcı (gelgit quencher 2, TQ2) ile çevrilidir (Tablo 1). Enzimatik bozulmanın kinetiğini ölçmek için bu floresan rezonans enerji transferi (FRET) bazlı florometrik testkullanarak, çekirdek aktivitesinin diferansiyel desenlerini ayırt etme potansiyeline sahip aday probları tanımlayabildik. sağlıklı veya hastalık durumları ile ilişkili. Yeni kütüphanelerin en iyi aday sondalarına dayalı olarak oluşturulduğu ve sonraki tarama adımlarında her zamankinden daha spesifik aday sondalarının belirlenmesine olanak tanıyan yinelemeli bir süreç tasarladık. Ayrıca bu yaklaşım, duyarlılığı artırmak için nükleazların katalitik yapısından yararlanır. Bu, muhabir sondaların aktivize edici doğasından ve çekirdeklerin alternatif antikor veya küçük molekül tabanlı tarama yöntemlerine göre önemli avantajları temsil eden substrat moleküllerini sürekli olarak işleme yeteneğinden yararlanarak elde edilir.
Bu yaklaşım, sağlıklı ve hastalık durumları arasında ayrım yapabilen spesifik nükleik asit problarının tanımlanması için son derece modüler, esnek ve uygulanması kolay bir tarama aracı ve yeni gelecekteki klinik uygulamalar için uyarlanabilen tanı araçları. Bu yaklaşım, salmonella Typhimurium'dan (burada Salmonellaolarak anılacaktır) bu bakterilerin spesifik olarak tanımlanması için elde edilen nükleaz aktivitesini tanımlamak için kullanılmıştır. Aşağıdaki protokolde, kinetik analiz kullanarak bakteriyel nükleaz aktivitesini taramak için bir yöntem rapor ediyoruz.
Protokol
1. Oligonükleotid kütüphane tasarımı ve hazırlanması
- Kütüphane tasarımı
- Aşağıdaki kriterlere göre, tüm problar için eşit uzunlukta, ikincil yapı oluşumunu önlemek için 8 ile 12-mer uzunluğunda ki söndürülen floresan oligonükleotid problardan oluşan bir kütüphane tasarlayın.
- Adenin (A), guanin (G), sitozin (C) ve timin (T)/urasil (U)(Tablo 1'de DNA ve RNA probları)kombinasyonunu içeren en az bir DNA ve bir RNA rasgele dizisini ekleyin.
NOT: Tablo 1'de açıklanan DNA ve RNA dizileri, bilinmeyen bir nükleaz aktivitesini sınıflandırmak için ilk yüzeyler olarak yararlı olmuştur, DNase veya RNase. Bakteri ve doku örneklerindeki nükleaz aktivite profillerini tespit etmek için geniş bir yetenek gösterdikleri için bu iki dizi herhangi bir tarama için başlangıç noktası olarak tavsiye edilir. Bu DNA ve RNA dizileri ile nükleaz aktivitesi gözlenmezise ek oligonükleotidlerin tasarımı gereklidir. - Belirli bir nükleaz aktivitesi (DNA-Poly A, DNA-Poly T, DNA-Poly C, DNA-Poly G, RNA-Poly A, RNA-Poly U, RNA-Poly C ve Tablo 1'deRNA-Poly G) dizi bağımlılığını belirlemek için tek bir nükleotit türünden oluşan polinükleotid dizileri ekleyin.
- İlk DNA ve RNA dizileri ile aynı nükleotit kalıntıdiziligisi kullanılarak, riboz şekerinin 2'-Floro ve 2'-O-Metil gibi 2'-pozisyonunda kimyasal modifikasyonlar barındıran nükleozit analogları içeren diziler, sıkı bir adım olarak nükleazların seçiciliğini artırmak için.
- Tamamen 2'-Floro nükleozit analogları veya 2'-O-Metil nükleozit analoglarından oluşan tam modifiye dizileri içerir (Tüm 2'-F ve Tüm 2'-OMe, Tablo 1)
- Değiştirilmemiş doğal pürinler ve 2'-Floro pirimidin nükleozit analoglarından oluşan şimerik dizileri içerir (RNA Pyr-2'F, Tablo 1).
- Değiştirilmemiş doğal pürinler ve 2'-O-Metil pirimidin nükleozit analoglarından oluşan şimerik dizileri içerir (RNA Pyr-2'OMe, Tablo 1).
- Değiştirilmemiş doğal pirimidinler ve 2'-Floro pürin nükleozit analoglarından oluşan şimerik dizileri içerir (RNA Pur-2'F, Tablo 1).
- Değiştirilmemiş doğal pirimidinler ve 2'-O-Metil pürin nükleozit analoglarından oluşan şimerik dizileri içerir (RNA Pur-2'OMe, Tablo 1).
- Adenin (A), guanin (G), sitozin (C) ve timin (T)/urasil (U)(Tablo 1'de DNA ve RNA probları)kombinasyonunu içeren en az bir DNA ve bir RNA rasgele dizisini ekleyin.
- Aşağıdaki kriterlere göre, tüm problar için eşit uzunlukta, ikincil yapı oluşumunu önlemek için 8 ile 12-mer uzunluğunda ki söndürülen floresan oligonükleotid problardan oluşan bir kütüphane tasarlayın.
- Oligonükleotid prob hazırlama ve depolama
NOT: Oligonükleotidler fosforamidit yöntemi ile sentezlenir. Sentezden sonra, oligonükleotidler yüksek performanslı sıvı kromatografisi (HPLC) kullanılarak saflaştırılır ve kütle kütle spektrometresi ile ölçülür.- Bir santrifüj kullanarak lyophilized oligonükleotid probları aşağı spin ve nükleaz bozulmasını önlemek için Tris-EDTA (TE) tampon seyreltmek. 500 pmol/μL konsantrasyonu olan bir stok çözeltisi işlemek için her probun verime göre seyreltme hacmini belirleyin.
- Lyophilized oligonükleotidleri 4 °C'de veya oda sıcaklığında kısa bir süre saklayın. Uzun süreli depolama için (aylar- yıllar) -20 °C'de liofili oligonükleotidleri saklayın. Te'de liyofilize oligonükleotidlerin yeniden askıya alınması üzerine stok çözeltilerini -20 °C'de veya tercihen -80 °C'de saklayın.
2. Bakteri kültürü
- Steril koşullar altında kriyojenik depolama şişesinden bir gözenekli cam boncuk alın.
- Bireysel bakteri kolonilerini izole etmek için(Salmonella ve E. coli),kurbağayı doğrudan Tryptic Soya Agar (TSA) içeren bir Petri kabına doğru serileştirerek/yuvarlayarak24 kadranda yöntemini kullanın. Kan.
- Kültürü 37 °C'de 24 saat kuluçkaya yatırın.
3. Supernatant hazırlık
- Tek bir koloniyi katı ortamdan 50 mL Triptik Soya Suyu'na (TSB) aktarın ve 37 °C'de 24 saat 200 rpm'de kuluçkaya yatırın.
- TSB'de (alt kültür) kültürü (1:500) seyreltin ve 37 °C'de 24 saat 200 rpm'de bir titreyen kuluçka makinesinde kuluçkaya yatırın.
NOT: 24 saat inkübasyondan sonra bakteri kültürlerinin sabit fazda 109 CFU/mL'den yüksek değerlere ulaşması beklenmektedir. - Kuluçka döneminden sonra kültürleri 30 dk boyunca 4.500 x g'da kapaklı steril tüplere ve santrifüjlere aktarın.
- Supernatant toplamak ve hemen kullanın. Alternatif olarak, supernatants 4 °C veya -20 °C'de saklayın.
4. Nükleaz Aktivitesi Tsası
- Çalışma çözümlerinin hazırlanması
- Stok çözeltisini (1:10 oran) 50 pmol/μL'lik son çalışma konsantrasyonu için seyrelterek (1:10 oranı) 1,5 mL nükleazsız mikrosantrifüj tüplerinde her prob için 20 μL çalışma çözeltisi hazırlayın. Bu amaçla, MgCl2 ve CaCl2 içeren 18 μL Fosfat Tampon Salini (PBS) ile 2 μL probu stok çözeltisini karıştırın.
NOT: Çalışma çözümünde oligonükleotidlerin nükleaz bozulmasına karşı daha savunmasız olduğunu unutmayın, bu nedenle reaksiyonu ayarlamadan hemen önce çalışma çözümlerini hazırlamaları önerilir. - Hazırlık sırasında doğrudan ışık temasından kaçının ve floroforlarla uğraşırken karanlıkta (folyoya sarılı) tutun.
- Stok çözeltisini (1:10 oran) 50 pmol/μL'lik son çalışma konsantrasyonu için seyrelterek (1:10 oranı) 1,5 mL nükleazsız mikrosantrifüj tüplerinde her prob için 20 μL çalışma çözeltisi hazırlayın. Bu amaçla, MgCl2 ve CaCl2 içeren 18 μL Fosfat Tampon Salini (PBS) ile 2 μL probu stok çözeltisini karıştırın.
- Reaksiyon kurulumu
- Florometreyi (örneğin Cytation 1) 37 °C'ye Kadar ısıtın.
- Her numune (TSB, E. coli ve Salmonella)için 1,5 mL nükleaz içermeyen mikrosantrifüj tüplerinden oluşan bir set (bir tüp/prob) hazırlayın ve buna göre etiketlendirin.
- Dikkatle TSB steril kültür medya, Salmonella supernatant veya E. coli supernatant (adım 3.4)96 μL ekleyin. Daha sonra, buna göre 4 μL/tüp prob çalışma çözeltisi ekleyin. Bu adımı oda sıcaklığında gerçekleştirin.
NOT: Taramanın ilk turunda 10 sonda, ikinci tur için 6 prob kullanıldı. - Homojen bir çözüm elde etmek için yukarı ve aşağı borular oluşturarak iyice karıştırın. Karıştırma sırasında örnekleriçine hava kabarcıkları tanıtmak kaçının.
- Her çözeltinin 95 μL'sini (prob + supernatant veya kültür ortamı) siyah bir alt, tedavi edilmeyen 96 kuyu plakasının ayrı bir kuyuya yükleyin. Kuyunun duvarına yakın ucu ile dikkatlice dağıtarak yükleme üzerine kuyularda kabarcıkların oluşumunu en aza indirin.
- Tabağı kapağıile kapatın. Kapağı görsel olarak inceleyin ve ölçüm yapıtlarına neden olabilecek kalem işaretleri veya toz parçacık birikimi olup olmadığını kontrol edin. Bu durumda yeni bir kapak için kapağı değiştirin.
- Ölçüm kurulumu
- Satın alma yazılımını (Gen5 3.05) yazılım kısayol simgesine(Şekil S1A)tıklayarak açın.
- Görev yöneticisi penceresinden Şimdi Oku'yu seçin ve kinetik ölçüm protokolü(Şekil S1B)oluşturmak için Yeni... seçeneğini belirleyin.
- Yordam başlıklı iletişim penceresinde Sıcaklığı Ayarla'ya tıklayın ve 37 °C'yi seçin. Tamam (Şekil S1C)tuşuna basarak ayarları onaylayın ve kaydedin.
- Yordambaşlıklı iletişim penceresinde Kinetik Başlat'ı tıklatın. Ardından, Kinetik Adımbaşlıklı açılır iletişim penceresinde, Çalışma Zamanı giriş kutusunda 2 saat ve Aralık giriş kutusunda 2 dakika seçin. Tamam (Şekil S1D)tuşuna basarak ayarları onaylayın ve kaydedin.
- Yordambaşlıklı iletişim penceresinde Oku'ya tıklayın. Daha sonra okuma yöntemibaşlıklı açılan iletişim penceresinde, bir algılama yöntemi olarak Floresan Yoğunluğu, okuma türü olarak Uç nokta/Kinetik ve optik türü olarak Filtreler'i seçin. Tamam (Şekil S1E)tuşuna basarak ayarları onaylayın ve kaydedin.
- Okuma Adımı (Kinetik)başlıklı açılan iletişim penceresinde, Filtre Kümesi'nden Yeşil'i seçin. Tamam (Şekil S2A)tuşuna basarak ayarları onaylayın ve kaydedin.
- Yordambaşlıklı iletişim penceresinde, oluşturulan protokolün geçerli olduğundan emin olmak için kullanım kapağını seçin ve doğrulama'yı tıklatın. Bu bir açılır iletişim penceresi(Şekil S2B)tarafından doğrulanır.
- Menü çubuğunda protokolü seçin ve yordamı seçin(Şekil S2C).
- Yordambaşlıklı iletişim penceresinde, ölçülecek kuyuları tanımlayın (Şekil S2D).
- Dosya adı giriş kutusuna denemenin adını girin (Şekil S2E).
- Plakayı kapağıyla plaka okuyucusuna yükleyin. Plakanın doğru yönde olduğundan emin olun.
- Araç çubuğundaki yeni oku düğmesini tıklatarak edinmeye başlayın (Şekil S2F).
- Veri Analizi
- İletişim penceresinde plaka 1 (Şekil S3A)başlıklı ölçülen kuyulardan birine tıklayın.
- Kuyuları seçin'i tıklatın ve kuyu seçimi iletişim penceresine ölçülen tüm kuyuları ekleyin. Tamamtuşuna basarak ayarları onaylayın ve kaydedin. (Şekil S3B).
- Tablolanmış sonuçları görselleştirmek için Plaka 1 başlıklı iletişim penceresinde verileri seçin(Şekil S3C).
- Bağlam menüsünden(Şekil S3C) hızlı dışa aktarma seçerek verileri bir elektronik sayfaya dışa aktarın.
- Elektronik tabloda, her örnek ve sonda için veri sütunlarını buna göre etiketlendirin.
- Göreceli floresan birimlerini (RFU) zamana karşı (x ekseni: reaksiyon ve y ekseninin zaman çizelgesi: RUK'lar) çizerek işaretçi stilini kullanarak kinetik grafikler oluşturun.
NOT: Elektronik tablo programları (örneğin, Excel) kullanırken grafik oluşturma açıklaması uygulanabilir. Ancak, diğer programlar, RFU'yu zamana karşı çizerek elde edilen ham verilerden grafik oluşturmak için kullanılabilir. - Aşağıdaki formül25göre ölçülen her bir aralık için enzimatik
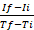 reaksiyon oranını hesaplamak : oran= , Maksimal aralık zaman noktasında RFU ise ve Ii minimum aralık zaman noktasında RFU ise, ve Tf ve Ti sırasıyla maksimal ve minimum aralıklı zaman noktalarıdır.
reaksiyon oranını hesaplamak : oran= , Maksimal aralık zaman noktasında RFU ise ve Ii minimum aralık zaman noktasında RFU ise, ve Tf ve Ti sırasıyla maksimal ve minimum aralıklı zaman noktalarıdır. - Her eğri için en yüksek değere (Rmax)sahip oranı seçin. Örnek başına enfazla r varsa, en erken ölçüm zaman noktasında oluşan r'yi seçin.
- Rmax ile Rmax'in oluştuğu zaman aralığının ortalama değeri arasındaki oranı hesaplayın: oran katsayısı =
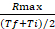
- Salmonella ve E. coli oranı katsayısı arasındaki kat farkı (FD) her prob için hesaplayın.
- 3'ten yüksek bir kat farkı değerini önemli olarak düşünün.
- En yüksek kat farkı değerlerine (FD) sahip önemli sondaları aday sondalar olarak ve bir sonraki tarama turunda kütüphane tasarımıiçin temel olarak düşünün.
5. Eleme Turları Seçim Kriterleri (Şekil 1)
- İlk Tarama Turu
- DNA, RNA ve polinükleotid probları kullanarak nükleaz aktivitesi testi (bölüm 4'te açıklandığı gibi) yapın.
- DNA ve RNA aday sondalarının sayısını ve performansını (FD değeri) karşılaştırarak DNA kimyası veya RNA kimyası tercihini değerlendirin.
- Şekil 1'degösterildiği gibi, en fazla sayıda aday prob işleyen nükleik asit kimyasının türünü seçin.
- İkinci Tur Gösterim
- İlk tarama turunda seçilen nükleik asit substratına göre tasarlanmış tam modifiye edilmiş problar ve şimerik problar kullanarak nükleaz aktivitesi testini (bölüm 4'te açıklandığı gibi) gerçekleştirin.
- 2'-Floro ve 2'-O-Metil nükleozit analogları içeren sekanslara yönelik tercihi 2'-Floro ve 2'-O-Metil modifiye aday problar için elde edilen sonuçları karşılaştırarak değerlendirin.
- Şekil 1'degösterildiği gibi en fazla sayıda aday prob işleyen nükleozit analog modifikasyon türünü seçin.
Sonuçlar
Şekil 1, iki tarama turuna ayrılan bu metodolojinin iş akışını göstermektedir. İlk tarama turunda 5 DNA sondası (DNA, DNA Poli A, DNA Poly T, DNA Poly C ve DNA Poly G) ve ayrıca 5 RNA probu (RNA, RNA Poly A, RNA Poly U RNA Poly C ve RNA Poly G) kullanıldı. Bu tarama turunun ham verileri Ek Tablo 1'de bulunabilir. İkinci turda, kimyasal olarak modifiye edilmiş problar, RNA dizisini kimyasal olarak değiştirilmiş nükleozitlerle (Tümü 2'-Floro ve Tüm 2'-OMt...
Tartışmalar
Nükleaz aktivitesindeki değişiklikler, farklı kanser türleri ve bakteriyel enfeksiyonlar da dahil olmak üzere çok çeşitli hastalık fenotipleri ile ilişkilendirilmiştir. Bu değişiklikler bir durumun nedensel ajan olduğu önerilmektedir14, diğer durumlarda onlar zararlı bir fizyolojik olayın sonucu iken20 veya patojenik ajan16,26. Beklendiği gibi, bir tanıbiyomarker olarak nükleaz ve nükleaz akti...
Açıklamalar
Yazarların açıklayacak bir şeyi yok.
Teşekkürler
Yazarlar luiza I. Hernandez (Linköping Üniversitesi) el yazması ve değerli tavsiyeler onu dikkatli revizyon için kabul etmek istiyorum. Bu çalışma, Linköping Üniversitesi'nde (Fakülte Hibe SFO-Mat-LiU No. 2009-00971) Knut ve Alice Wallenberg Vakfı ve İsveç Hükümeti Malzeme Bilimi Stratejik Araştırma Alanı tarafından desteklenmiştir.
Malzemeler
| Name | Company | Catalog Number | Comments |
| Black bottom, non-treated 96 well plate | Fisher Scientific | 10000631 | |
| Cytation1 | BioTek | CYT1FAV | |
| Eppendorf tubes | Thermofisher | 11926955 | |
| Escherichia coli | ATCC | 25922 | |
| Microbank cryogenic storage vial containing beads | Pro-Lab Diagnostics | 22-286-155 | |
| Nucleic acid probes | Biomers.net | # | |
| Phosphate Buffer Saline containing MgCl2 and CaCl2 | Gibco™ | 14040117 | |
| Salmonella enterica subs. Enterica | ATCC | 14028 | |
| Tris-EDTA | Fisher Scientific | 10647633 | |
| Tryptone Soya Agar with defibrinated sheep blood | Thermo Fisher Scientific | 10362223 | |
| Tryptic Soy Broth | Sigma Aldrich | 22092 |
Referanslar
- Yang, W. Nucleases: diversity of structure, function and mechanism. Quarterly Reviews of Biophysics. 44 (1), 1-93 (2011).
- Adli, M. The CRISPR tool kit for genome editing and beyond. Nature Communication. 9 (1), 1911 (2018).
- Mason, P. A., Cox, L. S. The role of DNA exonucleases in protecting genome stability and their impact on ageing. Age (Dordr). 34 (6), 1317-1340 (2012).
- Berends, E. T., et al. Nuclease expression by Staphylococcus aureus facilitates escape from neutrophil extracellular traps. Journal of Innate Immunity. 2 (6), 576-586 (2010).
- Kiedrowski, M. R., et al. Staphylococcus aureus Nuc2 is a functional, surface-attached extracellular nuclease. PLoS One. 9 (4), 95574 (2014).
- Morita, C., et al. Cell wall-anchored nuclease of Streptococcus sanguinis contributes to escape from neutrophil extracellular trap-mediated bacteriocidal activity. PLoS One. 9 (8), 103125 (2014).
- Hasegawa, T., et al. Characterization of a virulence-associated and cell-wall-located DNase of Streptococcus pyogenes. Microbiology. 156, 184-190 (2010).
- Moon, A. F., et al. Structural insights into catalytic and substrate binding mechanisms of the strategic EndA nuclease from Streptococcus pneumoniae. Nucleic Acids Research. 39 (7), 2943-2953 (2011).
- Li, L. Y., Luo, X., Wang, X. Endonuclease G is an apoptotic DNase when released from mitochondria. Nature. 412 (6842), 95-99 (2001).
- McDermott-Roe, C., et al. Endonuclease G is a novel determinant of cardiac hypertrophy and mitochondrial function. Nature. 478 (7367), 114-118 (2011).
- Wang, Y. T., et al. A link between adipogenesis and innate immunity: RNase-L promotes 3T3-L1 adipogenesis by destabilizing Pref-1 mRNA. Cell Death & Disease. 7 (11), 2458 (2016).
- Li, C. L., Yang, W. Z., Shi, Z., Yuan, H. S. Tudor staphylococcal nuclease is a structure-specific ribonuclease that degrades RNA at unstructured regions during microRNA decay. RNA. 24 (5), 739-748 (2018).
- Wan, L., et al. MTDH-SND1 interaction is crucial for expansion and activity of tumor-initiating cells in diverse oncogene- and carcinogen-induced mammary tumors. Cancer Cell. 26 (1), 92-105 (2014).
- Keyel, P. A. Dnases in health and disease. Developmental Biology. 429 (1), 1-11 (2017).
- Hernandez, F. J., et al. Noninvasive imaging of Staphylococcus aureus infections with a nuclease-activated probe. Nature Medicine. 20 (3), 301-306 (2014).
- Flenker, K. S., et al. Rapid Detection of Urinary Tract Infections via Bacterial Nuclease Activity. Molecular Therapy. 25 (6), 1353-1362 (2017).
- Kannan, N., Eaves, C. J. Tipping the balance: MTDH-SND1 curbs oncogene-induced apoptosis and promotes tumorigenesis. Cell Stem Cell. 15 (2), 118-120 (2014).
- Kemmer, T. P., Malfertheiner, P., Buchler, M., Kemmer, M. L., Ditschuneit, H. Serum ribonuclease activity in the diagnosis of pancreatic disease. International Journal of Pancreatology. 8 (1), 23-33 (1991).
- Fernandez-Salas, E., Peracaula, R., Frazier, M. L., de Llorens, R. Ribonucleases expressed by human pancreatic adenocarcinoma cell lines. European Journal of Biochemistry. 267 (5), 1484-1494 (2000).
- Fujibayashi, K., et al. Serum deoxyribonuclease I activity can be a useful diagnostic marker for the early diagnosis of unstable angina pectoris or non-ST-segment elevation myocardial infarction. Journal of Cardiology. 59 (3), 258-265 (2012).
- Kawai, Y., et al. Diagnostic use of serum deoxyribonuclease I activity as a novel early-phase marker in acute myocardial infarction. Circulation. 109 (20), 2398-2400 (2004).
- Hernandez, L. I., Ozalp, V. C., Hernandez, F. J. Nuclease activity as a specific biomarker for breast cancer. Chemical Communication (Cambridge). 52 (83), 12346-12349 (2016).
- Machado, I., et al. Rapid and specific detection of Salmonella infections using chemically modified nucleic acid probes. Analytica Chimica Acta. 1054, 157-166 (2019).
- Sanders, E. R. Aseptic laboratory techniques: plating methods. Journal of Visualized Experiment. (63), e3064 (2012).
- Worthington Biochemical Corporation. . Manual of Clinical Enzyme Measurements. , (1972).
- Burghardt, E. L., et al. Rapid, Culture-Free Detection of Staphylococcus aureus Bacteremia. PLoS One. 11 (6), 0157234 (2016).
- Bibette, J. Gaining confidence in high-throughput screening. Proceedings of the National Academy of Science U. S. A. 109 (3), 649-650 (2012).
- Hernandez, L. I., Machado, I., Schafer, T., Hernandez, F. J. Aptamers overview: selection, features and applications. Current Topics in Medicinal Chemistry. 15 (12), 1066-1081 (2015).
- Pande, J., Szewczyk, M. M., Grover, A. K. Phage display: concept, innovations, applications and future. Biotechnology Advances. 28 (6), 849-858 (2010).
- Skerra, A. Alternative binding proteins: anticalins - harnessing the structural plasticity of the lipocalin ligand pocket to engineer novel binding activities. FEBS Journal. 275 (11), 2677-2683 (2008).
- Ku, T. H., et al. Nucleic Acid Aptamers: An Emerging Tool for Biotechnology and Biomedical Sensing. Sensors (Basel). 15 (7), 16281-16313 (2015).
- Jayasena, S. D. Aptamers: an emerging class of molecules that rival antibodies in diagnostics. Clinical Chemistry. 45 (9), 1628-1650 (1999).
- Gray, B. P., Brown, K. C. Combinatorial peptide libraries: mining for cell-binding peptides. Chemical Reviews. 114 (2), 1020-1081 (2014).
Yeniden Basımlar ve İzinler
Bu JoVE makalesinin metnini veya resimlerini yeniden kullanma izni talebi
Izin talebiThis article has been published
Video Coming Soon
JoVE Hakkında
Telif Hakkı © 2020 MyJove Corporation. Tüm hakları saklıdır