Для просмотра этого контента требуется подписка на Jove Войдите в систему или начните бесплатную пробную версию.
Method Article
Кинетический скрининг nuclease деятельности с использованием нуклеиновой кислоты зондов
* Эти авторы внесли равный вклад
В этой статье
Резюме
Измененная деятельность нуклеаза была связана с различными человеческими условиями, лежащими в основе ее потенциала в качестве биомаркера. Модульная и простая в ней методология скрининга, представленная в настоящем документе, позволяет подбирать специфические нуклеиновые кислотные зонды для использования нуклеаза в качестве биомаркера заболевания.
Аннотация
Nucleases являются класс ферментов, которые расщепляют нуклеиновые кислоты путем катализирования гидролиза фосфодидерных связей, которые связывают рибоза сахара. Nucleases отображают различные жизненно важные физиологические роли в прокариотических и эукариотических организмов, начиная от поддержания стабильности генома для обеспечения защиты от патогенных микроорганизмов. Измененная активность нуклеаза была связана с несколькими патологическими состояниями, включая бактериальные инфекции и рак. С этой целью деятельность по нуклеаде показала большой потенциал для использования в качестве конкретного биомаркера. Однако надежный и воспроизводимый метод скрининга, основанный на этой деятельности, остается весьма желательным.
В этой связи мы внедряем метод, позволяющий проводить скрининг на нуклеализную деятельность с использованием зондов нуклеиновой кислоты в качестве субстратов с разновидностью между патологическими и здоровыми состояниями. Этот метод предлагает возможность проектирования новых библиотек зонда, с увеличением специфичности, в итеративной манере. Таким образом, несколько раундов скрининга необходимы для уточнения конструкции зондов с расширенными функциями, пользуясь наличием химически модифицированных нуклеиновых кислот. Значительный потенциал предлагаемой технологии заключается в ее гибкости, высокой воспроизводимости и универсальности скрининга активности нуклеаза, связанной с заболеваниями. Ожидается, что эта технология позволит разработать перспективные диагностические инструменты с большим потенциалом в клинике.
Введение
Nucleases являются класс ферментов, способных расщепления фосфодиестерных связей, которые образуют основную структуру нуклеиновой кислоты молекул. Огромное разнообразие нуклеаз делает их классификацию довольно трудно. Тем не менее, Есть некоторые общие критерии, используемые для описания нуклеазы, такие как предпочтения субстрата (дезоксибонуклеиновая кислота (ДНК) или рибонуклеиновой кислоты (РНК)), расщепление сайта (эндонуклезаили или exonucleases), или ионной зависимости металла, среди других1. Nucleases являются высоко сохранены каталитические ферменты, которые имеют основные роли в обоих, прокариотических и эукариотических организмов и были использованы, и продолжают использоваться, как инструменты редактирования генов2. Они также являются основополагающими действующими лицами в поддержании ДНК и репликации, помогая сохранить стабильность генома и участвуя в процессах проверки чтения3. В бактериях, например, нуклеазы были определены в качестве важных факторов вирулентности, способных способствовать выживаемости бактерий за счет снижения эффективности иммунной системы хозяина4,5,6,7 ,8. У млекопитающих, нуклеазы были предложены быть вовлечены в апоптоз9, митохондриальный биогенез и содержание10 и посредничество антибактериальных и противовирусных врожденных иммунных ответов11. Неудивительно, что изменения активности нуклеаза, будь то повышение или отсутствие, были вовлечены в широкий спектр заболеваний человека. Эти заболевания варьируются от широкого спектра раковых заболеваний12,13 до сердечной гипертрофии10 или аутоиммунных заболеваний14. Таким образом, нуклеазы стали интересными кандидатами в качестве биомаркеров для неоднородной группы человеческих условий. В самом деле, nucleases уже показали свой потенциал в качестве успешных диагностических инструментов для выявления инфекций, вызванных конкретными бактериальными агентами, такими как золотистый стафилококк или Escherichia coli15, 16. Во многих типах рака, выражение стафилококковой нуклеи домен-содержащих белков 1 (SND1) рибонуклеазы свидетельствует о плохой прогноз17. У больных раком поджелудочной железы, повышенный рибонуклеаI (RNase I) сыворотки были зарегистрированы18 и предлагается быть связаны с раковыми фенотипами клеток19. В ишемических условиях сердца, таких как инфаркт миокарда или нестабильный стенокардия pectoris, дезоксирибонуклея I (DNase I) уровни сыворотки были показаны, чтобы быть действительным диагностическим маркером20,21.
Было высказывано что глобальный план деятельности nuclease может быть по-разному в здоровых и положениях заболевания. В самом деле, последние доклады использовали различия в активности нуклеаза различать здоровые и раковые фенотипы22 или для выявления патогенных бактериальных инфекций в видов-специфических образом15,23. Эти выводы открыли новый путь для использования нуклеаз в качестве биомаркеров болезни. Поэтому существует необходимость в разработке комплексного метода скрининга, способного систематически выявлять связанные с заболеваниями различия в деятельности нуклеаза, которые могут иметь ключевое значение при разработке новых диагностических средств.
В этом виде мы вводим и описываем новый подход к скринингу in vitro(рисунок 1)для выявления чувствительных и специфических зондов, способных дискриминировать деятельность нуклеазы в здоровой и нездоровой, или деятельность, характерную для типа клеток или бактерий. Воспользовавшись модульностью нуклеиевых кислот, мы разработали начальную библиотеку утоленных флуоресцентных олигонуклеотидных зондов, состоящих из всеобъемлющего набора различных последовательностей и химии, оба являющиеся важными параметрами для оформления библиотеки. Эти олигонуклеотидные зонды окружены флюорофором (флуоресцеин амидитом, FAM) и утоляжем (tide quencher 2, T'2) на 5' и 3' заканчивается соответственно (Таблица 1). С помощью этого флуоресцентного резонанса передачи энергии (FRET) на основе флюорометрического исследования для измерения кинетики ферментативной деградации, мы смогли определить кандидат зондов с потенциалом для дискриминации дифференциальных моделей нуклеаза деятельности связанных со здоровыми или болезнями. Мы разработали итеративный процесс, в котором создаются новые библиотеки на основе лучших зондов-кандидатов, что позволяет идентифицировать все более конкретные кандидаты зондов в последующих шагах скрининга. Кроме того, этот подход использует каталитический характер нуклеаз для повышения чувствительности. Это достигается за счет использования активируемого характера репортер зондов и способность nucleases постоянно обрабатывать молекулы субстрата, как представляющие ключевые преимущества по сравнению с альтернативными антителами или малых молекул на основе методов скрининга.
Этот подход предлагает весьма модульный, гибкий и простой в реализации инструмент скрининга для идентификации конкретных зондов нуклеиновой кислоты, способных различать здоровые и болезни, и отличную платформу для разработки новых диагностические инструменты, которые могут быть адаптированы для будущих клинических применений. Таким образом, этот подход был использован для выявления нуклеаза деятельности, полученных от Salmonella Typhimurium (здесь называют salmonella) для конкретной идентификации этой бактерии. В следующем протоколе мы сообщаем о методе проверки на бактериальную активность нуклеаза с помощью кинетической экспертизы.
протокол
1. Дизайн и подготовка библиотеки олигонуклеотида
- Дизайн библиотеки
- Основываясь на следующих критериях дизайн библиотеки утоленных флуоресцентных олигонуклеотидных зондов между 8 и 12-Mer долго, чтобы избежать вторичного формирования структуры, с одинаковой длиной для всех зондов.
- Включите по крайней мере одну ДНК и одну РАН случайную последовательность, содержащую комбинацию аденина (A), гуанина (G), цитозина (C) и тимина (T)/uracil (U)(ДНК и РНК-зондов в таблице 1).
ПРИМЕЧАНИЕ: Последовательности ДНК и РНК, описанные в таблице 1, были полезны в качестве первоначальных субстратов для классификации неизвестного типа нуклеаза, либо DNase, либо RNase. Эти две последовательности рекомендуется в качестве отправной точки для любого скрининга, так как они показали широкую возможность для обнаружения nuclease профилей деятельности в бактериях и образцах тканей. Если активность нуклеаза не наблюдается с этими последовательностями ДНК и РНК, требуется разработка дополнительных олигонуклеотидов. - Включите полинуклеотидные последовательности, состоящие из одного типа нуклеотида для определения зависимости последовательности для данной нуклеаза деятельности (ДНК-Поли А, ДНК-Поли Т, ДНК-Поли С, ДНК-Поли G, РНК-Поли А, РНК-Поли У, РНК-Поли С и РНК-Поли G в таблице 1).
- Использование той же последовательности нуклеотидных остатков, как первоначальные последовательности ДНК и РНК, включают последовательности, которые содержат нуклеозидные аналоги, укрывающие химические модификации на 2'-позиции сахара рибоза, такие как 2'-Fluoro и 2'-O-Methyl, как строгий шаг для повышения избирательности нуклеаз.
- Включите полностью модифицированные последовательности, полностью состоящие из аналогов нуклеозидов 2'-Fluoro или 2'-O-Methyl нуклеозидных аналогов (Все 2'-F и Все 2'-OMe, таблица 1)
- Включите химерные последовательности, состоящие из неизмененных натуральных пуринов и 2'-Флуоро пиримидино в нуклеозидных аналогах (RNA Pyr-2'F, таблица 1).
- Включите химерные последовательности, состоящие из неизмененных натуральных пуринов и аналогов нуклеозида 2'O-Methyl pyrimidine (RNA Pyr-2'OMe, таблица 1).
- Включите химерные последовательности, состоящие из неизмененных природных пиримидинов и 2'-Fluoro пуриновые нуклеозидные аналоги (RNA Pur-2'F, Таблица 1).
- Включите химерные последовательности, состоящие из неизмененных природных пиримидинов и 2'O-Метил пуринового нуклеозида аналогов (RNA Pur-2'OMe, Таблица 1).
- Включите по крайней мере одну ДНК и одну РАН случайную последовательность, содержащую комбинацию аденина (A), гуанина (G), цитозина (C) и тимина (T)/uracil (U)(ДНК и РНК-зондов в таблице 1).
- Основываясь на следующих критериях дизайн библиотеки утоленных флуоресцентных олигонуклеотидных зондов между 8 и 12-Mer долго, чтобы избежать вторичного формирования структуры, с одинаковой длиной для всех зондов.
- Подготовка и хранение олигонуклеотидного зонда
ПРИМЕЧАНИЕ: Олигонуклеотиды синтезируются методом фосфорамидита. После синтеза олигонуклеотиды очищаются с помощью высокопроизводительной жидкой хроматографии (HPLC), а масса измеряется массой спектрометрии.- Спин вниз лиофилизированных олигонуклеотидных зондов с помощью центрифуги и разбавить их в Tris-EDTA (TE) буфер для предотвращения деградации нуклеаза. Определите объем разбавления в соответствии с выходом каждого зонда, чтобы сделать складраствор с концентрацией 500 рмоль/ЗЛ.
- Храните лиофилизированные олигонуклеотиды при температуре 4 градуса Цельсия или комнатной температуре в течение короткого срока. Для длительного хранения (от месяцев до лет) храните лиофилизированные олигонуклеотиды при -20 градусов по Цельсию. При повторной приостановке лиофилизированных олигонуклеотидов в TE храните на складе растворы при -20 градусах цельсия или, желательно, при -80 градусов по Цельсию.
2. Бактериальная культура
- Возьмите один пористый стеклянный бисер из криогенного флакона хранения в стерильных условиях.
- Чтобы изолировать отдельные бактериальные колонии(Salmonella и E. coli),используйте метод квадранта24, полосируя/катаясь на бисе прямо на блюдо Петри, содержащее триптической соевой агар (TSA) среды, дополненной дефибринатными овцами Крови.
- Инкубировать культуру при 37 градусах по Цельсию на 24 ч.
3. Супернатанпрепарат препарат
- Перенесите одну колонию из твердых носителей на 50 мл триптической соевого бульона (TSB) и инкубировать при 37 градусах по Цельсию при 24 л.с. при 200 об/мин.
- Разбавить культуру (1:500) в TSB (суб-культура) и инкубировать при 37 градусах по Цельсию на 24 ч при 200 об/мин в тряся инкубаторе.
ПРИМЕЧАНИЕ: Ожидается, что после 24 ч инкубации бактериальные культуры находятся в стационарной фазе, достигая значений выше 109 CFU/mL. - После инкубационного периода, передача культур на ограниченные стерильные трубки и центрифуги на 4500 х г в течение 30 мин.
- Соберите супернатант и используйте его немедленно. Кроме того, храните супернационты при 4 градусах или -20 градусов по Цельсию.
4. Нуклиз едкие действия Ассаи
- Подготовка рабочих решений
- Подготовьте рабочий раствор мощностью 20 л для каждого зонда в нуклеаузных трубах мощностью 1,5 мл, разбавляя (1:10 соотношение) стоковый раствор (500 моль/Л) для конечной рабочей концентрации 50 пмоль/Л. Для этой цели, смесь 18 л фосфатного буферного солен (PBS), содержащего MgCl2 и CaCl2 с 2 злителя раствора запаса зонда.
ПРИМЕЧАНИЕ: Имейте в виду, что в рабочем решении, олигонуклеотиды более уязвимы для деградации нуклеаны, поэтому, рекомендуется подготовить рабочие решения непосредственно перед созданием реакции. - Избегайте прямого контакта света во время подготовки и держать в темноте (завернутый в фольгу), при работе с флюорофорами.
- Подготовьте рабочий раствор мощностью 20 л для каждого зонда в нуклеаузных трубах мощностью 1,5 мл, разбавляя (1:10 соотношение) стоковый раствор (500 моль/Л) для конечной рабочей концентрации 50 пмоль/Л. Для этой цели, смесь 18 л фосфатного буферного солен (PBS), содержащего MgCl2 и CaCl2 с 2 злителя раствора запаса зонда.
- Реакция настроена
- Предварительно разогреть фторметр (например, цитирование 1) до 37 градусов по Цельсию.
- Подготовьте набор (одна трубка/зонд) из 1,5 мл нуклеаза бесплатно микроцентрифуговых труб для каждого образца (TSB, E. coli и Salmonella)и этикетки соответственно.
- Аккуратно добавьте 96 зЛ стерильных средств массовой информации tSB, супернатант сальмонеллы или супернатант E. coli (с шага 3.4.). Впоследствии, добавить 4 Л /трубки зонда рабочего решения соответственно. Выполните этот шаг при комнатной температуре.
ПРИМЕЧАНИЕ: 10 зондов были использованы для первого раунда скрининга и 6 зондов для второго раунда. - Тщательно перемешайте, повысив вверх и вниз, чтобы получить однородное решение. Избегайте введения пузырьков воздуха в образцы при смешивании.
- Загрузите 95 зл каждого раствора (зондик - супернатант или культурные носители) в отдельный колодец черного дна, не обработанную пластину 96 скважин. Свести к минимуму образование пузырьков в колодцах при загрузке, тщательно раздавсив кончик близко к стене колодца.
- Накройте тарелку крышкой. Визуально осматривать крышку и проверять на наличие маркировки пера или накопления частиц пыли, которые могут ввести артефакты измерения. Замените крышку на новую, если это так.
- Настройка измерения
- Откройте приобретение программного обеспечения (Gen5 3.05), нажав на значок ярлыка программного обеспечения(рисунок S1A).
- Выберите Read Now из окна диспетчера задач и выберите Новый...
- Нажмите на Установить Температуру в окне диалога под названием Процедура и выберите 37 градусов по Цельсию. Подтвердите и сохраните настройки, нажав OK (Рисунок S1C).
- Нажмите на кнопку «Пуск кинетика» в окне диалога под названием «Процедура». Затем в всплывающем диалоговом окне, озаглавленном Kinetic Step,выберите 2 ч в входной коробке Run Time и 2 мин в входной коробке Interval. Подтвердите и сохраните настройки, нажав OK (Рисунок S1D).
- Нажмите на Чтение в окне диалога под названием Процедура. Затем в всплывающем диалоговом окне, озаглавленном Read Method, выберите интенсивность флуоресценции в качестве метода обнаружения, Endpoint/Kinetic как тип чтения и фильтры как тип оптики. Подтвердите и сохраните настройки, нажав OK (Рисунок S1E).
- В всплывающем окне диалога под названием Read Step (Кинетическая)выберите зеленый цвет из набора фильтров. Подтвердите и сохраните настройки, нажав OK (Рисунок S2A).
- В окне диалога под названием Процедуравыберите крышку и нажмите на проверку, чтобы убедиться, что созданный протокол действителен. Это подтверждается всплывающее окно диалога(рисунок S2B).
- Выберите протокол в меню бар и выбрать процедуру (рисунок S2C).
- В окне диалога под названием Процедура, определить скважины должны быть измерены(рисунок S2D).
- Введите название эксперимента в поле ввода имени файла(рисунок S2E).
- Загрузите тарелку крышкой в считыватель пластины. Убедитесь, что пластина находится в нужной ориентации.
- Начните приобретение, нажав на новую кнопку чтения в панели инструментов(Рисунок S2F).
- Анализ данных
- Нажмите на одну из измеренных скважин в диалоговом окне под названием Плита 1 (рисунок S3A).
- Нажмите выберите скважины и включите все измеренные скважины в окно диалога выбора скважины. Подтвердите и сохраните настройки, нажав OK. (Рисунок S3B).
- Выберите данные в окне диалога под названием Плита 1, чтобы визуализировать результаты, сформулированные(рисунок S3C).
- Экспорт данных в спредлист, выбрав быстрый экспорт из контекстного меню(рисунок S3C).
- В спред-листе пометьте столбцы данных соответствующим образом для каждого образца и зонда.
- Создание кинетических графиков, используя линию со стилем маркеров, путем построения относительных флуоресценции (RFU) по сравнению со временем (x-оси: хронология реакции и y-оси: RFUs).
ПРИМЕЧАНИЕ: Описание создания графика применимо при использовании программ электронных таблиц (например, Excel). Тем не менее, любые другие программы могут быть использованы для создания графиков из необработанных данных, полученных, путем построения РФС по сравнению со временем. - Рассчитайте скорость ферментативной реакции для каждого измеренного интервала в соответствии со следующей формулой25: скорость
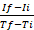 , где если РФС в максимальной временной точке времени и Ii является РФС в минимальном интервале времени, и Tf и Ti являются максимальными и минимальными временными точками интервала, соответственно.
, где если РФС в максимальной временной точке времени и Ii является РФС в минимальном интервале времени, и Tf и Ti являются максимальными и минимальными временными точками интервала, соответственно. - Выберите скорость с наивысшим значением (Rmax)для каждой кривой. Если на образец приходится более одногоR max, выберите тот, который происходит в самой ранней точке времени измерения.
- Рассчитайте соотношение между Rmax и средним значением интервала времени, с которым происходит Rmax: коэффициент скорости
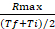
- Рассчитайте разницу в раз (FD) между коэффициентом скорости salmonella и E. coli для каждого зонда.
- Учитывайте значение разницы в развыше 3 как значительное.
- Рассмотрим значительные зонды с самыми высокими значениями разницы складов (FD) в качестве кандидатов зондов и в качестве основы для разработки библиотеки в следующем раунде отбора.
5. Критерии отбора раундов скрининга(рисунок 1)
- Первый раунд скрининга
- Выполните анализ активности нуклеаза (как описано в разделе 4) с использованием ДНК, РНК и полинуклеотидов зондов.
- Оцените предпочтение химии ДНК или ХИМИи РНК, сравнив количество и производительность (значение FD) днк и зондов-кандидатов НА РНК.
- Выберите тип химии нуклеиновой кислоты, что делает наибольшее количество зондов-кандидатов, как показано на рисунке 1.
- Второй раунд скрининга
- Выполните анализ активности нуклеаза (как описано в разделе 4) с использованием полностью модифицированных зондов и химерных зондов, разработанных на основе субстрата нуклеиновой кислоты, выбранного в первом раунде отбора.
- Оцените предпочтение последовательностям, содержащим 2'-Fluoro и 2'-O-Methyl нуклеозидные аналоги, сравнивая результаты, полученные для 2'-Fluoro и 2'-O-Methyl модифицированных зондов-кандидатов.
- Выберите тип нуклеозидной аналоговой модификации, оказывая наибольшее количество зондов-кандидатов, как показано на рисунке 1.
Результаты
На рисунке 1 показан рабочий поток этой методологии, которая делится на два раунда отбора. В первом раунде скрининга, мы использовали 5 ДНК зондов (ДНК, ДНК Поли A, ДНК Поли T, ДНК Поли C и ДНК Поли G), а также 5 РНК зондов (РНК, РНК Поли A, РНК Поли U РНК Поли C и РНК Поли G). Необработ?...
Обсуждение
Изменения активности нуклеаны были связаны с широким спектром фенотипов заболеваний, в ключая различные виды рака и бактериальных инфекций. Эти изменения предлагается быть возбудителем состояния14, в то время как в других случаях они являются следствием пагубного физиоло...
Раскрытие информации
Авторам нечего раскрывать.
Благодарности
Авторы хотели бы отметить Луису И. Эрнандес (Университет Линчепинга) за ее тщательный пересмотр рукописи и ценные советы. Эта работа была поддержана Фондом Кнута и Алисы Валленберг и Шведским правительством по стратегическим исследованиям в области материаловедения по перспективным функциональным материалам в Университете Линчепинга (Faculty Grant SFO-Mat-LiU No 2009-00971).
Материалы
| Name | Company | Catalog Number | Comments |
| Black bottom, non-treated 96 well plate | Fisher Scientific | 10000631 | |
| Cytation1 | BioTek | CYT1FAV | |
| Eppendorf tubes | Thermofisher | 11926955 | |
| Escherichia coli | ATCC | 25922 | |
| Microbank cryogenic storage vial containing beads | Pro-Lab Diagnostics | 22-286-155 | |
| Nucleic acid probes | Biomers.net | # | |
| Phosphate Buffer Saline containing MgCl2 and CaCl2 | Gibco™ | 14040117 | |
| Salmonella enterica subs. Enterica | ATCC | 14028 | |
| Tris-EDTA | Fisher Scientific | 10647633 | |
| Tryptone Soya Agar with defibrinated sheep blood | Thermo Fisher Scientific | 10362223 | |
| Tryptic Soy Broth | Sigma Aldrich | 22092 |
Ссылки
- Yang, W. Nucleases: diversity of structure, function and mechanism. Quarterly Reviews of Biophysics. 44 (1), 1-93 (2011).
- Adli, M. The CRISPR tool kit for genome editing and beyond. Nature Communication. 9 (1), 1911 (2018).
- Mason, P. A., Cox, L. S. The role of DNA exonucleases in protecting genome stability and their impact on ageing. Age (Dordr). 34 (6), 1317-1340 (2012).
- Berends, E. T., et al. Nuclease expression by Staphylococcus aureus facilitates escape from neutrophil extracellular traps. Journal of Innate Immunity. 2 (6), 576-586 (2010).
- Kiedrowski, M. R., et al. Staphylococcus aureus Nuc2 is a functional, surface-attached extracellular nuclease. PLoS One. 9 (4), 95574 (2014).
- Morita, C., et al. Cell wall-anchored nuclease of Streptococcus sanguinis contributes to escape from neutrophil extracellular trap-mediated bacteriocidal activity. PLoS One. 9 (8), 103125 (2014).
- Hasegawa, T., et al. Characterization of a virulence-associated and cell-wall-located DNase of Streptococcus pyogenes. Microbiology. 156, 184-190 (2010).
- Moon, A. F., et al. Structural insights into catalytic and substrate binding mechanisms of the strategic EndA nuclease from Streptococcus pneumoniae. Nucleic Acids Research. 39 (7), 2943-2953 (2011).
- Li, L. Y., Luo, X., Wang, X. Endonuclease G is an apoptotic DNase when released from mitochondria. Nature. 412 (6842), 95-99 (2001).
- McDermott-Roe, C., et al. Endonuclease G is a novel determinant of cardiac hypertrophy and mitochondrial function. Nature. 478 (7367), 114-118 (2011).
- Wang, Y. T., et al. A link between adipogenesis and innate immunity: RNase-L promotes 3T3-L1 adipogenesis by destabilizing Pref-1 mRNA. Cell Death & Disease. 7 (11), 2458 (2016).
- Li, C. L., Yang, W. Z., Shi, Z., Yuan, H. S. Tudor staphylococcal nuclease is a structure-specific ribonuclease that degrades RNA at unstructured regions during microRNA decay. RNA. 24 (5), 739-748 (2018).
- Wan, L., et al. MTDH-SND1 interaction is crucial for expansion and activity of tumor-initiating cells in diverse oncogene- and carcinogen-induced mammary tumors. Cancer Cell. 26 (1), 92-105 (2014).
- Keyel, P. A. Dnases in health and disease. Developmental Biology. 429 (1), 1-11 (2017).
- Hernandez, F. J., et al. Noninvasive imaging of Staphylococcus aureus infections with a nuclease-activated probe. Nature Medicine. 20 (3), 301-306 (2014).
- Flenker, K. S., et al. Rapid Detection of Urinary Tract Infections via Bacterial Nuclease Activity. Molecular Therapy. 25 (6), 1353-1362 (2017).
- Kannan, N., Eaves, C. J. Tipping the balance: MTDH-SND1 curbs oncogene-induced apoptosis and promotes tumorigenesis. Cell Stem Cell. 15 (2), 118-120 (2014).
- Kemmer, T. P., Malfertheiner, P., Buchler, M., Kemmer, M. L., Ditschuneit, H. Serum ribonuclease activity in the diagnosis of pancreatic disease. International Journal of Pancreatology. 8 (1), 23-33 (1991).
- Fernandez-Salas, E., Peracaula, R., Frazier, M. L., de Llorens, R. Ribonucleases expressed by human pancreatic adenocarcinoma cell lines. European Journal of Biochemistry. 267 (5), 1484-1494 (2000).
- Fujibayashi, K., et al. Serum deoxyribonuclease I activity can be a useful diagnostic marker for the early diagnosis of unstable angina pectoris or non-ST-segment elevation myocardial infarction. Journal of Cardiology. 59 (3), 258-265 (2012).
- Kawai, Y., et al. Diagnostic use of serum deoxyribonuclease I activity as a novel early-phase marker in acute myocardial infarction. Circulation. 109 (20), 2398-2400 (2004).
- Hernandez, L. I., Ozalp, V. C., Hernandez, F. J. Nuclease activity as a specific biomarker for breast cancer. Chemical Communication (Cambridge). 52 (83), 12346-12349 (2016).
- Machado, I., et al. Rapid and specific detection of Salmonella infections using chemically modified nucleic acid probes. Analytica Chimica Acta. 1054, 157-166 (2019).
- Sanders, E. R. Aseptic laboratory techniques: plating methods. Journal of Visualized Experiment. (63), e3064 (2012).
- Worthington Biochemical Corporation. . Manual of Clinical Enzyme Measurements. , (1972).
- Burghardt, E. L., et al. Rapid, Culture-Free Detection of Staphylococcus aureus Bacteremia. PLoS One. 11 (6), 0157234 (2016).
- Bibette, J. Gaining confidence in high-throughput screening. Proceedings of the National Academy of Science U. S. A. 109 (3), 649-650 (2012).
- Hernandez, L. I., Machado, I., Schafer, T., Hernandez, F. J. Aptamers overview: selection, features and applications. Current Topics in Medicinal Chemistry. 15 (12), 1066-1081 (2015).
- Pande, J., Szewczyk, M. M., Grover, A. K. Phage display: concept, innovations, applications and future. Biotechnology Advances. 28 (6), 849-858 (2010).
- Skerra, A. Alternative binding proteins: anticalins - harnessing the structural plasticity of the lipocalin ligand pocket to engineer novel binding activities. FEBS Journal. 275 (11), 2677-2683 (2008).
- Ku, T. H., et al. Nucleic Acid Aptamers: An Emerging Tool for Biotechnology and Biomedical Sensing. Sensors (Basel). 15 (7), 16281-16313 (2015).
- Jayasena, S. D. Aptamers: an emerging class of molecules that rival antibodies in diagnostics. Clinical Chemistry. 45 (9), 1628-1650 (1999).
- Gray, B. P., Brown, K. C. Combinatorial peptide libraries: mining for cell-binding peptides. Chemical Reviews. 114 (2), 1020-1081 (2014).
Перепечатки и разрешения
Запросить разрешение на использование текста или рисунков этого JoVE статьи
Запросить разрешениеСмотреть дополнительные статьи
This article has been published
Video Coming Soon
Авторские права © 2025 MyJoVE Corporation. Все права защищены