Zum Anzeigen dieser Inhalte ist ein JoVE-Abonnement erforderlich. Melden Sie sich an oder starten Sie Ihre kostenlose Testversion.
Method Article
Kinetisches Screening der Nuklease-Aktivität mit Nukleinsäuresonden
* Diese Autoren haben gleichermaßen beigetragen
In diesem Artikel
Zusammenfassung
Veränderte Nuklease-Aktivität wurde mit verschiedenen menschlichen Bedingungen verbunden, die ihr Potenzial als Biomarker zugrunde liegen. Die modulare und einfach zu implementierende Screening-Methodik, die in diesem Papier vorgestellt wird, ermöglicht die Auswahl spezifischer Nukleinsäuresonden zur Nutzung der Nukleaseaktivität als Biomarker von Krankheiten.
Zusammenfassung
Nukleasen sind eine Klasse von Enzymen, die Nukleinsäuren abbauen, indem sie die Hydrolyse der Phosphodiesterbindungen katalysieren, die die Ribosezucker verbinden. Nukleasen weisen eine Vielzahl lebenswichtiger physiologischer Rollen in prokaryotischen und eukaryotischen Organismen auf, die von der Aufrechterhaltung der Genomstabilität bis hin zum Schutz vor Krankheitserregern reichen. Veränderte Nuklease-Aktivität wurde mit mehreren pathologischen Bedingungen verbunden, einschließlich bakterieller Infektionen und Krebs. Zu diesem Zweck hat die Nukleaseaktivität ein großes Potenzial gezeigt, als spezifischer Biomarker genutzt zu werden. Ein robustes und reproduzierbares Screening-Verfahren auf der Grundlage dieser Aktivität ist jedoch nach wie vor sehr wünschenswert.
Hierin führen wir ein Verfahren ein, das das Screening auf Nukleaseaktivität mit Nukleinsäuresonden als Substraten ermöglicht, mit dem Umfang der Unterscheidung zwischen pathologischen und gesunden Bedingungen. Diese Methode bietet die Möglichkeit, neue Sondenbibliotheken mit zunehmender Spezifität iterativ zu gestalten. Daher sind mehrere Screening-Runden notwendig, um das Design der Sonden mit verbesserten Eigenschaften zu verfeinern und dabei die Verfügbarkeit chemisch modifizierter Nukleinsäuren zu nutzen. Das beträchtliche Potenzial der vorgeschlagenen Technologie liegt in ihrer Flexibilität, hohen Reproduzierbarkeit und Vielseitigkeit für das Screening von Nukleaseaktivitäten im Zusammenhang mit Krankheitszuständen. Es wird erwartet, dass diese Technologie die Entwicklung vielversprechender diagnostischer Werkzeuge mit großem Potenzial in der Klinik ermöglichen wird.
Einleitung
Nukleasen sind eine Klasse von Enzymen, die in der Lage sind, die Phosphodiesterbindungen zu spalten, die die Rückgratstruktur von Nukleinsäuremolekülen bilden. Die große Vielfalt der Nukleasen macht ihre Klassifizierung ziemlich schwierig. Es gibt jedoch einige gängige Kriterien, die zur Beschreibung von Nukleasen verwendet werden, wie substratpräferenzium (Deoxyribonukleinsäure (DNA) oder Ribonukleinsäure (RNA)), Spaltungsstelle (Endonukleasen oder Exonukleasen) oder Metallionenabhängigkeit, unter anderem1. Nukleasen sind hochkonservierte katalytische Enzyme, die sowohl in prokaryotischen als auch in eukaryotischen Organismen eine grundlegendeRolle spielen und als Genbearbeitungswerkzeuge verwendet wurden und weiterhin verwendet werden 2 . Sie sind auch grundlegende Akteure in der DNA-Wartung und -Replikation, helfen, die Genomstabilität zu erhalten und nehmen an Korrekturverfahren teil3. Bei Bakterien wurden beispielsweise Nukleasen als wichtige Virulenzfaktoren identifiziert, die das überleben der Bakterien fördern können, indem sie die Wirksamkeit desImmunsystemsdes Wirts 4,5,6,7 ,8. Bei Säugetieren, Nukleasen wurden vorgeschlagen, in Apoptose9verwickelt werden , mitochondriale Biogenese und Wartung10 und Vermittlung von antibakteriellen und antiviralen angeborenen Immunantworten11. Es überrascht nicht, dass Nuklease-Aktivitätsänderungen, ob Verbesserung oder Fehlen von, in eine Vielzahl von menschlichen Krankheiten verwickelt sind. Diese Krankheiten reichen von einer Vielzahl von Krebsarten12,13 bis zu Herzhypertrophie10 oder Autoimmunerkrankungen14. Daher sind Nukleasen zu interessanten Kandidaten als Biomarker für eine heterogene Gruppe menschlicher Bedingungen geworden. Tatsächlich haben Nukleasen bereits ihr Potenzial als erfolgreiche diagnostische Instrumente zum Nachweis von Infektionen gezeigt, die durch bestimmte bakterielle Wirkstoffe wie Staphylococcus aureus oder Escherichia coli15 verursacht werden. 16. Bei vielen Krebsarten ist die Expression von Staphylokokkennuklease-Domänen-haltigem Protein 1 (SND1) Ribonuklease ein Hinweis auf eine schlechte Prognose17. Bei Patienten mit Bauchspeicheldrüsenkrebs wurden erhöhte Ribonuklease-I-Serumspiegel18 berichtet und vorgeschlagen, mit Krebszellphänotypen in Verbindung gebracht zu werden19. Bei ischämischen Herzerkrankungen, wie Myokardinfarkt oder instabiler Angina pectoris, desoxyribonuklease I (DNase I) Serumspiegel haben sich als ein gültiger diagnostischer Marker20,21.
Es wurde vermutet, dass die globale Blaupause der Nuklease-Aktivität in gesunden und Krankheitszuständen unterschiedlich sein kann. Tatsächlich haben jüngste Berichte Unterschiede in der Nukleaseaktivität verwendet, um zwischen gesunden und krebsernen Phänotypen22 zu unterscheiden oder pathogene bakterielle Infektionen artspezifisch zu identifizieren15,23. Diese Erkenntnisse haben einen neuen Weg für den Einsatz von Nukleasen als Biomarker von Krankheiten eröffnet. Daher besteht die Notwendigkeit für die Entwicklung einer umfassenden Screening-Methode, die in der Lage ist, krankheitsassoziierte Unterschiede in der Nukleaseaktivität systematisch zu identifizieren, die bei der Entwicklung neuer diagnostischer Instrumente von entscheidender Bedeutung sein können.
Hierin führen wir einen neuen In-vitro-Screening-Ansatz ein (Abbildung 1), um empfindliche und spezifische Sonden zu identifizieren, die in der Lage sind, zwischen Nukleaseaktivität bei gesunder und ungesundem Oder einer spezifischen Aktivität einer Zell- oder Bakterienart zu unterscheiden. Unter Ausnutzung der Modularität von Nukleinsäuren haben wir eine erste Bibliothek von abgeschreckten fluoreszierenden Oligonukleotid-Sonden entwickelt, die aus einem umfassenden Satz verschiedener Sequenzen und Chemikalien bestehen, die beide wichtige Parameter für die Bibliotheksgestaltung sind. Diese Oligonukleotid-Sonden werden von einem Fluorophor (Fluorescein-Amidit, FAM) und einem Quencher (Tide Quencher 2, TQ2) an den 5' bzw. 3' Enden(Tabelle 1) flankiert. Durch die Verwendung dieses fluoreszierenden Resonanzenergietransfers (FRET) basierenden fluormetrischen Assays zur Messung der Kinetik des enzymatischen Abbaus konnten wir Kandidatensonden mit dem Potenzial identifizieren, differentiale Muster der Nukleaseaktivität zu unterscheiden. mit gesunden oder Krankheitszuständen verbunden sind. Wir haben einen iterativen Prozess entwickelt, bei dem neue Bibliotheken auf der Grundlage der besten Kandidatensonden erstellt werden, der es ermöglicht, immer spezifischere Kandidatensonden in nachfolgenden Screening-Schritten zu identifizieren. Darüber hinaus nutzt dieser Ansatz die katalytische Natur von Nukleasen, um die Empfindlichkeit zu erhöhen. Dies wird erreicht, indem die aktivierbare Natur der Reportersonden und die Fähigkeit von Nukleasen genutzt werden, Substratmoleküle kontinuierlich zu verarbeiten, die beide wichtige Vorteile gegenüber alternativen Antikörpern oder kleinen molekülbasierten Screening-Methoden darstellen.
Dieser Ansatz bietet ein hochmodulares, flexibles und einfach zu implementierendes Screening-Tool zur Identifizierung spezifischer Nukleinsäuresonden, die in der Lage sind, gesunde und krankheitsfördernde Erkrankungen zu unterscheiden, und eine hervorragende Plattform für die Entwicklung neuer diagnostische Nagenzwerkzeuge, die für zukünftige klinische Anwendungen angepasst werden können. Als solcher wurde dieser Ansatz verwendet, um die Nuklease-Aktivität von Salmonella Typhimurium (hier als Salmonellabezeichnet ) für die spezifische Identifizierung dieser Bakterien abgeleitet zu identifizieren. Im folgenden Protokoll berichten wir über eine Methode zum Abschirmen auf bakterielle Nukleaseaktivität mittels kinetischer Analyse.
Protokoll
1. Oligonukleotid Bibliothek Design und Vorbereitung
- Bibliotheksdesign
- Basierend auf den folgenden Kriterien entwerfen Sie eine Bibliothek von abgeschreckten fluoreszierenden Oligonukleotid-Sonden zwischen 8 und 12-mer lang, um sekundäre Strukturbildung zu vermeiden, mit gleicher Länge für alle Sonden.
- Schließen Sie mindestens eine DNA- und eine RNA-Zufallssequenz ein, die eine Kombination aus Adenin (A), Guanin (G), Cytosin (C) und Thymin (T)/Uracil (U) enthält (DNA- und RNA-Sonden in Tabelle 1).
HINWEIS: Die in Tabelle 1 beschriebenen DNA- und RNA-Sequenzen waren als Anfangssubstrate für die Klassifizierung einer unbekannten Art von Nukleaseaktivität, entweder DNase oder RNase, nützlich. Diese beiden Sequenzen werden als Ausgangspunkt für jedes Screening empfohlen, da sie eine breite Fähigkeit gezeigt haben, Nuklease-Aktivitätsprofile in Bakterien und Gewebeproben zu erkennen. Wenn die Nukleaseaktivität mit diesen DNA- und RNA-Sequenzen nicht beobachtet wird, ist die Entwicklung zusätzlicher Oligonukleotide erforderlich. - Umfassen Sie Polynukleotidsequenzen, die aus einem einzigen Nukleotidtyp bestehen, um die Sequenzabhängigkeit für eine gegebene Nukleaseaktivität zu bestimmen (DNA-Poly A, DNA-Poly T, DNA-Poly C, DNA-Poly G, RNA-Poly A, RNA-Poly U, RNA-Poly C und RNA-Poly G in Tabelle 1).
- Unter Verwendung der gleichen Sequenz von Nukleotid-Rückständen wie die ursprünglichen DNA- und RNA-Sequenzen umfassen Sequenzen, die Nukleosidanaloge enthalten, die chemische Modifikationen an der 2'-Position des Ribosezuckers, wie 2'-Fluoro und 2'-O-Methyl, als einen strengen Schritt enthalten. zur Erhöhung der Selektivität der Nukleasen.
- Vollständige modifizierte Sequenzen, die vollständig aus 2'-Fluorosid-Analogen oder 2'-O-Methyl-Nukleosid-Analogen bestehen (Alle 2'-F und Alle 2'-OMe, Tabelle 1)
- Schließen Sie chimäre Sequenzen ein, die aus unveränderten natürlichen Purinen und 2'-Fluoropyrimidin-Nukleosid-Analogen bestehen (RNA Pyr-2'F, Tabelle 1).
- Schließen Sie chimäre Sequenzen ein, die aus unveränderten natürlichen Purinen und 2'-O-Methylpyrimidin-Nukleosid-Analogen bestehen (RNA Pyr-2'OMe, Tabelle 1).
- Schließen Sie chimäre Sequenzen ein, die aus unveränderten natürlichen Pyrimiden und 2'-Fluor-Purin-Nukleosid-Analogen bestehen (RNA Pur-2'F, Tabelle 1).
- Schließen Sie chimäre Sequenzen ein, die aus unveränderten natürlichen Pyrimiden und 2'-O-Methyl-Purin-Nukleosid-Analogen bestehen (RNA Pur-2'OMe, Tabelle 1).
- Schließen Sie mindestens eine DNA- und eine RNA-Zufallssequenz ein, die eine Kombination aus Adenin (A), Guanin (G), Cytosin (C) und Thymin (T)/Uracil (U) enthält (DNA- und RNA-Sonden in Tabelle 1).
- Basierend auf den folgenden Kriterien entwerfen Sie eine Bibliothek von abgeschreckten fluoreszierenden Oligonukleotid-Sonden zwischen 8 und 12-mer lang, um sekundäre Strukturbildung zu vermeiden, mit gleicher Länge für alle Sonden.
- Oligonukleotid-Sonde Vorbereitung und Lagerung
HINWEIS: Oligonukleotide werden mit der Phosphoramidit-Methode synthetisiert. Nach der Synthese werden die Oligonukleotide mittels Hochleistungsflüssigkeitschromatographie (HPLC) gereinigt und die Masse mittels Massenspektrometrie gemessen.- Drehen Sie die lyophilisierten Oligonukleotid-Sonden mit einer Zentrifuge herunter und verdünnen Sie sie im Tris-EDTA (TE)-Puffer, um einen Nukleaseabbau zu verhindern. Bestimmen Sie das Verdünnungsvolumen entsprechend der Ausbeute jeder Sonde, um eine Lagerlösung mit einer Konzentration von 500 pmol/l zu rendern.
- Lyophilisierte Oligonukleotide bei 4 °C oder raumtemperatur kurzfristig lagern. Für die Langzeitlagerung (Monate bis Jahre) lyophilisierte Oligonukleotide bei -20 °C lagern. Bei Resuspension lyophilisierter Oligonukleotide in TE die Lagerlösungen bei -20 °C oder vorzugsweise bei -80 °C lagern.
2. Bakterielle Kultur
- Nehmen Sie eine poröse Glasperle aus der kryogenen Durchstechflasche unter sterilen Bedingungen.
- Um einzelne Bakterienkolonien (Salmonellen und E. coli)zu isolieren, verwenden Sie die Quadrantenmethode24, indem Sie die Perle direkt auf eine Petrischale mit Tryptic Soy Agar (TSA) Medium stutzen, das mit defibrinierten Schafen ergänzt wird. blut.
- Inkubieren Sie die Kultur bei 37 °C für 24 h.
3. Supernatante Vorbereitung
- Übertragen Sie eine einzelne Kolonie von festen Medien auf 50 ml Tryptic Soy Broth (TSB) und brüten bei 37 °C für 24 h bei 200 Rpm.
- Die Kultur (1:500) in TSB (Subkultur) verdünnen und bei 37 °C für 24 h bei 200 U/min in einem Schüttelinkubator brüten.
HINWEIS: Es wird erwartet, dass sich die Bakterienkulturen nach 24 h Inkubation in einer stationären Phase befinden und Werte über 109 KBE/ml erreichen. - Nach der Inkubationszeit die Kulturen 30 min auf gekapselte sterile Schläuche und Zentrifugen bei 4.500 x g übertragen.
- Sammeln Sie den Überstand und verwenden Sie ihn sofort. Alternativ können Sie die Überräube bei 4 °C oder -20 °C lagern.
4. Nuklease Aktivität Assay
- Vorbereitung von Arbeitslösungen
- Bereiten Sie für jede Sonde in 1,5 ml Nuklease-freien Mikrozentrifugenrohren eine 20-L-Arbeitslösung vor, indem Sie die Stammlösung (500 pmol/l) für eine Endarbeitskonzentration von 50 pmol/l verdünnen (1:10 Verhältnis). Zu diesem Zweck mischen Sie 18 l Phosphatpuffer-Saline (PBS), die MgCl2 und CaCl2 enthalten, mit 2 l Sondenlagerlösung.
HINWEIS: Beachten Sie, dass in der Arbeitslösung die Oligonukleotide anfälliger für Nukleaseabbau sind, daher wird empfohlen, die Arbeitslösungen kurz vor der Einrichtung der Reaktion vorzubereiten. - Vermeiden Sie direkten Lichtkontakt während der Vorbereitung und halten Sie im Dunkeln (in Folie gewickelt), wenn Sie mit Fluorophoren umgehen.
- Bereiten Sie für jede Sonde in 1,5 ml Nuklease-freien Mikrozentrifugenrohren eine 20-L-Arbeitslösung vor, indem Sie die Stammlösung (500 pmol/l) für eine Endarbeitskonzentration von 50 pmol/l verdünnen (1:10 Verhältnis). Zu diesem Zweck mischen Sie 18 l Phosphatpuffer-Saline (PBS), die MgCl2 und CaCl2 enthalten, mit 2 l Sondenlagerlösung.
- Reaktion eingerichtet
- Das Fluorometer (z.B. Cytation 1) auf 37 °C vorwärmen.
- Bereiten Sie für jede Probe (TSB, E. coli und Salmonella ) ein Set (ein Rohr/Sonde) von 1,5 ml Nuklease-freien Mikrozentrifugenröhrchen vor und etikettierenSie entsprechend.
- Fügen Sie vorsichtig 96 L TSB sterile Kulturmedien, Salmonellen-Überstand oder E. coli Überstand hinzu (ab Schritt 3.4.). Anschließend fügen Sie die Sondenarbeitslösung 4 l/L/Rohr entsprechend hinzu. Führen Sie diesen Schritt bei Raumtemperatur aus.
HINWEIS: 10 Sonden wurden für die erste Runde des Screenings und 6 Sonden für die zweite Runde verwendet. - Mischen Sie gründlich durch Pipettieren nach oben und unten, um eine homogene Lösung zu erhalten. Vermeiden Sie das Einführen von Luftblasen in die Proben während des Mischens.
- Laden Sie 95 l jeder Lösung (Sonde + Überstand oder Kulturmedien) in einen separaten Brunnen einer schwarzen, nicht behandelten 96-Wellplatte. Minimieren Sie die Bildung von Blasen in den Brunnen beim Beladen, indem Sie sorgfältig mit der Spitze in der Nähe der Wand des Brunnens verzichten.
- Bedecken Sie die Platte mit ihrem Deckel. Überprüfen Sie den Deckel visuell und prüfen Sie, ob Stiftmarkierungen oder Staubpartikelansammlungen, die Messartefakte einführen können, überprüft werden. Ersetzen Sie den Deckel gegen einen neuen, wenn dies der Fall ist.
- Messeinrichtung
- Öffnen Sie die Erfassungssoftware (Gen5 3.05), indem Sie auf das Symbol für die Softwareverknüpfung klicken (Abbildung S1A).
- Wählen Sie Jetzt lesen aus dem Aufgaben-Manager-Fenster und wählen Sie Neu... um das kinetische Messprotokoll zu erstellen (Abbildung S1B).
- Klicken Sie im Dialogfenster prozedural auf Temperatur festlegen und wählen Sie 37 °C aus. Bestätigen und speichern Sie die Einstellungen, indem Sie OK drücken (Abbildung S1C).
- Klicken Sie im Dialogfenster mit dem Titel Procedureauf Kinetics starten. Wählen Sie dann im Popup-Dialogfenster mit dem Titel Kinetic Step2 h im Eingabefeld Laufzeit und 2 min im Intervalleingabefeld aus. Bestätigen und speichern Sie die Einstellungen, indem Sie OK drücken (Abbildung S1D).
- Klicken Sie im Dialogfenster mit dem Titel Prozedurauf Lesen . Wählen Sie dann im Popup-Dialogfenster mit dem Titel Lesemethode Fluoreszenzintensität als Erkennungsmethode, Endpunkt/Kinetik als Lesetyp und Filter als Optiktyp aus. Bestätigen und speichern Sie die Einstellungen, indem Sie OK drücken (Abbildung S1E).
- Wählen Sie im Popup-Dialogfenster Leseschritt (Kinetisch) Grün aus dem Filtersatzaus. Bestätigen und speichern Sie die Einstellungen, indem Sie OK drücken (Abbildung S2A).
- Wählen Sie im Dialogfeld prozedural den Deckel aus, und klicken Sie auf Überprüfen, um sicherzustellen, dass das erstellte Protokoll gültig ist. Dies wird durch ein Popup-Dialogfenster bestätigt (Abbildung S2B).
- Wählen Sie Protokoll in der Menüleiste und wählen Sie Prozedur (Abbildung S2C).
- Definieren Sie im Dialogfenster prozedurierenSie die zu messenden Brunnen (Abbildung S2D).
- Geben Sie den Namen des Experiments in das Eingabefeld Dateiname ein (Abbildung S2E).
- Laden Sie die Platte mit dem Deckel in den Plattenleser. Stellen Sie sicher, dass sich die Platte in der richtigen Ausrichtung befindet.
- Starten Sie die Erfassung, indem Sie auf die Schaltfläche "Neue Taste lesen" in der Symbolleiste klicken (Abbildung S2F).
- Datenanalyse
- Klicken Sie auf eine der gemessenen Brunnen im Dialogfenster mit dem Titel Platte 1 (Abbildung S3A).
- Klicken Sie auf Brunnen auswählen und alle gemessenen Brunnen in das Dialogfenster Brunnenauswahl ein. Bestätigen und speichern Sie die Einstellungen, indem Sie OKdrücken. (Abbildung S3B).
- Wählen Sie Daten im Dialogfeld mit dem Titel Platte 1 aus, um die tabellarischen Ergebnisse zu visualisieren (Abbildung S3C).
- Exportieren Sie die Daten in ein Spreadsheet, indem Sie den Schnellexport aus dem Kontextmenü auswählen (Abbildung S3C).
- Beschriften Sie im Spreadsheet die Datenspalten für jede Probe und jeden Prüfpunkt entsprechend.
- Generieren Sie kinetische Diagramme unter Verwendung des Linienstils mit Markern, indem Sie relative Fluoreszenzeinheiten (RFU) im Vergleich zur Zeit (x-Achse: Zeitachse der Reaktion und y-Achse: RFUs) darstellen.
HINWEIS: Die Beschreibung der Erstellung eines Diagramms ist anwendbar, wenn Tabellenkalkulationsprogramme (z. B. Excel) verwendet werden. Alle anderen Programme können jedoch verwendet werden, um Diagramme aus den erhaltenen Rohdaten zu generieren, indem RFU im Vergleich zur Zeit dargestellt wird. - Berechnen Sie die Rate der enzymatischen Reaktion für jedes gemessene
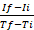 Intervall nach der folgenden Formel25: rate= , wobei If is RFU beim maximalen Intervallzeitpunkt und Ii RFU beim minimalen Intervallzeitpunkt ist, Und Tf und Ti sind die maximalen bzw. minimalen Intervallzeitpunkte.
Intervall nach der folgenden Formel25: rate= , wobei If is RFU beim maximalen Intervallzeitpunkt und Ii RFU beim minimalen Intervallzeitpunkt ist, Und Tf und Ti sind die maximalen bzw. minimalen Intervallzeitpunkte. - Wählen Sie die Rate mit dem höchsten Wert (Rmax) für jede Kurve aus. Wenn pro Probe mehr als ein Rmax vorhanden ist, wählen Sie den Wert aus, der am frühesten Messzeitpunkt auftritt.
- Berechnen Sie das Verhältnis zwischen Rmax und dem Durchschnittswert des Zeitintervalls, in dem Rmax auftritt: Ratenkoeffizient =
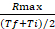
- Berechnen Sie die Faltendifferenz (FD) zwischen dem Ratenkoeffizienten von Salmonellen und E. coli für jede Sonde.
- Betrachten Sie einen Faltendifferenzwert, der höher als 3 ist, als signifikant.
- Betrachten Sie die signifikanten Sonden mit den höchsten Falzdifferenzwerten (FD) als Kandidatensonden und als Grundlage für das Bibliotheksdesign in der nächsten Screening-Runde.
5. Auswahlkriterien der Screening-Runden (Abbildung 1)
- Erste Runde des Screenings
- Führen Sie den Nukleaseaktivitätstest (wie in Abschnitt 4 beschrieben) mit DNA-, RNA- und Polynukleotid-Sonden durch.
- Bewerten Sie die Präferenz für DNA-Chemie oder RNA-Chemie, indem Sie die Anzahl und Leistung (FD-Wert) von DNA- und RNA-Kandidatensonden vergleichen.
- Wählen Sie die Art der Nukleinsäurechemie aus, die die größte Anzahl von Kandidatensonden ausgibt, wie in Abbildung 1dargestellt.
- Zweite Runde des Screenings
- Führen Sie den Nukleaseaktivitätstest (wie in Abschnitt 4 beschrieben) mit vollständig modifizierten Sonden und chimären Sonden durch, die auf dem in der ersten Screening-Runde ausgewählten Nukleinsäuresubstrat basieren.
- Bewerten Sie die Präferenz für Sequenzen, die 2'-Fluoro- und 2'-O-Methyl-Nukleosid-Analoge enthalten, indem Sie die Ergebnisse für 2'-Fluor- und 2'-O-Methylmodifizierte Kandidatensonden vergleichen.
- Wählen Sie den Typ der analogen Nukleosidmodifikation aus, wodurch die größte Anzahl von Kandidatensonden dargestellt wird, wie in Abbildung 1dargestellt.
Ergebnisse
Abbildung 1 zeigt den Arbeitsablauf dieser Methode, die in zwei Screening-Runden unterteilt ist. In der ersten Runde des Screenings verwendeten wir 5 DNA-Sonden (DNA, DNA Poly A, DNA Poly T, DNA Poly C und DNA Poly G) sowie 5 RNA-Sonden (RNA, RNA Poly A, RNA Poly U RNA Poly C und RNA Poly G). Die Rohdaten dieser Screening-Runde sind in der Ergänzenden Tabelle 1 zu finden. In der zweiten Runde wurden chemisch modifizierte Sonden synthetisiert, indem die RNA-Sequenz durch che...
Diskussion
Veränderungen der Nuklease-Aktivität wurden mit einer Vielzahl von Krankheitsphänotypen in Verbindung gebracht, einschließlich verschiedener Krebsarten und bakterieller Infektionen. Diese Veränderungen werden als Erreger einer Erkrankung14vorgeschlagen, während sie in anderen Fällen die Folge eines schädlichen physiologischen Ereignisses20 oder des Pathogens16,26sind. Es überrascht nicht, dass Versuche, Nuk...
Offenlegungen
Die Autoren haben nichts zu verraten.
Danksagungen
Die Autoren würdigen Luiza I. Hernandez (Linköping University) für ihre sorgfältige Überarbeitung des Manuskripts und wertvolle Ratschläge. Diese Arbeit wurde von der Knut and Alice Wallenberg Foundation und dem Strategic Research Area in Materials Science on Advanced Functional Materials der Universität Linköping (Faculty Grant SFO-Mat-LiU No. 2009-00971) unterstützt.
Materialien
| Name | Company | Catalog Number | Comments |
| Black bottom, non-treated 96 well plate | Fisher Scientific | 10000631 | |
| Cytation1 | BioTek | CYT1FAV | |
| Eppendorf tubes | Thermofisher | 11926955 | |
| Escherichia coli | ATCC | 25922 | |
| Microbank cryogenic storage vial containing beads | Pro-Lab Diagnostics | 22-286-155 | |
| Nucleic acid probes | Biomers.net | # | |
| Phosphate Buffer Saline containing MgCl2 and CaCl2 | Gibco™ | 14040117 | |
| Salmonella enterica subs. Enterica | ATCC | 14028 | |
| Tris-EDTA | Fisher Scientific | 10647633 | |
| Tryptone Soya Agar with defibrinated sheep blood | Thermo Fisher Scientific | 10362223 | |
| Tryptic Soy Broth | Sigma Aldrich | 22092 |
Referenzen
- Yang, W. Nucleases: diversity of structure, function and mechanism. Quarterly Reviews of Biophysics. 44 (1), 1-93 (2011).
- Adli, M. The CRISPR tool kit for genome editing and beyond. Nature Communication. 9 (1), 1911 (2018).
- Mason, P. A., Cox, L. S. The role of DNA exonucleases in protecting genome stability and their impact on ageing. Age (Dordr). 34 (6), 1317-1340 (2012).
- Berends, E. T., et al. Nuclease expression by Staphylococcus aureus facilitates escape from neutrophil extracellular traps. Journal of Innate Immunity. 2 (6), 576-586 (2010).
- Kiedrowski, M. R., et al. Staphylococcus aureus Nuc2 is a functional, surface-attached extracellular nuclease. PLoS One. 9 (4), 95574 (2014).
- Morita, C., et al. Cell wall-anchored nuclease of Streptococcus sanguinis contributes to escape from neutrophil extracellular trap-mediated bacteriocidal activity. PLoS One. 9 (8), 103125 (2014).
- Hasegawa, T., et al. Characterization of a virulence-associated and cell-wall-located DNase of Streptococcus pyogenes. Microbiology. 156, 184-190 (2010).
- Moon, A. F., et al. Structural insights into catalytic and substrate binding mechanisms of the strategic EndA nuclease from Streptococcus pneumoniae. Nucleic Acids Research. 39 (7), 2943-2953 (2011).
- Li, L. Y., Luo, X., Wang, X. Endonuclease G is an apoptotic DNase when released from mitochondria. Nature. 412 (6842), 95-99 (2001).
- McDermott-Roe, C., et al. Endonuclease G is a novel determinant of cardiac hypertrophy and mitochondrial function. Nature. 478 (7367), 114-118 (2011).
- Wang, Y. T., et al. A link between adipogenesis and innate immunity: RNase-L promotes 3T3-L1 adipogenesis by destabilizing Pref-1 mRNA. Cell Death & Disease. 7 (11), 2458 (2016).
- Li, C. L., Yang, W. Z., Shi, Z., Yuan, H. S. Tudor staphylococcal nuclease is a structure-specific ribonuclease that degrades RNA at unstructured regions during microRNA decay. RNA. 24 (5), 739-748 (2018).
- Wan, L., et al. MTDH-SND1 interaction is crucial for expansion and activity of tumor-initiating cells in diverse oncogene- and carcinogen-induced mammary tumors. Cancer Cell. 26 (1), 92-105 (2014).
- Keyel, P. A. Dnases in health and disease. Developmental Biology. 429 (1), 1-11 (2017).
- Hernandez, F. J., et al. Noninvasive imaging of Staphylococcus aureus infections with a nuclease-activated probe. Nature Medicine. 20 (3), 301-306 (2014).
- Flenker, K. S., et al. Rapid Detection of Urinary Tract Infections via Bacterial Nuclease Activity. Molecular Therapy. 25 (6), 1353-1362 (2017).
- Kannan, N., Eaves, C. J. Tipping the balance: MTDH-SND1 curbs oncogene-induced apoptosis and promotes tumorigenesis. Cell Stem Cell. 15 (2), 118-120 (2014).
- Kemmer, T. P., Malfertheiner, P., Buchler, M., Kemmer, M. L., Ditschuneit, H. Serum ribonuclease activity in the diagnosis of pancreatic disease. International Journal of Pancreatology. 8 (1), 23-33 (1991).
- Fernandez-Salas, E., Peracaula, R., Frazier, M. L., de Llorens, R. Ribonucleases expressed by human pancreatic adenocarcinoma cell lines. European Journal of Biochemistry. 267 (5), 1484-1494 (2000).
- Fujibayashi, K., et al. Serum deoxyribonuclease I activity can be a useful diagnostic marker for the early diagnosis of unstable angina pectoris or non-ST-segment elevation myocardial infarction. Journal of Cardiology. 59 (3), 258-265 (2012).
- Kawai, Y., et al. Diagnostic use of serum deoxyribonuclease I activity as a novel early-phase marker in acute myocardial infarction. Circulation. 109 (20), 2398-2400 (2004).
- Hernandez, L. I., Ozalp, V. C., Hernandez, F. J. Nuclease activity as a specific biomarker for breast cancer. Chemical Communication (Cambridge). 52 (83), 12346-12349 (2016).
- Machado, I., et al. Rapid and specific detection of Salmonella infections using chemically modified nucleic acid probes. Analytica Chimica Acta. 1054, 157-166 (2019).
- Sanders, E. R. Aseptic laboratory techniques: plating methods. Journal of Visualized Experiment. (63), e3064 (2012).
- Worthington Biochemical Corporation. . Manual of Clinical Enzyme Measurements. , (1972).
- Burghardt, E. L., et al. Rapid, Culture-Free Detection of Staphylococcus aureus Bacteremia. PLoS One. 11 (6), 0157234 (2016).
- Bibette, J. Gaining confidence in high-throughput screening. Proceedings of the National Academy of Science U. S. A. 109 (3), 649-650 (2012).
- Hernandez, L. I., Machado, I., Schafer, T., Hernandez, F. J. Aptamers overview: selection, features and applications. Current Topics in Medicinal Chemistry. 15 (12), 1066-1081 (2015).
- Pande, J., Szewczyk, M. M., Grover, A. K. Phage display: concept, innovations, applications and future. Biotechnology Advances. 28 (6), 849-858 (2010).
- Skerra, A. Alternative binding proteins: anticalins - harnessing the structural plasticity of the lipocalin ligand pocket to engineer novel binding activities. FEBS Journal. 275 (11), 2677-2683 (2008).
- Ku, T. H., et al. Nucleic Acid Aptamers: An Emerging Tool for Biotechnology and Biomedical Sensing. Sensors (Basel). 15 (7), 16281-16313 (2015).
- Jayasena, S. D. Aptamers: an emerging class of molecules that rival antibodies in diagnostics. Clinical Chemistry. 45 (9), 1628-1650 (1999).
- Gray, B. P., Brown, K. C. Combinatorial peptide libraries: mining for cell-binding peptides. Chemical Reviews. 114 (2), 1020-1081 (2014).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten