このコンテンツを視聴するには、JoVE 購読が必要です。 サインイン又は無料トライアルを申し込む。
Method Article
レジオネラ肺球菌ドット/Icm分泌系の空間的特徴を解決するためのライブ細胞イメージングとクライオ電子断層撮影の適用
要約
細菌細胞のイメージングは、大きな高分子機械の機能を決定する静的および動的プロセスの定義に焦点を当てた新しいシステム生物学アプローチです。ここでは、定量的ライブ細胞イメージングとクライオ電子断層撮影の統合により、レジオネラ菌型肺炎球体系IV型分泌系のアーキテクチャと機能を研究しています。
要約
レジオネラ・ニューモフィルのドット/Icm分泌系は、細菌極で局所化し、タンパク質およびDNA基質を標的細胞に送達する複雑なIV分泌系(T4SS)ナノマシンであり、一般的に直接細胞間接触を必要とするプロセスである。我々は最近、凍結電子断層撮影(cryo-ET)によってドット/Icm装置の構造を解明し、細胞質複合体に接続する細胞エンベロープスパンチャネルを形成することを示した。生きた細胞とcryo-ETにおける蛍光顕微鏡法を用いて、試料のネイティブ構造を保存する2つの相補的アプローチを適用することで、タンパク質の可視化と、他のDot/Icmサブユニットに対する各機械成分の生産時期と同化をその時点で可能にします。極位置の要件を調べ、T4SS機械生物発生に関連する動的特徴を特徴付けるために、染色体上のネイティブ位置でスーパーフォルダグリーン蛍光タンパク質をドット/Icm ATPase遺伝子にコードする遺伝子を融合しました。以下の方法は、生体細胞の定量的蛍光顕微鏡とcryo-ETを統合し、これらのタンパク質の細胞内の極地の局在、ダイナミクス、および構造を定量化します。レジオネラ・ニューモフィラT4SSを研究するためにこれらのアプローチを適用することは、ドット/Icmシステムの機能を特徴付けるために有用であり、T4SSまたは他のタイプの細菌分泌複合体を利用する多種多様な細菌病原体を研究するために適応することができる。
概要
レジオネラ菌(L.肺炎球菌)は、レジオネラ症の病因である淡水貯留層に生息し、水生自由水泳原生動物の中で細菌が感染して複製することによって増殖する。L. 気球菌は、飲料水源からのエアロゾル化細菌の吸入が発生すると、ヒトで病気の発生を引き起こす。感染した細胞では、宿主経路の転覆により、L.肺炎球菌は、それが存在する液胞のエンドサイト成熟を遅らせ、細菌複製をサポートする細胞コンパートメントの生生生成を促進することができる。このプロセスは、Dot/Icmとして知られている特殊な細菌タイプIVB分泌システム(T4BSS)と、感染中に宿主サイトゾルに転化される300以上の「エフェクター」タンパク質のレパートリーによって駆動され、細胞機能1、2、3、4、5の操作を容易にする。機能的なDot/Icm装置を欠いている変異体は、宿主サイトゾルにエフェクターを送達できず、細胞内複製に欠陥があり、疾患6,7の動物モデルにおいてアビリエントである。
多くの細菌種は、感染プロセスに必要な非常に複雑で動的なマルチコンポーネントマシンを開発してきました。Dot/Icmシステムのような他のT4BSSも、コクシエラ・ブルネティやリケッチエラ・グリリなどの細菌病原体の細胞内複製に不可欠である。T4BSSは、DNAの伝達を媒介し、エフェクタータンパク質の限られたレパートリーを提供できる原型型IVA系と進化的に関連していますが、Dot/Icmシステムは機械成分のほぼ2倍を有し、多種多様なエフェクターを提供します。おそらく、コンポーネント数のこの拡大は、Dot / Icm装置が新しいエフェクタを容易に8,9に収容し、統合することを可能にしました。
我々は最近、クライオ電子断層撮影(cryo-ET)を使用して、その場でドット/Icm装置の構造を解き、細胞質複合体に接続する細胞エンベロープスパンチャネルを形成することを示した。さらなる分析は、細胞体ATPase DotOとの相互作用を通じて、細胞内のTPCとの関連がL.ニューモフィラ細胞極のドット/Icm系と関連することを明らかにした。DotBは、ほとんどの細菌細胞に細胞間運動を示すことを発見し、このATPaseが動的な細胞構造集団に存在するが、極性ドット/Icm複合体にも関連していることを示している。さらに、DotOは、内膜複合体に関連するDotOダイマーの六方体集合体を形成し、この細胞質複合体の基部にDotBヘキサマーが結合する。DotB-DotOエネルギー複合体のアセンブリは、T4SS(図1)10を介して基板の転座を指示する細胞質チャネルを作成する。
これらの最近の進歩にもかかわらず、Dot/Icmシステムがどのように機能し、各タンパク質がどのように組み立てて活性装置8を形成するかについてはほとんど知られていない。Dot/Icm T4SSの制御回路を明らかにすることは、宿主と病原体の相互作用の分子メカニズムを理解する上で重要です。そこで、スーパーフォルダGFP(sfGFP)でタグ付けされた必須L.肺炎球菌ドット/Icmシステムコンポーネントを検出し、特徴付けるために、生細胞顕微鏡とcryo-ETを使用する方法について説明します。定量的蛍光顕微鏡を用いて、DotBの極地局在化は野生型のバックグラウンドで、またはタイプIV系が削除されたときに定義される。タイムラプス顕微鏡は、ドット/Icm細胞体ATPases間の局在およびダイナミクスの違いを定量化するために使用されます。
ライブイメージングやクライオETなどの2つの相補的アプローチを組み合わせることで、他のインビトロシステムと比較して利点があります。両方の方法は無傷の細胞で行われ、T4BSSの自然環境を維持し、サンプル調製中のネイティブ構造の破壊を最小限に抑えます。タンパク質の過剰発現は分泌装置の染色法を損なう可能性があるため、sfGFP融合は、各融合が単一コピーでコードされ、発現が内因性プロモーターによって駆動されるように、レジオネラ染色体へのアレジオン交換を介して返される。染色体でコード化された融合の可視化により、定義された時点で発現しているタンパク質の正確なレベルを定量化することができます。Cryo-ETには、分泌システムの構造を決定するための多くの利点があります。最も顕著な利点は、cryo-ETサンプルが細菌細胞アーキテクチャの文脈で天然複合体を保存する凍結された無傷の細胞で構成されているということです。その結果、cryo-ETは、膜複合体を抽出し、コア装置から末梢タンパク質を取り除くか、または全体的な構造を変更することができる生化学的精製アプローチよりも好ましい場合があります。さらに、関心のあるタンパク質にsfGFPなどのかさばるタンパク質をタグ付けすると、cryo-ETによって検出可能な質量が追加され、Dot/Icm装置の異なるサブコンプレックスをcryo-ETによって得られた構造にマッピングするのに役立ちます。
このアプローチは、細菌細胞膜に組み立てる多分子複合体に関する構造情報を明らかにするための強力なツールです。これらの手法を用いて解明された構造の解釈は、T4BSS コンポーネントの機能、機能に必要なコンポーネントの数、コンポーネントの相互作用、およびこれらの機能を理解するのに役立ちます。サブアセンブリが実行されます。
プロトコル
注:L.肺炎球菌の成長、操作、およびイメージングを含むすべての手順は、局所的なガイドラインに従って生物学的安全レベル2の実験室で行われるべきです。
1. アレル交換と二重選択戦略を用いたL.気球菌染色体へのsfGFPの挿入(図2、図3)
- 遺伝子置換ベクターpSR47S11にクローンを作成し、次の配列:目的の部位の1,000bp上流、次いでsfGFP配列、次いで1,000bp下流の対象部位(図2)。sfGFP配列は、4〜8個のアミノ酸を含むリンカーで終わるN末端またはC末端にフレーム内に配置する必要があります。得られたベクターを大腸菌DH5αλpirに変換します。その後、L.ストリークL.肺炎球菌(レシピエント)は、100μg/mLストレプトマイシンを含む100μg/mLストレプトマイシンを含む寒天12に対する単一コロニーの37°C(図3)に対して行う。
- CYE-寒天連鎖菌のストリークL.肺炎球菌は、37°C(重パッチ)10で2日間成長する。25μg/mLクロラムフェニコールを含むLB寒天上にpRK600ヘルパープラスミド(ヘルパー)13で変換されたストリーク大腸菌DH5α。50 μg/mL カナマイシンを含む LB 寒天上の大腸菌DH5αλpir(ドナー)をストリークする。
- トライペアレンタル交配を行う:選択せずにCYE寒天プレートに3つの株のパッチを重ね、37°Cで4〜8時間インキュベートすることによって、ヘルパーのコロニー、ドナーのコロニー、およびレシピエントをインキュベートする。否定的なコントロールとして、ヘルパー+レシピエント株ミックスとドナー+レシピエント株ミックスを同じ期間にインキュベートします。
- 交配反応をddH2O.プレート20 μLの500 μLおよび100 μg/mLストレプトマイシンおよび10 μg/mLカナマイシンを含むCYE寒天反応の500 μLで再中断し、37°Cで5日間成長させる。得られたクローンの4個を100μg/mLストレプトマイシンを含むCYE寒天にストリークし、37°Cで5日間増殖する。
- 5%スクロースと100 μg/mLストレプトマイシンを含むCYE寒天上の16クローンをストリークし、37°Cで5日間増殖します。次いで、100μg/mLストレプトマイシンを含むCYE寒天上のこれらのクローンの32を、100μg/mLストレプトマイシンおよび10μg/mLカナマイシンを含むCYE寒天上で、37°Cで5日間増殖する。
2. L.肺炎球菌染色体にsfGFPを統合したクローンの分離
- CYE-寒天連鎖マイシンプレート上のカナマイシンに感受性であったストリーククローンとコロニーPCRによる染色体へのsfGFPの挿入を確認した。sfGFP遺伝子と目的の染色体領域と相補的なプライマーを使用して、挿入接合部を増幅する。
- 10 μM プライマー溶液の各0.5 μLとコロニー1個を最終体積12.5 μLに混合し、95°Cで10分間変性します。氷の上で10分間冷却し、12.5 μLの2倍 PCR マスターミックス溶液を加え、PCR 分析を実行します。
- CYE-寒天-ストレプトマイシンプレート上の分離されたコロニーの重いパッチを37°Cで2日間成長させます。抗GFP抗体による免疫ブロット法によるsfGFP融合の発現レベルと安定性を調べる。
3. 蛍光タグ付きドット/Icm成分を用いたL.肺炎球菌の生細胞イメージング
- アガロースパッドの調製
- 水中に1%低融解アガロース溶液の約30mLを作ります。ガラスフラスコにマイクロ波を約90s、時折渦巻き、アガロースが完全に溶解するまで。
- 22 x 22 x 0.15 mm3ガラス スライドを 25 x 75 x 1.1 mm3ガラス スライドの端に 2 枚配置し、1 枚をもう一方のスライドの上に配置します。もう一方の端にさらに22 x 22 x 0.15 mm3ガラススライドを積み重ねなさい。
- 2つの上のガラススライドの間の中央スライドに溶融アガロースの約1mLのピペット、溶融アガロースの上に別の25 x 75 x 1.1 mm3スライドを置きます。気泡の形成を避けるようにしてください。スライドを4°Cで15分間冷まします。
- メスやカミソリの刃を使って、パッドを小さな正方形にそっと切り、〜5 x 5 mm2。両面接着剤17 x 28 x 0.25 mm3フレームを25 x 75 x 1.1 mm3ガラススライドに固定し、スライド上にパッドを複数配置します。
- 画像取得
注:次の手順は、スライドブック6.0の制御下にあり、ソリッドステートイルミエータ、CCDモノクロカメラ、および100x対物レンズ(1.4個の開口)を備えた顕微鏡について説明します。必要に応じて、プロトコル設定に従ってカスタマイズできる適切なハードウェア構成およびソフトウェア構成を備えた代替の顕微鏡装置を使用します。- L.肺炎球菌の重いパッチをddH2Oの1 mL、渦およびピペット2~3 μLの希釈液をパッドに溶解します。50 x 24 x 0.15 mm3のカバースリップを接着剤フレームの上にそっと置きます。
- キャプチャウィンドウで、NDを180に調整します。ビニングを 2×2 に調整し、488 nm チャネルを使用してサンプルを 500 ~ 1,000 ミリ秒の間で公開します。
4. 極地の局在化とドット/Icm成分のダイナミクスの定量化
注:次の手順は、2 x 2 ビニングで取得したピクセルあたり 0.129 μm のイメージ用に設計されています。
- sfGFP融合タンパク質の極性の定量 (図 5)
- 細菌がはっきりと見えるように画像のコントラストを調整します。領域ツールを使用して、極から始めて細胞質に伸びる0.25 x 1.3 μm 2の長方形を配置します。四角形は、細菌の境界内に正確に残る必要があります。
- 少なくとも 200 個の細菌をマークし、[マスクする領域] ボタンを使用して、対象地域にマスクを作成します。[マスク統計とマスクスコープ] で、[オブジェクト] を選択します。次に、[特徴と強度] で、[平均強度と分散] をマークします。
- データをエクスポートし、各細菌の極性スコアを平均強度に対する分散の比率として計算します。
- ハイスループットアプリケーションの場合は、位相と488 nmチャネルで画像を取得するために、位相目標と適切なコンデンサ設定を使用します。細菌が完全に分離されている視野を選択してください。
- 画像の位相チャネルのコントラストを、細菌がはっきりと見えるレベルに調整します。デュアルチャンネルイメージを開き、セグメントマスク作成ウィンドウを起動し、チャンネルをフェーズに変更します。
- 適切なしきい値を調整し、[オブジェクトの定義]ボタンを使用して小さなオブジェクトを削除します。[マスクをリファイン]で、[エッジ オブジェクトを除去]を選択し、後で隣接するバクテリアのマスクを分離します。
- ステップ 4.1.2 ~ 4.1.3 で説明したように、各セルの 488 チャネルの信号の極性スコアを計算します。
- sfGFP融合タンパク質のダイナミクスの定量化 (図 6)
注: セクション 3 の指示に従って、画像取得のサンプルを準備します。ダイナミクスは時間の経過に伴う強度の変化として定義され、次の手順は短いイメージング期間(すなわち、数分)のために設計されています。より長いイメージング期間が望ましい場合は、適切なサプリメントをパッドに加えます。- [イメージキャプチャ]ウィンドウで、[Timelapse]をマークし、[間隔]ボックスに間隔の時間を入力し、[時間ポイントの数]ボックスに2を入力します。目的の蛍光タンパク質を発現するL.肺炎球菌の2つの連続した画像を取得します。
- セルがはっきりと見えるまで、画像のコントラストを調整します。図 6Aと以下の説明に従って、3 つの異なるマスクを配置します。領域ツールを使用して、少なくとも400個のセルの中央に0.25 x 0.25 μmの正方形を配置します。
- [マスクする領域] ボタンを使用して、対象となる四角形のマスク (マスク 1) を作成します。新しい空のマスク(マスク 2)を作成し、ピクセルツールまたはポリゴンツールを使用して、少なくとも25個のランダムセルのセル領域全体をマークし、蛍光漂白の計算に使用します。新しい空のマスク(マスク 3)を作成し、大きなブラシツールを使用してセル間の領域をマークします。
- [マスク統計とマスク範囲] で、[マスク1] の [オブジェクト]を選択し、マスク 2 を選択します。次に、[フィーチャと強度] で [平均強度] を選択し、2 つのマスクのデータをエクスポートします。マスク 3 の場合、マスク全体の平均強度をエクスポートします。
- マスク 1 の各オブジェクトの蛍光強度の変化を次の式を使用して計算します。

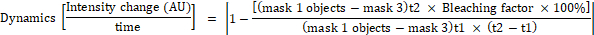
ここでマスク1は、セル中心に0.25μm×0.25μmの正方形を、マスク2はセル全体を覆い、マスク3はセル間の背景、t1は最初のタイムポイントの平均強度、t2は2番目の時間ポイントの平均強度である。
クライオ-ETによるsfGFP質量密度の検出
- サンプルの準備、データ収集、および再構築
- CYE-寒天ストレプトマイシンプレート上の蛍光タグ付きDot/Icmタンパク質を37°Cで48時間発現するL.肺炎球菌の重いパッチを成長させます。ddH2Oの細胞をOD600〜0.7に再懸濁する。5 μLのコロイド状金粒子(BSAトレーサー、10 nm)を細胞懸濁液の20 μLに加えます。
- ピペット5μLの細胞混合物を、新たにグロー排出された穴あきカーボングリッド(Cu 200メッシュ上のR2/1)に、前述のように重力駆動プランジャー装置を用いて濾紙でブロットを1分間放置し、液体エタンで凍結させる。
- フィールドエミッションガン、エネルギーフィルター、ボルタ相プレート、直接検出装置を搭載した300kV透過型電子顕微鏡で凍結水和標本を画像化します。26,000x と 42,000x の倍率で単軸傾斜系列を収集し、その結果、それぞれ 5.4 Å/ピクセルまたは 3.4 Å/ピクセルの標本レベルでピクセル サイズが得られます。
- 断層図パッケージSerialEMを使用して、-0 μmの焦点がずれた画像スタックを収集し、3°ステップインクリメントと累積用量で-60°~+60°の間の傾斜角の範囲で、~60 e-/Å2,16を収集します。MotionCor217を使用して、各スタック内の線量分画ムービーイメージを整列させます。TOMOAUTO14を使用してドリフト補正スタックを組み立てます。
- ドリフト補正スタックを IMOD マーカー依存の位置合わせ18で整列します。セグメンテーションおよびダイレクト画像解析用のSIRT法19とWBP法20を用いて、トモグラムを再構築する。
- sfGFP融合サンプルのサブトモグラム解析
- サブモグラム解析14、21、22の場合は、断層図パッケージ I3 (0.9.9.3) を使用します。
注: 線形は反復的に進行し、各反復は参照と分類マスクが生成され、サブトモグラムが整列および分類され、クラス平均が互いに整列される 3 つの部分で構成されます。 - 最初の位置合わせには、4 x 4 x 4 ビン分割サブトモグラムを使用します。クラス平均に属する粒子を結合し、sfGFPに対応する電子密度を示します。sfGFP 融合で粒子を選別した後、2 x 2 x 2 ビン分割サブモグラムを使用して、対象領域(Dot/Icm 細胞質 ATPase 複合体など)の焦点を合わせ、高解像度の構造を得る。
- サブモグラム解析14、21、22の場合は、断層図パッケージ I3 (0.9.9.3) を使用します。
結果
2段階で二重選択との相同組換えがsfGFPの定義された挿入を構築するために使用された。第1のステップでは、大腸菌ヘルパー株MT616からのpRK600共役プラスミド(IncPプラスミド)を2つの相同領域に横たわるsfGFP遺伝子を含む自殺ベクターpSR47Sを用いてドナー大腸菌株に動員したトライペアレンタル交配を行った。次に、pRK600からの共役系は、pSR47S誘導体をL.肺炎球菌
ディスカッション
細菌分泌系の機能を解明することは、宿主と病原体の相互作用を完全に理解する鍵です。分泌系は、エフェクタータンパク質を宿主細胞に注入できる複雑な機械であり、場合によっては細菌複製をサポートする細胞内ニッチの確立を促進する。上記の方法は、呼吸器病原体レジオネラ肺炎球菌のDot/Icm分泌システムを研究するための重要な新しいツールを提供し、病原性に不可欠なエフ...
開示事項
著者らは開示するものは何もない。
謝辞
D.C.とC.R.R.はNIH(R37AI041699およびR21AI130671)によってサポートされました。D.P.、B.H.、J.Lは国立衛生研究所(R01AI087946およびR01GM107629)によって支援されました。
資料
| Name | Company | Catalog Number | Comments |
| 10 nm colloidal gold particles | Aurion | 25486 | |
| 100x Plan Apo objective (1.4 NA) | Nikon | ||
| ACES | Sigma-Aldrich | A9758 | |
| Activated charcoal | Sigma-Aldrich | C5510 | |
| Agaroze GPG/LMP, low melt | American bioanalytical | AB00981 | |
| Bacto dehydrated agar | BD | 214010 | |
| CoolSNAP EZ 20 MHz digital monochrome camera | Photometrics | ||
| Gene Frame, 1.7x2.8 cm, 125 µL | Fisher Scientific | AB-0578 | |
| Holey Carbon grid R 2/1 Cu 200 mesh | Quantifoil | Q225-CR1 | |
| Iron(III) nitrate nonahydrate | Sigma-Aldrich | 216828 | |
| K2 Summit camera for cryo-EM | GATAN | ||
| L-Cysteine | Sigma-Aldrich | C7352 | |
| Microscope cover slides 22x22 mm | Fisher Scientific | 12-542B | |
| Microscope cover slides 24x50 mm | Fisher Scientific | 12-545K | |
| Microscope slides 25x75x1 mm | Globe Scientific | 1380 | |
| SlideBook 6.0 | Intelligent Imaging Innovations | ||
| Spectra X light engine | Lumencor | ||
| Taq 2X Master Mix | New England BioLabs | M0270 | |
| Titan Krios | Thermo Fisher Scientific | ||
| Yeast Extract | BD | 212750 |
参考文献
- Franco, I. S., Shuman, H. A., Charpentier, X. The perplexing functions and surprising origins of Legionella pneumophila type IV secretion effectors. Cellular Microbiology. 11, 1435-1443 (2009).
- Burstein, D., et al. Genome-scale identification of Legionella pneumophila effectors using a machine learning approach. PLOS Pathogens. 5, 1000508 (2009).
- Ninio, S., Roy, C. R. Effector proteins translocated by Legionella pneumophila: strength in numbers. Trends in Microbiology. 15, 372-380 (2007).
- Vogel, J. P., Andrews, H. L., Wong, S. K., Isberg, R. R. Conjugative transfer by the virulence system of Legionella pneumophila. Science. 279, 873-876 (1998).
- Isberg, R. R., O'Connor, T. J., Heidtman, M. The Legionella pneumophila replication vacuole: making a cosy niche inside host cells. Nature Reviews Microbiology. 7, 13-24 (2009).
- Roy, C. R., Berger, K. H., Isberg, R. R. Legionella pneumophila DotA protein is required for early phagosome trafficking decisions that occur within min of bacterial uptake. Molecular Microbiology. 28, 663-674 (1998).
- Archer, K. A., Roy, C. R. MyD88-Dependent Responses Involving Toll-Like Receptor 2 Are Important for Protection and Clearance of Legionella pneumophila in a Mouse Model of Legionnaires' Disease. Infection and Immunity. 74, 3325-3333 (2006).
- Nagai, H., Kubori, T. Type IVB Secretion Systems of Legionella and Other Gram-Negative Bacteria. Frontiers in Microbiology. 2, 136 (2011).
- Kubori, T., Nagai, H. The Type IVB secretion system: an enigmatic chimera. Current Opinion in Microbiology. 29, 22-29 (2016).
- Chetrit, D., Hu, B., Christie, P. J., Roy, C. R., Liu, J. A unique cytoplasmic ATPase complex defines the Legionella pneumophila type IV secretion channel. Nature Microbiology. 3, 678-686 (2018).
- Merriam, J. J., Mathur, R., Maxfield-Boumil, R., Isberg, R. R. Analysis of the Legionella pneumophila fliI gene: intracellular growth of a defined mutant defective for flagellum biosynthesis. Infection and Immunity. 65, 2497-2501 (1997).
- Feeley, J. C., et al. Charcoal-yeast extract agar: primary isolation medium for Legionella pneumophila. Journal of Clinical Microbiology. 10, 437-441 (1979).
- Andrews, H. L., Vogel, J. P., Isberg, R. R. Identification of linked Legionella pneumophila genes essential for intracellular growth and evasion of the endocytic pathway. Infection and Immunity. 66, 950-958 (1998).
- Morado, D. R., Hu, B., Liu, J. Using Tomoauto: A Protocol for High-throughput Automated Cryo-electron Tomography. Journal of Visualized Experiments. (107), e53608 (2016).
- Hu, B., Lara-Tejero, M., Kong, Q., Galan, J. E., Liu, J. In Situ Molecular Architecture of the Salmonella Type III Secretion Machine. Cell. 168, 1065-1074 (2017).
- Mastronarde, D. N. Automated electron microscope tomography using robust prediction of specimen movements. Journal of Structural Biology. 152, 36-51 (2005).
- Zheng, S. Q., et al. MotionCor2: anisotropic correction of beam-induced motion for improved cryo-electron microscopy. Nature Methods. 14, 331-332 (2017).
- Kremer, J. R., Mastronarde, D. N., McIntosh, J. R. Computer visualization of three-dimensional image data using IMOD. Journal of Structural Biology. 116, 71-76 (1996).
- Gilbert, P. Iterative methods for the three-dimensional reconstruction of an object from projections. Journal of Theoretical Biology. 36, 105-117 (1972).
- Radermacher, M. Weighted Back-projection Methods. Electron Tomography. , 245-273 (2007).
- Winkler, H., et al. Tomographic subvolume alignment and subvolume classification applied to myosin V and SIV envelope spikes. Journal of Structural Biology. 165, 64-77 (2009).
- Winkler, H., Taylor, K. A. Accurate marker-free alignment with simultaneous geometry determination and reconstruction of tilt series in electron tomography. Ultramicroscopy. 106, 240-254 (2006).
- Prevost, M. S., Waksman, G. X-ray crystal structures of the type IVb secretion system DotB ATPases. Protein Science. 27, 1464-1475 (2018).
- Miklos, G. L., Rubin, G. M. The role of the genome project in determining gene function: insights from model organisms. Cell. 86, 521-529 (1996).
- Reyrat, J. M., Pelicic, V., Gicquel, B., Rappuoli, R. Counterselectable markers: untapped tools for bacterial genetics and pathogenesis. Infection and Immunity. 66, 4011-4017 (1998).
- Yu, J. Single-Molecule Studies in Live Cells. Annual Review of Physical Chemistry. 67, 565-585 (2016).
- Stewart, P. L. Cryo-electron microscopy and cryo-electron tomography of nanoparticles. Wiley Interdisciplinary Reviews: Nanomedicine and Nanobiotechnology. 9, (2017).
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved