Zum Anzeigen dieser Inhalte ist ein JoVE-Abonnement erforderlich. Melden Sie sich an oder starten Sie Ihre kostenlose Testversion.
Method Article
Anwendung von Live Cell Imaging und Cryo-Electron Tomography zur Auflösung der raumzeitlichen Merkmale des Legionella pneumophila Dot/Icm Sekretionssystems
In diesem Artikel
Zusammenfassung
Die Bildgebung von Bakterienzellen ist ein aufkommendem systembiologischen Ansatz, der sich auf die Definition statischer und dynamischer Prozesse konzentriert, die die Funktion großer makromolekularer Maschinen diktieren. Hier wird die Integration von quantitativer Live-Zell-Bildgebung und Kryo-Elektronentomographie verwendet, um Legionella pneumophila Typ IV Sekretionssystemarchitektur und -funktionen zu untersuchen.
Zusammenfassung
Das Dot/Icm-Sekretionssystem von Legionella pneumophila ist eine komplexe Typ-IV-Sekretionssystem (T4SS)-Nanomaschine, die am Bakteriellen Pol lokalisiert und die Lieferung von Protein- und DNA-Substraten an Zielzellen vermittelt, ein Prozess, der in der Regel einen direkten Zell-zu-Zell-Kontakt erfordert. Wir haben vor kurzem die Struktur des Dot/Icm-Apparats mittels Kryo-Elektronentomographie (Cryo-ET) gelöst und gezeigt, dass es einen Zellhüllen-Spannkanal bildet, der sich mit einem zytoplasmatischen Komplex verbindet. Die Anwendung von zwei komplementären Ansätzen, die die native Struktur der Probe erhalten, die fluoreszierende Mikroskopie in lebenden Zellen und Kryo-ET, ermöglicht die in situ-Visualisierung von Proteinen und die Assimilation der Stoiterminatrie und das Timing der Produktion jeder Maschinenkomponente im Vergleich zu anderen Dot/Icm-Untereinheiten. Um die Anforderungen an die polare Positionierung zu untersuchen und dynamische Merkmale im Zusammenhang mit der T4SS-Maschinenbiogenese zu charakterisieren, haben wir ein Gen kodiert, das superfolder green fluorescent protein zu Dot/Icm ATPase-Genen an ihren nativen Positionen auf dem Chromosom kodiert. Die folgende Methode integriert die quantitative Fluoreszenzmikroskopie lebender Zellen und Kryo-ET, um polare Lokalisation, Dynamik und Struktur dieser Proteine in intakten Bakterienzellen zu quantifizieren. Die Anwendung dieser Ansätze für die Untersuchung der Legionella pneumophila T4SS ist nützlich für die Charakterisierung der Funktion des Dot/Icm-Systems und kann angepasst werden, um eine Vielzahl von bakteriellen Krankheitserregern zu untersuchen, die die T4SS oder andere Arten von bakteriellen Sekretionskomplexen nutzen.
Einleitung
Legionella pneumophila (L. pneumophila), das ätiologische Mittel der Legionärskrankheit, bewohnt Süßwasserreservoirs, in denen sich die Bakterien vermehren, indem sie sich in aquatischen, freischwimmenden Protozoen animieren. L. pneumophila verursacht Krankheitsausbrüche beim Menschen, wenn das Einatmen von aerosolisierten Bakterien aus Trinkwasserquellen auftritt. In infizierten Zellen ermöglicht die Subversion von Wirtswegen L. pneumophila, die endozytische Reifung des Vakuums, in dem es sich befindet, zu verzögern und die Biogenese eines Zellfachs zu fördern, das die bakterielle Replikation unterstützt. Dieser Prozess wird durch ein spezialisiertes bakterielles Sekretionssystem des Typs IVB (T4BSS) angetrieben, das als Dot/Icm bekannt ist, und seinem Repertoire von über 300 "Effektor"-Proteinen, die während der Infektion in den Wirtcytosol transloziert werden, um die Manipulation der Zellfunktionen1,2,3,4,5zu erleichtern. Mutanten ohne funktionelles Dot/Icm-Gerät liefern keine Effektoren in das Wirtszytosol, sind defekt für die intrazelluläre Replikation und sind in Tiermodellen der Krankheit6,7aviär.
Viele Bakterienarten haben extrem komplexe und dynamische Mehrkomponenten-Maschinen entwickelt, die für Infektionsprozesse benötigt werden. Andere T4BSS wie das Dot/Icm-System sind auch wichtig für die intrazelluläre Replikation von bakteriellen Krankheitserregern wie Coxiella burnetii und Rickettsiella grylli. Obwohl T4BSS evolutionär mit prototypischen IVA-Systemen des Typs verwandt sind, die DNA-Übertragung vermitteln und ein begrenztes Repertoire an Effektorproteinen liefern können, verfügt das Dot/Icm-System über fast doppelt so viele Maschinenkomponenten und liefert eine Vielzahl von Effektoren. Vermutlich hat diese Erweiterung der Anzahl der Komponenten es dem Dot/Icm-Gerät ermöglicht, neue Effektoren einfach unterzubringen und zu integrieren8,9.
Kürzlich haben wir die Kryo-Elektronen-Tomographie (Cryo-ET) verwendet, um die Struktur des Dot/Icm-Geräts in situ zu lösen und zeigten, dass es einen Zellhüllen-Spannkanal bildet, der sich mit einem zytoplasmatischen Komplex verbindet. Weitere Analysen ergaben, dass der zytosolische ATPase DotB durch Wechselwirkungen mit dem zytosolischen ATPase DotO mit dem Dot/Icm-System am L. pneumophila-Zellpol assoziiert. Wir haben entdeckt, dass DotB eine zytosolische Bewegung in den meisten Bakterienzellen zeigt, was darauf hindeutet, dass diese ATPase in einer dynamischen zytosolischen Population vorhanden ist, aber auch mit den polaren Dot/Icm-Komplexen assoziiert wird. Darüber hinaus bildet DotO eine hexamerische Baugruppe von DotO-Dimeren, die mit dem inneren Membrankomplex verbunden sind, und ein DotB-Hexamer verbindet sich mit der Basis dieses zytoplasmatischen Komplexes. Die Montage des DotB-DotO-Energiekomplexes erzeugt einen zytoplasmatischen Kanal, der die Translokation von Substraten durch das T4SS leitet (Abbildung 1)10.
Trotz dieser jüngsten Fortschritte ist wenig darüber bekannt, wie das Dot/Icm-System funktioniert und wie sich jedes Protein zu einem aktiven Apparat zusammenfügt8. Die Aufdeckung der regulatorischen Schaltungen des Dot/Icm T4SS ist von grundlegender Bedeutung für das Verständnis der molekularen Mechanismen von Wirts-Pathogen-Wechselwirkungen. Daher besprechen wir, wie man mit Live-Zellmikroskopie und Kryo-ET wesentliche L. pneumophila Dot/Icm-Systemkomponenten erkennt und charakterisiert, die mit Super-Ordner GFP (sfGFP) markiert sind. Mittels quantitativer Fluoreszenzmikroskopie wird die polare Lokalisation von DotB in einem Wildtyp-Hintergrund oder beim Löschen des Typ-IV-Systems definiert. Die Zeitraffermikroskopie wird verwendet, um Unterschiede in lokalisierung und Dynamik zwischen den zytosolischen AtPases Dot/Icm zu quantifizieren.
Die kombinierte Anwendung von zwei sich ergänzenden Ansätzen wie Live Imaging und Kryo-ET bietet einen Vorteil gegenüber anderen In-vitro-Systemen. Beide Methoden werden in intakten Zellen durchgeführt und erhalten die natürliche Umgebung des T4BSS, wodurch Störungen der nativen Struktur während der Probenvorbereitung minimiert werden. Da die Überexpression von Proteinen die Stoiiometrie des Sekretionsapparates beeinträchtigen kann, werden sfGFP-Fusionen über allelischen Austausch an das Legionella-Chromosom zurückgegeben, so dass jede Fusion in einer Kopie kodiert ist und die Expression vom endogenen Promotor angetrieben wird. Die Visualisierung chromosomal-kodierter Fusionen ermöglicht die Quantifizierung des genauen Proteinniveaus, das zu einem definierten Zeitpunkt exprimiert wird. Cryo-ET hat auch viele Vorteile bei der Bestimmung der Struktur von Sekretionssystemen. Der bemerkenswerteste Vorteil ist, dass Kryo-ET-Proben aus gefrorenen intakten Zellen bestehen, die native Komplexe im Kontext der bakteriellen Zellarchitektur bewahren. Daher kann Kryo-ET biochemischen Reinigungsansätzen vorzuziehen sein, die Membrankomplexe extrahieren und periphere Proteine aus dem Kernapparat entfernen oder die Gesamtstruktur verändern können. Darüber hinaus fügt die Kennzeichnung eines von Interesse sindden Proteins mit einem sperrigen Protein wie sfGFP eine Masse hinzu, die durch Cryo-ET nachweisbar ist und bei der Kartierung der verschiedenen Unterkomplexe des Dot/Icm-Geräts auf die durch Cryo-ET erhaltene Struktur helfen kann.
Dieser Ansatz ist ein leistungsfähiges Werkzeug, um strukturelle Informationen über multimolekulare Komplexe aufzudecken, die sich in der bakteriellen Zellmembran zusammensetzen. Die Interpretation von Strukturen, die mit diesen Techniken aufgeklärt werden, wird dem Feld helfen zu verstehen, wie T4BSS-Komponenten funktionieren, warum so viele Komponenten für die Funktion benötigt werden, wie die Komponenten innerhalb des größeren Komplexes interagieren und welche Funktionen diese Unterbaugruppen ausführen.
Protokoll
HINWEIS: Alle Verfahren, die das Wachstum, die Manipulation und die Bildgebung von L. pneumophila beinhalten, sollten in einem Labor der biologischen Sicherheitsstufe 2 in Übereinstimmung mit den lokalen Richtlinien durchgeführt werden.
1. Einfügung von sfGFP in L. pneumophila Chromosom mit Allelic Exchange und Double Selection Strategy (Abbildung 2, Abbildung 3)
- Klonen Sie in den Genersatzvektor pSR47S11 die folgende Sequenz: die 1.000 bp vor dem Standort von Interesse, dann die sfGFP-Sequenz, dann die 1.000 bp flussabwärts des Sehenswürdigkeiten (Abbildung 2). Die sfGFP-Sequenz sollte in Frame zu den N-Terminus- oder C-Terminus-Enden mit einem Linker platziert werden, der vier bis acht Aminosäuren enthält. Verwandeln Sie den resultierenden Vektor in E. coli DH5-pir. Später, Streifen L. pneumophila (der Empfänger) für einzelne Kolonien auf Holzkohle-Hefe-Extrakt (CYE) Agar12 enthält 100 g/ml Streptomycin und wachsen für 5 Tage bei 37 °C (Abbildung 3).
- Streak L. pneumophila auf CYE-Agar-Streptomycin und wachsen für 2 Tage bei 37 °C (schweres Pflaster)10. Streak E. coli DH5- transformiert mit pRK600 Helfer-Plasmid (Helfer)13 auf LB-Agar mit 25 g/ml Chloramphenicol. Streichen Sie den E. coli DH5-pir (Spender) auf LB-Agar, der 50 g/ml Kanamycin enthält.
- Führen Sie eine triparentale Paarung durch: inkubieren Sie eine Kolonie des Helfers, einer Kolonie des Spenders und des Empfängers, indem Sie Flecken der drei Stämme auf einer CYE-Agarplatte ohne Auswahl überlagern und 4–8 h bei 37 °C bebrüten. Als negative Kontrollen inkubieren eine Helfer +Empfänger-Stamm-Mix und ein Spender + Empfänger Stamm-Mix für den gleichen Zeitraum.
- Setzen Sie die Paarungsreaktionen in 500 l ddH2O. Platte 20 l und 50 l der Reaktionen auf CYE-Agar mit 100 g/ml Streptomycin und 10 g/ml Kanamycin wieder auf und wachsen 5 Tage bei 37 °C. Streifen vier der resultierenden Klone auf CYE-Agar, der 100 g/ml Streptomycin enthält, und wachsen 5 Tage lang bei 37 °C.
- Streak 16 Klone auf CYE-Agar mit 5% Saccharose und 100 g/ml Streptomycin und wachsen 5 Tage bei 37 °C. Dann Streifen 32 dieser Klone auf CYE-Agar, der 100 g/ml Streptomycin enthält, und auf CYE-Agar, der 100 g/ml Streptomycin und 10 g/ml Kanamycin enthält, und 5 Tage bei 37 °C wachsen.
2. Isolierung von Klonen, die sfGFP in das L. pneumophila Chromosom integriert haben
- Streifenklone, die empfindlich auf Kanamycin auf CYE-Agar-Streptomycin-Platten waren und bestätigen das Einbringen von sfGFP in das Chromosom mit Kolonie PCR. Verwenden Sie Primer, die das sfGFP-Gen und den chromosomalen Bereich von Interesse ergänzen, um die Einfügekreuzung zu verstärken.
- Mischen Sie 0,5 l der 10-M-Primer-Lösungen und eine Kolonie auf ein Endvolumen von 12,5 l und denaturieren Sie 10 min bei 95 °C. 10 min auf Eis abkühlen, 12,5 L 2x PCR-Master-Mix-Lösung hinzufügen und eine PCR-Analyse durchführen.
- Wachsen Sie schwere Flecken der isolierten Kolonien auf CYE-Agar-Streptomycin-Platten für 2 Tage bei 37 °C. Untersuchen Sie den Expressionsgehalt und die Stabilität der sfGFP-Fusionen durch Immunoblotting mit einem Anti-GFP-Antikörper.
3. Live Cell Imaging von L. pneumophila mit fluoreszierend tagged Dot/Icm Komponenten
- Zubereitung von Agarose-Pads
- Machen Sie ca. 30 ml einer 1% niedrigschmelzenden Agarose-Lösung in Wasser. Mikrowelle in einem Glaskolben für ca. 90 s, gelegentlich wirbelnd, bis die Agarose vollständig aufgelöst ist.
- Legen Sie zwei 22 x 22 x 0,15 mm3 Glasschlitten am Rand eines 25 x 75 x 1,1 mm3 Glasschlittens, einer über dem anderen. Stapeln Sie zwei weitere 22 x 22 x 0,15 mm3 Glasschlitten auf der anderen Kante.
- Pipette ca. 1 ml der geschmolzenen Agarose in die Mittelrutsche zwischen den beiden oberen Glasrutschen, dann legen Sie eine weitere 25 x 75 x 1,1 mm3 Rutsche auf die geschmolzene Agarose. Versuchen Sie, die Bildung von Luftblasen zu vermeiden. Kühlen Sie die Dias bei 4 °C für 15 min.
- Schneiden Sie das Pad mit einem Skalpell oder einer Rasierklinge vorsichtig in kleine Quadrate, 5 x 5 mm2. Befestigen Sie einen doppelseitigen Klebstoff 17 x 28 x 0,25 mm3 Rahmen auf einem 25 x 75 x 1,1 mm3 Glasschlitten und legen Sie mehrere Pads auf dem Schlitten.
- Bildaufnahme
HINWEIS: Die folgenden Schritte werden für ein Mikroskop beschrieben, das unter der Kontrolle von SlideBook 6.0 steht und mit Festkörper-Beleuchtungskörpern, EINER CCD-Monochromkamera und einem 100-fachen Objektiv (1,4 numerische Blende) ausgestattet ist. Verwenden Sie bei Bedarf alternative Mikroskopiegeräte mit entsprechenden Hardware- und Softwarekonfigurationen, die an die Protokolleinstellungen angepasst werden können.- Lösen Sie ein schweres Pflaster von L. pneumophila in 1 ml ddH2O, Wirbel und Pipet 2–3 l der Verdünnung auf die Pads. Legen Sie einen 50 x 24 x 0,15 mm 3-Abdeckungsrutsch sanft über den Kleberahmen.
- Passen Sie im Aufnahmefenster die ND auf 180 an. Passen Sie das Binning auf 2x2 an und verwenden Sie den 488 nm-Kanal, um die Probe zwischen 500–1.000 ms freizulegen. Validieren Sie die Spezifität des Fluoreszenzsignals, indem Sie nicht markierte L. pneumophila mit den gleichen Parametern abbilden (Abbildung 4).
4. Quantifizierung der Polarlokalisierung und Dynamik von Punkt/Icm-Komponenten
HINWEIS: Die folgenden Schritte sind für Bilder mit 0,129 m pro Pixel konzipiert, die mit 2 x 2 Binning aufgenommen wurden.
- Quantifizierung der Polarität für sfGFP-Fusionsproteine (Abbildung 5)
- Passen Sie den Bildkontrast so an, dass die Bakterien deutlich sichtbar sind. Verwenden Sie das Regionswerkzeug, um ein Rechteck von 0,25 x 1,3 m2 am Pol zu platzieren, das sich in das Zytoplasma ausdehnt. Das Rechteck muss genau innerhalb der bakteriellen Grenzen bleiben.
- Markieren Sie mindestens 200 Bakterien, und verwenden Sie die Schaltfläche "Region zum Maskieren", um Masken für die Interessengebiete zu erstellen. Wählen Sie unter Maskenstatistik und Maskenbereich Objektaus. Markieren Sie dann unter Features und Intensität die mittlere Intensität und Varianz.
- Exportieren Sie die Daten und berechnen Sie die Polaritätswerte jedes Bakteriums als Verhältnis zwischen der Varianz und der mittleren Intensität.
- Für Anwendungen mit hohem Durchsatz verwenden Sie ein Phasenobjektiv und ein entsprechendes Kondensator-Setup, um Bilder mit den Phasen- und 488 nm-Kanälen zu erfassen. Achten Sie darauf, Sichtfelder zu wählen, in denen die Bakterien vollständig getrennt sind.
- Passen Sie den Phasenkanalkontrast des Bildes auf eine Ebene an, auf der die Bakterien deutlich sichtbar sind. Öffnen Sie das Dual-Channel-Bild, starten Sie das Fenster Segmentmaske erstellen und ändern Sie den Kanal in Phase.
- Passen Sie einen geeigneten Schwellenwert an, und entfernen Sie kleine Objekte mit der Schaltfläche Objekte definieren. Wählen Sie unter Maske verfeinern die Option Kantenobjekte entfernen und später separate Masken von Bakterien, die nebeneinander liegen.
- Berechnen Sie die Polaritätswerte des Signals im 488-Kanal für jede Zelle, wie zuvor in den Schritten 4.1.2–4.1.3 beschrieben.
- Quantifizierung der Dynamik für sfGFP-Fusionsproteine (Abbildung 6)
HINWEIS: Befolgen Sie die Anweisungen in Abschnitt 3, um ein Beispiel für die Bildaufnahme vorzubereiten. Dynamik ist definiert als Änderungen der Intensität im Laufe der Zeit und die folgenden Schritte sind für kurze Bildgebungszeiträume (d. h. mehrere Minuten) ausgelegt. Fügen Sie dem Pad die entsprechenden Ergänzungen hinzu, wenn längere Bildgebungszeiten gewünscht werden.- Markieren Sie im Fenster Bildaufnahme Zeitraffer, geben Sie die Zeit der Intervalle in das Intervallfeld ein, und geben Sie 2 in das Feld Zeitpunkte ein. Erwerben Sie zwei aufeinanderfolgende Bilder von L. pneumophila, die das fluoreszierende Protein von Interesse ausdrücken.
- Passen Sie den Bildkontrast an, bis die Zellen deutlich sichtbar sind. Folgen Sie Abbildung 6A und den folgenden Beschreibungen, um drei verschiedene Masken zu platzieren. Verwenden Sie das Regionswerkzeug, um ein Quadrat von 0,25 x 0,25 m in der Mitte von mindestens 400 Zellen zu platzieren.
- Verwenden Sie die Schaltfläche "Zu maskieren Region", um eine Maske (Maske 1) der interessenden Quadrate zu erstellen. Erstellen Sie eine neue leere Maske (Maske 2) und verwenden Sie das Pixelwerkzeug oder das Polygonwerkzeug, um die gesamte Zellfläche von mindestens 25 zufälligen Zellen zu markieren, die zum Berechnen der Fluoreszenzbleiche verwendet wird. Erstellen Sie eine neue leere Maske (Maske 3), und markieren Sie mit dem großen Pinselwerkzeug Bereiche zwischen den Zellen, die für die Hintergrundsubtraktion verwendet werden.
- Wählen Sie unter Maskenstatistik und Maskenbereich Objekt für Maske 1 und Maske 2 aus. Wählen Sie dann unter Features und Intensität die mittlere Intensität aus und exportieren Sie die Daten der beiden Masken. Exportieren Sie für Maske 3 die mittlere Intensität der gesamten Maske.
- Berechnen Sie die Änderung der Fluoreszenzintensität für jedes Objekt in Maske 1 mit den folgenden Formeln:

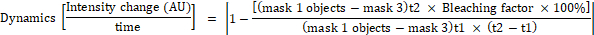
wobei Maske 1 ein quadratisch von 0,25 m bei 0,25 m in der Zellenmitte ist, Maske 2 die gesamte Zelle abdeckt, Maske 3 der Hintergrund zwischen den Zellen, t1 die mittlere Intensität im ersten Zeitpunkt und t2 die mittlere Intensität im zweiten Zeitpunkt ist.
5. Detektion der sfGFP-Massendichte mit Cryo-ET
- Probenvorbereitung, Datenerfassung und -rekonstruktion
- Wachsen Sie einen schweren Fleck L. pneumophila, der ein fluoreszierend markiertes Dot/Icm-Protein auf CYE-Agar-Streptomycin-Platten 48 Stunden lang bei 37 °C ausdrückt. Setzen Sie die Zellen in ddH2O bis OD600 0,7 wieder aus. Fügen Sie 5 l kolloidale Goldpartikel (BSA Tracer, 10 nm) bis 20 l der Zellsuspension hinzu.
- Pipette 5 l des Zellgemisches auf frisch glühend entludtes holey Kohlenstoffgitter (R 2/1 auf Cu 200 Mesh) und 1 min stehen lassen. Mit Filterpapier bloten und in flüssigem Ethan mit einem schwerkraftgetriebenen Kolbengerät einfrieren, wie zuvorbeschrieben 14,15.
- Stellen Sie die gefrorenen Hydratten mit einem 300 kV Transmissionselektronenmikroskop auf, das mit einer Feldemissionspistole, einem Energiefilter, einer Volta-Phasenplatte und einer Direktdetektionsvorrichtung ausgestattet ist. Sammeln Sie einachsige Neigungsreihen mit 26.000x bzw. 42.000x Vergrößerungen, die zu Pixelgrößen auf der Probenebene von 5,4 x/Pixel bzw. 3,4 X/Pixel führen.
- Verwenden Sie das tomographische Paket SerialEM, um Bildstapel mit einem Neigungswinkelbereich zwischen -60° und +60° mit 3° Schrittinkrement und einer akkumulativen Dosis von 60 e-/2,16zu sammeln. Richten Sie dosisfraktionierte Filmbilder in jedem Stapel mit MotionCor217aus. Montieren Sie driftkorrigierte Stapel mit TOMOAUTO14.
- Ausrichtung von driftkorrigierten Stapeln durch die IMOD-Marker-abhängige Ausrichtung18. Rekonstruieren Sie Tomogramme mit der SIRT-Methode19 für Segmentierungen und direkte Bildanalyse und der WBP-Methode20.
- Subtomogrammanalyse von sfGFP-Fusionsproben
- Verwenden Sie das tomographische Paket I3 (0.9.9.3) für die Subtomogrammanalyse14,21,22.
ANMERKUNG: Die Ausrichtung verläuft iterativ, wobei jede Iteration aus drei Teilen besteht, in denen Referenzen und Klassifizierungsmasken generiert, Subtomogramme ausgerichtet und klassifiziert werden und Klassendurchschnitte aneinander ausgerichtet werden. - Verwenden Sie 4 x 4 x 4 binned Subtomogramme für eine anfängliche Ausrichtung. Verschmelzen Sie Partikel, die zu den Klassendurchschnitten gehören, und zeigen Sie eine Elektronendichte, die sfGFP entspricht. Nach dem Sortieren von Partikeln mit sfGFP-Fusionen verwenden Sie 2 x 2 x 2 binned Subtomogramme für eine fokussierte Ausrichtung eines Bereichs von Interesse (wie den Dot/Icm zytoplasmatischen ATPase-Komplex), um eine hochauflösende Struktur zu erhalten.
- Verwenden Sie das tomographische Paket I3 (0.9.9.3) für die Subtomogrammanalyse14,21,22.
Ergebnisse
Homologe Rekombination mit doppelter Auswahl in zwei Schritten wurde verwendet, um die definierte Einfügung von sfGFP zu konstruieren. Im ersten Schritt wurde eine triparentale Paarung durchgeführt, bei der das konjugative Plasmid pRK600 (ein IncP-Plasmid) aus dem E. coli-Helferstamm MT616 mit dem Selbstmordvektor pSR47S, der das sfGFP-Gen enthält, das von den beiden homologen Regionen, dem Ursprung des TransferoriTs und dem Bacillus subtilis-Gegenselektions-SacB, flankiert wurde, zum Spender E. ...
Diskussion
Die Aufklärung der Funktionen bakterieller Sekretionssysteme ist der Schlüssel zu einem vollständigen Verständnis der Wirts-Pathogen-Wechselwirkungen. Sekretionssysteme sind komplexe Maschinen, die Effektorenproteine in Wirtszellen injizieren können und in einigen Fällen die Etablierung einer subzellulären Nische fördern, die die bakterielle Replikation unterstützt. Die obige Methode bietet wichtige neue Werkzeuge für die Untersuchung des Dot/Icm-Sekretionssystems des reinspiratorischen bakteriellen Erregers
Offenlegungen
Die Autoren haben nichts zu verraten.
Danksagungen
D.C. und C.R.R. wurden vom NIH (R37AI041699 und R21AI130671) unterstützt. D.P., B.H. und J.L wurden von den National Institutes of Health (R01AI087946 und R01GM107629) unterstützt.
Materialien
| Name | Company | Catalog Number | Comments |
| 10 nm colloidal gold particles | Aurion | 25486 | |
| 100x Plan Apo objective (1.4 NA) | Nikon | ||
| ACES | Sigma-Aldrich | A9758 | |
| Activated charcoal | Sigma-Aldrich | C5510 | |
| Agaroze GPG/LMP, low melt | American bioanalytical | AB00981 | |
| Bacto dehydrated agar | BD | 214010 | |
| CoolSNAP EZ 20 MHz digital monochrome camera | Photometrics | ||
| Gene Frame, 1.7x2.8 cm, 125 µL | Fisher Scientific | AB-0578 | |
| Holey Carbon grid R 2/1 Cu 200 mesh | Quantifoil | Q225-CR1 | |
| Iron(III) nitrate nonahydrate | Sigma-Aldrich | 216828 | |
| K2 Summit camera for cryo-EM | GATAN | ||
| L-Cysteine | Sigma-Aldrich | C7352 | |
| Microscope cover slides 22x22 mm | Fisher Scientific | 12-542B | |
| Microscope cover slides 24x50 mm | Fisher Scientific | 12-545K | |
| Microscope slides 25x75x1 mm | Globe Scientific | 1380 | |
| SlideBook 6.0 | Intelligent Imaging Innovations | ||
| Spectra X light engine | Lumencor | ||
| Taq 2X Master Mix | New England BioLabs | M0270 | |
| Titan Krios | Thermo Fisher Scientific | ||
| Yeast Extract | BD | 212750 |
Referenzen
- Franco, I. S., Shuman, H. A., Charpentier, X. The perplexing functions and surprising origins of Legionella pneumophila type IV secretion effectors. Cellular Microbiology. 11, 1435-1443 (2009).
- Burstein, D., et al. Genome-scale identification of Legionella pneumophila effectors using a machine learning approach. PLOS Pathogens. 5, 1000508 (2009).
- Ninio, S., Roy, C. R. Effector proteins translocated by Legionella pneumophila: strength in numbers. Trends in Microbiology. 15, 372-380 (2007).
- Vogel, J. P., Andrews, H. L., Wong, S. K., Isberg, R. R. Conjugative transfer by the virulence system of Legionella pneumophila. Science. 279, 873-876 (1998).
- Isberg, R. R., O'Connor, T. J., Heidtman, M. The Legionella pneumophila replication vacuole: making a cosy niche inside host cells. Nature Reviews Microbiology. 7, 13-24 (2009).
- Roy, C. R., Berger, K. H., Isberg, R. R. Legionella pneumophila DotA protein is required for early phagosome trafficking decisions that occur within min of bacterial uptake. Molecular Microbiology. 28, 663-674 (1998).
- Archer, K. A., Roy, C. R. MyD88-Dependent Responses Involving Toll-Like Receptor 2 Are Important for Protection and Clearance of Legionella pneumophila in a Mouse Model of Legionnaires' Disease. Infection and Immunity. 74, 3325-3333 (2006).
- Nagai, H., Kubori, T. Type IVB Secretion Systems of Legionella and Other Gram-Negative Bacteria. Frontiers in Microbiology. 2, 136 (2011).
- Kubori, T., Nagai, H. The Type IVB secretion system: an enigmatic chimera. Current Opinion in Microbiology. 29, 22-29 (2016).
- Chetrit, D., Hu, B., Christie, P. J., Roy, C. R., Liu, J. A unique cytoplasmic ATPase complex defines the Legionella pneumophila type IV secretion channel. Nature Microbiology. 3, 678-686 (2018).
- Merriam, J. J., Mathur, R., Maxfield-Boumil, R., Isberg, R. R. Analysis of the Legionella pneumophila fliI gene: intracellular growth of a defined mutant defective for flagellum biosynthesis. Infection and Immunity. 65, 2497-2501 (1997).
- Feeley, J. C., et al. Charcoal-yeast extract agar: primary isolation medium for Legionella pneumophila. Journal of Clinical Microbiology. 10, 437-441 (1979).
- Andrews, H. L., Vogel, J. P., Isberg, R. R. Identification of linked Legionella pneumophila genes essential for intracellular growth and evasion of the endocytic pathway. Infection and Immunity. 66, 950-958 (1998).
- Morado, D. R., Hu, B., Liu, J. Using Tomoauto: A Protocol for High-throughput Automated Cryo-electron Tomography. Journal of Visualized Experiments. (107), e53608 (2016).
- Hu, B., Lara-Tejero, M., Kong, Q., Galan, J. E., Liu, J. In Situ Molecular Architecture of the Salmonella Type III Secretion Machine. Cell. 168, 1065-1074 (2017).
- Mastronarde, D. N. Automated electron microscope tomography using robust prediction of specimen movements. Journal of Structural Biology. 152, 36-51 (2005).
- Zheng, S. Q., et al. MotionCor2: anisotropic correction of beam-induced motion for improved cryo-electron microscopy. Nature Methods. 14, 331-332 (2017).
- Kremer, J. R., Mastronarde, D. N., McIntosh, J. R. Computer visualization of three-dimensional image data using IMOD. Journal of Structural Biology. 116, 71-76 (1996).
- Gilbert, P. Iterative methods for the three-dimensional reconstruction of an object from projections. Journal of Theoretical Biology. 36, 105-117 (1972).
- Radermacher, M. Weighted Back-projection Methods. Electron Tomography. , 245-273 (2007).
- Winkler, H., et al. Tomographic subvolume alignment and subvolume classification applied to myosin V and SIV envelope spikes. Journal of Structural Biology. 165, 64-77 (2009).
- Winkler, H., Taylor, K. A. Accurate marker-free alignment with simultaneous geometry determination and reconstruction of tilt series in electron tomography. Ultramicroscopy. 106, 240-254 (2006).
- Prevost, M. S., Waksman, G. X-ray crystal structures of the type IVb secretion system DotB ATPases. Protein Science. 27, 1464-1475 (2018).
- Miklos, G. L., Rubin, G. M. The role of the genome project in determining gene function: insights from model organisms. Cell. 86, 521-529 (1996).
- Reyrat, J. M., Pelicic, V., Gicquel, B., Rappuoli, R. Counterselectable markers: untapped tools for bacterial genetics and pathogenesis. Infection and Immunity. 66, 4011-4017 (1998).
- Yu, J. Single-Molecule Studies in Live Cells. Annual Review of Physical Chemistry. 67, 565-585 (2016).
- Stewart, P. L. Cryo-electron microscopy and cryo-electron tomography of nanoparticles. Wiley Interdisciplinary Reviews: Nanomedicine and Nanobiotechnology. 9, (2017).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenWeitere Artikel entdecken
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten