Se requiere una suscripción a JoVE para ver este contenido. Inicie sesión o comience su prueba gratuita.
Method Article
Aplicación de imágenes de células vivas y tomografía crioelectrónica para resolver las características espaciotemporales del sistema de secreción de puntos de legionela pneumofilia/Icm
En este artículo
Resumen
La imagen de células bacterianas es un enfoque de biología de sistemas emergentes centrado en la definición de procesos estáticos y dinámicos que dictan la función de grandes máquinas macromoleculares. Aquí, la integración de imágenes cuantitativas de células vivas y tomografía crioelectrónica se utiliza para estudiar la arquitectura y las funciones del sistema de secreción de legionela pneumophila tipo IV.
Resumen
El sistema de secreción de Punto/Cm de Legionella pneumophila es una nanomáquina compleja del sistema de secreción tipo IV (T4SS) que se localiza en el polo bacteriano y media la entrega de proteínas y sustratos de ADN a las células diana, un proceso que generalmente requiere contacto directo de célula a célula. Recientemente hemos resuelto la estructura del aparato Dot/Icm mediante tomografía crioelectrónica (crio-ET) y hemos demostrado que forma un canal de expansión celular que se conecta a un complejo citoplasmático. La aplicación de dos enfoques complementarios que preservan la estructura nativa de la muestra, la microscopía fluorescente en células vivas y crio-ET, permite la visualización in situ de proteínas y la asimilación de la estequiometría y el momento de producción de cada componente de la máquina en relación con otras subunidades Dot/Icm. Para investigar los requisitos para el posicionamiento polar y caracterizar las características dinámicas asociadas con la biogénesis de la máquina T4SS, hemos fusionado un gen que codifica la proteína fluorescente verde de la supercarpeta a los genes DOT/Icm ATPase en sus posiciones nativas en el cromosoma. El siguiente método integra la microscopía cuantitativa de fluorescencia de células vivas y crio-ET para cuantificar la localización polar, la dinámica y la estructura de estas proteínas en células bacterianas intactas. La aplicación de estos enfoques para el estudio de la Legionella pneumophila T4SS es útil para caracterizar la función del sistema Punto/Icm y se puede adaptar para estudiar una amplia variedad de patógenos bacterianos que utilizan el T4SS u otros tipos de complejos de secreción bacteriana.
Introducción
Legionella pneumophila (L. pneumophila), el agente etiológico de la enfermedad de los legionarios, habita en los reservorios de agua dulce, donde las bacterias se propagan infectando y replicando dentro de los protozoos acuáticos de natación libre. L. pneumophila causa brotes de enfermedades en los seres humanos cuando se produce la inhalación de bacterias aerosolizadas de fuentes de agua potable. En las células infectadas, la subversión de las vías huésped permite a L. pneumophila retrasar la maduración endocítica de la vacuola en la que reside y promover la biogénesis de un compartimento celular que apoya la replicación bacteriana. Este proceso es impulsado por un sistema especializado de secreción IVB de tipo bacteriano (T4BSS) conocido como Dot/Icm y su repertorio de más de 300 proteínas "efectoras" que se trasladan al citosol huésped durante la infección para facilitar la manipulación de las funciones celulares1,2,3,4,5. Los mutantes que carecen de un aparato funcional Dedo/Cm no entregan efectores en el citosol del huésped, son defectuosos para la replicación intracelular y son avirulentes en modelos animales de enfermedad6,7.
Muchas especies bacterianas han desarrollado máquinas multicomponente extremadamente complejas y dinámicas que son necesarias para los procesos de infección. Otros T4BSS como el sistema Dot/Icm también son esenciales para la replicación intracelular de patógenos bacterianos como Coxiella burnetii y Rickettsiella grylli. Aunque el T4BSS está relacionado evolutivamente con los sistemas prototípicos de tipo IVA, que median la transferencia de ADN y pueden ofrecer un repertorio limitado de proteínas de efectores, el sistema Dot/Icm tiene casi el doble de componentes de la máquina y ofrece una amplia variedad de efectores. Presumiblemente, esta expansión en el número de componentes ha permitido que el aparato Dot/Icm acomode e integre nuevos efectores fácilmente8,9.
Recientemente usamos la tomografía crioelectrónica (crio-ET) para resolver la estructura del aparato Dot/Icm in situ y mostramos que forma un canal de expansión celular que se conecta a un complejo citoplasmático. Análisis posterior reveló que el citosólico ATPase DotB se asocia con el sistema Dot/Icm en el polo celular L. pneumophila a través de interacciones con el citosólico ATPase DotO. Hemos descubierto que DotB muestra un movimiento citosólico en la mayoría de las células bacterianas, lo que indica que este ATPase está presente en una población citosólica dinámica, pero también se asocia con los complejos de puntos polares/icm. Además, DotO forma un conjunto hexamerico de dimers DotO asociados con el complejo de membrana interna, y un hexamer DotB se une a la base de este complejo citoplasmático. El conjunto de la energía DotB-DotO crea un canal citoplasmático que dirige la translocación de sustratos a través del T4SS(Figura 1)10.
A pesar de estos avances recientes, poco se sabe sobre cómo funciona el sistema Dot/Icm y cómo cada proteína se ensambla para formar un aparato activo8. Descubrir los circuitos reguladores del Dot/Icm T4SS es fundamental para entender los mecanismos moleculares de las interacciones huésped-patógeno. Por lo tanto, analizamos cómo utilizar la microscopía de células vivas y crio-ET para detectar y caracterizar los componentes esenciales del sistema L. pneumophila Dot/Icm que están etiquetados con la supercarpeta GFP (sfGFP). Mediante la microscopía cuantitativa de fluorescencia, la localización polar de DotB se definirá en un fondo de tipo salvaje o cuando se elimine el sistema de tipo IV. La microscopía de lapso de tiempo se utilizará para cuantificar las diferencias en la localización y la dinámica entre las ATPases citosólicas Dot/Icm.
La aplicación combinada de dos enfoques complementarios como la imagen en vivo y el crio-ET proporciona una ventaja en comparación con otros sistemas in vitro. Ambos métodos se realizan en celdas intactas y preservan el entorno natural del T4BSS, minimizando así la interrupción de la estructura nativa durante la preparación de la muestra. Debido a que la sobreexpresión de proteínas puede afectar a la estequiometría del aparato de secreción, las fusiones sfGFP se devuelven a través del intercambio alélico al cromosoma Legionella para que cada fusión esté codificada en una sola copia y la expresión sea impulsada por el promotor endógeno. La visualización de fusiones codificadas cromosómicamente permite cuantificar el nivel exacto de proteína expresada en un momento dado definido. Cryo-ET también tiene muchas ventajas para determinar la estructura de los sistemas de secreción. La ventaja más notable es que las muestras crio-ET se componen de células intactas congeladas que preservan complejos nativos en el contexto de la arquitectura celular bacteriana. En consecuencia, el crio-ET puede ser preferible a los enfoques de purificación bioquímica, que extraen complejos de membrana y pueden extraer proteínas periféricas del aparato central o modificar la estructura general. Además, el etiquetado de una proteína de interés con una proteína voluminosa como el sfGFP añade una masa que es detectable por crio-ET y puede ayudar a mapear los diferentes subcomplejos del aparato Punto/Cm en la estructura obtenida por crio-ET.
Este enfoque es una poderosa herramienta para descubrir información estructural sobre complejos multimoleculares que se ensamblan en la membrana celular bacteriana. La interpretación de las estructuras dilucidadas utilizando estas técnicas ayudará al campo a entender cómo funcionan los componentes t4BSS, por qué se requieren tantos componentes para la función, cómo interactúan los componentes dentro del complejo más grande y qué funciones estos subensamblajes de ejecución.
Protocolo
NOTA: Todos los procedimientos relacionados con el crecimiento, manipulación e imágenes de L. pneumophila deben realizarse en un laboratorio de nivel de seguridad biológica 2 de conformidad con las directrices locales.
1. Inserción de sfGFP en el cromosoma L. pneumophila utilizando el intercambio alélico y la estrategia de doble selección(Figura 2, Figura 3)
- Clonar en el vector de reemplazo genético pSR47S11 la siguiente secuencia: la 1.000 bp aguas arriba del sitio de interés, luego la secuencia sfGFP, luego la 1.000 bp aguas abajo del sitio de interés(Figura 2). La secuencia sfGFP debe colocarse en el marco de los extremos De N-terminus o C-terminus con un vinculador que contenga de cuatro a ocho aminoácidos. Transforme el vector resultante en E. coli DH5-pir. Más tarde, la raya L. pneumophila (el receptor) para colonias individuales en el agar extracto de carbón-levadura (CYE)12 que contiene 100 g/ml de estreptomicina y crecer durante 5 días a 37 oC(Figura 3).
- Racha L. pneumophila en CYE-agar-estreptomicina y crecer durante 2 días a 37 oC (parche pesado)10. La racha E. coli DH5, transformada con plásmido auxiliar pRK600 (ayudante)13 en el agar LB que contiene 25 g/ml de cloramphenicol. Raya el E. coli DH5-pir (donante) en el agar LB que contiene 50 g/ml de kanamicina.
- Realizar apareamiento triparental: incubar una colonia del ayudante, una colonia del donante y el receptor superponiendo parches de las tres cepas en una placa de agar CYE sin selección e incubando durante 4-8 h a 37oC. A medida que los controles negativos incuban una mezcla de cepa auxiliar+receptor y una mezcla de cepa donante+receptor durante los mismos períodos de tiempo.
- Resuspenda las reacciones de apareamiento en 500 sL de ddH2O. Placa 20 l y 50 l de las reacciones en el agar CYE que contenga 100 g/ml de estreptomicina y 10 g/ml de kanamicina y crezca durante 5 días a 37 oC. Raya cuatro de los clones resultantes en el agar CYE que contiene 100 g/ml de estreptomicina y crece durante 5 días a 37 oC.
- Racha 16 clones en agar CYE que contiene 5% de sacarosa y 100 g/ml de estreptomicina y crece durante 5 días a 37 oC. Luego, la raya 32 de estos clones en el agar CYE que contiene 100 g/ml de estreptomicina y en el agar CYE que contiene 100 g/ml de estreptomicina y 10 g/ml de kanamicina y crece durante 5 días a 37 oC.
2. Aislamiento de clones que integraron sfGFP en el cromosoma L. pneumophila
- Clones de rayas que eran sensibles a la kanamicina en placas CYE-agar-estreptomicina y confirman la inserción de sfGFP en el cromosoma con PCR de colonia. Utilice imprimadores complementarios al gen sfGFP y a la región cromosómica de interés para amplificar la unión de inserción.
- Mezclar 0,5 ml de cada una de las soluciones de imprimación de 10 m y una colonia a un volumen final de 12,5 ml y desnaturalizar durante 10 min a 95 oC. Enfríe en hielo durante 10 min, agregue 12,5 l de solución de mezcla maestra de PCR 2 x y realice un análisis de PCR.
- Cultivar manchas pesadas de las colonias aisladas en placas CYE-agar-estreptomicina durante 2 días a 37 oC. Examine los niveles de expresión y la estabilidad de las fusiones sfGFP inmunoblotando con un anticuerpo anti-GFP.
3. Imagen de celda viva de L. pneumophila con componentes de punto/icm etiquetados fluorescentemente
- Preparación de almohadillas de agarosa
- Hacer unos 30 ml de una solución de agarosa 1% baja en agua. Microondas en un matraz de vidrio durante unos 90 s, girando ocasionalmente, hasta que la agarosa se disuelva por completo.
- Coloque dos portaobjetos de vidrio de 22 x 22 x 0,15 mm3 en el borde de un portaobjetos de vidrio de 25 x 75 x 1,1 mm3, uno encima del otro. Apilar dos diapositivas de vidrio más de 22 x 22 x 0,15 mm3 en el otro borde.
- Pipetear aproximadamente 1 ml de la agarosa fundida en el tobogán central entre los dos portaobjetos de vidrio superior, luego coloque otro tobogán de 25 x 75 x 1,1 mm3 en la parte superior de la agarosa fundida. Trate de evitar la formación de burbujas de aire. Enfríe los portaobjetos a 4 oC durante 15 min.
- Usando un bisturí o una cuchilla de afeitar, corte suavemente la almohadilla en cuadrados pequeños, 5 x 5 mm2. Fije un adhesivo de doble cara 17 x 28 x 0.25 mm3 marco en un portacristales de 25 x 75 x 1.1 mm3 y coloque varias almohadillas en la diapositiva.
- Adquisición de imágenes
NOTA: Los siguientes pasos se describen para un microscopio que está bajo el control de SlideBook 6.0 y está equipado con iluminadores de estado sólido, cámara monocromática CCD y una lente objetivo de 100x (1.4 apertura numérica). Si es necesario, utilice dispositivos de microscopía alternativos con configuraciones de hardware y software adecuadas que se pueden personalizar de acuerdo con la configuración del protocolo.- Disolver un parche pesado de L. pneumophila en 1 ml de ddH2O, vórtice e pipeteo 2–3 l de la dilución en las almohadillas. Coloque un cubreobjetos de 50 x 24 x 0,15 mm3 suavemente sobre el marco adhesivo.
- En la ventana de captura ajuste el ND a 180. Ajustar el binning a 2 x 2 y utilizar el canal de 488 nm para exponer la muestra entre 500–1.000 ms. Validar la especificidad de la señal de fluorescencia mediante imágenes sin etiquetar L. pneumophila con los mismos parámetros (Figura 4).
4. Cuantificación de la Localización Polar y Dinámica de Componentes De Punto/Icm
NOTA: Los siguientes pasos están diseñados para imágenes con 0,129 m por píxel que se adquirieron con 2 x 2 binning.
- Cuantificación de la polaridad para proteínas de fusión sfGFP (Figura 5)
- Ajuste el contraste de la imagen para que las bacterias sean claramente visibles. Utilice la herramienta de región para colocar un rectángulo de 0,25 x 1,3m2 comenzando en el polo y extendiéndose hasta el citoplasma. El rectángulo debe permanecer precisamente dentro de los bordes bacterianos.
- Marca al menos 200 bacterias y usa el botón Región para enmascarar para crear máscaras para las regiones de interés. En Estadísticas de máscara y Alcance de máscara, elija Objeto. A continuación, en Características e intensidad, marque Intensidad media y varianza.
- Exporte los datos y calcule las puntuaciones de polaridad de cada bacteria como la relación entre la varianza y la intensidad media.
- Para aplicaciones de alto rendimiento, utilice un objetivo de fase y una configuración de condensador adecuada para adquirir imágenes con los canales de fase y 488 nm. Asegúrese de elegir los campos de visión donde las bacterias están completamente separadas.
- Ajuste el contraste del canal de fase de la imagen a un nivel donde las bacterias sean claramente visibles. Abra la imagen de canal dual, inicie la ventana Crear máscara de segmento y cambie el canal a fase.
- Ajuste un umbral adecuado y elimine objetos pequeños con el botón Definir objetos. En Refine Mask,elija Remove Edges Objects y, posteriormente, máscaras separadas de bacterias adyacentes entre sí.
- Calcule las puntuaciones de polaridad de la señal en el canal 488 para cada celda como se describió anteriormente en los pasos 4.1.2–4.1.3.
- Cuantificación de la dinámica de las proteínas de fusión sfGFP (Figura 6)
NOTA: Siga las instrucciones de la sección 3 para preparar una muestra para la adquisición de imágenes. La dinámica se define como cambios en la intensidad a lo largo del tiempo y los pasos siguientes están diseñados para períodos de imágenes cortos (es decir, varios minutos). Agregue a la almohadilla los suplementos apropiados si se desean períodos de diagnóstico por imágenes más largos.- En la ventana Captura de imagen, marque Timelapse, escriba el tiempo de intervalos en el cuadro intervalo e introduzca 2 en el cuadro Puntos de tiempo. Adquirir dos imágenes sucesivas de L. pneumophila expresando la proteína fluorescente de interés.
- Ajuste el contraste de la imagen hasta que las celdas sean claramente visibles. Siga la Figura 6A y las descripciones a continuación para colocar tres máscaras diferentes. Utilice la herramienta de región para colocar un cuadrado de 0,25 x 0,25 m en el centro de al menos 400 celdas.
- Utilice el botón Región para enmascarar para crear una máscara (máscara 1) de los cuadrados de interés. Cree una nueva máscara vacía (máscara 2) y utilice la herramienta píxel o la herramienta poligonal para marcar toda el área de celda de al menos 25 celdas aleatorias, que se utilizarán para calcular el blanqueo de fluorescencia. Cree una nueva máscara vacía (máscara 3) y utilice la herramienta de pincel grande para marcar áreas entre las celdas, que se utilizarán para la resta de fondo.
- En Estadísticas de máscara y Alcance de máscara, elija Objeto para máscara 1 y máscara 2. A continuación, en Características e intensidad, elija Intensidad media y exporte los datos de las dos máscaras. Para la máscara 3, exporte la intensidad media de toda la máscara.
- Calcule el cambio en la intensidad de la fluorescencia para cada objeto en la máscara 1 utilizando las siguientes fórmulas:

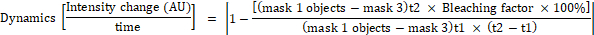
donde la máscara 1 es un cuadrado de 0,25 m a 0,25 m colocado en el centro de la célula, la máscara 2 cubre toda la celda, la máscara 3 es el fondo entre las células, t1 es la intensidad media en el primer punto de tiempo, y t2 es la intensidad media en el segundo punto de tiempo.
5. Detección de densidad de masa sfGFP con Cryo-ET
- Preparación de muestras, recopilación de datos y reconstrucción
- Cultivar un parche pesado de L. pneumophila expresando una proteína Dot/Icm etiquetada fluorescentemente en placas CYE-agar-streptomicina durante 48 horas a 37 oC. Resuspenda las celdas en ddH2O a OD600 a 0,7. Añadir 5 l de partículas de oro coloidal (BSA Tracer, 10 nm) a 20 l de la suspensión celular.
- Pipetear 5 l de la mezcla de células en una rejilla de carbono perforada recién descargada por el brillo (R 2/1 en malla Cu 200) y dejar reposar durante 1 min. Blot con papel de filtro y congelar en etantano líquido utilizando un aparato de émbolo impulsado por gravedad como se describió anteriormente14,15.
- Imagen de las muestras hidratadas congeladas con un microscopio electrónico de transmisión de 300 kV equipado con una pistola de emisión de campo, un filtro de energía, una placa de fase Volta y un dispositivo de detección directa. Recoge series de inclinación de un solo eje con aumentos de 26.000x y 42.000x, que dan como resultado tamaños de píxeles a un nivel de muestra de 5,4 o píxeles o 3,4 o píxeles, respectivamente.
- Utilice el paquete tomográfico SerialEM para recoger pilas de imágenes a un desenfoque de 0 m, con una gama de ángulos de inclinación entre -60o y +60o con incremento de paso de 3o y dosis acumulada de 60e -/a2,16. Alinee las imágenes de película fraccionadas por dosis en cada pila utilizando MotionCor217. Ensamble pilas corregidas a la deriva utilizando TOMOAUTO14.
- Alinee las pilas corregidas por deriva mediante la alineación dependiente del marcador IMOD18. Reconstruir mamografías con el método SIRT19 para segmentaciones y análisis directo de imágenes y el método WBP20.
- Análisis de subtomogramas de muestras de fusiones sfGFP
- Utilice el paquete tomográfico I3 (0.9.9.3) para el análisis de subtomogramas14,21,22.
NOTA: La alineación continúa de forma iterativa, con cada iteración que consta de tres partes en las que se generan referencias y máscaras de clasificación, los subtomogramas se alinean y clasifican, y los promedios de clase se alinean entre sí. - Utilice 4 x 4 x 4 subtomogramas binned para una alineación inicial. Combinar partículas que pertenecen a promedios de clase y mostrar la densidad de electrones correspondiente a sfGFP. Después de clasificar partículas con fusiones sfGFP, utilice subtogramas de 2 x 2 x 2 binned para una alineación enfocada de una región de interés (como el complejo ATPase citoplasma Dedo/Icm) para obtener una estructura de alta resolución.
- Utilice el paquete tomográfico I3 (0.9.9.3) para el análisis de subtomogramas14,21,22.
Resultados
La recombinación homóloga con doble selección en dos pasos se utilizó para construir la inserción definida de sfGFP. En el primer paso, se realizó el acoplamiento triparental, donde el plásmido conjugado pRK600 (un plásmido IncP) de la cepa auxiliar E. coli MT616 se movilizó a la cepa del donante E. coli con el vector suicida pSR47S que contiene el flanco sfGFP geneado por las dos regiones homólogas, el origen de la orita de transferencia y el gen de la subselección Bacillus subistitulaci...
Discusión
Elaclarar las funciones de los sistemas de secreción bacteriana es clave para una comprensión completa de las interacciones huésped-patógeno. Los sistemas de secreción son máquinas complejas que pueden inyectar proteínas efectoras en las células huésped, y en algunos casos promueven el establecimiento de un nicho subcelular que apoya la replicación bacteriana. El método anterior proporciona nuevas herramientas importantes para el estudio del sistema de secreción de puntos/mic del patógeno bacteriano respirat...
Divulgaciones
Los autores no tienen nada que revelar.
Agradecimientos
D.C. y C.R.R. fueron apoyados por los NIH (R37AI041699 y R21AI130671). D.P., B.H. y J.L fueron apoyados por los Institutos Nacionales de Salud (R01AI087946 y R01GM107629).
Materiales
| Name | Company | Catalog Number | Comments |
| 10 nm colloidal gold particles | Aurion | 25486 | |
| 100x Plan Apo objective (1.4 NA) | Nikon | ||
| ACES | Sigma-Aldrich | A9758 | |
| Activated charcoal | Sigma-Aldrich | C5510 | |
| Agaroze GPG/LMP, low melt | American bioanalytical | AB00981 | |
| Bacto dehydrated agar | BD | 214010 | |
| CoolSNAP EZ 20 MHz digital monochrome camera | Photometrics | ||
| Gene Frame, 1.7x2.8 cm, 125 µL | Fisher Scientific | AB-0578 | |
| Holey Carbon grid R 2/1 Cu 200 mesh | Quantifoil | Q225-CR1 | |
| Iron(III) nitrate nonahydrate | Sigma-Aldrich | 216828 | |
| K2 Summit camera for cryo-EM | GATAN | ||
| L-Cysteine | Sigma-Aldrich | C7352 | |
| Microscope cover slides 22x22 mm | Fisher Scientific | 12-542B | |
| Microscope cover slides 24x50 mm | Fisher Scientific | 12-545K | |
| Microscope slides 25x75x1 mm | Globe Scientific | 1380 | |
| SlideBook 6.0 | Intelligent Imaging Innovations | ||
| Spectra X light engine | Lumencor | ||
| Taq 2X Master Mix | New England BioLabs | M0270 | |
| Titan Krios | Thermo Fisher Scientific | ||
| Yeast Extract | BD | 212750 |
Referencias
- Franco, I. S., Shuman, H. A., Charpentier, X. The perplexing functions and surprising origins of Legionella pneumophila type IV secretion effectors. Cellular Microbiology. 11, 1435-1443 (2009).
- Burstein, D., et al. Genome-scale identification of Legionella pneumophila effectors using a machine learning approach. PLOS Pathogens. 5, 1000508 (2009).
- Ninio, S., Roy, C. R. Effector proteins translocated by Legionella pneumophila: strength in numbers. Trends in Microbiology. 15, 372-380 (2007).
- Vogel, J. P., Andrews, H. L., Wong, S. K., Isberg, R. R. Conjugative transfer by the virulence system of Legionella pneumophila. Science. 279, 873-876 (1998).
- Isberg, R. R., O'Connor, T. J., Heidtman, M. The Legionella pneumophila replication vacuole: making a cosy niche inside host cells. Nature Reviews Microbiology. 7, 13-24 (2009).
- Roy, C. R., Berger, K. H., Isberg, R. R. Legionella pneumophila DotA protein is required for early phagosome trafficking decisions that occur within min of bacterial uptake. Molecular Microbiology. 28, 663-674 (1998).
- Archer, K. A., Roy, C. R. MyD88-Dependent Responses Involving Toll-Like Receptor 2 Are Important for Protection and Clearance of Legionella pneumophila in a Mouse Model of Legionnaires' Disease. Infection and Immunity. 74, 3325-3333 (2006).
- Nagai, H., Kubori, T. Type IVB Secretion Systems of Legionella and Other Gram-Negative Bacteria. Frontiers in Microbiology. 2, 136 (2011).
- Kubori, T., Nagai, H. The Type IVB secretion system: an enigmatic chimera. Current Opinion in Microbiology. 29, 22-29 (2016).
- Chetrit, D., Hu, B., Christie, P. J., Roy, C. R., Liu, J. A unique cytoplasmic ATPase complex defines the Legionella pneumophila type IV secretion channel. Nature Microbiology. 3, 678-686 (2018).
- Merriam, J. J., Mathur, R., Maxfield-Boumil, R., Isberg, R. R. Analysis of the Legionella pneumophila fliI gene: intracellular growth of a defined mutant defective for flagellum biosynthesis. Infection and Immunity. 65, 2497-2501 (1997).
- Feeley, J. C., et al. Charcoal-yeast extract agar: primary isolation medium for Legionella pneumophila. Journal of Clinical Microbiology. 10, 437-441 (1979).
- Andrews, H. L., Vogel, J. P., Isberg, R. R. Identification of linked Legionella pneumophila genes essential for intracellular growth and evasion of the endocytic pathway. Infection and Immunity. 66, 950-958 (1998).
- Morado, D. R., Hu, B., Liu, J. Using Tomoauto: A Protocol for High-throughput Automated Cryo-electron Tomography. Journal of Visualized Experiments. (107), e53608 (2016).
- Hu, B., Lara-Tejero, M., Kong, Q., Galan, J. E., Liu, J. In Situ Molecular Architecture of the Salmonella Type III Secretion Machine. Cell. 168, 1065-1074 (2017).
- Mastronarde, D. N. Automated electron microscope tomography using robust prediction of specimen movements. Journal of Structural Biology. 152, 36-51 (2005).
- Zheng, S. Q., et al. MotionCor2: anisotropic correction of beam-induced motion for improved cryo-electron microscopy. Nature Methods. 14, 331-332 (2017).
- Kremer, J. R., Mastronarde, D. N., McIntosh, J. R. Computer visualization of three-dimensional image data using IMOD. Journal of Structural Biology. 116, 71-76 (1996).
- Gilbert, P. Iterative methods for the three-dimensional reconstruction of an object from projections. Journal of Theoretical Biology. 36, 105-117 (1972).
- Radermacher, M. Weighted Back-projection Methods. Electron Tomography. , 245-273 (2007).
- Winkler, H., et al. Tomographic subvolume alignment and subvolume classification applied to myosin V and SIV envelope spikes. Journal of Structural Biology. 165, 64-77 (2009).
- Winkler, H., Taylor, K. A. Accurate marker-free alignment with simultaneous geometry determination and reconstruction of tilt series in electron tomography. Ultramicroscopy. 106, 240-254 (2006).
- Prevost, M. S., Waksman, G. X-ray crystal structures of the type IVb secretion system DotB ATPases. Protein Science. 27, 1464-1475 (2018).
- Miklos, G. L., Rubin, G. M. The role of the genome project in determining gene function: insights from model organisms. Cell. 86, 521-529 (1996).
- Reyrat, J. M., Pelicic, V., Gicquel, B., Rappuoli, R. Counterselectable markers: untapped tools for bacterial genetics and pathogenesis. Infection and Immunity. 66, 4011-4017 (1998).
- Yu, J. Single-Molecule Studies in Live Cells. Annual Review of Physical Chemistry. 67, 565-585 (2016).
- Stewart, P. L. Cryo-electron microscopy and cryo-electron tomography of nanoparticles. Wiley Interdisciplinary Reviews: Nanomedicine and Nanobiotechnology. 9, (2017).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoExplorar más artículos
This article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados