Un abonnement à JoVE est nécessaire pour voir ce contenu. Connectez-vous ou commencez votre essai gratuit.
Method Article
Appliquer l’imagerie cellulaire en direct et la tomographie cryo-électronique pour résoudre les caractéristiques spatiotemporales du système de sécrétion Legionella pneumophila Dot/Icm
Dans cet article
Résumé
L’imagerie des cellules bactériennes est une approche émergente de biologie des systèmes axée sur la définition de processus statiques et dynamiques qui dictent la fonction de grandes machines macromoléculaires. Ici, l’intégration de l’imagerie cellulaire vivante quantitative et de la tomographie cryo-électronique est utilisée pour étudier l’architecture et les fonctions du système de sécrétion de type IV de Legionella pneumophila.
Résumé
Le système de sécrétion Dot/Icm de Legionella pneumophila est une nanomachine complexe du système de sécrétion DE type IV (T4SS) qui localise au pôle bactérien et favorise la livraison de protéines et de substrats d’ADN aux cellules cibles, un processus nécessitant généralement un contact direct de cellule à cellule. Nous avons récemment résolu la structure de l’appareil Dot/Icm par la tomographie cryo-électronique (cryo-ET) et avons montré qu’il forme un canal de cellule qui s’étend sur une enveloppe cellulaire qui se connecte à un complexe cytoplasmique. L’application de deux approches complémentaires qui préservent la structure indigène du spécimen, la microscopie fluorescente dans les cellules vivantes et cryo-ET, permet la visualisation in situ des protéines et l’assimilation de la stoichiométrie et le moment de production de chaque composant de machine par rapport à d’autres sous-unités Dot/Icm. Pour étudier les exigences en matière de positionnement polaire et pour caractériser les caractéristiques dynamiques associées à la biogenèse de la machine T4SS, nous avons fusionné un gène codant des protéines fluorescentes vertes superpliantes aux gènes Dot/Icm ATPase à leurs positions indigènes sur le chromosome. La méthode suivante intègre la microscopie quantitative de fluorescence des cellules vivantes et cryo-ET pour quantifier la localisation polaire, la dynamique et la structure de ces protéines dans les cellules bactériennes intactes. L’application de ces approches pour étudier le Legionella pneumophila T4SS est utile pour caractériser la fonction du système Dot/Icm et peut être adaptée pour étudier une grande variété d’agents pathogènes bactériens qui utilisent le T4SS ou d’autres types de complexes de sécrétion bactérienne.
Introduction
Legionella pneumophila (L. pneumophila), l’agent éétiologique de la maladie des légionnaires, habite les réservoirs d’eau douce, où les bactéries se propagent en infectant et en se reproduisant dans les protozoaires aquatiques nageant librement. L. pneumophila provoque des éclosions de maladie chez l’homme lorsque l’inhalation de bactéries aérosolisées provenant de sources d’eau potable se produit. Dans les cellules infectées, la subversion des voies hôtes permet à L. pneumophila de retarder la maturation endocytique du vacuole dans lequel elle réside et de promouvoir la biogenèse d’un compartiment cellulaire qui soutient la réplication bactérienne. Ce processus est piloté par un système spécialisé de sécrétion de type bactérien IVB (T4BSS) connu sous le nom de Dot/Icm et son répertoire de plus de 300 protéines « effecteurs » qui sont translocalisés dans le cytosol hôte pendant l’infection pour faciliter la manipulation des fonctions cellulaires1,2,3,4,5. Les mutants dépourvus d’un appareil fonctionnel Dot/Icm ne parviennent pas à délivrer des effecteurs dans le cytosol hôte, sont défectueux pour la réplication intracellulaire, et sont avirulents dans les modèles animaux de la maladie6,7.
De nombreuses espèces bactériennes ont mis au point des machines multicomposantes extrêmement complexes et dynamiques qui sont nécessaires pour les processus d’infection. D’autres T4BSS comme le système Dot/Icm sont également essentiels pour la réplication intracellulaire d’agents pathogènes bactériens tels que Coxiella burnetii et Rickettsiella grylli. Bien que les T4BSS soient évolutivement liés aux systèmes IVA de type prototypique, qui servent de médiateur au transfert d’ADN et peuvent fournir un répertoire limité de protéines effectrices, le système Dot/Icm a presque deux fois plus de composants de machine et offre une grande variété d’effecteurs. Vraisemblablement, cette expansion du nombre de composants a permis à l’appareil Dot/Icm d’accueillir et d’intégrer facilement de nouveaux effecteurs8,9.
Nous avons récemment utilisé la tomographie cryo-électronique (cryo-ET) pour résoudre la structure de l’appareil Dot/Icm in situ et avons montré qu’il forme un canal de cellule qui s’étend sur une enveloppe cellulaire qui se connecte à un complexe cytoplasmique. Une analyse plus approfondie a révélé que le cytosolic ATPase DotB s’associe au système Dot/Icm au pôle cellulaire L. pneumophila par des interactions avec le cytosolic ATPase DotO. Nous avons découvert que DotB affiche un mouvement cytosolique dans la plupart des cellules bactériennes, ce qui indique que cet ATPase est présent dans une population cytosolique dynamique, mais s’associe également avec les complexes polar Dot/Icm. En outre, DotO forme un assemblage hexamerique de dimers DotO associés au complexe interne de membrane, et un hexamer DotB se joint à la base de ce complexe cytoplasmique. L’assemblage du complexe énergétique DotB-DotO crée un canal cytoplasmique qui dirige la translocation des substrats à travers le T4SS(figure 1)10.
Malgré ces progrès récents, on sait peu de choses sur le fonctionnement du système Dot/Icm et sur la façon dont chaque protéine s’assemble pour former un appareil actif8. La découverte des circuits réglementaires du T4SS Dot/Icm est fondamentale pour comprendre les mécanismes moléculaires des interactions hôte-pathogène. Par conséquent, nous discutons de la façon d’utiliser la microscopie cellulaire vivante et cryo-ET pour détecter et caractériser les composants essentiels du système L. pneumophila Dot/Icm qui sont étiquetés avec le super-folder GFP (sfGFP). À l’aide de la microscopie quantitative de fluorescence, la localisation polaire de DotB sera définie dans un fond de type sauvage ou lorsque le système de type IV est supprimé. La microscopie en accéléré sera utilisée pour quantifier les différences de localisation et de dynamique entre les ATPases cytosoliques Dot/Icm.
L’application combinée de deux approches complémentaires telles que l’imagerie en direct et le cryo-ET offre un avantage par rapport à d’autres systèmes in vitro. Les deux méthodes sont effectuées dans des cellules intactes et préservent l’environnement naturel du T4BSS, minimisant ainsi la perturbation de la structure indigène pendant la préparation de l’échantillon. Parce que la surexpression des protéines peut altérer la stoichiométrie de l’appareil de sécrétion, les fusions sfGFP sont retournées par échange allélique au chromosome Legionella de sorte que chaque fusion est codée en une seule copie et l’expression est conduite par le promoteur endogène. La visualisation des fusions codées chromosomalement permet de quantifier le niveau exact de protéines exprimés à un moment défini. Cryo-ET a également de nombreux avantages pour déterminer la structure des systèmes de sécrétion. L’avantage le plus notable est que les échantillons cryo-ET sont composés de cellules intactes congelées qui préservent les complexes indigènes dans le contexte de l’architecture des cellules bactériennes. Par conséquent, le cryo-ET peut être préférable aux approches de purification biochimique, qui extraient des complexes membranaires et peuvent dépouiller les protéines périphériques de l’appareil de base ou modifier la structure globale. En outre, le marquage d’une protéine d’intérêt avec une protéine encombrante comme le sfGFP ajoute une masse qui est détectable par cryo-ET et peut aider à cartographier les différents sous-modèles de l’appareil Dot/Icm sur la structure obtenue par cryo-ET.
Cette approche est un outil puissant pour découvrir des informations structurelles sur les complexes multimoléculaires qui s’assemblent dans la membrane cellulaire bactérienne. L’interprétation des structures élucidées à l’aide de ces techniques aidera le terrain à comprendre comment fonctionnent les composants T4BSS, pourquoi tant de composants sont nécessaires pour fonctionner, comment les composants interagissent dans le plus grand complexe, et quelles fonctions ces sous-assemblages effectuer.
Protocole
REMARQUE : Toutes les procédures impliquant la croissance, la manipulation et l’imagerie de L. pneumophila doivent être effectuées dans un laboratoire biologique de niveau de sécurité 2 conformément aux directives locales.
1. Insertion de sfGFP dans le chromosome L. pneumophila à l’aide de la stratégie d’échange allégique et de sélection double (figure 2, figure 3)
- Clonez dans le vecteur de remplacement génétique pSR47S11 la séquence suivante : les 1 000 bp en amont du site d’intérêt, puis la séquence sfGFP, puis les 1 000 pb en aval du site d’intérêt(figure 2). La séquence sfGFP doit être placée dans le cadre du N-terminus ou du C-terminus se termine par un lien contenant quatre à huit acides aminés. Transformez le vecteur résultant en E. coli DH5-pir. Plus tard, strie L. pneumophila (le receveur) pour les colonies individuelles sur l’extrait de charbon de bois-levure (CYE) agar12 contenant 100 g/mL streptomycine et se développent pendant 5 jours à 37 oC(figure 3).
- Streak L. pneumophila sur CYE-agar-streptomycin et se développer pendant 2 jours à 37 oC (patch lourd)10. Streak E. coli DH5MD transformé avec pRK600 aide plasmid (aide)13 sur LB agar contenant 25 g/mL chloramphenicol. Streak l’E. coli DH5 -pir (donneur) sur l’agar LB contenant 50 g/mL kanamycine.
- Effectuer l’accouplement triparental : incuber une colonie de l’aide, une colonie du donneur, et le receveur en superposant les plaques des trois souches sur une plaque d’agar CYE sans sélection et incuber pendant 4 à 8 h à 37 oC. Au fur et à mesure que les contrôles négatifs couvent un mélange de souches aide-bénéficiaire et un mélange de souches donneurs-bénéficiaires pendant les mêmes périodes.
- Resuspendez les réactions d’accouplement dans 500 l’utérus de ddH2O. Plaque 20 L et 50 l des réactions sur CYE agar contenant 100 g/mL streptomycine et 10 g/mL kanamycin et poussent pendant 5 jours à 37 oC. Streak quatre des clones résultant sur L’agar CYE contenant 100 g/mL streptomycine et se développent pendant 5 jours à 37 oC.
- Streak 16 clones sur CYE agar contenant 5% de saccharose et 100 g/mL streptomycine et se développent pendant 5 jours à 37 oC. Ensuite, stries 32 de ces clones sur l’agar CYE contenant 100 g/mL streptomycine et sur l’agar CYE contenant 100 g/mL streptomycine et 10 g/mL kanamycine et poussent pendant 5 jours à 37 oC.
2. Isolation des clones qui ont intégré le sfGFP dans le chromosome de L. pneumophila
- Clones de streak qui étaient sensibles à la kanamycine sur les plaques de CYE-agar-streptomycine et confirment l’insertion du sfGFP dans le chromosome avec la colonie PCR. Utilisez des amorces complémentaires au gène sfGFP et à la région chromosomique d’intérêt pour amplifier la jonction d’insertion.
- Mélanger 0,5 L de chacune des solutions d’amorce de 10 M et une colonie à un volume final de 12,5 L et à la dénature pendant 10 min à 95 oC. Laisser refroidir sur la glace pendant 10 min, ajouter 12,5 L de la solution de mélange principal PCR 2x et effectuer une analyse PCR.
- Cultivez de lourdes parcelles des colonies isolées sur des plaques CYE-agar-streptomycine pendant 2 jours à 37 oC. Examinez les niveaux d’expression et la stabilité des fusions de sfGFP en immunoblotting avec un anticorps anti-GFP.
3. Imagerie cellulaire en direct de L. pneumophila avec des composants fluorescents Tagged Dot/Icm
- Préparation de tampons agaroses
- Faire environ 30 ml d’une solution d’agarose à faible fondre de 1 % dans l’eau. Cuire au micro-ondes dans un flacon de verre pendant environ 90 s, tourbillonnant de temps en temps, jusqu’à ce que l’agarose soit complètement dissoute.
- Placez deux 22 x 22 x 0,15 mm3 toboggans en verre sur le bord d’une glissière en verre de 25 x 75 x 1,1 mm3, l’une sur l’autre. Empilez deux autres 22 x 22 x 0,15 mm3 toboggans en verre sur l’autre bord.
- Pipette environ 1 mL de l’agarose fondue dans la glissière centrale entre les deux toboggans en verre supérieur, puis placez une autre glissade de 25 x 75 x 1,1 mm3 sur le dessus de l’agarose fondue. Essayez d’éviter la formation de bulles d’air. Refroidir les lames à 4 oC pendant 15 min.
- À l’aide d’un scalpel ou d’une lame de rasoir, coupez délicatement le tampon en petits carrés, soit 5 x 5 mm2. Fixez un adhésif recôtit à double face 17 x 28 x 0,25 mm3 sur une glissière en verre de 25 x 75 x 1,1 mm3 et placez plusieurs garnitures sur la glissière.
- Acquisition d’images
REMARQUE : Les étapes suivantes sont décrites pour un microscope qui est sous le contrôle du SlideBook 6.0 et équipé d’enlumineurs à l’état solide, d’une caméra monochrome CCD et d’une lentille objective 100x (1,4 ouverture numérique). Si nécessaire, utilisez d’autres appareils de microscopie avec des configurations matérielles et logicielles appropriées qui peuvent être personnalisées en fonction des paramètres du protocole.- Dissoudre une lourde parcelle de L. pneumophila dans 1 ml de ddH2O, vortex et pipet 2-3 'L de la dilution sur les garnitures. Placer un 50 x 24 x 0,15 mm3 couverture doucement sur le cadre adhésif.
- Dans la fenêtre de capture ajuster le ND à 180. Ajustez le binning à 2/2 et utilisez le canal de 488 nm pour exposer l’échantillon entre 500 et 1 000 ms. Valider la spécificité du signal de fluorescence en imagerie L. pneumophila non étiqueté avec les mêmes paramètres(figure 4).
4. Quantification de la localisation polaire et de la dynamique des composants Dot/Icm
REMARQUE : Les étapes suivantes sont conçues pour les images de 0,129 m par pixel qui ont été acquises avec 2 x 2 binning.
- Quantification de la polarité pour les protéines de fusion sfGFP (figure 5)
- Ajustez le contraste d’image de sorte que les bactéries sont clairement visibles. Utilisez l’outil de la région pour placer un rectangle de 0,25 x 1,3 m2 à partir du pôle et s’étendant dans le cytoplasme. Le rectangle doit rester précisément à l’intérieur des frontières bactériennes.
- Marquez au moins 200 bactéries et utilisez le bouton Région pour masquer pour créer des masques dans les régions d’intérêt. Sous les statistiques de masque et la portée de masque, choisissez l’objet. Puis, sous les caractéristiques et l’intensité, marquez l’intensité moyenne et la variance.
- Exporter les données et calculer les scores de polarité de chaque bactérie comme le rapport entre l’écart à l’intensité moyenne.
- Pour les applications à haut débit, utilisez un objectif de phase et une configuration de condensateur appropriée pour acquérir des images avec les canaux de phase et 488 nm. Assurez-vous de choisir les champs de vision où les bactéries sont entièrement séparées.
- Ajuster le contraste du canal de phase de l’image à un niveau où les bactéries sont clairement visibles. Ouvrez l’image double canal, lancez la fenêtre Create Segment Mask et changez le canal en phase.
- Ajustez un seuil approprié et supprimez les petits objets avec le bouton Définir les objets. Sous le masque de raffinage, choisissez des objets supprimer les bords et plus tard séparer les masques de bactéries qui sont adjacents les uns aux autres.
- Calculez les scores de polarité du signal dans le canal 488 pour chaque cellule comme cela a été décrit précédemment dans les étapes 4.1.2-4.1.3.
- Quantification de la dynamique des protéines de fusion sfGFP(figure 6)
REMARQUE : Suivez les instructions de la section 3 pour préparer un échantillon pour l’acquisition d’images. La dynamique est définie comme des changements d’intensité au fil du temps et les étapes suivantes sont conçues pour de courtes périodes d’imagerie (c.-à-d. plusieurs minutes). Ajouter à la garniture les suppléments appropriés si de plus longues périodes d’imagerie sont désirées.- Dans la fenêtre Image Capture, marquez Timelapse,entrez le temps des intervalles dans la boîte d’intervalle, et entrez 2 dans la boîte de points de temps. Acquérir deux images successives de L. pneumophila exprimant la protéine fluorescente d’intérêt.
- Ajuster le contraste d’image jusqu’à ce que les cellules soient clairement visibles. Suivez la figure 6A et les descriptions ci-dessous pour placer trois masques différents. Utilisez l’outil de la région pour placer un carré de 0,25 x 0,25 m au milieu d’au moins 400 cellules.
- Utilisez le bouton Région pour masquer pour créer un masque (masque 1) des carrés d’intérêt. Créez un nouveau masque vide (masque 2) et utilisez l’outil pixel ou l’outil polygone pour marquer l’ensemble de la zone cellulaire d’au moins 25 cellules aléatoires, qui seront utilisées pour calculer le blanchiment de fluorescence. Créez un nouveau masque vide (masque 3) et utilisez le grand outil de brosse pour marquer les zones entre les cellules, qui seront utilisées pour la soustraction de fond.
- Sous les statistiques de masque et la portée de masque,choisissez l’objet pour le masque 1 et masque 2. Ensuite, sous Les caractéristiques et l’intensité, choisissez l’intensité moyenne et exportez les données des deux masques. Pour le masque 3, exportez l’intensité moyenne de l’ensemble du masque.
- Calculer le changement d’intensité de fluorescence pour chaque objet dans le masque 1 à l’aide des formules suivantes :

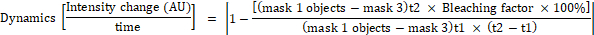
où le masque 1 est un carré de 0,25 m à 0,25 m placé au centre cellulaire, le masque 2 couvre toute la cellule, le masque 3 est l’arrière-plan entre les cellules, t1 est l’intensité moyenne dans le premier point de temps, et t2 est l’intensité moyenne dans le deuxième point de temps.
5. Détection de la densité de masse sfGFP avec Cryo-ET
- Préparation des échantillons, collecte de données et reconstruction
- Cultivez une lourde parcelle de L. pneumophila exprimant une protéine Point/Icm étiquetée fluorescente sur des plaques CYE-agar-streptomycine pendant 48 heures à 37 oC. Résuspendez les cellules dans ddH2O à600 OD 0,7. Ajouter 5 L de particules d’or colloïdales (BSA Tracer, 10 nm) à 20 l de la suspension cellulaire.
- Pipette 5 L du mélange cellulaire sur le réseau de carbone troué fraîchement rougeoyé (R 2/1 sur le maillage Cu 200) et laisser reposer 1 min. Blot avec du papier filtre et congeler dans l’éthane liquide à l’aide d’un appareil de piston gravitationnel tel que décrit précédemment14,15.
- Imagez les spécimens hydratés congelés avec un microscope électronique de transmission de 300 kV équipé d’un pistolet à émission de champ, d’un filtre d’énergie, d’une plaque de phase Volta et d’un dispositif de détection directe. Recueillir des séries d’inclinaison à axe unique à 26 000x et 42 000x grossissements, qui se traduisent par des pixels de taille au niveau du spécimen de 5,4 euros/pixel ou 3,4 euros/pixel, respectivement.
- Utilisez le paquet tomographique SerialEM pour collecter des piles d’images à 0 m defocus, avec une gamme d’angles d’inclinaison entre -60 et 60 euros avec une augmentation de 3 degrés et une dose cumulative de 60 e-/2,16. Alignez les images de films fractionnées de dose dans chaque pile à l’aide de MotionCor217. Assembler les piles corrigées à la dérive à l’aide de TOMOAUTO14.
- Alignez les piles corrigées de la dérive par l’alignement IMOD dépendant des marqueurs18. Reconstruire les tomogrammes avec la méthode SIRT19 pour les segmentations et l’analyse directe de l’image et la méthode WBP20.
- Analyse de sous-tomogrammes des échantillons de fusions de sfGFP
- Utilisez le paquet tomographique I3 (0.9.9.3) pour l’analyse de sous-biogramme14,21,22.
REMARQUE : L’alignement procède de façon itérative, à chaque itération composée de trois parties dans lesquelles des références et des masques de classification sont générés, des sous-biogrammes sont alignés et classifiés, et les moyennes de classe sont alignées les unes aux autres. - Utilisez 4 x 4 x 4 sous-tomgrammes binned pour un alignement initial. Fusionner les particules qui appartiennent à des moyennes de classe et montrer la densité d’électrons correspondant à sfGFP. Après avoir trié des particules avec des fusions sfGFP, utilisez 2 sous-bigrammes binned 2 x 2 pour un alignement ciblé d’une région d’intérêt (comme le complexe ATPase cytoplasmique Dot/Icm) pour obtenir une structure haute résolution.
- Utilisez le paquet tomographique I3 (0.9.9.3) pour l’analyse de sous-biogramme14,21,22.
Résultats
La recombinaison homologue avec une double sélection en deux étapes a été utilisée pour construire l’insertion définie de sfGFP. Dans la première étape, l’accouplement triparental a été effectué, où le pRK600 plasmide conjugal (un plasmide IncP) de la souche d’aide E. coli MT616 a été mobilisé à la souche de donneur E. coli avec le vecteur suicide pSR47S contenant le gène SfGFP flanqué des deux régions homologues, l’origine du transfert oriT et le Bacillus subtilis co...
Discussion
L’élucidation des fonctions des systèmes de sécrétion bactérienne est la clé d’une compréhension complète des interactions hôte-pathogène. Les systèmes de sécrétion sont des machines complexes qui peuvent injecter des protéines effecteurs dans les cellules hôtes, et dans certains cas promouvoir l’établissement d’une niche subcellulaire qui soutient la réplication bactérienne. La méthode ci-dessus fournit de nouveaux outils importants pour étudier le système de sécrétion Dot/Icm de l’agent...
Déclarations de divulgation
Les auteurs n’ont rien à divulguer.
Remerciements
D.C. et C.R.R. ont été soutenus par les NIH (R37AI041699 et R21AI130671). D.P., B.H., et J.L. ont été soutenus par les National Institutes of Health (R01AI087946 et R01GM107629).
matériels
| Name | Company | Catalog Number | Comments |
| 10 nm colloidal gold particles | Aurion | 25486 | |
| 100x Plan Apo objective (1.4 NA) | Nikon | ||
| ACES | Sigma-Aldrich | A9758 | |
| Activated charcoal | Sigma-Aldrich | C5510 | |
| Agaroze GPG/LMP, low melt | American bioanalytical | AB00981 | |
| Bacto dehydrated agar | BD | 214010 | |
| CoolSNAP EZ 20 MHz digital monochrome camera | Photometrics | ||
| Gene Frame, 1.7x2.8 cm, 125 µL | Fisher Scientific | AB-0578 | |
| Holey Carbon grid R 2/1 Cu 200 mesh | Quantifoil | Q225-CR1 | |
| Iron(III) nitrate nonahydrate | Sigma-Aldrich | 216828 | |
| K2 Summit camera for cryo-EM | GATAN | ||
| L-Cysteine | Sigma-Aldrich | C7352 | |
| Microscope cover slides 22x22 mm | Fisher Scientific | 12-542B | |
| Microscope cover slides 24x50 mm | Fisher Scientific | 12-545K | |
| Microscope slides 25x75x1 mm | Globe Scientific | 1380 | |
| SlideBook 6.0 | Intelligent Imaging Innovations | ||
| Spectra X light engine | Lumencor | ||
| Taq 2X Master Mix | New England BioLabs | M0270 | |
| Titan Krios | Thermo Fisher Scientific | ||
| Yeast Extract | BD | 212750 |
Références
- Franco, I. S., Shuman, H. A., Charpentier, X. The perplexing functions and surprising origins of Legionella pneumophila type IV secretion effectors. Cellular Microbiology. 11, 1435-1443 (2009).
- Burstein, D., et al. Genome-scale identification of Legionella pneumophila effectors using a machine learning approach. PLOS Pathogens. 5, 1000508 (2009).
- Ninio, S., Roy, C. R. Effector proteins translocated by Legionella pneumophila: strength in numbers. Trends in Microbiology. 15, 372-380 (2007).
- Vogel, J. P., Andrews, H. L., Wong, S. K., Isberg, R. R. Conjugative transfer by the virulence system of Legionella pneumophila. Science. 279, 873-876 (1998).
- Isberg, R. R., O'Connor, T. J., Heidtman, M. The Legionella pneumophila replication vacuole: making a cosy niche inside host cells. Nature Reviews Microbiology. 7, 13-24 (2009).
- Roy, C. R., Berger, K. H., Isberg, R. R. Legionella pneumophila DotA protein is required for early phagosome trafficking decisions that occur within min of bacterial uptake. Molecular Microbiology. 28, 663-674 (1998).
- Archer, K. A., Roy, C. R. MyD88-Dependent Responses Involving Toll-Like Receptor 2 Are Important for Protection and Clearance of Legionella pneumophila in a Mouse Model of Legionnaires' Disease. Infection and Immunity. 74, 3325-3333 (2006).
- Nagai, H., Kubori, T. Type IVB Secretion Systems of Legionella and Other Gram-Negative Bacteria. Frontiers in Microbiology. 2, 136 (2011).
- Kubori, T., Nagai, H. The Type IVB secretion system: an enigmatic chimera. Current Opinion in Microbiology. 29, 22-29 (2016).
- Chetrit, D., Hu, B., Christie, P. J., Roy, C. R., Liu, J. A unique cytoplasmic ATPase complex defines the Legionella pneumophila type IV secretion channel. Nature Microbiology. 3, 678-686 (2018).
- Merriam, J. J., Mathur, R., Maxfield-Boumil, R., Isberg, R. R. Analysis of the Legionella pneumophila fliI gene: intracellular growth of a defined mutant defective for flagellum biosynthesis. Infection and Immunity. 65, 2497-2501 (1997).
- Feeley, J. C., et al. Charcoal-yeast extract agar: primary isolation medium for Legionella pneumophila. Journal of Clinical Microbiology. 10, 437-441 (1979).
- Andrews, H. L., Vogel, J. P., Isberg, R. R. Identification of linked Legionella pneumophila genes essential for intracellular growth and evasion of the endocytic pathway. Infection and Immunity. 66, 950-958 (1998).
- Morado, D. R., Hu, B., Liu, J. Using Tomoauto: A Protocol for High-throughput Automated Cryo-electron Tomography. Journal of Visualized Experiments. (107), e53608 (2016).
- Hu, B., Lara-Tejero, M., Kong, Q., Galan, J. E., Liu, J. In Situ Molecular Architecture of the Salmonella Type III Secretion Machine. Cell. 168, 1065-1074 (2017).
- Mastronarde, D. N. Automated electron microscope tomography using robust prediction of specimen movements. Journal of Structural Biology. 152, 36-51 (2005).
- Zheng, S. Q., et al. MotionCor2: anisotropic correction of beam-induced motion for improved cryo-electron microscopy. Nature Methods. 14, 331-332 (2017).
- Kremer, J. R., Mastronarde, D. N., McIntosh, J. R. Computer visualization of three-dimensional image data using IMOD. Journal of Structural Biology. 116, 71-76 (1996).
- Gilbert, P. Iterative methods for the three-dimensional reconstruction of an object from projections. Journal of Theoretical Biology. 36, 105-117 (1972).
- Radermacher, M. Weighted Back-projection Methods. Electron Tomography. , 245-273 (2007).
- Winkler, H., et al. Tomographic subvolume alignment and subvolume classification applied to myosin V and SIV envelope spikes. Journal of Structural Biology. 165, 64-77 (2009).
- Winkler, H., Taylor, K. A. Accurate marker-free alignment with simultaneous geometry determination and reconstruction of tilt series in electron tomography. Ultramicroscopy. 106, 240-254 (2006).
- Prevost, M. S., Waksman, G. X-ray crystal structures of the type IVb secretion system DotB ATPases. Protein Science. 27, 1464-1475 (2018).
- Miklos, G. L., Rubin, G. M. The role of the genome project in determining gene function: insights from model organisms. Cell. 86, 521-529 (1996).
- Reyrat, J. M., Pelicic, V., Gicquel, B., Rappuoli, R. Counterselectable markers: untapped tools for bacterial genetics and pathogenesis. Infection and Immunity. 66, 4011-4017 (1998).
- Yu, J. Single-Molecule Studies in Live Cells. Annual Review of Physical Chemistry. 67, 565-585 (2016).
- Stewart, P. L. Cryo-electron microscopy and cryo-electron tomography of nanoparticles. Wiley Interdisciplinary Reviews: Nanomedicine and Nanobiotechnology. 9, (2017).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationExplorer plus d’articles
This article has been published
Video Coming Soon