Method Article
電子顕微鏡によるピソメーク精度原子位置追跡
要約
この研究は、原子分解能透過電子顕微鏡イメージングにおける原子位置追跡のワークフローを提示する。このワークフローは、オープンソースのMatlabアプリ(EASY-STEM)を使用して実行されます。
要約
現代の収差補正走査型透過電子顕微鏡(AC-STEM)は、サブオングストローム分解能を有する原子カラムの直接可視化に成功しました。この大きな進歩により、高度な画像の定量と分析は未だ初期段階にあります。本研究では、原子分解能走査透過電子顕微鏡(STEM)画像の計測の完全経路を提示する。これには、(1)高品質のSTEM画像を取得するためのヒントが含まれます。(2) 測定精度を高めるための、非ノイズおよびドリフト補正(3) 初期原子位置の取得(4) 単位セルベクターに基づいて原子のインデックスを作成する。(5)原子列を2D-Gaussianシングルピークフィッティングまたは(6)少し重なっている原子列のためのマルチピークフィッティングルーチンのいずれかで原子列位置を定量化する。(7)結晶構造内または格子周期性が破壊される欠陥/インターフェースにおける格子歪み/歪みの定量化。(8)解析を視覚化して提示するいくつかの一般的な方法。
さらに、グラフィカルユーザーインターフェース(GUI)を備えたシンプルな自己開発無料MATLABアプリ(EASY-STEM)が提示されます。GUIは、専用の分析コードやソフトウェアを作成することなく、STEMイメージの分析を支援できます。ここに示す高度なデータ解析方法は、欠陥緩和、局所構造歪み、局所相変換、非中心対称性の局所的な定量化に適用できます。
概要
現代の走査型透過電子顕微鏡(STEM)における球面収差補正の開発により、マイクロスコピストは、サブオングストロームサイズの電子ビーム1,2で結晶を探査することを可能にした。これにより、重い要素と軽い要素3、4の両方に対して解釈可能な原子分解能画像を持つ多種多様な結晶中の個々の原子カラムのイメージングが可能になった。画素化直接電子検出器およびデータ解析アルゴリズムの最近の進展により、ptychographyなどの位相再構成イメージング技術が可能になり、空間分解能のさらなる改善が午後30時頃、6、7となっている。さらに、STEM断層撮影における最近の進歩は、単一ナノ粒子8の三次元原子分解再構成を可能にした。電子顕微鏡は、高精度と部位特異性の両方で材料の構造特性を定量化するための非常に強力なツールとなっています。
超高解像度のSTEM画像をデータ入力として、構造歪みの直接測定を行い、原子スケール9,10で結晶から物理的情報を抽出した。例えば、WS2単層内のMoドーパントと単一のS空孔との間の欠陥結合を、原子位置を測定し、次いで投影結合長11を計算することによって直接可視化した。さらに、単層WS2における結晶界面上の測定は、単層WS2における合体粒子境界など、局所原子配置12を示すことができる。LiNbO3の強誘電体ドメイン壁に対して行われた界面解析により、ドメイン壁がイジング状態とニール状態13の組み合わせであることが明らかになった。別の例は、SrTiO 3-PbTiO3超格子で達成された極渦構造の視覚化であり、ストロンチウムおよび鉛カラム位置14に対するチタン原子カラム変位の計算によって達成される。最後に、非局所原理成分分析15による画像の脱明などのコンピュータビジョンアルゴリズムの進歩により、リチャードソンとルーシーの沈降16、非線形登録17によるドリフト補正、およびディープラーニングによるパターン認識により、サブピソメータ精度18に対する測定の精度が著しく強化されている。そのような例の1つは、シグナル対ノイズ比を高めるために、複数の高速スキャン極低温-STEM画像のアライメントと画像登録です。続いて、Fourierマスキング技術を適用し、周期格子歪み19を直接可視化して結晶中の電荷密度波を解析した。信じられないほどの収差補正されたSTEM計測器は、世界中の研究者にますますアクセス可能ですが、高度なデータ分析手順と方法は依然として珍しく、データ分析の経験がない人にとっては大きな障壁です。
今回の研究では、原子分解能のSTEM画像の計測の完全な経路を紹介しています。このプロセスには、まず収差補正顕微鏡でSTEM画像を取得し、続いて取得後の脱ノイズ/ドリフト補正を行い、測定精度を向上させることが含まれます。次に、原子柱20,21をわずかに重ね合わせるための2D-Gaussianシングルピークフィッティングまたはマルチピークフィッティングルーチンのいずれかで、原子列位置を明確に解決し、正確に定量化する既存の方法についてさらに議論する。最後に、このチュートリアルでは、結晶構造内の格子歪み/歪みの定量方法、または格子周期が乱される欠陥/インタフェースでの方法について説明します。また、専用の分析コードやソフトウェアを書くことなく、STEM画像の分析に役立つグラフィカルユーザーインターフェース(GUI)を備えた簡単な自己開発無料MATLABアプリ(EASY-STEM)を紹介します。ここに示す高度なデータ解析方法は、欠陥緩和、局所構造歪み、局所相変換、非中心対称性の局所的な定量化に適用できます。
プロトコル
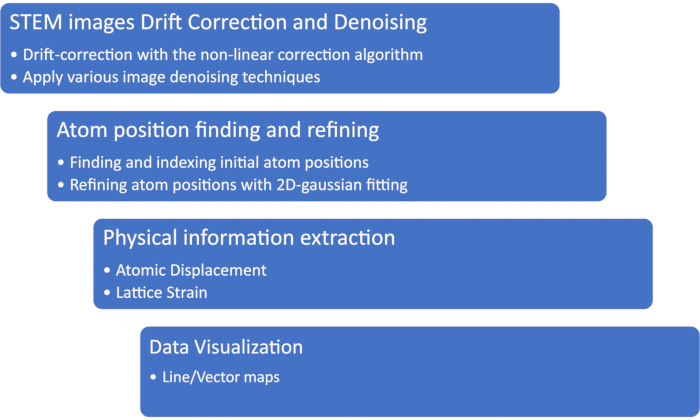
注: 図 1 のフローチャートは、アトミック位置の定量の一般的な手順を示しています。
1. STEM画像のドリフト補正とデノイズ
- 高品質の環状暗視野(ADF)/環状明視野(ABF)のSTEM画像を取得します。
注: 入力データの品質は、データ分析の精度を確保するための鍵であるため、良い画像データを取得するためのヒントをいくつか使用してプロトコルを開始します。- 高品質の TEM サンプルを確認します。サンプルの品質は非常に重要です。イメージングには、ビーム損傷のない薄くてクリーンなTEMサンプルを使用します。サンプル汚染の原因となる可能性がありますので、取り扱いおよびローディング中にサンプルに触れないようにしてください。
- 挿入前にサンプルをクリーニングします (可能な場合)。プラズマクリーナーを使用してサンプルをきれいにし、真空中で焼く、または顕微鏡に挿入した後に電子ビームを広げて低倍率でサンプルに関心のある領域を照射する(「ビームシャワー」)。画像撮影時に損傷や汚染された領域を避けてください。
- 顕微鏡を合わせ、収差補正器を調整してレンズの収差をできるだけ最小限に抑えます。標準的なサンプルでいくつかのSTEM画像を取得して解像度をテストし、空間分解能が特定の結晶構造を解決し、画像内の収差をさらに微調整できることを確認します。
- 光軸が結晶の特定のゾーン軸に合うようにサンプルを傾けます。特定の結晶については、必要なゾーン軸から観測を行います。たとえば、測定のために、強誘電体結晶のドメイン壁の平面に表示軸を合わせます。
- 電子線量を最適化しながら、電子線の損傷とイメージング中のサンプルドリフトを制限します。試料が電子線下で安定していて、取得中にドリフトや損傷を示さない場合は、より高い電子線量を試すか、同じ領域の複数の画像を取得して信号対雑音比を高めることができる可能性があります。ここでの目標は、ビーム損傷や画像アーティファクトのない、より高い信号対雑音比を持つものを持つ。
- 異なるスキャン方向のSTEM画像を取得し、取得時の潜在的なドリフトを修正します。まず、画像を取得し、スキャン方向を90°回転させた直後に同じ領域から2番目のものを取ります。
- スキャンの方向以外は、同じイメージング条件を使用して画像を撮ります。このステップの目的は、最近開発されたドリフト補正アルゴリズムに回転した画像を供給することです 17.
注: また、アルゴリズムに(任意の角度で)より変化するスキャン方向を持つ2つ以上の画像を入力することもできます。しかし、同じ領域を連続してスキャンすると、その領域で格子損傷やドリフトが発生する可能性があります。さらに、スキャン方向と低インデックスラティス平面は、互いに平行または垂直な方向を維持せず、代わりに斜めの角度を維持することをお勧めします。スキャン方向が特定の水平または垂直フィーチャ(ラティス 平面、インターフェイスなど)と一致する場合、強い垂直方向または横方向に変化するフィーチャの方向に沿ったドリフトは、イメージの登録中にアーティファクトを引き起こす可能性があります。
- スキャンの方向以外は、同じイメージング条件を使用して画像を撮ります。このステップの目的は、最近開発されたドリフト補正アルゴリズムに回転した画像を供給することです 17.
- 非線形補正アルゴリズムでドリフト補正を実行します。
注:非線形ドリフト補正アルゴリズムは、C. Ophusららによって提案され、構築され、オープンソースMatlabコードは、論文で見つけることができます。スキャン方向が異なる複数の画像が補正アルゴリズムに送られ、このアルゴリズムはドリフト補正されたSTEM画像を出力します。ダウンロードしたコード パッケージには、実装の詳細でありながら簡単な手順が含まれています。プロセスのより詳細なアルゴリズムと説明は、元の論文で見つけることができます。 - さまざまな画像の破壊技術を適用します。
注: ドリフト補正後、画像のデノイズを実行して、今後の解析の精度を向上させます。一般的なデノイズ技法の一部を以下に示します。さらに、分析を支援するグラフィカルユーザーインターフェイスを備えたEASY-STEMという名前の無料のインタラクティブなMatlabアプリを紹介します。インターフェイスは 図 2に示され、対応するボタンにすべてのステップがラベル付けされています。
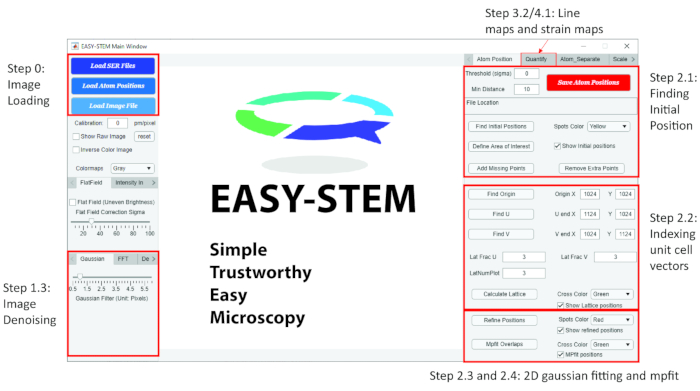
図2MatlabアプリEASY-STEMのグラフィカルユーザーインターフェース(GUI) プロトコルセクションで説明されているすべてのステップに従ってラベルが付けられます。 この図の大きなバージョンを表示するには、ここをクリックしてください。
- ガウス フィルタリングを適用します。EASY-STEMアプリで、左下に ガウシアン と呼ばれるタブを見つけます。スライダーを使用して、近くにあるピクセルの平均の数を選択します。スライダを動かして、ガウスフィルタを画像に適用します。
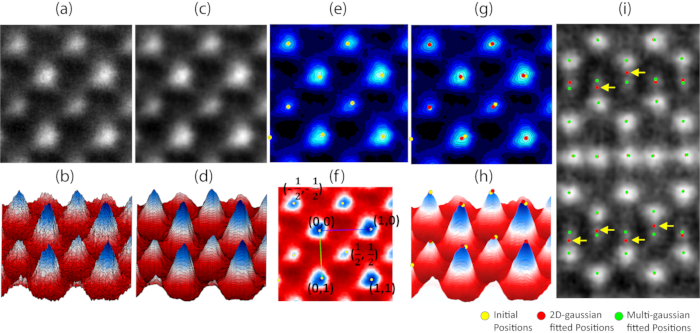
図3: 原子位置追跡の結果例(i) mp-fit アルゴリズムによる位置の精緻化の例。通常の2D-ガウスフィッティングとmpfitアルゴリズムの結果は、それぞれ赤と緑の円で示されています。黄色の矢印は、隣接する原子からの強度による通常の2Dガウスフィッティングの失敗を強調しています。() ドリフト補正されたADF-STEM画像は、ABO3ペロブスカイトの典型的な単位セルを示す。(b) での強度の 3D プロット (a)(c) ガウスフィルターでノイズ除去された同じ画像。(d)(c)の強度の 3D プロット(e) 初期原子位置(黄色の円)が重なった(c)の強度の輪郭図。(f)画像内の原子位置のインデックスを示す単位セルベクターインデックス化システムの例。(g) 初期原子位置(黄色の円)と細分化された原子位置(赤い円)を重ねた(c)の強度の輪郭プロット、および(h)黄色と赤の円で示された初期および洗練された原子位置を持つ強度の3Dプロット。この図の大きなバージョンを表示するには、ここをクリックしてください。
注: この手法では、画像の近くのピクセルの強度を平均するフィルターを使用します。ガウス フィルタリングの効果を 図 3a-dに示します。
- フーリエ フィルタリングを適用します。EASY-STEMアプリで、左下に FFT というタブを見つけてください。高周波ノイズを低減するために空間周波数を制限するスライダがあります。スライダーを動かして、フーリエフィルターを画像に適用します。
注: この手法では、画像の空間周波数を制限して、画像の高周波ノイズを除去します。 - リチャードソン・ルーシーの装飾を適用します。EASY-STEMアプリでは、左下に 「デコンボリューション 」と呼ばれるタブがあり、ブラインドデコンボリューションとリチャードソン・ルーシーのデコンボリューションの反復のための2つの入力ボックスがあります。値を変更し、ボタンをクリックしてこの削除アルゴリズムを適用します。
注: この手法は、点広がり関数を計算して画像内のノイズを効果的に除去するためのデコンボリューションアルゴリズムです。
2. 原子位置の検索と精製
- 初期アトミック位置を見つけます。
注:取得後の画像処理後、初期原子位置は、ADFまたはABF STEM画像の局所強度の最大値または最小値として抽出することができます。隣接するアトミック列間の最小距離を定義して、余分な位置を削除する必要があります。- 隣接するピーク間の距離を決定する入力ボックスの値を変更して、最小距離をピクセル単位で定義します。
- EASY-STEMアプリの 「初期位置を検索 」ボタンをクリックします。結果は 図 3eに示されています。
注: 多くの場合、余分な位置や欠落した位置は、単純なローカルの最大/分の検出アルゴリズムで観察されます。このように、手動修正モードは、さらに原子の位置を絞り込むためにEASY-STEMアプリで作成されます (追加欠損/余分なポイントを削除 ボタン).この機能により、マウス カーソルを使用して、初期位置の追加と削除が可能になります。
- ユニット・セル・ベクトル・ベースのシステムで初期アトミック位置を索引付けします。
- イメージ内の原点を定義します。EASY-STEMアプリで、[ 原点を検索 ]ボタンをクリックします。ボタンをクリックした後、ポインタを最初のアトミック位置のいずれかにドラッグして、原点として定義します。
- 2D 単位セルのユーと v ベクトルと単位セルの分数を定義します。
- [U/V を検索]ボタンをクリックし、ポインタをユニットセルの末尾までドラッグします。
- ラティスの小数部の値を定義するには、ラティスの値を変更して、ラティスの小数部の値を入力ボックスで 、LatFrac U と Lat Frac V 入力ボックスで値を変更します。
注: この値は、単位セル ベクトルに沿ったラティスの小数の値を決定します。例えば、ABO3 ペロブスカイト単位セルにおいて、単位セルは、2つの垂直単位セルベクター方向に沿って2つの半分に均等に分割することができる。したがって、各単位セルのベクトル方向に 2 つの分数があるため、単位セルの分数の値は、それぞれ 2 と 2 の方向と v 方向です。インデックス化の結果の例と、対応する i および v 単位のセル ベクターを 図 3fに示します。たとえば、図 3fでは、角の原子に (0, 0), (1, 0), (0, 1), (1, 1), (1, 1);そして、中央の原子を(1/2、1/2)としてインデックスします。このインデックス作成システムは、以下の手順で情報の抽出に役立ちます。 - [ ラティスの計算 ]ボタンをクリックして、すべての原子にインデックスを付けます。
- EASY-STEMアプリの 「位置を調整 」ボタンをクリックして、2D-ガウスフィッティングで原子位置を絞り込みます。
注: 初期原子位置を取得し、画像内の原子をインデックス化した後、各原子列の周囲に 2D ガウスのフィッティングを適用して、解析でサブピクセル レベルの精度を実現する必要があります。このアルゴリズムを使用すると、最初に画像内の初期原子位置の周りの領域をトリミングし、次に 2D ガウスピークをトリミングされた画像に収めます。その後、適合した2Dガウスピークの中心を洗練された原子位置として使用します。このアルゴリズムは、2D ガウス関数を画像の各原子列に適合させ、フィットしたピークの中心がフィッティング後にプロットされます。2D ガウス継ぎの結果を図 3g,hに示します。 - (オプション)EASY-STEMの 「mpfitオーバーラップ 」ボタンをクリックして、2D-ガウスのマルチピークフィッティング(mp-fit)で原子位置を調整します。
注: 隣接するアトミック列の強度が重なっている場合は、mp-fit アルゴリズムを使用してアトミックの位置を調整します。mp-fit アルゴリズムとその有効性は D. Mukherjeeら21. によって詳細に議論されています。EASY-STEMアプリは、このアルゴリズムを組み込み、重なり合う強度を持つ隣接原子を分離するために使用することができます。mp-fit の結果を図 3iに示します。 - [アトミックの位置を保存]ボタンをクリックして、結果を保存します。
注: アプリは、場所の保存とファイル名のユーザーを求めます。保存されたすべての結果は、変数「atom_pos」に含まれます。
3. 物理的情報の抽出
- 単位セル ベクトルのインデックス化と原子位置に基づいて、原子変位を測定します。
- 単位セルの中心を定義します。
注: たとえば、ABO3 ペロブスカイトユニットセルの場合、その[100]軸から見ると、ユニットセルの中心は4つのAサイト原子の平均位置として定義できます。第1の単位セルにおいて、これらのA部位原子は、以前に(0,0),(1,0),(0,1),(1,1),(1,1)としてインデックス付けされている。 - 変位原子の位置を見つける。
注:ABO3 ペロブスカイトユニットセルの場合、変位原子はB部位原子であり、以前は(1/2,1/2)とラベル付けされていました。 - 参照単位セルの中心と変位原子の位置を、画像内の完全な単位セルの位置を反復的に見つけます。
メモ:ユニットセルは、TEM画像の端付近で不完全である可能性があります。これらの単位セル内のアトミック位置は破棄されます。 - 次のコマンドを入力して、変位ベクトルを測定します。
d = pos(B) - 平均(pos(A))
- 単位セルの中心を定義します。
- ラティスの歪みを定量化します。
- 原子位置に基づいて、各ユニットセルから単位セルベクターを抽出します。
注: ベクトル行列 "C"を抽出します。これは、x 方向と y 方向の各単位セルのユーベクトルと v ベクトルからなる 2x2 行列です。 - 参照ベクトル「C0」を定義します。
注: C0 は、イメージの一部から平均単位セル ベクトルとして定義できます (推奨) または理論的に計算された単位セル ベクトル値。 - 次の式を使用して、2x2 変換行列 "T" を計算します。
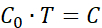 または
または 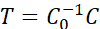 (1)
(1) - 歪み行列"D"を計算します。
D = T - I (2)
ここで"I"は単位行列です。 - 歪み "D" を対称歪み行列 "ε" と反対称回転行列 "ω" に分解します。
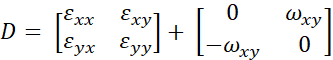 (3)
(3)
注: 歪み行列 "ε" と回転行列 "ω" は、次の式を使用して抽出できます。
ε =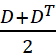 (4) および ω =
(4) および ω = 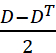 (5).
(5). - すべての単位セルのひずみを反復計算します。
- EASY-STEMアプリで、インターフェースの左上にある[定量]タブの下にある原子位置に基づいてひずみを計算ボタンをクリックします。
注: ユーザーは、[ひずみ上限/下限] 入力ボックス内の値を変更することで 、ひずみ マップの表示範囲をカスタマイズできます。
- 原子位置に基づいて、各ユニットセルから単位セルベクターを抽出します。
4. データの可視化
- 色付きの線マップを作成します。
注: 原子結合の色付き線マッピングは、近くの原子間の距離を表す簡単な方法です。Matlab では、2 つのポイントの間に線を引くコマンドは 、ライン([x1 x2]、[y1 y2]、'Color'、[r g b]) です。 入力[x1 x2]と[y1 y2]は、第1および第2位置の座標値である。距離の変化は、ライン マップの色を変えて表示できます。[r g b] 値は、赤、緑、青の各色の値を表し、それぞれ 0 ~ 1 の範囲です。その後、色付きの線で近くのすべての原子を繰りつながるように接続します。- EASY-STEMアプリで色付きのラインマップを生成します。
注:EASY-STEMアプリでは、ラインマップは、インターフェイスの右上にある 数量 タブの下にあるシンプルなボタンクリックで生成できます。- [ 平均距離 ] 入力ボックスの値 (pm) と、EASY-STEM の [計測範囲 ] 入力ボックスで値を調整します。この 2 つの値は、投影された原子距離の平均距離と測定の距離範囲を定義します。
- EASY-STEMアプリで、[ 近傍に基づいて結合長を計算 ]ボタンをクリックします。
注: ライン マップは自動的に生成されます。ユーザーは、カラーマップ、線のスタイル、線幅を調整して、視覚化を改善できます。
- EASY-STEMアプリで色付きのラインマップを生成します。
- ベクトルマップを作成します。
注: ベクトルマップは、結晶の領域に原子変位を示すことができます。変位解析は個々のシステムに固有のものであるため、コードをEASY-STEMアプリに統合したのではなく、ここでは標準のABO3 ペロブスカイトユニットセルに基づいてこのような解析のためのMatlabコマンドを紹介します。- 変位測定の基準位置を計算します。
注: ABO3 ペロブスカイトの例では、(0,0),(0,0),(0,1),(1,1),中央の原子(Bサイト)として角(Aサイト)上の原子を(1/2,1/2)としてインデックス付けしました。単位セルの中心に対する変位を計算するには、まず、基準位置を角(A-サイト)原子の平均位置として計算します。この計算の Matlab コマンドは次のとおりです。
ref_center=(ポジションA1+ポジションA2+ポジションA3+
ポスティオンA4)/4 - 次のコマンドを入力して変位を計算します。
[displace_x displace_y] = 位置B - ref_center - ベクトルマップを実装します。
クイバー(x、y、displace_x、displace_y)
注: 入力 x と y は変位原子の位置です。変数 displace_x と displace_y は、x 方向と y 方向の変位の大きさです。ベクトルマップは、均一に色付け(例えば、黄色、白、赤..)、または変位の大きさに基づいてシェーディングすることができます。
- 変位測定の基準位置を計算します。
- 偽色のマップを作成します。
- アップサンプリングによって偽色のマップを生成し、イメージ内の各ピクセルの測定値(変位、歪みなど)を推定します。
イメージサイズ = サイズ(イメージ);
[xi,yi] = メッシュグリッド(1:1:ImageSize(1)、1:1:ImageSize(2));
Upsampled_Data = グリッドデータ(x,y,YourData,xi,yi,'v4');
注: "griddata" 関数は、画像全体の各ピクセルの値を推定するために、位置 (x,y) のデータをアップサンプリングします。入力 xi と yi はグリッド座標で、'v4' は二方晶のアップサンプリング方式です。 - ユーザー定義のカラースケールを使用して、アップサンプルデータをプロットします。
- アップサンプリングによって偽色のマップを生成し、イメージ内の各ピクセルの測定値(変位、歪みなど)を推定します。
結果
図 3は、プロトコルの手順 1 と 2 に従って、アトミック位置の追跡の結果の例を示しています。ABO3ペロブスカイトのユニットセルの生のADF-STEM画像を図3aに示し、その強度プロファイルは図3bの3-Dにプロットされている。図3cは、ガウスフィルタリングが図3aのSTEM画像に適用された後の結果を示し、強度プロファイルは図3dにプロットされている。初期位置はイメージ内の局所的な最大値を見つけることによって決定され、位置は図 3eの黄色い円で示されます。アトミック位置は、単位セルベクトルに基づいてインデックスが作成され、図 3fに示されています。初期位置が見つまり、インデックスが作成された後、2D ガウスフィッティングが適用され、計測をさらに洗練します。図3gおよび図3hでは、フィット位置が赤い円として示され、初期位置(黄色の円)に比べて絞り込まれた位置が中心に近づくにつれて測定精度が向上する。最後に、重なり合う強度にmpfitアルゴリズムを適用する利点は、BaMnSb2結晶のADF-STEM画像に示される(図3i)。通常の 2D ガウスフィッティング (赤い円) は黄色の矢印で強調表示されている Mn 列で重大な障害を起こし、mpfit アルゴリズムは位置をより正確に決定できます (緑の円)。

図4:CA3Ru2O7(CRO)のHAADF-STEM画像(a)を結晶構造概略に重ね合わせたCa3Ru2O7(CRO)結晶のADF-STEM画像の拡大画像。ペロブスカイト層におけるCa原子の相対変位は黄色の矢印で強調表示されます。(b) ドリフト補正およびノイズ除去された ADF-STEM 画像(c) を重ね合わせ、洗練された原子位置 (赤い点) を重ねた。(d) ペロブスカイト層の上部(赤)、中央(青)、下(黄色)Ca原子を同定するためにインデックス化システムを用いた例。この図の大きなバージョンを表示するには、ここをクリックしてください。
Ca3Ru2O 7(CRO)のHAADF-STEM画像を図4aおよび図4bに示す(オーバーレイされた結晶構造を有する)。CROは、極空間群A21amを有するラドルスデン・ポッパー相ペロブスカイト結晶である。ADF-STEMイメージングは、より重い元素(CaとRu)とのコントラストを示していますが、O原子は、HAADF検出器で見えるように十分な強度のビームを散乱させないように、より軽い原子として視覚化されません。結晶構造の非中心対称性は、O八面体の傾きによって引き起こされ、二重ペロブスカイト層の中央にあるCa原子の変位を解析することによってADF-STEM画像で可視化することができる。「プロトコル」セクションに記載されている手順に従うことで、図 4cに示すように、このイメージ内のすべての原子位置を、適合した 2D ガウスピークの中心を見つけることによって見つけられる。さらに、インデックス化システムを用いて、ステップ3.2において、単位セル内の各タイプの原子を特定し、更なる処理に使用することができる。例えば、ペロブスカイト二重層の上側、中央部、下側のCa原子を容易に識別でき、その位置は、図4dに示すように、異なる色で満たされた円で提示される。
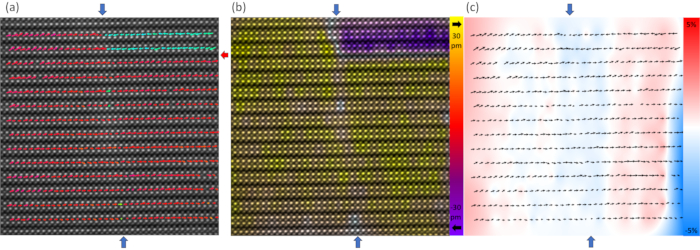
図5: 物理的な情報(a ) 中心Ca変位パターンから得られた偏光を示すベクトルマップの実装例。矢印は、向き(右に赤、左に青)に基づいて色付けされます。垂直の頭から頭への垂直および頭から尾へのドメインの壁は青い矢印で示され、水平180°ドメイン壁は赤い矢印で示される。(b) 偏光を示す偽色マップの実装例。色は、左(黄色)と右(紫)の方向の大きさを示します。強度が小さくなると、色が薄くなります。(c)イメージ内のεxx歪みを示す偽色マップの実装例。色は引張り(赤)と圧縮(青)のひずみの値を示します。この図の大きなバージョンを表示するには、ここをクリックしてください。
STEM画像内の原子の位置付けとインデックス作成後、図5に示すように、物理情報を抽出し、さまざまな種類のプロットを介して可視化することができる。偏光方向を示すベクトルマップを図5aに示します。矢印は、投影された偏光方向に向かって向き、向きに基づいて矢印を色付けすることで、縦方向の頭から頭に向かう 90° ドメインの壁 (青い矢印で示されています) と水平方向の 180° ドメイン壁 (赤い矢印で示されています) が画像の上部に表示されます。図5bに示すように偽色の地図を構築することで、中心のフェーディングカラーを通して極変位の大きさが減少し、頭から尾までのドメインの壁を視覚化することができます。ベクトルマップと偽色マップを組み合わせることで、3つのドメイン壁によって形成されたTジャンクションがADF-STEM画像に示されています。さらに、画像内の各単位セルの寸法を測定すると、図5cに示すように、ε xxひずみマップを構築できます。
ディスカッション
取得後の処理に取り組む場合は、注意が必要です。まず、画像のドリフト補正中に、アルゴリズムは0°画像が水平高速スキャン方向を持つものと仮定するため、計算の前に方向を再確認します。スキャン方向が正しく設定されていない場合、ドリフト補正アルゴリズムは失敗し、出力17にアーティファクトが生じる可能性があります。次に、イメージングのノイズ化中に、特定の方法でアーティファクトを導入する可能性があります。たとえば、フーリエ フィルタリングでは、空間解像度が適切に制限されていない場合、空室サイトでアトム列のコントラストを作成したり、画像内の微細なフィーチャを削除したりできます。その結果、ノイズ除去された画像が元の生の入力イメージに近いかどうかを確認することが極めて重要です。
次に、ローカルの最大値/最小に基づいて初期原子位置を決定する場合、アトミック列間の冗長な位置を作成しないように、ピーク間の制限最小距離を調整してみてください。これらの冗長な位置は、アルゴリズムが誤ってイメージ内のローカルの最大/最小値を原子列として認識するために生成されるアーティファクトです。さらに、画像内の様々な原子種間に大きなコントラスト差がある場合(例えば、WS2のADF-STEM画像)の位置の大部分を見つけるためにしきい値を調整することができる。イメージの初期アトミック位置のほとんどを取得した後、不足しているものを手動で追加するか、または最善の努力で余分なものを削除してみてください。また、画像内の周期性に大きな中断がない場合には、原子のインデックス化方法が最も効果的である。画像に表示される粒子境界や位相境界などの中断がある場合、インデックス化が失敗することがあります。この問題の解決策は、画像内の対象領域を定義し(EASY-STEMアプリで [対象領域の定義 ]ボタンをクリック)、各領域内の位置を個別にインデックス化して絞り込みます。その後、同じ画像内の異なる領域のデータセットを 1 つのデータセットに簡単に結合し、解析に取り組むことができます。
最後に、2D-Gaussian のピーク 継手を適用した後、入力イメージに微調整された位置ポイントを分散して、適合結果を検証し、精製された位置が原子列から逸脱しているかどうかを確認します。単一のガウスフィッティングアルゴリズムによって提供される精度は、ほとんどのSTEM実験で十分です。しかし、隣接する原子からの強度により位置が逸脱する場合は、マルチピークフィッティング(mpfit)アルゴリズムを使用して、隣接する原子列21から強度を分離する。それ以外の場合、特定の原子列から画質の問題や低強度に起因する位置が逸脱した場合、その位置で適合位置を破棄することが示唆される。
原子位置測定のためのいくつかの既存の特殊なアルゴリズムがあり、例えば、酸素オクタヘドラピッカーソフトウェア22、Atomapパイソンパッケージ23、およびStatSTEM Matlabパッケージ24。ただし、これらのアルゴリズムには、特定の面でいくつかの制限があります。例えば、酸素オクタヘドラピッカーは、明らかに解決された原子列のみを含むSTEM画像の入力を必要とし、したがって強度21を重ね合う原子列を有する画像の問題に対処できなかった。一方、Atomapは「ダンベルのような」原子列の位置を計算できますが、このプロセスはあまり簡単ではありません。さらに、StatSTEM は重複する強度を定量化するための優れたアルゴリズムですが、反復モデルベースのフィッティングプロセスは計算上高価な21 です。対照的に、高度なmpfitアルゴリズムと統合されたMatlabアプリEASY-STEMと共にこの研究で導入された当社のアプローチは、重なり合う強度の問題に対処することができ、競争力のある測定精度を提供しながら、StatSTEMよりも計算上コストが低い。さらに、Atomapおよび酸素八面体ピッカーソフトウェアパッケージからの分析は、ABO3 ペロブスカイト結晶からのデータを分析するために設計され、特殊化されていますが、この研究で示されているインデックスシステムは、さまざまな材料システムについてはるかに柔軟です。この方法を使用すると、ユーザーは、洗練された原子位置とユニットセルベクトルインデックスの両方を含む出力結果に基づいて、独自の材料システムのデータ分析を完全に設計し、カスタマイズすることができます。
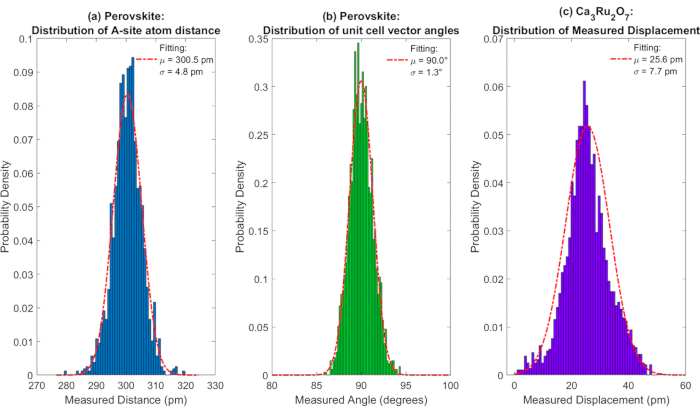
図6: 原子位置発見の統計的定量法. (a ) ヒストグラムで提示されたペロブスカイトAサイトからAサイトまでの距離の分布正規分布フィッティングは、午後300.5時の平均と4.8pmの標準偏差を示す赤い破線としてプロットされ、重ね合わされます。正規分布継手は、90.0°の平均と1.3°の標準偏差を示す赤い破線としてプロットされ、重ね合わせられます。(c)Ca3Ru2O7(CRO)における極偏位測定の統計的定量をヒストグラムとして提示する。正規分布継手は、25.6 pmの平均と7.7 pmの標準偏差を示す赤い破線としてプロットされ、重ね合わされています。
ここで紹介するメソッドは、実装のためのピクメータレベルの精度とシンプルさを提供します。測定精度を実証するために、原子位置発見の統計的定量を 図6に示す。立方体ABO3 ペロブスカイトAサイト距離分布と単位細胞ベクトル角度分布の測定値は、それぞれ 図6a および 図6bのヒストグラムを用いてプロットされる。正規分布曲線を分布に合わせることによって、Aサイト距離分布は平均300.5 pmを示し、標準偏差は午後4.8時、単位セルベクトル角度分布は平均90.0°、標準偏差は1.3°を示します。統計的定量は、ここで提案された方法がピクメータレベルの精度を可能にし、イメージング中のドリフトによる歪みを大幅に軽減できることを示しています。この結果は、測定する物理的情報がおよそ午後10時以上である場合に、この測定が信頼できることを示唆しています。例えば、前述のCRO結晶の場合、極偏位の大きさの測定を 図6cに示す。測定は、午後7時の標準偏差である25.6pmの平均を示し、CRO STEM画像の極偏差測定が固体であることを示しています。また、ビーム感度サンプルを撮像する場合、低信号対雑音比などの実験上の制限がある場合は、より注意が必要です。そのような場合、測定された原子位置は、測定の妥当性を確認するために生画像に対して綿密に調べる必要があります。したがって、ここで紹介した解析方法は、より最近の高度なアルゴリズムと比較して、測定精度に限界があります。サブピコメータレベルで精度が必要な場合、この方法は不十分であるため、画像内で抽出される機能が一定の閾値を下回る場合は、より高度な解析ルーチンが必要です。例えば、非剛性登録アルゴリズムは、シリコン上でサブピソメーターの精密測定を示しており、単一のPtナノ粒子25上で結合長変動を正確に測定することが可能である。最近では、ディープラーニングアルゴリズムを用いて、膨大な量のSTEM画像データから2次元遷移金属ジカルコゲニド単層の様々なタイプの点欠陥を同定しました。その後、異なるタイプの欠陥の平均画像に対して測定を行い、この方法は、それらの欠陥18の周囲の歪みにサブピコメータレベルの精度を実証しました。そのため、分析能力を向上させる将来の計画として、ディープラーニングなどの高度なアルゴリズムの開発と実装が進んでいます。また、今後のデータ解析ツールの更新に統合することも試みています。
開示事項
著者らは開示するものは何もない。
謝辞
L.MとN.A.の研究は、ペン州ナノスケール科学センター(助成金番号DMR-2011839(2020-2026)のNSF MRSECによってサポートされています。D.Mは、米国エネルギー省(DOE)のUT-Battelle(LLC)が管理するORNLの研究所主導型研究開発(LDRD)プログラムによって支援されました。A.CとN.A.は、空軍科学研究局(AFOSR)プログラムFA9550-18-1-0277とゲームMURI、10059059-PENNのサポートを認めています。
資料
| Name | Company | Catalog Number | Comments |
| EASY-STEM | Nasim Alem Group, Pennsylvania State University | Matlab app for STEM image processing; Download link: https://github.com/miaoleixin1994/EASY-STEM.git | |
| JoVE article example script | Nasim Alem Group, Pennsylvania State University | Example Script for sorting atoms in unit cells | |
| Matlab Optimization Tool Box | MathWorks | Optimization add-on packge in Matlab | |
| Matlab | MathWorks | Numerical calculation software | |
| Matlab: Image Processing Tool Box | MathWorks | Image processing add-on packge in Matlab |
参考文献
- Batson, P. E., Dellby, N., Krivanek, O. L. Sub-aångstrom resolution using aberration corrected electron optics. Nature. , (2002).
- Haider, M., et al. Electron microscopy image enhanced. Nature. , (1998).
- Muller, D. A., Nakagawa, N., Ohtomo, A., Grazul, J. L., Hwang, H. Y. Atomic-scale imaging of nanoengineered oxygen vacancy profiles in SrTiO3. Nature. , (2004).
- Findlay, S. D., et al. Robust atomic resolution imaging of light elements using scanning transmission electron microscopy. Applied Physics Letters. , (2009).
- Tate, M. W., et al. High Dynamic Range Pixel Array Detector for Scanning Transmission Electron Microscopy. Microscopy and Microanalysis. , (2016).
- Rodenburg, J. M., McCallum, B. C., Nellist, P. D. Experimental tests on double-resolution coherent imaging via STEM. Ultramicroscopy. 48, 304-314 (1993).
- Jiang, Y., et al. Electron ptychography of 2D materials to deep sub-ångström resolution. Nature. 559, 343-349 (2018).
- Yang, Y., et al. Deciphering chemical order/disorder and material properties at the single-atom level. Nature. , (2017).
- Bals, S., Van Aert, S., Van Tendeloo, G., Ávila-Brande, D. Statistical estimation of atomic positions from exit wave reconstruction with a precision in the picometer range. Physics Review Letters. , (2006).
- Kim, Y. M., He, J., Biegalski, M., et al. Probing oxygen vacancy concentration and homogeneity in solid-oxide fuel-cell cathode materials on the subunit-cell level. Nature Mater. 11, (2012).
- Azizi, A., et al. Defect Coupling and Sub-Angstrom Structural Distortions in W1-xMoxS2 Monolayers. Nano Letters. , (2017).
- Reifsnyder Hickey, D., et al. Illuminating Invisible Grain Boundaries in Coalesced Single-Orientation WS2 Monolayer Films. arXiv. , (2020).
- Mukherjee, D., et al. Atomic-scale measurement of polar entropy. Physics Review B. 100, 1-21 (2019).
- Yadav, A. K., et al. Observation of polar vortices in oxide superlattices. Nature. , (2016).
- Yankovich, A. B., et al. Non-rigid registration and non-local principle component analysis to improve electron microscopy spectrum images. Nanotechnology. , (2016).
- Ishizuka, K., Abe, E. Improvement of Spatial Resolution of STEM-HAADF Image by Maximum-Entropy and Richardson-Lucy Deconvolution. EMC. , (2004).
- Ophus, C., Ciston, J., Nelson, C. T. Correcting nonlinear drift distortion of scanning probe and scanning transmission electron microscopies from image pairs with orthogonal scan directions. Ultramicroscopy. , (2016).
- Lee, C. H., et al. Deep learning enabled strain mapping of single-atom defects in two-dimensional transition metal dichalcogenides with sub-picometer precision. Nano Letters. , (2020).
- Savitzky, B. H., et al. Bending and breaking of stripes in a charge ordered manganite. Nature Communications. 8, 1-6 (2017).
- Stone, G., et al. Atomic scale imaging of competing polar states in a Ruddlesden-Popper layered oxide. Natature Communications. 7, 1-9 (2016).
- Mukherjee, D., Miao, L., Stone, G., Alem, N. mpfit: a robust method for fitting atomic resolution images with multiple Gaussian peaks. Advanced Structural and Chemical Imaging. , (2020).
- Wang, Y., Salzberger, U., Sigle, W., Eren Suyolcu, Y., van Aken, P. A. Oxygen octahedra picker: A software tool to extract quantitative information from STEM images. Ultramicroscopy. 168, 46-52 (2016).
- Nord, M., Vullum, P. E., MacLaren, I., Tybell, T., Holmestad, R. Atomap: a new software tool for the automated analysis of atomic resolution images using two-dimensional Gaussian fitting. Advanced Structral and Chemical Imaging. 3, 9(2017).
- De Backer, A., vanden Bos, K. H. W., Vanden Broek, W., Sijbers, J., Van Aert, S. StatSTEM: An efficient approach for accurate and precise model-based quantification of atomic resolution electron microscopy images. Ultramicroscopy. 171, 104-116 (2016).
- Yankovich, A. B., et al. Picometre-precision analysis of scanning transmission electron microscopy images of platinum nanocatalysts. Nature Communications. , (2014).
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved