Method Article
オリゴヌクレオチドの化学的三リン酸化
要約
オリゴヌクレオチド5'-三リン酸は、必須の生物学的経路におけるユビキタス成分であり、バイオテクノロジー用途での使用が増加している。ここでは、標準的な自動合成技術によって調製されたオリゴヌクレオチドから出発して、オリゴヌクレオチド5'-三リン酸の日常的な合成および精製のための技術について述べる。
要約
5'-三リン酸は、すべての生命を通して見られる必須の核酸修飾であり、バイオテクノロジーおよび合成生物学におけるオリゴヌクレオチドの機能修飾としてますます使用されている。オリゴヌクレオチド5'-三リン酸は、歴史的に酵素的方法によって インビトロで 調製されてきた。しかしながら、これらの方法は天然RNAオリゴヌクレオチドに限定され、強い配列選好を有し、そして不均一な産物を産生する傾向がある。化学的三リン酸化の新しい方法は、ホスホロアミダイト化学による自動オリゴヌクレオチド合成のコスト削減と、現在利用可能な多様なヌクレオチド修飾の両方を補完します。したがって、任意の配列および長さのオリゴヌクレオチド三リン酸の合成、および任意選択で様々な非天然修飾を含む、現在アクセス可能である。
この論文では、サリチルホスホロクロリダイトおよびピロリン酸を用いたオリゴヌクレオチドの化学的三リン酸化のための適切な方法および技術を提示する。この方法は、市販の試薬を使用し、標準的な固相合成法によって調製されたほとんどのオリゴヌクレオチドと適合性があり、脱保護および精製の前に、オリゴヌクレオチド合成後2時間で完了することができる。非生物学的L-RNA三リン酸からのハンマーヘッドリボザイムの鏡像版の合成を含む、触媒RNA酵素の基質としての化学的に三リン酸化されたオリゴヌクレオチドの2つの使用が実証されている。
概要
RNAの5'-トリリン酸化型は、生命のすべてのドメインにおけるRNA転写および多くのRNAウイルスのライフサイクル中のRNA複製によって生成されるため、生物学において遍在している。これらの三リン酸は、真核生物における7-メチルグアニル酸キャップ付きmRNAの形成のための基質として働き、したがって、タンパク質発現において不可欠な役割を果たす1。対照的に、三リン酸は細菌およびウイルスに保持される。したがって、RNA 5'-三リン酸は、真核生物2、3、4、5、6、7の自然免疫応答調節因子によって認識される。生物学の外では、RNAリガーゼリボザイムのホストは、インビトロ8で5'-三リン酸を使用するように進化し、診断アッセイ9、10、11、12、13、14、15で使用するために修飾されています。そのようなリボザイムの1つは、小さなL−RNAオリゴヌクレオチド5'−三リン酸16,17,18からの天然D−RNAの非生物学的「鏡像」鏡像異性体であるL−RNAの鋳型依存的合成に使用することができる。様々な配列および骨格組成のトリリン酸化オリゴヌクレオチドの日常的な調製は、これらの系を調査するために不可欠である。
実験室でRNA 5'-三リン酸を調製するための最も一般的でアクセス可能な方法は、in vitro転写によるものです。しかしながら、この方法により産生されるRNAは、RNAポリメラーゼ酵素のプロモーターや基質要件によって配列やサイズに制限がある。T7 RNAポリメラーゼおよび特殊化誘導体は、この目的に使用される最も一般的なポリメラーゼである19、20、21、22。これらの酵素で調製されたインビトロ転写RNAは、5'末端プリンで開始されなければならず、最初の10ヌクレオチド23,24のプリン体に強く偏っている。さらに、塩基または骨格修飾ヌクレオチドの酵素的組み込みは、せいぜい非効率的であり、天然ポリメラーゼではよりしばしば不可能であり、天然D-RNA以外のものからなるオリゴヌクレオチド5'-三リン酸を生成する機会を制限する。もう一つの制限要因は、インビトロ転写によって生成されたRNAが実質的な5'-および3'-不均一性を含み得、20nt 23,24,25,26,27より短いときに非常に不均一な産物として産生されることである。
対照的に、固相ホスホロアミダイト合成28、29、30、31、32、33、34、35によって調製されたオリゴヌクレオチドの化学的三リン酸化は、任意の配列の3〜50nt長のオリゴヌクレオチド5'−三リン酸を調製するために使用することができる。さらに、ホスホロアミダイト合成にアクセス可能な膨大な数の核酸修飾を、5'-三リン酸化14,15,16,17,18,29,36の前にオリゴヌクレオチドに付加することができる。これらの方法の多くは、モノヌクレオシド37の溶液相三リン酸化のためにルートヴィヒとエクシュタインによって開発された亜リン酸ホスホロクロリダイトを使用する。この試薬によるオリゴヌクレオチドの三リン酸化は、オリゴヌクレオチド5'-ヒドロキシルのホスフィチル化、ピロリン酸との反応および酸化による三リン酸への変換、続いて固体支持体からのオリゴヌクレオチドの切断、脱保護、および精製のための標準的な手順によって固相上で達成される(図1)28。
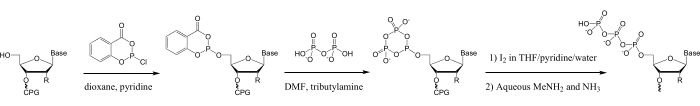
図1:合成オリゴヌクレオチドの三リン酸化のスキーム。第1工程では、オリゴヌクレオチド5'-ヒドロキシルをSalPClでホスフィチル化する。次のステップでは、5'-サリチルホスファイトをTBAPと反応させて環状メタホスファイトを形成し、次いで第3工程で酸化してDNA/RNA合成機酸化溶液(0.1 M ヨウ素/ピリジン/H2 O/THF)中で環状5'-トリメタリン酸を生成し、これを急速に加水分解して同じ溶液中の直鎖状5'-三リン酸を生成する28,33、37。その後の固体CPG担体からのアルカリ切断および水性MeNH2/アンモニア中でのオリゴヌクレオチドの脱保護は、残留環状トリメタリン酸を直鎖状形態に加水分解する。略語:SalPCl=サリチルホスホロクロリダイト;TBAP = トリブチルアンモニウムピロホスフェート;THF=テトラヒドロフラン;CPG = 制御された細孔ガラス;MeNH2=メチルアミン。この図の拡大版を表示するには、ここをクリックしてください。
この方法を用いた早期に発表された報告は、しばしば収率の悪さおよび望ましくない副産物28,37,38に苦しんだが、無水条件の慎重な維持は、日常的に高い収率を得るために必要なすべてである。これは、試薬の慎重な調製と、標準的なプラスチック部品から組み立てられた簡単な反応装置の使用によって達成することができる。ここでは、試薬の調製、反応チャンバーの組み立て、トリリン酸化反応、およびその後のトリリン酸化オリゴヌクレオチドの脱保護および精製を含む、オリゴヌクレオチドの化学的トリリン酸化のための適切なステップを実証する。また、天然のD-RNAおよび非生物的L-RNA骨格を有するより大きな核酸産物を合成するためのリガーゼリボザイムの基質としての5'-トリリン酸化オリゴヌクレオチドの代表的な使用も含まれる。
プロトコル
1. 固体支持体上での5'-ヒドロキシルオリゴヌクレオチドの自動固相合成
- 自動DNA/RNA合成機を試薬およびホスホロアミダイトとともに調製し、標的オリゴヌクレオチド組成および機器の指示に従ってください。
- 固体支持体を含む合成カラムをシンセサイザーにロードし、シンセサイザーの機器プロトコルに従ってオリゴヌクレオチドを合成する。
注:三リン酸化手順は、1μmoleスケールで調製したオリゴヌクレオチド用に最適化されています。 - 5'-ジメトキシトリチル保護基を除去し、前の工程でオリゴヌクレオチド合成の一部として、または合成器器具のプロトコルに従って末端脱トリチル化工程を行うことによって、固体支持オリゴヌクレオチド5'-ヒドロキシルを得る。
- 固体支持体上の5'-ヒドロキシルオリゴヌクレオチドを含むカラムをシンセサイザーから取り出し、ハウス真空下で10分間乾燥させて残留溶媒を除去し、トリリン酸化のための材料が調製されたら(セクション3および4)に進む(セクション2)。
注:すぐに使用しない場合、乾燥カラムは、-20°Cの乾燥剤を含む密閉プラスチック容器に通常の雰囲気下で保存することができます。 セクション 3 のトリリン酸化の前にカラムが完全に乾燥されるため、この段階ではさらなる乾燥は必要ありません。
2. 三リン酸化材料の準備
- 圧力調節可能なドライアルゴン源を少なくとも2本のラインでガスマニホールドに取り付け、バブラーに接続します。反応装置への接続を容易にするために、ラインが1mLのプラスチックシリンジで終端していることを確認してください。
- 1 mL のプラスチックシリンジ、三方ポリプロピレン活栓、ノンコアリング針、1.5 mL ポリプロピレンチューブ、小型金属ヘラなど、三リン酸化時に使用する収集装置。使用前に少なくとも1日間、室温で乾燥剤と一緒に密閉容器またはデシケータに保管してください。
- 少なくとも1日前に、無水1,4-ジオキサン、3:1ジオキサン:ピリジン、N,N-ジメチルホルムアミド(DMF)、およびアセトニトリル(ACN)を、乾燥トラップ(密閉膜パケット中の4Åモレキュラーシーブ)を備えた乾燥30mLガラス瓶にそれぞれ30mLずつ調製する。ゴムセプタでシールし、乾燥剤を入れたデシケーターに保管してください。
- 固体2-クロロ-4H-1,3,2-ベンゾジオキサホスホリン-4-オン(サリチルホスホロクロリダイト、SalPCl)を、4°Cで乾燥剤を入れた密閉瓶の中の元の容器に保管してください。 使用の合間には常にコンテナをアルゴンでフラッシュしてください。
- トリフェニル化反応の少なくとも5日前にトリブチルアンモニウムピロホスフェート(TBAP)溶液を調製する:
- 乾燥した30mLガラス瓶に1〜5gの固体TBAPを計量し、TBAP1gあたり1mLのDMFおよび0.5mLのトリブチルアミンに溶解します。
- 3つの乾燥トラップを追加し、アルゴン下のゴム中隔でボトルを密封し、アルゴンで30分間泡立てて脱気します。
- モレキュラーシーブがすべての微量水を吸収できるように、4°Cの乾燥剤を入れた密封瓶の中に5日間保管してください。瓶を-20°Cで保管し、6ヶ月後に新鮮なものを用意してください。
3. 三リン酸化装置の組み立てと使用
- -20°Cで貯蔵から取り出す場合は、合成カラムを室温まで昇温させます。
- 図 2 に示す反応チャンバーを組み立てます。
- 前室を準備する:乾燥した1mLシリンジからプランジャーを取り外し、はさみまたはカミソリブレードを使用してシリンジの上部を切り取り、シリンジを合成カラムに取り付けます。三方活栓をシリンジの上部に取り付け、活栓の側面入口をバブラーで乾燥アルゴン源に取り付け、活栓の上部入口が試薬注入口になるようにします。
- この装置をクランプ付きのスタンドに固定し、すべての上流ジョイントをワックスシーリングフィルムでシールします。活栓を調整して、注入口が閉じ、装置がアルゴン源に対して開かれるようにします。バブラーを閉じ、低圧(<10psi)のアルゴンが反応チャンバ内を5分間流れるようにします。
注:複数の反応チャンバは、トリホスホリル酸2〜4オリゴヌクレオチドに並列に設定することができる。ただし、マニホールドからの1本のラインは、試薬ボトルにアルゴンを供給するために予約する必要があります。 - バブラーを再度開き、合成カラムの底部にシリンジを取り付けます。廃棄物シリンジを使用してカラムにアルゴンを繰り返し引き出します。次に、プランジャーを完全に押し込んだ状態でシリンジを取り付け直します。
メモ: 図2Aに示すように、試薬をロードしない限り、活栓は注入ポートが閉じられ、装置がアルゴン源に対して開くように設定する必要があります。同様に、廃シリンジを取り付け、試薬を積極的に除去しない限り、合成カラムにジョイントをワックス封止フィルムで密封する必要があります。
- 試薬または溶媒を追加するには:
- 針を乾燥アルゴン源に取り付け、試薬または溶媒ボトルの隔壁に挿入し、針をボトルの内容物に浸さないように注意してください。
- 針で乾いたシリンジを組み立て、ボトルの内容物に浸さずに試薬または溶媒ボトルセプタムに挿入します。シリンジをアルゴンで満たし、中隔から針を抜き取り、アルゴンを排出する。シリンジをアルゴンで満たし、再び排出する。次に、アルゴン圧力下で必要な量の溶媒または試薬をシリンジに充填する。
- アルゴン源が閉じ、注入ポートが開くように装置の活栓を調整します(図2B)。充填されたシリンジと針をソースボトルから素早く取り出し、針の側面または先端に付着した溶媒を拭き取り、針を注射口に挿入します。試薬を装置の前室に排出し、針を取り外し、注入口を閉じ、アルゴン源に装置を再度開放する。
- すべての液体が廃棄物シリンジに保持されるように、廃液シリンジを使用して、前室から合成カラムを通って液体を静かに引き出します。次に、溶液をゆっくりと合成カラムに押し戻し、カラムに気泡がないことを確認します。混合または攪拌するには、廃シリンジで溶液をカラム上で静かに上下に引っ張ります。
メモ:液体を反応チャンバ内をゆっくりと穏やかに動かして、シールが破られないようにして、空気が装置に入るようにしてください。
- カラムから試薬または溶媒を除去するには:
- 溶液をゆっくりと廃シリンジに引き込みます。溶液の大部分が廃シリンジに渡されたら、アルゴンを通してカラムから残りの溶媒を洗い流します。
- 廃シリンジジョイントの周りのワックスシーリングフィルムを取り外し、シリンジを取り外して廃液を捨てます。廃シリンジを新しいドライシリンジに交換し、ワックスシーリングフィルムでジョイントを再シールします。
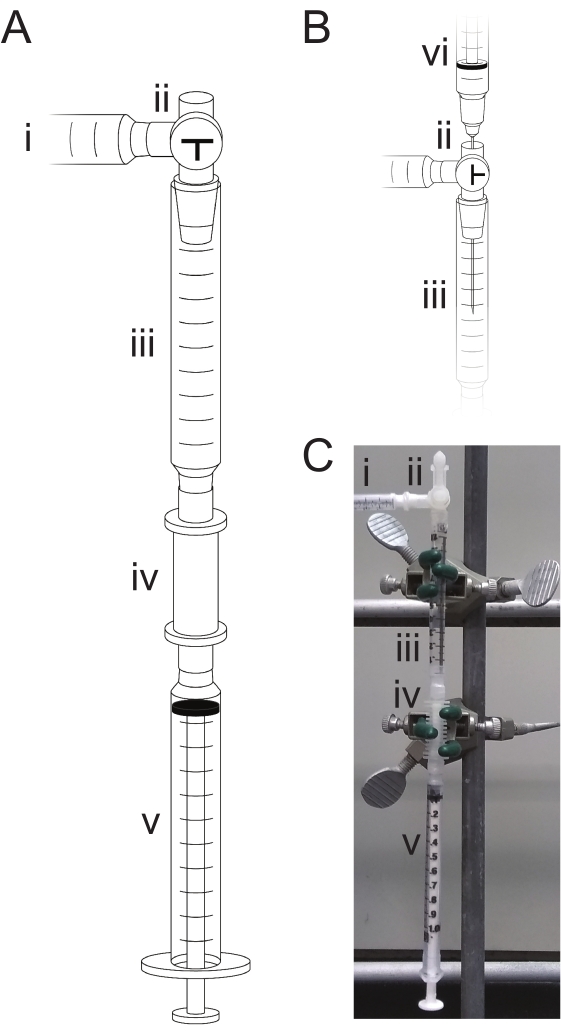
図2:三リン酸化装置。 混合中または反応中、装置(A)はアルゴン源(i)に対して開放され、三方活栓(ii)を調整することによって空気に対して閉じられる。試薬は、廃棄物シリンジ(v)を用いて前室(iii)から合成カラム(iv)に引き出される。試薬は、すべての液体を廃シリンジ(v)に引き込み、それを廃棄することによって除去される。試薬(B)を装填する場合、三方活栓(ii)は大気に開放され、試薬はシリンジと針(vi)によって前室(iii)に装填される。(C)(A)のように試薬混合反応用に組み立てられた装置の写真。 この図の拡大版を表示するには、ここをクリックしてください。
4. 合成 5'-ヒドロキシルオリゴヌクレオチドのオンカラムトリリン酸化
- SalPClおよびTBAP溶液を-20°Cの保管から取り出し、使用前に室温まで温めるようにします。
- 200 μLのピリジン/ジオキサンを、ステップ3.3.1-3.3.3に従って前室に加える。ただし、ステップ 4.4 まで合成カラムに溶媒をロードしないでください。
- 乾燥金属ヘラを使用して、乾燥した1.5mLの微量遠心管に6〜12mgのSalPClを計量し、微量遠心管内で溶媒を上下に静かにシリンディングして100μLのジオキサンに溶解する。
- 溶解したSalPClを前室に加え、ステップ3.3に従って合成カラムにロードします。15分間反応させ、5分ごとに溶液を攪拌する。手順 3.4 に従って SalPCl ソリューションを取り出し、廃棄します。
注:SalPClは大過剰に添加され、調製および反応チャンバへの装填中に吸収された水を捕捉します。しかし、ステップ4.5および4.6の間に水分を導入すると、最終的なオリゴヌクレオチド5'-三リン酸収率が損なわれます。 - 250 μL の TBAP 溶液を前室に加え、ステップ 3.3 に従って合成カラムにロードします。5分ごとにかき混ぜながら、20分間反応させます。手順 3.4 に従って TBAP ソリューションを取り外して破棄します。
- カラムを 0.5 mL の DMF、次いで 0.5 mL の ACN で洗浄し、ステップ 3.3 および 3.4 に従って各添加後に溶媒を除去します。
- 250 μL の酸化剤溶液 (0.1 M ヨウ素 in テトラヒドロフラン (THF)/ピリジン/水、88:10:2) をアンテチャンバーに加え、ステップ 3.3 に従って合成カラムにロードします。10分ごとに攪拌しながら、30分間反応させます。ステップ3.4に従って酸化剤溶液を取り出して廃棄する。
- カラムを 0.5 mL の ACN で洗浄し、ステップ 3.3 および 3.4 に従って除去します。
- 反応装置を分解します。合成カラムを 5 mL の ACN で洗浄し、カラムを乾燥させます。
5. 固体支持体からの切断、脱保護、精製
- 乾燥した固体支持樹脂を合成カラムから取り出し、シリコーンOリング付きの1.5mLポリプロピレン製スクリューキャップシールチューブに移します。
- 28%-30%アンモニア水と40%メチルアミン水溶液(AMA)の1:1混合物1mLに樹脂を懸濁させ、チューブをしっかりと密封します。穏やかに反転39による間欠混合で65°Cで10分間インキュベートする。40 ntより長いオリゴヌクレオチドについては、室温で2時間、より穏やかな処理を使用する。
警告: AMA 溶液を加熱すると、チューブに高圧がかかります。チューブがしっかりと密封されていない場合、またはアンモニアと相溶する(シリコーン)Oリングを使用しない場合、チューブはガスを排出したり、溶媒を漏らしたりして、安全性や最終製品の歩留まりを損なう可能性があります。高温のAMA溶液はガスを激しく発生させる可能性があるため、周囲温度を超えている間はチューブを開けないでください。 - チューブを氷の上で冷やし、短時間遠心分離します(6,000-12,000 × g を10秒間)。樹脂から上清を取り出し、0.2μmフィルターを備えたシリンジでろ過し、新しい滅菌ポリプロピレンチューブに移します。アンモニア中和化学トラップを取り付けた真空遠心分離機を使用して、溶液を蒸発乾固させる。
注:合成オリゴヌクレオチドに2'-シリル保護基を有するRNAヌクレオチドが含まれていない場合は、ステップ5.8に進んでください。 - 乾燥した材料を1mLの1 Mテトラブチルアンモニウムフルオリド(TBAF)をTHFに溶解し、55°Cに加熱し、必要に応じて振とうしてオリゴヌクレオチドを完全に溶解させ、室温で16〜24時間インキュベートすることによってシリル保護基を除去する40、41。
- TBAF溶液を1 mLの1 M Tris緩衝液(pH 7.5)でクエンチし、真空遠心分離機を使用してTHFを除去します。
- TBAF塩は、製造元の指示に従って、使い捨てのサイズ除外カラムを使用して除去します。15ntより短いオリゴヌクレオチドについては、溶出液を画分に集めて生成物の溶出を確認し、UV-Vis分光光度計上の260nmの吸光度により主生成物画分を同定する。
- 脱保護RNAオリゴヌクレオチドを、15nt未満の場合は凍結乾燥または真空遠心分離機によって、または15ntを超える場合はエタノール沈殿によって濃縮する。
- オリゴヌクレオチドサイズ、ゲルプレート、およびスタンドの適切なプロトコルに従って、19:1 mono:bisアクリルアミドストックを使用して、分取スケール10%〜20%ポリアクリルアミド/8 M 尿素/1x TBEゲルを調製します。ゲルプレートを1x TBEのリザーバーでゲルスタンドに組み立て、35W(またはゲルプレートフォーマットの場合は適宜)で少なくとも30分間プレランします。
- 固体オリゴヌクレオチドを尿素ゲルローディングバッファー(8 M 尿素、10% スクロース、50 mM Tris、ブロモフェノールおよびキシレンシアノールランニング色素と共にpH 8、1 mM EDTA)に溶解し、80°Cに加熱する。 ポリアクリルアミドゲルに負荷をかけ、25〜35W(またはゲルプレートフォーマットおよびオリゴヌクレオチドサイズに適したもの)で1〜2時間実行する。
注:ブロモフェノールブルーまたはキシレンシアノールは、エタノール沈殿なしで溶出オリゴヌクレオチドからこれらの色素を除去することは困難であるため、どちらかが生成物と共移行する場合、15ntより短いオリゴヌクレオチドのゲルローディングバッファーから除外する必要があります。 - ゲル電気泳動が終了したら、ゲルスタンドからゲルプレートを取り外し、ゲルプレートを分解し、ゲルをポリ塩化ビニルフィルムで包みます。254 nm UV光でバックシャドーイングして製品バンドを特定し、カミソリブレードを使用して主要な製品バンドを切除します。
- 破砕・浸漬法42により切り出したゲルからオリゴヌクレオチドを抽出する。
- 切り出したゲルをプラスチックシリンジを通して押し出すか、機械的に押し出して粉砕する。
- 15 ntより長いオリゴヌクレオチドの場合:
- 3倍量の破砕バッファー(300 mM NaCl、10 mM Tris、pH 8、1 mM EDTA)に、攪拌または振とうしながら少なくとも12時間浸します。
- 溶液を0.2 μmフィルターを取り付けたシリンジに通して固体ゲル片を除去し、エタノール沈殿によりオリゴヌクレオチドを濃縮する。
- 15 ntより短いオリゴヌクレオチドの場合:
- 3倍の量のヌクレアーゼフリーの水を少なくとも12時間、攪拌または振盪しながら溶出する。
- 0.2 μmフィルターを取り付けたシリンジに溶液を通して固体ゲル片を除去し、凍結乾燥により濃縮する。
- ステップ 5.6 のように使い捨てサイズ排除カラムを使用して、残留塩および溶質を除去します。凍結乾燥により濃縮する。
- 精製トリリン酸化オリゴヌクレオチドを -20 °C で TE バッファー (10 mM Tris、pH 8; 1 mM EDTA) または類似のオリゴヌクレオチド保存バッファーに保存します。
- UV-Vis分光光度計を用いて260nmにおける吸光度を測定することによりオリゴヌクレオチドの濃度を求める。
注:オリゴヌクレオチドの推定吸光係数は、その5'-リン酸化状態の影響を受けるべきではなく、オリゴヌクレオチド吸光係数計算機を使用してその配列から計算することができる。 - 質量分析により三リン酸化を検証する。5'-ヒドロキシルオリゴヌクレオチドの質量に対して+239.94Daの期待質量を探します。
注:マトリックス支援レーザー脱離/イオン化またはエレクトロスプレーイオン化質量分析(それぞれMALDI-MSまたはESI-MS)は、核酸に最適化されたプロトコルを使用する場合、オリゴヌクレオチドの三リン酸化状態を特定するのに適しています。しかし、ESI-MSはイオンフラグメンテーションの速度が低いため、より一貫した結果を提供します。代表的な商用サービスは、 材料表で提供されています。
- UV-Vis分光光度計を用いて260nmにおける吸光度を測定することによりオリゴヌクレオチドの濃度を求める。
6. リボザイムの自己複製の基質としてのトリリン酸化オリゴヌクレオチド
警告: 32P は放射性同位元素であり、実験室で放射性物質を扱うための標準的な安全プロトコルを使用して、および関連する環境安全衛生部門によって放射性物質の使用が認定された研究者によって、次の手順を実行する必要があります。代替として、自己複製リボザイム基質Aを、5'−フルオレセイン標識14 を用いて合成的に調製し、ステップ7.9のように蛍光で画像化することができる。
- 溶液Aを、自己複製子リボザイムEおよび5'-32P標識RNA基質A14の混合物として、それぞれ0.30μMおよび30μMの濃度に調製する。75 mM EPPS緩衝液、pH 8.5、および37.5MgCl2中で、それぞれ上記のようにインビトロ転写14または化学的トリリン酸化によって調製した、15 μMトリリン酸化RNA基質Bを用いてB転写およびB合成した溶液を調製する。両方の溶液を42°Cにしてください。
注: すべての RNA コンポーネントは、 材料表に記載されています。 - 自己複製を開始するには、5 μL の A 溶液と、転写または B 合成の 10 μL を終濃度 0.1 μM E、10 μM A、10 μM B、25 mM MgCl2、および 50 mM EPPS バッファー pH 8.5 に迅速に混合します。反応混合物を42°Cでインキュベートする。
- 一定の間隔で、0.5 μL のアリコートを取り、9.5 μL のホルムアミドゲルローディングバッファー (95% ホルムアミド; 10 mM EDTA、pH 8) でクエンチします。
- 分析用ポリアクリルアミド/8 M 尿素/1x TBEゲルを、ゲルプレートおよびスタンドの適切なプロトコールに従って調製します。キャストゲルとプレートをゲルスタンドに組み立て、リザーバを1x TBEで満たし、40W(または異なるゲルプレートとスタンドに適宜)で30分間プリランします。
- クエンチした反応サンプルを80°Cに加熱し、各ウェルに5 μLのサンプルを負荷し、ゲルを40 Wで約40分間(または異なるゲルプレートおよびスタンドに適宜)実行します。
- ゲルスタンドからゲルプレートを取り出し、分解し、ゲルをポリ塩化ビニルフィルムで包みます。ゲルを蛍光体スクリーンで覆い、1時間(または 32Pcpmの場合)露光する。蛍光/燐光ゲルスキャナーを使用して画面をスキャンします。
- ゲル画像定量ソフトを用いて反応収率を定量する。
- 分析ツールボックスを使用してゲル画像を開き、[分析|[形状定義]で、[長方形の形状|領域]を選択し、各時間および両方の反応について、未反応Aと生成物Eに対応するバンドの周りに同じサイズの長方形を描画します。
- 分析|の選択背景減算で、長方形の形状|領域を選択し、ゲル画像の空の部分に同じサイズの長方形を描画します。すべての四角形の背景減算方法を [画像四角形] に変更します。
- 「ウィンドウ」|「2領域ウィンドウ」を選択し、「|を編集」を選択します。Excelにエクスポートして、定量化されたバンドピクセルボリュームをスプレッドシートファイルにエクスポートします。
- 時間に対する製品Eの濃度をプロットし、統計データフィッティングソフトウェアを使用してデータをロジスティック成長Eq(1)に当てはめます。
[E] =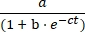 (1)
(1)
ここで、a は反応最大程度、b はシグモイディシティの程度、c は指数関数的成長速度です。- エクスポートされたスプレッドシートで、生成物 E 体積を基質 A と生成物 E 体積の合計で除算し、各時間および両方の反応の生成物の分数収率を決定します。基質Aの初期濃度(10 μM)を掛けて、生成物Eの収率を時間の関数として求めます。
- 統計データフィッティングソフトウェアで、[ファイル|] を選択します。新|新しいプロジェクト ファイル、[新しいテーブルとグラフ] の下の [XY] を選択して、[作成] をクリックします。隣接するカラム、およびラベルカラムに対応する両方の反応の反応時間と生成物E収率を入力します(例えば、「時間」、「転写B」、「合成B」)。
- [|の挿入] を選択します。[新しい解析]、[XY解析]|選択非線形回帰 (カーブフィット) をクリックし、「OK」をクリックします。成長曲線|を選択ロジスティック成長をクリックし、「OK」をクリックします。他のパラメータは調整しないでください。結果の下の適合パラメータと信頼区間を、グラフの下のデータ点と適合曲線のプロットを観察します。
7. L-RNAのクロスキラルコピー
- 20 μM D-RNA 27.3t クロスキラルポリメラーゼ、2 μM 5′-フルオレセイン標識 L-RNA プライマー、4 μM ビオチン化 L-RNA ハンマーヘッド テンプレート、および 20 μM の L-RNA pppCUG、pppAUG、pppAGG、および pppCGC の各 20 μM を含む RNA 溶液 10 μL を、50 mM Tris、pH 8.3 の 10 μL で調製します。RNA を 90 °C に 1 分間加熱し、PCR サーモサイクラーで 0.2 °C/s で 23 °C まで冷却してアニーリングします。ポリメラーゼ、プライマー、およびテンプレートの詳細については、 材料表 を参照してください。
- RNA 溶液を 17 °C でインキュベートし、10 μL の 2x 開始バッファー (400 mM MgCl2、500 mM NaCl、および 50 mM Tris、pH 8.3) を加えて反応を開始します。反応のすべての成分の最終濃度が、10 μM ポリメラーゼ、1 μM プライマー、2 μM テンプレート、10 μM 各トリヌクレオチド 5'-三リン酸、200 mM MgCl2、250 mM NaCl、および 50 mM トリス、pH 8.3 であることを確認します。
- 反応が進行するにつれて、10 μLのアリコートを取り、5 μLの0.5 M EDTA、pH 8でクエンチする。
- クエンチした各反応サンプルに、0.05%中性洗剤を含むTEバッファー中の1 M NaCl 10 μLに懸濁した0.1 mgのストレプトアビジン被覆磁気ビーズ(20 pmolビオチン-オリゴヌクレオチド結合能)を加え、ビオチン化テンプレート を介して 3量体延長プライマーを捕捉し、振とうしながら室温で30分間インキュベートします。
- ビーズをビーズ捕捉磁石に捕捉し、液体を除去して廃棄し、50 μL の洗浄液 (0.05% 中性洗剤を含む TE 中の 250 mM NaCl) を加えます。ビーズを混ぜ合わせ、もう一度捕まえて、洗浄液を取り除きます。もう一度繰り返します。
- ビーズから伸長プライマーを溶出するには、50 μL の溶出溶液 (0.05% 中性洗剤を含む 25 mM NaOH) を加え、ビーズを混合します。ビーズを再回収し、溶出溶液を除去し、100mMトリス(pH7.5)でクエンチし、エタノールで沈殿させた。
- 分析用ポリアクリルアミド/8 M 尿素/1x TBEゲルを、ゲルプレートおよびスタンドの適切なプロトコールに従って調製します。キャストゲルとプレートをゲルスタンドに組み立て、リザーバを1x TBEで満たし、40W(または異なるゲルプレートとスタンドに適宜)で30分間プリランします。
- 沈殿したRNAを10 μLのホルムアミドゲルローディングバッファーに溶解し、ホルムアミドゲルローディングバッファーに0.5 μMで未反応末端標識プライマーを調製した。サンプルを80°Cに加熱し、各ウェルにサンプルの5μLを負荷し、ゲルを40Wで約40分間(または異なるゲルプレートおよびスタンドに適宜)流す。
- ゲルプレートをスタンドから取り出し、蛍光/燐光ゲルスキャナーを使用してスキャンし、クロスキラルL-RNA伸長生成物を視覚化します。
結果
オリゴヌクレオチドは、ホスホロアミダイトおよび自動DNA/RNA合成装置に適した標準プロトコールを使用して合成し、元のプラスチック合成カラムで固体支持体から生成物オリゴヌクレオチドを抜き去り、5'末端ジメトキシトリチル基を除去して遊離の5'-ヒドロキシルを得る(セクション1)。この実証で使用したすべてのオリゴヌクレオチドは、固体支持体として1,000 Å制御細孔ガラス(CPG)樹脂を使用して調製し、0.2または1 μmoleスケールで実施しました。シンセサイザーカラム、樹脂、試薬、およびホスホロアミダイトの代表例は、 材料の表に提供される。より大規模な反応の場合、後続のステップで使用される量と時間を調整する必要があるかもしれません。
トリリン酸化反応は、材料表に記載されている標準的な市販の成分を使用して、特注の反応チャンバ(図2、セクション3)でオンカラムで実施され、図1(セクション4)28に示されているスキームに従います。トリリン酸化中は条件を厳密に無水に保ち、すべての溶媒と試薬を事前にモレキュラーシーブで調製し、使用前に完全に乾燥させることが不可欠です(セクション2)。トリリン酸化が起こるのに通常2時間かかり、その後、洗浄および乾燥されたカラムは、標準的なオリゴヌクレオチドの脱保護および精製手順に従って処理することができる(セクション5)。
脱保護後、オリゴヌクレオチド三リン酸を変性ポリアクリルアミドゲル電気泳動(PAGE)によって精製し、ゲルから切り出して溶出することができるUVバックシャドウイングによって単一の主要な生成物バンドを示す。 図3A、BにDNAトリヌクレオチド5ʹ-三リン酸、pppAAAおよびpppCCC、およびL-RNAトリヌクレオチド5ʹ-三リン酸pppGAAについて示されているように、5'-三リン酸生成物は、短いオリゴヌクレオチドの反応側生成物から容易に分離される。AAAおよびCCC DNA三量体の5'-ヒドロキシルおよび5'-三リン酸生成物の両方を摘出し、質量分析によって同定し、それに対応して 図3Aに標識した。追加のバンドは、AAA DNA三量体について見られるように、一般に、回収および同定するのに十分な物質を含まない。しかしながら、これらのバンドの存在は、以下で説明するように、典型的には5'-二リン酸、一リン酸、およびH-ホスホネート副生成物を表す未精製反応生成物(図3C)中の追加の生成物質量と相関する。
PAGE精製後、より大きなオリゴヌクレオチドを、破砕および浸漬法42 およびそれに続くエタノール沈殿を用いて溶出することができる。しかしながら、15nt未満のオリゴヌクレオチドはエタノールで効率的に沈殿させることができず、したがって、ゲル溶出のための修飾手順を必要とする(ステップ5.11.3)。 材料表 に記載されている使い捨てサイズ除外欄は、10 ntより長いオリゴヌクレオチドでの使用についてのみ評価されています。しかし、三量体ほど短いオリゴヌクレオチドは、製造業者の推奨プロトコルを使用して効果的に脱塩できることを見出した。それにもかかわらず、短いオリゴヌクレオチドを脱塩する場合(ステップ5.6および5.11.3のように)、カラム溶出液をフラクションで収集し、製品フラクションをUV-Vis分光光度計を使用した260nmの吸光度で同定することが推奨されます。より短いオリゴヌクレオチド用に最適化されたサイズ除外カラムは、代替選択肢として 材料表 に用意されています。精製後のオリゴヌクレオチド合成の1μmoleスケールからの最終収率は50〜300nmolである。
トリリン酸化は質量分析によって確認することができ、トリリン酸化生成物は5'-ヒドロキシルオリゴヌクレオチドよりも質量+239.94Da大きいが、5'-ジリン酸および一リン酸(+159.96および+79.98Da)に対応する物質の存在がしばしば観察される。5'-OH質量から質量+63.98Daの5'-H-ホスホネート副生成物も観察される可能性があり、この生成物の高レベルは、トリリン酸化中の状態が十分に無水ではなかったことを示している。精製の前に、脱保護されたオリゴヌクレオチドは通常、これらすべての生成物(図3C)を示すが、精製された物質は、5'-ジおよびモノリン酸とともに5'-三リン酸生成物に対応するピークを示す(図3D、E)。
質量分析法だけでは、イオン化の微分速度およびイオン化中の三リン酸の断片化のために、典型的には5'-三リン酸純度の厳密な測定値は得られない。最終生成物の純度を測定するには、特に長いオリゴヌクレオチドの場合、逆相液体クロマトグラフィーおよびタンデムESI-MS(RP-LC/ESI-MS)を推奨します。RP-LC/ESI-MS による D-RNA 5'-三リン酸 pppACGAGG および pppGAGACCGCAACUUA の分析 (それぞれ図 4A,B) は、より長いオリゴヌクレオチド上に存在する場合、これら 2 つの種を分離することが困難であるため、20% の 5'-二リン酸を含む典型的な最終生成物純度を示しています。
合成5'-三リン酸オリゴヌクレオチドは、典型的には、生化学的研究において酵素的に調製された材料と同程度またはそれ以上に機能する。セクション6では、例として、合成またはインビトロ転写によって調製された5'−三リン酸14nt RNA基質を、RNA触媒自己複製反応14、15、43、44、45において比較した。リボザイムEは、基質AとBの接合を触媒し、指数関数的成長が可能な自己触媒反応においてEの新しいコピーを生成する(図5A)。Eおよび32P標識A成分をインビトロ転写によって調製し、そしてトリリン酸化基質Bを、上述のように合成的に、またはインビトロ転写によって調製した14。自己複製反応の進行は、変性PAGEによって分析され、蛍光/燐光ゲルスキャナを介して定量された定期的なサンプルを採取することによってモニターされた。得られたデータは、ロジスティック成長関数に適合し、転写された基質または合成B基質のいずれかが指数関数的成長を支持するが、合成Bはわずかに多くの生成物を与えることを明らかにした(図5B)。この結果は、in vitro転写によって調製されたRNAの5'末端における組成の不均一性を反映している可能性がある23,24。
化学的三リン酸化はまた、 インビトロ または細胞内で生物学的に調製することができないオリゴヌクレオチド三リン酸の合成を可能にする。セクション7では、セクション1〜5のように調製された天然D-RNAの鏡像異性体であるL-RNAからなる非生物学的オリゴヌクレオチド三リン酸を、短いL-RNAオリゴヌクレオチド5'-三リン酸からのより長いL-RNA産物の鋳型指向重合を配列一般の様式で触媒するD-RNA「クロスキラル」ポリメラーゼリボザイム27.3t(図6A)の基質として使用した。一例として、リボザイムはハンマーヘッド自己切断モチーフのL-RNAバージョンを合成することができる(図6B)18。精製されたL-RNAトリヌクレオチド三リン酸をフルオレセイン標識L-RNAプライマーおよびL-RNAテンプレート(図6C)と組み合わせ、クロスキラルリガーゼと反応させた。反応の過程でサンプルをPAGEで分析し、蛍光/燐光ゲルスキャナーを使用して画像化し、テンプレートによってコードされたハンマーヘッドリボザイムのL-RNAバージョンの合成を実証しました(図6D)。
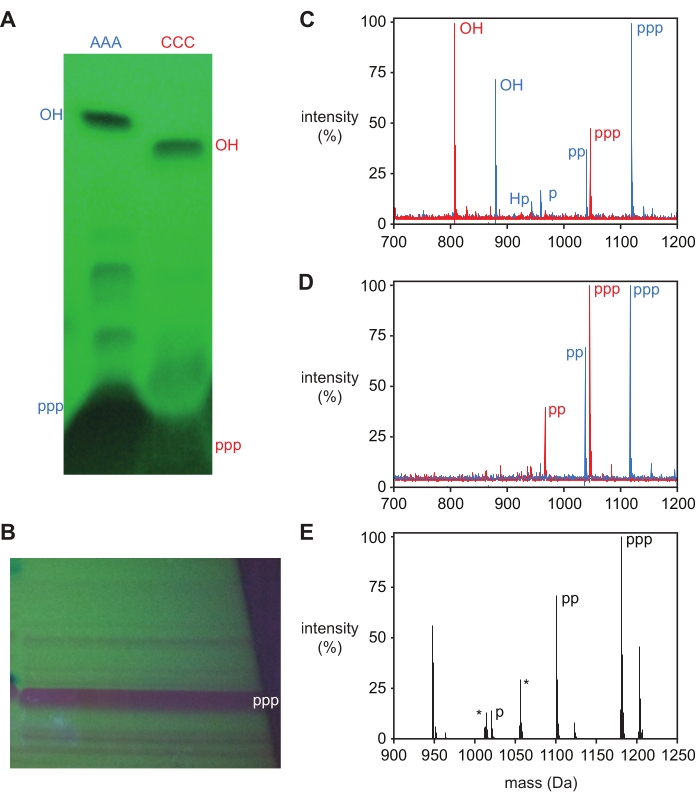
図3:トリヌクレオチド5'-三リン酸の精製 (A)DNAトリヌクレオチドトリデオキシアデノシン(AAA、青)およびトリデオキシシチジン(CCC、赤)のトリリン酸化のPAGE分析(UVバックシャドーイングによって視覚化)は、マイナーな副産物を視覚化するために意図的に過負荷をかける。5'-三リン酸生成物(ppp)および5'-ヒドロキシル(OH)出発物質の両方を切除し、MALDI-MSによって同定した。(B)L-RNAトリヌクレオチドGAAのトリリン酸化の分取PAGEを、主要産物バンドを切除し、ESI-MSによって5ʹ-三リン酸(ppp)として同定した。(C)(A)からの脱保護後の粗反応生成物及び(D)精製品のMALDI−MS。5ʹ-三リン酸(ppp;pppAAAは1,119Da、観測値1,118Da;pppCCCは1,047Da、観測値1,046Da);5ʹ-二リン酸(pp)、5'-一リン酸(p)、5'-ヒドロキシル(OH)、および5'-H-ホスホネート(Hp)が標識されている。(e)(B)から単離された5'−三リン酸生成物の直接注入ESI−MSからのデコンボリューションマススペクトル、標識された同定されたピークを有する(予想1,181.6Da、観測された1,181.0Da)。5ʹ-二リン酸(pp)生成物も観察され、三リン酸生成物と二リン酸生成物(+22Da)の両方のナトリウムイオンピークも観察される。一般的な汚染物質ピークは、アスタリスクでラベル付けされています。比較を容易にするために、マススペクトルは各スペクトルで測定された最も高い強度に正規化され、その値に対する百分率として報告される。略語: PAGE = ポリアクリルアミドゲル電気泳動;MALDI-MS = マトリックス支援レーザー脱離/イオン化;ESI-MS = エレクトロスプレーイオン化質量分析。この図の拡大版を表示するには、ここをクリックしてください。
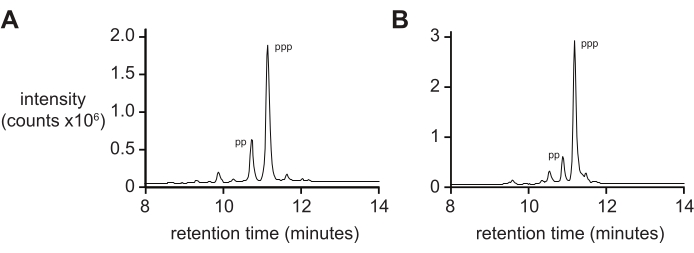
図4:6 ntおよび14 nt D-RNAオリゴヌクレオチド三リン酸の分析RP-LCである。 (A) 5ʹ-pppACGAGG-3ʹおよび(B) 5ʹ-pppGAGACCGCAACUUA-3ʹ.タンデムESI-MSは、両方の主要なピーク(〜70%)を5'-三リン酸(ppp)と同定し、5'-二リン酸(pp)の量が少ない。略語:RP-LC=逆相液体クロマトグラフィー;nt = ヌクレオチド;ESI-MS = エレクトロスプレーイオン化質量分析。 この図の拡大版を表示するには、ここをクリックしてください。
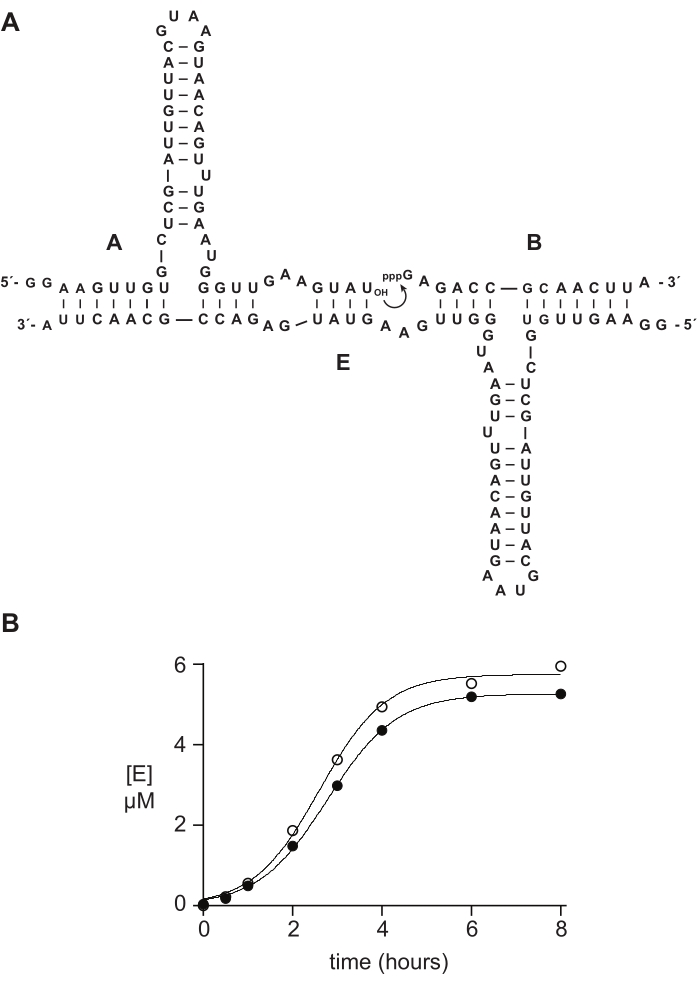
図5:化学合成またはインビトロ転写によって調製されたオリゴヌクレオチド5ʹ-三リン酸基質の比較(A)自己複製リボザイムEは、RNA Aおよび5'-トリリン酸化RNA Bを連結する。 (B)合成(白丸)またはインビトロ転写(塗り丸)のいずれかで、10μM Aおよび10μM Bを用いた自己複製反応の比較。(B)データはロジスティック成長方程式に当てはまりました:[E] = a / (1 + b e-ct)、ここでaは最終収率、bはシグモイシティの程度、cは指数関数的な成長率です。2つの反応の成長率は1.14時間-1で同じでしたが、合成Bの反応では最終範囲が10%高かったため、この図のより大きなバージョンを表示するにはここをクリックしてください。
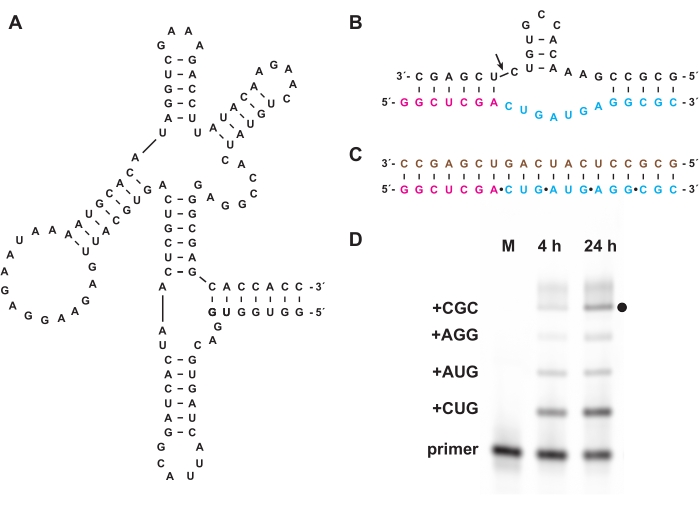
図6:リボザイムによるクロスキラルL-RNA重合(A)L-RNAの鋳型依存的ライゲーションを触媒するD-RNA 27.3tポリメラーゼリボザイム。(b)27.3tで合成されたL-RNA産物がハンマーヘッドエンドヌクレアーゼモチーフの一部を形成している。3tで触媒されるL-RNA重合は、ビオチン化L-RNA鋳型(茶色)、末端標識L-RNAプライマー(マゼンタ)、および4つのL-RNAトリヌクレオチド三リン酸(シアン)を用いて、合成的に調製した。(D)(B)の伸長産物のPAGE分析は、4時間および24時間における、全長産物(黒点)までの各トリヌクレオチドの組み込みを示す。未反応のL-RNAプライマーは、参照マーカーとして含まれる。略語: PAGE = ポリアクリルアミドゲル電気泳動;M = 参照マーカー。この図の拡大版を表示するには、ここをクリックしてください。
ディスカッション
ここで説明するトリリン酸化手順は、標準的なホスホロアミダイト化学を用いたオリゴヌクレオチド合成と広く適合性がある。ヌクレオシドホスホロアミダイトは、亜リン酸エステル上の標準β-シアノエチル、および核酸塩基の環外アミン上のイソブチリル、ジメチルホルムアミジル、アセチル、フェノキシアセチル、または4-イソプロピルフェノキシアセチル基を含む、AMA39における迅速な脱保護と相溶する塩基不安定性保護基を有するべきである。リボース2'-ヒドロキシル基は、t-ブチルジメチルシリル(TBDMS)またはトリイソプロピルシリルオキシメチル(TOM)40,41のいずれかのシリル保護基によって保護されるべきである。塩基不安定性ピバロイルオキシメチル(PivOM)基はまた、化学的三リン酸化30と相溶性であることが報告されている。
合成オリゴヌクレオチド28、29、30、31、32、33、34、35の化学的三リン酸化について複数の方法が記載されている。我々は、ルートヴィヒ・エクスタイン試薬37を用いた三リン酸化が最もアクセスしやすいものの1つであり、試薬の特殊な合成や特殊な装置を必要としないことを発見しました。この方法で調製されたオリゴヌクレオチド5'-三リン酸は、この「ミラーイメージ」核酸の鋳型依存的な合成および複製を達成するために酵素的にアクセスできないL-RNAオリゴヌクレオチド三リン酸の使用を含む、RNAリガーゼリボザイムの基質として日常的に使用されている14,16,17,18.この方法は、脊椎動物における自然免疫応答の強力な活性化因子である小さな5'-トリリン酸化ステムループRNAの調製にも適している6,7。
ルートヴィヒ・エクスタイン試薬であるサリチルホスホロクロリダイト37は、水に対する反応性が高く、オリゴヌクレオチドカラムにロードする前に試薬を溶解するときに導入された水を効果的に除去します。しかし、この時点以降、5'-ホスフィチル化オリゴヌクレオチドは、ピロリン酸よりも系に導入された水と優先的に反応し、後処理後に5'-H-ホスホネート副生成物を形成する28,37,38。トリリン酸化試薬とトリリン酸化反応チャンバの慎重な調製は、この副生成物が形成されないようにします。溶媒乾燥のために、タイプ4 Åモレキュラーシーブは、ほとんどのオリゴヌクレオチド合成試薬会社によって、さまざまなブランド名でほとんどの有機溶媒と互換性のあるテフロン(登録商標)バッグにあらかじめ包装されて販売されています。無水雰囲気下でグローブボックス内で三リン酸化を行うなどの追加の予防措置は、一般に必要ではない。
5'-ホスフィチル化オリゴヌクレオチドとTBAPとの反応は、環状5'-トリメタホスファイト中間体を形成し、次いで、オリゴヌクレオチド合成酸化剤溶液(水/ピリジン/THF中のヨウ素)を用いて環状5'-トリメタリン酸に酸化される。市販の酸化剤溶液は様々な量のヨウ素を使用するので、三リン酸への完全な酸化を確実にするために高い0.1 Mヨウ素濃度を使用することが不可欠であることに留意すべきである。環状生成物は、同じ溶液37中で最終的な直鎖状5'-三リン酸に加水分解され、水以外の求核剤による線状化が望まれる場合、代替の無水酸化剤溶液を使用しなければならない(用途については下記参照)33。しかしながら、残留環状トリメタリン酸は、オリゴヌクレオチドのその後のアルカリ脱保護中に線形化される。環状5'-トリメタリン酸の加水分解は、分枝状の三リン酸37,46ではなく、直鎖状のもののみを生じる。
オリゴヌクレオチドの脱保護は、典型的には、5'-トリリン酸化を収容するように修飾される必要はないが、いくつかの予防措置がとられるべきである。三リン酸はアルカリ性条件への短時間の曝露に対して比較的安定であるが、三リン酸を必要以上に長くAMAに曝露しないように注意すべきである。アンモニアまたはAMA中で65°Cで10分以上長時間処理する必要がある保護基は避けるべきです。室温でアンモニア中で2時間などのより穏やかな処理は、他のホスホロアミダイト保護基と相溶する場合に許容される。シリル保護合成RNAオリゴヌクレオチドの一般的で高速な脱保護方法は、トリエチルアミン三フッ化水素塩および高温を使用する47;しかし、RNAの5'-三リン酸を調製する際には、長時間の酸性条件が三リン酸加水分解を促進することが判明しているため、これは避けるべきである31,32。
分取PAGEは、5'-トリリン酸化オリゴヌクレオチドの脱保護後精製のための最も簡単で信頼性の高い方法であることが証明されています(図3および図4)。しかしながら、分取逆相HPLCは、トリリン酸化生成物を精製するためにも使用することができる。5'-二リン酸および5'-一リン酸生成物の存在は、質量分析によって三リン酸化を検証する際に日常的に観察される。我々は、特に装置がオリゴヌクレオチドの分析に最適化されていない場合、化学合成または転写によって調製された高純度材料からの質量分析中に5'-三リン酸断片化を観察した。それにもかかわらず、RP-LC分析は、5'-二リン酸側生成物の10%〜20%が、より長い5'-トリリン酸化オリゴヌクレオチド中に存在することをしばしば示している(図4)。トリブチルアンモニウムピロホスフェートの市販の調製物は、20%のモノホスフェートで汚染される可能性があり、トリリン酸化中に副生成物として5'-二リン酸を生成する30,31。この試薬を社内で慎重に調製することにより、はるかに純粋なTBAPストック31を産出することができる。しかしながら、我々は、TBAPの市販供給源を用いてトリリン酸化されたオリゴヌクレオチドが、in vitro転写によって調製された物質と比較して、酵素反応において基質として使用された場合(図5B)に依然として同等以上の反応性を示すことを見出した。
ルートヴィヒ・エクシュタイン試薬を用いたオリゴヌクレオチド三リン酸化の注目すべきさらなる使用の1つは、環状トリメタホスファイト中間体33を利用する。無水条件下でのオリゴヌクレオチド酸化によく使用されるヘキサン中で 1Mt-ブチルパーオキサイドを用いてその後の酸化工程を行うと、開環加水分解することなく亜リン酸の酸化が起こり、環状トリメタリン酸を生じる。次いで、この中間体を第一級アミンまたはアルコール求核剤と反応させて、γリン酸で修飾した5'-三リン酸塩を生成することができる。これらの修飾には、ホスホラミデート結合によって連結された親油性タグの付加が含まれ、これはRP−LCによる迅速な三リン酸特異的精製を容易にし、続いて三リン酸33からのタグの酸性加水分解を含む。γリン酸位置における蛍光修飾はまた、リボザイム触媒ライゲーション反応15、33のためのリアルタイム蛍光レポーターとして使用するために導入され得る。
開示事項
著者らは、競合する金銭的利益はないと宣言している。
謝辞
著者らは、化学的三リン酸化反応のベストプラクティスに関する有益な議論をしてくれたGreg Springsteen、Natasha Paul、Charles Olea, Jr.、Jonathan Sczepanski、Katrina Tjhung、およびGerald Joyceに有益なコメントを寄せて感謝している。この研究は、国立科学財団からの助成金MCB 2114588によって支援されました。
資料
| Name | Company | Catalog Number | Comments |
| 0.22 µm polyethersulfone syringe filter | MilliporeSigma | SLMP025SS | Syringe filter for removing crushed polyacrylamide gel particles (Section 5) |
| 0.22 µm PTFE syringe filter | MilliporeSigma | SLLG013SL | Syringe filter for removing CPG resin (Section 5) |
| 1 mL plastic syringes | ThermoFisher Scientific | 14-823-434 (BD 309659) | Components of triphosphorylation apparatus (sections 2–4) |
| 1,4-Dioxane, anhydrous | MilliporeSigma | 296309 | Triphosphorylation solvent (sections 2–4) |
| 2-Chloro-4H-1,3,2-benzodioxaphosphorin-4-one, Salicyl Phosphorochloridite (SalPCl) | MilliporeSigma | 324124 | Triphosphorylation reagent (sections 2–4) |
| 30 mL glass bottles | MilliporeSigma | 23232 | Bottles for preparing triphosphorylation solvents and TBAP solution (section 2) |
| 3-way Stopcock, polycarbonate/polypropylene | Bio-Rad Laboratories | 7328103 | Component of triphosphorylation apparatus (sections 2–4) |
| 40% acrylamide/bis-acrylamide solution, 19:1 | Bio-Rad Laboratories | 1610144 | For PAGE (sections 5–7) |
| Acetonitrile, anhydrous, 100 mL | Glen Research | 40-4050-50 | Triphosphorylation solvent (sections 2–4) |
| Ammonia-neutralizing Trap | ThermoFisher Scientific | ANT100 and ANS121 | For use with Speedvac DNA130 (section 5) |
| Ammonium persulfate (APS) | Bio-Rad Laboratories | 1610700 | For PAGE, catalyst for acrylamide polymerization (sections 5–7) |
| Aqueous ammonia, 28% | MilliporeSigma | 338818 | For preparing AMA deprotection reagent (section 5) |
| Aqueous methylamine, 40% | TCI America | TCI-M0137 | For preparing AMA deprotection reagent (section 5) |
| Automated DNA/RNA oligonucleotide synthesizer | PerSeptive Biosystems | Expedite 8909 DNA/RNA Synthesizer | any column-based synthesizer is acceptable (section 1) |
| Bead-capture magnet | ThermoFisher Scientific | 12320D | For streptavidin bead capture (section 7) |
| Bromophenol blue | Bio-Rad Laboratories | 1610404 | For PAGE urea loading buffer (section 5) |
| Deep vacuum oil pump | ThermoFisher Scientific | VLP200-115 | For use with lyophilizer (section 5) |
| Drierite dessicant, 10-20 mesh | MilliporeSigma | 737828 | Desiccant for storing triphosphorylation chemicals and equipment (sections 1–2) |
| D-RNA 27.3t cross-chiral polymerase | prepared in house18 | 5′-GGUGGUGGAC GUGAUCAUUA CGGAUCACUA ACUCGUCAGU GCAUUGAGAA GGAGAAUAAA AUGCACAUAG GUCGAAAGAC CUUAUACAAG AACUGUAUCA CCGGAGGGCG AGCACCACC-3′ | For cross-chiral ribozyme reactions (section 7) |
| D-RNA CPG solid supports, 1,000Å, prepackaged 1 µmole synthesis columns | Glen Research | 20-3404-41E, 20-3415-41E, 20-3424-41E, 20-3430-41E | representative, for D-RNA oligonucleotide synthesis (section 1) |
| D-RNA TOM-protected phosphoramidites | ChemGenes | ANP-3201, 3202, 3203, 3205 | representative, for D-RNA oligonucleotide synthesis (section 1) |
| Empty Expedite Synthesis Columns, 1µm | Glen Research | 20-0021-01 | Synthesis columns for use with Expedite DNA/RNA synthesizer (section 1) |
| EPPS, N-(2-Hydroxyethyl)piperazine-N′-(3-propanesulfonic acid), solid | MilliporeSigma | E1894 | Ribozyme reaction buffer component (section 6) |
| Ethylenediaminetetraacetic acid (EDTA), solid | MilliporeSigma | EDS | Divalent metal ion chelator for use in various buffers (sections 5–7) |
| Filters for Expedite synthesis columns | Glen Research | 20-0021-0F | Expedite-style synthesis column filters, for use with empty synthesis columns (section 1) |
| Fluorescent/phosphorescent gel scanner | Cytiva | Amersham Typhoon RGB, 29187193 | For visualizing analytical PAGE (sections 6–7) |
| Formamide, deionized | VWR Life Science | 97062 | For PAGE formamide gel loading buffer (sections 6–7) |
| Gel image quantitation software | Cytiva | ImageQuant TL | For quantifying scanned gel images (section 6) |
| Glass desiccator | MilliporeSigma | CLS3121150 | Triphosphorylation solvent storage (section 2) |
| L-RNA CPG solid supports, 1,000Å, bulk | ChemGenes | N-4691-10, N-4692-10, N-4693-10, N-4694-10 | L-RNA oligonucleotide synthesis (section 1) |
| L-RNA hammerhead template | prepared in house18 | 5′-GCGCCUCAUC AGUCGAGCC-3′ | For cross-chiral ribozyme reactions (section 7) |
| L-RNA primer | prepared in house18 | 5′-fluorescein-GGCUCGA-3′ | For cross-chiral ribozyme reactions (section 7) |
| L-RNA TOM-protected phosphoramidites | ChemGenes | OP ANP-5201, 5202, 5203, 5205 | L-RNA oligonucleotide synthesis (section 1) |
| Lyophilizer/Freeze Dryer | VirTis | Benchtop K | For concentrating oligonucleotides (section 5) |
| Magnesium Chloride Hexahydrate, solid | MilliporeSigma | M2670 | For ribozyme reactions (sections 6–7) |
| N,N-Dimethylformamide, anhydrous | MilliporeSigma | 227056 | Triphosphorylation solvent (section 2) |
| NAP-25 Desalting column (Sephadex G-25 resin) | ThermoFisher Scientific | 45000150 | Disposable gravity-flow size exclusion chromatography columns containing Sephadex G-25 resin (section 5) |
| Non-coring stainless steel needle, 20 G | ThermoFisher Scientific | 14-815-410 | Needles for piercing rubber septa (sections 2–4) |
| Oligonucleotide extinction coefficient calculator | Integrated DNA Technologies | OligoAnalyzer Tool | Nearest-Neighbor Model Short Oligonucleotide 260nm extinction coefficient calculator (section 5) |
| Oxidizer solution, 0.1 M Iodine in THF/pyridine/water | ChemGenes | RN-1456 | Triphosphorylation reagent (section 4) |
| PAGE plates | Timberrock/CBS | NGP-250-BO and NO | For PAGE (sections 5–7) |
| PAGE power supply | Bio-Rad Laboratories | PowerPac HV 1645056 | For PAGE (sections 5–7) |
| PAGE spacers and combs (analytical) | Timberrock/CBS | VGS-0725 and VGC-0714 | For PAGE (sections 6–7) |
| PAGE spacers and combs (preparative) | Timberrock/CBS | VGS-3025R and VGC-3001 | For PAGE (section 5) |
| PAGE stand | Timberrock/CBS | ASG-250 | For PAGE (sections 5–7) |
| Parafilm M | ThermoFisher Scientific | 13-374-12 (Bemis PM999) | Wax sealing film for triphosphorylation apparatus (sections 2–4) |
| PCR thermocycler | Bio-Rad Laboratories | C1000 Touch Thermalcycler | For cross-chiral ribozyme reactions (section 7) |
| PD 10 Desalting column (Sephadex G-10 resin) | MilliporeSigma | GE17-0010-01 | Disposable gravity-flow size exclusion chromatography columns containing Sephadex G-10 resin, for oligonucleotides < 15 nt (section 5) |
| Phosphor screens | Cytiva | 28956480 | For visualizing 32P-labeled RNA (section 6) |
| Phosphoramidite synthesis reagents | Glen Research | 30-3142-52, 40-4050-53, 40-4012-52, 40-4122-52, 40-4132-52, 40-4060-62 | representative, for standard RNA/DNA synthesis (section 1) |
| Polypropylene screw-cap sealable tube | MilliporeSigma | BR780752 | 1.5 mL microcentrifuge tubes with screw-cap and silicone O-ring, for safe AMA deprotection (section 5) |
| Pyridine, anhydrous | MilliporeSigma | 270970 | Triphosphorylation solvent (section 2) |
| Reverse-phase liquid chromatography/electrospray ionization mass spectrometry (RP-LC/ESI-MS) | Novatia | n/a | Commercial service for LC/MS specializing in oligonucleotides (section 5) |
| Rubber Septa (ID x OD 7.9 mm x 14 mm), white | MilliporeSigma | Z564702 | Septa for preparing triphosphorylation solvents and TBAP (section 2) |
| Self-replicator ribozyme E | prepared in house14 | 5′-GGAAGUUGUG CUCGAUUGUU ACGUAAGUAA CAGUUUGAAU GGUUGAAGUA UGAGACCGCA ACUUA-3′ | For self-replicator ribozyme reactions (section 6) |
| Self-replicator substrate A | prepared in house14 | 5′-32P-GGAAGUUGUG CUCGAUUGUU ACGUAAGUAA CAGUUUGAAU GGUUGAAGUA U-3′-OH | For self-replicator ribozyme reactions (section 6) |
| Self-replicator substrate B, transcribed | prepared in house14 | 5′-pppGAGACCGCAA CUUA-3′ | For self-replicator ribozyme reactions (section 6) |
| Small Drying Traps, 4 Å molecular sieves | ChemGenes | DMT-1975 | Drying traps for DNA/RNA synthesizer phosphoramidites and triphosphorylation reagents (sections 1–2) |
| Sodium Chloride (NaCl), solid | MilliporeSigma | S7653 | Salt for use in various buffers (sections 5–7) |
| Sodium Hydroxide (NaOH), solid | MilliporeSigma | S8045 | Salt for use in various buffers (sections 5–7) |
| Statistical data-fitting software | GraphPad | Prism | For fitting data from analytical PAGE to kinetic models (section 6) |
| Streptavidin-coated magnetic beads | ThermoFisher Scientific | 65002 | For capturing biotin-labeled RNA in cross-chiral ribozyme reactions (section 7) |
| Sucrose | MilliporeSigma | 84097 | For PAGE urea loading buffer (section 5) |
| TBE running buffer, 10x | ThermoFisher Scientific | AAJ62788K3 | For PAGE (sections 5–7) |
| Tetrabutylammonium Fluoride, 1.0 M solution in Tetrahydrofuran | Aldrich | 216143 | For removing 2′-silyl protecting groups (section 5) |
| Tetramethylethylenediamine (TEMED) | Bio-Rad Laboratories | 1610801 | For polymerizing acrylamide for PAGE (sections 5–7) |
| Tributylamine | MilliporeSigma | 90781 | Triphosphorylation reagent (section 2) |
| Tributylammonium pyrophosphate (TBAP) | MilliporeSigma | P8533 | Triphosphorylation reagent (section 2) |
| Tris base | MilliporeSigma | T6666 | Buffering agent for use in various buffers (sections 5–7) |
| TWEEN20 polysorbate detergent | MilliporeSigma | P7949 | Neutral detergent for use with magnetic beads (Section 7) |
| Urea | MilliporeSigma | U5378 | For PAGE and gel loading buffer (sections 5–7) |
| UV-Vis spectrophotometer | ThermoFisher Scientific | NanoDrop 2000, ND2000 | For measuring oligonucleotide concentrations (section 5) |
| Vacuum centrifuge | ThermoFisher Scientific | Savant Speedvac DNA130-115 Vacuum Concentrator | For removing AMA and THF (section 5) |
| Xylene cyanol | Bio-Rad Laboratories | 1610423 | For PAGE urea loading buffer (section 5) |
参考文献
- Shuman, S. What messenger RNA capping tells us about eukaryotic evolution. Nature Reviews. Molecular Cell Biology. 3 (8), 619-625 (2002).
- Pichlmair, A., et al. RIG-I-mediated antiviral responses single-stranded RNA bearing 5'-phosphates. Science. 314 (5801), 997-1001 (2006).
- Hornung, V., et al. 5'-Triphosphate RNA is the ligand for RIG-I. Science. 314 (5801), 994-997 (2006).
- Myong, S., et al. Cytosolic viral sensor RIG-I is a 5'-triphosphate-dependent translocase on double-stranded RNA. Science. 323 (5917), 1070-1074 (2009).
- Takeuchi, O., Akira, S. Pattern recognition receptors and inflammation. Cell. 140, 805-820 (2010).
- Wang, Y., et al. Structural and functional insights into 5'-ppp RNA pattern recognition by the innate immune receptor RIG-I. Nature Structural & Molecular Biology. 17 (7), 781-787 (2010).
- Goubau, D., et al. Antiviral immunity via RIG-I-mediated recognition of RNA bearing 5'-diphosphates. Nature. 514 (7522), 372-375 (2014).
- Joyce, G. F. Forty years of in vitro evolution. Angewandte Chemie. 46 (34), 6420-6436 (2007).
- Robertson, M. P., Ellington, A. D. In vitro selection of an allosteric ribozyme that transduces analytes to amplicons. Nature Biotechnology. 17 (1), 62-66 (1999).
- Robertson, M. P., Hesselberth, J. R., Ellington, A. D. Optimization and optimality of a short ribozyme ligase that joins non-Watson-Crick base pairings. RNA. 7 (4), 513-523 (2001).
- Hesselberth, J. R., Robertson, M. P., Knudsen, S. M., Ellington, A. D. Simultaneous detection of diverse analytes with an aptazyme ligase array. Analytical Biochemistry. 312 (2), 106-112 (2003).
- Lam, B. J., Joyce, G. F. Autocatalytic aptazymes enable ligand-dependent exponential amplification of RNA. Nature Biotechnology. 27 (3), 288-292 (2009).
- Lam, B. J., Joyce, G. F. An isothermal system that couples ligand-dependent catalysis to ligand-independent exponential amplification. Journal of the American Chemical Society. 133 (9), 3191-3197 (2011).
- Olea, C., Horning, D. P., Joyce, G. F. Ligand-dependent exponential amplification of a self-replicating L-RNA enzyme. Journal of the American Chemical Society. 134 (19), 8050-8053 (2012).
- Olea, C., Joyce, G. F. Real-Time Detection of a Self-Replicating RNA Enzyme. Molecules. 21 (10), (2016).
- Sczepanski, J. T., Joyce, G. F. A cross-chiral RNA polymerase ribozyme. Nature. 515 (7527), 440-442 (2014).
- Tjhung, K. F., Sczepanski, J. T., Murtfeldt, E. R., Joyce, G. F. RNA-catalyzed cross-chiral polymerization of RNA. Journal of the American Chemical Society. 142 (36), 15331-15339 (2020).
- Bare, G. A. L., Joyce, G. F. Cross-chiral, RNA-catalyzed exponential amplification of RNA. Journal of the American Chemical Society. 143 (45), 19160-19166 (2021).
- Milligan, J. F., Groebe, D. R., Witherell, G. W., Uhlenbeck, O. C. Oligoribonucleotide synthesis using T7 RNA polymerase and synthetic DNA templates. Nucleic Acids Research. 15 (21), 8783-8798 (1987).
- Chelliserrykattil, J., Ellington, A. D. Evolution of a T7 RNA polymerase variant that transcribes 2'-O-methyl RNA. Nature Biotechnology. 22 (9), 1155-1160 (2004).
- Ibach, J., et al. Identification of a T7 RNA polymerase variant that permits the enzymatic synthesis of fully 2′-O-methyl-modified RNA. Journal of Biotechnology. 167 (3), 287-295 (2013).
- Esvelt, K. M., Carlson, J. C., Liu, D. R. A system for the continuous directed evolution of biomolecules. Nature. 472 (7344), 499-503 (2011).
- Pleiss, J. A., Derrick, M. L., Uhlenbeck, O. C. T7 RNA polymerase produces 5' end heterogeneity during in vitro transcription from certain templates. RNA. 4 (10), 1313-1317 (1998).
- Schenborn, E. T., Mierendorf, R. C. A novel transcription property of SP6 and T7 RNA polymerases: dependence on template structure. Nucleic Acids Research. 13 (17), 6223-6236 (1985).
- Martin, C. T., Muller, D. K., Coleman, J. E. Processivity in early stages of transcription by T7 RNA polymerase. Biochemistry. 27 (11), 3966-3974 (1988).
- Gholamalipour, Y., Karunanayake Mudiyanselage, A., Martin, C. T. 3' end additions by T7 RNA polymerase are RNA self-templated, distributive and diverse in character-RNA-Seq analyses. Nucleic Acids Research. 46 (18), 9253-9263 (2018).
- Vasilyev, N., Serganov, A. Preparation of short 5′-triphosphorylated oligoribonucleotides for crystallographic and biochemical studies. Nucleic Acid Crystallography: Methods and Protocols. , 11-20 (2016).
- Lebedev, A. V., Koukhareva, I. I., Beck, T., Vaghefi, M. M. Preparation of oligodeoxynucleotide 5'-triphosphates using solid support approach. Nucleosides, Nucleotides & Nucleic Acids. 20 (4-7), 1403-1409 (2001).
- Paul, N., Springsteen, G., Joyce, G. F. Conversion of a ribozyme to a deoxyribozyme through in vitro evolution. Chemistry & Biology. 13 (3), 329-338 (2006).
- Zlatev, I., et al. Efficient solid-phase chemical synthesis of 5'-triphosphates of DNA, RNA, and their analogues. Organic Letters. 12 (10), 2190-2193 (2010).
- Zlatev, I., Manoharan, M., Vasseur, J. -J., Morvan, F. Solid-phase chemical synthesis of 5'-triphosphate DNA, RNA, and chemically modified oligonucleotides. Current Protocols in Nucleic Acid Chemistry. , Chapter 1, Unit1.28 (2012).
- Zlatev, I., et al. Automated parallel synthesis of 5'-triphosphate oligonucleotides and preparation of chemically modified 5'-triphosphate small interfering RNA. Bioorganic & Medicinal Chemistry. 21 (3), 722-732 (2013).
- Goldeck, M., Tuschl, T., Hartmann, G., Ludwig, J. Efficient solid-phase synthesis of pppRNA by using product-specific labeling. Angewandte Chemie. 53 (18), 4694-4698 (2014).
- Sarac, I., Meier, C. Efficient automated solid-phase synthesis of DNA and RNA 5′-triphosphates. Chemistry-A European Journal. 21 (46), 16421-16426 (2015).
- Sarac, I., Meier, C. Solid-phase synthesis of DNA and RNA 5'-O-triphosphates using cycloSal chemistry. Current Protocols in Nucleic Acid Chemistry. 64 (1), 4-67 (2016).
- Perez, J. T., et al. Influenza A virus-generated small RNAs regulate the switch from transcription to replication. Proceedings of the National Academy of Sciences of the United States of America. 107 (25), 11525-11530 (2010).
- Ludwig, J., Eckstein, F. Rapid and efficient synthesis of nucleoside 5'-0-(1-thiotriphosphates), 5'-triphosphates and 2',3'-cyclophosphorothioates using 2-chloro-4H-1,3,2-benzodioxaphosphorin-4-one. The Journal of Organic Chemistry. 54 (3), 631-635 (1989).
- Gaur, R. K., Sproat, B. S., Krupp, G. Novel solid phase synthesis of 2'-o-methylribonucleoside 5'-triphosphates and their α-thio analogues. Tetrahedron Letters. 33 (23), 3301-3304 (1992).
- Reddy, M. P., Hanna, N. B., Farooqui, F. Fast cleavage and deprotection of oligonucleotides. Tetrahedron Letters. 35 (25), 4311-4314 (1994).
- Hogrefe, R. I., McCaffrey, A. P., Borozdina, L. U., McCampbell, E. S., Vaghefi, M. M. Effect of excess water on the desilylation of oligoribonucleotides using tetrabutylammonium fluoride. Nucleic Acids Research. 21 (20), 4739-4741 (1993).
- Pitsch, S., Weiss, P. A., Jenny, L., Stutz, A., Wu, X. Reliable chemical synthesis of oligoribonucleotides (RNA) with 2′-O-[(Triisopropylsilyl)oxy]methyl(2′-O-tom)-protected phosphoramidites. Helvetica Chimica Acta. 84 (12), 3773-3795 (2001).
- Green, M. R., Sambrook, J. Isolation of DNA fragments from polyacrylamide gels by the crush and soak method. Cold Spring Harbor Protocols. 2019 (2), (2019).
- Paul, N., Joyce, G. F. A self-replicating ligase ribozyme. Proceedings of the National Academy of Sciences of the United States of America. 99 (20), 12733-12740 (2002).
- Lincoln, T. A., Joyce, G. F. Self-sustained replication of an RNA enzyme. Science. 323 (5918), 1229-1232 (2009).
- Robertson, M. P., Joyce, G. F. Highly efficient self-replicating RNA enzymes. Chemistry & Biology. 21 (2), 238-245 (2014).
- Singh, J., et al. Synthesis of modified nucleoside oligophosphates simplified: fast, pure, and protecting group free. Journal of the American Chemical Society. 141 (38), 15013-15017 (2019).
- Bellon, L. Oligoribonucleotides with 2'-O-(tert-butyldimethylsilyl) groups. Current Protocols in Nucleic Acid Chemistry. , Chapter 3., Unit 3.6 (2001).
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請さらに記事を探す
This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved