Method Article
Trifosforilación química de oligonucleótidos
En este artículo
Resumen
Los oligonucleótidos 5′-trifosfatos son componentes ubicuos en vías biológicas esenciales y han visto un uso creciente en aplicaciones biotecnológicas. Aquí, describimos técnicas para la síntesis y purificación rutinaria de oligonucleótidos 5′-trifosfatos, a partir de oligonucleótidos preparados por técnicas de síntesis automatizadas estándar.
Resumen
El 5′-trifosfato es una modificación esencial del ácido nucleico que se encuentra a lo largo de toda la vida y se utiliza cada vez más como una modificación funcional de los oligonucleótidos en biotecnología y biología sintética. Los oligonucleótidos 5′-trifosfatos se han preparado históricamente in vitro por métodos enzimáticos. Sin embargo, estos métodos se limitan a los oligonucleótidos naturales de ARN, tienen fuertes preferencias de secuencia y tienden a producir productos heterogéneos. Los nuevos métodos de trifosforilación química complementan tanto el costo reducido de la síntesis automatizada de oligonucleótidos por la química de la fosforamida como la amplia gama de modificaciones de nucleótidos ahora disponibles. Por lo tanto, la síntesis de trifosfatos de oligonucleótidos de secuencia y longitud arbitrarias, y que opcionalmente contienen varias modificaciones no naturales, es ahora accesible.
Este artículo presenta los métodos y técnicas apropiadas para la trifosforilación química de oligonucleótidos utilizando fosforocloridina de salicil y pirofosfato. Este método utiliza reactivos disponibles comercialmente, es compatible con la mayoría de los oligonucleótidos preparados por métodos estándar de síntesis en fase sólida y se puede completar en 2 h después de la síntesis de oligonucleótidos, antes de la desprotección y purificación. Se han demostrado dos usos de oligonucleótidos trinucleótidos químicamente trifosforilados como sustratos para enzimas catalíticas de ARN, incluida la síntesis de una versión de imagen especular de la ribozima hammerhead a partir de trifosfatos de L-ARN no biológicos.
Introducción
La forma 5′ trifosforilada de ARN es omnipresente en biología, ya que es generada por la transcripción de ARN en todos los dominios de la vida y por la replicación de ARN durante el ciclo de vida de muchos virus de ARN. Estos trifosfatos sirven como sustrato para la formación de ARNm con tapa de 7-metilguanilato en eucariotas y, por lo tanto, desempeñan un papel esencial en la expresión de proteínas1. En contraste, el trifosfato se retiene en bacterias y virus; por lo tanto, los ARN 5′-trifosfatos son reconocidos por reguladores de respuesta de inmunidad innata en eucariotas 2,3,4,5,6,7. Fuera de la biología, una gran cantidad de ribozimas de ARN ligasa han sido evolucionadas para usar el 5′-trifosfato in vitro8 y modificadas para su uso en ensayos de diagnóstico 9,10,11,12,13,14,15. Una de estas ribozimas se puede utilizar para la síntesis dependiente de plantillas de L-ARN, el enantiómero no biológico de "imagen especular" del ARN D natural, a partir de pequeños oligonucleótidos de L-ARN 5′-trifosfatos 16,17,18. La preparación rutinaria de oligonucleótidos trifosforilados de secuencia variable y composición de la columna vertebral es esencial para investigar estos sistemas.
El método más común y accesible para preparar ARN 5′-trifosfatos en el laboratorio es la transcripción in vitro. Sin embargo, el ARN producido por este método está restringido en secuencia y tamaño por los requisitos de promotor y sustrato de la enzima ARN polimerasa. La ARN polimerasa T7 y sus derivados especializados son las polimerasas más comunes utilizadas para este fin 19,20,21,22. El ARN transcrito in vitro preparado con estas enzimas debe iniciarse con una purina 5′-terminal y está fuertemente sesgado hacia las purinas en los primeros 10 nucleótidos23,24. Además, la incorporación enzimática de nucleótidos modificados por la base o la columna vertebral es, en el mejor de los casos, ineficiente y más a menudo imposible con las polimerasas naturales, lo que limita la oportunidad de producir oligonucleótidos 5′-trifosfatos compuestos de cualquier cosa menos D-ARN natural. Otro factor limitante es que el ARN generado por la transcripción in vitro puede contener una heterogeneidad sustancial de 5′ y 3′ y se produce como productos extremadamente heterogéneos cuando es inferior a 20 nt 23,24,25,26,27.
Por el contrario, la trifosforilación química de oligonucleótidos preparados por síntesis de fosforamidadito en fase sólida 28,29,30,31,32,33,34,35 puede utilizarse para preparar oligonucleótidos 5′-trifosfatos de 3-50 nt de largo, de cualquier secuencia. Además, se puede agregar una amplia gama de modificaciones de ácidos nucleicos accesibles a la síntesis de fosforamidas a los oligonucleótidos antes de la trifosforilación 5′-14,15,16,17,18,29,36. Muchos de estos métodos utilizan el reactivo fosfocloridita de fosfocloridita de fosfilofilación, que fue desarrollado por Ludwig y Eckstein para la trifosforilación en fase de solución de mononucleósidos37. La trifosforilación de oligonucleótidos con este reactivo se logra en la fase sólida por fosfilación del oligonucleótido 5′-hidroxilo, conversión al trifosfato por reacción con pirofosfato y oxidación, seguido de procedimientos estándar para la escisión del oligonucleótido del soporte sólido, desprotección y purificación (Figura 1)28.
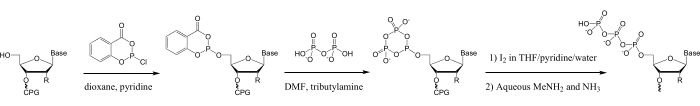
Figura 1: Esquema para la trifosforilación de oligonucleótidos sintéticos. En el primer paso, el oligonucleótido 5ʹ-hidroxilo se fosfitila con SalPCl. En el siguiente paso, el fosfito de 5ʹ-salicil se reacciona con TBAP para formar el metafosfito cíclico, luego en el tercer paso se oxida para generar el 5ʹ-trimetafosfato cíclico en la solución de oxidación del sintetizador de ADN / ARN (0.1 M de yodo / piridina / H2O / THF), que se hidroliza rápidamente para producir el 5ʹ-trifosfato lineal en la misma solución28,33, 37. La escisión alcalina posterior del soporte sólido de la CPG y la desprotección del oligonucleótido en MeNH2/amoníaco acuoso hidrolizarán cualquier trimetafosfato cíclico residual a la forma lineal. Abreviaturas: SalPCl = salicil fosforocloridita; TBAP = pirofosfato de tributilamonio; THF = tetrahidrofurano; CPG = vidrio de poro controlado; MenH2 = metilamina. Haga clic aquí para ver una versión más grande de esta figura.
Aunque los primeros informes publicados que utilizan este método a menudo sufrían de bajos rendimientos y productos secundarios no deseados 28,37,38, el mantenimiento cuidadoso de las condiciones anhidras es todo lo que se necesita para obtener rutinariamente altos rendimientos. Esto se puede lograr mediante la preparación cuidadosa de reactivos y el uso de un dispositivo de reacción simple ensamblado a partir de componentes de plástico estándar. Aquí, demostramos los pasos apropiados para la trifosforilación química de oligonucleótidos, incluida la preparación de reactivos, el ensamblaje de la cámara de reacción, la reacción de trifosforilación y la posterior desprotección y purificación de los oligonucleótidos trifosforilados. También se incluye el uso representativo de oligonucleótidos trifosforilados de 5′ como sustratos para ribozimas ligasas para la síntesis de productos de ácido nucleico más grandes con columna vertebral natural de D-ARN y L-ARN abiótico.
Protocolo
1. Síntesis automatizada en fase sólida de oligonucleótidos de 5′-hidroxilo en un soporte sólido
- Prepare el sintetizador automatizado de ADN / ARN con reactivos y fosforamidatis de acuerdo con la composición del oligonucleótido objetivo y las instrucciones del instrumento.
- Cargue una columna de síntesis que contenga soporte sólido en el sintetizador y sintetice oligonucleótidos de acuerdo con los protocolos del instrumento del sintetizador.
NOTA: El procedimiento de trifosforilación ha sido optimizado para oligonucleótidos preparados a escala de 1 μmol. - Elimine el grupo protector 5′-dimetoxitrilo para producir el oligonucleótido 5′-hidroxilo soportado por sólidos como parte de la síntesis de oligonucleótidos en el paso anterior, o realizando un paso de detritilación terminal de acuerdo con los protocolos del instrumento del sintetizador.
- Retire la columna que contiene el oligonucleótido 5′-hidroxilo en soporte sólido del sintetizador, seque al vacío doméstico durante 10 minutos para eliminar el disolvente residual y proceda a la trifosforilación (secciones 3 y 4) una vez que se preparen los materiales para la trifosforilación (sección 2).
NOTA: Si no se utiliza inmediatamente, la columna seca se puede almacenar bajo una atmósfera normal en un recipiente de plástico sellado con desecante a -20 °C. No se necesita un secado adicional en esta etapa, ya que la columna se seca completamente antes de la trifosforilación en la sección 3.
2. Preparación de materiales para la trifosforilación
- Conecte una fuente de argón seco con presión ajustable a un colector de gas con al menos dos líneas y conéctela a un burbujeador. Asegúrese de que las líneas terminan en jeringas de plástico de 1 ml para facilitar la conexión al aparato de reacción.
- Reúna el equipo que se utilizará durante la trifosforilación, incluidas jeringas de plástico de 1 ml, una llave de paso de polipropileno de tres vías, agujas no punteras, tubos de polipropileno de 1,5 ml y una pequeña espátula de metal. Guárdelos en un recipiente sellado o desecante con desecante a temperatura ambiente durante al menos 1 día antes de su uso.
- Preparar 30 ml de 1,4-dioxano anhidro, dioxano 3:1: piridina por volumen, N, N-dimetilformamida (DMF) y acetonitrilo (ACN) en botellas de vidrio secas de 30 ml con trampas de secado (tamices moleculares de 4 Å en paquetes de membrana sellados) al menos 1 día antes de su uso. Selle con septos de goma y guárdelo en un desecador con desecante.
- Guarde el sólido 2-cloro-4H-1,3,2-benzodioxafosforina-4-ona (salicil fosforocloridita, SalPCl) en su envase original dentro de un frasco sellado con desecante a 4 °C. Siempre enjuague el recipiente con argón entre usos.
- Prepare una solución de pirofosfato de tributilamonio (TBAP) al menos 5 días antes de la reacción de trifosforilación:
- Pesar 1-5 g de TBAP sólido en una botella de vidrio seca de 30 ml y disolver en 1 ml de DMF y 0,5 ml de tributilamina por g de TBAP.
- Agregue tres trampas de secado, selle la botella con un tabique de goma debajo del argón y burbujee con argón durante 30 minutos para desgasificar.
- Almacene dentro de un frasco sellado con desecante a 4 °C durante 5 días para permitir que los tamices moleculares absorban toda el agua traza. Guarde el frasco a -20 °C y prepárelo fresco después de 6 meses.
3. Montaje y uso del aparato de trifosforilación
- Permita que la columna de síntesis se caliente a temperatura ambiente si se recupera del almacenamiento a -20 °C.
- Ensamble la cámara de reacción que se muestra en la Figura 2:
- Prepare la antecámara: retire el émbolo de una jeringa seca de 1 ml, corte la parte superior de la jeringa con tijeras o una cuchilla de afeitar y conecte la jeringa a la columna de síntesis. Conecte la llave de paso de tres vías a la parte superior de la jeringa y conecte la entrada lateral de la llave de paso a la fuente de argón seco con el burbujeador, de modo que la entrada superior de la llave de paso sea el puerto de inyección de reactivo.
- Asegure este aparato a un soporte con abrazaderas y selle todas las juntas aguas arriba con una película de sellado de cera. Ajuste la llave de paso para que el puerto de inyección esté cerrado y el aparato esté abierto a la fuente de argón. Cierre el burbujeador y permita que el argón a baja presión (<10 psi) fluya a través de la cámara de reacción durante 5 minutos.
NOTA: Las cámaras de reacción múltiples se pueden configurar en paralelo a los oligonucleótidos trifosforilato 2-4. Sin embargo, se debe reservar una línea del colector para suministrar argón a las botellas de reactivo. - Vuelva a abrir el burbujeador y coloque una jeringa en la parte inferior de la columna de síntesis, que será la jeringa de desecho. Tire del argón a través de la columna repetidamente usando la jeringa de desecho; luego, vuelva a conectar la jeringa con el émbolo completamente empujado.
NOTA: A menos que se carguen reactivos, la llave de paso debe ajustarse de modo que el puerto de inyección esté cerrado y el aparato abierto a la fuente de argón, como se muestra en la Figura 2A. Del mismo modo, la jeringa de desecho debe fijarse y la unión a la columna de síntesis sellada con una película de sellado de cera a menos que se eliminen activamente los reactivos.
- Para añadir un reactivo o disolvente:
- Coloque una aguja en la fuente de argón seco e insértela en el reactivo o en el tabique de la botella de disolvente, teniendo cuidado de no sumergir la aguja en el contenido de la botella.
- Ensamble una jeringa seca con una aguja e insértela en el tabique del reactivo o del frasco de disolvente, sin sumergirlo en el contenido del frasco. Llene la jeringa con argón, retire la aguja del tabique y expulse el argón. Llene la jeringa con argón y expulse de nuevo; luego, llene la jeringa con el volumen requerido de disolvente o reactivo bajo presión de argón.
- Ajuste la llave de paso en el aparato para que la fuente de argón esté cerrada y el puerto de inyección esté abierto (Figura 2B). Retire rápidamente la jeringa y la aguja llenas del frasco de origen, limpie cualquier disolvente pegado al costado o la punta de la aguja e inserte la aguja en el puerto de inyección. Expulse el reactivo en la antecámara del aparato, retire la aguja y cierre el puerto de inyección, reabriendo el aparato a la fuente de argón.
- Extraiga suavemente el líquido de la antecámara hasta la columna de síntesis utilizando la jeringa de desecho para que todo el líquido se mantenga ahora en la jeringa de desecho. Ahora, empuje lentamente la solución hacia arriba en la columna de síntesis, asegurándose de que no haya burbujas de gas en la columna. Para mezclar o agitar, tire suavemente de la solución hacia arriba y hacia abajo sobre la columna con la jeringa de desecho.
NOTA: Siempre mueva el líquido lenta y suavemente a través de la cámara de reacción para asegurarse de que no se rompan los sellos, lo que permite que el aire ingrese al aparato.
- Para extraer un reactivo o disolvente de la columna:
- Tire lentamente de la solución en la jeringa de desecho. Después de que la mayor parte de la solución haya pasado a la jeringa de desecho, tire del argón para eliminar el disolvente restante de la columna.
- Retire la película de sellado de cera alrededor de la junta de la jeringa de desecho, luego retire la jeringa y deseche la solución de desecho. Reemplace la jeringa de desecho con una jeringa nueva y seca y vuelva a sellar la junta con una película de sellado de cera.
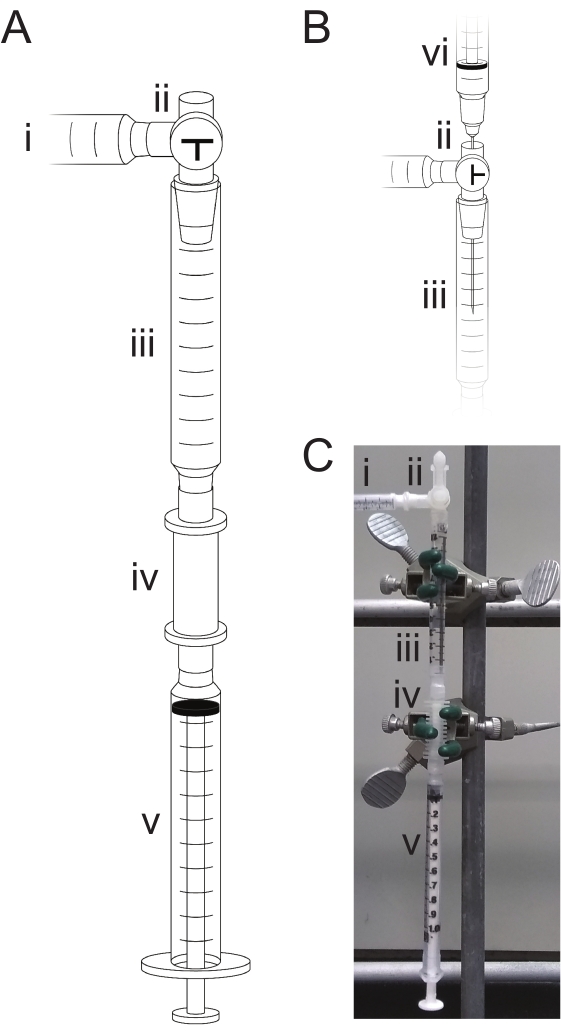
Figura 2: Aparato de trifosforilación. Durante la mezcla o las reacciones, el dispositivo (A) está abierto a la fuente de argón (i) y cerrado al aire ajustando la llave de paso de tres vías (ii). Los reactivos se extraen de la antecámara iii) a la columna de síntesis iv) por medio de la jeringa de desecho (v). Los reactivos se eliminan extrayendo todo el líquido en la jeringa de desecho (v) y desechándolo. Al cargar reactivos (B), la llave de paso de tres vías (ii) está abierta a la atmósfera, y el reactivo se carga en la antecámara (iii) por medio de una jeringa y una aguja (vi). (C) Una fotografía del aparato ensamblado como en (A) para mezclar y reaccionar con reactivos. Haga clic aquí para ver una versión más grande de esta figura.
4. Trifosforilación en columna del oligonucleótido sintético 5′-hidroxilo
- Retire la solución de SalPCl y TBAP del almacenamiento a -20 °C y deje que se calienten a temperatura ambiente antes de su uso.
- Añadir 200 μL de piridina/dioxano a la antecámara, de acuerdo con los pasos 3.3.1-3.3.3. Sin embargo, no cargue disolvente en la columna de síntesis hasta el paso 4.4.
- Use una espátula de metal seco para pesar 6-12 mg de SalPCl en un tubo de microcentrífuga seco de 1,5 ml, y disuelva en 100 μL de dioxano jeringando suavemente el disolvente hacia arriba y hacia abajo dentro del tubo de la microcentrífuga.
- Añadir el SalPCl disuelto a la antecámara y cargarlo en la columna de síntesis, siguiendo el paso 3.3. Deja que reaccione durante 15 min, agitando la solución cada 5 min. Retire y deseche la solución de SalPCl de acuerdo con el paso 3.4.
NOTA: SalPCl se agrega en gran exceso y eliminará cualquier agua absorbida durante la preparación y la carga en la cámara de reacción. Sin embargo, la introducción de cualquier humedad durante los pasos 4.5 y 4.6 comprometerá el rendimiento final del oligonucleótido 5′-trifosfato. - Añadir 250 μL de solución TBAP a la antecámara y cargarla en la columna de síntesis, siguiendo el paso 3.3. Deja que reaccione durante 20 min, agitando cada 5 min. Retire y deseche la solución TBAP de acuerdo con el paso 3.4.
- Lave la columna con 0,5 ml de DMF, luego 0,5 ml de ACN, eliminando el disolvente después de cada adición de acuerdo con los pasos 3.3 y 3.4.
- Añadir 250 μL de solución oxidante (0,1 M de yodo en tetrahidrofurano (THF)/piridina/agua, 88:10:2) a la antecámara y cargarlo en la columna de síntesis, siguiendo el paso 3.3. Deja que reaccione durante 30 min, agitando cada 10 min. Retire y deseche la solución oxidante de acuerdo con el paso 3.4.
- Lave la columna con 0,5 ml de ACN y retire, de acuerdo con los pasos 3.3 y 3.4.
- Desmontar el aparato de reacción. Lavar la columna de síntesis con 5 ml de ACN y secar la columna.
5. Escisión del soporte sólido, desprotección y purificación
- Retire la resina de soporte sólido seco de la columna de síntesis y transfiérala a un tubo sellable de tapa de rosca de polipropileno de 1,5 ml con una junta tórica de silicona.
- Suspenda la resina en 1 ml de una mezcla 1:1 de amoníaco acuoso al 28%-30% y metilamina acuosa (AMA) al 40% y selle el tubo herméticamente. Incubar a 65 °C durante 10 min con mezcla intermitente mediante inversión suave39. Utilice un tratamiento más suave a temperatura ambiente durante 2 h para oligonucleótidos de más de 40 nt.
PRECAUCIÓN: El calentamiento de la solución AMA pondrá el tubo bajo alta presión. Si el tubo no está sellado herméticamente o no utiliza una junta tórica compatible con amoníaco (silicona), el tubo puede ventilar gas o fuga de disolvente, lo que podría comprometer la seguridad o el rendimiento del producto final. Nunca abra el tubo mientras esté por encima de la temperatura ambiente, ya que la solución AMA caliente puede evolucionar violentamente el gas. - Enfríe el tubo sobre hielo y centrímelo brevemente (6.000-12.000 × g durante 10 s). Retire el sobrenadante de la resina, filtre a través de una jeringa equipada con un filtro de 0,2 μm y transfiéralo a un nuevo tubo de polipropileno estéril. Evapore la solución a la sequedad utilizando una centrífuga de vacío equipada con una trampa química neutralizante de amoníaco.
NOTA: Si el oligonucleótido sintético no contiene ningún nucleótido de ARN con grupos protectores de 2′-sililo, vaya al paso 5.8. - Eliminar los grupos protectores de sililo disolviendo el material seco en 1 mL de 1 M de fluoruro de tetrabutilamonio (TBAF) en THF, calentando a 55 °C, agitando si es necesario para disolver completamente el oligonucleótido e incubando a temperatura ambiente durante 16-24 h40,41.
- Apague la solución TBAF con 1 ml de tampón Tris de 1 M, pH 7.5, y retire THF con una centrífuga de vacío.
- Retire las sales de TBAF utilizando una columna de exclusión de tamaño desechable, siguiendo las instrucciones del fabricante. Para oligonucleótidos inferiores a 15 nt, confirme la elución del producto recogiendo el eluido en fracciones e identificando las principales fracciones del producto mediante absorbancia a 260 nm en un espectrofotómetro UV-Vis.
- Concentrar el oligonucleótido de ARN desprotegido por liofilización o centrífuga al vacío si es inferior a 15 nt, o por precipitación de etanol si es mayor de 15 nt.
- Prepare una escala preparativa 10% -20% poliacrilamida / 8 M urea / 1x gel TBE utilizando stock de acrilamida mono: bis 19: 1, de acuerdo con los protocolos apropiados para el tamaño del oligonucleótido, la placa de gel y el soporte. Ensamble la placa de gel en el soporte de gel con 1x TBE en depósitos y precorra a 35 W (o según corresponda para el formato de placa de gel) durante al menos 30 min.
- Disolver el oligonucleótido sólido en tampón de carga de gel de urea (8 M de urea, 10% de sacarosa, 50 mM de Tris, pH 8, 1 mM de EDTA con bromofenol y cianol de xileno) y calentar a 80 °C. Cargue en el gel de poliacrilamida y corra durante 1-2 h a 25-35 W (o según corresponda para el formato de la placa de gel y el tamaño del oligonucleótido).
NOTA: El azul de bromofenol o el cianol de xileno deben excluirse del tampón de carga de gel para oligonucleótidos inferiores a 15 nt si alguno de los dos comigra con el producto, ya que es difícil eliminar estos colorantes del oligonucleótido eluido sin precipitación de etanol. - Cuando se complete la electroforesis del gel, retire la placa de gel del soporte de gel, desmonte la placa de gel y envuelva el gel en una película de cloruro de polivinilo. Identifique las bandas del producto mediante el sombreado posterior con luz UV de 254 nm y elimine la banda principal del producto con una cuchilla de afeitar.
- Extraiga el oligonucleótido del gel extirpado por el método de trituración y remojo42.
- Triture el gel extirpado extruyéndolo a través de una jeringa de plástico o mecánicamente.
- Para oligonucleótidos de más de 15 nt:
- Elute en 3x volúmenes de tampón de trituración y remojo (300 mM NaCl; 10 mM Tris, pH 8; 1 mM EDTA) durante al menos 12 h con agitación o agitación.
- Retire las piezas de gel sólido pasando la solución a través de una jeringa equipada con un filtro de 0,2 μm y concentre el oligonucleótido por precipitación de etanol.
- Para oligonucleótidos inferiores a 15 nt:
- Elute en 3 volúmenes de agua libre de nucleasa durante al menos 12 h con agitación o agitación.
- Retire las piezas de gel sólido pasando la solución a través de una jeringa equipada con un filtro de 0,2 μm y concéntrese por liofilización.
- Elimine las sales residuales y los solutos utilizando una columna de exclusión de tamaño desechable como en el paso 5.6. Concentrado por liofilización.
- Almacene los oligonucleótidos trifosforilados purificados a -20 °C en el tampón TE (10 mM Tris, pH 8; 1 mM EDTA) o en un tampón de almacenamiento de oligonucleótidos similar.
- Determine la concentración del oligonucleótido midiendo la absorbancia a 260 nm utilizando un espectrofotómetro UV-Vis.
NOTA: El coeficiente de extinción estimado del oligonucleótido no debe verse afectado por su estado de fosforilación de 5′, y se puede calcular a partir de su secuencia utilizando una calculadora de coeficiente de extinción de oligonucleótidos. - Verificar la trifosforilación por espectrometría de masas. Busque una masa esperada de +239.94 Da en relación con la del oligonucleótido 5′-hidroxilo.
NOTA: La desorción/ionización por láser asistida por matriz o la espectrometría de masas de ionización por electropulverización (MALDI-MS o ESI-MS, respectivamente) son adecuadas para identificar el estado de trifosforilación del oligonucleótido cuando se utilizan protocolos optimizados para ácidos nucleicos. SIN EMBARGO, ESI-MS proporciona resultados más consistentes debido a las menores tasas de fragmentación iónica; en la Tabla de Materiales se proporciona un servicio comercial representativo.
- Determine la concentración del oligonucleótido midiendo la absorbancia a 260 nm utilizando un espectrofotómetro UV-Vis.
6. Oligonucleótidos trifosforilados como sustratos para la autorreplicación de la ribozima
PRECAUCIÓN: 32P es un isótopo radiactivo y los siguientes pasos deben realizarse utilizando protocolos de seguridad estándar para trabajar con materiales radiactivos en un laboratorio y por un investigador certificado para el uso de materiales radiactivos por los departamentos de Salud y Seguridad Ambiental pertinentes. Como alternativa, el sustrato de ribozima A autorreplicante se puede preparar sintéticamente con una etiqueta de 5′-fluoresceína14 y obtener imágenes fluorescentes, como en el paso 7.9.
- Preparar la solución A como una mezcla de ribozima E autorreplicante y sustrato de ARN marcado con 5′-32P A14 a concentraciones de 0,30 μM y 30 μM, respectivamente. Preparar soluciones B-transcritas y B-sintéticas con 15 μM de ARN trifosforilado sustrato B, preparadas por transcripción in vitro 14 o trifosforilación química como la anterior, respectivamente, en tampón EPPS de 75 mM, pH 8.5, y 37.5 MgCl2. Llevar ambas soluciones a 42 °C.
NOTA: Todos los componentes de ARN se enumeran en la Tabla de materiales. - Para iniciar la autorreplicación, mezcle rápidamente 5 μL de solución A con 10 μL de solución B transcrita o B sintética a una concentración final de 0,1 μM E, 10 μM A, 10 μM B, 25 mM MgCl2 y 50 mM tampón EPPS, pH 8,5. Incubar la mezcla de reacción a 42 °C.
- A intervalos regulares, tomar 0,5 μL de alícuotas y apagar en 9,5 μL de tampón de carga de gel de formamida (95% de formamida; 10 mM de EDTA, pH 8).
- Preparar un gel analítico de poliacrilamida/8 M urea/1x TBE, de acuerdo con los protocolos apropiados para la placa de gel y el soporte. Ensamble el gel fundido y la placa en un soporte de gel, llene los depósitos con 1x TBE y ejecute previamente a 40 W (o según corresponda para diferentes placas y soportes de gel) durante 30 minutos.
- Calentar las muestras de reacción apagadas a 80 °C, cargar 5 μL de la muestra en cada pozo y hacer funcionar el gel a 40 W durante aproximadamente 40 min (o según corresponda para diferentes placas y soportes de gel).
- Retire la placa de gel del soporte de gel, desmonte y envuelva el gel en una película de policloruro de vinilo. Cubra el gel con una pantalla de fósforo y expóngalo durante 1 h (o según corresponda para 32P cpm); escanear la pantalla usando un escáner de gel fluorescente/fosforescente.
- Cuantificar el rendimiento de la reacción utilizando un software de cuantificación de imágenes en gel.
- Abra la imagen del gel con analysis Toolbox, elija Analysis | Definición de forma, seleccione Áreas | la forma del rectángulo y dibuje rectángulos del mismo tamaño alrededor de las bandas correspondientes a A y E no reaccionadas para cada vez y ambas reacciones.
- Elija | de análisis Resta de fondo, seleccione Áreas | la forma del rectángulo y dibuje un rectángulo del mismo tamaño en una parte vacía de la imagen del gel. Cambie el método de sustracción de fondo para todos los rectángulos a Rectángulo de imagen.
- Elija Ventana | 2 y, a continuación, Editar | Exporte a Excel para exportar los volúmenes de píxeles de banda cuantificados a un archivo de hoja de cálculo.
- Trazar la concentración del producto E frente al tiempo, y ajustar los datos al crecimiento logístico Eq (1) utilizando un software de ajuste de datos estadísticos:
[E] =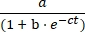 (1)
(1)
Donde a es la extensión máxima de la reacción, b es el grado de sigmoidicidad y c es la tasa de crecimiento exponencial.- En la hoja de cálculo exportada, divida el volumen E del producto por la suma de los volúmenes del sustrato A y del producto E para determinar el rendimiento fraccional del producto para cada momento y ambas reacciones. Multiplique por la concentración inicial de sustrato A (10 μM) para determinar el rendimiento del producto E en función del tiempo.
- En el software de ajuste de datos estadísticos, elija File | Nuevas | Nuevo archivo de proyecto, seleccione XY en Nueva tabla y gráfico y haga clic en Crear. Introduzca los tiempos de reacción y los rendimientos del producto E para ambas reacciones en columnas adyacentes y etiquete las columnas correspondientemente (por ejemplo, "tiempo", "transcrito B", "sintético B").
- Elija Insertar | Nuevo análisis, seleccione análisis XY | Regresión no lineal (ajuste de curva) y, a continuación, haga clic en Aceptar. Elija Curvas de crecimiento | Crecimiento logístico y haga clic en Aceptar; no ajuste ningún otro parámetro. Observe los parámetros de ajuste y los intervalos de confianza en Resultados y la gráfica de puntos de datos y curvas ajustadas en Gráficos.
7. Copia quiral cruzada de L-ARN
- Preparar 10 μL de solución de ARN que contenga 20 μM de D-ARN 27,3t polimerasa quiral cruzada, 2 μM de cebador de L-ARN marcado con 5′-fluoresceína, plantilla de cabeza de martillo L-ARN biotinilada de 4 μM y 20 μM cada una de L-RNA pppCUG, pppAUG, pppAGG y pppCGC, en 10 μL de 50 mM Tris, pH 8.3. Anneal el ARN calentándolo a 90 °C durante 1 min y enfriándolo a 23 °C a 0,2 °C/s en un termociclador PCR. Consulte la Tabla de materiales para obtener detalles sobre la polimerasa, la imprimación y la plantilla.
- Incubar la solución de ARN a 17 °C y comenzar la reacción añadiendo 10 μL de tampón de inicio 2x (400 mM MgCl2, 500 mM NaCl y 50 mM Tris, pH 8.3). Asegúrese de que las concentraciones finales de todos los componentes de la reacción sean 10 μM polimerasa, 1 μM de imprimación, 2 μM de plantilla, 10 μM de cada trinucleótido 5′-trifosfato, 200 mM mgCl2, 250 mM de NaCl y 50 mM de Tris, pH 8,3.
- A medida que avanza la reacción, tomar 10 μL de alícuotas y apagar con 5 μL de 0,5 M de EDTA, pH 8.
- A cada muestra de reacción apagada, agregue 0,1 mg de perlas magnéticas recubiertas de estreptavidina (capacidad de unión de biotina-oligonucleótido de 20 pmol) suspendidas en 10 μL de 1 M de NaCl en tampón TE con detergente neutro al 0,05% para capturar imprimaciones extendidas por trímero a través de la plantilla biotinilada e incubar durante 30 min a temperatura ambiente con agitación.
- Capture las perlas en un imán de captura de perlas, retire y deseche el líquido y agregue 50 μL de solución de lavado (250 mM de NaCl en TE con detergente neutro al 0,05%). Mezcle las perlas, recójalas de nuevo y retire la solución de lavado. Repite una vez más.
- Para eluir imprimaciones extendidas de perlas, agregue 50 μL de solución de elución (25 mM NaOH con detergente neutro al 0.05%) y mezcle las perlas. Vuelva a capturar las perlas, retire la solución de elución, apague con 100 mM Tris (pH 7.5) y precipite con etanol.
- Preparar un gel analítico de poliacrilamida/8 M urea/1x TBE, de acuerdo con los protocolos apropiados para la placa de gel y el soporte. Ensamble el gel fundido y la placa en un soporte de gel, llene los depósitos con 1x TBE y ejecute previamente a 40 W (o según corresponda para diferentes placas y soportes de gel) durante 30 minutos.
- Disuelva el ARN precipitado en 10 μL de tampón de carga de gel de formamida y prepare un cebador final marcado sin reaccionar a 0,5 μM en tampón de carga de gel de formamida. Caliente las muestras a 80 °C, cargue 5 μL de la muestra en cada pozo y haga funcionar el gel a 40 W durante aproximadamente 40 minutos (o según corresponda para diferentes placas y soportes de gel).
- Retire la placa de gel del soporte y escanee con un escáner de gel fluorescente / fosforescente para visualizar los productos de extensión de L-ARN quiral cruzado.
Resultados
Los oligonucleótidos deben sintetizarse utilizando protocolos estándar apropiados para las fosforamidas y el sintetizador automatizado de ADN/ARN, dejando el oligonucleótido del producto extraído del soporte sólido en la columna de síntesis plástica original, con el grupo dimetoxitritil 5ʹ-terminal eliminado para producir el 5ʹ-hidroxilo libre (sección 1). Todos los oligonucleótidos utilizados en esta demostración se prepararon utilizando resina de vidrio de poro controlado (CPG) de 1.000 Å como soporte sólido y se realizaron a escala de 0,2 o 1 μmol. Ejemplos representativos de columnas de sintetizador, resinas, reactivos y fosforamidatis se proporcionan en la Tabla de Materiales. Para reacciones a mayor escala, es posible que sea necesario ajustar los volúmenes y los tiempos utilizados en los pasos posteriores.
La reacción de trifosforilación se lleva a cabo en columna en una cámara de reacción personalizada (Figura 2, sección 3) utilizando componentes estándar disponibles comercialmente enumerados en la Tabla de Materiales y sigue el esquema ilustrado en la Figura 1 (sección 4)28. Es esencial que las condiciones se mantengan estrictamente anhidras durante la trifosforilación, y que todos los disolventes y reactivos se preparen previamente sobre tamices moleculares y se dejen secar completamente antes de su uso (sección 2). La trifosforilación suele tardar 2 h en producirse y, posteriormente, la columna lavada y seca puede tratarse de acuerdo con los procedimientos estándar de desprotección y purificación de oligonucleótidos (sección 5).
Después de la desprotección, los trifosfatos de oligonucleótidos se purifican mediante electroforesis en gel de poliacrilamida desnaturalizante (PAGE), mostrando una sola banda principal del producto mediante retrosombrecimiento UV que se puede extirpar y eludir del gel. El producto 5′-trifosfato se separa fácilmente de los productos secundarios de reacción para oligonucleótidos cortos, como se muestra para el trinucleótido de ADN 5ʹ-trifosfatos, pppAAA y pppCCC, y el trinucleótido L-ARN 5ʹ-trifosfato pppGAA en la Figura 3A, B. Tanto los productos de 5′-hidroxilo como 5′-trifosfato para los trímeros de ADN AAA y CCC fueron extirpados e identificados por espectrometría de masas y etiquetados correspondientemente en la Figura 3A. Las bandas adicionales, como son visibles para el trímero de ADN AAA, generalmente no contienen suficiente material para recuperarse e identificarse. La presencia de estas bandas, sin embargo, se correlaciona con masas de producto adicionales en los productos de reacción no purificados (Figura 3C), que típicamente representan productos secundarios de 5′-difosfato, monofosfato y H-fosfonato, como se discute a continuación.
Después de la purificación de PAGE, se pueden eluvir oligonucleótidos más grandes utilizando el método de trituración y remojo42 y la posterior precipitación de etanol. Sin embargo, los oligonucleótidos inferiores a 15 nt no pueden precipitarse etanol de manera eficiente y, por lo tanto, requieren un procedimiento modificado para la elución en gel (paso 5.11.3). La columna de exclusión de tamaño desechable enumerada en la Tabla de materiales está clasificada solo para su uso con oligonucleótidos de más de 10 nt. Sin embargo, hemos encontrado que los oligonucleótidos tan cortos como los trímeros se pueden desalinizar de manera efectiva utilizando el protocolo recomendado por el fabricante. Sin embargo, se recomienda al desalinizar oligonucleótidos cortos (como en los pasos 5.6 y 5.11.3) que el eluido de la columna se recoja en fracciones y las fracciones de producto se identifiquen por absorbancia a 260 nm utilizando un espectrofotómetro UV-Vis. Una columna de exclusión de tamaño optimizada para oligonucleótidos más cortos se proporciona en la Tabla de Materiales como una opción alternativa. El rendimiento final de la síntesis de oligonucleótidos a escala de 1 μmole después de la purificación es de 50-300 nmol.
La trifosforilación puede confirmarse mediante espectrometría de masas, donde el producto trifosforilado tiene una masa +239,94 Da mayor que el oligonucleótido 5′-hidroxilo, aunque a menudo se observa la presencia de materiales correspondientes al 5′-di- y monofosfato (+159,96 y +79,98 Da, respectivamente). También se puede observar un producto lateral de 5′-H-fosfonato con una masa +63.98 Da de la masa 5′-OH, y los altos niveles de este producto indican que las condiciones durante la trifosforilación no fueron lo suficientemente anhidras. Antes de la purificación, los oligonucleótidos desprotegidos generalmente mostrarán todos estos productos (Figura 3C), mientras que el material purificado mostrará un pico correspondiente al producto 5′-trifosfato junto con 5'-di- y monofosfatos (Figura 3D, E).
La espectrometría de masas por sí sola generalmente no dará una medida rigurosa de la pureza de 5'-trifosfato debido a las tasas diferenciales de ionización y fragmentación del trifosfato durante la ionización. Para medir la pureza del producto final, se recomienda la cromatografía líquida de fase inversa y la ESI-MS en tándem (RP-LC/ESI-MS), especialmente para oligonucleótidos más largos. El análisis de D-RNA 5ʹ-trifosfatos pppACGAGG y pppGAGACCGCAACUUA por RP-LC/ESI-MS (Figura 4A,B, respectivamente) muestra la pureza típica del producto final, que contiene 20% de 5ʹ-difosfato ya que estas dos especies son difíciles de separar cuando están presentes en oligonucleótidos más largos.
Los oligonucleótidos sintéticos de 5′-trifosfato suelen funcionar tan bien o mejor que los materiales preparados enzimáticamente en estudios bioquímicos. En la sección 6, a modo de ejemplo, se compararon sustratos de ARN de 5′-trifosfato de 14 nt preparados sintéticamente o mediante transcripción in vitro en una reacción de autorreplicación catalizada por ARN 14,15,43,44,45. La ribozima E cataliza la unión de los sustratos A y B para producir una nueva copia de E en una reacción autocatalítica capaz de crecimiento exponencial (Figura 5A). Los componentes E y 32A marcados con P se prepararon mediante transcripción in vitro, y el sustrato trifosforilado B se preparó sintéticamente, como se describió anteriormente, o mediante transcripción in vitro 14. El progreso de la reacción de autorreplicación se monitoreó mediante la toma de muestras periódicas que se analizaron mediante la desnaturalización de PAGE y se cuantificaron a través de un escáner de gel fluorescente / fosforescente. Los datos resultantes, ajustados a una función de crecimiento logístico, revelaron que el sustrato B transcrito o sintético soporta un crecimiento exponencial, pero el B sintético da una cantidad ligeramente mayor de producto (Figura 5B). Este resultado puede reflejar heterogeneidad compositiva en el extremo 5′ del ARN preparado por transcripción in vitro 23,24.
La trifosforilación química también permite la síntesis de trifosfatos de oligonucleótidos que no se pueden preparar biológicamente, ya sea in vitro o en células. En la sección 7, se utilizaron trifosfatos de oligonucleótidos no biológicos compuestos de L-ARN, el enantiómero del ARN-D natural, preparado como en las secciones 1-5, como sustratos para la ribozima 27.3t de la polimerasa D-ARN "cross-quiral" (Figura 6A), que cataliza la polimerización dirigida por plantilla de un producto de L-ARN más largo a partir de oligonucleótidos cortos de L-ARN 5′-trifosfatos de manera general de secuencia. Como ejemplo, la ribozima puede sintetizar una versión L-ARN del motivo de autoesclava del martillo (Figura 6B)18. Los trifosfatos de trinucleótidos de L-ARN purificados se combinaron con un cebador de L-ARN marcado con fluoresceína y una plantilla de L-ARN (Figura 6C) y reaccionaron con la ligasa quiral cruzada. Las muestras en el transcurso de la reacción fueron analizadas por PAGE y fotografiadas utilizando un escáner de gel fluorescente / fosforescente para demostrar la síntesis de una versión de L-ARN de la ribozima de martillo codificada por la plantilla (Figura 6D).
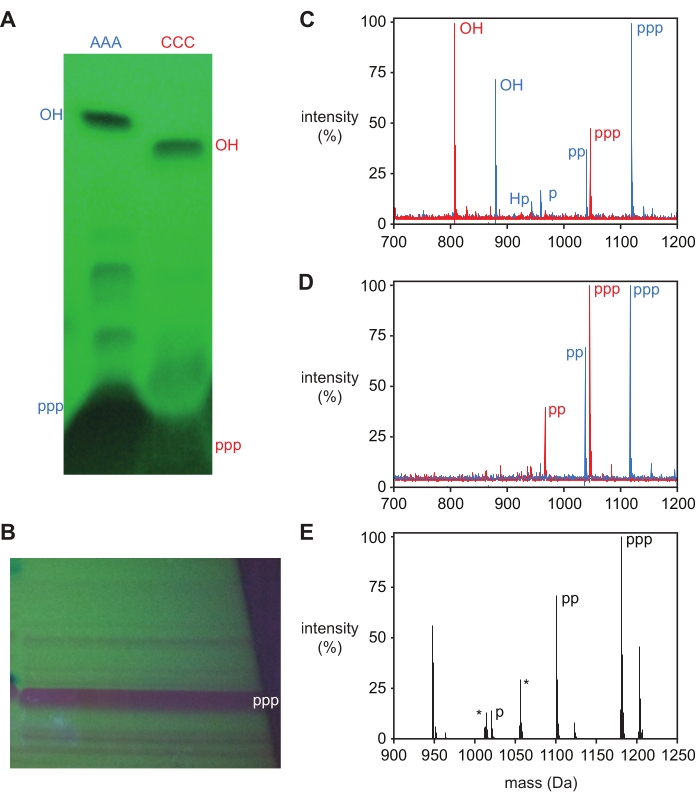
Figura 3: Purificación de trinucleótidos 5ʹ-trifosfatos. (A) Análisis PAGE (visualizado por UV-back-shadowing) de trifosforilación de trinucleótidos de ADN tri-desoxiadenosina (AAA, azul) y tri-desoxicitidina (CCC, rojo), sobrecargados intencionalmente para visualizar productos secundarios menores. Tanto el producto 5ʹ-trifosfato (ppp) como el material de partida 5ʹ-hidroxilo (OH) fueron extirpados e identificados por MALDI-MS. (B) PÁGINA preparatoria de trifosforilación de L-ARN trinucleótido GAA, con la banda principal del producto extirpada e identificada como el 5ʹ-trifosfato (ppp) por ESI-MS. (C) MALDI-MS de productos de reacción crudos después de la desprotección y (D) productos purificados de (A). 5ʹ-trifosfato (ppp; pppAAA esperado 1.119 Da, observado 1.118 Da; pppCCC esperado 1.047 Da, observado 1.046); 5ʹ-difosfato (pp), 5ʹ-monofosfato (p), 5ʹ-hidroxilo (OH) y 5ʹ-H-fosfonato (Hp) están etiquetados. (E) Espectro de masas descontornado de inyección directa ESI-MS de producto aislado de 5ʹ-trifosfato de (B), con picos identificados marcados (esperado 1,181.6 Da, observado 1,181.0 Da). También se observan productos de 5ʹ-difosfato (pp), al igual que picos de iones de sodio para los productos tri- y di-fosfato (+22 Da). Los picos de contaminantes comunes están etiquetados con un asterisco. Para facilitar la comparación, los espectros de masas se normalizaron a la intensidad más alta medida en cada espectro y se informan como un porcentaje relativo a ese valor. Abreviaturas: PAGE = electroforesis en gel de poliacrilamida; MALDI-MS = desorción/ionización láser asistida por matriz; ESI-MS = espectrometría de masas de ionización por electropulverización. Haga clic aquí para ver una versión más grande de esta figura.
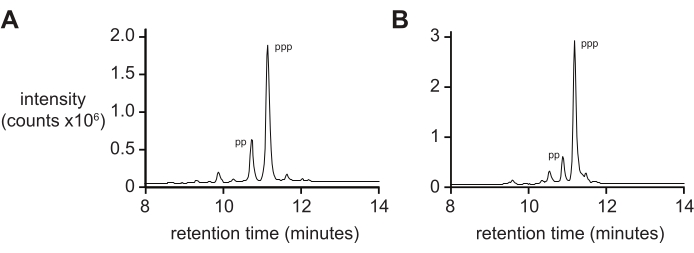
Figura 4: RP-LC analítico de 6 nt y 14 nt D-RNA oligonucleótidos trifosfatos. (A) 5ʹ-pppACGAGG-3ʹ y (B) 5ʹ-pppGAGACCGCAACUUA-3ʹ. Tandem ESI-MS identificó el pico principal de ambos (~ 70%) como el 5ʹ-trifosfato (ppp), con menores cantidades del 5ʹ-difosfato (pp). Abreviaturas: RP-LC = cromatografía líquida de fase inversa; nt = nucleótidos; ESI-MS = espectrometría de masas de ionización por electropulverización. Haga clic aquí para ver una versión más grande de esta figura.
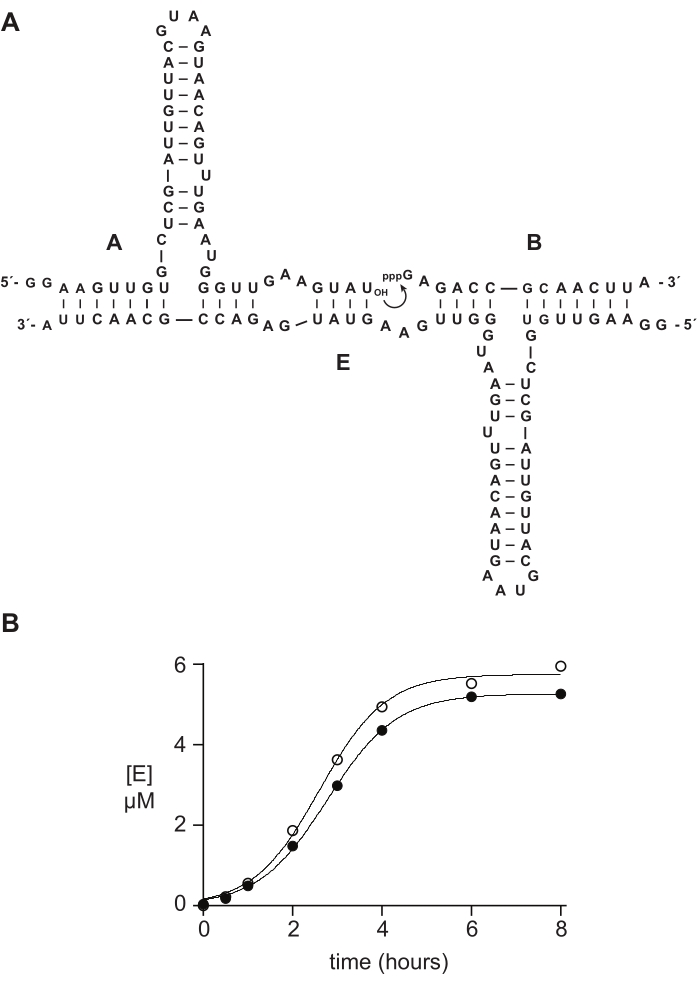
Figura 5: Comparación de sustratos de oligonucleótidos 5ʹ-trifosfato preparados por síntesis química o transcripción in vitro. (A) La ribozima E autorreplicante liga el ARN A y el ARN B trifosforilado 5′. (B) Comparación de reacciones de autorreplicación utilizando 10 μM A y 10 μM B, ya sea sintético (círculos abiertos) o transcrito in vitro (círculos llenos). (B) Los datos se ajustaron a la ecuación de crecimiento logístico: [E] = a / (1 + b e-ct), donde a es el rendimiento final, b es el grado de sigmoidicidad y c es la tasa de crecimiento exponencial. Las tasas de crecimiento para las dos reacciones fueron idénticas, a las 1.14 h-1, mientras que la extensión final fue un 10% más alta para las reacciones con B sintético. Haga clic aquí para ver una versión más grande de esta figura.
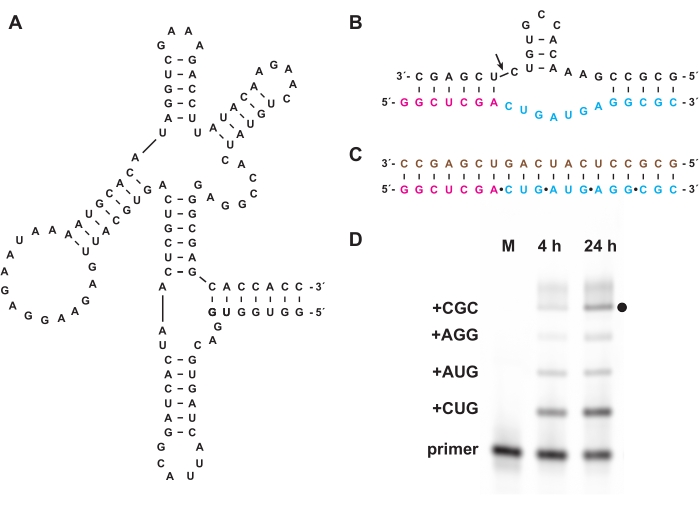
Figura 6: Polimerización de L-ARN quiral cruzado con una ribozima. (A) La ribozima de la polimerasa D-RNA 27.3t, que cataliza la ligadura dependiente de plantillas de L-ARN. (B) El producto L-ARN sintetizado por 27.3t forma parte de un motivo de endonucleasa de martillo. (C) Polimerización de L-ARN catalizada por 27.3t utilizando una plantilla de L-ARN biotinilada (marrón), un cebador de L-ARN marcado al final (magenta) y cuatro trifosfatos de trinucleótidos de L-ARN (cian), preparados sintéticamente. (D) Análisis PAGE de productos de extensión de (B) a las 4 h y 24 h, mostrando cada incorporación de trinucleótidos hasta el producto de longitud completa (punto negro). El cebador de L-ARN no reaccionado se incluye como marcador de referencia. Abreviaturas: PAGE = electroforesis en gel de poliacrilamida; M = marcador de referencia. Haga clic aquí para ver una versión más grande de esta figura.
Discusión
El procedimiento de trifosforilación descrito aquí es ampliamente compatible con la síntesis de oligonucleótidos utilizando la química estándar de fosforamida. Las fosforamidatis nucleósidos deben tener grupos protectores lábiles de base compatibles con la desprotección rápida en AMA39, incluido el β-cianoetilo estándar en el fosfito, y los grupos isobutiril, dimetilformimidil, acetil, fenoxiacetilo o 4-isopropilfenoxiacetilo en las aminas exocíclicas de las nucleobases. Los grupos ribosa 2'-hidroxilo deben estar protegidos por grupos protectores de silil, ya sea t-butildimetilsilil (TBDMS) o tri-iso-propilsililoximetilo (TOM)40,41. También se ha informado que el grupo pivaloiloximetilometil (PivOM) base-lábil es compatible con la trifosforilación química30.
Se han descrito múltiples métodos para la trifosforilación química de oligonucleótidos sintéticos 28,29,30,31,32,33,34,35. Hemos encontrado que la trifosforilación utilizando el reactivoLudwig-Eckstein 37 es una de las más accesibles, ya que no requiere síntesis especializada de reactivos ni equipo especializado. Los oligonucleótidos 5′-trifosfatos preparados por este método se han utilizado rutinariamente como sustratos para ribozimas de ARN ligasa, incluido el uso de trifosfatos de oligonucleótidos de ARN L-ARN enzimáticamente inaccesibles para lograr la síntesis y replicación dependiente de plantillas de este ácido nucleico "imagen especular" 14,16,17,18 . El método también es adecuado para la preparación de pequeños ARN de asa del tallo trifosforilados de 5′ que son potentes activadores de la respuesta inmune innata en vertebrados 6,7.
El reactivo de Ludwig-Eckstein, salicil fosforocloridita37, es altamente reactivo al agua y elimina eficazmente cualquier agua introducida al disolver el reactivo antes de cargarlo en la columna de oligonucleótidos. Después de este punto, sin embargo, el oligonucleótido fosfitilado de 5′reaccionará preferentemente con cualquier agua introducida en el sistema sobre el pirofosfato, formando un producto lateral de 5′-H-fosfonato después del análisis 28,37,38. La preparación cuidadosa de los reactivos de trifosforilación y la cámara de reacción de trifosforilación aseguran que este producto secundario no se forme. Para el secado con disolvente, los tamices moleculares tipo 4 Å se venden preenvasados en bolsas de teflón compatibles con la mayoría de los disolventes orgánicos bajo diversas marcas por la mayoría de las empresas de reactivos de síntesis de oligonucleótidos. Las precauciones adicionales, como realizar la trifosforilación en una guantera bajo una atmósfera anhidra, generalmente no son necesarias.
La reacción del oligonucleótido 5′-fosfitilado con TBAP forma un intermedio cíclico de 5′-trimetafosfito, que luego se oxida al 5′-trimetafosfato cíclico utilizando una solución oxidante de síntesis de oligonucleótidos (yodo en agua / piridina / THF). Cabe señalar que las soluciones oxidantes comerciales utilizan cantidades variables de yodo, y es esencial utilizar la alta concentración de yodo de 0,1 M para garantizar la oxidación completa del trifosfato. El producto cíclico se hidroliza hasta el 5′-trifosfato lineal final en la misma solución37, y se deben utilizar soluciones oxidantes anhidras alternativas si se desea la linealización con nucleófilos distintos del agua (ver más abajo para aplicaciones)33. Cualquier trimetafosfato cíclico residual, sin embargo, se linealizará durante la posterior desprotección alcalina del oligonucleótido. La hidrólisis del 5′-trimetafosfato cíclico produce solo el trifosfato lineal, en lugar del ramificado37,46.
La desprotección de oligonucleótidos generalmente no necesita ser modificada para acomodar la trifosforilación de 5', pero se deben tomar algunas precauciones. El trifosfato es relativamente estable a la exposición breve a condiciones alcalinas, pero se debe tener cuidado de no exponer el trifosfato a AMA más tiempo del necesario. Se deben evitar los grupos protectores que requieren un tratamiento más prolongado en amoníaco o AMA durante más de 10 min a 65 °C. Los tratamientos más suaves, como 2 h en amoníaco a temperatura ambiente, son aceptables cuando son compatibles con otros grupos protectores de fosforamida. Un método común y rápido de desprotección para oligonucleótidos de ARN sintético protegido por sililo utiliza trihidrofluoruro de trietilamina y alta temperatura47; sin embargo, esto debe evitarse al preparar ARN 5′-trifosfatos, ya que las condiciones ácidas prolongadas aceleran la hidrólisis del trifosfato 31,32.
Preparative PAGE ha demostrado ser el método más simple y confiable para la purificación postdesprotección de oligonucleótidos trifosforilados de 5′ (Figura 3 y Figura 4). Sin embargo, la HPLC de fase inversa preparativa también se puede utilizar para purificar productos trifosforilados. La presencia de productos de 5′-difosfato y, en menor medida, de 5′-monofosfato se observa rutinariamente al verificar la trifosforilación por espectrometría de masas. Hemos observado fragmentación de 5′-trifosfato durante la espectrometría de masas a partir de material altamente puro preparado por síntesis química o transcripción, particularmente si el instrumento no está optimizado para el análisis de oligonucleótidos. Sin embargo, el análisis RP-LC a menudo muestra que el 10%-20% del producto secundario 5′-difosfato está presente en oligonucleótidos trifosforilados más largos (Figura 4). Las preparaciones comerciales de pirofosfato de tributilamonio pueden estar contaminadas con hasta un 20% de monofosfato, lo que producirá 5′-difosfato como producto secundario durante la trifosforilación30,31. La preparación cuidadosa de este reactivo internamente puede producir existencias de TBAP mucho más puras31. Sin embargo, hemos encontrado oligonucleótidos trifosforilados utilizando fuentes comerciales de TBAP que todavía muestran una reactividad comparable o mayor cuando se utilizan como sustratos en reacciones enzimáticas (Figura 5B), en comparación con el material preparado por transcripción in vitro.
Un uso adicional notable de la trifosforilación de oligonucleótidos con el reactivo de Ludwig-Eckstein aprovecha el intermedio cíclico trimetafosfito33. Si la etapa de oxidación posterior se lleva a cabo con peróxido de 1 M de t-butilo en hexanos, que a menudo se usa para la oxidación de oligonucleótidos en condiciones anhidras, la oxidación del fosfito ocurre sin hidrólisis de apertura de anillo, produciendo el trimetafosfato cíclico. Este intermediario puede reaccionar con nucleófilos primarios de amina o alcohol para producir 5′-trifosfatos con modificaciones en el γ-fosfato. Estas modificaciones incluyen la adición de una etiqueta lipofílica unida por un enlace fosforamidato, que facilita la rápida purificación específica de trifosfato por RP-LC, seguida de hidrólisis ácida de la etiqueta del trifosfato33. También se pueden introducir modificaciones fluorescentes en la posición de γ-fosfato para su uso como reporteros fluorescentes en tiempo real para reacciones de ligadura catalizadas por ribozimas15,33.
Divulgaciones
Los autores declaran que no tienen intereses financieros contrapuestos.
Agradecimientos
Los autores agradecen a Greg Springsteen, Natasha Paul, Charles Olea, Jr., Jonathan Sczepanski y Katrina Tjhung por las útiles discusiones sobre las mejores prácticas para las reacciones de trifosforilación química y a Gerald Joyce por sus útiles comentarios. Este trabajo fue apoyado por la subvención MCB 2114588 de la National Science Foundation.
Materiales
| Name | Company | Catalog Number | Comments |
| 0.22 µm polyethersulfone syringe filter | MilliporeSigma | SLMP025SS | Syringe filter for removing crushed polyacrylamide gel particles (Section 5) |
| 0.22 µm PTFE syringe filter | MilliporeSigma | SLLG013SL | Syringe filter for removing CPG resin (Section 5) |
| 1 mL plastic syringes | ThermoFisher Scientific | 14-823-434 (BD 309659) | Components of triphosphorylation apparatus (sections 2–4) |
| 1,4-Dioxane, anhydrous | MilliporeSigma | 296309 | Triphosphorylation solvent (sections 2–4) |
| 2-Chloro-4H-1,3,2-benzodioxaphosphorin-4-one, Salicyl Phosphorochloridite (SalPCl) | MilliporeSigma | 324124 | Triphosphorylation reagent (sections 2–4) |
| 30 mL glass bottles | MilliporeSigma | 23232 | Bottles for preparing triphosphorylation solvents and TBAP solution (section 2) |
| 3-way Stopcock, polycarbonate/polypropylene | Bio-Rad Laboratories | 7328103 | Component of triphosphorylation apparatus (sections 2–4) |
| 40% acrylamide/bis-acrylamide solution, 19:1 | Bio-Rad Laboratories | 1610144 | For PAGE (sections 5–7) |
| Acetonitrile, anhydrous, 100 mL | Glen Research | 40-4050-50 | Triphosphorylation solvent (sections 2–4) |
| Ammonia-neutralizing Trap | ThermoFisher Scientific | ANT100 and ANS121 | For use with Speedvac DNA130 (section 5) |
| Ammonium persulfate (APS) | Bio-Rad Laboratories | 1610700 | For PAGE, catalyst for acrylamide polymerization (sections 5–7) |
| Aqueous ammonia, 28% | MilliporeSigma | 338818 | For preparing AMA deprotection reagent (section 5) |
| Aqueous methylamine, 40% | TCI America | TCI-M0137 | For preparing AMA deprotection reagent (section 5) |
| Automated DNA/RNA oligonucleotide synthesizer | PerSeptive Biosystems | Expedite 8909 DNA/RNA Synthesizer | any column-based synthesizer is acceptable (section 1) |
| Bead-capture magnet | ThermoFisher Scientific | 12320D | For streptavidin bead capture (section 7) |
| Bromophenol blue | Bio-Rad Laboratories | 1610404 | For PAGE urea loading buffer (section 5) |
| Deep vacuum oil pump | ThermoFisher Scientific | VLP200-115 | For use with lyophilizer (section 5) |
| Drierite dessicant, 10-20 mesh | MilliporeSigma | 737828 | Desiccant for storing triphosphorylation chemicals and equipment (sections 1–2) |
| D-RNA 27.3t cross-chiral polymerase | prepared in house18 | 5′-GGUGGUGGAC GUGAUCAUUA CGGAUCACUA ACUCGUCAGU GCAUUGAGAA GGAGAAUAAA AUGCACAUAG GUCGAAAGAC CUUAUACAAG AACUGUAUCA CCGGAGGGCG AGCACCACC-3′ | For cross-chiral ribozyme reactions (section 7) |
| D-RNA CPG solid supports, 1,000Å, prepackaged 1 µmole synthesis columns | Glen Research | 20-3404-41E, 20-3415-41E, 20-3424-41E, 20-3430-41E | representative, for D-RNA oligonucleotide synthesis (section 1) |
| D-RNA TOM-protected phosphoramidites | ChemGenes | ANP-3201, 3202, 3203, 3205 | representative, for D-RNA oligonucleotide synthesis (section 1) |
| Empty Expedite Synthesis Columns, 1µm | Glen Research | 20-0021-01 | Synthesis columns for use with Expedite DNA/RNA synthesizer (section 1) |
| EPPS, N-(2-Hydroxyethyl)piperazine-N′-(3-propanesulfonic acid), solid | MilliporeSigma | E1894 | Ribozyme reaction buffer component (section 6) |
| Ethylenediaminetetraacetic acid (EDTA), solid | MilliporeSigma | EDS | Divalent metal ion chelator for use in various buffers (sections 5–7) |
| Filters for Expedite synthesis columns | Glen Research | 20-0021-0F | Expedite-style synthesis column filters, for use with empty synthesis columns (section 1) |
| Fluorescent/phosphorescent gel scanner | Cytiva | Amersham Typhoon RGB, 29187193 | For visualizing analytical PAGE (sections 6–7) |
| Formamide, deionized | VWR Life Science | 97062 | For PAGE formamide gel loading buffer (sections 6–7) |
| Gel image quantitation software | Cytiva | ImageQuant TL | For quantifying scanned gel images (section 6) |
| Glass desiccator | MilliporeSigma | CLS3121150 | Triphosphorylation solvent storage (section 2) |
| L-RNA CPG solid supports, 1,000Å, bulk | ChemGenes | N-4691-10, N-4692-10, N-4693-10, N-4694-10 | L-RNA oligonucleotide synthesis (section 1) |
| L-RNA hammerhead template | prepared in house18 | 5′-GCGCCUCAUC AGUCGAGCC-3′ | For cross-chiral ribozyme reactions (section 7) |
| L-RNA primer | prepared in house18 | 5′-fluorescein-GGCUCGA-3′ | For cross-chiral ribozyme reactions (section 7) |
| L-RNA TOM-protected phosphoramidites | ChemGenes | OP ANP-5201, 5202, 5203, 5205 | L-RNA oligonucleotide synthesis (section 1) |
| Lyophilizer/Freeze Dryer | VirTis | Benchtop K | For concentrating oligonucleotides (section 5) |
| Magnesium Chloride Hexahydrate, solid | MilliporeSigma | M2670 | For ribozyme reactions (sections 6–7) |
| N,N-Dimethylformamide, anhydrous | MilliporeSigma | 227056 | Triphosphorylation solvent (section 2) |
| NAP-25 Desalting column (Sephadex G-25 resin) | ThermoFisher Scientific | 45000150 | Disposable gravity-flow size exclusion chromatography columns containing Sephadex G-25 resin (section 5) |
| Non-coring stainless steel needle, 20 G | ThermoFisher Scientific | 14-815-410 | Needles for piercing rubber septa (sections 2–4) |
| Oligonucleotide extinction coefficient calculator | Integrated DNA Technologies | OligoAnalyzer Tool | Nearest-Neighbor Model Short Oligonucleotide 260nm extinction coefficient calculator (section 5) |
| Oxidizer solution, 0.1 M Iodine in THF/pyridine/water | ChemGenes | RN-1456 | Triphosphorylation reagent (section 4) |
| PAGE plates | Timberrock/CBS | NGP-250-BO and NO | For PAGE (sections 5–7) |
| PAGE power supply | Bio-Rad Laboratories | PowerPac HV 1645056 | For PAGE (sections 5–7) |
| PAGE spacers and combs (analytical) | Timberrock/CBS | VGS-0725 and VGC-0714 | For PAGE (sections 6–7) |
| PAGE spacers and combs (preparative) | Timberrock/CBS | VGS-3025R and VGC-3001 | For PAGE (section 5) |
| PAGE stand | Timberrock/CBS | ASG-250 | For PAGE (sections 5–7) |
| Parafilm M | ThermoFisher Scientific | 13-374-12 (Bemis PM999) | Wax sealing film for triphosphorylation apparatus (sections 2–4) |
| PCR thermocycler | Bio-Rad Laboratories | C1000 Touch Thermalcycler | For cross-chiral ribozyme reactions (section 7) |
| PD 10 Desalting column (Sephadex G-10 resin) | MilliporeSigma | GE17-0010-01 | Disposable gravity-flow size exclusion chromatography columns containing Sephadex G-10 resin, for oligonucleotides < 15 nt (section 5) |
| Phosphor screens | Cytiva | 28956480 | For visualizing 32P-labeled RNA (section 6) |
| Phosphoramidite synthesis reagents | Glen Research | 30-3142-52, 40-4050-53, 40-4012-52, 40-4122-52, 40-4132-52, 40-4060-62 | representative, for standard RNA/DNA synthesis (section 1) |
| Polypropylene screw-cap sealable tube | MilliporeSigma | BR780752 | 1.5 mL microcentrifuge tubes with screw-cap and silicone O-ring, for safe AMA deprotection (section 5) |
| Pyridine, anhydrous | MilliporeSigma | 270970 | Triphosphorylation solvent (section 2) |
| Reverse-phase liquid chromatography/electrospray ionization mass spectrometry (RP-LC/ESI-MS) | Novatia | n/a | Commercial service for LC/MS specializing in oligonucleotides (section 5) |
| Rubber Septa (ID x OD 7.9 mm x 14 mm), white | MilliporeSigma | Z564702 | Septa for preparing triphosphorylation solvents and TBAP (section 2) |
| Self-replicator ribozyme E | prepared in house14 | 5′-GGAAGUUGUG CUCGAUUGUU ACGUAAGUAA CAGUUUGAAU GGUUGAAGUA UGAGACCGCA ACUUA-3′ | For self-replicator ribozyme reactions (section 6) |
| Self-replicator substrate A | prepared in house14 | 5′-32P-GGAAGUUGUG CUCGAUUGUU ACGUAAGUAA CAGUUUGAAU GGUUGAAGUA U-3′-OH | For self-replicator ribozyme reactions (section 6) |
| Self-replicator substrate B, transcribed | prepared in house14 | 5′-pppGAGACCGCAA CUUA-3′ | For self-replicator ribozyme reactions (section 6) |
| Small Drying Traps, 4 Å molecular sieves | ChemGenes | DMT-1975 | Drying traps for DNA/RNA synthesizer phosphoramidites and triphosphorylation reagents (sections 1–2) |
| Sodium Chloride (NaCl), solid | MilliporeSigma | S7653 | Salt for use in various buffers (sections 5–7) |
| Sodium Hydroxide (NaOH), solid | MilliporeSigma | S8045 | Salt for use in various buffers (sections 5–7) |
| Statistical data-fitting software | GraphPad | Prism | For fitting data from analytical PAGE to kinetic models (section 6) |
| Streptavidin-coated magnetic beads | ThermoFisher Scientific | 65002 | For capturing biotin-labeled RNA in cross-chiral ribozyme reactions (section 7) |
| Sucrose | MilliporeSigma | 84097 | For PAGE urea loading buffer (section 5) |
| TBE running buffer, 10x | ThermoFisher Scientific | AAJ62788K3 | For PAGE (sections 5–7) |
| Tetrabutylammonium Fluoride, 1.0 M solution in Tetrahydrofuran | Aldrich | 216143 | For removing 2′-silyl protecting groups (section 5) |
| Tetramethylethylenediamine (TEMED) | Bio-Rad Laboratories | 1610801 | For polymerizing acrylamide for PAGE (sections 5–7) |
| Tributylamine | MilliporeSigma | 90781 | Triphosphorylation reagent (section 2) |
| Tributylammonium pyrophosphate (TBAP) | MilliporeSigma | P8533 | Triphosphorylation reagent (section 2) |
| Tris base | MilliporeSigma | T6666 | Buffering agent for use in various buffers (sections 5–7) |
| TWEEN20 polysorbate detergent | MilliporeSigma | P7949 | Neutral detergent for use with magnetic beads (Section 7) |
| Urea | MilliporeSigma | U5378 | For PAGE and gel loading buffer (sections 5–7) |
| UV-Vis spectrophotometer | ThermoFisher Scientific | NanoDrop 2000, ND2000 | For measuring oligonucleotide concentrations (section 5) |
| Vacuum centrifuge | ThermoFisher Scientific | Savant Speedvac DNA130-115 Vacuum Concentrator | For removing AMA and THF (section 5) |
| Xylene cyanol | Bio-Rad Laboratories | 1610423 | For PAGE urea loading buffer (section 5) |
Referencias
- Shuman, S. What messenger RNA capping tells us about eukaryotic evolution. Nature Reviews. Molecular Cell Biology. 3 (8), 619-625 (2002).
- Pichlmair, A., et al. RIG-I-mediated antiviral responses single-stranded RNA bearing 5'-phosphates. Science. 314 (5801), 997-1001 (2006).
- Hornung, V., et al. 5'-Triphosphate RNA is the ligand for RIG-I. Science. 314 (5801), 994-997 (2006).
- Myong, S., et al. Cytosolic viral sensor RIG-I is a 5'-triphosphate-dependent translocase on double-stranded RNA. Science. 323 (5917), 1070-1074 (2009).
- Takeuchi, O., Akira, S. Pattern recognition receptors and inflammation. Cell. 140, 805-820 (2010).
- Wang, Y., et al. Structural and functional insights into 5'-ppp RNA pattern recognition by the innate immune receptor RIG-I. Nature Structural & Molecular Biology. 17 (7), 781-787 (2010).
- Goubau, D., et al. Antiviral immunity via RIG-I-mediated recognition of RNA bearing 5'-diphosphates. Nature. 514 (7522), 372-375 (2014).
- Joyce, G. F. Forty years of in vitro evolution. Angewandte Chemie. 46 (34), 6420-6436 (2007).
- Robertson, M. P., Ellington, A. D. In vitro selection of an allosteric ribozyme that transduces analytes to amplicons. Nature Biotechnology. 17 (1), 62-66 (1999).
- Robertson, M. P., Hesselberth, J. R., Ellington, A. D. Optimization and optimality of a short ribozyme ligase that joins non-Watson-Crick base pairings. RNA. 7 (4), 513-523 (2001).
- Hesselberth, J. R., Robertson, M. P., Knudsen, S. M., Ellington, A. D. Simultaneous detection of diverse analytes with an aptazyme ligase array. Analytical Biochemistry. 312 (2), 106-112 (2003).
- Lam, B. J., Joyce, G. F. Autocatalytic aptazymes enable ligand-dependent exponential amplification of RNA. Nature Biotechnology. 27 (3), 288-292 (2009).
- Lam, B. J., Joyce, G. F. An isothermal system that couples ligand-dependent catalysis to ligand-independent exponential amplification. Journal of the American Chemical Society. 133 (9), 3191-3197 (2011).
- Olea, C., Horning, D. P., Joyce, G. F. Ligand-dependent exponential amplification of a self-replicating L-RNA enzyme. Journal of the American Chemical Society. 134 (19), 8050-8053 (2012).
- Olea, C., Joyce, G. F. Real-Time Detection of a Self-Replicating RNA Enzyme. Molecules. 21 (10), (2016).
- Sczepanski, J. T., Joyce, G. F. A cross-chiral RNA polymerase ribozyme. Nature. 515 (7527), 440-442 (2014).
- Tjhung, K. F., Sczepanski, J. T., Murtfeldt, E. R., Joyce, G. F. RNA-catalyzed cross-chiral polymerization of RNA. Journal of the American Chemical Society. 142 (36), 15331-15339 (2020).
- Bare, G. A. L., Joyce, G. F. Cross-chiral, RNA-catalyzed exponential amplification of RNA. Journal of the American Chemical Society. 143 (45), 19160-19166 (2021).
- Milligan, J. F., Groebe, D. R., Witherell, G. W., Uhlenbeck, O. C. Oligoribonucleotide synthesis using T7 RNA polymerase and synthetic DNA templates. Nucleic Acids Research. 15 (21), 8783-8798 (1987).
- Chelliserrykattil, J., Ellington, A. D. Evolution of a T7 RNA polymerase variant that transcribes 2'-O-methyl RNA. Nature Biotechnology. 22 (9), 1155-1160 (2004).
- Ibach, J., et al. Identification of a T7 RNA polymerase variant that permits the enzymatic synthesis of fully 2′-O-methyl-modified RNA. Journal of Biotechnology. 167 (3), 287-295 (2013).
- Esvelt, K. M., Carlson, J. C., Liu, D. R. A system for the continuous directed evolution of biomolecules. Nature. 472 (7344), 499-503 (2011).
- Pleiss, J. A., Derrick, M. L., Uhlenbeck, O. C. T7 RNA polymerase produces 5' end heterogeneity during in vitro transcription from certain templates. RNA. 4 (10), 1313-1317 (1998).
- Schenborn, E. T., Mierendorf, R. C. A novel transcription property of SP6 and T7 RNA polymerases: dependence on template structure. Nucleic Acids Research. 13 (17), 6223-6236 (1985).
- Martin, C. T., Muller, D. K., Coleman, J. E. Processivity in early stages of transcription by T7 RNA polymerase. Biochemistry. 27 (11), 3966-3974 (1988).
- Gholamalipour, Y., Karunanayake Mudiyanselage, A., Martin, C. T. 3' end additions by T7 RNA polymerase are RNA self-templated, distributive and diverse in character-RNA-Seq analyses. Nucleic Acids Research. 46 (18), 9253-9263 (2018).
- Vasilyev, N., Serganov, A. Preparation of short 5′-triphosphorylated oligoribonucleotides for crystallographic and biochemical studies. Nucleic Acid Crystallography: Methods and Protocols. , 11-20 (2016).
- Lebedev, A. V., Koukhareva, I. I., Beck, T., Vaghefi, M. M. Preparation of oligodeoxynucleotide 5'-triphosphates using solid support approach. Nucleosides, Nucleotides & Nucleic Acids. 20 (4-7), 1403-1409 (2001).
- Paul, N., Springsteen, G., Joyce, G. F. Conversion of a ribozyme to a deoxyribozyme through in vitro evolution. Chemistry & Biology. 13 (3), 329-338 (2006).
- Zlatev, I., et al. Efficient solid-phase chemical synthesis of 5'-triphosphates of DNA, RNA, and their analogues. Organic Letters. 12 (10), 2190-2193 (2010).
- Zlatev, I., Manoharan, M., Vasseur, J. -. J., Morvan, F. Solid-phase chemical synthesis of 5'-triphosphate DNA, RNA, and chemically modified oligonucleotides. Current Protocols in Nucleic Acid Chemistry. , (2012).
- Zlatev, I., et al. Automated parallel synthesis of 5'-triphosphate oligonucleotides and preparation of chemically modified 5'-triphosphate small interfering RNA. Bioorganic & Medicinal Chemistry. 21 (3), 722-732 (2013).
- Goldeck, M., Tuschl, T., Hartmann, G., Ludwig, J. Efficient solid-phase synthesis of pppRNA by using product-specific labeling. Angewandte Chemie. 53 (18), 4694-4698 (2014).
- Sarac, I., Meier, C. Efficient automated solid-phase synthesis of DNA and RNA 5′-triphosphates. Chemistry-A European Journal. 21 (46), 16421-16426 (2015).
- Sarac, I., Meier, C. Solid-phase synthesis of DNA and RNA 5'-O-triphosphates using cycloSal chemistry. Current Protocols in Nucleic Acid Chemistry. 64 (1), 4-67 (2016).
- Perez, J. T., et al. Influenza A virus-generated small RNAs regulate the switch from transcription to replication. Proceedings of the National Academy of Sciences of the United States of America. 107 (25), 11525-11530 (2010).
- Ludwig, J., Eckstein, F. Rapid and efficient synthesis of nucleoside 5'-0-(1-thiotriphosphates), 5'-triphosphates and 2',3'-cyclophosphorothioates using 2-chloro-4H-1,3,2-benzodioxaphosphorin-4-one. The Journal of Organic Chemistry. 54 (3), 631-635 (1989).
- Gaur, R. K., Sproat, B. S., Krupp, G. Novel solid phase synthesis of 2'-o-methylribonucleoside 5'-triphosphates and their α-thio analogues. Tetrahedron Letters. 33 (23), 3301-3304 (1992).
- Reddy, M. P., Hanna, N. B., Farooqui, F. Fast cleavage and deprotection of oligonucleotides. Tetrahedron Letters. 35 (25), 4311-4314 (1994).
- Hogrefe, R. I., McCaffrey, A. P., Borozdina, L. U., McCampbell, E. S., Vaghefi, M. M. Effect of excess water on the desilylation of oligoribonucleotides using tetrabutylammonium fluoride. Nucleic Acids Research. 21 (20), 4739-4741 (1993).
- Pitsch, S., Weiss, P. A., Jenny, L., Stutz, A., Wu, X. Reliable chemical synthesis of oligoribonucleotides (RNA) with 2′-O-[(Triisopropylsilyl)oxy]methyl(2′-O-tom)-protected phosphoramidites. Helvetica Chimica Acta. 84 (12), 3773-3795 (2001).
- Green, M. R., Sambrook, J. Isolation of DNA fragments from polyacrylamide gels by the crush and soak method. Cold Spring Harbor Protocols. 2019 (2), (2019).
- Paul, N., Joyce, G. F. A self-replicating ligase ribozyme. Proceedings of the National Academy of Sciences of the United States of America. 99 (20), 12733-12740 (2002).
- Lincoln, T. A., Joyce, G. F. Self-sustained replication of an RNA enzyme. Science. 323 (5918), 1229-1232 (2009).
- Robertson, M. P., Joyce, G. F. Highly efficient self-replicating RNA enzymes. Chemistry & Biology. 21 (2), 238-245 (2014).
- Singh, J., et al. Synthesis of modified nucleoside oligophosphates simplified: fast, pure, and protecting group free. Journal of the American Chemical Society. 141 (38), 15013-15017 (2019).
- Bellon, L. Oligoribonucleotides with 2'-O-(tert-butyldimethylsilyl) groups. Current Protocols in Nucleic Acid Chemistry. , (2001).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoExplorar más artículos
This article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados