Method Article
Trifosforilazione chimica degli oligonucleotidi
In questo articolo
Riepilogo
Gli oligonucleotidi 5′-trifosfati sono componenti onnipresenti nei percorsi biologici essenziali e hanno visto un uso crescente nelle applicazioni biotecnologiche. Qui descriviamo le tecniche per la sintesi e la purificazione di routine degli oligonucleotidi 5′-trifosfati, a partire da oligonucleotidi preparati con tecniche di sintesi automatizzate standard.
Abstract
Il 5′-trifosfato è una modifica essenziale dell'acido nucleico che si trova per tutta la vita e sempre più utilizzata come modificazione funzionale degli oligonucleotidi nelle biotecnologie e nella biologia sintetica. Gli oligonucleotidi 5′-trifosfati sono stati storicamente preparati in vitro con metodi enzimatici. Tuttavia, questi metodi sono limitati agli oligonucleotidi naturali dell'RNA, hanno forti preferenze di sequenza e tendono a produrre prodotti eterogenei. Nuovi metodi di trifosforilazione chimica completano sia il costo ridotto della sintesi automatizzata di oligonucleotidi mediante la chimica della fosforamidite sia la vasta gamma di modificazioni nucleotidiche ora disponibili. Pertanto, la sintesi di oligonucleotidi trifosfati di sequenza e lunghezza arbitrarie, e facoltativamente contenenti varie modifiche non naturali, è ora accessibile.
Questo documento presenta i metodi e le tecniche appropriate per la trifosforilazione chimica degli oligonucleotidi utilizzando salicil fosforocloridite e pirofosfato. Questo metodo utilizza reagenti disponibili in commercio, è compatibile con la maggior parte degli oligonucleotidi preparati con metodi standard di sintesi in fase solida e può essere completato in 2 ore dopo la sintesi degli oligonucleotidi, prima della deprotezione e della purificazione. Sono stati dimostrati due usi di oligonucleotidi chimicamente trifosforilati come substrati per gli enzimi dell'RNA catalitico, compresa la sintesi di una versione speculare del ribozima testa di martello da trifosfati non biologici di L-RNA.
Introduzione
La forma 5′-tripfosforilata dell'RNA è onnipresente in biologia in quanto è generata dalla trascrizione dell'RNA in tutti i domini della vita e dalla replicazione dell'RNA durante il ciclo di vita di molti virus a RNA. Questi trifosfati fungono da substrato per la formazione di mRNA con 7-metilguanilato negli eucarioti e, quindi, svolgono un ruolo essenziale nell'espressione proteica1. Al contrario, il trifosfato viene trattenuto in batteri e virus; pertanto, l'RNA 5′-trifosfati è riconosciuto dai regolatori di risposta immunitaria innati negli eucarioti 2,3,4,5,6,7. Al di fuori della biologia, una serie di ribozimi di RNA ligasi sono stati evoluti per utilizzare il 5′-trifosfato in vitro8 e modificati per l'uso nei saggi diagnostici 9,10,11,12,13,14,15. Uno di questi ribozimi può essere utilizzato per la sintesi template-dipendente di L-RNA, l'enantiomero non biologico "mirror-image" del D-RNA naturale, da piccoli oligonucleotidi L-RNA 5′-trifosfati 16,17,18. La preparazione di routine di oligonucleotidi trifosforilati di varia sequenza e composizione della spina dorsale è essenziale per studiare questi sistemi.
Il metodo più comune e accessibile per preparare l'RNA 5′-trifosfati in laboratorio è la trascrizione in vitro. Tuttavia, l'RNA prodotto con questo metodo è limitato in sequenza e dimensioni dai requisiti del promotore e del substrato dell'enzima RNA polimerasi. T7 RNA polimerasi e derivati specializzati sono le polimerasi più comuni utilizzate a questo scopo 19,20,21,22. L'RNA trascritto in vitro preparato con questi enzimi deve essere iniziato con una purina 5′-terminale ed è fortemente sbilanciato verso le purine nei primi 10 nucleotidi23,24. Inoltre, l'incorporazione enzimatica di nucleotidi modificati dalla base o dalla spina dorsale è nel migliore dei casi inefficiente e più spesso impossibile con le polimerasi naturali, limitando l'opportunità di produrre oligonucleotidi 5′-trifosfati composti da tutt'altro che D-RNA naturale. Un altro fattore limitante è che l'RNA generato dalla trascrizione in vitro può contenere una sostanziale eterogeneità di 5′ e 3′ ed è prodotto come prodotti estremamente eterogenei quando è inferiore a 20 nt 23,24,25,26,27.
Al contrario, la trifosforilazione chimica degli oligonucleotidi preparati mediante sintesi di fosforamidite in fase solida 28,29,30,31,32,33,34,35 può essere utilizzata per preparare oligonucleotidi 5′-trifosfati lunghi 3-50 nt, di qualsiasi sequenza. Inoltre, una vasta gamma di modificazioni dell'acido nucleico accessibili alla sintesi della fosforamidite possono essere aggiunte agli oligonucleotidi prima della 5′-tripfosforilazione 14,15,16,17,18,29,36. Molti di questi metodi utilizzano il reagente di fosfolilazione salicil fosforocloridite, sviluppato da Ludwig ed Eckstein per la trifosforilazione in fase di soluzione dei mononucleosidi37. La trifosforilazione degli oligonucleotidi con questo reagente si ottiene in fase solida mediante fosfolilazione dell'oligonucleotide 5′-idrossile, conversione al trifosfato per reazione con pirofosfato e ossidazione, seguita da procedure standard per la scissione dell'oligonucleotide dal supporto solido, la deprotezione e la purificazione (Figura 1)28.
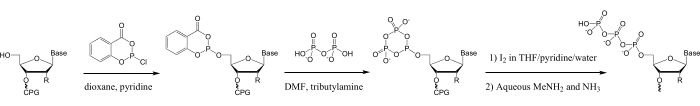
Figura 1: Schema per la trifosforilazione di oligonucleotidi sintetici. Nella prima fase, l'oligonucleotide 5ʹ-idrossile viene fosfolilato con SalPCl. Nella fase successiva, il fosfito 5ʹ-salicile viene fatto reagire con TBAP per formare il metafosfito ciclico, quindi nella terza fase ossidato per generare il ciclico 5ʹ-trimetafosfato nella soluzione di ossidazione del sintetizzatore DNA/RNA (0,1 M Iodio/piridina/H2O/THF), che viene rapidamente idrolizzato per produrre il 5ʹ-trifosfato lineare nella stessa soluzione28,33, 37. La successiva scissione alcalina dal supporto solido del CPG e la deprotezione dell'oligonucleotide nel MeNH2/ammoniaca acquosa idrolizzeranno qualsiasi trimetafosfato ciclico residuo nella forma lineare. Abbreviazioni: SalPCl = salicyl phosphorochloridite; TBAP = tributilammonio pirofosfato; THF = tetraidrofurano; CPG = vetro a pori controllati; MeNH2 = metilammina. Fare clic qui per visualizzare una versione più grande di questa figura.
Sebbene i primi rapporti pubblicati che utilizzano questo metodo spesso soffrissero di scarse rese e prodotti collaterali indesiderati 28,37,38, un attento mantenimento delle condizioni anidre è tutto ciò che è necessario per ottenere regolarmente rese elevate. Ciò può essere ottenuto mediante un'attenta preparazione dei reagenti e l'uso di un semplice dispositivo di reazione assemblato da componenti in plastica standard. Qui, dimostriamo i passaggi appropriati per la trifosforilazione chimica degli oligonucleotidi, compresa la preparazione dei reagenti, l'assemblaggio della camera di reazione, la reazione di trifosforilazione e la successiva deprotezione e purificazione degli oligonucleotidi trifosforilati. È incluso anche l'uso rappresentativo di oligonucleotidi trifosforilati 5′ come substrati per ribozimi ligasi per la sintesi di prodotti di acidi nucleici più grandi con dorsali naturali di D-RNA e L-RNA abiotico.
Protocollo
1. Sintesi automatizzata in fase solida di oligonucleotidi 5′-idrossilici su un supporto solido
- Preparare il sintetizzatore automatico DNA/RNA con reagenti e fosforamimiti in base alla composizione oligonucleotidica target e alle istruzioni dello strumento.
- Caricare una colonna di sintesi contenente supporto solido sul sintetizzatore e sintetizzare oligonucleotidi secondo i protocolli dello strumento del sintetizzatore.
NOTA: La procedura di trifosforilazione è stata ottimizzata per oligonucleotidi preparati alla scala 1 μmole. - Rimuovere il gruppo protettivo 5′-dimetossitrityl per produrre l'oligonucleotide 5′-idrossile supportato da solidi come parte della sintesi oligonucleotidica nella fase precedente, o eseguendo una fase di detritolazione terminale secondo i protocolli dello strumento del sintetizzatore.
- Rimuovere la colonna contenente l'oligonucleotide 5′-idrossile sul supporto solido dal sintetizzatore, asciugare sotto vuoto per 10 minuti per rimuovere il solvente residuo e procedere alla trifosforilazione (sezioni 3 e 4) una volta preparati i materiali per la trifosforilazione (sezione 2).
NOTA: Se non utilizzata immediatamente, la colonna essiccata può essere conservata in atmosfera normale in un contenitore di plastica sigillato con essiccante a -20 °C. In questa fase non è necessaria un'ulteriore essiccazione poiché la colonna viene accuratamente asciugata prima della trifosforilazione nel paragrafo 3.
2. Preparazione dei materiali per la trifosforilazione
- Collegare una sorgente di argon secco con pressione regolabile a un collettore di gas con almeno due linee e collegarsi a un gorgogliatore. Assicurarsi che le linee terminino in siringhe di plastica da 1 mL per facilitare il collegamento all'apparato di reazione.
- Raccogliere attrezzature da utilizzare durante la trifosforilazione, tra cui siringhe di plastica da 1 mL, un rubinetto in polipropilene a tre vie, aghi noncoranti, tubi in polipropilene da 1,5 ml e una piccola spatola metallica. Conservarli in un contenitore o essiccatore sigillato con essiccante a temperatura ambiente per almeno 1 giorno prima dell'uso.
- Preparare 30 mL ciascuno di 1,4-diossano anidro, 3:1 diossano:piridina in volume, N,N-dimetilformammide (DMF) e acetonitrile (ACN) in flaconi di vetro essiccati da 30 mL con trappole per essiccazione (setacci molecolari da 4 Å in pacchetti di membrana sigillati) almeno 1 giorno prima dell'uso. Sigillare con setti di gomma e conservare in un essiccatore con essiccante.
- Conservare la 2-cloro-4H-1,3,2-benzodioxafosforina-4-one solida (salicil fosforocloridite, SalPCl) nel suo contenitore originale all'interno di un barattolo sigillato con essiccante a 4 °C. Lavare sempre il contenitore con argon tra un uso e l'altro.
- Preparare una soluzione di pirofosfato di tributilammonio (TBAP) almeno 5 giorni prima della reazione di trifosforilazione:
- Pesare 1-5 g di TBAP solido in una bottiglia di vetro essiccata da 30 mL e sciogliere in 1 mL di DMF e 0,5 mL di tributilamina per g di TBAP.
- Aggiungere tre trappole per l'essiccazione, sigillare la bottiglia con un setto di gomma sotto argon e bollare con argon per 30 minuti a degassare.
- Conservare all'interno di un barattolo sigillato con essiccante a 4 °C per 5 giorni per consentire ai setacci molecolari di assorbire tutta l'acqua traccia. Conservare il barattolo a -20 °C e preparare fresco dopo 6 mesi.
3. Assemblaggio e utilizzo dell'apparato di trifosforilazione
- Lasciare che la colonna di sintesi si riscaldi a temperatura ambiente se si recupera dallo stoccaggio a -20 °C.
- Assemblate la camera di reazione mostrata nella Figura 2:
- Preparare l'anticamera: rimuovere lo stantuffo da una siringa asciutta da 1 mL, tagliare la parte superiore della siringa con forbici o una lama di rasoio e attaccare la siringa alla colonna di sintesi. Attaccare il rubinetto a tre vie alla parte superiore della siringa e fissare l'ingresso laterale del rubinetto alla fonte di argon secco con il gorgogliatore, in modo che l'ingresso superiore del rubinetto sia la porta di iniezione del reagente.
- Fissare questo apparecchio a un supporto con morsetti e sigillare tutti i giunti a monte con film sigillante a cera. Regolare il rubinetto in modo che la porta di iniezione sia chiusa e l'apparecchio sia aperto alla sorgente di argon. Chiudere il gorgogliatore e lasciare che l'argon a bassa pressione (<10 psi) scorra attraverso la camera di reazione per 5 minuti.
NOTA: Più camere di reazione possono essere impostate in parallelo agli oligonucleotidi tripfosforilato 2-4. Tuttavia, una linea dal collettore deve essere riservata alla fornitura di argon alle bottiglie di reagente. - Riaprire il gorgogliatore e attaccare una siringa sul fondo della colonna di sintesi, che sarà la siringa di scarto. Tirare l'argon attraverso la colonna ripetutamente usando la siringa di scarto; quindi, riattaccare la siringa con lo stantuffo spinto completamente dentro.
NOTA: a meno che non si carichino reagenti, il rubinetto deve essere impostato in modo che la porta di iniezione sia chiusa e l'apparecchio aperto alla sorgente di argon, come mostrato nella Figura 2A. Allo stesso modo, la siringa di scarto deve essere attaccata e il giunto alla colonna di sintesi sigillato con pellicola sigillante a cera, a meno che non si rimuovano attivamente i reagenti.
- Per aggiungere un reagente o un solvente:
- Attaccare un ago alla fonte di argon secco e inserirlo nel setto del reagente o del flacone di solvente, facendo attenzione a non immergere l'ago nel contenuto del flacone.
- Assemblare una siringa asciutta con un ago e inserirla nel setto del reagente o del flacone di solvente, senza immergerla nel contenuto del flacone. Riempire la siringa con argon, estrarre l'ago dal setto ed espellere l'argon. Riempire la siringa con argon ed espellere di nuovo; quindi, riempire la siringa con il volume richiesto di solvente o reagente sotto pressione di argon.
- Regolare il rubinetto di arresto sull'apparecchio in modo che la sorgente di argon sia chiusa e la porta di iniezione sia aperta (Figura 2B). Rimuovere rapidamente la siringa e l'ago riempiti dal flacone di origine, pulire via il solvente attaccato al lato o alla punta dell'ago e inserire l'ago nella porta di iniezione. Espellere il reagente nell'anticamera dell'apparecchio, rimuovere l'ago e chiudere la porta di iniezione, riaprendo l'apparecchio alla fonte di argon.
- Aspirare delicatamente il liquido dall'anticamera fino alla colonna di sintesi utilizzando la siringa di scarto in modo che tutto il liquido sia ora trattenuto nella siringa di scarto. Ora, spingere lentamente la soluzione verso l'alto nella colonna di sintesi, assicurandosi che non ci siano bolle di gas nella colonna. Per mescolare o agitare, tirare delicatamente la soluzione su e giù sopra la colonna con la siringa di scarto.
NOTA: Spostare sempre il liquido lentamente e delicatamente attraverso la camera di reazione per assicurarsi che non si rompano le guarnizioni, il che consente all'aria di entrare nell'apparecchio.
- Per rimuovere un reagente o un solvente dalla colonna:
- Estrarre lentamente la soluzione nella siringa di scarto. Dopo che la maggior parte della soluzione è passata nella siringa di scarto, estrarre l'argon per lavare il solvente rimanente dalla colonna.
- Rimuovere il film sigillante in cera attorno al giunto della siringa di scarto, quindi rimuovere la siringa ed eliminare la soluzione di scarto. Sostituire la siringa di scarto con una nuova siringa asciutta e richiudere l'articolazione con un film sigillante a cera.
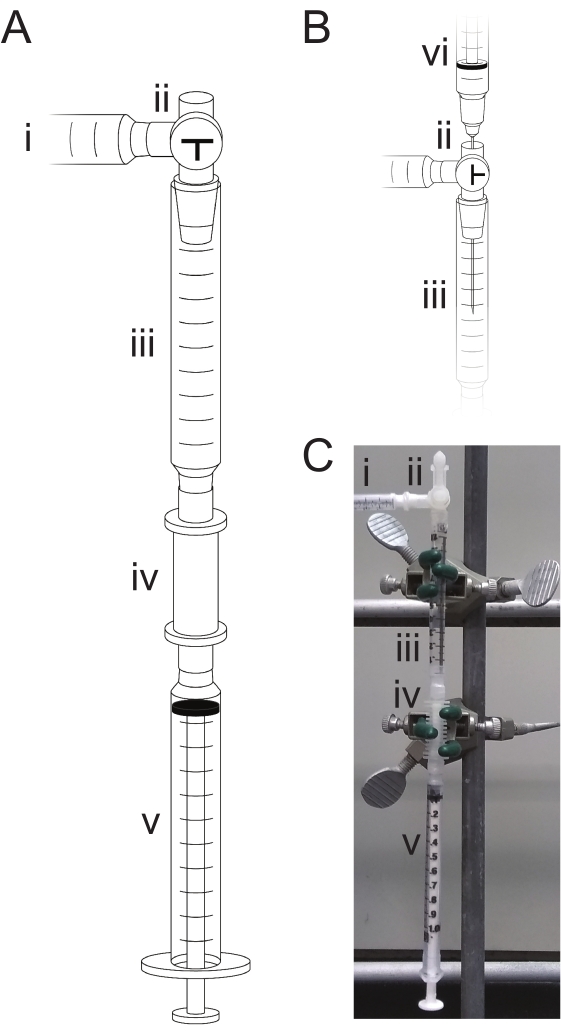
Figura 2: Apparato di trifosforilazione. Durante la miscelazione o le reazioni, il dispositivo (A) è aperto alla sorgente di argon (i) e chiuso all'aria regolando il rubinetto a tre vie (ii). I reagenti sono prelevati dall'anticamera (iii) nella colonna di sintesi (iv) per mezzo della siringa di scarto (v). I reagenti vengono rimossi aspirando tutto il liquido nella siringa di scarto (v) e scartandolo. Quando si caricano i reagenti (B), il rubinetto a tre vie (ii) è aperto all'atmosfera e il reagente viene caricato nell'anticamera (iii) per mezzo di una siringa e di un ago (vi). (C) Una fotografia dell'apparecchio assemblato come al punto A) per la miscelazione e la reazione dei reagenti. Fare clic qui per visualizzare una versione più grande di questa figura.
4. Trifosforilazione su colonna dell'oligonucleotide sintetico 5′-idrossile
- Rimuovere la soluzione SalPCl e TBAP dallo stoccaggio a -20 °C e lasciarli riscaldare a temperatura ambiente prima dell'uso.
- Aggiungere 200 μL di piridina/diossano all'anticamera, conformemente ai passaggi 3.3.1-3.3.3. Tuttavia, non caricare il solvente sulla colonna di sintesi fino al punto 4.4.
- Utilizzare una spatola metallica secca per pesare 6-12 mg di SalPCl in un tubo microcentrifuga asciutto da 1,5 mL e sciogliere in 100 μL di diossano siringando delicatamente il solvente su e giù all'interno del tubo microcentrifuga.
- Aggiungere il SalPCl disciolto all'anticamera e caricarlo sulla colonna di sintesi, seguendo il passaggio 3.3. Lasciare reagire per 15 minuti, agitando la soluzione ogni 5 minuti. Rimuovere ed eliminare la soluzione SalPCl secondo il passaggio 3.4.
NOTA: SalPCl viene aggiunto in grande eccesso e eliminerà l'acqua assorbita durante la preparazione e il caricamento nella camera di reazione. Tuttavia, l'introduzione di qualsiasi umidità durante le fasi 4.5 e 4.6 comprometterà la resa finale dell'oligonucleotide 5′-trifosfato. - Aggiungere 250 μL di soluzione TBAP all'anticamera e caricarla sulla colonna di sintesi, seguendo il passaggio 3.3. Lasciare reagire per 20 minuti, agitando ogni 5 minuti. Rimuovere ed eliminare la soluzione TBAP in base al passaggio 3.4.
- Lavare la colonna con 0,5 mL di DMF, quindi 0,5 mL di ACN, rimuovendo il solvente dopo ogni aggiunta secondo i passaggi 3.3 e 3.4.
- Aggiungere 250 μL di soluzione ossidante (0,1 M di iodio in tetraidrofurano (THF)/piridina/acqua, 88:10:2) all'anticamera e caricarlo sulla colonna di sintesi, seguendo il passaggio 3.3. Lasciare reagire per 30 minuti, agitando ogni 10 minuti. Rimuovere ed eliminare la soluzione ossidante secondo il punto 3.4.
- Lavare la colonna con 0,5 ml di ACN e rimuovere, secondo i passaggi 3.3 e 3.4.
- Smontare l'apparato di reazione. Lavare la colonna di sintesi con 5 ml di ACN e asciugare la colonna.
5. Scissione da supporto solido, deprotezione e purificazione
- Rimuovere la resina di supporto solida essiccata dalla colonna di sintesi e trasferirla in un tubo sigillabile con tappo a vite in polipropilene da 1,5 mL con un o-ring in silicone.
- Sospendere la resina in 1 mL di una miscela 1:1 di ammoniaca acquosa al 28%-30% e metilammina acquosa al 40% (AMA) e sigillare saldamente il tubo. Incubare a 65 °C per 10 minuti con miscelazione intermittente mediante inversione delicata39. Utilizzare un trattamento più delicato a temperatura ambiente per 2 ore per oligonucleotidi più lunghi di 40 nt.
ATTENZIONE: il riscaldamento della soluzione AMA metterà il tubo ad alta pressione. Se il tubo non è sigillato ermeticamente o non utilizza un o-ring compatibile con l'ammoniaca (silicone), il tubo potrebbe sfiatare gas o perdere solvente, compromettendo potenzialmente la sicurezza o la resa del prodotto finale. Non aprire mai il tubo mentre è al di sopra della temperatura ambiente, poiché la soluzione AMA calda può evolvere violentemente il gas. - Raffreddare il tubo sul ghiaccio e centrifugarlo brevemente (6.000-12.000 × g per 10 s). Rimuovere il surnatante dalla resina, filtrare attraverso una siringa dotata di un filtro da 0,2 μm e trasferirlo in un nuovo tubo sterile in polipropilene. Evaporare la soluzione a secco utilizzando una centrifuga sottovuoto dotata di una trappola chimica neutralizzante per ammoniaca.
NOTA: Se l'oligonucleotide sintetico non contiene nucleotidi di RNA con gruppi protettivi 2′-sililici, andare al passaggio 5.8. - Rimuovere i gruppi di protezione del silile sciogliendo il materiale essiccato in 1 mL di fluoruro di tetrabutilammonio 1 M (TBAF) in THF, riscaldando a 55 °C, agitando se necessario per sciogliere completamente l'oligonucleotide e incubando a temperatura ambiente per 16-24 ore40,41.
- Spegnere la soluzione TBAF con 1 mL di tampone Tris da 1 M, pH 7,5, e rimuovere THF utilizzando una centrifuga a vuoto.
- Rimuovere i sali TBAF utilizzando una colonna di esclusione delle dimensioni usa e getta, seguendo le istruzioni del produttore. Per oligonucleotidi inferiori a 15 nt, confermare l'eluizione del prodotto raccogliendo l'eluato in frazioni e identificando le principali frazioni di prodotto per assorbanza a 260 nm su uno spettrofotometro UV-Vis.
- Concentrare l'oligonucleotide dell'RNA deprotetto mediante liofilizzazione o centrifuga sotto vuoto se inferiore a 15 nt, o per precipitazione di etanolo se superiore a 15 nt.
- Preparare una scala preparativa 10%-20% poliacrilammide/8 M urea/1x TBE gel utilizzando 19:1 mono:bis acrilammide stock, secondo i protocolli appropriati per dimensione oligonucleotide, piastra gel e supporto. Assemblare la piastra gel nel supporto gel con 1x TBE nei serbatoi e pre-eseguire a 35 W (o come appropriato per il formato della piastra gel) per almeno 30 minuti.
- Sciogliere l'oligonucleotide solido nel tampone di carico del gel ureico (8 M urea, 10% saccarosio, 50 mM Tris, pH 8, 1 mM EDTA con bromofenolo e xilene cianolo) e riscaldare a 80 °C. Caricare sul gel di poliacrilammide e correre per 1-2 ore a 25-35 W (o come appropriato per il formato della piastra gel e la dimensione dell'oligonucleotide).
NOTA: il blu di bromofenolo o il cianolo xilene devono essere esclusi dal tampone di carico del gel per oligonucleotidi inferiori a 15 nt se uno dei due co-migra con il prodotto, poiché è difficile rimuovere questi coloranti dall'oligonucleotide eluito senza precipitazione di etanolo. - Quando l'elettroforesi su gel è completata, rimuovere la piastra gel dal supporto gel, smontare la piastra gel e avvolgere il gel in un film di polivinilcloruro. Identificare le bande di prodotto mediante back-shadowing con luce UV a 254 nm e asportare la banda principale del prodotto utilizzando una lama di rasoio.
- Estrarre l'oligonucleotide dal gel asportato con il metodo di frantumazione e ammollo42.
- Schiacciare il gel asportato estrudendolo attraverso una siringa di plastica o meccanicamente.
- Per oligonucleotidi di lunghezza superiore a 15 nt:
- Eluire in 3x volumi di tampone di schiacciamento e ammollo (300 mM NaCl; 10 mM Tris, pH 8; 1 mM EDTA) per almeno 12 ore con agitazione o agitazione.
- Rimuovere i pezzi di gel solido facendo passare la soluzione attraverso una siringa dotata di un filtro da 0,2 μm e concentrare l'oligonucleotide mediante precipitazione di etanolo.
- Per oligonucleotide inferiore a 15 nt:
- Eluire in 3x volumi di acqua priva di nucleasi per almeno 12 ore con agitazione o agitazione.
- Rimuovere i pezzi di gel solido facendo passare la soluzione attraverso una siringa dotata di un filtro da 0,2 μm e concentrare mediante liofilizzazione.
- Rimuovere i sali e i soluti residui utilizzando una colonna di esclusione delle dimensioni usa e getta come nel passaggio 5.6. Concentrato per liofilizzazione.
- Conservare oligonucleotidi trifosforilati purificati a -20 °C in tampone TE (10 mM Tris, pH 8; 1 mM EDTA) o in un tampone di stoccaggio oligonucleotidico simile.
- Determinare la concentrazione dell'oligonucleotide misurando l'assorbanza a 260 nm utilizzando uno spettrofotometro UV-Vis.
NOTA: Il coefficiente di estinzione stimato dell'oligonucleotide non deve essere influenzato dal suo stato di fosforilazione 5′ e può essere calcolato dalla sua sequenza utilizzando un calcolatore del coefficiente di estinzione degli oligonucleotidi. - Verificare la trifosforilazione mediante spettrometria di massa. Cercare una massa attesa di +239,94 Da rispetto a quella dell'oligonucleotide 5′-idrossile.
NOTA: La spettrometria di massa a laser assistito da matrice o la spettrometria di massa a ionizzazione elettrospray (MALDI-MS o ESI-MS, rispettivamente) sono adatte per identificare lo stato di trifosforilazione dell'oligonucleotide quando si utilizzano protocolli ottimizzati per gli acidi nucleici. ESI-MS fornisce risultati più coerenti, tuttavia, a causa dei minori tassi di frammentazione degli ioni; un servizio commerciale rappresentativo è fornito nella Tabella dei Materiali.
- Determinare la concentrazione dell'oligonucleotide misurando l'assorbanza a 260 nm utilizzando uno spettrofotometro UV-Vis.
6. Oligonucleotidi trifosforilati come substrati per l'autoreplicazione del ribozima
ATTENZIONE: 32P è un isotopo radioattivo e le seguenti fasi devono essere eseguite utilizzando protocolli di sicurezza standard per lavorare con materiali radioattivi in un laboratorio e da un ricercatore certificato per l'uso di materiali radioattivi dai dipartimenti competenti per la salute e la sicurezza ambientale. In alternativa, il substrato di ribozima A autoreplicante può essere preparato sinteticamente con un'etichetta di 5′-fluoresceina14 e ripreso in modo fluorescente, come nel passaggio 7.9.
- Preparare la soluzione A come miscela di ribozima E autoreplicatore e substrato di RNA A5′-32marcato P a concentrazioni rispettivamente di 0,30 μM e 30 μM. Preparare soluzioni B-trascritte e B-sintetiche con substrato di RNA trifosforilato B da 15 μM, preparate mediante trascrizione in vitro 14 o trifosforilazione chimica come sopra, rispettivamente, in tampone EPPS da 75 mM, pH 8,5 e 37,5 MgCl2. Portare entrambe le soluzioni a 42 °C.
NOTA: Tutti i componenti dell'RNA sono elencati nella Tabella dei materiali. - Per avviare l'autoreplicazione, mescolare rapidamente 5 μL di soluzione A con 10 μL di soluzione B-trascritta o B-sintetica ad una concentrazione finale di 0,1 μM E, 10 μM A, 10 μM B, 25 mM MgCl2 e tampone EPPS da 50 mM, pH 8,5. Incubare la miscela di reazione a 42 °C.
- A intervalli regolari, prelevare aliquote da 0,5 μL e spegnere in 9,5 μL di tampone di carico in gel di formammide (95% formammide; 10 mM EDTA, pH 8).
- Preparare un gel analitico di poliacrilammide/8 M urea/1x TBE, secondo protocolli appropriati per la piastra gel e il supporto. Assemblare il gel fuso e la piastra in un supporto in gel, riempire i serbatoi con 1x TBE e prerun a 40 W (o come appropriato per diverse piastre e supporti in gel) per 30 min.
- Riscaldare i campioni di reazione temprati a 80 °C, caricare 5 μL del campione in ciascun pozzetto e far funzionare il gel a 40 W per circa 40 minuti (o come appropriato per diverse piastre e supporti di gel).
- Rimuovere la piastra di gel dal supporto del gel, smontare e avvolgere il gel in un film di polivinilcloruro. Coprire il gel con uno schermo al fosforo ed esporre per 1 ora (o come appropriato per 32P cpm); scansionare lo schermo utilizzando uno scanner gel fluorescente/fosforescente.
- Quantificare la resa di reazione utilizzando il software di quantificazione dell'immagine in gel.
- Aprire l'immagine gel utilizzando la Casella degli strumenti Analisi, scegliere Analisi | Definizione forma, selezionare Aree | forma rettangolare e disegnare rettangoli delle stesse dimensioni attorno a bande corrispondenti a A non reagita e al prodotto E per ogni volta ed entrambe le reazioni.
- Scegli analisi | Sottrazione dello sfondo, selezionare Aree | forma rettangolare e disegnare un rettangolo delle stesse dimensioni in una parte vuota dell'immagine gel. Modificare il metodo di sottrazione dello sfondo per tutti i rettangoli in Rettangolo immagine.
- Scegli Finestra | Finestra 2 Area, quindi Modifica | Esporta in Excel per esportare i volumi di pixel di banda quantificati in un file di foglio di calcolo.
- Tracciare la concentrazione del prodotto E rispetto al tempo e adattare i dati alla crescita logistica Eq (1) utilizzando un software di adattamento statistico dei dati:
[E] =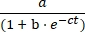 (1)
(1)
Dove a è l'estensione massima della reazione, b è il grado di sigmoidicità e c è il tasso di crescita esponenziale.- Nel foglio di calcolo esportato, dividere il volume E del prodotto per la somma dei volumi del substrato A e del prodotto E per determinare la resa frazionaria del prodotto per ogni volta ed entrambe le reazioni. Moltiplicare per la concentrazione iniziale del substrato A (10 μM) per determinare la resa del prodotto E in funzione del tempo.
- Nel software di adattamento dei dati statistici, scegliere File | Nuovi | Nuovo file di progetto, selezionare XY in Nuova tabella e grafico e fare clic su Crea. Immettere i tempi di reazione e i rendimenti del prodotto E per entrambe le reazioni in colonne adiacenti e etichettare le colonne in modo corrispondente (ad esempio, "tempo", "trascritto B", "B sintetico").
- Scegli Inserisci | Nuova analisi, seleziona analisi XY | Regressione non lineare (adattamento curva), quindi fate clic su OK. Scegliere curve di crescita | Crescita logistica e clicca OK; non regolare altri parametri. Osservare i parametri di adattamento e gli intervalli di confidenza in Risultati e il grafico dei punti dati e delle curve adattate in Grafici.
7. Copia cross-chirale di L-RNA
- Preparare 10 μL di soluzione di RNA contenente 20 μM D-RNA 27.3t polimerasi cross-chirale, 2 μM 5′-fluoresceina-marcato L-RNA primer, 4 μM biotinilato L-RNA hammerhead template e 20 μM ciascuno di L-RNA pppCUG, pppAUG, pppAGG e pppCGC, in 10 μL di 50 mM Tris, pH 8.3. Ricottura dell'RNA riscaldandolo a 90 °C per 1 minuto e raffreddandolo a 23 °C a 0,2 °C/s in un termociclatore PCR. Vedere la Tabella dei materiali per i dettagli sulla polimerasi, il primer e il modello.
- Incubare la soluzione di RNA a 17 °C e iniziare la reazione aggiungendo 10 μL di 2x tampone di avvio (400 mM MgCl2, 500 mM NaCl e 50 mM Tris, pH 8,3). Assicurarsi che le concentrazioni finali di tutti i componenti della reazione siano 10 μM di polimerasi, 1 μM di primer, 2 μM di modello, 10 μM ciascun trinucleotide 5′-trifosfato, 200 mM MgCl2, 250 mM NaCl e 50 mM Tris, pH 8,3.
- Mentre la reazione procede, prendere aliquote da 10 μL e spegnere con 5 μL di 0,5 M EDTA, pH 8.
- A ciascun campione di reazione estinto, aggiungere 0,1 mg di perline magnetiche rivestite con streptavidina (capacità di legame biotina-oligonucleotide di 20 pmol) sospese in 10 μL di 1 M NaCl in tampone TE con detergente neutro allo 0,05% per catturare primer estesi al trimero tramite il modello biotinilato e incubare per 30 minuti a temperatura ambiente con agitazione.
- Catturare le perline su un magnete di cattura del tallone, rimuovere e scartare il liquido e aggiungere 50 μL di soluzione di lavaggio (250 mM NaCl in TE con detergente neutro allo 0,05%). Mescolare le perline, catturarle di nuovo e rimuovere la soluzione di lavaggio. Ripeti ancora una volta.
- Per eluire i primer estesi dalle perline, aggiungere 50 μL di soluzione di eluizione (25 mM NaOH con detergente neutro allo 0,05%) e mescolare le perline. Ricatturare le perline, rimuovere la soluzione di eluizione, spegnere con 100 mM Tris (pH 7,5) e precipitare con etanolo.
- Preparare un gel analitico di poliacrilammide/8 M urea/1x TBE, secondo i protocolli appropriati per la piastra gel e il supporto. Assemblare il gel fuso e la piastra in un supporto in gel, riempire i serbatoi con 1x TBE e prerun a 40 W (o come appropriato per diverse piastre e supporti in gel) per 30 min.
- Sciogliere l'RNA precipitato in 10 μL di tampone di carico del gel di formammide e preparare il primer con etichetta finale non reagita a 0,5 μM nel tampone di carico del gel di formammide. Riscaldare i campioni a 80 °C, caricare 5 μL del campione in ciascun pozzetto ed eseguire il gel a 40 W per circa 40 minuti (o come appropriato per diverse piastre e supporti di gel).
- Rimuovere la piastra di gel dal supporto e scansionare utilizzando uno scanner di gel fluorescente / fosforescente per visualizzare i prodotti di estensione L-RNA cross-chiral.
Risultati
Gli oligonucleotidi devono essere sintetizzati utilizzando protocolli standard appropriati alle fosforamimiti e sintetizzatori automatici DNA/RNA, lasciando l'oligonucleotide prodotto allontanato dal supporto solido nella colonna di sintesi plastica originale, con il gruppo dimetossitrilcino terminale a 5ʹ rimosso per produrre il 5ʹ-idrossile libero (sezione 1). Tutti gli oligonucleotidi utilizzati in questa dimostrazione sono stati preparati utilizzando resina di vetro a pori controllati (CPG) da 1.000 Å come supporto solido e condotti alla scala 0,2 o 1 μmole. Esempi rappresentativi di colonne di sintetizzatori, resine, reagenti e fosforamimiti sono forniti nella Tabella dei materiali. Per le reazioni su larga scala, potrebbe essere necessario regolare i volumi e i tempi utilizzati nelle fasi successive.
La reazione di trifosforilazione viene condotta su colonna in una camera di reazione costruita su misura (Figura 2, sezione 3) utilizzando componenti standard disponibili in commercio elencati nella Tabella dei materiali e segue lo schema illustrato nella Figura 1 (sezione 4)28. È essenziale che le condizioni siano mantenute rigorosamente anidre durante la trifosforilazione e che tutti i solventi e i reagenti siano preparati in anticipo su setacci molecolari e lasciati asciugare completamente prima dell'uso (paragrafo 2). La trifosforilazione richiede in genere 2 ore per verificarsi e, successivamente, la colonna lavata e asciugata può essere trattata secondo le procedure standard di deprotezione e purificazione degli oligonucleotidi (sezione 5).
Dopo la deprotezione, gli oligonucleotidi trifosfati vengono purificati mediante elettroforesi su gel di poliacrilammide denaturante (PAGE), mostrando una singola banda di prodotto principale mediante back-shadowing UV che può essere asportata ed eluita dal gel. Il prodotto 5′-trifosfato è facilmente separato dai prodotti collaterali di reazione per oligonucleotidi corti, come mostrato per DNA trinucleotide 5ʹ-trifosfati, pppAAA e pppCCC, e L-RNA trinucleotide 5ʹ-trifosfato pppGAA in Figura 3A,B. Entrambi i prodotti 5′-idrossile e 5′-trifosfato per i trimeri di DNA AAA e CCC sono stati asportati e identificati mediante spettrometria di massa e corrispondentemente etichettati nella Figura 3A. Bande aggiuntive, come visibile per il trimero di DNA AAA, generalmente non contengono abbastanza materiale per recuperare e identificare. La presenza di queste bande, tuttavia, è correlata con masse di prodotto aggiuntive nei prodotti di reazione non purificati (Figura 3C), che rappresentano tipicamente prodotti collaterali 5′-difosfato, monofosfato e H-fosfonato, come discusso di seguito.
Dopo la purificazione di PAGE, gli oligonucleotidi più grandi possono essere eluiti utilizzando il metodo di schiacciamento e immersione42 e la successiva precipitazione di etanolo. Tuttavia, gli oligonucleotidi inferiori a 15 nt non possono essere precipitati in modo efficiente con etanolo e, pertanto, richiedono una procedura modificata per l'eluizione del gel (fase 5.11.3). La colonna di esclusione delle dimensioni usa e getta elencata nella Tabella dei materiali è classificata solo per l'uso con oligonucleotidi di lunghezza superiore a 10 nt. Tuttavia, abbiamo scoperto che gli oligonucleotidi corti come i trimeri possono essere efficacemente dissalati utilizzando il protocollo raccomandato dal produttore. Tuttavia, si raccomanda quando si dissalazione di oligonucleotidi corti (come nei passaggi 5.6 e 5.11.3) che l'eluato di colonna sia raccolto in frazioni e che le frazioni di prodotto siano identificate per assorbanza a 260 nm utilizzando uno spettrofotometro UV-Vis. Una colonna di esclusione dimensionale ottimizzata per oligonucleotidi più corti è fornita nella Tabella dei materiali come scelta alternativa. La resa finale dalla sintesi di oligonucleotidi in scala 1 μmole dopo la purificazione è di 50-300 nmol.
La trifosforilazione può essere confermata dalla spettrometria di massa, dove il prodotto trifosforilato ha una massa +239,94 Da maggiore dell'oligonucleotide 5′-idrossile, sebbene si osservi spesso la presenza di materiali corrispondenti al 5′-di- e al monofosfato (+159,96 e +79,98 Da, rispettivamente). Si può anche osservare un prodotto laterale 5′-H-fosfonato con una massa +63,98 Da dalla massa 5′-OH, e alti livelli di questo prodotto indicano che le condizioni durante la trifosforilazione non erano sufficientemente anidre. Prima della purificazione, gli oligonucleotidi deprotetti mostreranno tipicamente tutti questi prodotti (Figura 3C), mentre il materiale purificato mostrerà un picco corrispondente al prodotto 5′-trifosfato insieme a 5′-di- e monofosfati (Figura 3D,E).
La spettrometria di massa da sola in genere non fornisce una misura rigorosa della purezza del 5′-trifosfato a causa dei tassi differenziali di ionizzazione e frammentazione del trifosfato durante la ionizzazione. Per misurare la purezza del prodotto finale, si raccomanda la cromatografia liquida in fase inversa e l'ESI-MS tandem (RP-LC/ESI-MS), in particolare per gli oligonucleotidi più lunghi. L'analisi dei D-RNA 5ʹ-trifosfati pppACGAGG e pppGAGACCGCAACUUA da parte di RP-LC/ESI-MS (Figura 4A,B, rispettivamente) mostra una purezza tipica del prodotto finale, contenente il 20% di 5ʹ-difosfato poiché queste due specie sono difficili da separare quando presenti su oligonucleotidi più lunghi.
Gli oligonucleotidi sintetici 5′-trifosfato funzionano tipicamente bene o meglio dei materiali preparati enzimaticamente negli studi biochimici. Nella sezione 6, ad esempio, substrati di RNA 5′-trifosfato 14 nt preparati sinteticamente o mediante trascrizione in vitro sono stati confrontati in una reazione di autoreplicazione catalizzata dall'RNA 14,15,43,44,45. Il ribozima E catalizza l'unione dei substrati A e B per produrre una nuova copia di E in una reazione autocatalitica capace di crescita esponenziale (Figura 5A). I componenti A marcati E e 32P sono stati preparati mediante trascrizione in vitro e il substrato B trifosforilato è stato preparato sinteticamente, come descritto sopra, o mediante trascrizione in vitro 14. Il progresso della reazione di autoreplicazione è stato monitorato prelevando campioni periodici che sono stati analizzati mediante DENATURAZIONE DI PAGE e quantificati tramite uno scanner gel fluorescente/fosforescente. I dati risultanti, adatti a una funzione di crescita logistica, hanno rivelato che il substrato B trascritto o sintetico supporta la crescita esponenziale, ma il B sintetico fornisce una quantità leggermente maggiore di prodotto (Figura 5B). Questo risultato può riflettere l'eterogeneità compositiva alla fine 5′-end dell'RNA preparato mediante trascrizione in vitro 23,24.
La trifosforilazione chimica consente anche la sintesi di oligonucleotidi trifosfati che non possono essere preparati biologicamente, né in vitro né in cellule. Nella sezione 7, gli oligonucleotidi trifosfati non biologici composti da L-RNA, l'enantiomero del D-RNA naturale, preparato come nelle sezioni 1-5, sono stati utilizzati come substrati per il D-RNA "cross-chiral" polimerasi ribozima 27.3t (Figura 6A), che catalizza la polimerizzazione diretta dal modello di un prodotto L-RNA più lungo da oligonucleotide 5′-trifosfati L-RNA corto in modo generale di sequenza. Ad esempio, il ribozima può sintetizzare una versione L-RNA del motivo di auto-scissione della testa di martello (Figura 6B)18. I trifosfati di L-RNA trinucleotide purificati sono stati combinati con un primer L-RNA marcato con fluoresceina e un modello di L-RNA (Figura 6C) e hanno reagito con la ligasi cross-chirale. I campioni nel corso della reazione sono stati analizzati da PAGE e ripresi utilizzando uno scanner gel fluorescente / fosforescente per dimostrare la sintesi di una versione L-RNA del ribozima a martello codificato dal modello (Figura 6D).
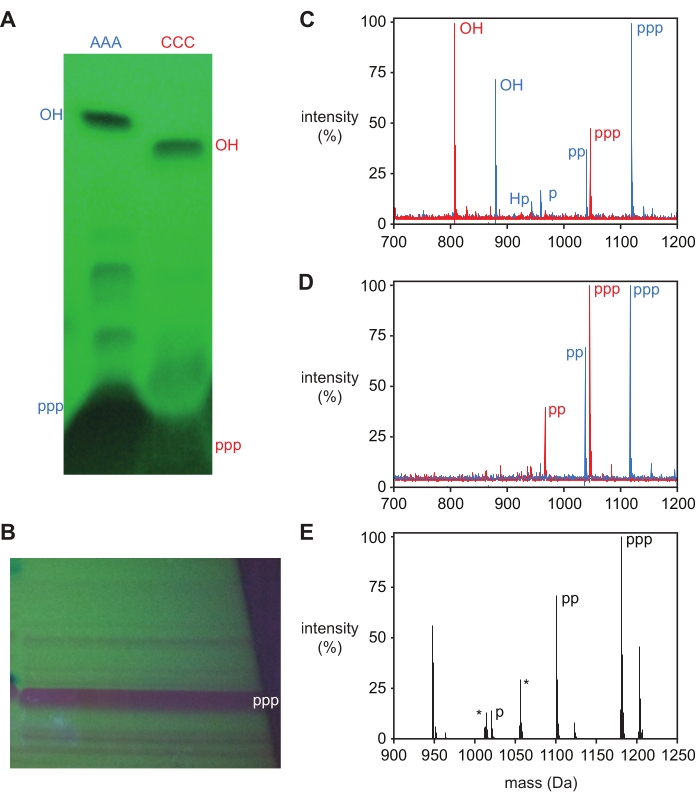
Figura 3: Purificazione del trinucleotide 5ʹ-trifosfati. (A) Analisi PAGE (visualizzata mediante UV-back-shadowing) della trifosforilazione dei trinucleotidi del DNA tri-deossiadenosina (AAA, blu) e tri-deossicitidina (CCC, rosso), intenzionalmente sovraccaricati per visualizzare prodotti collaterali minori. Sia il prodotto 5ʹ-trifosfato (ppp) che il materiale di partenza 5ʹ-idrossile (OH) sono stati asportati e identificati da MALDI-MS. (B) PAGINA preparatoria di trifosforilazione di L-RNA trinucleotide GAA, con banda di prodotto principale asportata e identificata come 5ʹ-trifosfato (ppp) da ESI-MS. C) MALDI-MS di prodotti grezzi di reazione dopo deprotezione e (D) prodotti purificati da (A). 5ʹ-trifosfato (ppp; pppAAA atteso 1.119 Da, osservato 1.118 Da; pppCCC atteso 1.047 Da, osservato 1.046); Sono etichettati 5ʹ-difosfato (pp), 5ʹ-monofosfato (p), 5ʹ-idrossile (OH) e 5ʹ-H-fosfonato (Hp). (E) Spettro di massa deconvoluto dall'iniezione diretta ESI-MS di prodotto isolato 5ʹ-trifosfato da (B), con picchi identificati etichettati (atteso 1.181,6 Da, osservato 1.181,0 Da). Si osservano anche prodotti a 5ʹ-difosfato (pp), così come picchi di ioni di sodio sia per i prodotti tri- che per quelli difosfato (+22 Da). I picchi di contaminanti comuni sono etichettati con un asterisco. Per facilitare il confronto, gli spettri di massa sono stati normalizzati alla massima intensità misurata in ciascuno spettro e sono riportati come percentuale rispetto a quel valore. Abbreviazioni: PAGE = elettroforesi su gel di poliacrilammide; MALDI-MS = desorbimento/ionizzazione laser assistita da matrice; ESI-MS = spettrometria di massa a ionizzazione elettrospray. Fare clic qui per visualizzare una versione più grande di questa figura.
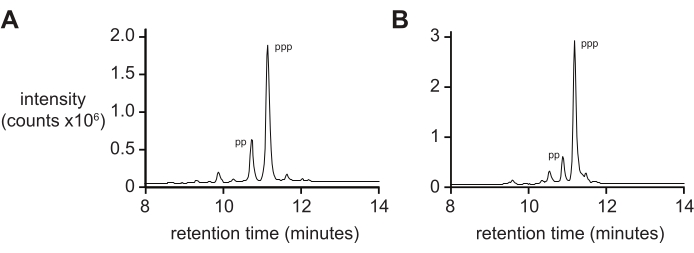
Figura 4: RP-LC analitico di 6 nt e 14 nt D-RNA oligonucleotide trifosfati. (A) 5ʹ-pppACGAGG-3ʹ e (B) 5ʹ-pppGAGACCGCAACUUA-3ʹ. Tandem ESI-MS ha identificato il picco maggiore di entrambi (~ 70%) come il 5ʹ-trifosfato (ppp), con minori quantità di 5ʹ-difosfato (pp). Abbreviazioni: RP-LC = cromatografia liquida in fase inversa; nt = nucleotidi; ESI-MS = spettrometria di massa a ionizzazione elettrospray. Fare clic qui per visualizzare una versione più grande di questa figura.
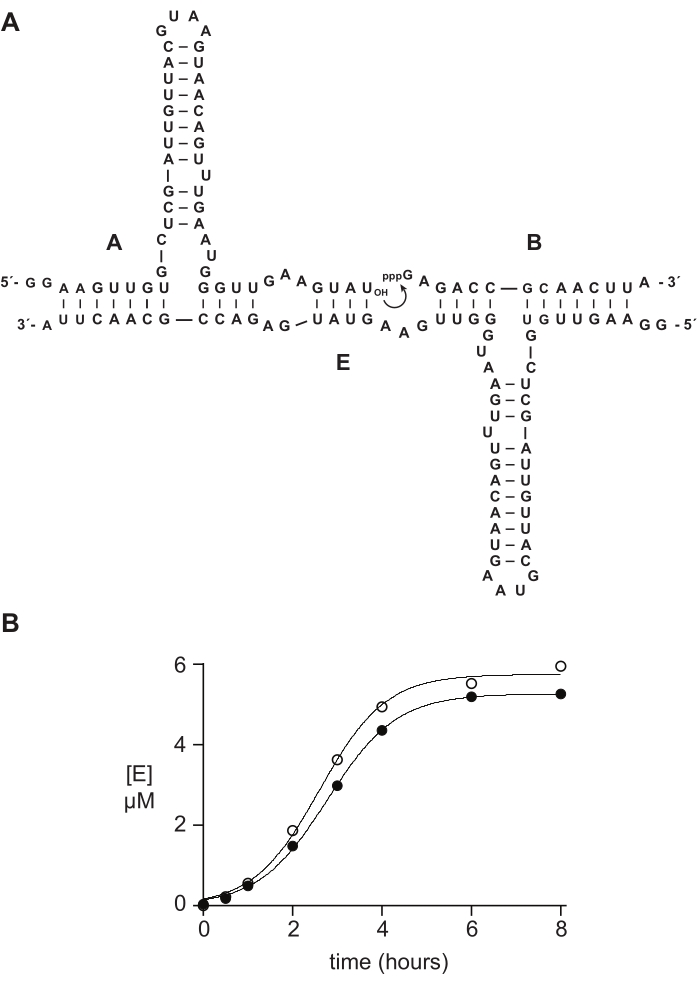
Figura 5: Confronto di substrati oligonucleotidi 5ʹ-trifosfato preparati mediante sintesi chimica o trascrizione in vitro. (A) Il ribozima E autoreplicante lega l'RNA A e l'RNA 5′-trifosforilato B. (B) Confronto delle reazioni di autoreplicazione utilizzando 10 μM A e 10 μM B, sia sintetici (cerchi aperti) che trascritti in vitro (cerchi riempiti). (B) I dati erano adatti all'equazione di crescita logistica: [E] = a / (1 + b e-ct), dove a è la resa finale, b è il grado di sigmoidicità e c è il tasso di crescita esponenziale. I tassi di crescita per le due reazioni erano identici, a 1,14 h-1, mentre l'estensione finale era superiore del 10% per le reazioni con B sintetico. Fare clic qui per visualizzare una versione più grande di questa figura.
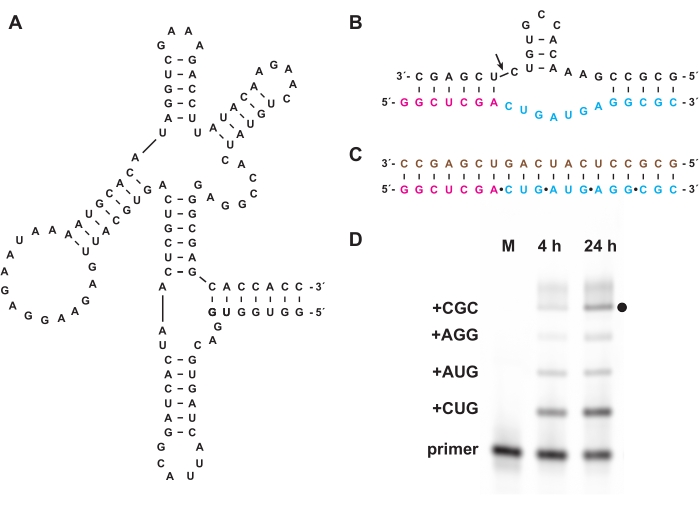
Figura 6: Polimerizzazione cross-chirale di L-RNA con un ribozima. (A) Il D-RNA 27.3t polimerasi ribozima, che catalizza la legatura template-dipendente di L-RNA. (B) Il prodotto L-RNA sintetizzato da 27.3t fa parte di un motivo di endonucleasi a martello. (C) Polimerizzazione L-RNA catalizzata di 27,3 t utilizzando un modello di L-RNA biotinilato (marrone), un primer L-RNA (magenta) marcato alla fine e quattro trifosfati trinucleotidici L-RNA (ciano), preparati sinteticamente. (D) ANALISI PAGE dei prodotti di estensione di (B) a 4 h e 24 h, mostrando ogni incorporazione di trinucleotide fino al prodotto a lunghezza intera (punto nero). Il primer L-RNA non reagito è incluso come marcatore di riferimento. Abbreviazioni: PAGE = elettroforesi su gel di poliacrilammide; M = marcatore di riferimento. Fare clic qui per visualizzare una versione più grande di questa figura.
Discussione
La procedura di trifosforilazione qui descritta è ampiamente compatibile con la sintesi di oligonucleotidi utilizzando la chimica standard della fosforamidite. I nucleosidici fosfomiditi dovrebbero avere gruppi protettivi labili di base compatibili con la deprotezione rapida in AMA39, compresi i gruppi standard β-cianoetile sul fosfito e isobutirrile, dimetilformamidile, acetil, fenossiacetile o 4-isopropilfenossiacetil sulle ammine esocicliche delle nucleobasi. I gruppi ribosio 2'-idrossile devono essere protetti da gruppi che proteggono il silile, t-butildimetilsilil (TBDMS) o tri-iso-propilsillilossimetile (TOM)40,41. Anche il gruppo pivaloilossimetile (PivOM) labile di base è stato segnalato per essere compatibile con la trifosforilazione chimica30.
Sono stati descritti diversi metodi per la trifosforilazione chimica degli oligonucleotidi sintetici 28,29,30,31,32,33,34,35. Abbiamo scoperto che la trifosforilazione con il reagente ludwig-Eckstein37 è una delle più accessibili, non richiedendo alcuna sintesi specializzata di reagenti e nessuna attrezzatura specializzata. Gli oligonucleotidi 5′-trifosfati preparati con questo metodo sono stati utilizzati di routine come substrati per i ribozimi dell'RNA ligasi, incluso l'uso di trifosfati oligonucleotidici L-RNA enzimaticamente inaccessibili per ottenere la sintesi dipendente dal modello e la replicazione di questo acido nucleico "mirror-image" 14,16,17,18 . Il metodo è adatto anche per la preparazione di piccoli RNA stem-loop 5′-trifosforilati che sono potenti attivatori della risposta immunitaria innata nei vertebrati 6,7.
Il reagente di Ludwig-Eckstein, salicil fosforocloridite37, è altamente reattivo all'acqua e elimina efficacemente l'acqua introdotta durante la dissoluzione del reagente prima del caricamento sulla colonna oligonucleotidica. Dopo questo punto, tuttavia, l'oligonucleotide 5′-fosfolilato reagirà preferenzialmente con qualsiasi acqua introdotta nel sistema su pirofosfato, formando un prodotto laterale 5′-H-fosfonato dopo il workup 28,37,38. Un'attenta preparazione dei reagenti di trifosforilazione e la camera di reazione di trifosforilazione assicurano che questo prodotto laterale non si formi. Per l'essiccazione con solvente, i setacci molecolari di tipo 4 Å sono venduti preconfezionati in sacchetti di teflon compatibili con la maggior parte dei solventi organici con vari marchi dalla maggior parte delle aziende di reagenti di sintesi di oligonucleotidi. Ulteriori precauzioni, come l'esecuzione della trifosforilazione in un vano portaoggetti in atmosfera anidra, non sono generalmente necessarie.
La reazione dell'oligonucleotide 5′-fosfolilato con TBAP forma un intermedio ciclico 5′-trimetafosfito, che viene poi ossidato al ciclico 5′-trimetafosfato usando una soluzione ossidante di sintesi oligonucleotidica (iodio in acqua/piridina/THF). Va notato che le soluzioni ossidanti commerciali utilizzano quantità variabili di iodio ed è essenziale utilizzare l'alta concentrazione di iodio di 0,1 M per garantire la completa ossidazione al trifosfato. Il prodotto ciclico viene idrolizzato al 5′-trifosfato lineare finale nella stessa soluzione37 e devono essere utilizzate soluzioni ossidanti anidre alternative se si desidera la linearizzazione con nucleofili diversi dall'acqua (vedi sotto per le applicazioni)33. L'eventuale trimetafosfato ciclico residuo, tuttavia, sarà linearizzato durante la successiva deprotezione alcalina dell'oligonucleotide. L'idrolisi del 5′-trimetafosfato ciclico produce solo il trifosfato lineare, piuttosto che ramificato37,46.
La deprotezione degli oligonucleotidi in genere non ha bisogno di essere modificata per ospitare la 5′ trifosforilazione, ma devono essere prese alcune precauzioni. Il trifosfato è relativamente stabile a breve esposizione a condizioni alcaline, ma bisogna fare attenzione a non esporre il trifosfato all'AMA più a lungo del necessario. Dovrebbe essere evitata la protezione dei gruppi che richiedono un trattamento più prolungato in ammoniaca o AMA per più di 10 minuti a 65 °C. Trattamenti più delicati, come 2 ore di ammoniaca a temperatura ambiente sono accettabili se compatibili con altri gruppi protettivi di fosforamidite. Un metodo di deprotezione comune e veloce per oligonucleotidi di RNA sintetico protetti da silile utilizza trietilammina triidrofluoruro e alta temperatura47; tuttavia, questo dovrebbe essere evitato quando si preparano RNA 5′-trifosfati poiché le condizioni acide prolungate accelerano l'idrolisi del trifosfato31,32.
Preparative PAGE ha dimostrato di essere il metodo più semplice e affidabile per la purificazione post-protezione degli oligonucleotidi trifosforilati 5′ (Figura 3 e Figura 4). Tuttavia, l'HPLC preparativo in fase inversa può essere utilizzato anche per purificare i prodotti trifosforilati. La presenza di prodotti a base di 5′-difosfato e, in misura minore, di 5′-monofosfato viene osservata di routine quando si verifica la trifosforilazione mediante spettrometria di massa. Abbiamo osservato la frammentazione del 5′-trifosfato durante la spettrometria di massa da materiale altamente puro preparato per sintesi chimica o trascrizione, in particolare se lo strumento non è ottimizzato per l'analisi degli oligonucleotidi. Tuttavia, l'analisi RP-LC mostra spesso che il 10%-20% del prodotto laterale 5′-difosfato è presente in oligonucleotidi 5′-trifosforilati più lunghi (Figura 4). I preparati commerciali di pirofosfato di tributilammonio possono essere contaminati fino al 20% di monofosfato, che produrrà 5′-difosfato come prodotto collaterale durante la trifosforilazione30,31. Un'attenta preparazione di questo reagente in-house può produrre scorte TBAP molto più pure31. Tuttavia, abbiamo scoperto che gli oligonucleotidi trifosforilati utilizzando fonti commerciali di TBAP mostrano ancora una reattività comparabile o maggiore se usati come substrati nelle reazioni enzimatiche (Figura 5B), rispetto al materiale preparato mediante trascrizione in vitro.
Un notevole ulteriore uso della trifosforilazione oligonucleotidica con il reagente di Ludwig-Eckstein sfrutta l'intermedio ciclico trimetafosfito33. Se la successiva fase di ossidazione viene condotta con perossido di t-butile 1 M negli esani, che viene spesso utilizzato per l'ossidazione degli oligonucleotidi in condizioni anidre, l'ossidazione del fosfito avviene senza idrolisi di apertura dell'anello, producendo il trimetafosfato ciclico. Questo intermedio può quindi essere fatto reagire con nucleofili primari di ammina o alcol per produrre 5′-trifosfati con modifiche al γ-fosfato. Queste modifiche includono l'aggiunta di un tag lipofilo collegato da un legame fosforamidato, che facilita la rapida purificazione specifica del trifosfato mediante RP-LC, seguita da idrolisi acida del tag dal trifosfato33. Le modifiche fluorescenti nella posizione γ-fosfato possono anche essere introdotte per l'uso come reporter fluorescenti in tempo reale per le reazioni di legatura catalizzate da ribozima15,33.
Divulgazioni
Gli autori dichiarano di non avere interessi finanziari concorrenti.
Riconoscimenti
Gli autori sono grati a Greg Springsteen, Natasha Paul, Charles Olea, Jr., Jonathan Sczepanski e Katrina Tjhung per le utili discussioni sulle migliori pratiche per le reazioni di trifosforilazione chimica e a Gerald Joyce per i commenti utili. Questo lavoro è stato sostenuto dalla sovvenzione MCB 2114588 dalla National Science Foundation.
Materiali
| Name | Company | Catalog Number | Comments |
| 0.22 µm polyethersulfone syringe filter | MilliporeSigma | SLMP025SS | Syringe filter for removing crushed polyacrylamide gel particles (Section 5) |
| 0.22 µm PTFE syringe filter | MilliporeSigma | SLLG013SL | Syringe filter for removing CPG resin (Section 5) |
| 1 mL plastic syringes | ThermoFisher Scientific | 14-823-434 (BD 309659) | Components of triphosphorylation apparatus (sections 2–4) |
| 1,4-Dioxane, anhydrous | MilliporeSigma | 296309 | Triphosphorylation solvent (sections 2–4) |
| 2-Chloro-4H-1,3,2-benzodioxaphosphorin-4-one, Salicyl Phosphorochloridite (SalPCl) | MilliporeSigma | 324124 | Triphosphorylation reagent (sections 2–4) |
| 30 mL glass bottles | MilliporeSigma | 23232 | Bottles for preparing triphosphorylation solvents and TBAP solution (section 2) |
| 3-way Stopcock, polycarbonate/polypropylene | Bio-Rad Laboratories | 7328103 | Component of triphosphorylation apparatus (sections 2–4) |
| 40% acrylamide/bis-acrylamide solution, 19:1 | Bio-Rad Laboratories | 1610144 | For PAGE (sections 5–7) |
| Acetonitrile, anhydrous, 100 mL | Glen Research | 40-4050-50 | Triphosphorylation solvent (sections 2–4) |
| Ammonia-neutralizing Trap | ThermoFisher Scientific | ANT100 and ANS121 | For use with Speedvac DNA130 (section 5) |
| Ammonium persulfate (APS) | Bio-Rad Laboratories | 1610700 | For PAGE, catalyst for acrylamide polymerization (sections 5–7) |
| Aqueous ammonia, 28% | MilliporeSigma | 338818 | For preparing AMA deprotection reagent (section 5) |
| Aqueous methylamine, 40% | TCI America | TCI-M0137 | For preparing AMA deprotection reagent (section 5) |
| Automated DNA/RNA oligonucleotide synthesizer | PerSeptive Biosystems | Expedite 8909 DNA/RNA Synthesizer | any column-based synthesizer is acceptable (section 1) |
| Bead-capture magnet | ThermoFisher Scientific | 12320D | For streptavidin bead capture (section 7) |
| Bromophenol blue | Bio-Rad Laboratories | 1610404 | For PAGE urea loading buffer (section 5) |
| Deep vacuum oil pump | ThermoFisher Scientific | VLP200-115 | For use with lyophilizer (section 5) |
| Drierite dessicant, 10-20 mesh | MilliporeSigma | 737828 | Desiccant for storing triphosphorylation chemicals and equipment (sections 1–2) |
| D-RNA 27.3t cross-chiral polymerase | prepared in house18 | 5′-GGUGGUGGAC GUGAUCAUUA CGGAUCACUA ACUCGUCAGU GCAUUGAGAA GGAGAAUAAA AUGCACAUAG GUCGAAAGAC CUUAUACAAG AACUGUAUCA CCGGAGGGCG AGCACCACC-3′ | For cross-chiral ribozyme reactions (section 7) |
| D-RNA CPG solid supports, 1,000Å, prepackaged 1 µmole synthesis columns | Glen Research | 20-3404-41E, 20-3415-41E, 20-3424-41E, 20-3430-41E | representative, for D-RNA oligonucleotide synthesis (section 1) |
| D-RNA TOM-protected phosphoramidites | ChemGenes | ANP-3201, 3202, 3203, 3205 | representative, for D-RNA oligonucleotide synthesis (section 1) |
| Empty Expedite Synthesis Columns, 1µm | Glen Research | 20-0021-01 | Synthesis columns for use with Expedite DNA/RNA synthesizer (section 1) |
| EPPS, N-(2-Hydroxyethyl)piperazine-N′-(3-propanesulfonic acid), solid | MilliporeSigma | E1894 | Ribozyme reaction buffer component (section 6) |
| Ethylenediaminetetraacetic acid (EDTA), solid | MilliporeSigma | EDS | Divalent metal ion chelator for use in various buffers (sections 5–7) |
| Filters for Expedite synthesis columns | Glen Research | 20-0021-0F | Expedite-style synthesis column filters, for use with empty synthesis columns (section 1) |
| Fluorescent/phosphorescent gel scanner | Cytiva | Amersham Typhoon RGB, 29187193 | For visualizing analytical PAGE (sections 6–7) |
| Formamide, deionized | VWR Life Science | 97062 | For PAGE formamide gel loading buffer (sections 6–7) |
| Gel image quantitation software | Cytiva | ImageQuant TL | For quantifying scanned gel images (section 6) |
| Glass desiccator | MilliporeSigma | CLS3121150 | Triphosphorylation solvent storage (section 2) |
| L-RNA CPG solid supports, 1,000Å, bulk | ChemGenes | N-4691-10, N-4692-10, N-4693-10, N-4694-10 | L-RNA oligonucleotide synthesis (section 1) |
| L-RNA hammerhead template | prepared in house18 | 5′-GCGCCUCAUC AGUCGAGCC-3′ | For cross-chiral ribozyme reactions (section 7) |
| L-RNA primer | prepared in house18 | 5′-fluorescein-GGCUCGA-3′ | For cross-chiral ribozyme reactions (section 7) |
| L-RNA TOM-protected phosphoramidites | ChemGenes | OP ANP-5201, 5202, 5203, 5205 | L-RNA oligonucleotide synthesis (section 1) |
| Lyophilizer/Freeze Dryer | VirTis | Benchtop K | For concentrating oligonucleotides (section 5) |
| Magnesium Chloride Hexahydrate, solid | MilliporeSigma | M2670 | For ribozyme reactions (sections 6–7) |
| N,N-Dimethylformamide, anhydrous | MilliporeSigma | 227056 | Triphosphorylation solvent (section 2) |
| NAP-25 Desalting column (Sephadex G-25 resin) | ThermoFisher Scientific | 45000150 | Disposable gravity-flow size exclusion chromatography columns containing Sephadex G-25 resin (section 5) |
| Non-coring stainless steel needle, 20 G | ThermoFisher Scientific | 14-815-410 | Needles for piercing rubber septa (sections 2–4) |
| Oligonucleotide extinction coefficient calculator | Integrated DNA Technologies | OligoAnalyzer Tool | Nearest-Neighbor Model Short Oligonucleotide 260nm extinction coefficient calculator (section 5) |
| Oxidizer solution, 0.1 M Iodine in THF/pyridine/water | ChemGenes | RN-1456 | Triphosphorylation reagent (section 4) |
| PAGE plates | Timberrock/CBS | NGP-250-BO and NO | For PAGE (sections 5–7) |
| PAGE power supply | Bio-Rad Laboratories | PowerPac HV 1645056 | For PAGE (sections 5–7) |
| PAGE spacers and combs (analytical) | Timberrock/CBS | VGS-0725 and VGC-0714 | For PAGE (sections 6–7) |
| PAGE spacers and combs (preparative) | Timberrock/CBS | VGS-3025R and VGC-3001 | For PAGE (section 5) |
| PAGE stand | Timberrock/CBS | ASG-250 | For PAGE (sections 5–7) |
| Parafilm M | ThermoFisher Scientific | 13-374-12 (Bemis PM999) | Wax sealing film for triphosphorylation apparatus (sections 2–4) |
| PCR thermocycler | Bio-Rad Laboratories | C1000 Touch Thermalcycler | For cross-chiral ribozyme reactions (section 7) |
| PD 10 Desalting column (Sephadex G-10 resin) | MilliporeSigma | GE17-0010-01 | Disposable gravity-flow size exclusion chromatography columns containing Sephadex G-10 resin, for oligonucleotides < 15 nt (section 5) |
| Phosphor screens | Cytiva | 28956480 | For visualizing 32P-labeled RNA (section 6) |
| Phosphoramidite synthesis reagents | Glen Research | 30-3142-52, 40-4050-53, 40-4012-52, 40-4122-52, 40-4132-52, 40-4060-62 | representative, for standard RNA/DNA synthesis (section 1) |
| Polypropylene screw-cap sealable tube | MilliporeSigma | BR780752 | 1.5 mL microcentrifuge tubes with screw-cap and silicone O-ring, for safe AMA deprotection (section 5) |
| Pyridine, anhydrous | MilliporeSigma | 270970 | Triphosphorylation solvent (section 2) |
| Reverse-phase liquid chromatography/electrospray ionization mass spectrometry (RP-LC/ESI-MS) | Novatia | n/a | Commercial service for LC/MS specializing in oligonucleotides (section 5) |
| Rubber Septa (ID x OD 7.9 mm x 14 mm), white | MilliporeSigma | Z564702 | Septa for preparing triphosphorylation solvents and TBAP (section 2) |
| Self-replicator ribozyme E | prepared in house14 | 5′-GGAAGUUGUG CUCGAUUGUU ACGUAAGUAA CAGUUUGAAU GGUUGAAGUA UGAGACCGCA ACUUA-3′ | For self-replicator ribozyme reactions (section 6) |
| Self-replicator substrate A | prepared in house14 | 5′-32P-GGAAGUUGUG CUCGAUUGUU ACGUAAGUAA CAGUUUGAAU GGUUGAAGUA U-3′-OH | For self-replicator ribozyme reactions (section 6) |
| Self-replicator substrate B, transcribed | prepared in house14 | 5′-pppGAGACCGCAA CUUA-3′ | For self-replicator ribozyme reactions (section 6) |
| Small Drying Traps, 4 Å molecular sieves | ChemGenes | DMT-1975 | Drying traps for DNA/RNA synthesizer phosphoramidites and triphosphorylation reagents (sections 1–2) |
| Sodium Chloride (NaCl), solid | MilliporeSigma | S7653 | Salt for use in various buffers (sections 5–7) |
| Sodium Hydroxide (NaOH), solid | MilliporeSigma | S8045 | Salt for use in various buffers (sections 5–7) |
| Statistical data-fitting software | GraphPad | Prism | For fitting data from analytical PAGE to kinetic models (section 6) |
| Streptavidin-coated magnetic beads | ThermoFisher Scientific | 65002 | For capturing biotin-labeled RNA in cross-chiral ribozyme reactions (section 7) |
| Sucrose | MilliporeSigma | 84097 | For PAGE urea loading buffer (section 5) |
| TBE running buffer, 10x | ThermoFisher Scientific | AAJ62788K3 | For PAGE (sections 5–7) |
| Tetrabutylammonium Fluoride, 1.0 M solution in Tetrahydrofuran | Aldrich | 216143 | For removing 2′-silyl protecting groups (section 5) |
| Tetramethylethylenediamine (TEMED) | Bio-Rad Laboratories | 1610801 | For polymerizing acrylamide for PAGE (sections 5–7) |
| Tributylamine | MilliporeSigma | 90781 | Triphosphorylation reagent (section 2) |
| Tributylammonium pyrophosphate (TBAP) | MilliporeSigma | P8533 | Triphosphorylation reagent (section 2) |
| Tris base | MilliporeSigma | T6666 | Buffering agent for use in various buffers (sections 5–7) |
| TWEEN20 polysorbate detergent | MilliporeSigma | P7949 | Neutral detergent for use with magnetic beads (Section 7) |
| Urea | MilliporeSigma | U5378 | For PAGE and gel loading buffer (sections 5–7) |
| UV-Vis spectrophotometer | ThermoFisher Scientific | NanoDrop 2000, ND2000 | For measuring oligonucleotide concentrations (section 5) |
| Vacuum centrifuge | ThermoFisher Scientific | Savant Speedvac DNA130-115 Vacuum Concentrator | For removing AMA and THF (section 5) |
| Xylene cyanol | Bio-Rad Laboratories | 1610423 | For PAGE urea loading buffer (section 5) |
Riferimenti
- Shuman, S. What messenger RNA capping tells us about eukaryotic evolution. Nature Reviews. Molecular Cell Biology. 3 (8), 619-625 (2002).
- Pichlmair, A., et al. RIG-I-mediated antiviral responses single-stranded RNA bearing 5'-phosphates. Science. 314 (5801), 997-1001 (2006).
- Hornung, V., et al. 5'-Triphosphate RNA is the ligand for RIG-I. Science. 314 (5801), 994-997 (2006).
- Myong, S., et al. Cytosolic viral sensor RIG-I is a 5'-triphosphate-dependent translocase on double-stranded RNA. Science. 323 (5917), 1070-1074 (2009).
- Takeuchi, O., Akira, S. Pattern recognition receptors and inflammation. Cell. 140, 805-820 (2010).
- Wang, Y., et al. Structural and functional insights into 5'-ppp RNA pattern recognition by the innate immune receptor RIG-I. Nature Structural & Molecular Biology. 17 (7), 781-787 (2010).
- Goubau, D., et al. Antiviral immunity via RIG-I-mediated recognition of RNA bearing 5'-diphosphates. Nature. 514 (7522), 372-375 (2014).
- Joyce, G. F. Forty years of in vitro evolution. Angewandte Chemie. 46 (34), 6420-6436 (2007).
- Robertson, M. P., Ellington, A. D. In vitro selection of an allosteric ribozyme that transduces analytes to amplicons. Nature Biotechnology. 17 (1), 62-66 (1999).
- Robertson, M. P., Hesselberth, J. R., Ellington, A. D. Optimization and optimality of a short ribozyme ligase that joins non-Watson-Crick base pairings. RNA. 7 (4), 513-523 (2001).
- Hesselberth, J. R., Robertson, M. P., Knudsen, S. M., Ellington, A. D. Simultaneous detection of diverse analytes with an aptazyme ligase array. Analytical Biochemistry. 312 (2), 106-112 (2003).
- Lam, B. J., Joyce, G. F. Autocatalytic aptazymes enable ligand-dependent exponential amplification of RNA. Nature Biotechnology. 27 (3), 288-292 (2009).
- Lam, B. J., Joyce, G. F. An isothermal system that couples ligand-dependent catalysis to ligand-independent exponential amplification. Journal of the American Chemical Society. 133 (9), 3191-3197 (2011).
- Olea, C., Horning, D. P., Joyce, G. F. Ligand-dependent exponential amplification of a self-replicating L-RNA enzyme. Journal of the American Chemical Society. 134 (19), 8050-8053 (2012).
- Olea, C., Joyce, G. F. Real-Time Detection of a Self-Replicating RNA Enzyme. Molecules. 21 (10), (2016).
- Sczepanski, J. T., Joyce, G. F. A cross-chiral RNA polymerase ribozyme. Nature. 515 (7527), 440-442 (2014).
- Tjhung, K. F., Sczepanski, J. T., Murtfeldt, E. R., Joyce, G. F. RNA-catalyzed cross-chiral polymerization of RNA. Journal of the American Chemical Society. 142 (36), 15331-15339 (2020).
- Bare, G. A. L., Joyce, G. F. Cross-chiral, RNA-catalyzed exponential amplification of RNA. Journal of the American Chemical Society. 143 (45), 19160-19166 (2021).
- Milligan, J. F., Groebe, D. R., Witherell, G. W., Uhlenbeck, O. C. Oligoribonucleotide synthesis using T7 RNA polymerase and synthetic DNA templates. Nucleic Acids Research. 15 (21), 8783-8798 (1987).
- Chelliserrykattil, J., Ellington, A. D. Evolution of a T7 RNA polymerase variant that transcribes 2'-O-methyl RNA. Nature Biotechnology. 22 (9), 1155-1160 (2004).
- Ibach, J., et al. Identification of a T7 RNA polymerase variant that permits the enzymatic synthesis of fully 2′-O-methyl-modified RNA. Journal of Biotechnology. 167 (3), 287-295 (2013).
- Esvelt, K. M., Carlson, J. C., Liu, D. R. A system for the continuous directed evolution of biomolecules. Nature. 472 (7344), 499-503 (2011).
- Pleiss, J. A., Derrick, M. L., Uhlenbeck, O. C. T7 RNA polymerase produces 5' end heterogeneity during in vitro transcription from certain templates. RNA. 4 (10), 1313-1317 (1998).
- Schenborn, E. T., Mierendorf, R. C. A novel transcription property of SP6 and T7 RNA polymerases: dependence on template structure. Nucleic Acids Research. 13 (17), 6223-6236 (1985).
- Martin, C. T., Muller, D. K., Coleman, J. E. Processivity in early stages of transcription by T7 RNA polymerase. Biochemistry. 27 (11), 3966-3974 (1988).
- Gholamalipour, Y., Karunanayake Mudiyanselage, A., Martin, C. T. 3' end additions by T7 RNA polymerase are RNA self-templated, distributive and diverse in character-RNA-Seq analyses. Nucleic Acids Research. 46 (18), 9253-9263 (2018).
- Vasilyev, N., Serganov, A. Preparation of short 5′-triphosphorylated oligoribonucleotides for crystallographic and biochemical studies. Nucleic Acid Crystallography: Methods and Protocols. , 11-20 (2016).
- Lebedev, A. V., Koukhareva, I. I., Beck, T., Vaghefi, M. M. Preparation of oligodeoxynucleotide 5'-triphosphates using solid support approach. Nucleosides, Nucleotides & Nucleic Acids. 20 (4-7), 1403-1409 (2001).
- Paul, N., Springsteen, G., Joyce, G. F. Conversion of a ribozyme to a deoxyribozyme through in vitro evolution. Chemistry & Biology. 13 (3), 329-338 (2006).
- Zlatev, I., et al. Efficient solid-phase chemical synthesis of 5'-triphosphates of DNA, RNA, and their analogues. Organic Letters. 12 (10), 2190-2193 (2010).
- Zlatev, I., Manoharan, M., Vasseur, J. -. J., Morvan, F. Solid-phase chemical synthesis of 5'-triphosphate DNA, RNA, and chemically modified oligonucleotides. Current Protocols in Nucleic Acid Chemistry. , (2012).
- Zlatev, I., et al. Automated parallel synthesis of 5'-triphosphate oligonucleotides and preparation of chemically modified 5'-triphosphate small interfering RNA. Bioorganic & Medicinal Chemistry. 21 (3), 722-732 (2013).
- Goldeck, M., Tuschl, T., Hartmann, G., Ludwig, J. Efficient solid-phase synthesis of pppRNA by using product-specific labeling. Angewandte Chemie. 53 (18), 4694-4698 (2014).
- Sarac, I., Meier, C. Efficient automated solid-phase synthesis of DNA and RNA 5′-triphosphates. Chemistry-A European Journal. 21 (46), 16421-16426 (2015).
- Sarac, I., Meier, C. Solid-phase synthesis of DNA and RNA 5'-O-triphosphates using cycloSal chemistry. Current Protocols in Nucleic Acid Chemistry. 64 (1), 4-67 (2016).
- Perez, J. T., et al. Influenza A virus-generated small RNAs regulate the switch from transcription to replication. Proceedings of the National Academy of Sciences of the United States of America. 107 (25), 11525-11530 (2010).
- Ludwig, J., Eckstein, F. Rapid and efficient synthesis of nucleoside 5'-0-(1-thiotriphosphates), 5'-triphosphates and 2',3'-cyclophosphorothioates using 2-chloro-4H-1,3,2-benzodioxaphosphorin-4-one. The Journal of Organic Chemistry. 54 (3), 631-635 (1989).
- Gaur, R. K., Sproat, B. S., Krupp, G. Novel solid phase synthesis of 2'-o-methylribonucleoside 5'-triphosphates and their α-thio analogues. Tetrahedron Letters. 33 (23), 3301-3304 (1992).
- Reddy, M. P., Hanna, N. B., Farooqui, F. Fast cleavage and deprotection of oligonucleotides. Tetrahedron Letters. 35 (25), 4311-4314 (1994).
- Hogrefe, R. I., McCaffrey, A. P., Borozdina, L. U., McCampbell, E. S., Vaghefi, M. M. Effect of excess water on the desilylation of oligoribonucleotides using tetrabutylammonium fluoride. Nucleic Acids Research. 21 (20), 4739-4741 (1993).
- Pitsch, S., Weiss, P. A., Jenny, L., Stutz, A., Wu, X. Reliable chemical synthesis of oligoribonucleotides (RNA) with 2′-O-[(Triisopropylsilyl)oxy]methyl(2′-O-tom)-protected phosphoramidites. Helvetica Chimica Acta. 84 (12), 3773-3795 (2001).
- Green, M. R., Sambrook, J. Isolation of DNA fragments from polyacrylamide gels by the crush and soak method. Cold Spring Harbor Protocols. 2019 (2), (2019).
- Paul, N., Joyce, G. F. A self-replicating ligase ribozyme. Proceedings of the National Academy of Sciences of the United States of America. 99 (20), 12733-12740 (2002).
- Lincoln, T. A., Joyce, G. F. Self-sustained replication of an RNA enzyme. Science. 323 (5918), 1229-1232 (2009).
- Robertson, M. P., Joyce, G. F. Highly efficient self-replicating RNA enzymes. Chemistry & Biology. 21 (2), 238-245 (2014).
- Singh, J., et al. Synthesis of modified nucleoside oligophosphates simplified: fast, pure, and protecting group free. Journal of the American Chemical Society. 141 (38), 15013-15017 (2019).
- Bellon, L. Oligoribonucleotides with 2'-O-(tert-butyldimethylsilyl) groups. Current Protocols in Nucleic Acid Chemistry. , (2001).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneEsplora altri articoli
This article has been published
Video Coming Soon