このコンテンツを視聴するには、JoVE 購読が必要です。 サインイン又は無料トライアルを申し込む。
Method Article
遺伝子コードされたカルシウムインジケーターを用いたウイルス感染ヒト腸オルガノイド単分子膜のライブカルシウムイメージング
要約
このプロトコルでは、ウイルスに感染したヒト腸オルガノイドでカルシウムイメージングを行うためのアプローチを説明し、分析へのアプローチを提供します。
要約
カルシウムシグナル伝達は、ほぼすべての組織に不可欠な調節因子です。腸管上皮内では、カルシウムは分泌活動、アクチン動態、炎症反応、幹細胞増殖、その他多くの特徴不明の細胞機能の調節に関与しています。そのため、腸上皮内のカルシウムシグナル伝達動態をマッピングすることで、恒常性細胞プロセスに関する洞察が得られ、さまざまな刺激に対する独自の応答が明らかになります。ヒト腸管オルガノイド(HIO)は、腸上皮を研究するためのハイスループットなヒト由来モデルであり、カルシウム動態を調べるための有用なシステムです。この論文では、遺伝子コードカルシウムインジケーター(GECI)を用いてHIOを安定的に形質導入し、ライブ蛍光顕微鏡法を実施し、イメージングデータを解析してカルシウムシグナルを有意義に特徴付けるためのプロトコルについて説明します。代表例として、3次元HIOをレンチウイルスで形質導入し、緑色蛍光タンパク質をベースとした細胞質GECIであるGCaMP6sを安定的に発現させました。次に、操作されたHIOを単一細胞懸濁液に分散させ、単層として播種しました。分化後、HIO単層をロタウイルスに感染させ、および/またはカルシウム応答を刺激することが知られている薬剤で処理しました。温度制御され加湿されたライブイメージングチャンバーを備えた落射蛍光顕微鏡は、感染した単分子膜や薬物処理された単分子膜の長期イメージングを可能にしました。イメージングに続いて、取得した画像は、無料で入手できる解析ソフトウェアImageJを使用して解析されました。全体として、この研究は、HIOにおける細胞シグナル伝達を特徴付けるための適応可能なパイプラインを確立するものである。
概要
カルシウムは広く保存されているセカンドメッセンジャーであり、細胞生理機能の調節に重要な役割を果たしています1。カルシウムは、その強い電荷、小型、および生理学的条件での高い溶解性を考えると、タンパク質のコンフォメーションの理想的なマニピュレーターです。これにより、カルシウムは電気化学的シグナルを酵素的、転写的、または転写後の変化に変換する強力な手段になります。小胞体(ER)と原形質膜を横切る厳密なカルシウム濃度勾配は、細胞質カルシウム濃度の急速な変化を可能にする高い駆動力を生み出します。バッファリングとアクティブトランスポートの両方を含む複数のメカニズムが、この勾配を厳密に維持します。正常な細胞機能には必要であるが、この維持はエネルギー的に高価であり 、ストレス状態において特に影響を受けやすい2。
このように、細胞質内のカルシウムの調節不全は、多くの種類の細胞ストレスのほぼ普遍的なシグナルです。代謝障害、毒素、病原体、機械的損傷、遺伝的摂動はすべて、カルシウムシグナル伝達を混乱させる可能性があります。刺激に関係なく、細胞レベルでは、細胞質カルシウムの持続的で制御不能な上昇は、アポトーシスを促進し、最終的には壊死を促進する可能性があります3,4。しかし、低振幅または高頻度の細胞質カルシウムレベルの変化は、さまざまな影響を及ぼします2。同様に、カルシウム変動の結果は、変動が発生する空間的ミクロドメインによって異なる可能性があります5。したがって、カルシウムレベルをモニタリングすることで、動的シグナル伝達プロセスに関する洞察を得ることができますが、そのためには比較的高い時間分解能と空間分解能でのサンプリングが必要です。
遺伝子コードされたカルシウム指標(GECI)は、生細胞システムでの連続サンプリングのための強力なツールです6。最も広く使用されているGECIには、GCaMPとして知られるGFPベースのカルシウム応答性蛍光タンパク質があります7。標準的なGCaMPは、環状に順列されたGFP(cpGFP)、カルモジュリン、およびM136の3つの異なるタンパク質ドメインの融合です。カルモジュリンドメインは、カルシウムの結合時にコンフォメーション変化を起こし、M13との相互作用を可能にします。カルモジュリン-M13相互作用は、cpGFPの立体構造変化を誘発し、励起時の蛍光発光を増加させます。そのため、カルシウム濃度の上昇はGCaMP蛍光強度の増加と相関します。これらのセンサーは、細胞質である場合もあれば、特定の細胞小器官を標的とする場合もある8。
ほとんどの組織と同様に、カルシウムは消化管上皮内のさまざまな機能を調節します。腸上皮は栄養素と体液の吸収に不可欠ですが、病原体の侵入や有毒な侮辱を避けるために、タイトなバリアと免疫インターフェースを形成する必要があります。カルシウム依存性経路は、これらの生命機能のほぼすべてに影響を与えます9,10,11。しかし、腸上皮内のカルシウムシグナル伝達は未開拓のフロンティアであり、治療標的として有望な可能性を秘めています。in vivoでの腸上皮内のカルシウム動態のモニタリングは引き続き課題となっていますが、ヒト腸オルガノイド(HIO)は実験に適応可能なex vivoシステムを提供します12。HIOは、ヒト腸管幹細胞に由来する3次元(3D)スフェロイドであり、分化すると、天然の腸上皮の細胞多様性の多くを再現する12。
このプロトコールでは、GECIを発現するHIOを設計し、生細胞カルシウムイメージング用の単層として設計されたHIOを調製するための包括的な方法について説明します。カルシウムシグナル伝達を破壊する病理学的操作の例としてウイルス感染を提供し、これらの変化を定量化するための分析的アプローチを提供します。
プロトコル
このプロトコルおよび代表的な実験で使用されるすべてのヒト腸オルガノイド(HIO)は、Texas Medical Center Digestive Diseases Enteroid Coreによって取得および維持されたヒト組織に由来しました。すべてのサンプルは、ベイラー医科大学の治験審査委員会によって承認されたプロトコルに従って収集されました。
1. 材料・試薬の調製
- オルガノイドの維持には、細胞培養処理した24ウェルプレート、基底膜マトリックス(BMM)、15 mLコニカルチューブ、および1.5 mLコニカルチューブを用意します。
- 成長因子(CMGF-)を含まない完全な培地を調製するには、500 mLの高度なDMEM F12に、5 mLの1M HEPES、5 mLの100x抗生物質-抗真菌薬、5 mLの100xグルタミンサプリメントを加えます。
- Wnt、R-spondin、およびNoggin含有(WRNE)培地を調製するには、CMGFおよびWnt馴化培地を等量混合し、Noggin馴化培地(10容量%)、R-spondin馴化培地(20容量%)、50 ng/mLヒト上皮成長因子、10 mMニコチンアミド、10 nM [Leu15]-ガストリンI、500 nM A-83-01、10 μM SB202190、1x B27サプリメント、1x N2サプリメント、および1 mM N-アセチルシステインを添加します。
- レンチウイルス形質導入については、ウシ胎児血清(FBS)、0.05%トリプシン-EDTAを1xリン酸緩衝生理食塩水(PBS)、CMGF-10%FBSを含む、滅菌済み1x PBS、ポリブレン、10 μM Y-27632、レンチウイルス、高Wnt WRNE + 10 μM Y-27632を調製します。
- オルガノイド単分子膜を作製するには、ガラス底10ウェル細胞培養スライド、FBS、コラーゲンIV(脱イオン化(ジ)H2O中1 mg/mL)、基底膜マトリックス、0.5 mM EDTAを1x PBS、5 mM EDTAを1x PBS、酵素解離バッファー、CMGF-、10% FBS、WRNE + 10 μM Y-27632で調製します。
- オルガノイド単分子膜のウイルス感染には、トリプシン、ロタウイルスストック、CMGF-、25Gニードル、滅菌済み1x PBS、およびフェノールレッドフリー分化培地を調製します。
- フェノールレッドフリー分化培地を調製するには、500 mLのフェノールレッドフリー細胞培養培地を採取し、5 mLの100x MEM非必須アミノ酸、5 mLの100x L-グルタミン、5 mLの100 nMピルビン酸ナトリウム、および7.5 mLの1M HEPESを加えます。
- オルガノイドの免疫蛍光染色には、4%ホルムアルデヒド(1x PBSで希釈した16%ホルムアルデヒド)、Triton X-100(0.1% Triton X-100 in 1x PBS)、ウシ血清アルブミン(3%ウシ血清アルブミン1x PBS中)、NH4Cl溶液(50 mM)、DAPI(1x PBS中1 μg/mL DAPI溶液)を調製します。
2. 遺伝子にコードされたカルシウムセンサーを発現するオルガノイドのエンジニアリング
注:このプロトコルは、24ウェルプレート13に30μLの基底膜マトリックス(BMM)にめっきされた3次元ヒト腸オルガノイドのシングルウェルをトランスデューシングする手順を説明しています。ほとんどのラインには、ウェルあたり約 400,000 個のセルが含まれます。2番目の形質導入されていないウェルをコントロールとして含める必要があります。すべての試薬と細胞懸濁液を氷上に保管してください。
- 最後の継代から2〜5日後、HIOの2つのウェルからメンテナンスWRNE培地を除去します。ウェルあたり 300 μL の 0.05% トリプシン-EDTA と交換し、5 回ピペットで静かに上下させて BMM をプレートから取り外します。37°Cのインキュベーターに4分間入れます。
- ウェルあたり500 μLのCMGF-+10%FBSを添加します。1 mLのFBSを上下2回ピペッティングすることにより、1 mLのローバウンドピペットチップをFBSでプレコートします。コーティング済みのチップを使用して、HIOを10回上下にピペットで移動します。
- 各ウェルの内容物を、事前にコーティングされた1.5 mL微量遠心チューブに移します。さらに500 μLのCMGF-で各ウェルをすすぎ、洗浄液をそれぞれのチューブに加えます。
- チューブを100 x g でスイングバケット遠心分離機で4°Cで5分間遠心分離します。 上清と残留BMMを取り除きます。
- 1 mLの1x PBSに再懸濁します。各チューブを2つの微量遠心チューブに分割し、合計4本のチューブにします。チューブを100 x g で4°Cで5分間遠心分離します。 上澄みを取り除きます。
- 各チューブを1x PBSにさらに1回再懸濁し、100 x g で4°Cで5分間遠心分離します。
- 形質導入培地とコントロール培地を400μL調製します(表1)。2本のチューブを200 μLのコントロール培地に、2本のチューブを200 μLの形質導入培地に再懸濁します。
- 37°Cの細胞培養インキュベーターで24時間インキュベートします。均一な形質導入を促進するために、コーティングされたチップで定期的にピペッティングして再懸濁します(例:形質導入後2時間(hpt)、12 hpt、18 hpt)。
- 24時間後、チューブを100 x g で4°Cで5分間遠心分離します。 上澄みを取り除きます。
- ペレットを500 μLの1x PBSに再懸濁して洗浄します。4°Cで100 x g で5分間遠心分離します。 上澄みを取り除きます。
- 氷冷した200 μLのピペットチップを使用して、4つのペレットをそれぞれ30 μLのBMMに再懸濁します。ピペットで上下に静かに動かし、均一に分散させます。
- 各チューブの内容物を24ウェルプレート上の独自のウェルにプレートします。37°Cで10分間インキュベートしてBMMを固化させてから、500 μLのHighWnt WRNE + 10 μM Y-27632を添加します。
- 形質導入したHIOを1週間増殖させ、培地(HighWnt WRNE + 10 μM Y-27632)を1日おきにリフレッシュします。1週間後、顕微鏡で蛍光指示薬の発現を確認します。シグナルが強い場合は、薬剤の選択を開始します。シグナルが弱い場合は、上記のように形質導入を繰り返します。
- ラインが確立されたら、アゴニスト処理により蛍光指示薬の機能を確認します。100nM ADPは、GCaMPバリデーションのための信頼性の高いアゴニストです。初期バリデーションのために3Dオルガノイドを試験するには、以下で説明します。
- 継代後、オルガノイドのウェル(または複数)をBMMで別のイメージングボトムプレートにプレーティングします。オルガノイドを通常の密度の約1/3でプレーティングし、イメージング時に過剰な重なりを避けます。無菌性を確保することは難しいため、アゴニスト治療後もこれらのHIOの継代を続けないでください。
- HIOが通過後に回復するまで2〜3日待ちます。イメージングする前に、培地をフェノール赤色フリー分化培地に切り替えてください。
- 蛍光顕微鏡を使用して、488 nm励起とFITC/GFPフィルターセットを使用して5秒ごとに取得した画像で3分間の実行をセットアップします。30秒のイメージング後、100 nM ADPまたは車両制御を追加します。信号がベースライン近くに戻るまで、~2分間イメージングを続けます。ADP処理によるGCaMP蛍光の一過性~2倍の増加は、形質導入とバイオセンサー機能が成功したことを示しています。形質導入効率をより正確に推定するには、パート3で説明したプロセスで生成した単分子膜を使用してイメージングでアゴニスト試験を繰り返します。
3. ライブ蛍光イメージングのためのHIO単分子膜の作製
- 10ウェルイメージングボトムチャンバースライドのすべてのウェルをコラーゲンIVでコーティングします。これを行うには、34 μL の 1 mg/mL コラーゲン IV を 960 μL の滅菌脱イオン水と混合します。希釈したコラーゲンIV溶液95 μLを各ウェルに加え、37°Cで0.5〜2時間インキュベートします。
- 3D HIOの4つのウェルからWRNE維持培地を除去します。HIOは、最後の通過から5〜7日後である必要があります。
注:1 10ウェルプレートには、通常、~1.25 x 106 セルが必要です。これには通常、それぞれ30 μLのBMMで播種された3D HIOのウェルが2〜4個必要ですが、密度によって異なります。 - ウェルあたり 500 μL の 1x PBS + 0.5 mM EDTA を添加します。コーティング済みの1 mLチップで、ピペットを静かに上下に動かし、BMMをプレートから取り外します。懸濁液をプレコートされた15 mLコニカルチューブに移し、同様のウェルを同じチューブに組み合わせます。
- さらに500 μLのPBS + 0.5 mM EDTAで各ウェルをすすぎます。4°Cで5分間、300 x g で遠心分離します。 上澄み液と残留BMMを取り除きます。
- プレコートチップを使用して、残りのペレットを 3 mL の PBS + 5 mM EDTA に再懸濁します(これは最初の洗浄の 10 倍の EDTA であることに注意してください)。
- 4°Cで5分間、300 x g で遠心分離します。 上澄み液と残留BMMを取り除きます。ペレットを2 mLの酵素解離緩衝液に再懸濁します。
- 37°Cのビーズ/ウォーターバスで5分間インキュベートします。3 mLのCMGF-+10%FBSを加え、ピペットで静かに混合します。
- 4°Cで5分間、300 x g で遠心分離します。 上澄みを取り除きます。1 mL の CMGF- を追加します。
- プレコートチップ80x-100xで活発に上下にピペット操作し、HIOを機械的に単一細胞に分解します。4°Cで5分間、300 x g で遠心分離します。 上澄みを取り除きます。
- 1 mL の WRNE + 10 μM Y-27632 に再懸濁します。1 mL 懸濁液の細胞数を取得します。
- 細胞懸濁液をWRNE + 10 μM Y-27632で希釈し、1.25 x 105 cells/100 μL(1.25 x 106 cells/mL)の濃度にします。
- 200 μLのピペットを使用して、コラーゲンがウェルの底に沈殿したので、ステップ1で調製したプレートからコラーゲン溶液を取り除きます。ピペットチップでウェルの底に触れないようにしてください。
- プレコートされた200 μLのピペットチップを使用して、ウェルあたりステップ3.11の細胞溶液100 μL(1.25 x 105 セル)を添加します。
- 37°Cの細胞培養インキュベーターで24時間インキュベートします。24時間後、すべてのウェルから培地を取り出し、ウェルあたり100 μLの分化培地と交換します。
注:この時点で、セルはプレートに付着しているはずです。単層は、コンフルエントまたは完全に平面ではない可能性が高いことに注意してください。 - スライドを37°Cの細胞培養インキュベーターに戻します。単分子膜がコンフルエントになるまで、24時間ごとに分化培地をリフレッシュします。これには通常、メッキから3〜5日かかります。この時点を過ぎると、単層は下流のアプリケーションの準備が整います。
4. HIO単分子膜のウイルス感染
- ウイルス接種を準備します。必要に応じて、トリプシンはウイルスストックを活性化します。ロタウイルスの場合は、10 μg/mL のワージントントリプシンをウイルスストックに加え、37 °C で 1 時間インキュベートします。
- 活性化したウイルスストックを、以下の2つの方法のいずれかで希釈します。
- 1つのウイルス株のみを使用して最大数の感染細胞を目指す場合は、活性化ウイルスストック50 μLと50 μLのCMGF-を混合します。
- 複数の株を比較する場合は、同等の感染多重度(MOI)を達成するように接種物を準備します。次の式を使用して、目的のMOIに必要なウェルあたりのウイルスストックの量を決定します。計算したウイルスストックの量に CMGF- を添加し、ウェルあたりの最終容量が 100 μL になるようにします。
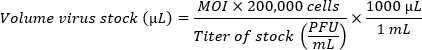
注:直径 5.46 mm のウェル(96 ウェルプレート)にめっきされた 1 つの HIO 単層には、約 200,000 個の細胞が含まれています。単層での感染効率が低いため、高MOI(細胞あたり1億〜100万個のウイルス粒子)が好まれます。最適なMOIは経験的に決定する必要があります。
- 200 mLピペットを使用してHIO単分子膜から培地を静かに除去し、単分子膜を感染させます。100mLのウイルス接種または模擬接種物と交換してください。
- ほとんどのロタウイルス株はMA104細胞で増殖するため、模擬接種物には感染していないMA104細胞ライセートを使用します。感染したウェルに使用したウイルスストックの量と同等の容量を使用し、ウェルあたりの最終容量が 100 mL になるように CMGF- を添加します。
- ロタウイルス14など、細胞に基底側から感染するウイルスの場合は、25Gの針を使用して単層にスコアを付けます。井戸の底から上まで、単層の長さにわたって単一のスコアを作成するのが最も簡単です。針を斜角を上にして、動きの方向と反対の角度で単分子膜にそっと押し込み、単層の長さを横切ってドラッグして瘢痕を作成します(図2A)。
- 37°Cのインキュベーターで2時間インキュベートします。ウイルスを除去し、模擬接種を行います。単層を1x PBSで1回洗浄します。
- 100 μLのFenol Red-free differentiation Mediaを添加します。イメージングの準備ができるまで、スライドを37°Cのインキュベーターに入れます。最適なイメージングウィンドウは、ウイルス感染の動態によって異なります。ロタウイルスの場合、感染後6〜8時間でイメージングを開始します。
- 活性化したウイルスストックを、以下の2つの方法のいずれかで希釈します。
5. 感染した単分子膜のCa2+ イメージング
- ステージトップインキュベーターを37°Cに予熱します。 加湿したCO2 を0.02L/minで流動します。感染した単層を含むスライドをインキュベーターチャンバーに入れ、蓋を密封します。
- 明視野(BF)照明と20倍対物レンズを使用して、画像化する単層内の視野のX座標とY座標を選択します。ほとんどの感染細胞は傷の近くにあるため、スコアリングされた領域を視野に入れてポイントを選択するのが最善です。
- イメージングパラメータを最適化し、488 nm励起を使用して画像を取得します。光毒性を最小限に抑えるには、光源を50%の出力に設定し、露光時間を50msにします。適切なアクイジションパラメータは異なる場合があり、複数のアクイジションによって最適化する必要があります。ピクセルが飽和していないことを確認します。必要に応じて露光時間と光量を調整し、蛍光カルシウムセンサーのダイナミックレンジ全体を検出できるようにします。
- 他の蛍光色素(蛍光タグ付きウイルスなど)を使用する場合は、これらのチャネルで最適化を繰り返します。イメージングループを設定し、選択したすべてのX、Y座標で488 nmの画像を1分間のループで取得します。このループを連続的にイメージ化します。蛍光タグ付きウイルスを使用する場合は、10ループ ごとに適切なチャンネルで画像を取得します(つまり、1画像/10分)。
- ~18時間画像を収集します。蛍光タグのないウイルスを使用する場合は、単層を固定し、免疫蛍光染色を行って感染細胞を同定します。イメージングの実行が完了したら、以下で説明するようにこれを実行します。
- イメージング培地を取り出し、1x PBSに100 μLの4%ホルムアルデヒドと交換します。室温で30分間インキュベートします。
- 固定液を除去し、100 μL の 50 mM NH4Cl で 10 分間クエンチします。 NH4Cl を除去します。 単分子膜を 100 μL の 0.1% Triton X-100 で 1x PBS で室温で 1 時間、または 4 °C で一晩透過処理します。
- 透過処理バッファーを除去します。100 μL の 3% ウシ血清アルブミンを 1x PBS で 1 時間ブロックします。
- ブロッキング溶液を取り除きます。推奨/最適化濃度に希釈した一次抗体を 1x PBS に添加します。ウェルあたり100 μLの抗体溶液を添加します。
- 穏やかに揺らしながら4°Cで一晩インキュベートします。一次抗体を除去します。1x PBSで3回洗浄します。
- 推奨濃度/最適化濃度に希釈した蛍光標識二次抗体を1x PBSに添加します。ウェルあたり100 μLを添加します。室温で2時間インキュベートします。
注: FPBase15 は、多重化時のブリードスルーを回避するセカンダリを選択するのに役立つ優れたリソースです。ほとんどの二次剤は、1x PBSで1:1000希釈すると効果的です。 - 二次抗体を除去します。1x PBSで1 μg/mLのDAPI溶液を調製し、ウェルあたり100 μLを添加します。20分間インキュベートし、DAPIを除去します。1x PBSで3回洗浄します。
- 固定単分子膜を100 μLの1x PBSに保持してイメージングします。スライドを顕微鏡ステージに戻し、ライブイメージングランからX座標とY座標を再ロードし、染色に使用した二次抗体に対応するチャンネル上の各マルチポイントを画像化します。ライブイメージング実行の画像を参照して、同じポイントがキャプチャされるようにします。
6. 細胞間カルシウム波の定量
- Fiji is Just ImageJ (FIJI) が次のプラグインでインストールされていることを確認します: Bio-Formats (FIJI にはプリインストールされているはずですが、ImageJ には含まれていません)。クックブック: インストールするには、FIJI メニューから [ヘルプ] > [更新] > [更新サイトの管理] に移動します。 クックブックを確認してください。FIJIを再起動します。
- ライブイメージング実行のデータファイルを複数のファイルに分割し、各X、Y座標を分離します。Nikon NES Elements で、[ファイル] >[インポート/エクスポート]>[マルチポイントを分割]を選択します。エクスポートするフォルダと適切な接頭辞を選択します。
- フィジーを開きます。分割されたマルチポイントから 1 つのファイルを読み込みます。マルチチャンネル画像を使用する場合は、チャンネルを分割します。488 nmチャンネル(GCaMP)の画像を含むウィンドウを選択します。
- DeltaF Up 関数を実行して、ある時点から次の時点への各ピクセル値の変化を判断します。これを行うには、[ Cookbook > T-functions] > [Delta F Up] を選択します。
- カルシウムの波が描かれた画像が見つかるまで、スタックをスクロールします。カルシウム波を表示した状態で、[ 画像] > [> しきい値の調整] をクリックしてしきい値を設定します。下限を調整して、スレッショルドを超える波形の一部ではない信号の量を最小限に抑えます。上記のパラメータを使用して取得した 16 ビット画像の場合は、下限しきい値の 600 から開始し、必要に応じて調整します。
- 粒子アナライザーを実行して波をセグメント化するには、[解析] > [粒子の解析] をクリックします。範囲を調整して、波の最小サイズをμm2で設定します。ベースラインでは 0 から無限大に設定されます。20倍対物レンズを使用して取得したMA104細胞の画像については、下限を10,000μm2に増やすことから始め、必要に応じて調整します。
- [ 結果の表示] と [結果のクリア] のチェックボックスをオンにします。[表示] プルダウン メニューから [ アウトライン] を選択し、[ OK] をクリックします。これにより、2 つの出力が得られます。 1 つのウィンドウ [結果] には、検出された各波、波の面積、およびその平均、最小、および最大の強度値 (デルタ F に基づく) が一覧表示されます。[ファイル名] の描画というタイトルのもう 1 つのウィンドウには、セグメント化された波のアウトラインを含む新しいスタックが含まれます。これを使用して、波形検出を確認します。必要に応じてしきい値とサイズ範囲を調整し、ウェーブ呼び出しを再確認します。
- バッチ処理には、付属のマクロスクリプト(補助コーディングファイル1)を使用します。これを利用するには、以下に説明する手順に従います。
- 手順 6.2 の説明に従って、マルチポイントを 1 つのファイルに分割します。フィジーを開きます。「 処理>バッチ>マクロ」を選択します。[入力] ボックスに、分割されたマルチポイント ファイルを含むフォルダーへのマップを指定します。「出力」ボックスは空のままにします。
- 補足コーディングファイル1のスクリプトをボックスに貼り付けます。スクリプトの強度しきい値とサイズしきい値を調整して、手順 6.5 と 6.6 で最適化されたパラメーターを反映させます。
- [ プロセス] をクリックします。マルチポイントの数やパソコンの処理速度によっては、すべての画像を処理するのに30分以上かかる場合があります。完了すると、データはデスクトップ上の「書き込み後に名前を変更する」という名前の新しいスプレッドシートファイルに書き込まれます。出力ファイルには、マルチポイントごとに、波数と波のメトリック (面積、平均強度、最小強度と最大強度、強度密度) が含まれている必要があります。
結果
図1A は、GCaMP6を安定に発現するように形質導入された3次元ヒト腸管オルガノイドを含むBMMドームを示しています。 図1B は、播種後24時間、48時間、72時間で単層として再播種したオルガノイドの同じラインを示しています。GCaMP6sの機能を検証するために、単分子膜を蛍光顕微鏡で2秒ごとに4分間イメージングし、~20秒後に100 nM ADPを培地に添加しま?...
ディスカッション
細胞質Ca2+レベルの変化は、上皮内の病状の原因と結果の両方である可能性があります10,16,17。細胞質カルシウムの増加は、カルシウム依存性塩化物チャネルの活性化を介して分泌を直接促進TMEM16A18,19。Ca2+に応答したTMEM16Aの活性化は、塩化物の頂端流出を可能に?...
開示事項
著者は、開示すべき競合する金銭的利害関係を有していません。
謝辞
この研究は、米国国立衛生研究所(NIH)の助成金R01DK115507およびR01AI158683(PI:J. M. Hyser)の支援を受けました。研修生のサポートは、NIHの助成金F30DK131828(PI:J.T. Gebert)、F31DK132942(PI:F. J. Scribano)、F32DK130288(PI:K.A. Engevik)によって提供されました。オルガノイド維持培地を提供してくださったTexas Medical Center Digestive Diseases Enteroid Coreに感謝します。
資料
| Name | Company | Catalog Number | Comments |
| Advanced DMEM F12 | Gibco | 12634028 | |
| [Leu15]-Gastrin I | Sigma-Aldrich | G9145 | |
| 0.05% Trypsin EDTA | Gibco | 25300054 | |
| 0.05% Trypsin EDTA | Gibco | 25300054 | |
| 1.5mL microcentrifuge tubes | Fisherbrand | 5408137 | |
| 15mL conical tubes | Thermofisher Scientific | 0553859A | |
| 16% formaldehyde | Thermofisher Scientific | 28906 | |
| 1M HEPES | Gibco | 15630080 | |
| 1M HEPES | Gibco | 15630080 | |
| 1X PBS | Corning | 21-040-CV | |
| 25 gauge needle | Thermofisher Scientific | 1482113D | |
| A-83-01 | Tocris | 2939 | |
| ADP | Sigma-Aldrich | A2754 | |
| Advanced DMEM F12 | Gibco | 12634028 | |
| Antibiotic-antimycocytic | Gibco | 15240062 | |
| Antibiotic-antimycotic | Gibco | 15240062 | |
| B27 Supplement | Gibco | 17504-044 | |
| Bovine serum albumin | FisherScientific | BP1600100 | |
| CellView Cell Culture Slide, PS, 75/25 MM, Glass Bottom, 10 compartments | Greiner | 543979 | |
| Collagen IV | Sigma Aldrich | C5533 | |
| DAPI | Thermofisher Scientific | D1306 | |
| EDTA | Corning | 46-034-CI | |
| Fetal bovine serum | Corning | 35010CV | |
| Fetal bovine serum | Corning | 35010CV | |
| Fluorobrite | Gibco | A1896701 | |
| GlutaMAX | Gibco | 35050079 | |
| GlutaMAX | Gibco | 35050079 | |
| Human epidermal growth factor | ProteinTech | HZ-1326 | |
| Lentivirus | VectorBuilder | (variable) | |
| Matrigel | BD Biosceicen | 356231/CB40230C | |
| N2 Supplement | Gibco | 17502-048 | |
| N-acetylcysteine | Sigma-Aldrich | A9165-5G | |
| NH4Cl | Sigma-Aldrich | A9434 | |
| Nicotinamide | Sigma-Aldrich | N0636 | |
| Nunc Cell Culture Treated 24-well Plates | Thermofisher Scientific | 142475 | |
| Polybrene | MilliporeSigma | TR1003G | |
| SB202190 | Sigma-Aldrich | S70767 | |
| Triton X-100 | Fisher BioReagents | BP151100 | |
| TrypLE Express Enzyme, no phenol red | Thermofisher Scientific | 12604013 | |
| Trypsin | Worthington Biochemical | NC9811754 | |
| Y-27632 | Tocris | 1254 |
参考文献
- Bootman, M. D., Bultynck, G. Fundamentals of cellular calcium signaling: A primer. Cold Spring Harb Perspect Biol. 12 (1), a038802 (2020).
- Clapham, D. E. Calcium signaling. Cell. 131 (6), 1047-1058 (2007).
- Danese, A., et al. Cell death as a result of calcium signaling modulation: A cancer-centric prospective. Biochim Biophys Acta Mol Cell Res. 1868 (8), 119061 (2021).
- Harr, M. W., Distelhorst, C. W. Apoptosis and autophagy: Decoding calcium signals that mediate life or death. Cold Spring Harb Perspect Biol. 2 (10), a005579 (2010).
- Barak, P., Parekh, A. B. Signaling through Ca2+ microdomains from store-operated CRAC channels. Cold Spring Harb Perspect Biol. 12 (7), a035097 (2020).
- Nakai, J., Ohkura, M., Imoto, K. A high signal-to-noise Ca(2+) probe composed of a single green fluorescent protein. Nat Biotechnol. 19 (2), 137-141 (2001).
- Erofeev, A. I., Vinokurov, E. K., Vlasova, O. L., Bezprozvanny, I. B. GCaMP, a family of single-fluorophore genetically encoded calcium indicators. J Evol Biochem Phys. 59 (4), 1195-1214 (2023).
- Suzuki, J., Kanemaru, K., Iino, M. Genetically encoded fluorescent indicators for organellar calcium imaging. Biophys J. 111 (6), 1119-1131 (2016).
- Nászai, M., Cordero, J. B. Intestinal stem cells: Got calcium. Curr Biol. 26 (3), R117-R119 (2016).
- Barrett, K. E. Calcium-mediated chloride secretion in the intestinal epithelium: Significance and regulation. Curr Top Membr. 53, 257-282 (2002).
- Xu, J., et al. Calcium-sensing receptor regulates intestinal dipeptide absorption via Ca2+ signaling and IKCa activation. Physiol Rep. 8 (1), e14337 (2020).
- Clevers, H. Modeling development and disease with organoids. Cell. 165 (7), 1586-1597 (2016).
- Lin, S. C., Haga, K., Zeng, X. L., Estes, M. K. Generation of CRISPR–Cas9-mediated genetic knockout human intestinal tissue–derived enteroid lines by lentivirus transduction and single-cell cloning. Nat Protoc. 17 (4), 1004-127 (2022).
- Crawford, S. E., Ramani, S., Blutt, S. E., Estes, M. K. Organoids to dissect gastrointestinal virus-host interactions: What have we learned. Viruses. 13 (6), 999 (2021).
- Lambert, T. J. FPbase: a community-editable fluorescent protein database. Nat Methods. 16 (4), 277-278 (2019).
- Lai, Y., et al. Inhibition of calcium-triggered secretion by hydrocarbon-stapled peptides. Nature. 603 (7903), 949-956 (2022).
- Chang-Graham, A. L., et al. Rotavirus induces intercellular calcium waves through ADP signaling. Science. 370 (6519), eabc3621 (2020).
- Lee, B., et al. Anoctamin 1/TMEM16A controls intestinal Cl− secretion induced by carbachol and cholera toxin. Exp Mol Med. 51 (8), 1-14 (2019).
- Saha, T., et al. Intestinal TMEM16A control luminal chloride secretion in a NHERF1 dependent manner. Biochem Biophys Rep. 25, 100912 (2021).
- Mroz, M. S., Keely, S. J. Epidermal growth factor chronically upregulates Ca2+-dependent Cl− conductance and TMEM16A expression in intestinal epithelial cells. J Physiol. 590 (8), 1907-1920 (2012).
- Sui, J., et al. Dual role of Ca2+-activated Cl− channel transmembrane member 16A in lipopolysaccharide-induced intestinal epithelial barrier dysfunction in vitro. Cell Death Dis. 11 (5), 404 (2020).
- Bellono, N. W., et al. Enterochromaffin cells are gut chemosensors that couple to sensory neural pathways. Cell. 170 (1), 185-198.e16 (2017).
- Paradis, T., Bègue, H., Basmaciyan, L., Dalle, F., Bon, F. Tight junctions as a key for pathogens invasion in intestinal epithelial cells. Int J Mol Sci. 22 (5), 2506 (2021).
- Samak, G., et al. Calcium/Ask1/MKK7/JNK2/c-Src signalling cascade mediates disruption of intestinal epithelial tight junctions by dextran sulfate sodium. Biochem J. 465 (3), 503-515 (2015).
- Deng, H., Gerencser, A. A., Jasper, H. Signal integration by Ca2+ regulates intestinal stem cell activity. Nature. 528 (7581), 212-217 (2015).
- Saurav, S., Tanwar, J., Ahuja, K., Motiani, R. K. Dysregulation of host cell calcium signaling during viral infections: Emerging paradigm with high clinical relevance. Mol Aspects Med. 81, 101004 (2021).
- Chang-Graham, A. L., et al. Rotavirus calcium dysregulation manifests as dynamic calcium signaling in the cytoplasm and endoplasmic reticulum. Sci Rep. 9 (1), 10822 (2019).
- Hyser, J. M., Collinson-Pautz, M. R., Utama, B., Estes, M. K. Rotavirus disrupts calcium homeostasis by NSP4 viroporin activity. mBio. 1 (5), e00265-e00310 (2010).
- Pham, T., Perry, J. L., Dosey, T. L., Delcour, A. H., Hyser, J. M. The Rotavirus NSP4 viroporin domain is a calcium-conducting ion channel. Sci Rep. 7, 43487 (2017).
- Crawford, S. E., Hyser, J. M., Utama, B., Estes, M. K. Autophagy hijacked through viroporin-activated calcium/calmodulin-dependent kinase kinase-β signaling is required for rotavirus replication. Proc Natl Acad Sci U S A. 109 (50), E3405-E3413 (2012).
- Crawford, S. E., Criglar, J. M., Liu, Z., Broughman, J. R., Estes, M. K. COPII vesicle transport is required for Rotavirus NSP4 interaction with the autophagy protein LC3 II and trafficking to viroplasms. J Virol. 94 (1), e01341 (2019).
- Pando, V., Iša, P., Arias, C. F., Ló Pez, S. Influence of calcium on the early steps of Rotavirus infection. Virology. 295 (1), 190-200 (2002).
- Hyser, J. M., Estes, M. K. Pathophysiological consequences of calcium-conducting viroporins. Annu Rev Virol. 2 (1), 473-496 (2015).
- Strtak, A. C., et al. Recovirus NS1-2 has viroporin activity that induces aberrant cellular calcium signaling to facilitate virus replication. mSphere. 4 (5), e00506-e00519 (2019).
- In, J. G., Foulke-Abel, J., Clarke, E., Kovbasnjuk, O. Human colonoid monolayers to study interactions between pathogens, commensals, and host intestinal epithelium. J Vis Exp. (146), 59357 (2019).
- Hirota, A., AlMusawi, S., Nateri, A. S., Ordóñez-Morán, P., Imajo, M. Biomaterials for intestinal organoid technology and personalized disease modeling. Acta Biomater. 132, 272-287 (2021).
- Cevallos Porta, D., López, S., Arias, C. F., Isa, P. Polarized rotavirus entry and release from differentiated small intestinal cells. Virology. 499, 65-71 (2016).
- Mirabelli, C., et al. Human Norovirus efficiently replicates in differentiated 3D-human intestinal enteroids. J Virol. 96 (22), e0085522 (2022).
- Icha, J., Weber, M., Waters, J. C., Norden, C. Phototoxicity in live fluorescence microscopy, and how to avoid it. Bioessays. 39 (8), 28749075 (2017).
- Li, J., et al. Engineering of NEMO as calcium indicators with large dynamics and high sensitivity. Nat Methods. 20 (6), 918-924 (2023).
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請さらに記事を探す
This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved