JoVE 비디오를 활용하시려면 도서관을 통한 기관 구독이 필요합니다. 전체 비디오를 보시려면 로그인하거나 무료 트라이얼을 시작하세요.
Method Article
유전적으로 인코딩된 칼슘 지표를 사용한 바이러스 감염 인간 장 오가노이드 단층의 라이브 칼슘 이미징
요약
이 프로토콜은 바이러스에 감염된 인간 장 오가노이드에서 칼슘 이미징을 수행하기 위한 접근 방식을 설명하고 분석에 대한 접근 방식을 제공합니다.
초록
칼슘 신호전달은 거의 모든 조직의 필수 조절자입니다. 장 상피 내에서 칼슘은 분비 활동, 액틴 역학, 염증 반응, 줄기 세포 증식 및 기타 많은 특성화되지 않은 세포 기능의 조절에 관여합니다. 따라서 장 상피 내의 칼슘 신호 역학을 매핑하면 항상성 세포 과정에 대한 통찰력을 제공하고 다양한 자극에 대한 고유한 반응을 밝힐 수 있습니다. 인간 장 오가노이드(HIO)는 장 상피를 연구하기 위한 고처리량 인간 유래 모델로, 칼슘 역학을 조사하는 데 유용한 시스템입니다. 이 논문에서는 유전적으로 인코딩된 칼슘 표지자(GECI)를 사용하여 HIO를 안정적으로 transduction하고, 실시간 형광 현미경 검사를 수행하고, 이미징 데이터를 분석하여 칼슘 신호를 의미 있게 특성화하는 프로토콜을 설명합니다. 대표적인 예로, 3차원 HIO를 렌티바이러스로 transduction하여 녹색 형광 단백질 기반 세포질 GECI인 GCaMP6를 안정적으로 발현시켰습니다. 그런 다음 엔지니어링된 HIO를 단일 세포 현탁액으로 분산하고 단층으로 파종했습니다. 분화 후, HIO 단층을 로타바이러스에 감염시키거나 칼슘 반응을 자극하는 것으로 알려진 약물로 처리하였다. 온도 제어, 가습 라이브 이미징 챔버가 장착된 형광 현미경을 사용하면 감염되거나 약물로 처리된 단층의 장기 이미징이 가능합니다. 이미징 후 무료로 제공되는 분석 소프트웨어인 ImageJ를 사용하여 획득한 이미지를 분석했습니다. 전반적으로, 이 연구는 HIO에서 세포 신호 전달을 특성화하기 위한 적응 가능한 파이프라인을 구축합니다.
서문
칼슘은 세포 생리학을 조절하는 데 중요한 역할을 하는 널리 보존된 두 번째 전달자이다1. 생리적 조건에서 강한 전하, 작은 크기 및 높은 용해도를 감안할 때 칼슘은 단백질 구조의 이상적인 조작제입니다. 따라서 칼슘은 전기화학적 신호를 효소, 전사 또는 전사 후 변화로 변환하는 강력한 수단이 됩니다. 소포체(ER)와 원형질막을 가로지르는 엄격한 칼슘 농도 구배는 세포질 칼슘 농도의 급격한 변화를 가능하게 하는 높은 추진력을 생성합니다. 버퍼링과 활성 전송을 모두 포함하는 여러 메커니즘은 이 기울기를 긴밀하게 유지합니다. 정상적인 세포 기능을 위해 필요하기는 하지만, 이러한 유지는 에너지 비용이 많이 들기 때문에 스트레스 상태에서 특히 취약하다 2.
이와 같이, 세포질 내의 칼슘 조절 장애는 많은 종류의 세포 스트레스의 거의 보편적인 신호입니다. 대사 장애, 독소, 병원균, 기계적 손상 및 유전적 교란은 모두 칼슘 신호 전달을 방해할 수 있습니다. 자극에 관계없이, 전체 세포 수준에서 세포질 칼슘의 지속적이고 통제되지 않은 상승은 세포 사멸을 촉진하고 결국 괴사를 촉진할 수 있습니다 3,4. 그러나 진폭이 낮거나 주파수가 높은 세포질 칼슘 수치의 변화는 다양한 영향을 미친다2. 마찬가지로, 칼슘 변동의 결과는 그것이 발생하는 공간적 미세영역에 따라 달라질 수 있다5. 따라서 칼슘 수치를 모니터링하면 동적 신호 전달 과정에 대한 통찰력을 얻을 수 있지만, 이를 위해서는 상대적으로 높은 시간적 및 공간적 분해능을 사용한 샘플링이 필요합니다.
유전자 인코딩 칼슘 지표(GECI)는 살아있는 세포 시스템에서 연속 샘플링을 위한 강력한 도구입니다6. 가장 널리 사용되는 GECI는 GCaMP로 알려진 GFP 기반 칼슘 반응성 형광 단백질입니다7. 표준 GCaMP는 세 가지 뚜렷한 단백질 도메인, 즉 원형으로 치환된 GFP(cpGFP), 칼모듈린 및 M136의 융합입니다. 칼모듈린 도메인은 칼슘과 결합할 때 형태 변화를 겪어 M13과 상호 작용할 수 있습니다. 칼모듈린-M13 상호작용은 cpGFP의 구조적 변화를 유도하여 여기(excitation) 시 형광 방출을 증가시킵니다. 따라서 칼슘 농도의 증가는 GCaMP 형광 강도의 증가와 상관관계가 있습니다. 이들 센서는 세포질(cytosoli)이거나 특정 세포소기관(specific organelle)을 표적으로 할 수 있다8.
대부분의 조직과 마찬가지로 칼슘은 위장 상피 내에서 다양한 기능을 조절합니다. 장 상피는 영양분과 체액 흡수에 필수적이지만 병원균 침입이나 독성 물질을 피하기 위해 단단한 장벽과 면역 인터페이스를 형성해야 합니다. 칼슘 의존 경로는 이러한 거의 모든 중요한 기능에 영향을 미칩니다 9,10,11. 그러나 장 상피 내의 칼슘 신호 전달은 치료 표적으로서 유망한 잠재력과 함께 아직 미개척 분야로 남아 있습니다. 생체 내 장 상피 내 칼슘 역학을 모니터링하는 것은 여전히 어려운 일이지만, 인간 장 오가노이드(HIO)는 실험을 위한 적응 가능한 생체 외 시스템을 제공합니다12. HIO는 인간 장 줄기 세포에서 유래한 3차원(3D) 스페로이드이며, 분화 시 천연 장 상피의 세포 다양성 대부분을 재현합니다12.
이 프로토콜은 GECI를 발현하는 HIO를 엔지니어링한 다음 엔지니어링된 HIO를 라이브 셀 칼슘 이미징을 위한 단층으로 준비하는 포괄적인 방법을 설명합니다. 칼슘 신호 전달을 방해하는 병리학적 조작의 예로 바이러스 감염을 제시하고 이러한 변화를 정량화하기 위한 분석적 접근 방식을 제공합니다.
프로토콜
이 프로토콜과 대표 실험에 사용된 모든 인간 장 오가노이드(HIO)는 Texas Medical Center Digestive Diseases Enteroid Core에서 획득하고 유지 관리하는 인체 조직에서 파생되었습니다. 모든 샘플은 Baylor College of Medicine의 Institutional Review Board에서 승인한 프로토콜에 따라 수집되었습니다.
1. 재료 및 시약의 준비
- 오가노이드 유지를 위해 세포 배양 처리된 24웰 플레이트, 기저막 매트릭스(BMM), 15mL 코니컬 튜브 및 1.5mL 코니컬 튜브를 수집합니다.
- 성장 인자(CMGF-)가 없는 완전한 배지를 준비하려면 500mL의 고급 DMEM F12에 1M HEPES 5mL, 100x 항생제-항진균제 5mL, 100x 글루타민 보충제 5mL를 추가합니다.
- Wnt-, R-spondin- 및 Noggin 함유 (WRNE) 배지를 제조하려면 CMGF 및 Wnt 조절 배지를 동일한 비율로 혼합하고 Noggin 조절 배지, 10 부피 %, R-spondin 조절 배지, 20 부피 %, 50 ng / mL 인간 표피 성장 인자, 10 mM 니코틴아미드, 10 nM [Leu15] -Gastrin I, 500 nM A-83-01, 10 μM SB202190, 1x B27 보충제, 1x N2 보충제 및 1 mM N- 아세틸 시스테인.
- 렌티바이러스 형질도입을 위해 1x 인산염 완충 식염수(PBS)에 0.05% 트립신-EDTA, 10% FBS가 포함된 CMGF-, 멸균 1x PBS, 폴리브렌, 10μM Y-27632, 렌티바이러스, 높은 Wnt WRNE + 10μM Y-27632를 준비합니다.
- 오가노이드 단층을 생성하기 위해 유리 바닥 10웰 세포 배양 슬라이드, FBS, 콜라겐 IV(탈이온화된(di)H2O에서 1mg/mL), 기저막 매트릭스, 1x PBS에서 0.5mM EDTA, 1x PBS에서 5mM EDTA, 효소 해리 완충액, 10% FBS가 포함된 CMGF-, WRNE + 10μM Y-27632를 준비합니다.
- 오가노이드 단층의 바이러스 감염을 위해 트립신, 로타바이러스 스톡, CMGF-, 25G 바늘, 멸균 1x PBS 및 페놀 레드-프리 분화 배지를 준비합니다.
- 페놀 레드-프리 분화 배지를 준비하려면 페놀 레드-프리 세포 배양 배지 500mL를 취하고 100x MEM 비필수 아미노산 5mL, 100x L-글루타민 5mL, 100nM 피루브산나트륨 5mL 및 1M HEPES 7.5mL를 추가합니다.
- 오가노이드의 면역형광 염색을 위해 4% 포름알데히드(1x PBS에 희석된 16% 포름알데히드), Triton X-100(1x PBS에서 0.1% Triton X-100), 소 혈청 알부민(1x PBS에서 3% 소 혈청 알부민), NH4Cl용액(50mM), DAPI(1x PBS에서 1μg/mL DAPI 용액)를 준비합니다.
2. 유전적으로 인코딩된 칼슘 센서를 발현하기 위한 오가노이드 엔지니어링
참고: 이 프로토콜은 24웰 플레이트(13)에서 30μL의 기저막 매트릭스(BMM)로 도금된 3차원 인간 장 오가노이드의 단일 웰을 형질도입하는 단계를 설명합니다. 대부분의 라인에는 웰당 약 400,000개의 셀이 포함됩니다. 형질도입되지 않은 두 번째 웰이 대조군으로 포함되어야 합니다. 모든 시약과 세포 현탁액을 얼음 위에 보관하십시오.
- 마지막 통과로부터 2-5일 후, HIO의 두 웰에서 유지 보수 WRNE 배지를 제거합니다. 웰당 300μL의 0.05% 트립신-EDTA로 교체하고 위아래로 5배 부드럽게 피펫팅하여 플레이트에서 BMM을 분리합니다. 37°C 인큐베이터에 4분 동안 넣습니다.
- 웰당 500μL의 CMGF- + 10% FBS를 추가합니다. 1mL의 FBS를 위아래로 2회 피펫팅하여 1mL 저결합 피펫 팁을 FBS로 사전 코팅합니다. FBS는 여러 팁에 재사용할 수 있습니다. 사전 코팅된 팁을 사용하여 HIO를 위아래로 10배 피펫팅합니다.
- 각 웰의 내용물을 사전 코팅된 1.5mL 마이크로 원심분리 튜브로 옮깁니다. 추가로 500μL의 CMGF-로 각각을 잘 헹구고 세척액을 해당 튜브에 추가합니다.
- 스윙 버킷 원심분리기에서 100 x g 의 튜브를 4°C에서 5분 동안 원심분리합니다. 상층액과 잔여 BMM을 제거합니다.
- 1x PBS 1mL에 재현탁합니다. 각 튜브를 총 4개의 튜브에 대해 2개의 마이크로 원심분리기 튜브로 나눕니다. 튜브를 100 x g 에서 4°C에서 5분 동안 원심분리합니다. 상층액을 제거합니다.
- 각 튜브를 1x PBS에 한 번 더 재현탁하고 100 x g 에서 4°C에서 5분 동안 원심분리합니다.
- 400μL의 형질주입 배지 및 대조 배지를 준비합니다(표 1). 대조 배지 200μL에 튜브 2개를 재현탁시키고 형질 주입 배지 200μL에 튜브 2개를 재현탁시킵니다.
- 37°C 세포 배양 인큐베이터에서 24시간 동안 배양합니다. 균일한 형질도입을 촉진하기 위해 주기적으로 코팅된 팁으로 위아래로 피펫팅하여 재현탁합니다(예: 형질도입 후 2시간(hpt), 12hpt, 18hpt).
- 24시간 후 100 x g 에서 4°C에서 5분 동안 원심분리기 튜브를 사용합니다. 상층액을 제거합니다.
- 펠릿을 500μL의 1x PBS에 재현탁시켜 세척합니다. 4 °C에서 5분 동안 100 x g 의 원심분리기. 상층액을 제거합니다.
- 얼음처럼 차가운 200 μL 피펫 팁을 사용하여 4 μL의 BMM에 30 개의 펠릿을 각각 재현탁시킵니다. 고르게 분산되도록 위아래로 부드럽게 피펫팅합니다.
- 각 튜브의 내용물을 24웰 플레이트의 자체 웰에 플레이트로 만듭니다. 500 μL의 HighWnt WRNE + 10μM Y-27632를 추가하기 전에 BMM이 응고되도록 37°C에서 10분 동안 배양합니다.
- 형질도입된 HIO를 1주일 동안 성장시켜 이틀에 한 번씩 배지(HighWnt WRNE + 10 μM Y-27632)를 새로 고칩니다. 1주일 후 현미경으로 형광지시약의 발현을 확인합니다. 신호가 강하면 약물 선택을 시작합니다. 신호가 약하면 위에서 설명한 대로 변환을 반복합니다.
- 라인이 설정되면 작용제 처리를 통해 형광 지시약의 기능을 확인합니다. 100nM ADP는 GCaMP 검증을 위한 신뢰할 수 있는 작용제입니다. 아래 설명된 대로 초기 검증을 위해 3D 오가노이드를 테스트합니다.
- 통과 후 BMM의 오가노이드 웰(또는 여러 개)을 별도의 이미징 바닥 플레이트에 플레이트합니다. 오가노이드를 정상 밀도의 약 1/3로 도금하여 이미징 시 과도한 겹침을 방지합니다. 무균 상태를 보장하기 어렵기 때문에 작용제 치료 후 이러한 HIO를 계속 통과시키지 마십시오.
- 통과 후 HIO가 회복되는 데 2-3일이 소요됩니다. 이미징하기 전에 배지를 페놀 적색 무함유 분화 배지로 전환합니다.
- 형광 현미경을 사용하여 488nm 여기와 FITC/GFP 필터 세트를 사용하여 5초마다 획득한 이미지로 3분 실행을 설정합니다. 30초 동안 이미징한 후 100nM ADP 또는 차량 제어를 추가합니다. 신호가 기준선에 가까워질 때까지 ~2분 동안 이미징을 계속합니다. ADP 처리로 GCaMP 형광이 ~2배 증가한 것은 성공적인 transduction 및 바이오센서 기능을 나타냅니다. 형질도입 효율을 보다 정확하게 추정하려면 파트 3에 설명된 프로세스를 통해 생성된 단층을 사용하여 이미징으로 작용제 테스트를 반복합니다.
3. 살아있는 형광 이미징을 위한 HIO 단층 준비
- 10웰 이미징 하단 챔버 슬라이드의 모든 웰을 Collagen IV로 코팅합니다. 이렇게 하려면 34μL의 1mg/mL 콜라겐 IV와 960μL의 멸균 탈이온수를 혼합합니다. 희석된 콜라겐 IV 용액 95μL를 각 웰에 추가하고 37°C에서 0.5-2시간 동안 배양합니다.
- 3D HIO의 4웰에서 WRNE 유지 관리 매체를 제거합니다. HIO는 마지막 통과로부터 5-7일 후여야 합니다.
참고: 1 10웰 플레이트에는 일반적으로 ~1.25 x 106 셀이 필요합니다. 이를 위해서는 일반적으로 각각 30μL의 BMM으로 도금된 3D HIO의 2-4웰이 필요하지만 밀도에 따라 달라집니다. - 웰당 500μL의 1x PBS + 0.5mM EDTA를 추가합니다. 사전 코팅된 1mL 팁을 사용하여 피펫을 위아래로 부드럽게 움직여 플레이트에서 BMM을 분리합니다. 현탁액을 사전 코팅된 15mL 코니컬 튜브로 옮기고 웰처럼 결합하여 동일한 튜브에 넣습니다.
- 추가로 500μL의 PBS + 0.5mM EDTA로 각각을 잘 헹굽니다. 300 x g 에서 4°C에서 5분 동안 원심분리합니다. 상층액과 잔여 BMM을 제거합니다.
- 사전 코팅된 팁을 사용하여 나머지 펠릿을 3mL의 PBS + 5mM EDTA에 재현탁합니다(이는 첫 번째 세척보다 10배 더 많은 EDTA입니다).
- 300 x g 에서 4°C에서 5분 동안 원심분리합니다. 상층액과 잔여 BMM을 제거합니다. 펠릿을 2mL의 효소 해리 완충액에 재현탁시킵니다.
- 37°C 비드/수조에서 5분 동안 배양합니다. CMGF- + 10% FBS 3mL와 피펫을 부드럽게 첨가하여 섞습니다.
- 300 x g 에서 4°C에서 5분 동안 원심분리합니다. 상층액을 제거합니다. CMGF-의 1mL를 추가합니다.
- 사전 코팅된 팁 80x-100x로 위아래로 세게 피펫팅하여 HIO를 기계적으로 단일 셀로 분해합니다. 300 x g 에서 4°C에서 5분 동안 원심분리합니다. 상층액을 제거합니다.
- 1 mL의 WRNE + 10 μM Y-27632에 재현탁 1mL 현탁액에 대한 세포 계수를 구합니다.
- 셀 현탁액을 WRNE + 10μM Y-27632로 희석하여 1.25 x 105 cells/100 μL(1.25 x 106 cells/mL)의 농도를 달성합니다.
- 200μL 피펫을 사용하여 콜라겐이 이제 웰 바닥에 가라앉았으므로 1단계에서 준비한 플레이트에서 콜라겐 용액을 제거합니다. 피펫 팁으로 웰 바닥을 만지지 마십시오.
- 사전 코팅된 200μL 피펫 팁을 사용하여 웰당 3.11단계(1.25 x 105 개 세포)의 세포 용액 100μL를 추가합니다.
- 37°C 세포 배양 인큐베이터에서 24시간 동안 배양합니다. 24시간 후 모든 웰에서 배양 배지를 제거하고 웰당 100μL의 분화 배지로 교체합니다.
알림: 이 시점에서 셀이 플레이트에 부착되어야 합니다. 단층은 합류하거나 완전히 평면이 아닐 수 있습니다. - 슬라이드를 37°C 세포 배양 인큐베이터에 다시 넣습니다. 단층이 합류할 때까지 24시간마다 분화 배지를 새로 고칩니다. 이것은 일반적으로 도금 후 3-5일이 필요합니다. 이 시점 이후에는 단층이 다운스트림 응용 분야에 사용할 준비가 된 것입니다.
4. HIO 단층의 바이러스 감염
- 바이러스 접종물을 준비합니다. 필요한 경우 트립신은 바이러스 스톡을 활성화합니다. 로타바이러스의 경우 10μg/mL Worthington's Trypsin을 바이러스 스톡에 추가하고 37°C에서 1시간 동안 배양합니다.
- 다음 두 가지 방법 중 하나를 사용하여 활성화된 바이러스 스톡을 희석합니다.
- 하나의 바이러스 균주만 사용하여 감염된 세포의 최대 수를 목표로 하는 경우 50μL의 활성 바이러스 스톡과 50μL CMGF-를 혼합합니다.
- 여러 균주를 비교하는 경우 동등한 감염 다중성(MOI)을 달성하기 위해 접종물을 준비합니다. 다음 공식을 사용하여 원하는 MOI에 필요한 웰당 바이러스 스톡의 양을 결정합니다. 웰당 100μL의 최종 부피를 달성하기 위해 계산된 바이러스 스톡의 부피에 CMGF-를 추가합니다.
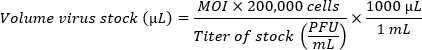
참고: 직경이 5.46mm인 웰(96웰 플레이트)에 도금된 단일 HIO 단층에는 약 200,000개의 셀이 포함되어 있습니다. 단층에서 감염 효율이 낮기 때문에 높은 MOI(세포당 1억-100만 개의 바이러스 입자)가 선호됩니다. 최적의 MOI는 경험적으로 결정되어야 합니다.
- 200mL 피펫을 사용하여 HIO 단층에서 배지를 부드럽게 제거하여 단층을 감염시킵니다. 100mL의 바이러스 접종물 또는 모의 접종물로 교체합니다.
- 대부분의 로타바이러스 스톡은 MA104 세포에서 증식하기 때문에 모의 접종에는 감염되지 않은 MA104 세포 용해물을 사용하십시오. 감염된 웰에 사용된 바이러스 스톡의 부피와 동일한 부피를 사용하고 웰당 최종 부피 100mL에 대해 CMGF-를 추가합니다.
- 로타바이러스14와 같이 기저변 세포를 감염시키는 바이러스의 경우 25G 바늘을 사용하여 단층을 채점합니다. 우물의 바닥에서 상단까지 단층의 길이를 가로질러 단일 점수를 만드는 것이 가장 쉽습니다. 바늘을 비스듬한 면이 위로 향하게 하여 운동 방향과 반대 방향으로 단층에 부드럽게 밀어 넣고 단층 길이를 가로질러 끌어 흉터를 만듭니다(그림 2A).
- 37 °C 인큐베이터에서 2 시간 동안 배양합니다. 바이러스를 제거하고 모의 접종을합니다. 단층을 1x PBS로 한 번 세척합니다.
- 100μL의 페놀 적색이 없는 분화 배지를 추가합니다. 이미징할 준비가 될 때까지 슬라이드를 37°C 인큐베이터에 넣습니다. 최적의 이미징 창은 바이러스 감염의 동역학에 따라 달라집니다. 로타바이러스의 경우, 감염 후 6-8시간 후에 영상 촬영을 시작합니다.
- 다음 두 가지 방법 중 하나를 사용하여 활성화된 바이러스 스톡을 희석합니다.
5. 감염된 단층의 Ca2+ 이미징
- 스테이지 탑 인큐베이터를 37°C로 예열합니다. 가습 CO2 를 0.02L/min으로 실행합니다. 감염된 단층이 있는 슬라이드를 인큐베이터 챔버에 넣고 뚜껑을 밀봉합니다.
- 명시야(BF) 조명과 20x 대물렌즈를 사용하여 이미징할 단층 내에서 시야의 X 및 Y 좌표를 선택합니다. 대부분의 감염된 세포는 스크래치 근처에 있으므로 점수가 매겨진 영역을 고려하여 포인트를 선택하는 것이 가장 좋습니다.
- 이미징 파라미터를 최적화하고 488nm excitation을 사용하여 이미지를 획득합니다. 광독성을 최소화하려면 광원을 50ms 노출 시간으로 50% 전력으로 설정하십시오. 적절한 획득 매개변수는 다를 수 있으며 여러 획득을 획득하여 최적화해야 합니다. 포화 상태의 픽셀이 없는지 확인합니다. 형광 칼슘 센서의 전체 동적 범위를 감지할 수 있도록 필요에 따라 노출 시간과 광력을 조정합니다.
- 다른 형광단(예: 형광 태그가 부착된 바이러스)을 사용하는 경우 이러한 채널에서 최적화를 반복합니다. 이미징 루프를 설정하고 1분 루프에서 선택한 모든 X, Y 좌표에서 488nm 이미지를 획득합니다. 이 루프를 연속적으로 이미지화합니다. 형광 태그가 부착된 바이러스를 사용하는 경우 10번째 루프마다 적절한 채널에서 이미지를 획득합니다(즉, 10분당 1개의 이미지).
- ~18시간 동안 이미지를 수집합니다. 형광 태그가 없는 바이러스를 사용하는 경우 단층을 고정하고 면역형광 염색을 수행하여 감염된 세포를 식별합니다. 아래 설명된 대로 이미징 실행을 완료한 후 이 작업을 수행합니다.
- 이미징 매체를 제거하고 1x PBS에서 100μL의 4% 포름알데히드로 교체합니다. 실온에서 30분 동안 배양합니다.
- 고정액을 제거하고 100μL의 50mM NH4Cl로 10분 동안 담금질합니다. NH4Cl을 제거합니다. 실온에서 1시간 동안 또는 4°C에서 하룻밤 동안 1x PBS에서 0.1% Triton X-100의 100μL로 단층을 투과시킵니다.
- 투과화 버퍼를 제거합니다. 100μL의 3% 소 혈청 알부민을 1x PBS에 넣고 1시간 동안 차단합니다.
- 차단 용액을 제거합니다. 1x PBS에서 권장/최적화된 농도로 희석된 1차 항체를 추가합니다. 웰당 100μL의 항체 용액을 추가합니다.
- 4 °C에서 부드럽게 흔들면서 하룻밤 동안 배양합니다. 1차 항체를 제거합니다. 1x PBS로 3번 세척합니다.
- 1x PBS에서 권장/최적화된 농도로 희석된 형광 접합 2차 항체를 추가합니다. 웰당 100μL를 추가합니다. 실온에서 2시간 동안 배양합니다.
참고: FPBase15 는 멀티플렉싱 시 블리드스루(bleed-through)를 방지하는 2차측을 선택하는 데 도움이 되는 훌륭한 리소스입니다. 대부분의 보조 복제본은 1x PBS에서 1:1000 희석으로 효과적입니다. - 2차 항체를 제거합니다. 1x PBS에서 1μg/mL DAPI 용액을 만들고 웰당 100μL를 추가합니다. 20분 동안 배양합니다. DAPI를 제거합니다. 1x PBS로 3번 세척합니다.
- 이미징을 위해 고정 단층을 100μL의 1x PBS에 보관합니다. 슬라이드를 현미경 스테이지에 다시 놓고, 라이브 이미징 실행에서 X 및 Y 좌표를 다시 로드하고, 염색에 사용된 2차 항체에 해당하는 채널의 각 다지점을 이미지화합니다. 라이브 이미징 실행의 이미지를 참조하여 동일한 지점이 캡처되었는지 확인합니다.
6. 세포간 칼슘파의 정량 분석
- Fiji가 다음 플러그인과 함께 설치된 FIJI(Fiji is Just ImageJ)인지 확인합니다. Bio-Formats(FIJI에는 사전 설치되어 있어야 하지만 ImageJ에는 설치되지 않음); 쿡북: 설치하려면 FIJI 메뉴에서 도움말 > 업데이트 > 업데이트 사이트 관리로 이동합니다. 쿡북(Cookbook)을 확인합니다. FIJI를 다시 시작합니다.
- 라이브 이미징 실행의 데이터 파일을 여러 파일로 분할하여 각 X, Y 좌표를 분리합니다. Nikon NES Elements에서 파일 > 가져오기/내보내기 > 멀티포인트 분할을 선택합니다. 내보낼 폴더와 적절한 접두어를 선택합니다.
- 피지를 엽니다. 분할된 멀티포인트에서 단일 파일을 로드합니다. 다중 채널 이미지를 사용하는 경우 채널을 분할합니다. 488nm 채널(GCaMP)의 이미지가 있는 창을 선택합니다.
- DeltaF Up 함수를 실행하여 한 시점에서 다음 시점까지 각 픽셀 값의 변화를 확인합니다. 이렇게 하려면 [Cookbook] > T-Functions > Delta F Up을 선택합니다.
- 칼슘 파동이 있는 이미지를 찾을 때까지 스택을 스크롤합니다. 칼슘파를 표시한 상태에서 Image > Adjust > Threshold(임계값 조정)를 클릭하여 임계값을 설정합니다. 하한을 조정하여 임계값을 초과하는 파동의 일부가 아닌 신호의 양을 최소화합니다. 위에서 설명한 파라미터를 사용하여 획득한 16비트 이미지의 경우 낮은 임계값인 600으로 시작하여 필요에 따라 조정합니다.
- 입자 분석기를 실행하여 분석(Analyze) > 입자 분석(Analyze Particles)을 클릭하여 파동을 분할합니다. 범위를 조정하여 파동의 최소 크기를 μm2 단위로 설정합니다. 기준선에서 0-무한대로 설정됩니다. 20x 대물렌즈를 사용하여 획득한 MA104 세포 이미지의 경우 먼저 하한을 10,000μm2로 늘리고 적절하게 조정합니다.
- 결과 표시 및 결과 지우기 확인란을 선택합니다. '표시' 드롭다운 메뉴에서 개요를 선택한 다음 확인을 클릭합니다. 이렇게 하면 2개의 출력이 생성됩니다: 하나의 창인 Results는 감지된 각 파동, 파동의 면적, 평균, 최소 및 최대 강도 값(델타 F 기준)을 나열합니다. [파일 이름]의 그리기라는 제목의 다른 창에는 분할된 파도의 윤곽선이 있는 새 스택이 포함됩니다. 이를 사용하여 웨이브 감지를 확인합니다. 필요한 경우 임계값과 크기 범위를 조정하고 웨이브 호출을 다시 확인합니다.
- 일괄 처리의 경우 포함된 매크로 스크립트(보충 코딩 파일 1)를 사용합니다. 이를 활용하려면 아래 설명된 단계를 따르십시오.
- 6.2단계에서 설명한 대로 다중 지점을 단일 파일로 분할합니다. 피지를 엽니다. Batch > 매크로> 프로세스를 선택합니다. "입력" 상자에서 분할된 멀티포인트 파일이 포함된 폴더에 맵을 제공합니다. "출력" 상자는 비워 둡니다.
- 보조 코딩 파일 1의 스크립트를 상자에 붙여넣습니다. 스크립트에서 강도 임계값 및 크기 임계값을 조정하여 6.5단계와 6.6단계에서 최적화된 매개 변수를 반영합니다.
- 프로세스를 클릭합니다. 멀티 포인트 수와 컴퓨터의 처리 속도에 따라 모든 이미지를 처리하는 데 30 분 이상 걸릴 수 있습니다. 완료되면 데이터가 바탕 화면에 "쓰기 후 이름 바꾸기"라는 새 스프레드시트 파일에 기록됩니다. 출력 파일에는 각 멀티포인트에 대한 웨이브 카운트와 웨이브 메트릭(면적, 평균 강도, 최소 및 최대 강도, 강도 밀도)이 포함되어야 합니다.
결과
그림 1A 는 GCaMP6를 안정적으로 발현하기 위해 형질도입된 3차원 인간 장 오가노이드를 포함하는 BMM 돔을 보여줍니다. 그림 1B 는 파종 후 24시간, 48시간 및 72시간에 단층으로 재도금된 동일한 오가노이드 라인을 보여줍니다. GCaMP6의 기능을 검증하기 위해 단층을 4분 동안 2초마다 형광 현미경으로 이미지화하고 ~20초 후에 100nM ADP를 배지에 첨가했습니다. ...
토론
세포질 Ca2+ 수준의 변화는 상피 내 병리학의 원인이자 결과일 수 있습니다10,16,17. 세포질 칼슘의 증가는 칼슘 의존성 염화물 채널의 활성화를 통해 분비를 직접 유도할 수 있습니다18,19 TMEM16A. Ca2 +에 대한 반응으로 TMEM16A의 활성화는 염화물의 정점 유출을 허용하여 ?...
공개
저자는 공개할 재정적 이해관계가 없습니다.
감사의 말
이 연구는 미국 국립보건원(NIH)의 보조금 R01DK115507 및 R01AI158683(PI: J. M. Hyser)의 지원을 받았습니다. 연수생 지원은 NIH 보조금 F30DK131828(PI: J.T. Gebert), F31DK132942(PI: F. J. Scribano) 및 F32DK130288(PI: K.A. Engevik)에서 제공했습니다. 오가노이드 유지 배지를 제공해 주신 Texas Medical Center Digestive Diseases Enteroid Core에 감사드립니다.
자료
| Name | Company | Catalog Number | Comments |
| Advanced DMEM F12 | Gibco | 12634028 | |
| [Leu15]-Gastrin I | Sigma-Aldrich | G9145 | |
| 0.05% Trypsin EDTA | Gibco | 25300054 | |
| 0.05% Trypsin EDTA | Gibco | 25300054 | |
| 1.5mL microcentrifuge tubes | Fisherbrand | 5408137 | |
| 15mL conical tubes | Thermofisher Scientific | 0553859A | |
| 16% formaldehyde | Thermofisher Scientific | 28906 | |
| 1M HEPES | Gibco | 15630080 | |
| 1M HEPES | Gibco | 15630080 | |
| 1X PBS | Corning | 21-040-CV | |
| 25 gauge needle | Thermofisher Scientific | 1482113D | |
| A-83-01 | Tocris | 2939 | |
| ADP | Sigma-Aldrich | A2754 | |
| Advanced DMEM F12 | Gibco | 12634028 | |
| Antibiotic-antimycocytic | Gibco | 15240062 | |
| Antibiotic-antimycotic | Gibco | 15240062 | |
| B27 Supplement | Gibco | 17504-044 | |
| Bovine serum albumin | FisherScientific | BP1600100 | |
| CellView Cell Culture Slide, PS, 75/25 MM, Glass Bottom, 10 compartments | Greiner | 543979 | |
| Collagen IV | Sigma Aldrich | C5533 | |
| DAPI | Thermofisher Scientific | D1306 | |
| EDTA | Corning | 46-034-CI | |
| Fetal bovine serum | Corning | 35010CV | |
| Fetal bovine serum | Corning | 35010CV | |
| Fluorobrite | Gibco | A1896701 | |
| GlutaMAX | Gibco | 35050079 | |
| GlutaMAX | Gibco | 35050079 | |
| Human epidermal growth factor | ProteinTech | HZ-1326 | |
| Lentivirus | VectorBuilder | (variable) | |
| Matrigel | BD Biosceicen | 356231/CB40230C | |
| N2 Supplement | Gibco | 17502-048 | |
| N-acetylcysteine | Sigma-Aldrich | A9165-5G | |
| NH4Cl | Sigma-Aldrich | A9434 | |
| Nicotinamide | Sigma-Aldrich | N0636 | |
| Nunc Cell Culture Treated 24-well Plates | Thermofisher Scientific | 142475 | |
| Polybrene | MilliporeSigma | TR1003G | |
| SB202190 | Sigma-Aldrich | S70767 | |
| Triton X-100 | Fisher BioReagents | BP151100 | |
| TrypLE Express Enzyme, no phenol red | Thermofisher Scientific | 12604013 | |
| Trypsin | Worthington Biochemical | NC9811754 | |
| Y-27632 | Tocris | 1254 |
참고문헌
- Bootman, M. D., Bultynck, G. Fundamentals of cellular calcium signaling: A primer. Cold Spring Harb Perspect Biol. 12 (1), a038802 (2020).
- Clapham, D. E. Calcium signaling. Cell. 131 (6), 1047-1058 (2007).
- Danese, A., et al. Cell death as a result of calcium signaling modulation: A cancer-centric prospective. Biochim Biophys Acta Mol Cell Res. 1868 (8), 119061 (2021).
- Harr, M. W., Distelhorst, C. W. Apoptosis and autophagy: Decoding calcium signals that mediate life or death. Cold Spring Harb Perspect Biol. 2 (10), a005579 (2010).
- Barak, P., Parekh, A. B. Signaling through Ca2+ microdomains from store-operated CRAC channels. Cold Spring Harb Perspect Biol. 12 (7), a035097 (2020).
- Nakai, J., Ohkura, M., Imoto, K. A high signal-to-noise Ca(2+) probe composed of a single green fluorescent protein. Nat Biotechnol. 19 (2), 137-141 (2001).
- Erofeev, A. I., Vinokurov, E. K., Vlasova, O. L., Bezprozvanny, I. B. GCaMP, a family of single-fluorophore genetically encoded calcium indicators. J Evol Biochem Phys. 59 (4), 1195-1214 (2023).
- Suzuki, J., Kanemaru, K., Iino, M. Genetically encoded fluorescent indicators for organellar calcium imaging. Biophys J. 111 (6), 1119-1131 (2016).
- Nászai, M., Cordero, J. B. Intestinal stem cells: Got calcium. Curr Biol. 26 (3), R117-R119 (2016).
- Barrett, K. E. Calcium-mediated chloride secretion in the intestinal epithelium: Significance and regulation. Curr Top Membr. 53, 257-282 (2002).
- Xu, J., et al. Calcium-sensing receptor regulates intestinal dipeptide absorption via Ca2+ signaling and IKCa activation. Physiol Rep. 8 (1), e14337 (2020).
- Clevers, H. Modeling development and disease with organoids. Cell. 165 (7), 1586-1597 (2016).
- Lin, S. C., Haga, K., Zeng, X. L., Estes, M. K. Generation of CRISPR–Cas9-mediated genetic knockout human intestinal tissue–derived enteroid lines by lentivirus transduction and single-cell cloning. Nat Protoc. 17 (4), 1004-127 (2022).
- Crawford, S. E., Ramani, S., Blutt, S. E., Estes, M. K. Organoids to dissect gastrointestinal virus-host interactions: What have we learned. Viruses. 13 (6), 999 (2021).
- Lambert, T. J. FPbase: a community-editable fluorescent protein database. Nat Methods. 16 (4), 277-278 (2019).
- Lai, Y., et al. Inhibition of calcium-triggered secretion by hydrocarbon-stapled peptides. Nature. 603 (7903), 949-956 (2022).
- Chang-Graham, A. L., et al. Rotavirus induces intercellular calcium waves through ADP signaling. Science. 370 (6519), eabc3621 (2020).
- Lee, B., et al. Anoctamin 1/TMEM16A controls intestinal Cl− secretion induced by carbachol and cholera toxin. Exp Mol Med. 51 (8), 1-14 (2019).
- Saha, T., et al. Intestinal TMEM16A control luminal chloride secretion in a NHERF1 dependent manner. Biochem Biophys Rep. 25, 100912 (2021).
- Mroz, M. S., Keely, S. J. Epidermal growth factor chronically upregulates Ca2+-dependent Cl− conductance and TMEM16A expression in intestinal epithelial cells. J Physiol. 590 (8), 1907-1920 (2012).
- Sui, J., et al. Dual role of Ca2+-activated Cl− channel transmembrane member 16A in lipopolysaccharide-induced intestinal epithelial barrier dysfunction in vitro. Cell Death Dis. 11 (5), 404 (2020).
- Bellono, N. W., et al. Enterochromaffin cells are gut chemosensors that couple to sensory neural pathways. Cell. 170 (1), 185-198.e16 (2017).
- Paradis, T., Bègue, H., Basmaciyan, L., Dalle, F., Bon, F. Tight junctions as a key for pathogens invasion in intestinal epithelial cells. Int J Mol Sci. 22 (5), 2506 (2021).
- Samak, G., et al. Calcium/Ask1/MKK7/JNK2/c-Src signalling cascade mediates disruption of intestinal epithelial tight junctions by dextran sulfate sodium. Biochem J. 465 (3), 503-515 (2015).
- Deng, H., Gerencser, A. A., Jasper, H. Signal integration by Ca2+ regulates intestinal stem cell activity. Nature. 528 (7581), 212-217 (2015).
- Saurav, S., Tanwar, J., Ahuja, K., Motiani, R. K. Dysregulation of host cell calcium signaling during viral infections: Emerging paradigm with high clinical relevance. Mol Aspects Med. 81, 101004 (2021).
- Chang-Graham, A. L., et al. Rotavirus calcium dysregulation manifests as dynamic calcium signaling in the cytoplasm and endoplasmic reticulum. Sci Rep. 9 (1), 10822 (2019).
- Hyser, J. M., Collinson-Pautz, M. R., Utama, B., Estes, M. K. Rotavirus disrupts calcium homeostasis by NSP4 viroporin activity. mBio. 1 (5), e00265-e00310 (2010).
- Pham, T., Perry, J. L., Dosey, T. L., Delcour, A. H., Hyser, J. M. The Rotavirus NSP4 viroporin domain is a calcium-conducting ion channel. Sci Rep. 7, 43487 (2017).
- Crawford, S. E., Hyser, J. M., Utama, B., Estes, M. K. Autophagy hijacked through viroporin-activated calcium/calmodulin-dependent kinase kinase-β signaling is required for rotavirus replication. Proc Natl Acad Sci U S A. 109 (50), E3405-E3413 (2012).
- Crawford, S. E., Criglar, J. M., Liu, Z., Broughman, J. R., Estes, M. K. COPII vesicle transport is required for Rotavirus NSP4 interaction with the autophagy protein LC3 II and trafficking to viroplasms. J Virol. 94 (1), e01341 (2019).
- Pando, V., Iša, P., Arias, C. F., Ló Pez, S. Influence of calcium on the early steps of Rotavirus infection. Virology. 295 (1), 190-200 (2002).
- Hyser, J. M., Estes, M. K. Pathophysiological consequences of calcium-conducting viroporins. Annu Rev Virol. 2 (1), 473-496 (2015).
- Strtak, A. C., et al. Recovirus NS1-2 has viroporin activity that induces aberrant cellular calcium signaling to facilitate virus replication. mSphere. 4 (5), e00506-e00519 (2019).
- In, J. G., Foulke-Abel, J., Clarke, E., Kovbasnjuk, O. Human colonoid monolayers to study interactions between pathogens, commensals, and host intestinal epithelium. J Vis Exp. (146), 59357 (2019).
- Hirota, A., AlMusawi, S., Nateri, A. S., Ordóñez-Morán, P., Imajo, M. Biomaterials for intestinal organoid technology and personalized disease modeling. Acta Biomater. 132, 272-287 (2021).
- Cevallos Porta, D., López, S., Arias, C. F., Isa, P. Polarized rotavirus entry and release from differentiated small intestinal cells. Virology. 499, 65-71 (2016).
- Mirabelli, C., et al. Human Norovirus efficiently replicates in differentiated 3D-human intestinal enteroids. J Virol. 96 (22), e0085522 (2022).
- Icha, J., Weber, M., Waters, J. C., Norden, C. Phototoxicity in live fluorescence microscopy, and how to avoid it. Bioessays. 39 (8), 28749075 (2017).
- Li, J., et al. Engineering of NEMO as calcium indicators with large dynamics and high sensitivity. Nat Methods. 20 (6), 918-924 (2023).
재인쇄 및 허가
JoVE'article의 텍스트 или 그림을 다시 사용하시려면 허가 살펴보기
허가 살펴보기This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. 판권 소유