Bu içeriği görüntülemek için JoVE aboneliği gereklidir. Oturum açın veya ücretsiz deneme sürümünü başlatın.
Method Article
Genetik Olarak Kodlanmış Kalsiyum İndikatörleri Kullanılarak Virüsle Enfekte İnsan Bağırsak Organoid Tek Tabakalarının Canlı Kalsiyum Görüntülemesi
Bu Makalede
Özet
Bu protokol, virüsle enfekte insan bağırsak organoidlerinde kalsiyum görüntüleme yapmak için bir yaklaşımı açıklar ve analize bir yaklaşım sunar.
Özet
Kalsiyum sinyali, hemen hemen her dokunun ayrılmaz bir düzenleyicisidir. Bağırsak epiteli içinde kalsiyum, salgı aktivitesinin, aktin dinamiklerinin, inflamatuar yanıtların, kök hücre proliferasyonunun ve diğer birçok karakterize edilmemiş hücresel fonksiyonun düzenlenmesinde rol oynar. Bu nedenle, bağırsak epiteli içindeki kalsiyum sinyal dinamiklerinin haritalanması, homeostatik hücresel süreçler hakkında fikir verebilir ve çeşitli uyaranlara benzersiz tepkileri ortaya çıkarabilir. İnsan bağırsak organoidleri (HIO'lar), bağırsak epitelini incelemek için yüksek verimli, insan kaynaklı bir modeldir ve bu nedenle kalsiyum dinamiklerini araştırmak için yararlı bir sistemi temsil eder. Bu makale, HIO'ları genetik olarak kodlanmış kalsiyum göstergeleri (GECI'ler) ile kararlı bir şekilde dönüştürmek, canlı floresan mikroskobu gerçekleştirmek ve kalsiyum sinyallerini anlamlı bir şekilde karakterize etmek için görüntüleme verilerini analiz etmek için bir protokolü açıklamaktadır. Temsili bir örnek olarak, 3 boyutlu HIO'lar, yeşil floresan protein bazlı sitozolik bir GECI olan GCaMP6'ları stabil bir şekilde eksprese etmek için lentivirüs ile dönüştürüldü. Tasarlanan HIO'lar daha sonra tek hücreli bir süspansiyona dağıtıldı ve tek katmanlar olarak tohumlandı. Farklılaşmadan sonra, HIO tek tabakaları rotavirüs ile enfekte edildi ve/veya kalsiyum yanıtını uyardığı bilinen ilaçlarla tedavi edildi. Sıcaklık kontrollü, nemlendirilmiş canlı görüntüleme odası ile donatılmış bir epifloresan mikroskobu, enfekte olmuş veya ilaçla tedavi edilmiş tek tabakaların uzun süreli görüntülenmesine izin verdi. Görüntülemenin ardından, elde edilen görüntüler ücretsiz olarak kullanılabilen analiz yazılımı ImageJ kullanılarak analiz edildi. Genel olarak, bu çalışma HIO'larda hücresel sinyallemeyi karakterize etmek için uyarlanabilir bir boru hattı oluşturur.
Giriş
Kalsiyum, hücresel fizyolojinin düzenlenmesinde kritik bir rol oynayan, yaygın olarak korunan ikinci bir habercidir1. Güçlü yükü, küçük boyutu ve fizyolojik koşullarda yüksek çözünürlüğü göz önüne alındığında, kalsiyum, protein konformasyonunun ideal bir manipülatörüdür. Bu, kalsiyumu elektrokimyasal sinyalleri enzimatik, transkripsiyonel veya transkripsiyon sonrası değişikliklere dönüştürmek için güçlü bir araç haline getirir. Endoplazmik retikulum (ER) ve plazma membranları boyunca katı kalsiyum konsantrasyonu gradyanları, sitozolik kalsiyum konsantrasyonunda hızlı değişikliklere izin veren yüksek bir itici güç oluşturur. Hem arabelleğe alma hem de aktif aktarım dahil olmak üzere birden çok mekanizma bu gradyanı sıkı bir şekilde korur. Normal hücresel fonksiyonlar için gerekli olsa da, bu bakım enerjik olarak pahalıdır ve bu da onu stres durumlarında özellikle duyarlı hale getirir 2.
Bu nedenle, sitozol içindeki kalsiyumun düzensizliği, birçok hücresel stres türünün neredeyse evrensel bir sinyalidir. Metabolik bozukluklar, toksinler, patojenler, mekanik hasar ve genetik bozulmaların tümü kalsiyum sinyalini bozabilir. Uyarandan bağımsız olarak, tüm hücre düzeyinde, sitozolik kalsiyumdaki sürekli, kontrolsüz artışlar apoptozu ve sonunda nekrozu teşvik edebilir 3,4. Bununla birlikte, daha düşük genlikli veya daha yüksek frekanslı sitozolik kalsiyum seviyelerindeki değişikliklerin değişen etkileri vardır2. Benzer şekilde, kalsiyum dalgalanmalarının sonuçları, meydana geldikleri uzamsal mikro alana bağlı olarak farklılık gösterebilir5. Bu nedenle kalsiyum seviyelerinin izlenmesi, dinamik sinyalleme süreçleri hakkında fikir verebilir, ancak bu, nispeten yüksek zamansal ve uzamsal çözünürlüğe sahip örnekleme gerektirir.
Genetik olarak kodlanmış kalsiyum indikatörleri (GECI'ler), canlı hücre sistemlerinde sürekli örnekleme için güçlü araçlardır6. En yaygın kullanılan GECI'lerden bazıları, GCaMPs7 olarak bilinen GFP bazlı kalsiyuma duyarlı floresan proteinlerdir. Kanonik GCaMP, üç farklı protein alanının bir birleşimidir: dairesel olarak permütasyonlu bir GFP (cpGFP), kalmodulin ve M136. Kalmodulin alanı, kalsiyumun bağlanması üzerine bir konformasyon değişikliğine uğrar ve M13 ile etkileşimine izin verir. Kalmodulin-M13 etkileşimi, cpGFP'de, uyarma üzerine floresan emisyonunu artıran konformasyonel bir değişikliğe neden olur. Bu nedenle, kalsiyum konsantrasyonundaki bir artış, GCaMP floresan yoğunluğundaki bir artışla ilişkilidir. Bu sensörler sitozolik olabilir veya belirli organellerihedefleyebilir 8.
Çoğu dokuya benzer şekilde, kalsiyum gastrointestinal epitel içindeki çeşitli işlevleri düzenler. Bağırsak epiteli, besin ve sıvı emilimi için ayrılmaz bir parçadır, ancak aynı zamanda patojen istilasını veya toksik hakaretleri önlemek için sıkı bir bariyer ve bağışıklık arayüzü oluşturmalıdır. Kalsiyuma bağımlı yollar bu hayati fonksiyonların neredeyse tamamını etkiler 9,10,11. Bununla birlikte, bağırsak epiteli içindeki kalsiyum sinyali, terapötik bir hedef olarak umut verici potansiyele sahip, yeterince keşfedilmemiş bir sınır olmaya devam etmektedir. Bağırsak epitelindeki kalsiyum dinamiklerinin in vivo olarak izlenmesi zorluklar sunmaya devam ederken, insan bağırsak organoidleri (HIO'lar) deney için uyarlanabilir bir ex vivo sistem sunar12. HIO'lar, insan bağırsak kök hücrelerinden türetilen 3 boyutlu (3D) sferoidlerdir ve farklılaşma üzerine, doğal bağırsak epitelinin hücresel çeşitliliğinin çoğunu özetler12.
Bu protokol, GECI'leri eksprese eden HIO'ları tasarlamak ve daha sonra tasarlanmış HIO'ları canlı hücreli kalsiyum görüntüleme için tek katmanlar olarak hazırlamak için kapsamlı yöntemleri açıklar. Kalsiyum sinyalizasyonunu bozan patolojik bir manipülasyon örneği olarak viral enfeksiyonu sunar ve bu değişiklikleri ölçmek için analitik bir yaklaşım sağlar.
Protokol
Bu protokolde kullanılan tüm insan bağırsak organoidleri (HIO'lar) ve temsili deneyler, Texas Tıp Merkezi Sindirim Hastalıkları Enteroid Çekirdeği tarafından elde edilen ve sürdürülen insan dokusundan türetilmiştir. Tüm örnekler, Baylor Tıp Fakültesi'ndeki Kurumsal İnceleme Kurulu tarafından onaylanan bir protokole uygun olarak toplandı.
1. Malzemelerin ve reaktiflerin hazırlanması
- Organoid bakımı için, hücre kültürü ile muamele edilmiş 24 oyuklu plakalar, bazal membran matrisi (BMM), 15 mL konik tüpler ve 1.5 mL konik tüpler toplayın.
- Büyüme faktörleri (CMGF-) olmadan tam ortam hazırlamak için, 500 mL gelişmiş DMEM F12'ye 5 mL 1M HEPES, 5 mL 100x antibiyotik-antimikotik, 5 mL 100x glutamin takviyesi ekleyin.
- Wnt-, R-spondin- ve Noggin içeren (WRNE) ortamı hazırlamak için, eşit miktarda CMGF ve Wnt koşullu ortamı karıştırın, Noggin koşullu ortam, hacimce %10, R-spondin koşullu ortam, hacimce %20, 50 ng/mL insan epidermal büyüme faktörü, 10 mM nikotinamid, 10 nM [Leu15]-Gastrin I, 500 nM A-83-01, 10 μM SB202190, 1x B27 takviyesi, 1x N2 takviyesi ve 1 mM N-asetilsistein.
- Lentiviral transdüksiyon için Fetal Sığır Serumu (FBS),% 0.05 Tripsin-EDTA 1x fosfat tamponlu salin (PBS),% 10 FBS ile CMGF, Steril 1x PBS, Polibren, 10 μM Y-27632, Lentivirus, Yüksek Wnt WRNE + 10 μM Y-27632 hazırlayın
- Organoid tek tabakalar oluşturmak için, cam tabanlı 10 oyuklu hücre kültürü slaytı, FBS, Kollajen IV (deiyonize (di)H2O'da1 mg/mL), bazal membran matrisi, 1x PBS'de 0,5 mM EDTA, 1x PBS'de 5 mM EDTA, enzimatik ayrışma tamponu, %10 FBS'li CMGF-, WRNE + 10 μM Y-27632.
- Organoid tek tabakaların viral enfeksiyonu için tripsin, rotavirüs stoğu, CMGF-, 25G iğne, Steril 1x PBS ve fenol kırmızısı içermeyen farklılaşma ortamı hazırlayın.
- Fenol kırmızısı içermeyen farklılaşma ortamı hazırlamak için 500 mL fenol kırmızısı içermeyen hücre kültürü ortamı alın, 5 mL 100x MEM esansiyel olmayan amino asit, 5 mL 100x L-Glutamin, 5 mL 100 nM sodyum piruvat ve 7.5 mL 1M HEPES ekleyin.
- Organoidlerin immünofloresan boyaması için,% 4 formaldehit (1x PBS'de seyreltilmiş% 16 formaldehit), Triton X-100 (1x PBS'de% 0.1 Triton X-100), Sığır serum albümini (1x PBS'de% 3 sığır serum albümini), NH4Cl çözeltisi (50 mM), DAPI (1x PBS'de 1 μg / mL DAPI çözeltisi).
2. Genetik olarak kodlanmış kalsiyum sensörlerini ifade etmek için mühendislik organoidleri
NOT: Bu protokol, 24 oyuklu bir plaka13 üzerinde 30 μL Bodrum Membran Matrisi (BMM) ile kaplanmış tek bir 3 boyutlu insan bağırsak organoidlerinin dönüştürülmesi adımlarını açıklar. Çoğu hat, kuyu başına yaklaşık 400.000 hücre içerecektir. İkinci, transdüksiyonsuz kuyu kontrol olarak dahil edilmelidir. Tüm reaktifleri ve hücre süspansiyonlarını buz üzerinde tutun.
- Son geçişten 2-5 gün sonra, bakım WRNE ortamını iki HIO kuyusundan çıkarın. Kuyucuk başına 300 μL% 0.05 Tripsin-EDTA ile değiştirin ve BMM'yi plakadan ayırmak için 5 kez yukarı ve aşağı hafifçe pipetleyin. 37 °C'lik bir inkübatörde 4 dakika bekletin.
- Oyuk başına 500 μL CMGF- +% 10 FBS ekleyin. 1 mL FBS'yi 2 kez yukarı ve aşağı pipetleyerek 1 mL düşük bağlanan pipet ucunu FBS ile önceden kaplayın. FBS, birden fazla uçta yeniden kullanılabilir. Önceden kaplanmış uçla HIO'ları 10 kat yukarı ve aşağı pipetleyin.
- Her kuyucuğun içeriğini kendi önceden kaplanmış 1.5 mL mikrosantrifüj tüpüne aktarın. Her birini ilave 500 μL CMGF- ile iyice durulayın ve yıkamayı ilgili tüpe ekleyin.
- Tüpleri 100 °C'de 5 dakika boyunca sallanan bir kova santrifüjünde 4 x g'da santrifüjleyin. Süpernatanı ve kalan BMM'yi çıkarın.
- 1 mL 1x PBS'de tekrar süspanse edin. Her tüpü toplam 4 tüp için iki mikrosantrifüj tüpüne bölün. Tüpleri 100 x g'da 4 °C'de 5 dakika santrifüjleyin. Süpernatanı çıkarın.
- Her tüpü 1x PBS'de bir kez daha tekrar süspanse edin ve 4 °C'de 5 dakika boyunca 100 x g'da santrifüjleyin.
- 400 μL transdüksiyon ortamı ve kontrol ortamı hazırlayın (Tablo 1). Kontrol ortamının 200 μL'sinde 2 tüpü ve transdüksiyon ortamının 200 μL'sinde 2 tüpü yeniden süspanse edin.
- 37 °C'lik bir hücre kültürü inkübatöründe 24 saat inkübe edin. Düzgün transdüksiyonu teşvik etmek için kaplanmış bir uçla periyodik olarak (örn. transdüksiyondan 2 saat sonra (hpt), 12 hpt, 18 hpt) yukarı ve aşağı pipetleyerek yeniden süspanse edin.
- 24 saat sonra, tüpleri 4 ° C'de 5 dakika boyunca 100 x g'de santrifüjleyin. Süpernatanı çıkarın.
- Yıkamak için peleti 500 μL 1x PBS'de yeniden süspanse edin. 4 °C'de 5 dakika boyunca 100 x g'da santrifüjleyin. Süpernatanı çıkarın.
- Buz gibi 200 μL'lik bir pipet ucu kullanarak, 4 peletin her birini 30 μL BMM'de yeniden süspanse edin. Eşit şekilde dağıtmak için hafifçe yukarı ve aşağı pipetleyin.
- Her tüpün içeriğini 24 oyuklu bir plaka üzerinde kendi kuyucuğuna yerleştirin. 500 μL HighWnt WRNE + 10μM Y-27632 eklemeden önce BMM'nin katılaşmasına izin vermek için 37 °C'de 10 dakika inkübe edin.
- Dönüştürülen HIO'ların 1 hafta boyunca büyümesine izin verin, ortamı (HighWnt WRNE + 10 μM Y-27632) her gün yenileyin. 1 hafta sonra, floresan indikatörün ifadesini mikroskopi ile kontrol edin. Sinyal güçlüyse, ilaç seçimine başlayın. Sinyal zayıfsa, iletimi yukarıda açıklandığı gibi tekrarlayın.
- Hat kurulduktan sonra, agonist tedavisi ile floresan göstergesinin işlevini doğrulayın. 100nM ADP, GCaMP doğrulaması için güvenilir bir agonisttir. Aşağıda açıklandığı gibi ilk doğrulama için 3D organoidleri test edin.
- Bir geçişten sonra, BMM'de bir kuyu (veya birden fazla) organoidi ayrı bir görüntüleme alt plakası üzerine yerleştirin. Görüntüleme sırasında aşırı örtüşmeyi önlemek için organoidleri normal yoğunluğun yaklaşık 1 / 3'ünde plakalayın. Steriliteyi sağlamak zor olduğundan, agonist tedaviyi takiben bu HIO'ları geçmeye devam etmeyin.
- HIO'ların geçiş sonrası iyileşmesi için 2-3 gün bekleyin. Görüntülemeden önce, ortamı fenol kırmızısı içermeyen farklılaşma ortamına geçirin.
- Bir floresan mikroskobu kullanarak, 488 nm uyarma ve bir FITC/GFP filtre seti kullanarak her 5 saniyede bir elde edilen görüntülerle 3 dakikalık bir çalışma ayarlayın. 30 saniyelik görüntülemeden sonra, 100 nM ADP veya araç kontrolü ekleyin. Sinyal taban çizgisine yakın olana kadar görüntülemeye devam edin, ~2 dakika. ADP tedavisi ile GCaMP floresansında geçici, ~2 kat artış, başarılı transdüksiyon ve biyosensör fonksiyonunu gösterir. Transdüksiyon verimliliğinin daha kesin bir tahmini için, bölüm 3'te açıklanan işlemle oluşturulan tek katmanları kullanarak görüntüleme ile agonist testini tekrarlayın.
3. Canlı floresan görüntüleme için HIO tek katmanlarının hazırlanması
- 10 oyuklu görüntüleme alt haznesi lamının tüm kuyucuklarını Kollajen IV ile kaplayın. Bunu yapmak için 34 μL 1 mg/mL Kollajen IV'ü 960 μL steril deiyonize su ile karıştırın. Her bir oyuğa 95 μL seyreltilmiş kollajen IV çözeltisi ekleyin ve 37 ° C'de 0.5-2 saat inkübe edin.
- WRNE bakım ortamını 4 kuyu 3D HIO'dan çıkarın. HIO'lar son geçişlerinden itibaren 5-7 gün olmalıdır.
NOT: 1 10 oyuklu plaka tipik olarak ~ 1.25 x 106 hücre gerektirir. Bu tipik olarak, her biri 30 μL BMM ile kaplanmış 2-4 kuyucuklu 3D HIO gerektirir, ancak yoğunluğa bağlı olarak değişecektir. - Kuyucuk başına 500 μL 1x PBS + 0.5 mM EDTA ekleyin. Önceden kaplanmış 1 mL'lik bir uçla, BMM'yi plakadan ayırmak için hafifçe yukarı ve aşağı pipetleyin. Süspansiyonu önceden kaplanmış 15 mL'lik konik bir tüpe aktarın ve benzer kuyucukları aynı tüpte birleştirin.
- Her bir kuyuyu ilave 500 μL PBS + 0,5 mM EDTA ile durulayın. 4 °C'de 5 dakika boyunca 300 x g'da santrifüjleyin. Süpernatan ve artık BMM'yi çıkarın.
- Önceden kaplanmış bir uç kullanarak, kalan peleti 3 mL PBS + 5 mM EDTA içinde yeniden süspanse edin (bunun ilk yıkamadan 10 kat daha fazla EDTA olduğunu unutmayın).
- 4 °C'de 5 dakika boyunca 300 x g'da santrifüjleyin. Süpernatan ve artık BMM'yi çıkarın. Peletin 2 mL enzimatik ayrışma tamponunda yeniden süspanse edilmesi.
- 37 °C boncuk/su banyosunda 5 dakika inkübe edin. 3 mL CMGF- + %10 FBS ekleyin ve karıştırmak için hafifçe pipetleyin.
- 4 °C'de 5 dakika boyunca 300 x g'da santrifüjleyin. Süpernatanı çıkarın. 1 mL CMGF- ekleyin.
- HIO'ları mekanik olarak tek hücrelere ayırmak için önceden kaplanmış bir 80x-100x uçla kuvvetlice yukarı ve aşağı pipetleyin. 4 °C'de 5 dakika boyunca 300 x g'da santrifüjleyin. Süpernatanı çıkarın.
- 1 mL WRNE + 10 μM Y-27632 içinde yeniden süspanse edin. 1 mL süspansiyon için bir hücre sayısı elde edin.
- 1.25 x 105 hücre/100 μL (1.25 x 10 6 hücre/mL) konsantrasyon elde etmek için hücre süspansiyonunu WRNE + 10μMY-27632 ile seyreltin.
- 200 μL'lik bir pipet kullanarak, kollajen çözeltisini 1. adımda hazırlanan plakadan çıkarın, çünkü kollajen şimdi kuyunun dibine yerleşmiştir. Pipet ucuyla kuyunun dibine dokunmaktan kaçının.
- Önceden kaplanmış 200 μL'lik bir pipet ucu kullanarak, oyuk başına 3.11. adımdan (1.25 x 105 hücre) 100 μL hücre çözeltisi ekleyin.
- 37 °C'lik bir hücre kültürü inkübatöründe 24 saat inkübe edin. 24 saat sonra, kültür ortamını tüm oyuklardan çıkarın ve oyuk başına 100 μL farklılaşma ortamı ile değiştirin.
NOT: Bu noktada, hücreler plakaya yapışmış olmalıdır. Tek katmanın büyük olasılıkla birleşik veya tamamen düzlemsel olmayacağını unutmayın. - Slaytı 37 °C hücre kültürü inkübatörüne geri yerleştirin. Farklılaşma ortamını, tek katman birleşene kadar her 24 saatte bir yenileyin. Bu genellikle kaplamadan 3-5 gün gerektirir. Bu noktadan sonra, tek katmanlar sonraki uygulamalar için hazırdır.
4. HIO tek katmanlarının viral enfeksiyonu
- Virüs aşısını hazırlayın. Gerekirse, tripsin virüs stoğunu aktive eder. Rotavirüs için, virüs stoklarına 10 μg / mL Worthington's Trypsin ekleyin ve 37 ° C'de 1 saat inkübe edin.
- Etkinleştirilmiş virüs stoğunu aşağıdaki iki yöntemden birini kullanarak seyreltin.
- Sadece bir viral suş kullanarak maksimum enfekte hücre sayısını hedefliyorsanız, 50 μL aktive edilmiş viral stoğu 50 μL CMGF- ile karıştırın.
- Birden fazla suş karşılaştırıldığında, eşdeğer enfeksiyon çoklukları (MOI'ler) elde etmek için aşıları hazırlayın. İstenen MOI için gerekli olan kuyu başına virüs stoğu hacmini belirlemek için aşağıdaki formülü kullanın. Kuyu başına 100 μL'lik bir nihai hacim elde etmek için hesaplanan virüs stoğu hacmine CMGF- ekleyin.
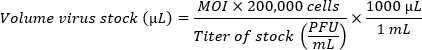
NOT: 5.46 mm çapında (96 oyuklu plaka) bir kuyuya kaplanmış tek bir HIO tek tabakası yaklaşık 200.000 hücre içerir. Tek tabakalarda enfeksiyon etkinliğinin düşük olması nedeniyle, yüksek MOI (hücre başına 100-1 milyon viral partikül) tercih edilir. Optimal MOI ampirik olarak belirlenmelidir.
- 200 mL'lik bir pipet kullanarak ortamı HIO tek katmanından nazikçe çıkararak tek katmanları enfekte edin. 100 mL virüs aşısı veya sahte aşı ile değiştirin.
- Çoğu rotavirüs stoğu MA104 hücrelerinde çoğaltıldığından, sahte aşılama için enfekte olmamış bir MA104 hücre lizatı kullanın. Enfekte kuyucuklar için kullanılan virüs stoğunun hacmine eşdeğer bir hacim kullanın ve kuyucuk başına 100 mL'lik bir son hacim için CMGF- ekleyin.
- Rotavirüs14 gibi hücreleri bazolateral olarak enfekte eden virüsler için, tek tabakayı puanlamak için 25G'lik bir iğne kullanın. Tek katmanın uzunluğu boyunca, kuyunun altından üstüne kadar tek bir puan yapmak en kolay yoldur. İğneyi, eğimli tarafı yukarı bakacak şekilde, hareket yönünün tersine bir açıyla tek katmana hafifçe bastırın ve bir yara izi oluşturmak için tek katmanın uzunluğu boyunca sürükleyin (Şekil 2A).
- 37 °C'lik bir inkübatörde 2 saat inkübe edin. Virüsü çıkarın ve sahte aşılama. Tek katmanları 1x PBS ile bir kez yıkayın.
- 100 μL fenol kırmızısı içermeyen farklılaştırma ortamı ekleyin. Slaytı görüntülemeye hazır olana kadar 37 °C'lik bir inkübatöre yerleştirin. Optimal görüntüleme penceresi, viral enfeksiyonun kinetiğine bağlı olarak değişecektir. Rotavirüs için, enfeksiyondan 6-8 saat sonra görüntülemeye başlayın.
- Etkinleştirilmiş virüs stoğunu aşağıdaki iki yöntemden birini kullanarak seyreltin.
5. Ca2+ Enfekte tek tabakaların görüntülenmesi
- Sahne üstü inkübatörü önceden 37 °C'ye ısıtın. Nemlendirilmiş CO2'yi 0,02 L/dk'da çalıştırın. Enfekte olmuş tek katmanlarla birlikte slaytı inkübatör odasına yerleştirin ve kapağı kapatın.
- Parlak alan (BF) aydınlatması ve 20x objektif kullanarak, görüntülenecek tek katman içindeki görüş alanlarının X ve Y koordinatlarını seçin. Enfekte olmuş hücrelerin çoğu çiziğe yakın olacağından, puanlanan bölge göz önünde olacak şekilde noktaları seçmek en iyisidir.
- Görüntüleme parametrelerini optimize edin ve 488 nm uyarma kullanarak bir görüntü elde edin. Fototoksisiteyi en aza indirmek için ışık kaynağını 50 ms pozlama süresiyle %50 güce ayarlayın. Uygun alım parametreleri değişebilir ve birden fazla alım alınarak optimize edilmelidir. Hiçbir pikselin doygun olmadığından emin olun. Floresan kalsiyum sensörünün tüm dinamik aralığının algılanabilmesini sağlamak için pozlama süresini ve ışık gücünü gerektiği gibi ayarlayın.
- Diğer floroforlar (örneğin, floresan etiketli virüsler) kullanıyorsanız, bu kanallarda optimizasyonu tekrarlayın. Görüntüleme döngüsünü ayarlayın ve 1 dakikalık bir döngüde seçilen her X, Y koordinatında 488 nm'lik bir görüntü elde edin. Bu döngüyü sürekli olarak görüntüleyin. Floresan etiketli bir virüs kullanırken, her 10. döngüde bir uygun kanalda bir görüntü elde edin (yani, 1 görüntü/10 dakika).
- ~ 18 saat boyunca görüntüleri toplayın. Floresan etiketi olmayan virüsler kullanıyorsanız, tek katmanları düzeltin ve enfekte olmuş hücreleri tanımlamak için immünofloresan boyama yapın. Bunu, aşağıda açıklandığı gibi görüntüleme çalıştırmasını tamamladıktan sonra gerçekleştirin.
- Görüntüleme ortamını çıkarın ve 1x PBS'de 100 μL %4 formaldehit ile değiştirin. Oda sıcaklığında 30 dakika inkübe edin.
- Sabitleyiciyi çıkarın ve 100 μL 50mM NH4Cl ile 10 dakika söndürün. NH4Cl'yi çıkarın. Tek katmanları 1x PBS'de 100 μL% 0.1 Triton X-100 ile oda sıcaklığında 1 saat veya gece boyunca 4 °C'de geçirgenleştirin.
- Geçirgenlik tamponunu çıkarın. 1 saat boyunca 1x PBS'de 100 μL% 3 sığır serum albümini ile bloklayın.
- Engelleme çözümünü çıkarın. 1x PBS'de önerilen/optimize edilmiş konsantrasyona seyreltilmiş birincil antikorlar ekleyin. Kuyucuk başına 100 μL antikor çözeltisi ekleyin.
- Gece boyunca 4 °C'de hafifçe sallayarak inkübe edin. Birincil antikorları çıkarın. 1x PBS ile 3x yıkayın.
- 1x PBS'de önerilen/optimize edilmiş konsantrasyona seyreltilmiş floresan konjuge ikincil antikorlar ekleyin. Oyuk başına 100 μL ekleyin. Oda sıcaklığında 2 saat inkübe edin.
NOT: FPBase15 , çoğullama sırasında taşmayı önleyecek ikincilleri seçmeye yardımcı olacak harika bir kaynaktır. İkincillerin çoğu, 1x PBS'de 1:1000 seyreltmede etkilidir. - İkincil antikorları çıkarın. 1x PBS'de 1 μg/mL DAPI çözeltisi yapın ve oyuk başına 100 μL ekleyin. 20 dakika inkübe edin. DAPI'yi çıkarın. 1x PBS ile 3x yıkayın.
- Görüntüleme için sabit tek katmanları 100 μL 1x PBS'de tutun. Slaytı mikroskop tablasına geri yerleştirin, canlı görüntüleme çalışmasından X ve Y koordinatlarını yeniden yükleyin ve boyama için kullanılan ikincil antikora karşılık gelen kanaldaki her çok noktayı görüntüleyin. Aynı noktaların yakalandığından emin olmak için canlı görüntüleme çalışmasından elde edilen görüntülere bakın.
6. Hücreler arası kalsiyum dalgalarının kantitasyonu
- Fiji'nin Just ImageJ (FIJI) olduğundan emin olun ve aşağıdaki eklentilerle birlikte yüklenmiştir: Bio-Formats (FIJI'ye önceden yüklenmiş olmalıdır ancak ImageJ'de olmayacaktır); Yemek Kitabı: Yüklemek için FIJI menüsünden Yardım > Güncellemeleri > Güncelleme sitelerini yönet'e gidin. Yemek Kitabını Kontrol Edin. FIJI'yi yeniden başlatın.
- Canlı görüntüleme çalışmasından gelen veri dosyasını, her X, Y koordinatını ayırarak birden çok dosyaya bölün. Nikon NES Elements'te, Dosya > İçe/Dışa Aktar > Çoklu Noktaları Böl'ü seçin. Dışa aktarmak için bir klasör ve uygun bir önek seçin.
- FIJI'yi açın. Bölünmüş çoklu noktalardan tek bir dosya yükleyin. Çok kanallı bir görüntü kullanıyorsanız, kanalları bölün. 488 nm kanalından (GCaMP) görüntülerin bulunduğu pencereyi seçin.
- Her piksel değerindeki değişimi bir zaman noktasından diğerine belirlemek için DeltaF Up işlevini çalıştırın. Bunu yapmak için, Delta F Up > Yemek Kitabı > T-fonksiyonlarını seçin.
- Kalsiyum dalgasına sahip bir görüntü bulunana kadar yığında ilerleyin. Kalsiyum dalgası görüntülenirken, Eşiği Ayarla > Görüntü > Ayarla'ya tıklayarak bir eşik ayarlayın. Eşiği geçmesini sağlayan dalganın parçası olmayan sinyal miktarını en aza indirmek için alt sınırı ayarlayın. Yukarıda açıklanan parametreler kullanılarak elde edilen 16 bit görüntüler için, 600 gibi daha düşük bir eşikle başlayın ve gerektiği gibi ayarlayın.
- Parçacıkları Analiz Et > Analiz Et'e tıklayarak dalgaları segmentlere ayırmak için partikül analizörünü çalıştırın. Aralığı ayarlayarak minimum dalga boyutunu μm2 olarak ayarlayın. Başlangıçta 0-sonsuz olarak ayarlanır. 20x objektif kullanılarak elde edilen MA104 hücrelerinin görüntüleri için, alt sınırı 10.000μm2'ye yükselterek başlayın ve uygun şekilde ayarlayın.
- Sonuçları görüntüle ve Sonuçları temizle kutularını işaretleyin. "Göster" açılır menüsünden Anahatlar'ı seçin ve ardından Tamam'ı tıklayın. Bu, 2 çıkışla sonuçlanmalıdır: Bir pencere, Sonuçlar, algılanan her dalgayı, dalganın alanını ve ortalama, minimum ve maksimum yoğunluk değerlerini (delta F'ye göre) listeleyecektir. [dosya adı] çizimi başlıklı diğer pencere, parçalı dalgaların ana hatlarını içeren yeni bir yığın içerecektir. Dalga algılamayı doğrulamak için bunu kullanın. Gerekirse eşik değerlerini ve boyut aralığını ayarlayın ve dalga çağrılarını yeniden kontrol edin.
- Toplu işleme için, birlikte verilen makro komut dosyasını kullanın (Ek Kodlama Dosyası 1). Bunu kullanmak için aşağıda açıklanan adımları izleyin.
- Birden çok noktayı adım 6.2'de açıklandığı gibi tek dosyalara bölün. FIJI'yi açın. Toplu > Makro> İşlem'i seçin. "Giriş" kutusunda, bölünmüş çok noktalı dosyaları içeren klasörün haritasını sağlayın. "Çıktı" kutusunu boş bırakın.
- Komut dosyasını Ek Kodlama Dosyası 1'den kutuya yapıştırın. Koddaki yoğunluk eşiğini ve boyut eşiğini, 6.5 ve 6.6 adımlarındaki en iyileştirilmiş parametreleri yansıtacak şekilde ayarlayın.
- İşlem'e tıklayın. Çoklu noktaların sayısına ve bilgisayarın işlem hızına bağlı olarak, tüm görüntülerin işlenmesi 30 dakika veya daha uzun sürebilir. Tamamlandığında, veriler Masaüstünde "Yazdıktan sonra beni yeniden adlandır" adlı yeni bir elektronik tablo dosyasına yazılacaktır. Çıktı dosyası, her çok noktalı için bir dalga sayısı ve dalga ölçümleri (alan, ortalama yoğunluk, minimum ve maksimum yoğunluk ve yoğunluk yoğunluğu) içermelidir.
Sonuçlar
Şekil 1A , GCaMP6'ları stabil bir şekilde ifade etmek için dönüştürülmüş 3 boyutlu insan bağırsak organoidlerini içeren bir BMM kubbesini göstermektedir. Şekil 1B , tohumlamadan 24, 48 ve 72 saat sonra tek tabaka olarak yeniden kaplanan aynı organoid hattını göstermektedir. GCaMP6'ların işlevini doğrulamak için, tek tabaka 4 dakika boyunca her 2 saniyede bir floresan mikroskobu ile görüntülendi ve ~20 saniye sonra ortama 100 nM ADP ekl...
Tartışmalar
Sitozolik Ca2+ seviyelerindeki değişiklikler,epitel 10,16,17 içindeki patolojilerin hem nedeni hem de sonucu olabilir. Sitozolik kalsiyumdaki artışlar, kalsiyuma bağımlı klorür kanalının aktivasyonu yoluyla doğrudan sekresyona neden olabilirTMEM16A 18,19. Ca2+'ya yanıt olarak TMEM16A aktivasyonu, apikal klorür akışına izin vererek, ...
Açıklamalar
Yazarların ifşa etmek için rekabet eden mali çıkarları yoktur.
Teşekkürler
Bu çalışma, Ulusal Sağlık Enstitüleri'nden (NIH) R01DK115507 ve R01AI158683 (PI: J. M. Hyser) hibeleri ile desteklenmiştir. Stajyer desteği, NIH hibe F30DK131828 (PI: J.T. Gebert), F31DK132942 (PI: F.J. Scribano) ve F32DK130288 (PI: K.A. Engevik) tarafından sağlandı. Organoid bakım ortamını sağladığı için Texas Tıp Merkezi Sindirim Hastalıkları Enteroid Çekirdeğine teşekkür ederiz.
Malzemeler
| Name | Company | Catalog Number | Comments |
| Advanced DMEM F12 | Gibco | 12634028 | |
| [Leu15]-Gastrin I | Sigma-Aldrich | G9145 | |
| 0.05% Trypsin EDTA | Gibco | 25300054 | |
| 0.05% Trypsin EDTA | Gibco | 25300054 | |
| 1.5mL microcentrifuge tubes | Fisherbrand | 5408137 | |
| 15mL conical tubes | Thermofisher Scientific | 0553859A | |
| 16% formaldehyde | Thermofisher Scientific | 28906 | |
| 1M HEPES | Gibco | 15630080 | |
| 1M HEPES | Gibco | 15630080 | |
| 1X PBS | Corning | 21-040-CV | |
| 25 gauge needle | Thermofisher Scientific | 1482113D | |
| A-83-01 | Tocris | 2939 | |
| ADP | Sigma-Aldrich | A2754 | |
| Advanced DMEM F12 | Gibco | 12634028 | |
| Antibiotic-antimycocytic | Gibco | 15240062 | |
| Antibiotic-antimycotic | Gibco | 15240062 | |
| B27 Supplement | Gibco | 17504-044 | |
| Bovine serum albumin | FisherScientific | BP1600100 | |
| CellView Cell Culture Slide, PS, 75/25 MM, Glass Bottom, 10 compartments | Greiner | 543979 | |
| Collagen IV | Sigma Aldrich | C5533 | |
| DAPI | Thermofisher Scientific | D1306 | |
| EDTA | Corning | 46-034-CI | |
| Fetal bovine serum | Corning | 35010CV | |
| Fetal bovine serum | Corning | 35010CV | |
| Fluorobrite | Gibco | A1896701 | |
| GlutaMAX | Gibco | 35050079 | |
| GlutaMAX | Gibco | 35050079 | |
| Human epidermal growth factor | ProteinTech | HZ-1326 | |
| Lentivirus | VectorBuilder | (variable) | |
| Matrigel | BD Biosceicen | 356231/CB40230C | |
| N2 Supplement | Gibco | 17502-048 | |
| N-acetylcysteine | Sigma-Aldrich | A9165-5G | |
| NH4Cl | Sigma-Aldrich | A9434 | |
| Nicotinamide | Sigma-Aldrich | N0636 | |
| Nunc Cell Culture Treated 24-well Plates | Thermofisher Scientific | 142475 | |
| Polybrene | MilliporeSigma | TR1003G | |
| SB202190 | Sigma-Aldrich | S70767 | |
| Triton X-100 | Fisher BioReagents | BP151100 | |
| TrypLE Express Enzyme, no phenol red | Thermofisher Scientific | 12604013 | |
| Trypsin | Worthington Biochemical | NC9811754 | |
| Y-27632 | Tocris | 1254 |
Referanslar
- Bootman, M. D., Bultynck, G. Fundamentals of cellular calcium signaling: A primer. Cold Spring Harb Perspect Biol. 12 (1), a038802 (2020).
- Clapham, D. E. Calcium signaling. Cell. 131 (6), 1047-1058 (2007).
- Danese, A., et al. Cell death as a result of calcium signaling modulation: A cancer-centric prospective. Biochim Biophys Acta Mol Cell Res. 1868 (8), 119061 (2021).
- Harr, M. W., Distelhorst, C. W. Apoptosis and autophagy: Decoding calcium signals that mediate life or death. Cold Spring Harb Perspect Biol. 2 (10), a005579 (2010).
- Barak, P., Parekh, A. B. Signaling through Ca2+ microdomains from store-operated CRAC channels. Cold Spring Harb Perspect Biol. 12 (7), a035097 (2020).
- Nakai, J., Ohkura, M., Imoto, K. A high signal-to-noise Ca(2+) probe composed of a single green fluorescent protein. Nat Biotechnol. 19 (2), 137-141 (2001).
- Erofeev, A. I., Vinokurov, E. K., Vlasova, O. L., Bezprozvanny, I. B. GCaMP, a family of single-fluorophore genetically encoded calcium indicators. J Evol Biochem Phys. 59 (4), 1195-1214 (2023).
- Suzuki, J., Kanemaru, K., Iino, M. Genetically encoded fluorescent indicators for organellar calcium imaging. Biophys J. 111 (6), 1119-1131 (2016).
- Nászai, M., Cordero, J. B. Intestinal stem cells: Got calcium. Curr Biol. 26 (3), R117-R119 (2016).
- Barrett, K. E. Calcium-mediated chloride secretion in the intestinal epithelium: Significance and regulation. Curr Top Membr. 53, 257-282 (2002).
- Xu, J., et al. Calcium-sensing receptor regulates intestinal dipeptide absorption via Ca2+ signaling and IKCa activation. Physiol Rep. 8 (1), e14337 (2020).
- Clevers, H. Modeling development and disease with organoids. Cell. 165 (7), 1586-1597 (2016).
- Lin, S. C., Haga, K., Zeng, X. L., Estes, M. K. Generation of CRISPR–Cas9-mediated genetic knockout human intestinal tissue–derived enteroid lines by lentivirus transduction and single-cell cloning. Nat Protoc. 17 (4), 1004-127 (2022).
- Crawford, S. E., Ramani, S., Blutt, S. E., Estes, M. K. Organoids to dissect gastrointestinal virus-host interactions: What have we learned. Viruses. 13 (6), 999 (2021).
- Lambert, T. J. FPbase: a community-editable fluorescent protein database. Nat Methods. 16 (4), 277-278 (2019).
- Lai, Y., et al. Inhibition of calcium-triggered secretion by hydrocarbon-stapled peptides. Nature. 603 (7903), 949-956 (2022).
- Chang-Graham, A. L., et al. Rotavirus induces intercellular calcium waves through ADP signaling. Science. 370 (6519), eabc3621 (2020).
- Lee, B., et al. Anoctamin 1/TMEM16A controls intestinal Cl− secretion induced by carbachol and cholera toxin. Exp Mol Med. 51 (8), 1-14 (2019).
- Saha, T., et al. Intestinal TMEM16A control luminal chloride secretion in a NHERF1 dependent manner. Biochem Biophys Rep. 25, 100912 (2021).
- Mroz, M. S., Keely, S. J. Epidermal growth factor chronically upregulates Ca2+-dependent Cl− conductance and TMEM16A expression in intestinal epithelial cells. J Physiol. 590 (8), 1907-1920 (2012).
- Sui, J., et al. Dual role of Ca2+-activated Cl− channel transmembrane member 16A in lipopolysaccharide-induced intestinal epithelial barrier dysfunction in vitro. Cell Death Dis. 11 (5), 404 (2020).
- Bellono, N. W., et al. Enterochromaffin cells are gut chemosensors that couple to sensory neural pathways. Cell. 170 (1), 185-198.e16 (2017).
- Paradis, T., Bègue, H., Basmaciyan, L., Dalle, F., Bon, F. Tight junctions as a key for pathogens invasion in intestinal epithelial cells. Int J Mol Sci. 22 (5), 2506 (2021).
- Samak, G., et al. Calcium/Ask1/MKK7/JNK2/c-Src signalling cascade mediates disruption of intestinal epithelial tight junctions by dextran sulfate sodium. Biochem J. 465 (3), 503-515 (2015).
- Deng, H., Gerencser, A. A., Jasper, H. Signal integration by Ca2+ regulates intestinal stem cell activity. Nature. 528 (7581), 212-217 (2015).
- Saurav, S., Tanwar, J., Ahuja, K., Motiani, R. K. Dysregulation of host cell calcium signaling during viral infections: Emerging paradigm with high clinical relevance. Mol Aspects Med. 81, 101004 (2021).
- Chang-Graham, A. L., et al. Rotavirus calcium dysregulation manifests as dynamic calcium signaling in the cytoplasm and endoplasmic reticulum. Sci Rep. 9 (1), 10822 (2019).
- Hyser, J. M., Collinson-Pautz, M. R., Utama, B., Estes, M. K. Rotavirus disrupts calcium homeostasis by NSP4 viroporin activity. mBio. 1 (5), e00265-e00310 (2010).
- Pham, T., Perry, J. L., Dosey, T. L., Delcour, A. H., Hyser, J. M. The Rotavirus NSP4 viroporin domain is a calcium-conducting ion channel. Sci Rep. 7, 43487 (2017).
- Crawford, S. E., Hyser, J. M., Utama, B., Estes, M. K. Autophagy hijacked through viroporin-activated calcium/calmodulin-dependent kinase kinase-β signaling is required for rotavirus replication. Proc Natl Acad Sci U S A. 109 (50), E3405-E3413 (2012).
- Crawford, S. E., Criglar, J. M., Liu, Z., Broughman, J. R., Estes, M. K. COPII vesicle transport is required for Rotavirus NSP4 interaction with the autophagy protein LC3 II and trafficking to viroplasms. J Virol. 94 (1), e01341 (2019).
- Pando, V., Iša, P., Arias, C. F., Ló Pez, S. Influence of calcium on the early steps of Rotavirus infection. Virology. 295 (1), 190-200 (2002).
- Hyser, J. M., Estes, M. K. Pathophysiological consequences of calcium-conducting viroporins. Annu Rev Virol. 2 (1), 473-496 (2015).
- Strtak, A. C., et al. Recovirus NS1-2 has viroporin activity that induces aberrant cellular calcium signaling to facilitate virus replication. mSphere. 4 (5), e00506-e00519 (2019).
- In, J. G., Foulke-Abel, J., Clarke, E., Kovbasnjuk, O. Human colonoid monolayers to study interactions between pathogens, commensals, and host intestinal epithelium. J Vis Exp. (146), 59357 (2019).
- Hirota, A., AlMusawi, S., Nateri, A. S., Ordóñez-Morán, P., Imajo, M. Biomaterials for intestinal organoid technology and personalized disease modeling. Acta Biomater. 132, 272-287 (2021).
- Cevallos Porta, D., López, S., Arias, C. F., Isa, P. Polarized rotavirus entry and release from differentiated small intestinal cells. Virology. 499, 65-71 (2016).
- Mirabelli, C., et al. Human Norovirus efficiently replicates in differentiated 3D-human intestinal enteroids. J Virol. 96 (22), e0085522 (2022).
- Icha, J., Weber, M., Waters, J. C., Norden, C. Phototoxicity in live fluorescence microscopy, and how to avoid it. Bioessays. 39 (8), 28749075 (2017).
- Li, J., et al. Engineering of NEMO as calcium indicators with large dynamics and high sensitivity. Nat Methods. 20 (6), 918-924 (2023).
Yeniden Basımlar ve İzinler
Bu JoVE makalesinin metnini veya resimlerini yeniden kullanma izni talebi
Izin talebiDaha Fazla Makale Keşfet
This article has been published
Video Coming Soon
JoVE Hakkında
Telif Hakkı © 2020 MyJove Corporation. Tüm hakları saklıdır