Для просмотра этого контента требуется подписка на Jove Войдите в систему или начните бесплатную пробную версию.
Method Article
Визуализация кальция в режиме реального времени инфицированных вирусом монослоев органоидов кишечника человека с использованием генетически кодируемых кальциевых индикаторов
В этой статье
Резюме
Этот протокол описывает подход к выполнению визуализации кальция в инфицированных вирусом органоидах кишечника человека и предлагает подход к анализу.
Аннотация
Кальциевая сигнализация является неотъемлемым регулятором практически каждой ткани. В кишечном эпителии кальций участвует в регуляции секреторной активности, динамики актина, воспалительных реакций, пролиферации стволовых клеток и многих других нехарактерных клеточных функций. Таким образом, картирование динамики передачи сигналов кальция в кишечном эпителии может дать представление о гомеостатических клеточных процессах и выявить уникальные реакции на различные стимулы. Органоиды кишечника человека (HIO) представляют собой высокопроизводительную модель для изучения кишечного эпителия и, таким образом, представляют собой полезную систему для исследования динамики кальция. В данной статье описывается протокол для стабильной трансдукции HIO с генетически кодируемыми кальциевыми индикаторами (GECI), выполнения флуоресцентной микроскопии в реальном времени и анализа данных визуализации для осмысленной характеристики сигналов кальция. В качестве репрезентативного примера можно привести 3-мерные HIO, трансдуцированные лентивирусом для стабильной экспрессии GCaMP6s, цитозольного GECI на основе зеленого флуоресцентного белка. Затем сконструированные HIO были диспергированы в одноклеточную суспензию и высеяны в виде монослоев. После дифференцировки монослои HIO инфицировали ротавирусом и/или лечили препаратами, которые, как известно, стимулируют кальциевый ответ. Эпифлуоресцентный микроскоп, оснащенный камерой для визуализации в реальном времени с регулируемой температурой, позволил получить долгосрочную визуализацию инфицированных или обработанных лекарственными препаратами монослоев. После получения изображений полученные изображения были проанализированы с помощью свободно доступного аналитического программного обеспечения ImageJ. В целом, эта работа создает адаптируемый конвейер для характеристики клеточной сигнализации в HIO.
Введение
Кальций является широко консервативным вторичным мессенджером, который играет важнейшую роль в регуляции клеточной физиологии1. Учитывая его сильный заряд, небольшой размер и высокую растворимость в физиологических условиях, кальций является идеальным манипулятором конформации белка. Это делает кальций мощным средством для преобразования электрохимических сигналов в ферментативные, транскрипционные или посттранскрипционные изменения. Строгие градиенты концентрации кальция в эндоплазматическом ретикулуме (ER) и плазматических мембранах создают высокую движущую силу, которая позволяет быстро изменять концентрацию цитозольного кальция. Многочисленные механизмы, включая буферизацию и активный транспорт, жестко поддерживают этот градиент. Несмотря на то, что это необходимо для нормального функционирования клеток, это поддержание энергетически затратно, что делает его особенно восприимчивым в стрессовых состояниях.
Таким образом, дисрегуляция кальция в цитозоле является почти универсальным сигналом многих видов клеточного стресса. Метаболические нарушения, токсины, болезнетворные микроорганизмы, механические повреждения и генетические нарушения могут нарушить передачу сигналов кальция. Независимо от стимула, на уровне всей клетки устойчивое, неконтролируемое повышение цитозольного кальция может способствовать апоптозу и, в конечномитоге, некрозу. Однако изменения уровня кальция в цитозолье с более низкой амплитудой или более высокой частотой имеют различные эффекты2. Аналогичным образом, результаты флуктуаций кальция могут различаться в зависимости от пространственного микродомена, в которомони происходят. Таким образом, мониторинг уровня кальция может дать представление о динамических сигнальных процессах, но для этого требуется отбор проб с относительно высоким временным и пространственным разрешением.
Генетически кодируемые индикаторы кальция (GECI) являются мощными инструментами для непрерывного отбора проб в системах живых клеток6. Одними из наиболее широко используемых GECI являются кальций-чувствительные флуоресцентные белки на основе GFP, известные как GCaMPs7. Канонический GCaMP представляет собой слияние трех различных белковых доменов: циркулярно перестановочного GFP (cpGFP), кальмодулина и M136. Домен кальмодулина претерпевает конформационные изменения при связывании кальция, что позволяет ему взаимодействовать с М13. Взаимодействие кальмодулина и М13 индуцирует конформационное изменение cpGFP, которое увеличивает его флуоресцентное излучение при возбуждении. Таким образом, увеличение концентрации кальция коррелирует с увеличением интенсивности флуоресценции GCaMP. Эти сенсоры могут быть цитозольными или нацеленными на конкретные органеллы8.
Как и большинство тканей, кальций регулирует различные функции в эпителии желудочно-кишечного тракта. Кишечный эпителий является неотъемлемой частью всасывания питательных веществ и жидкости, но также должен образовывать плотный барьер и иммунный интерфейс, чтобы избежать инвазии патогенов или токсического раздражения. Кальций-зависимые пути влияют почти на все эти жизненно важные функции 9,10,11. Тем не менее, передача сигналов кальция в кишечном эпителии остается малоизученным рубежом с многообещающим потенциалом в качестве терапевтической мишени. В то время как мониторинг динамики кальция в кишечном эпителии in vivo по-прежнему представляет собой проблему, кишечные органоиды человека (HIO) предлагают адаптируемую систему ex vivo для экспериментов12. HIO представляют собой трехмерные (3D) сфероиды, полученные из стволовых клеток кишечника человека и при дифференцировке повторяют большую часть клеточного разнообразия нативного кишечного эпителия12.
Этот протокол описывает комплексные методы разработки HIO, экспрессирующих GECI, а затем подготовки сконструированных HIO в виде монослоев для визуализации живых клеток кальция. В качестве примера патологической манипуляции, которая нарушает передачу сигналов кальция, предлагается вирусная инфекция, а также аналитический подход к количественной оценке этих изменений.
протокол
Все органоиды кишечника человека (HIO), использованные в этом протоколе и репрезентативных экспериментах, были получены из тканей человека, полученных и поддерживаемых Техасским медицинским центром по заболеваниям пищеварительной системы. Все образцы были собраны в соответствии с протоколом, утвержденным Институциональным наблюдательным советом Медицинского колледжа Бэйлора.
1. Подготовка материалов и реагентов
- Для поддержания органоидов соберите обработанные клеточной культурой 24-луночные планшеты, матрицу базальной мембраны (BMM), конические пробирки объемом 15 мл и конические пробирки объемом 1,5 мл.
- Для приготовления полной среды без факторов роста (CMGF-) к 500 мл продвинутого DMEM F12 добавляют 5 мл 1M HEPES, 5 мл 100-кратного антибиотик-антимикотика, 5 мл 100-кратной добавки глютамина.
- Для приготовления Wnt-, R-спондин- и Noggin-содержащих (WRNE) сред смешайте равные части сред, кондиционированных CMGF и Wnt, добавьте среду, кондиционированную Noggin, 10% по объему, R-спондин-кондиционированную среду, 20% по объему, 50 нг/мл эпидермального фактора роста человека, 10 мМ никотинамида, 10 нМ [Leu15]-гастрина I, 500 нМ A-83-01, 10 мкМ SB202190, 1 добавку B27, 1 добавку N2 и 1 мМ N-ацетилцистеина.
- Для лентивирусной трансдукции приготовьте фетальную бычью сыворотку (FBS), 0,05% трипсин-ЭДТА в 1x фосфатно-солевом буфере (PBS), CMGF- с 10% FBS, стерильный 1x PBS, полибрен, 10 мкМ Y-27632, лентивирус, высокий Wnt WRNE + 10 мкМ Y-27632
- Для генерации органоидных монослоев готовят предметное стекло на дне 10-луночной клеточной культуры, FBS, коллаген IV (1 мг/мл в деионизированном (di)H2O), матрицу базальной мембраны, 0,5 мМ ЭДТА в 1x PBS, 5 мМ ЭДТА в 1x PBS, буфер ферментативной диссоциации, CMGF- с 10% FBS, WRNE + 10 мкМ Y-27632.
- Для вирусной инфекции монослоев органоидов приготовьте трипсин, ротавирусную массу, иглу CMGF-, 25G, стерильную 1x PBS и свободную от фенола красную дифференцировочную среду.
- Для приготовления дифференцировочных сред без фенольного красного берут 500 мл клеточных культуральных сред, не содержащих фенолового красного, добавляют 5 мл заменимых аминокислот 100x MEM, 5 мл 100-глютамина, 5 мл пирувата натрия 100 нМ и 7,5 мл 1M HEPES.
- Для иммунофлуоресцентного окрашивания органоидов готовят 4% формальдегид (16% формальдегид, разбавленный в 1х PBS), Triton X-100 (0,1% Triton X-100 в 1x PBS), бычий сывороточный альбумин (3% бычий сывороточный альбумин в 1x PBS), раствор NH4Cl (50 мМ), DAPI (1 мкг/мл раствор DAPI в 1x PBS).
2. Разработка органоидов для экспрессии генетически закодированных кальциевых сенсоров
ПРИМЕЧАНИЕ: В этом протоколе описаны этапы трансдукции одной лунки 3-мерных органоидов кишечника человека, покрытых 30 мкл матрицы базальной мембраны (BMM) на 24-луночном планшете13. Большинство линий будут содержать около 400 000 ячеек на скважину. Вторая, нетрансдуцированная скважина должна быть включена в качестве контрольной. Храните все реагенты и клеточные суспензии на льду.
- Через 2-5 суток после последнего прохода удалить поддерживающую среду WRNE из двух скважин HIO. Замените на 300 мкл 0,05% трипсина-ЭДТА на лунку и осторожно пипеткой вверх и вниз 5 раз, чтобы отсоединить BMM от планшета. Поместить в инкубатор при температуре 37 °C на 4 минуты.
- Добавьте 500 мкл CMGF- + 10% FBS на лунку. Предварительно покрыте наконечник для пипетки с низким связыванием 1 мл FBS, пипетируя 1 мл FBS вверх и вниз 2 раза. FBS можно повторно использовать на нескольких наконечниках. С помощью наконечника с предварительно покрытым пипеткой поднимайте и опускайте HIO вверх и вниз 10 раз.
- Перелейте содержимое каждой лунки в собственную предварительно покрытую пробирку для микроцентрифуг объемом 1,5 мл. Промойте каждую лунку дополнительным 500 мкл CMGF- и добавьте промывку в соответствующую пробирку.
- Центрифугируйте пробирки при давлении 100 x g в центрифуге с качающимся ковшом в течение 5 минут при 4 °C. Удалите надосадочную жидкость и все остатки BMM.
- Ресуспендировать в 1 мл 1x PBS. Разделите каждую пробирку на две пробирки для микроцентрифуги, чтобы получить в общей сложности 4 пробирки. Центрифугируйте пробирки при 100 x g в течение 5 мин при 4 °C. Удалите надосадочную жидкость.
- Повторно суспендируйте каждую пробирку еще один раз в 1x PBS и центрифугируйте при 100 x g в течение 5 минут при 4 °C.
- Приготовьте 400 мкл трансдукционной среды и контрольной среды (табл. 1). Ресуспендировать 2 пробирки в 200 мкл контрольной среды и 2 пробирки в 200 мкл трансдукционной среды.
- Инкубируют в инкубаторе клеточных культур при температуре 37 °C в течение 24 ч. Ресуспендирование путем периодического пипетирования вверх и вниз наконечником с покрытием (например, через 2 ч после трансдукции (hpt), 12 hpt, 18 hpt), чтобы обеспечить равномерную трансдукцию.
- Через 24 ч центрифужные пробирки при 100 x g в течение 5 мин при 4 °C. Удалите надосадочную жидкость.
- Ресуспендируйте гранулы в 500 мкл 1x PBS для стирки. Центрифуга при 100 x g в течение 5 мин при 4 °C. Удалите надосадочную жидкость.
- Используя ледяной наконечник дозатора объемом 200 мкл, повторно суспендируйте каждую из 4 гранул в 30 мкл BMM. Осторожно поднимите и опустите пипетку вверх и вниз, чтобы равномерно распределиться.
- Выложите содержимое каждой пробирки в отдельную лунку на 24-луночной пластине. Инкубируйте в течение 10 минут при 37 °C, чтобы BMM затвердел, прежде чем добавлять 500 мкл HighWnt WRNE + 10 мкМ Y-27632.
- Дайте трансдуцированным HIO расти в течение 1 недели, обновляя носитель (HighWnt WRNE + 10 мкМ Y-27632) через день. Через 1 неделю проверить экспрессию флуоресцентного индикатора с помощью микроскопии. Если сигнал сильный, начинайте подбор препарата. Если сигнал слабый, повторите трансдукцию, как описано выше.
- После того, как линия установлена, проверьте функцию индикатора флуоресценции с помощью лечения агонистами. 100 нМ АДФ является надежным агонистом для валидации GCaMP. Протестируйте 3D-органоиды для первоначальной проверки, как описано ниже.
- После прохода поместите лунку (или несколько) органоидов в ТММ на отдельную пластину с изображением дна. Нанесите органоиды примерно на 1/3 от нормальной плотности, чтобы избежать чрезмерного перекрытия при визуализации. Не следует продолжать прохождение этих HIO после лечения агонистами, так как трудно обеспечить стерильность.
- Подождите 2-3 дня, чтобы HIO восстановились после прохождения. Перед визуализацией переключите среду на дифференцирующую среду, не содержащую фенолового красителя.
- Используя флуоресцентный микроскоп, настройте 3-минутный прогон с изображениями, полученными каждые 5 с, с использованием возбуждения 488 нм и набора фильтров FITC/GFP. Через 30 секунд визуализации добавьте 100 нм ADP или управление транспортным средством. Продолжайте визуализацию до тех пор, пока сигнал не вернется к почти базовому уровню, ~2 мин. Преходящее, ~2-кратное увеличение флуоресценции GCaMP при лечении АДФ указывает на успешную трансдукцию и функцию биосенсора. Для более точной оценки эффективности трансдукции повторите тест агонистов с визуализацией с использованием монослоев, полученных с помощью процесса, описанного в части 3.
3. Подготовка монослоев HIO для флуоресцентной визуализации в реальном времени
- Покройте все лунки 10-луночного предметного стекла нижней камеры визуализации коллагеном внутривенно. Для этого смешайте 34 мкл 1 мг/мл коллагена внутривенно с 960 мкл стерильной деионизированной воды. Добавьте 95 мкл разбавленного раствора коллагена внутривенно в каждую лунку и инкубируйте при 37 °C в течение 0,5-2 ч.
- Удалите среду обслуживания WRNE из 4 лунок 3D HIO. HIO должны пройти 5-7 дней с момента их последнего прохождения.
ПРИМЕЧАНИЕ: 1 10-луночная пластина обычно требует ~1,25 x 106 ячеек. Обычно для этого требуется 2-4 лунки 3D HIO, покрытые 30 мкл BMM каждая, но это зависит от плотности. - Добавьте 500 мкл 1x PBS + 0,5 мМ ЭДТА на лунку. С предварительно покрытым наконечником объемом 1 мл осторожно поднимайте и опускайте пипетку, чтобы отсоединить BMM от планшета. Переложите суспензию в коническую пробирку объемом 15 мл с предварительно покрытым покрытием, соединив подобные лунки в одну и ту же пробирку.
- Промойте каждую лунку дополнительными 500 мкл PBS + 0,5 мМ ЭДТА. Центрифуга при 300 x g в течение 5 мин при 4 °C. Удалите надосадочную жидкость и остаточную БММ.
- Используя предварительно покрытый наконечник, повторно суспендируйте оставшуюся гранулу в 3 мл PBS + 5 мМ ЭДТА (обратите внимание, что это в 10 раз больше ЭДТА, чем при первой стирке).
- Центрифуга при 300 x g в течение 5 мин при 4 °C. Удалите надосадочную жидкость и остаточную БММ. Ресуспендируйте гранулу в 2 мл ферментативного диссоциационного буфера.
- Инкубировать на водяной бане при температуре 37 °C в течение 5 минут. Добавьте 3 мл CMGF- + 10% FBS и аккуратно перемешайте пипеткой.
- Центрифуга при 300 x g в течение 5 мин при 4 °C. Удалите надосадочную жидкость. Добавьте 1 мл ЦМГФ-.
- Энергично пипетка вверх и вниз с предварительно покрытым наконечником 80x-100x для механического дробления HIO на отдельные ячейки. Центрифуга при 300 x g в течение 5 мин при 4 °C. Удалите надосадочную жидкость.
- Ресуспендировать в 1 мл WRNE + 10 мкМ Y-27632. Получите количество клеток для 1 мл суспензии.
- Разбавьте клеточную суспензию WRNE + 10 мкМ Y-27632 до получения концентрации 1,25 x 105 клеток/100 мкл (1,25 x 106 клеток/мл).
- С помощью пипетки на 200 мкл удалите раствор коллагена из планшета, приготовленного на шаге 1, так как коллаген осел на дно лунки. Не прикасайтесь наконечником пипетки ко дну лунки.
- Используя предварительно покрытый наконечник дозатора объемом 200 мкл, добавьте 100 мкл клеточного раствора с шага 3,11 (1,25 x 105 ячеек) на лунку.
- Инкубируют в течение 24 ч в инкубаторе клеточных культур при температуре 37 °C. Через 24 ч извлеките питательную среду из всех лунок и замените ее 100 мкл дифференцирующей среды на лунку.
ПРИМЕЧАНИЕ: К этому моменту ячейки должны прилегать к пластине. Обратите внимание, что монослой, скорее всего, не будет сливающимся или полностью плоским. - Поместите предметное стекло обратно в инкубатор клеточных культур с температурой 37 °C. Обновляйте дифференцирующую среду каждые 24 часа до тех пор, пока монослой не станет сливающимся. Обычно для этого требуется 3-5 дней с момента нанесения покрытия. После этого монослои готовы к последующему применению.
4. Вирусная инфекция монослоев HIO
- Подготовьте вирусный посевной материал. При необходимости трипсин активирует вирусный запас. При ротавирусе добавляют 10 мкг/мл трипсина Уортингтона к запасам вируса и инкубируют при 37 °C в течение 1 ч.
- Разбавьте активированный запас вируса одним из двух следующих способов.
- Если вы стремитесь получить максимальное количество инфицированных клеток, используя только один вирусный штамм, смешайте 50 мкл активированного вирусного запаса с 50 мкл CMGF-.
- При сравнении нескольких штаммов подготавливайте инокулюмы для достижения эквивалентной кратности инфекции (MOIs). Используйте следующую формулу для определения объема запаса вируса на скважину, необходимого для требуемого MOI. Добавьте CMGF- к объему запаса вируса, рассчитанному для достижения конечного объема 100 мкл на лунку.
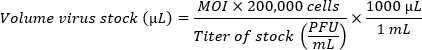
ПРИМЕЧАНИЕ: Один монослой HIO, покрытый в лунке диаметром 5,46 мм (96-луночная пластина), содержит около 200 000 ячеек. Из-за низкой эффективности инфекции в монослоях предпочтение отдается высокому MOI (100-1 млн вирусных частиц на клетку). Оптимальный MOI должен быть определен опытным путем.
- Чтобы заразить монослои, аккуратно удалите материал с монослоя HIO с помощью пипетки объемом 200 мл. Замените 100 мл вирусного посева или фиктивного посева.
- Поскольку большинство ротавирусов размножаются в клетках MA104, используйте неинфицированный клеточный лизат MA104 для имитации посевного материала. Используйте объем, эквивалентный объему запаса вируса, используемого для зараженных лунок, и добавьте CMGF- для получения конечного объема 100 мл на лунку.
- Для вирусов, которые инфицируют клетки базолатерально, таких как ротавирус14, используйте иглу 25G, чтобы надрезать монослой. Проще всего сделать одинарную насечку по всей длине монопласта, от дна до верха лунки. Аккуратно вдавите иглу в монослой под углом скошенной стороной вверх, противоположным направлению движения, и протяните ее по длине монослоя, чтобы создать рубец (рисунок 2А).
- Инкубируют в инкубаторе при температуре 37 °C в течение 2 ч. Удалите вирус и имитируйте посевной материал. Постирайте монослои один раз с помощью 1x PBS.
- Добавьте 100 мкл дифференцирующей среды, не содержащей фенола красного. Поместите предметное стекло в инкубатор с температурой 37 °C до получения изображения. Оптимальное окно визуализации зависит от кинетики вирусной инфекции. При ротавирусной инфекции следует начинать визуализацию через 6-8 ч после заражения.
- Разбавьте активированный запас вируса одним из двух следующих способов.
5. Ca2+ Визуализация инфицированных монослоев
- Предварительно нагрейте инкубатор до 37 °C. Запустите увлажненныйCO2 со скоростью 0,02 л/мин. Поместите предметное стекло с зараженными монослоями в камеру инкубатора и закройте крышкой.
- Используя яркопольную подсветку (BF) и 20-кратный объектив, выберите координаты X и Y полей зрения в пределах монослоя, который будет изображен. Лучше всего выбирать точки с учетом забитой области, так как большинство зараженных клеток будут находиться рядом с царапиной.
- Оптимизируйте параметры визуализации и получите изображение с помощью возбуждения с длиной волны 488 нм. Чтобы свести к минимуму фототоксичность, установите источник света на мощность 50% с временем экспозиции 50 мс. Соответствующие параметры сбора данных могут различаться и должны быть оптимизированы путем получения нескольких приобретений. Убедитесь, что пиксели не насыщены. При необходимости отрегулируйте время экспозиции и мощность света, чтобы обеспечить обнаружение всего динамического диапазона флуоресцентного кальциевого датчика.
- При использовании других флуорофоров (например, флуоресцентно меченных вирусов) повторите оптимизацию по этим каналам. Настройте контур визуализации и получите изображение с длиной волны 488 нм в каждой выбранной координате X, Y за 1 минуту. Изображайте этот цикл непрерывно. При использовании флуоресцентно помеченного вируса каждые 10циклов (т.е. 1 изображение в течение 10 минут) следует получать изображение по соответствующему каналу.
- Собирайте изображения в течение ~18 часов. При использовании вирусов без флуоресцентных меток фиксируйте монослои и выполняйте иммунофлуоресцентное окрашивание для выявления инфицированных клеток. Выполните это после завершения работы с образами, как описано ниже.
- Извлеките носитель изображения и замените его 100 мкл 4% формальдегида в 1 PBS. Выдерживают при комнатной температуре 30 мин.
- Удалить фиксатор и закалить 100 мкл 50 мМ NH4Cl в течение 10 мин. Удалить NH4Cl. Пермеабилизацию монослоев 100 мкл 0,1% Triton X-100 в 1x PBS в течение 1 ч при комнатной температуре или в течение ночи при 4 °C.
- Удалите буфер пермеабилизации. Блок со 100 мкл 3% бычьего сывороточного альбумина в 1x PBS в течение 1 ч.
- Удалите блокирующий раствор. Добавьте первичные антитела, разбавленные до рекомендуемой/оптимизированной концентрации в 1x PBS. Добавьте 100 мкл раствора антител на лунку.
- Инкубируйте в течение ночи при температуре 4 °C с легким покачиванием. Удалить первичные антитела. Стирайте 3 раза с 1 PBS.
- Добавьте флуоресцентно-конъюгированные вторичные антитела, разбавленные до рекомендуемой/оптимизированной концентрации в 1x PBS. Добавьте 100 мкл на лунку. Выдерживают 2 ч при комнатной температуре.
ПРИМЕЧАНИЕ: FPBase15 является отличным ресурсом, помогающим выбрать вторичные реплики, которые позволят избежать протекания при мультиплексировании. Большинство вторичных препаратов эффективны при разведении 1:1000 в 1x PBS. - Удалить вторичные антитела. Приготовьте раствор DAPI 1 мкг/мл в 1x PBS и добавьте 100 мкл на лунку. Инкубировать 20 мин. Удалить DAPI. Стирайте 3 раза с 1 PBS.
- Храните фиксированные монослои в 100 мкл 1x PBS для визуализации. Поместите предметное стекло обратно на столик микроскопа, перезагрузите координаты X и Y из цикла визуализации в реальном времени и визуализируйте каждую многоточечную точку на канале, который соответствует вторичному антителу, которое использовалось для окрашивания. Ссылайтесь на изображения из прогона изображений в реальном времени, чтобы убедиться, что захвачены одни и те же точки.
6. Количественное определение межклеточных кальциевых волн
- Убедитесь, что Fiji is Just ImageJ (FIJI) установлен со следующими плагинами: Bio-Formats (должен быть предустановлен в FIJI, но не будет в ImageJ); Поваренная книга: Для установки в меню FIJI перейдите в раздел «Справка» > «Обновления» > «Управление сайтами обновлений». Проверьте поваренную книгу. Перезапустите FIJI.
- Разделите файл данных из динамического образа на несколько файлов, разделив все координаты X, Y. В Nikon NES Elements выберите «Файл» > «Импорт/экспорт» > «Разделить мультиточки». Выберите папку для экспорта и соответствующий префикс.
- Откройте ФИДЖИ. Загрузите один файл из разделенных мультиточек. Если используется многоканальное изображение, разделите каналы. Выберите окно с изображениями из канала 488 нм (GCaMP).
- Запустите функцию DeltaF Up, чтобы определить изменение значения каждого пикселя от одной временной точки к другой. Для этого выберите Cookbook > T-функции > Delta F Up.
- Прокручивайте стопку, пока не найдете изображение с кальциевой волной. Увидев кальциевую волну, установите пороговое значение, щелкнув Изображение > Настроить > Порог. Отрегулируйте нижнюю границу, чтобы свести к минимуму количество сигнала, который не является частью волны, прошедшей пороговое значение. Для 16-битных изображений, полученных с использованием параметров, описанных выше, начните с нижнего порога, равного 600, и корректируйте его по мере необходимости.
- Запустите анализатор частиц для сегментации волн, нажав кнопку Анализ > Анализ частиц. Установите минимальный размер волны вмкм2 , отрегулировав диапазон. Для него установлено значение 0-бесконечность на базовом уровне. Для изображений клеток MA104, полученных с помощью 20-кратного объектива, начните с увеличения нижнего предела до 10 000мкм2 и скорректируйте его соответствующим образом.
- Установите флажки Отображать результаты и Очистить результаты. В раскрывающемся меню «Показать» выберите «Контуры» и нажмите кнопку «ОК». В результате должно получиться 2 выходных файла: В одном окне, Results, будут перечислены все обнаруженные волны, площадь волны, а также ее среднее, минимальное и максимальное значения интенсивности (на основе дельты F). В другом окне, озаглавленном Drawing of [file name], появится новый стек с контурами сегментированных волн. Используйте это для проверки обнаружения волн. При необходимости отрегулируйте пороговые значения и диапазон размеров, а также перепроверьте вызовы волн.
- Для пакетной обработки используйте прилагаемый сценарий макроса (Дополнительный файл кодирования 1). Чтобы использовать это, выполните действия, описанные ниже.
- Разделите многоточечные файлы на отдельные файлы, как описано в шаге 6.2. Откройте ФИДЖИ. Выберите Обработать > Пакетная > макрос. В поле "input" укажите карту папки, содержащей разделенные мультиточечные файлы. Оставьте поле "output" пустым.
- Вставьте скрипт из Дополнительного файла кодирования 1 в коробку. Настройте порог интенсивности и порог размера в сценарии, чтобы отразить оптимизированные параметры из шагов 6.5 и 6.6.
- Нажмите кнопку Обработать. В зависимости от количества мультиточек и скорости обработки компьютера, обработка всех изображений может занять 30 минут или больше. После завершения данные будут записаны в новый файл электронной таблицы под названием «Переименовать меня после записи» на рабочем столе. Выходной файл должен содержать, для каждой мультиточки, количество волн и волновые метрики (площадь, средняя интенсивность, минимальная и максимальная интенсивность, а также плотность интенсивности).
Результаты
На рисунке 1А показан купол BMM, содержащий 3-мерные органоиды кишечника человека, которые были трансдуцированы для стабильной экспрессии GCaMP6. На рисунке 1B показана та же линия органоида, покрытая в виде монослоя через 24, 48 и 72 ч после посева. Для валидации ф...
Обсуждение
Изменения цитозольного уровняCa2+ могут быть как причиной, так и следствием патологий в эпителии 10,16,17. Увеличение цитозольного кальция может напрямую стимулировать секрецию через активацию кальций-зависимого хлоридного канала TM...
Раскрытие информации
У авторов нет конкурирующих финансовых интересов, которые они могли бы раскрыть.
Благодарности
Эта работа была поддержана грантами R01DK115507 и R01AI158683 (PI: J. M. Hyser) от Национальных институтов здравоохранения (NIH). Поддержка стажеров была обеспечена грантами NIH F30DK131828 (PI: J.T. Gebert), F31DK132942 (PI: F.J. Scribano) и F32DK130288 (PI: K.A. Engevik). Мы хотели бы поблагодарить Texas Medical Center Digestive Diseases Enteroid Core за предоставление среды для поддержания органоидов.
Материалы
| Name | Company | Catalog Number | Comments |
| Advanced DMEM F12 | Gibco | 12634028 | |
| [Leu15]-Gastrin I | Sigma-Aldrich | G9145 | |
| 0.05% Trypsin EDTA | Gibco | 25300054 | |
| 0.05% Trypsin EDTA | Gibco | 25300054 | |
| 1.5mL microcentrifuge tubes | Fisherbrand | 5408137 | |
| 15mL conical tubes | Thermofisher Scientific | 0553859A | |
| 16% formaldehyde | Thermofisher Scientific | 28906 | |
| 1M HEPES | Gibco | 15630080 | |
| 1M HEPES | Gibco | 15630080 | |
| 1X PBS | Corning | 21-040-CV | |
| 25 gauge needle | Thermofisher Scientific | 1482113D | |
| A-83-01 | Tocris | 2939 | |
| ADP | Sigma-Aldrich | A2754 | |
| Advanced DMEM F12 | Gibco | 12634028 | |
| Antibiotic-antimycocytic | Gibco | 15240062 | |
| Antibiotic-antimycotic | Gibco | 15240062 | |
| B27 Supplement | Gibco | 17504-044 | |
| Bovine serum albumin | FisherScientific | BP1600100 | |
| CellView Cell Culture Slide, PS, 75/25 MM, Glass Bottom, 10 compartments | Greiner | 543979 | |
| Collagen IV | Sigma Aldrich | C5533 | |
| DAPI | Thermofisher Scientific | D1306 | |
| EDTA | Corning | 46-034-CI | |
| Fetal bovine serum | Corning | 35010CV | |
| Fetal bovine serum | Corning | 35010CV | |
| Fluorobrite | Gibco | A1896701 | |
| GlutaMAX | Gibco | 35050079 | |
| GlutaMAX | Gibco | 35050079 | |
| Human epidermal growth factor | ProteinTech | HZ-1326 | |
| Lentivirus | VectorBuilder | (variable) | |
| Matrigel | BD Biosceicen | 356231/CB40230C | |
| N2 Supplement | Gibco | 17502-048 | |
| N-acetylcysteine | Sigma-Aldrich | A9165-5G | |
| NH4Cl | Sigma-Aldrich | A9434 | |
| Nicotinamide | Sigma-Aldrich | N0636 | |
| Nunc Cell Culture Treated 24-well Plates | Thermofisher Scientific | 142475 | |
| Polybrene | MilliporeSigma | TR1003G | |
| SB202190 | Sigma-Aldrich | S70767 | |
| Triton X-100 | Fisher BioReagents | BP151100 | |
| TrypLE Express Enzyme, no phenol red | Thermofisher Scientific | 12604013 | |
| Trypsin | Worthington Biochemical | NC9811754 | |
| Y-27632 | Tocris | 1254 |
Ссылки
- Bootman, M. D., Bultynck, G. Fundamentals of cellular calcium signaling: A primer. Cold Spring Harb Perspect Biol. 12 (1), a038802 (2020).
- Clapham, D. E. Calcium signaling. Cell. 131 (6), 1047-1058 (2007).
- Danese, A., et al. Cell death as a result of calcium signaling modulation: A cancer-centric prospective. Biochim Biophys Acta Mol Cell Res. 1868 (8), 119061 (2021).
- Harr, M. W., Distelhorst, C. W. Apoptosis and autophagy: Decoding calcium signals that mediate life or death. Cold Spring Harb Perspect Biol. 2 (10), a005579 (2010).
- Barak, P., Parekh, A. B. Signaling through Ca2+ microdomains from store-operated CRAC channels. Cold Spring Harb Perspect Biol. 12 (7), a035097 (2020).
- Nakai, J., Ohkura, M., Imoto, K. A high signal-to-noise Ca(2+) probe composed of a single green fluorescent protein. Nat Biotechnol. 19 (2), 137-141 (2001).
- Erofeev, A. I., Vinokurov, E. K., Vlasova, O. L., Bezprozvanny, I. B. GCaMP, a family of single-fluorophore genetically encoded calcium indicators. J Evol Biochem Phys. 59 (4), 1195-1214 (2023).
- Suzuki, J., Kanemaru, K., Iino, M. Genetically encoded fluorescent indicators for organellar calcium imaging. Biophys J. 111 (6), 1119-1131 (2016).
- Nászai, M., Cordero, J. B. Intestinal stem cells: Got calcium. Curr Biol. 26 (3), R117-R119 (2016).
- Barrett, K. E. Calcium-mediated chloride secretion in the intestinal epithelium: Significance and regulation. Curr Top Membr. 53, 257-282 (2002).
- Xu, J., et al. Calcium-sensing receptor regulates intestinal dipeptide absorption via Ca2+ signaling and IKCa activation. Physiol Rep. 8 (1), e14337 (2020).
- Clevers, H. Modeling development and disease with organoids. Cell. 165 (7), 1586-1597 (2016).
- Lin, S. C., Haga, K., Zeng, X. L., Estes, M. K. Generation of CRISPR–Cas9-mediated genetic knockout human intestinal tissue–derived enteroid lines by lentivirus transduction and single-cell cloning. Nat Protoc. 17 (4), 1004-127 (2022).
- Crawford, S. E., Ramani, S., Blutt, S. E., Estes, M. K. Organoids to dissect gastrointestinal virus-host interactions: What have we learned. Viruses. 13 (6), 999 (2021).
- Lambert, T. J. FPbase: a community-editable fluorescent protein database. Nat Methods. 16 (4), 277-278 (2019).
- Lai, Y., et al. Inhibition of calcium-triggered secretion by hydrocarbon-stapled peptides. Nature. 603 (7903), 949-956 (2022).
- Chang-Graham, A. L., et al. Rotavirus induces intercellular calcium waves through ADP signaling. Science. 370 (6519), eabc3621 (2020).
- Lee, B., et al. Anoctamin 1/TMEM16A controls intestinal Cl− secretion induced by carbachol and cholera toxin. Exp Mol Med. 51 (8), 1-14 (2019).
- Saha, T., et al. Intestinal TMEM16A control luminal chloride secretion in a NHERF1 dependent manner. Biochem Biophys Rep. 25, 100912 (2021).
- Mroz, M. S., Keely, S. J. Epidermal growth factor chronically upregulates Ca2+-dependent Cl− conductance and TMEM16A expression in intestinal epithelial cells. J Physiol. 590 (8), 1907-1920 (2012).
- Sui, J., et al. Dual role of Ca2+-activated Cl− channel transmembrane member 16A in lipopolysaccharide-induced intestinal epithelial barrier dysfunction in vitro. Cell Death Dis. 11 (5), 404 (2020).
- Bellono, N. W., et al. Enterochromaffin cells are gut chemosensors that couple to sensory neural pathways. Cell. 170 (1), 185-198.e16 (2017).
- Paradis, T., Bègue, H., Basmaciyan, L., Dalle, F., Bon, F. Tight junctions as a key for pathogens invasion in intestinal epithelial cells. Int J Mol Sci. 22 (5), 2506 (2021).
- Samak, G., et al. Calcium/Ask1/MKK7/JNK2/c-Src signalling cascade mediates disruption of intestinal epithelial tight junctions by dextran sulfate sodium. Biochem J. 465 (3), 503-515 (2015).
- Deng, H., Gerencser, A. A., Jasper, H. Signal integration by Ca2+ regulates intestinal stem cell activity. Nature. 528 (7581), 212-217 (2015).
- Saurav, S., Tanwar, J., Ahuja, K., Motiani, R. K. Dysregulation of host cell calcium signaling during viral infections: Emerging paradigm with high clinical relevance. Mol Aspects Med. 81, 101004 (2021).
- Chang-Graham, A. L., et al. Rotavirus calcium dysregulation manifests as dynamic calcium signaling in the cytoplasm and endoplasmic reticulum. Sci Rep. 9 (1), 10822 (2019).
- Hyser, J. M., Collinson-Pautz, M. R., Utama, B., Estes, M. K. Rotavirus disrupts calcium homeostasis by NSP4 viroporin activity. mBio. 1 (5), e00265-e00310 (2010).
- Pham, T., Perry, J. L., Dosey, T. L., Delcour, A. H., Hyser, J. M. The Rotavirus NSP4 viroporin domain is a calcium-conducting ion channel. Sci Rep. 7, 43487 (2017).
- Crawford, S. E., Hyser, J. M., Utama, B., Estes, M. K. Autophagy hijacked through viroporin-activated calcium/calmodulin-dependent kinase kinase-β signaling is required for rotavirus replication. Proc Natl Acad Sci U S A. 109 (50), E3405-E3413 (2012).
- Crawford, S. E., Criglar, J. M., Liu, Z., Broughman, J. R., Estes, M. K. COPII vesicle transport is required for Rotavirus NSP4 interaction with the autophagy protein LC3 II and trafficking to viroplasms. J Virol. 94 (1), e01341 (2019).
- Pando, V., Iša, P., Arias, C. F., Ló Pez, S. Influence of calcium on the early steps of Rotavirus infection. Virology. 295 (1), 190-200 (2002).
- Hyser, J. M., Estes, M. K. Pathophysiological consequences of calcium-conducting viroporins. Annu Rev Virol. 2 (1), 473-496 (2015).
- Strtak, A. C., et al. Recovirus NS1-2 has viroporin activity that induces aberrant cellular calcium signaling to facilitate virus replication. mSphere. 4 (5), e00506-e00519 (2019).
- In, J. G., Foulke-Abel, J., Clarke, E., Kovbasnjuk, O. Human colonoid monolayers to study interactions between pathogens, commensals, and host intestinal epithelium. J Vis Exp. (146), 59357 (2019).
- Hirota, A., AlMusawi, S., Nateri, A. S., Ordóñez-Morán, P., Imajo, M. Biomaterials for intestinal organoid technology and personalized disease modeling. Acta Biomater. 132, 272-287 (2021).
- Cevallos Porta, D., López, S., Arias, C. F., Isa, P. Polarized rotavirus entry and release from differentiated small intestinal cells. Virology. 499, 65-71 (2016).
- Mirabelli, C., et al. Human Norovirus efficiently replicates in differentiated 3D-human intestinal enteroids. J Virol. 96 (22), e0085522 (2022).
- Icha, J., Weber, M., Waters, J. C., Norden, C. Phototoxicity in live fluorescence microscopy, and how to avoid it. Bioessays. 39 (8), 28749075 (2017).
- Li, J., et al. Engineering of NEMO as calcium indicators with large dynamics and high sensitivity. Nat Methods. 20 (6), 918-924 (2023).
Перепечатки и разрешения
Запросить разрешение на использование текста или рисунков этого JoVE статьи
Запросить разрешениеThis article has been published
Video Coming Soon
Авторские права © 2025 MyJoVE Corporation. Все права защищены