È necessario avere un abbonamento a JoVE per visualizzare questo. Accedi o inizia la tua prova gratuita.
Method Article
Imaging del calcio vivo di monostrati di organoidi intestinali umani infettati da virus utilizzando indicatori di calcio geneticamente codificati
In questo articolo
Riepilogo
Questo protocollo descrive un approccio per l'esecuzione dell'imaging del calcio in organoidi intestinali umani infettati da virus e offre un approccio all'analisi.
Abstract
La segnalazione del calcio è un regolatore integrale di quasi tutti i tessuti. All'interno dell'epitelio intestinale, il calcio è coinvolto nella regolazione dell'attività secretoria, nella dinamica dell'actina, nelle risposte infiammatorie, nella proliferazione delle cellule staminali e in molte altre funzioni cellulari non caratterizzate. Pertanto, la mappatura delle dinamiche di segnalazione del calcio all'interno dell'epitelio intestinale può fornire informazioni sui processi cellulari omeostatici e svelare risposte uniche a vari stimoli. Gli organoidi intestinali umani (HIO) sono un modello ad alto rendimento, derivato dall'uomo, per studiare l'epitelio intestinale e rappresentano quindi un sistema utile per studiare la dinamica del calcio. Questo documento descrive un protocollo per trasdurre stabilmente HIO con indicatori di calcio geneticamente codificati (GECI), eseguire microscopia a fluorescenza dal vivo e analizzare i dati di imaging per caratterizzare in modo significativo i segnali di calcio. Come esempio rappresentativo, gli HIO tridimensionali sono stati trasdotti con lentivirus per esprimere stabilmente GCaMP6s, un GECI citosolico basato su proteine fluorescenti verdi. Gli HIO ingegnerizzati sono stati poi dispersi in una sospensione a cellula singola e seminati come monostrati. Dopo la differenziazione, i monostrati HIO sono stati infettati con rotavirus e/o trattati con farmaci noti per stimolare una risposta al calcio. Un microscopio a epifluorescenza dotato di una camera di imaging dal vivo umidificata a temperatura controllata ha consentito l'imaging a lungo termine di monostrati infetti o trattati con farmaci. Dopo l'imaging, le immagini acquisite sono state analizzate utilizzando il software di analisi ImageJ, disponibile gratuitamente. Nel complesso, questo lavoro stabilisce una pipeline adattabile per la caratterizzazione della segnalazione cellulare negli HIO.
Introduzione
Il calcio è un secondo messaggero ampiamente conservato che svolge un ruolo fondamentale nella regolazione della fisiologia cellulare1. Data la sua forte carica, le piccole dimensioni e l'elevata solubilità in condizioni fisiologiche, il calcio è un manipolatore ideale della conformazione proteica. Ciò rende il calcio un potente mezzo per trasdurre i segnali elettrochimici in alterazioni enzimatiche, trascrizionali o post-trascrizionali. I rigidi gradienti di concentrazione di calcio attraverso il reticolo endoplasmatico (ER) e le membrane plasmatiche creano un'elevata forza motrice che consente rapidi cambiamenti nella concentrazione citosolica di calcio. Diversi meccanismi, tra cui sia il buffering che il trasporto attivo, mantengono strettamente questo gradiente. Sebbene sia necessaria per le normali funzioni cellulari, questa manutenzione è energeticamente costosa, il che la rende particolarmente suscettibile negli stati di stress 2.
In quanto tale, la disregolazione del calcio all'interno del citosol è un segnale quasi universale di molti tipi di stress cellulare. Disturbi metabolici, tossine, agenti patogeni, danni meccanici e perturbazioni genetiche possono interrompere la segnalazione del calcio. Indipendentemente dallo stimolo, a livello di cellula intera, aumenti sostenuti e incontrollati del calcio citosolico possono promuovere l'apoptosi e infine la necrosi 3,4. Alterazioni dei livelli citosolici di calcio di minore ampiezza o di maggiore frequenza, tuttavia, hanno effetti variabili2. Allo stesso modo, gli esiti delle fluttuazioni del calcio possono differire in base al microdominio spaziale in cui si verificano5. Il monitoraggio dei livelli di calcio può quindi offrire informazioni sui processi di segnalazione dinamica, ma ciò richiede un campionamento con una risoluzione temporale e spaziale relativamente elevata.
Gli indicatori di calcio geneticamente codificati (GECI) sono potenti strumenti per il campionamento continuo in sistemi di cellule vive6. Alcune delle GECI più utilizzate sono proteine fluorescenti reattive al calcio a base di GFP note come GCaMPs7. Il GCaMP canonico è una fusione di tre domini proteici distinti: una GFP permutata circolarmente (cpGFP), calmodulina e M136. Il dominio calmodulina subisce un cambiamento di conformazione al momento del legame con il calcio, permettendone l'interazione con M13. L'interazione calmodulina-M13 induce un cambiamento conformazionale nella cpGFP che aumenta la sua emissione fluorescente durante l'eccitazione. Pertanto, un aumento della concentrazione di calcio è correlato a un aumento dell'intensità della fluorescenza GCaMP. Questi sensori possono essere citosolici o mirati a specifici organelli8.
Simile alla maggior parte dei tessuti, il calcio regola una varietà di funzioni all'interno dell'epitelio gastrointestinale. L'epitelio intestinale è fondamentale per l'assorbimento dei nutrienti e dei liquidi, ma deve anche formare una barriera stretta e un'interfaccia immunitaria per evitare l'invasione di agenti patogeni o insulti tossici. Le vie calcio-dipendenti influenzano quasi tutte queste funzioni vitali 9,10,11. Tuttavia, la segnalazione del calcio all'interno dell'epitelio intestinale rimane una frontiera poco esplorata con un potenziale promettente come bersaglio terapeutico. Mentre il monitoraggio della dinamica del calcio all'interno dell'epitelio intestinale in vivo continua a presentare sfide, gli organoidi intestinali umani (HIO) offrono un sistema ex vivo adattabile per la sperimentazione12. Gli HIO sono sferoidi tridimensionali (3D) derivati da cellule staminali intestinali umane e, una volta differenziati, ricapitolano gran parte della diversità cellulare dell'epitelio intestinale nativo12.
Questo protocollo descrive metodi completi per ingegnerizzare HIO che esprimono GECI e quindi preparare HIO ingegnerizzati come monostrati per l'imaging del calcio su cellule vive. Offre l'infezione virale come esempio di manipolazione patologica che interrompe la segnalazione del calcio e fornisce un approccio analitico per quantificare questi cambiamenti.
Protocollo
Tutti gli organoidi intestinali umani (HIO) utilizzati in questo protocollo e negli esperimenti rappresentativi sono stati derivati da tessuto umano ottenuto e mantenuto dal Texas Medical Center Digestive Diseases Enteroid Core. Tutti i campioni sono stati raccolti in conformità con un protocollo approvato dall'Institutional Review Board del Baylor College of Medicine.
1. Preparazione dei materiali e dei reagenti
- Per il mantenimento degli organoidi, raccogliere piastre a 24 pozzetti trattate con colture cellulari, matrice di membrana basale (BMM), provette coniche da 15 mL e provette coniche da 1,5 mL.
- Per preparare terreni completi senza fattori di crescita (CMGF-), a 500 mL di DMEM F12 avanzato aggiungere 5 mL di 1M HEPES, 5 mL di 100x antibiotico-antimicotico, 5 mL di integratore di glutammina 100x.
- Per preparare terreni contenenti Wnt, R-spondina e Noggin (WRNE), mescolare parti uguali di terreni condizionati da CMGF e Wnt, aggiungere terreno condizionato da Noggin, 10% in volume, terreno condizionato da R-spondina, 20% in volume, 50 ng/mL di fattore di crescita epidermico umano, 10 mM di nicotinamide, 10 nM [Leu15]-Gastrina I, 500 nM A-83-01, 10 μM SB202190, 1x integratore di B27, 1x integratore di N2 e 1 mM di N-acetilcisteina.
- Per la trasduzione lentivirale, preparare Siero fetale bovino (FBS), 0,05% tripsina-EDTA in 1x soluzione salina tamponata con fosfato (PBS), CMGF- con 10% FBS, 1x PBS sterile, Polybrene, 10 μM Y-27632, Lentivirus, High Wnt WRNE + 10 μM Y-27632
- Per la generazione di monostrati di organoidi, preparare un vetrino per coltura cellulare a 10 pozzetti con fondo in vetro, FBS, collagene IV (1 mg/mL in (di)H2O deionizzato), matrice di membrana basale, EDTA 0,5 mM in 1x PBS, 5 mM EDTA in 1x PBS, tampone di dissociazione enzimatica, CMGF- con FBS al 10%, WRNE + 10 μM Y-27632.
- Per l'infezione virale dei monostrati di organoidi, preparare tripsina, rotavirus, CMGF-, ago 25G, PBS sterile 1x e terreni di differenziazione privi di rosso fenolo.
- Per preparare i terreni di differenziazione privi di rosso fenolo, prelevare 500 mL di terreno di coltura cellulare privo di rosso fenolo, aggiungere 5 mL di amminoacidi non essenziali MEM 100x, 5 mL di L-glutammina 100x, 5 mL di piruvato di sodio 100 nM e 7,5 mL di HEPES 1M.
- Per la colorazione in immunofluorescenza degli organoidi, preparare il 4% di formaldeide (16% di formaldeide diluita in 1x PBS), Triton X-100 (0,1% Triton X-100 in 1x PBS), albumina sierica bovina (3% di albumina sierica bovina in 1x PBS), NH4Cl soluzione (50 mM), DAPI (1 μg/mL di soluzione DAPI in 1x PBS).
2. Ingegnerizzazione di organoidi per esprimere sensori di calcio geneticamente codificati
NOTA: Questo protocollo descrive i passaggi per trasdurre un singolo pozzetto di organoidi intestinali umani tridimensionali placcati in 30 μL di matrice di membrana basale (BMM) su una piastra a 24 pozzetti13. La maggior parte delle linee conterrà circa 400.000 cellule per pozzetto. Un secondo pozzetto, non trasdotto, deve essere incluso come controllo. Conservare tutti i reagenti e le sospensioni cellulari in ghiaccio.
- Dopo 2-5 giorni dall'ultimo passaggio, rimuovere il terreno WRNE di mantenimento da due pozzetti di HIO. Sostituire con 300 μL di tripsina-EDTA allo 0,05% per pozzetto e pipettare delicatamente su e giù 5 volte per staccare il BMM dalla piastra. Mettere in incubatrice a 37 °C per 4 min.
- Aggiungere 500 μL di CMGF- + 10% FBS per pozzetto. Pre-rivestire un puntale per pipette a basso legame da 1 mL con FBS pipettando 1 mL di FBS su e giù 2 volte. FBS può essere riutilizzato su più puntali. Con il puntale preverniciato, pipettare gli HIO su e giù 10 volte.
- Trasferire il contenuto di ciascun pozzetto nella propria provetta per microcentrifuga prerivestita da 1,5 mL. Sciacquare ogni pozzetto con altri 500 μL di CMGF- e aggiungere il lavaggio alla rispettiva provetta.
- Centrifugare le provette a 100 x g in una centrifuga a secchiello oscillante per 5 minuti a 4 °C. Rimuovere il surnatante e l'eventuale BMM residuo.
- Risospendere in 1 ml di 1x PBS. Dividere ogni provetta in due provette per microcentrifuga per un totale di 4 provette. Centrifugare le provette a 100 x g per 5 minuti a 4 °C. Rimuovere il surnatante.
- Risospendere ogni provetta un'altra volta in 1x PBS e centrifugare a 100 x g per 5 minuti a 4 °C.
- Preparare 400 μL di mezzo di trasduzione e mezzo di controllo (Tabella 1). Risospendere 2 provette in 200 μL del mezzo di controllo e 2 provette in 200 μL del mezzo di trasduzione.
- Incubare in un incubatore per colture cellulari a 37 °C per 24 ore. Risospendere pipettando periodicamente verso l'alto e verso il basso con un puntale rivestito (ad es. 2 ore dopo la trasduzione (HPT), 12 HPT, 18 HPT) per favorire una trasduzione uniforme.
- Dopo 24 ore, centrifugare le provette a 100 x g per 5 minuti a 4 °C. Rimuovere il surnatante.
- Risospendere il pellet in 500 μL di 1x PBS per lavare. Centrifugare a 100 x g per 5 min a 4 °C. Rimuovere il surnatante.
- Utilizzando un puntale per pipette ghiacciato da 200 μL, risospendere ciascuno dei 4 pellet in 30 μL di BMM. Pipettare delicatamente verso l'alto e verso il basso per disperdersi in modo uniforme.
- Placcare il contenuto di ciascuna provetta nel proprio pozzetto su una piastra da 24 pozzetti. Incubare per 10 minuti a 37 °C per consentire al BMM di solidificarsi prima di aggiungere 500 μL di HighWnt WRNE + 10μM Y-27632.
- Lasciare crescere gli HIO trasdotti per 1 settimana, aggiornando il terreno (HighWnt WRNE + 10 μM Y-27632) a giorni alterni. Dopo 1 settimana, verificare l'espressione dell'indicatore fluorescente al microscopio. Se il segnale è forte, iniziare la selezione del farmaco. Se il segnale è debole, ripetere la trasduzione come descritto sopra.
- Una volta stabilita la linea, verificare la funzione dell'indicatore di fluorescenza tramite trattamento agonista. L'ADP da 100 nM è un agonista affidabile per la convalida GCaMP. Testare gli organoidi 3D per la convalida iniziale come descritto di seguito.
- Dopo un passaggio, placcare un pozzetto (o più di uno) di organoidi in BMM su una piastra inferiore di imaging separata. Placcare gli organoidi a circa 1/3 della densità normale per evitare sovrapposizioni eccessive durante l'imaging. Non continuare a passare questi HIO dopo il trattamento con agonisti, poiché è difficile garantire la sterilità.
- Attendere 2-3 giorni affinché gli HIO si riprendano dopo il passaggio. Prima dell'imaging, passare al terreno di differenziazione privo di rosso fenolo.
- Utilizzando un microscopio a fluorescenza, impostare una corsa di 3 minuti con immagini acquisite ogni 5 s utilizzando un'eccitazione a 488 nm e un set di filtri FITC/GFP. Dopo 30 secondi di imaging, aggiungere 100 nM ADP o controllo del veicolo. Continuare l'imaging fino a quando il segnale non ritorna vicino alla linea di base, ~2 min. Un aumento transitorio, ~2 volte, della fluorescenza GCaMP con il trattamento ADP indica il successo della trasduzione e della funzione del biosensore. Per una stima più precisa dell'efficienza di trasduzione, ripetere il test dell'agonista con l'imaging utilizzando monostrati generati tramite il processo descritto nella parte 3.
3. Preparazione di monostrati HIO per l'imaging in fluorescenza dal vivo
- Rivestire tutti i pozzetti di un vetrino a 10 pozzetti della camera inferiore con Collagen IV. Per fare ciò, mescolare 34 μL di 1 mg/mL di collagene EV con 960 μL di acqua deionizzata sterile. Aggiungere 95 μL di soluzione di collagene IV diluito in ciascun pozzetto e incubare a 37 °C per 0,5-2 ore.
- Rimuovere il fluido di manutenzione WRNE da 4 pozzetti di HIO 3D. Gli HIO dovrebbero essere 5-7 giorni dall'ultimo passaggio.
NOTA: 1 piastra da 10 pozzetti richiede in genere ~1,25 x 106 celle. Ciò richiede in genere 2-4 pozzetti di HIO 3D placcati in 30 μL di BMM ciascuno, ma varierà in base alla densità. - Aggiungere 500 μL di 1x PBS + 0,5 mM di EDTA per pozzetto. Con un puntale prerivestito da 1 mL, pipettare delicatamente verso l'alto e verso il basso per staccare il BMM dalla piastra. Trasferire la sospensione in una provetta conica da 15 mL prerivestita, combinando pozzetti simili nella stessa provetta.
- Risciacquare ogni pozzetto con altri 500 μL di PBS + 0,5 mM di EDTA. Centrifugare a 300 x g per 5 minuti a 4 °C. Rimuovere il surnatante e il BMM residuo.
- Utilizzando una punta preverniciata, risospendere il pellet rimanente in 3 mL di PBS + 5 mM di EDTA (si noti che si tratta di 10 volte più EDTA rispetto al primo lavaggio).
- Centrifugare a 300 x g per 5 minuti a 4 °C. Rimuovere il surnatante e il BMM residuo. Risospendere il pellet in 2 mL di tampone di dissociazione enzimatica.
- Incubare a bagnomaria a 37 °C per 5 min. Aggiungere 3 mL di CMGF- + 10% FBS e pipettare delicatamente per mescolare.
- Centrifugare a 300 x g per 5 minuti a 4 °C. Rimuovere il surnatante. Aggiungere 1 mL di CMGF-.
- Pipettare vigorosamente su e giù con un puntale prerivestito 80x-100x per rompere meccanicamente gli HIO in singole cellule. Centrifugare a 300 x g per 5 minuti a 4 °C. Rimuovere il surnatante.
- Risospendere in 1 mL di WRNE + 10 μM Y-27632. Ottenere un conteggio delle cellule per la sospensione da 1 mL.
- Diluire la sospensione cellulare con WRNE + 10μM Y-27632 per ottenere una concentrazione di 1,25 x 105 cellule/100 μL (1,25 x 106 cellule/mL).
- Utilizzando una pipetta da 200 μL, rimuovere la soluzione di collagene dalla piastra preparata nella fase 1, poiché il collagene si è ora depositato sul fondo del pozzetto. Evitare di toccare il fondo del pozzetto con il puntale della pipetta.
- Utilizzando un puntale per pipetta prerivestito da 200 μL, aggiungere 100 μL della soluzione cellulare del passaggio 3.11 (1,25 x 105 celle) per pozzetto.
- Incubare per 24 ore in un incubatore per colture cellulari a 37 °C. Dopo 24 ore, rimuovere il terreno di coltura da tutti i pozzetti e sostituirlo con 100 μL di terreno di differenziazione per pozzetto.
NOTA: A questo punto, le celle dovrebbero aderire alla piastra. Si noti che il monostrato probabilmente non sarà confluente o completamente planare. - Riposizionare il vetrino nell'incubatore per colture cellulari a 37 °C. Rinfrescare il mezzo di differenziazione ogni 24 ore fino a quando il monostrato è confluente. Questo di solito richiede 3-5 giorni dalla placcatura. A questo punto, i monostrati sono pronti per le applicazioni a valle.
4. Infezione virale dei monostrati HIO
- Preparare l'inoculo virale. Se necessario, la tripsina attiva il ceppo virale. Per il rotavirus, aggiungere 10 μg/mL di tripsina di Worthington alle scorte di virus e incubare a 37 °C per 1 ora.
- Diluire il virus attivo utilizzando uno dei due metodi seguenti.
- Se si punta al numero massimo di cellule infette utilizzando un solo ceppo virale, miscelare 50 μL di stock virale attivato con 50 μL di CMGF-.
- Se si confrontano più ceppi, preparare gli inoculi per ottenere una molteplicità equivalente di infezione (MOI). Utilizzare la seguente formula per determinare il volume delle scorte di virus per pozzetto necessarie per il MOI desiderato. Aggiungere CMGF- al volume di scorte virali calcolato per ottenere un volume finale di 100 μL per pozzetto.
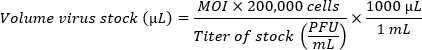
NOTA: Un singolo monostrato HIO placcato in un pozzetto con un diametro di 5,46 mm (piastra a 96 pozzetti) contiene circa 200.000 cellule. A causa della scarsa efficienza dell'infezione nei monostrati, è preferibile un MOI elevato (100-1 milione di particelle virali per cellula). Il MOI ottimale deve essere determinato empiricamente.
- Infettare i monostrati rimuovendo delicatamente il terreno dal monostrato HIO utilizzando una pipetta da 200 mL. Sostituire con 100 ml di inoculo virale o inoculo simulato.
- Poiché la maggior parte dei ceppi di rotavirus si propaga nelle cellule MA104, utilizzare un lisato di cellule MA104 non infetto per l'inoculo simulato. Utilizzare un volume equivalente al volume di scorte virali utilizzato per i pozzetti infetti e aggiungere CMGF- per un volume finale di 100 mL per pozzetto.
- Per i virus che infettano le cellule basolateralmente, come il rotavirus14, utilizzare un ago da 25G per incidere il monostrato. È più facile fare un singolo segno su tutta la lunghezza del monostrato, dal basso verso l'alto del pozzo. Premere delicatamente l'ago nel monostrato ad angolo con il lato smussato rivolto verso l'alto, opposto alla direzione del movimento, e trascinarlo per tutta la lunghezza del monostrato per creare una cicatrice (Figura 2A).
- Incubare in incubatrice a 37 °C per 2 ore. Rimuovere il virus e simulare l'inoculo. Lavare i monostrati una volta con 1x PBS.
- Aggiungere 100 μL di terreno di differenziazione privo di rosso fenolo. Posizionare il vetrino in un incubatore a 37 °C fino al momento di ottenere l'immagine. La finestra di imaging ottimale varia in base alla cinetica dell'infezione virale. Per il rotavirus, iniziare l'imaging a 6-8 ore dopo l'infezione.
- Diluire il virus attivo utilizzando uno dei due metodi seguenti.
5. Imaging Ca2+ di monostrati infetti
- Preriscaldare l'incubatrice a 37 °C. Funzionamento con CO2 umidificato a 0,02 L/min. Posizionare il vetrino con i monostrati infetti nella camera dell'incubatrice e sigillare il coperchio.
- Utilizzando l'illuminazione in campo chiaro (BF) e un obiettivo 20x, selezionare le coordinate X e Y dei campi visivi all'interno del monostrato che verrà ripreso. È meglio selezionare i punti con la regione segnata in vista, poiché la maggior parte delle cellule infette sarà vicina al graffio.
- Ottimizza i parametri di imaging e acquisisci un'immagine utilizzando l'eccitazione a 488 nm. Per ridurre al minimo la fototossicità, impostare la sorgente luminosa al 50% di potenza con un tempo di esposizione di 50 ms. I parametri di acquisizione appropriati possono variare e devono essere ottimizzati acquisendo più acquisizioni. Assicurati che nessun pixel sia saturo. Regolare il tempo di esposizione e la potenza della luce in base alle esigenze per garantire che l'intera gamma dinamica del sensore di calcio fluorescente possa essere rilevata.
- Se si utilizzano altri fluorofori (ad esempio, virus marcati con fluorescenza), ripetere l'ottimizzazione su questi canali. Impostare il loop di imaging e acquisire un'immagine a 488 nm ad ogni coordinata X, Y selezionata in un loop di 1 minuto. Immagina questo ciclo continuamente. Quando si utilizza un virus marcato con fluorescenza, acquisire un'immagine sul canale appropriato ogni 10loop (ad esempio, 1 immagine/10 min).
- Raccogli immagini per ~18 h. Se si utilizzano virus senza tag fluorescenti, fissare i monostrati ed eseguire la colorazione in immunofluorescenza per identificare le cellule infette. Eseguire questa operazione dopo aver completato l'esecuzione dell'imaging come descritto di seguito.
- Rimuovere i supporti di imaging e sostituirli con 100 μL di formaldeide al 4% in PBS 1x. Incubare a temperatura ambiente per 30 min.
- Rimuovere il fissativo e temprare con 100 μL di 50mM NH4Cl per 10 min. Rimuovere NH4Cl. Permeabilizzare i monostrati con 100 μL di Triton X-100 allo 0,1% in 1x PBS per 1 ora a temperatura ambiente o per una notte a 4 °C.
- Rimuovere il tampone di permeabilizzazione. Bloccare con 100 μL di albumina sierica bovina al 3% in 1x PBS per 1 ora.
- Rimuovere la soluzione bloccante. Aggiungere gli anticorpi primari diluiti alla concentrazione raccomandata/ottimizzata in 1x PBS. Aggiungere 100 μL di soluzione anticorpale per pozzetto.
- Incubare per una notte a 4 °C con un leggero dondolio. Rimuovere gli anticorpi primari. Lavare 3 volte con 1 PBS.
- Aggiungere anticorpi secondari coniugati con fluorescenza diluiti alla concentrazione raccomandata/ottimizzata in 1x PBS. Aggiungere 100 μL per pozzetto. Incubare per 2 ore a temperatura ambiente.
NOTA: FPBase15 è un'ottima risorsa per aiutare a selezionare i database secondari che eviteranno il bleed-through durante il multiplexing. La maggior parte dei secondari è efficace con una diluizione 1:1000 in PBS 1x. - Rimuovere gli anticorpi secondari. Preparare una soluzione DAPI da 1 μg/mL in 1x PBS e aggiungere 100 μL per pozzetto. Incubare per 20 min. Rimuovere DAPI. Lavare 3 volte con 1 PBS.
- Mantenere i monostrati fissi in 100 μL di PBS 1x per l'imaging. Riposizionare il vetrino sul tavolino del microscopio, ricaricare le coordinate X e Y dall'esecuzione dell'imaging dal vivo e visualizzare l'immagine di ciascun punto multiplo sul canale che corrisponde all'anticorpo secondario utilizzato per la colorazione. Fare riferimento alle immagini dell'esecuzione di immagini dal vivo per assicurarsi che vengano acquisiti gli stessi punti.
6. Quantificazione delle onde intercellulari di calcio
- Assicurati che Fiji is Just ImageJ (FIJI) sia installato con i seguenti plug-in: Bio-Formats (dovrebbe essere preinstallato in FIJI ma non sarà in ImageJ); Ricettario: per eseguire l'installazione, dal menu FIJI, vai a Guida > aggiornamenti > Gestisci siti di aggiornamento. Dai un'occhiata al ricettario. Riavvia le Figi.
- Suddividere il file di dati dall'esecuzione dell'imaging dal vivo in più file, separando ciascuna coordinata X, Y. In Nikon NES Elements, selezionare File > Importa/Esporta > Dividi punti multipli. Selezionare una cartella per l'esportazione e un prefisso appropriato.
- Aprire le FIJI. Caricare un singolo file dai punti multipli divisi. Se si utilizza un'immagine multicanale, dividere i canali. Selezionare la finestra con le immagini del canale a 488 nm (GCaMP).
- Eseguire la funzione DeltaF Up per determinare la variazione del valore di ciascun pixel da un punto temporale all'altro. A tale scopo, selezionare Ricettario > funzioni T > Delta F Up.
- Scorri la pila fino a trovare un'immagine con un'onda di calcio. Con l'onda di calcio in vista, impostare una soglia facendo clic su Immagine > Regola > soglia. Regolare il limite inferiore per ridurre al minimo la quantità di segnale che non fa parte dell'onda che supera la soglia. Per le immagini a 16 bit acquisite utilizzando i parametri sopra descritti, iniziare con una soglia inferiore di 600 e regolare in base alle esigenze.
- Eseguire l'analizzatore di particelle per segmentare le onde facendo clic su Analizza > Analizza particelle. Impostare la dimensione minima dell'onda in μm2 regolando l'intervallo. È impostato su 0-infinito al valore di base. Per le immagini di cellule MA104 acquisite utilizzando un obiettivo 20x, iniziare aumentando il limite inferiore a 10.000 μm2 e regolare in base alle esigenze.
- Selezionare le caselle Visualizza risultati e Cancella risultati. Dal menu a discesa "Mostra", seleziona Contorni, quindi fai clic su OK. Ciò dovrebbe comportare 2 output: Una finestra, Risultati, elencherà ogni onda rilevata, l'area dell'onda e i suoi valori di intensità media, minima e massima (in base al delta F). L'altra finestra, intitolata Disegno di [nome file], includerà una nuova pila con i contorni delle onde segmentate. Utilizzare questa opzione per verificare il rilevamento delle onde. Regolare i valori di soglia e l'intervallo di dimensioni, se necessario, e ricontrollare le chiamate ondata.
- Per l'elaborazione batch, utilizzare lo script macro incluso (file di codifica supplementare 1). Per utilizzarlo, segui i passaggi descritti di seguito.
- Suddividere i punti multipli in file singoli come descritto nel passaggio 6.2. Aprire le FIJI. Selezionare Elabora > Batch > Macro. Nella casella "input", fornisci la mappa alla cartella contenente i file multipoint divisi. Lasciare vuota la casella "output".
- Incollare lo script dal file di codifica supplementare 1 nella casella. Regolare la soglia di intensità e la soglia di dimensione nello script in modo che riflettano i parametri ottimizzati dei passaggi 6.5 e 6.6.
- Fare clic su Elabora. A seconda del numero di punti multipli e della velocità di elaborazione del computer, l'elaborazione di tutte le immagini può richiedere 30 minuti o più. Una volta completati, i dati verranno scritti in un nuovo foglio di calcolo denominato "Rinominami dopo la scrittura" sul desktop. Il file di output deve contenere, per ogni multipunto, un conteggio delle onde e le metriche delle onde (area, intensità media, intensità minima e massima e densità di intensità).
Risultati
La Figura 1A mostra una cupola BMM contenente organoidi intestinali umani tridimensionali che sono stati trasdotti per esprimere stabilmente GCaMP6. La Figura 1B mostra la stessa linea di organoidi riplaccati come monostrato a 24, 48 e 72 ore dopo la semina. Per convalidare la funzione di GCaMP6s, il monostrato è stato sottoposto a imaging mediante microscopia a fluorescenza ogni 2 secondi per 4 minuti e 100 nM di ADP sono stati aggiunti al terreno dopo ~ 20 se...
Discussione
Le alterazioni dei livelli citosolici di Ca2+ possono essere sia causa che effetto di patologie all'interno dell'epitelio 10,16,17. L'aumento del calcio citosolico può guidare direttamente la secrezione attraverso l'attivazione del canale del cloro calcio-dipendente TMEM16A18,19. L'attivazione di TMEM16A in risposta a Ca2+ consente l'efflusso apica...
Divulgazioni
Gli autori non hanno interessi finanziari concorrenti da divulgare.
Riconoscimenti
Questo lavoro è stato sostenuto da sovvenzioni R01DK115507 e R01AI158683 (PI: J. M. Hyser) del National Institutes of Health (NIH). Il supporto ai tirocinanti è stato fornito da NIH grants F30DK131828 (PI: J.T. Gebert), F31DK132942 (PI: F. J. Scribano) e F32DK130288 (PI: K.A. Engevik). Vorremmo ringraziare il Texas Medical Center Digestive Diseases Enteroid Core per aver fornito i terreni di mantenimento dell'organoide.
Materiali
| Name | Company | Catalog Number | Comments |
| Advanced DMEM F12 | Gibco | 12634028 | |
| [Leu15]-Gastrin I | Sigma-Aldrich | G9145 | |
| 0.05% Trypsin EDTA | Gibco | 25300054 | |
| 0.05% Trypsin EDTA | Gibco | 25300054 | |
| 1.5mL microcentrifuge tubes | Fisherbrand | 5408137 | |
| 15mL conical tubes | Thermofisher Scientific | 0553859A | |
| 16% formaldehyde | Thermofisher Scientific | 28906 | |
| 1M HEPES | Gibco | 15630080 | |
| 1M HEPES | Gibco | 15630080 | |
| 1X PBS | Corning | 21-040-CV | |
| 25 gauge needle | Thermofisher Scientific | 1482113D | |
| A-83-01 | Tocris | 2939 | |
| ADP | Sigma-Aldrich | A2754 | |
| Advanced DMEM F12 | Gibco | 12634028 | |
| Antibiotic-antimycocytic | Gibco | 15240062 | |
| Antibiotic-antimycotic | Gibco | 15240062 | |
| B27 Supplement | Gibco | 17504-044 | |
| Bovine serum albumin | FisherScientific | BP1600100 | |
| CellView Cell Culture Slide, PS, 75/25 MM, Glass Bottom, 10 compartments | Greiner | 543979 | |
| Collagen IV | Sigma Aldrich | C5533 | |
| DAPI | Thermofisher Scientific | D1306 | |
| EDTA | Corning | 46-034-CI | |
| Fetal bovine serum | Corning | 35010CV | |
| Fetal bovine serum | Corning | 35010CV | |
| Fluorobrite | Gibco | A1896701 | |
| GlutaMAX | Gibco | 35050079 | |
| GlutaMAX | Gibco | 35050079 | |
| Human epidermal growth factor | ProteinTech | HZ-1326 | |
| Lentivirus | VectorBuilder | (variable) | |
| Matrigel | BD Biosceicen | 356231/CB40230C | |
| N2 Supplement | Gibco | 17502-048 | |
| N-acetylcysteine | Sigma-Aldrich | A9165-5G | |
| NH4Cl | Sigma-Aldrich | A9434 | |
| Nicotinamide | Sigma-Aldrich | N0636 | |
| Nunc Cell Culture Treated 24-well Plates | Thermofisher Scientific | 142475 | |
| Polybrene | MilliporeSigma | TR1003G | |
| SB202190 | Sigma-Aldrich | S70767 | |
| Triton X-100 | Fisher BioReagents | BP151100 | |
| TrypLE Express Enzyme, no phenol red | Thermofisher Scientific | 12604013 | |
| Trypsin | Worthington Biochemical | NC9811754 | |
| Y-27632 | Tocris | 1254 |
Riferimenti
- Bootman, M. D., Bultynck, G. Fundamentals of cellular calcium signaling: A primer. Cold Spring Harb Perspect Biol. 12 (1), a038802 (2020).
- Clapham, D. E. Calcium signaling. Cell. 131 (6), 1047-1058 (2007).
- Danese, A., et al. Cell death as a result of calcium signaling modulation: A cancer-centric prospective. Biochim Biophys Acta Mol Cell Res. 1868 (8), 119061 (2021).
- Harr, M. W., Distelhorst, C. W. Apoptosis and autophagy: Decoding calcium signals that mediate life or death. Cold Spring Harb Perspect Biol. 2 (10), a005579 (2010).
- Barak, P., Parekh, A. B. Signaling through Ca2+ microdomains from store-operated CRAC channels. Cold Spring Harb Perspect Biol. 12 (7), a035097 (2020).
- Nakai, J., Ohkura, M., Imoto, K. A high signal-to-noise Ca(2+) probe composed of a single green fluorescent protein. Nat Biotechnol. 19 (2), 137-141 (2001).
- Erofeev, A. I., Vinokurov, E. K., Vlasova, O. L., Bezprozvanny, I. B. GCaMP, a family of single-fluorophore genetically encoded calcium indicators. J Evol Biochem Phys. 59 (4), 1195-1214 (2023).
- Suzuki, J., Kanemaru, K., Iino, M. Genetically encoded fluorescent indicators for organellar calcium imaging. Biophys J. 111 (6), 1119-1131 (2016).
- Nászai, M., Cordero, J. B. Intestinal stem cells: Got calcium. Curr Biol. 26 (3), R117-R119 (2016).
- Barrett, K. E. Calcium-mediated chloride secretion in the intestinal epithelium: Significance and regulation. Curr Top Membr. 53, 257-282 (2002).
- Xu, J., et al. Calcium-sensing receptor regulates intestinal dipeptide absorption via Ca2+ signaling and IKCa activation. Physiol Rep. 8 (1), e14337 (2020).
- Clevers, H. Modeling development and disease with organoids. Cell. 165 (7), 1586-1597 (2016).
- Lin, S. C., Haga, K., Zeng, X. L., Estes, M. K. Generation of CRISPR–Cas9-mediated genetic knockout human intestinal tissue–derived enteroid lines by lentivirus transduction and single-cell cloning. Nat Protoc. 17 (4), 1004-127 (2022).
- Crawford, S. E., Ramani, S., Blutt, S. E., Estes, M. K. Organoids to dissect gastrointestinal virus-host interactions: What have we learned. Viruses. 13 (6), 999 (2021).
- Lambert, T. J. FPbase: a community-editable fluorescent protein database. Nat Methods. 16 (4), 277-278 (2019).
- Lai, Y., et al. Inhibition of calcium-triggered secretion by hydrocarbon-stapled peptides. Nature. 603 (7903), 949-956 (2022).
- Chang-Graham, A. L., et al. Rotavirus induces intercellular calcium waves through ADP signaling. Science. 370 (6519), eabc3621 (2020).
- Lee, B., et al. Anoctamin 1/TMEM16A controls intestinal Cl− secretion induced by carbachol and cholera toxin. Exp Mol Med. 51 (8), 1-14 (2019).
- Saha, T., et al. Intestinal TMEM16A control luminal chloride secretion in a NHERF1 dependent manner. Biochem Biophys Rep. 25, 100912 (2021).
- Mroz, M. S., Keely, S. J. Epidermal growth factor chronically upregulates Ca2+-dependent Cl− conductance and TMEM16A expression in intestinal epithelial cells. J Physiol. 590 (8), 1907-1920 (2012).
- Sui, J., et al. Dual role of Ca2+-activated Cl− channel transmembrane member 16A in lipopolysaccharide-induced intestinal epithelial barrier dysfunction in vitro. Cell Death Dis. 11 (5), 404 (2020).
- Bellono, N. W., et al. Enterochromaffin cells are gut chemosensors that couple to sensory neural pathways. Cell. 170 (1), 185-198.e16 (2017).
- Paradis, T., Bègue, H., Basmaciyan, L., Dalle, F., Bon, F. Tight junctions as a key for pathogens invasion in intestinal epithelial cells. Int J Mol Sci. 22 (5), 2506 (2021).
- Samak, G., et al. Calcium/Ask1/MKK7/JNK2/c-Src signalling cascade mediates disruption of intestinal epithelial tight junctions by dextran sulfate sodium. Biochem J. 465 (3), 503-515 (2015).
- Deng, H., Gerencser, A. A., Jasper, H. Signal integration by Ca2+ regulates intestinal stem cell activity. Nature. 528 (7581), 212-217 (2015).
- Saurav, S., Tanwar, J., Ahuja, K., Motiani, R. K. Dysregulation of host cell calcium signaling during viral infections: Emerging paradigm with high clinical relevance. Mol Aspects Med. 81, 101004 (2021).
- Chang-Graham, A. L., et al. Rotavirus calcium dysregulation manifests as dynamic calcium signaling in the cytoplasm and endoplasmic reticulum. Sci Rep. 9 (1), 10822 (2019).
- Hyser, J. M., Collinson-Pautz, M. R., Utama, B., Estes, M. K. Rotavirus disrupts calcium homeostasis by NSP4 viroporin activity. mBio. 1 (5), e00265-e00310 (2010).
- Pham, T., Perry, J. L., Dosey, T. L., Delcour, A. H., Hyser, J. M. The Rotavirus NSP4 viroporin domain is a calcium-conducting ion channel. Sci Rep. 7, 43487 (2017).
- Crawford, S. E., Hyser, J. M., Utama, B., Estes, M. K. Autophagy hijacked through viroporin-activated calcium/calmodulin-dependent kinase kinase-β signaling is required for rotavirus replication. Proc Natl Acad Sci U S A. 109 (50), E3405-E3413 (2012).
- Crawford, S. E., Criglar, J. M., Liu, Z., Broughman, J. R., Estes, M. K. COPII vesicle transport is required for Rotavirus NSP4 interaction with the autophagy protein LC3 II and trafficking to viroplasms. J Virol. 94 (1), e01341 (2019).
- Pando, V., Iša, P., Arias, C. F., Ló Pez, S. Influence of calcium on the early steps of Rotavirus infection. Virology. 295 (1), 190-200 (2002).
- Hyser, J. M., Estes, M. K. Pathophysiological consequences of calcium-conducting viroporins. Annu Rev Virol. 2 (1), 473-496 (2015).
- Strtak, A. C., et al. Recovirus NS1-2 has viroporin activity that induces aberrant cellular calcium signaling to facilitate virus replication. mSphere. 4 (5), e00506-e00519 (2019).
- In, J. G., Foulke-Abel, J., Clarke, E., Kovbasnjuk, O. Human colonoid monolayers to study interactions between pathogens, commensals, and host intestinal epithelium. J Vis Exp. (146), 59357 (2019).
- Hirota, A., AlMusawi, S., Nateri, A. S., Ordóñez-Morán, P., Imajo, M. Biomaterials for intestinal organoid technology and personalized disease modeling. Acta Biomater. 132, 272-287 (2021).
- Cevallos Porta, D., López, S., Arias, C. F., Isa, P. Polarized rotavirus entry and release from differentiated small intestinal cells. Virology. 499, 65-71 (2016).
- Mirabelli, C., et al. Human Norovirus efficiently replicates in differentiated 3D-human intestinal enteroids. J Virol. 96 (22), e0085522 (2022).
- Icha, J., Weber, M., Waters, J. C., Norden, C. Phototoxicity in live fluorescence microscopy, and how to avoid it. Bioessays. 39 (8), 28749075 (2017).
- Li, J., et al. Engineering of NEMO as calcium indicators with large dynamics and high sensitivity. Nat Methods. 20 (6), 918-924 (2023).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneEsplora altri articoli
This article has been published
Video Coming Soon