Method Article
手術後の多発性原発性結腸直腸癌患者における癌特異的生存を予測するための競合リスクノモグラム
要約
この研究では、男性の性別、腫瘍の悪性度の低下、および進行した腫瘍リンパ節転移の病期が、手術後の複数の原発性結腸直腸癌(MPCC)患者の癌特異的生存率(CSS)の低下と関連していることがわかりました。MPCC患者のCSSを予測し、臨床治療の意思決定に貢献するためのノモグラムを開発しました。
要約
多発性原発性結腸直腸癌(MPCC)患者におけるがん特異的生存率(CSS)は、他の原因による死亡によって競争的に影響を受けます。この研究は、MPCC患者における競合リスク分析により、CSSおよび関連する危険因子を調査することを目的としていました。この研究のデータはSEERデータベースからのものです。競技リスクモデルの単変量および多変量解析を使用して、競技イベントの影響を弱めるために、CSSのリスク要因を調査し、ノモグラムモデルを開発します。次に、モデルの性能をROC曲線、検量線、およびDCAによって検証します。この研究には合計8931人の患者が含まれ、そのうち6255人がトレーニングコホート、2676人が検証コホートです。単変量解析および多変量解析により、性別、腫瘍リンパ節転移(TNM)病期、および腫瘍悪性度が、MPCC患者におけるがん特異的生存の独立した危険因子であることが示された。リスク要因に基づいて、CSSを予測するための図モデルを開発しました。ROC曲線、検量線、DCAも良好な結果を示しています。結論として、MPCC患者のCSSを予測するための貴重なツールとして機能するノモグラムが開発され、臨床医に個別の治療計画のための重要な洞察を提供します。
概要
結腸直腸癌は、世界で最も一般的な消化器系腫瘍の1つであり、過去数十年にわたってその発生率が継続的に増加しています。高い発生率と死亡率が持続していることは、大きな注目を集めています。最新の統計によると、結腸直腸癌は世界の一般的な悪性腫瘍の中で3位にランクされており、その死亡率は世界第2位にランクされています1。多発性原発性結腸直腸癌(MPCC)は、近年研究者からますます注目を集めている結腸直腸癌の特別なサブタイプです。これは、同じ患者内の2つ以上の独立した結腸直腸癌病変の診断として定義され、病変の端の間の距離が5cmを超えます。MPCCでは、複数の腫瘍が同時に診断された場合、または6か月以内に診断された場合は同時性結腸直腸癌(SCRC)と定義され、診断間隔が6か月を超える場合は異時性結腸直腸癌(MCRC)と定義されます2,3,4,5。
すべての結腸直腸がんにおけるMPCCの割合は比較的低く、その発生率に関する報告は地域や研究によって異なります。最近の研究では、MPCCが結腸直腸癌の2%〜10%を占めることが報告されています2,3,4,5。孤立性結腸直腸癌と比較して、MPCCの予後は3,6。現在、MPCCの予後の臨床評価は、主に米国がん合同委員会の腫瘍リンパ節転移(TNM)病期分類システムに依存しており、MPCCの実際の病期は複数の病変の最も進行した病期を使用しています。しかし、TNM病期分類のみに基づいて予後予測を行うだけでは不十分です。MPCCの術後の予後を予測するための効果的なツールはまだ不足しています。現在の精密医療の時代では、リスクを定量化するための臨床予測モデルは、臨床上の意思決定や患者の予後評価に広く使用されています7,8。近年、独立した予後危険因子に基づくノモグラムは、腫瘍の予後を予測するために広く受け入れられている9。ノモグラムは、複雑な統計モデルを視覚化できるため、臨床現場での適用が容易になります。腫瘍予後の評価では、全生存期間(OS)とがん特異的生存期間(CSS)が一般的に使用されるアウトカム指標です9。OSとは、患者のがんが確認されてから何らかの原因で死亡するまでの時間を指します。CSSは、腫瘍の診断から特に癌によって引き起こされた死亡までの時間を指し、癌による死亡のリスクをより正確に反映しています。CSSがアウトカム指標として使用される場合、他の因子による死亡はがん特異的な死亡の確率に影響を及ぼし、2つの事象の間に競合するリスク関係を導入する可能性がある10,11。したがって、腫瘍特異的生存率の研究では、競合するリスクモデルを使用して、競合するイベントの影響を排除する必要があります。以前の研究では、MPCCの予後を予測するモデルが構築されていましたが、これらは同時性結腸直腸癌に限定されており、競合するイベントがCSSに与える影響を説明するために競合するリスクモデルを使用していませんでした12,13,14。
この研究では、手術後のMPCCのがん特異的生存率に影響を与える競合する独立した危険因子を調査しました。競合するリスクモデルを採用する理論的根拠は、患者ががん以外の原因で死亡する可能性を説明する能力に由来しており、これは偏りのない生存推定値を得るために重要である15,16。Coxモデルのような従来の生存分析手法は、競合する事象の存在下での生存確率を過大評価する可能性があり、これらのシナリオでは競合するリスクモデルの使用がより適切になる17。
特定された独立したリスク要因に基づいて、生存確率を予測するためのノモグラムを構築し、その性能を検証しました。ノモグラムは、複雑な統計モデルのユーザーフレンドリーなグラフィカル表現を提供し、臨床医が個別のリスクスコア18,19を簡単に計算できるため、臨床現場で人気を博しています。他の予測ツールとは異なり、ノモグラムは複数のリスク要因を組み込み、より正確でパーソナライズされた予後推定を提供します。このアプローチは、さまざまながんでますます検証されており、従来の病期分類システムよりも優れた性能を示しています20,21,22。当社のツールは、臨床医が診断および治療中に個別化されたより正確な予後推定を行うのを支援し、それによってMPCCの管理における意思決定を強化することを目的としています。
プロトコル
この調査には 2 つのステップがあります。まず、SEERデータベースからMPCCの臨床データと生存データを取得しました。次に、Rソフトウェア(バージョン4.3.3)を使用して、競合するリスクモデルを分析および構築しました。この試験のワークフロー図を 図1に示します。この研究に参加するために倫理的な承認と同意は必要ありません。この研究で使用されたデータは、データベースから取得されました。
1. データ取得
- SEERをダウンロードします。SEERデータベースWebサイト(http://seer.cancer.gov/about/overview.html)のStat 8.4.3ソフトウェア。SEERに登録してログインします。関連する患者データを取得するための統計8.4.3。
- SEERにログイン後。Stat 8.4.3 で Case Listing Session > Data をクリックし 、Incidence SEER Research Data, 17 Registries, Nov 2022 Sub (2000-2020) データベースを選択します。
- [選択] > [編集] をクリックし、{人種、性別、年、Dx. 診断年} = '2004', '2005', '2006', '2007', '2008', '2009', '2010', '2011', '2012', '2013', '2014', '2015' AND {サイトと形態を選択します。サイト再コードICD-O-3 / WHO 2008} = '結腸と直腸' AND {サイトと形態。診断確認} = '陽性組織型' AND {複数のプライマリフィールド。連番}!= '1 つのプライマリのみ'。
- [OK]をクリックして、選択内容を保存します。[テーブル]をクリックし、[使用可能な変数]インターフェイスで、[単一の年齢と85+の年齢再コード]、[性別]、[サイト再コードICD-O-3 / WHO 2008]、[CS腫瘍サイズ]、[グレード再コード(2017年まで)]、[派生AJCC T、第6版(2004-2015)]、[派生AJCC N、第6版(2004-2015)]、[派生AJCC M、第6版(2004-2015)]、[放射線再コード化]、[化学療法再コード化(はい、いいえ/未解決)、SEER原因別死亡分類]、[ SEERその他の死因分類、生存期間、患者ID、RX Summ-Surg Prim Site (1998+)を選択し、列をクリックしてください。
- [出力] をクリックし、データに名前を付け、[実行] をクリックしてデータを出力して保存します。
- SEER データベースは、患者が MPCC に感染しているかどうかに関する情報を直接提供していません。上記のようにデータのダウンロードが完了したら、患者 ID を使用して MPCC の患者 (つまり、2 回以上の発生と診断された患者) を除外します。MPCC患者を特定した後、生存時間に基づいて腫瘍診断の間隔を計算します。6か月をカットオフとして、患者をSMPCCとMMPCCに分類します。
- 初期診断時の年齢に基づいて、患者の年齢を= 65歳と>65歳に分類します。腫瘍の悪性度については、悪性度Iが良好な分化、悪性度IIが中等度の分化、悪性度IIIが低分化、悪性度IVが未分化に対応する。複数の腫瘍の分布に基づいて、腫瘍の位置を右結腸、左結腸直腸、および結腸直腸全体として分類します。
- 近位結腸直腸と遠位結腸直腸の間には、胚発生および生物学的特性に明らかな違いがあり、脾臓屈曲23に基づいて腫瘍の位置を分割する。右結腸は、盲腸、上行結腸、肝屈曲、横行結腸を含む脾屈曲の近位と定義され、左結腸には脾屈曲、下行結腸、S状結腸、直腸S状結腸、および直腸が含まれます。患者の腫瘍が完全に右結腸にある場合は、腫瘍の位置を右結腸と定義します。すべての腫瘍が左結腸直腸にある場合は、その位置を左結腸直腸と定義します。右結腸と左結腸直腸の両方に腫瘍がある場合は、その位置を結腸直腸全体と定義します。
- 同じ患者の複数の腫瘍の中から最大の腫瘍径を選択することにより、腫瘍サイズを決定します。腫瘍の直径に基づいて、サイズを= 5cmまたは>5cmに分類します。この研究には、合計8,931人の患者が含まれていました。トレーニング コホートと検証コホートを作成するには、すべてのケースを 7:3 の比率でランダムに分割します。
2. モデル構築
- RStudio (2023.12.1+402) と R ソフトウェア (4.3.3) をダウンロードします。RStudio を開いて R ソフトウェアを実行します。 [新しいファイル ]をクリックし、[ Rスクリプト ]を選択して、新しいRプログラミングインターフェイスを作成します。コードエディタで関連するコードを入力し、[ 実行 ]をクリックしてコードを実行します。
注: R 言語とその関数には、さまざまなパラメータがあります。これらのパラメータを追加または変更すると、データの解析と視覚化を強化できます。 - 次のコードを使用して、単変量解析を実行し、CIF 曲線をプロットします。
図書館(tidycmprsk)
ライブラリ(gtsummary)
図書室(ggsurvfit)
図書館(GGPRISM)
データ <-read.csv('data.csv')"
data$status<-factor(data$status, levels=c(0,1,2),labels=c("0","CSS","OCS"))
CIF <- tidycmprsk::cuminc(Surv(時間, ステータス) ~ 性別, データ = データ)
ggcuminc(CIF,outcome= c("CSS", "OCS"),サイズ=1.5)
ここで、data.csv は SEER データベースから取得したデータです。 - 上記のコードを実行した後、[ エクスポート]をクリックし、[ 画像として保存]をクリックし、最後に[ 保存 ]をクリックして画像を保存します。後続の画像を保存する方法は、この手順と同じです。上記のコードの性別を1つずつ他の要因に置き換えて、すべての要因に対して単変量解析を実行します。
- 次のコードを使用して、BSR と多変量解析と視覚化を実行します。
図書室(LEPS)
ライブラリ(riskRegression)
図書室(Prodlim)
図書室(forestplot)
leaps<-regsubsets(status==0~性別+サイズ+学年+T+N+M,データ = データ)
plot(leaps,scale="adjr2")
multi <- FGR(Hist(時間,状態)~性別 + T + N + M +Grade+ サイズ,原因=1,データ=データ)
概要(マルチ)
マルチ<- read.csv("multi.csv",ヘッダ = T)
forestplot(labeltext=as.matrix(Multi [,1:4]), mean= Multi $HR_mean, ower= Multi $HR_1, upper= Multi $HR_2)
ここで、multi.csvのデータは前のコードの結果から取得されます。 - 手順2.3に従います。をクリックして画像を保存します。
- 次のコードを使用して、ノモグラム、ROC曲線、検量線、およびDCA曲線をプロットします。
図書室(QHScrnomo)
図書館(RMS)
ライブラリ(riskRegression)
図書室(Prodlim)
データ<-read.csv(以下「data.csv」)
D <- datadist(データ)
オプション(datadist = "d")
e <-cph(Surv(time,status==1)~Sex + T + N + M +Grade,data = data,
x=T, y=T, surv=T,time.inc=60)
野茂 <- crr.fit(e,failcode=1,cencode = 0)
nomogram.crr(fit =nomo, lp = F, xfrac = 0.5, fun.at =seq(from=0, to=1, by= 0.1) , failtime =c(12,36,60),funlabel = c("1-year CSS Cumulative Incidence", "3-year CSS Cumulative Incidence","5-year CSS Cumulative Incidence"))
セットシード(123)
data$pro <- tenf.crr(m3,time = 60)
groupci(x=data$pro, ftime = data$time, fstatus = data$status, failcode = 1, cencode = 0, ci = TRUE)
f <- CSC(Hist(時間,状態)~性別 + T + N + M +Grade,データ = データ)
x <- Score(list(model1=f), Hist(time,status)~1, data=data, cause=1, times=c(12,36,60), se.fit=1L, plots="roc", metrics="auc")
g <- as.data.frame(x$AUC$score)
h <- g[g$times %in% c(12,36,60),]
col = c("ダークシアン","トマト","パープル")
plotROC(x, xlab="1-特異性", ylab="感度",col=col[1], cex=1.5, legend="", auc.in.legend = F, times = 12)
plotROC(x,col=col[2],legend = '', cex=1.5,times =36,auc.in.legend = F,add=T)
plotROC(x,col=col[3], times =60, add=T, cex=1.5, legend = '',auc.in.legend = F)
leg <- paste0(c("1年: ","3年: ","5年: "),substr(a$AUC,1,5)) - 手順2.3に従います。をクリックして画像を保存します。
- ggscidca パッケージを使用して、DCA 曲線をプロットします。
図書館(GGSciDCA)
df_surv<-read.csv("data.csv",ヘッダ = T)
cox_model <- coxph(Surv(時間,ステータス==1)~性別 + T + N+ M +グレード, データ = df_surv)
cox_model1<-newcrr(cox_model)
scidca(cox_model1, newdata= df_surv,threshold.line = T,threshold.text = T)
ここで、data.csv は SEER データベースから取得したデータです。 - 手順2.3に従います。をクリックして画像を保存します。
結果
患者の特性
この研究には、合計8,931人の患者が含まれていました。性別分布では、女性患者(44%)に比べて男性患者(56%)の割合が高かった。腫瘍の位置分布に関しては、腫瘍の大部分は全結腸直腸に分布しており、腫瘍は最も少なかった(21%)。腫瘍の悪性度に関しては、最も一般的な悪性度はグレードIIで、患者数は6,251人で、全体の70%を占めました。次いでグレードIIIで、患者数は2,026人(23%)でした。グレードIとIVの患者数はそれぞれ355人、IVは299人で、全体の4%と3.3%を占めていました。腫瘍浸潤に関しては、T3およびT4の病期が最も一般的でした。患者の半数以上はリンパ節転移を認めず、17%の患者が遠隔転移を経験した。放射線療法は1,446人(16%)が受け、化学療法は3,843人(43%)が実施されました。SCRCはより一般的で、5,327人の患者が全体の60%を占めていました。異時性結腸直腸癌の患者は3,604人で、40%を占めていました。患者は、7:3の比率でトレーニングセットと検証セットにランダムに分けられました。 表1に示すように、トレーニングコホートと検証コホートの間でベースラインデータに統計的に有意な差はありませんでした。
単変数解析
競争イベントの影響を制御した後、単変量解析の結果は、性別、腫瘍の悪性度とサイズ、TNM病期、放射線、化学療法、同期または異時性の状態、および腫瘍の位置がMPCC患者のCSSに影響を与える予後因子であることを示しました。年齢だけがMPCC患者のCSSの予後因子ではありません。放射線療法、化学療法、部位、および同期性または異時性の状態においてCIFには有意な共通点があることに留意し、MPCC患者に対する放射線の短期的および長期的な予後的影響は、化学療法、部位、および同期または異時性の状態と同様に異なっていたことを示している。各サブグループの累積リスク曲線を 図2に示します。
多変数解析
単変量解析によって得られた予後因子は、競争リスクモデルの最適サブセット回帰(BSR)および多変量解析に組み込まれました。その中で、放射線療法、化学療法、場所、および同期性または異時性の状態は、予後に二重の影響を与えるため除外されます。BSRと多変量解析の結果は、性別、TNM病期、および腫瘍の悪性度がMPCC患者におけるCSSの独立した危険因子であることを示しました。(図2)
ノモグラムの構築と検証
多変量解析によって得られた独立したリスク要因に基づいて、CSSを予測するためのラインノモグラムを構築し、予測モデルの性能を検証します(図3)。次に、ROC曲線、検量線、およびDCAを使用してモデルを評価します。ROC曲線は、訓練コホートの1年、3年、5年のAUCが0.762、0.742、0.734であり、検証コホートの1年、3年、5年のAUCが0.801、0.740、0.743であったことを示した。トレーニングコホートと検証コホートでは、検量線は予測確率と実際のデータとの間に高い一致を示しました(図4)。モデルの臨床応用における性能を検証するために、DCAを用いてモデルの臨床的価値を評価しました。結果は、モデルが良好な純利益を示していることを示しています(図5)。
図1:試験のワークフロー図。 この研究は2つのステップから成り立っています:まず、SEERを用いてデータを取得しました。統計、およびデータ分析と視覚化は、Rを使用して実行されました。 この図の拡大版を表示するには、ここをクリックしてください。
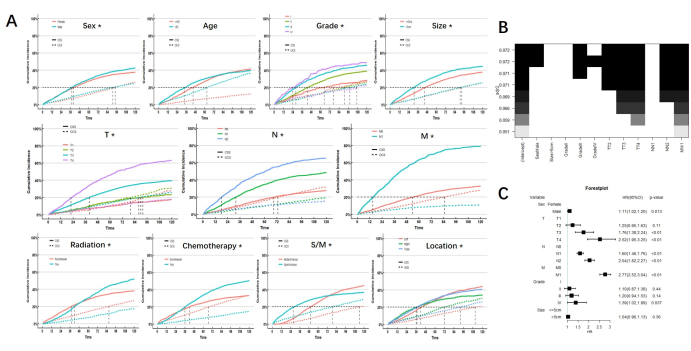
図2:分析結果 (A)サブグループのCIF。*はP<0.05を示します。(B) ベストサブセット回帰。適合度が最も高い場合、性別、グレード、およびTNMステージを含めることを検討しました(上部に黒いブロックで表示)。(C)多変量解析では、性別、グレード、およびTNMステージが独立した危険因子であることも示されました。 この図の拡大版を表示するには、ここをクリックしてください。
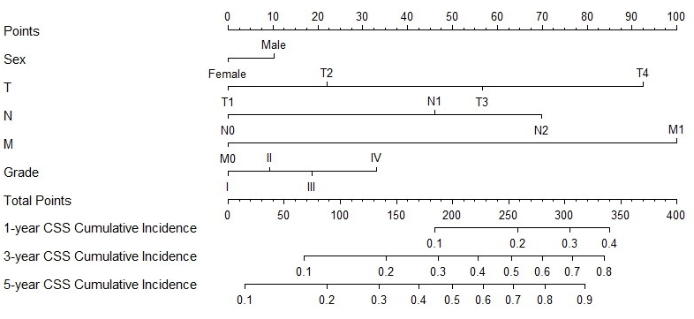
図3:MPCC患者におけるCSSのノモグラム。 患者の合計スコアは、各要因に対応するスコアを加算することで計算できます。合計スコアに基づいて、1年、3年、および5年でのがん特異的な死亡の確率を予測することができます この図の拡大版を表示するには、ここをクリックしてください。
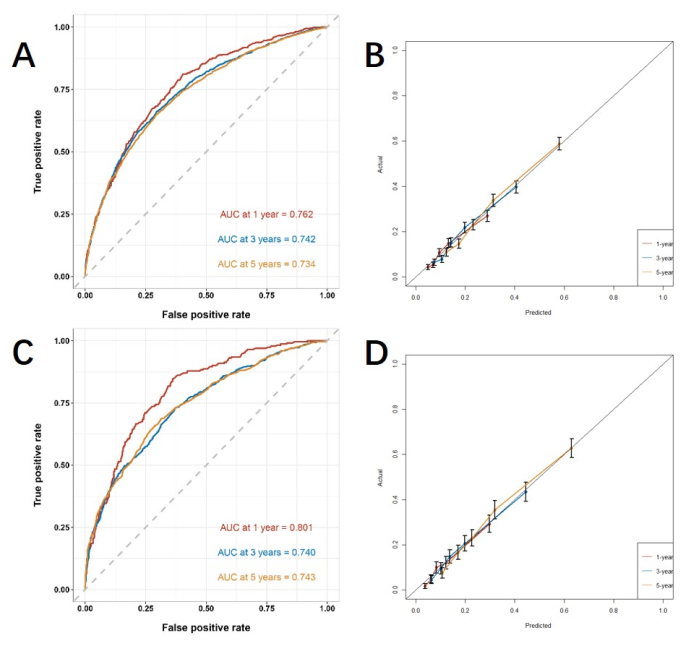
図4:ROC曲線と検量線 (A, B) トレーニングコホート (n=6255) と (C, D) 検証コホート (n=2676) の曲線。AUC 値が 1 に近いほど、モデルの分類パフォーマンスが向上します。エラーバーは、実際のイベントが発生する確率の95%信頼区間を示しています。 この図の拡大版を表示するには、ここをクリックしてください。
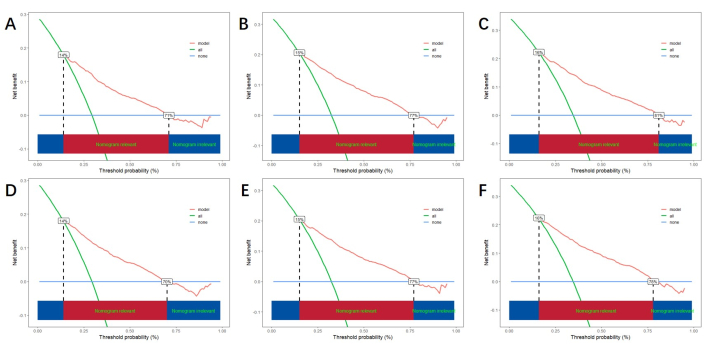
図5:1年、3年、5年のDCA( トレーニングコホートの(A、B、C)DCAおよび(D、E、F)検証コホート。緑の線はすべてのポジティブの正味の利益を表し、青い線は正の何もない正の利益を表し、赤の線はモデルの正味の利益を示します。下の赤い領域は、モデルのベネフィットがすべてのポジティブとなしのベネフィットを上回っていることを表しており、モデルの実際のベネフィット範囲を示しています。 この図の拡大版を表示するには、ここをクリックしてください。
表1:患者の臨床病理学的およびベースライン特性。この表をダウンロードするには、ここをクリックしてください。
ディスカッション
一般的な消化器系腫瘍として、孤立性結腸直腸癌の予後に影響を与える因子が以前の研究で研究され、確認されています24。しかし、MPCC患者の予後因子に関する研究は限られています。この研究には、2004年から2015年の間にSEERデータベースから手術を受けた8,931人のMPCC患者が含まれていました。競合するリスクモデルを使用して、CSSに影響を与えるリスク要因を調査し、予測モデルを構築しました。この研究では、死亡した患者の39.1%ががん以外の原因で死亡しており、CSSのリスク因子を分析するために競合するリスクモデルを使用する必要性を強調しています。
以前の研究では、高齢がMPCC患者のOSに影響を与える危険因子であることがわかっています12。しかし、この研究では、年齢はCSSの危険因子ではないことがわかりました。高齢の患者は、ベースラインの健康状態が不良で併存疾患が多いことが多く、若年患者と比較してOSが短くなる可能性があります。また、高齢者は心血管イベントや重篤な感染症などの他の原因で死亡する可能性が高く、若年者よりもこれらの要因の影響を受けやすくなっています。本研究では、他の原因による死亡とがん自体による死亡との間の競合関係に着目した。競合するリスクモデルを使用し、他の理由による死亡の干渉を除外することにより、これらの条件下では、高齢はもはやMPCCのCSSのリスク因子ではないことがわかりました。このことは、異なる年齢層のMPCC患者が同様の腫瘍量に直面する可能性があることを示している。全体として、この疾患の若年患者と高齢患者は同様の CSS を持っており、個別化された治療戦略を開発するための貴重なガイダンスを提供します。
また、本研究では、MPCCの発生率と予後が性別に関連する傾向を示し、男性患者の割合が高く、CSSが不良であることもわかりました。これは、孤立性結腸直腸癌で観察される状況と一致しています。以前の研究では、孤立性結腸直腸癌は男性に多く見られ、男性患者は女性患者よりも予後が悪いことが示されています。これは、結腸直腸癌の発生と進行に対するエストロゲンの影響によるものかもしれません25。さらに、他の研究では、男性患者の腸内細菌叢と腸内代謝産物が、結腸直腸癌患者で観察される性差の理由の1つである可能性があることが示唆されています26。ドイツでの研究では、2型糖尿病が女性の結腸直腸癌に大きな影響を与えることがわかりました27。さらに、ビタミンDは女性の結腸直腸腫瘍に対する保護効果がありますが、男性では同様の所見は見つかりませんでした28。
競合する単変量解析により、腫瘍サイズが大きいMPCC患者ほどCSSが不良であることがわかった。以前の研究では、腫瘍の大きさはしばしば腫瘍の攻撃性の指標と考えられてきました29,30,31。しかしながら、競合する多変量解析の結果は、腫瘍の大きさを独立した危険因子として支持していなかった。これは、結腸直腸のような中空器官では、腫瘍のサイズが腫瘍の攻撃性を反映する能力が限られている可能性があることを示唆しています。この現象は、腫瘍生物学の複雑さや、腫瘍の成長パターンや臓器の広がりの違いなど、さまざまな要因から生じている可能性があります。
腫瘍の悪性度が低いということは、腫瘍細胞の侵襲性および移動能力が強いことを示しており、これはこの研究の結論と一致しています:腫瘍の悪性度が悪いほど、MPCC患者のCSSが不良であることを示唆しています。TNM病期は、患者の治療を指導し、予後を予測するために最も一般的に使用される臨床方法です32,33。この研究では、腫瘍の浸潤が深いほど、転移性リンパ節の数が多くなり、臓器転移の存在が、患者のがん特異的生存率が低下することがわかりました。これは、臨床上のコンセンサスと完全に一致しています。
結腸直腸癌では、腫瘍の位置によって予後が異なります。多くの研究により、右結腸は左結腸直腸と比較して予後が不良であることがわかっています34,35,36,37。しかし、いくつかの研究は、切除可能な結腸直腸癌では、腫瘍の位置の側面が長期予後に影響を与えないことを示唆している38。この研究では、右結腸に位置するMPCCのCSSは短期的には不良でしたが、長期的には予後が良好でした。同様に、放射線療法と化学療法を受けたMPCC患者は、短期のCSSは良好であったが、長期のCSSは不良であった。SCRCはMCRCと比較して短期CSSが劣っていましたが、長期CSSは優れていました。これらの二重の影響が予後に及ぼす影響の理由はまだ不明である。
この研究にはいくつかの制限があります。SEERデータベースに固有の制約により、癌胎児性抗原(CEA)レベルやマイクロサテライトの状態など、一部の既知の予後指標を取得または分析することはできませんでした。さらに、炎症性腸疾患、遺伝性非ポリポーシス結腸直腸癌、および家族性大腸腺腫症の患者で、MPCC39のリスクが高い患者を除外することはできませんでした。また、免疫療法は大腸がん治療においてますます重要な側面となっていますが、これに関するデータは得られていませんでした。私たちの分析の遡及的な性質と単一のデータセットへの依存は、固有のバイアスをもたらす可能性があります。
SEERデータベースに基づく研究では、男性の性別、腫瘍の悪性度、および進行したTNMの病期が、手術後のMPCC患者のCSSの低下と関連していることがわかりました。診断および治療中に、これらの危険因子を持つ患者に細心の注意を払うことが重要です。さらに、本研究では、MPCC患者のCSSを予測するためのノモグラムを開発したことで、予後を正確に予測し、臨床治療の意思決定に貢献できるようになった。
開示事項
著者は何も開示していません。
謝辞
何一つ。
資料
| Name | Company | Catalog Number | Comments |
| forestplot package | Comprehensive R Archive Network (CRAN) | forest plot 3.1.3 | A forest plot that allows for multiple confidence intervals per row, custom fonts for each text element, custom confidence intervals, text mixed with expressions, and more. |
| ggprism package | Comprehensive R Archive Network (CRAN) | ggprism 1.0.5 | The ggprism package provides various themes, palettes, and other useful functions to customise ggplots and give them the ‘GraphPad Prism’ look. |
| ggscidca package | Comprehensive R Archive Network (CRAN) | ggscidca package | The 'ggscidca' package adds coloured bars of discriminant relevance to the traditional decision curve. Improved practicality and aesthetics. |
| ggsurvfit package | Comprehensive R Archive Network (CRAN) | ggsurvfit 1.1.0 | The ggsurvfit package eases the creation of time-to-event (aka survival) summary figures with ggplot2. The concise and modular code creates images that are ready for publication or sharing. |
| gtsummary package | Comprehensive R Archive Network (CRAN) | gtsummary 2.0.0 | The gtsummary package provides an elegant and flexible way to create publication-ready analytical and summary tables using the R programming language. The {gtsummary} package summarizes data sets, regression models, and more, using sensible defaults with highly customizable capabilities. |
| QHScrnomo package | Comprehensive R Archive Network (CRAN) | QHScrnomo 3.0.1 | The goal of QHScrnomo is to provide functionality to construct nomograms in the context of time-to-event (survival) analysis in the presence of competing risks. It also contains functions to build, validate, and summarize these models. |
| R Software | R Core Team | R 4.3.3 | Free software environment for statistical computing and graphics |
| riskRegression package | Comprehensive R Archive Network (CRAN) | riskRegression 1.3.7 | Risk Regression Models and Prediction Scores for Survival Analysis with Competing Risks |
| rms package | Comprehensive R Archive Network (CRAN) | rms 6.8-1 | rms does regression modeling, testing, estimation, validation, graphics, prediction, and typesetting by storing enhanced model design attributes in the fit. |
| RStudio | RStudio, Public Benefit Corporation(PBC) | Rstudio 2023.12.1+402 | Integrated Development Environment (IDE) used for running R scripts, data analysis, and model development. Provides a user-friendly interface for R programming with advanced features like script editing, debugging, and version control. |
| SEERstat | National Cancer Institute (NCI) | SEERstat 8.4.3 | Software for statistical analysis of SEER and other cancer-related databases |
| tidycmprsk package | Comprehensive R Archive Network (CRAN) | tidycmprsk 1.0.0 | The tidycmprsk package provides an intuitive interface for working with the competing risk endpoints |
参考文献
- Bray, F., et al. Global cancer statistics 2022: GLOBOCAN estimates of incidence and mortality worldwide for 36 cancers in 185 countries. CA Cancer J Clin. 74 (3), 229-263 (2024).
- Derwinger, K., Gustavsson, B. A study of aspects on gender and prognosis in synchronous colorectal cancer. Clin Med Insights Oncol. 5, 259-264 (2011).
- Chin, C. C., Kuo, Y. H., Chiang, J. M. Synchronous colorectal carcinoma: predisposing factors and characteristics. Colorectal Dis. 21 (4), 432-440 (2019).
- Nikoloudis, N., et al. Synchronous colorectal cancer. Tech Coloproctol. 8 (Suppl 1), s177-s179 (2004).
- Huang, C. S., et al. Synchronous and metachronous colorectal cancers: Distinct disease entities or different disease courses. Hepatogastroenterology. 62 (140), 838-842 (2015).
- He, W., et al. Prognosis of synchronous colorectal carcinoma compared to solitary colorectal carcinoma: a matched pair analysis. Eur J Gastroenterol Hepatol. 31 (12), 1489-1495 (2019).
- Ranstam, J., Cook, J. A., Collins, G. S. Clinical prediction models. Br J Surg. 103 (13), 1886 (2016).
- Smith, T., et al. Comparison of prognostic models to predict the occurrence of colorectal cancer in asymptomatic individuals: a systematic literature review and external validation in the EPIC and UK Biobank prospective cohort studies. Gut. 68 (4), 672-683 (2019).
- Iasonos, A., Schrag, D., Raj, G. V., Panageas, K. S. How to build and interpret a nomogram for cancer prognosis. J Clin Oncol. 26 (8), 1364-1370 (2008).
- Conroy, T., et al. Five-year outcomes of FOLFIRINOX vs Gemcitabine as adjuvant therapy for pancreatic cancer: A randomized clinical trial. JAMA Oncol. 8 (11), 1571-1578 (2022).
- Zhong, M., et al. Impact of lung metastasis versus metastasis of bone, brain, or liver on overall survival and thyroid cancer-specific survival of thyroid cancer patients: A population-based study. Cancers. 14 (13), 3133 (2022).
- Xu, Y., Wang, X., Huang, Y., Ye, D., Chi, P. A LASSO-based survival prediction model for patients with synchronous colorectal carcinomas based on SEER. Transl Cancer Res. 11 (8), 2795-2809 (2022).
- Zhang, X., Zhao, L., Hu, Y., Deng, K., Ren, W. A novel risk prediction nomogram for early death in patients with resected synchronous multiple primary colorectal cancer based on the SEER database. Int J Colorectal Dis. 38 (1), 130 (2023).
- Zhang, X., et al. Developing prognostic nomograms to predict overall survival and cancer-specific survival in synchronous multiple primary colorectal cancer based on the SEER database. J Cancer Res Clin Oncol. 149 (15), 14057-14070 (2023).
- Austin, P. C., Lee, D. S., Fine, J. P. Introduction to the analysis of survival data in the presence of competing risks. Circulation. 133 (6), 601-609 (2016).
- Lau, B., Cole, S. R., Gange, S. J. Competing risk regression models for epidemiologic data. Am J Epidemiol. 170 (2), 244-256 (2009).
- Nolan, E. K., Chen, H. Y. A comparison of the Cox model to the Fine-Gray model for survival analyses of re-fracture rates. Arch Osteoporos. 15 (1), 86 (2020).
- Park, S. Y. Nomogram: An analogue tool to deliver digital knowledge. J Thorac Cardiovasc Surg. 155 (4), 1793 (2018).
- Balachandran, V. P., Gonen, M., Smith, J. J., DeMatteo, R. P. Nomograms in oncology: more than meets the eye. Lancet Oncol. 16 (4), e173-e180 (2015).
- Liang, W., et al. Development and validation of a nomogram for predicting survival in patients with resected non-small-cell lung cancer. J Clin Oncol. 33 (8), 861-869 (2015).
- Chen, D., et al. Predicting postoperative peritoneal metastasis in gastric cancer with serosal invasion using a collagen nomogram. Nat Commun. 12 (1), 179 (2021).
- Niu, X., et al. A prognostic nomogram for patients with newly diagnosed adult thalamic glioma in a surgical cohort. Neuro Oncol. 23 (2), 337-338 (2021).
- Luo, S., et al. Comparison of left- and right-sided colorectal cancer to explore prognostic signatures related to pyroptosis. Heliyon. 10 (7), e28091 (2024).
- Keum, N., Giovannucci, E. Global burden of colorectal cancer: emerging trends, risk factors and prevention strategies. Nat Rev Gastroenterol Hepatol. 16 (12), 713-732 (2019).
- Foster, P. A. Oestrogen and colorectal cancer: mechanisms and controversies. Int J Colorectal Dis. 28 (6), 737-749 (2013).
- Wang, L., et al. Male-biased gut microbiome and metabolites aggravate colorectal cancer development. Adv Sci. 10 (25), e2206238 (2023).
- Krämer, H. U., et al. Type 2 diabetes mellitus and gender-specific risk for colorectal neoplasia. Eur J Epidemiol. 27 (5), 341-347 (2012).
- Aigner, E., et al. Gender- and site-specific differences of colorectal neoplasia relate to vitamin D. Aliment Pharmacol Ther. 40 (11-12), 1341-1348 (2014).
- Dai, W., et al. Does tumor size have its prognostic role in colorectal cancer? Re-evaluating its value in colorectal adenocarcinoma with different macroscopic growth pattern. Int J Surg. 45, 105-112 (2017).
- Rössler, O., et al. Tumor size, tumor location, and antitumor inflammatory response are associated with lymph node size in colorectal cancer patients. Mod Pathol. 30 (6), 897-904 (2017).
- Zhang, Q., et al. Prognostic impact of tumor size on patients with metastatic colorectal cancer: a large SEER-based retrospective cohort study. Updates Surg. 75 (5), 1135-1147 (2023).
- Greene, F. L. TNM: our language of cancer. CA Cancer J Clin. 54 (3), 129-130 (2004).
- Ueno, H., et al. Optimal colorectal cancer staging criteria in TNM classification. J Clin Oncol. 30 (13), 1519-1526 (2012).
- Yahagi, M., Okabayashi, K., Hasegawa, H., Tsuruta, M., Kitagawa, Y. The worse prognosis of right-sided compared with left-sided colon cancers: a systematic review and meta-analysis. J Gastrointest Surg. 20 (3), 648-655 (2016).
- Holch, J. W., Ricard, I., Stintzing, S., Modest, D. P., Heinemann, V. The relevance of primary tumour location in patients with metastatic colorectal cancer: A meta-analysis of first-line clinical trials. Eur J Cancer. 70, 87-98 (2017).
- Petrelli, F., et al. Prognostic survival associated with left-sided vs right-sided colon cancer: A systematic review and meta-analysis. JAMA Oncol. 3 (2), 211-219 (2017).
- Zheng, C., Jiang, F., Lin, H., Li, S. Clinical characteristics and prognosis of different primary tumor location in colorectal cancer: a population-based cohort study. Clin Transl Oncol. 21 (11), 1524-1531 (2019).
- Karim, S., Brennan, K., Nanji, S., Berry, S. R., Booth, C. M. Association between prognosis and tumor laterality in early-stage colon cancer. JAMA Oncol. 3 (10), 1386-1392 (2017).
- Lindberg, L. J., et al. Risk of multiple colorectal cancer development depends on age and subgroup in individuals with hereditary predisposition. Fam Cancer. 18 (2), 183-191 (2019).
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請さらに記事を探す
This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved